Table 2.
| Modified residue parent peptide | P1 | E2 | P3 | T4 | (A5)P6 | P7[c] | E8 | E9 | |||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 8 | 9 | 10 | 11 | 12 | 13 | 14 | 15 | 16 | 17 | 18 | 19 | ||
| – | Aldehyded (no aldehyde) | 37 | 56 | 22 | 68 | 214 | 64 | n.b. e | n.b. | 43 | 55 | 47 | 79 |
| a |
|
45 | 23 | 24 | 24 | 88 | 246 | n.b. | n.b. | 41 | 42 | 28 | 132 |
| b |
|
53 | 62 | 15 | 92 | 188 | n.f.f | n.b. | n.b. | 27 | 75 | 28 | 56 |
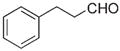
| c |
|
50 | 42 | 19 | 34 | 24 | n.f. | n.b. | n.b. | 26 | 83 | 18 | 77 |
| d |
|
45 | 44 | 20 | 42 | 16 | 400 | n.b. | n.b. | 30 | 48 | 25 | 78 |
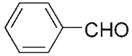
| e |
|
38 | 19 | 29 | 33 | 16 | n.f. | n.b. | n.b. | 26 | 64 | 23 | 88 |
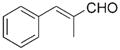
| f |
|
54 | 14 | 39 | 51 | 15 | n.f. | n.b. | n.b. | 13 | 58 | 26 | 107 |
| g |
|
35 | 105 | 17 | 19 | 58 | n.f. | n.b. | n.b. | 17 | 42 | 13 | 124 |
| h |
|
41 | 74 | 16 | 17 | 156 | n.f. | n.b. | n.b. | 21 | 91 | 28 | 82 |
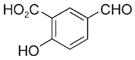
| i |
|
18 | 23 | 11 | 27 | 71 | n.f. | n.b. | n.b. | 16 | 93 | 18 | 89 |
| j |
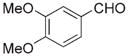
|
42 | 38 | 21 | 3.3 | 121 | n.f. | n.b. | n.b. | 16 | 114 | 20 | 54 |
| k |
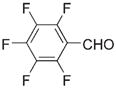
|
42 | 20 | 27 | 46 | 177 | n.f. | n.b. | n.b. | 32 | 139 | 21 | 33 |
| l |
|
54 | 23 | 26 | 9.5 | 87 | n.f. | n.b. | n.b. | 30 | 54.5 | 35 | 55 |
Numerical data represent Kd values [μm] obtained as indicated in ref. [13].
Wild-type peptide FITC-Ava-PEPTAPPEE-amide Kd ~ 50–60 μm.
Data previously reported in ref. [14].
Aldehyde used to form the indicated oxime-containing peptide.
n.b.= no binding.
n.f. = no fit, suggesting weak binding (Kd > 500 μm).
