James et al. 10.1073/pnas.0609174104. |

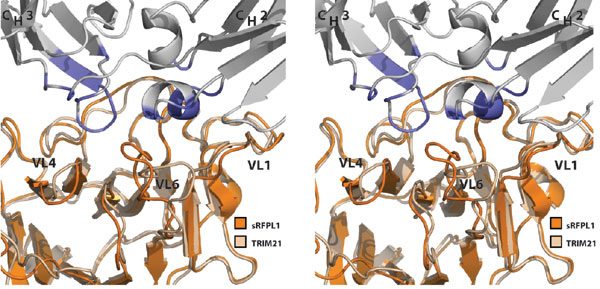
Fig. 6. Stereo representation of TRIM21:Fc complex superposed with sRFPL1 (1). Fc is in gray with interaction surface in blue, sRFPL1 is in orange,and TRIM21 is in wheat.
1. Grutter C, Briand C, Capitani G, Mittl PR, Papin S, Tschopp J, Grutter MG (2006) FEBS Lett 580:99-106.
Table 1. Data collection and refinement statistics
Data collection | |
Space group | P61 |
Cell dimensions | |
a, b, c, Å | 112.49,112.49,194.60 |
a, b, g, ° | 90.00,90.00,120.00 |
Resolution, Å | 97.6 - 2.35 |
R sym | 0.085(0.361) |
I / sI | 11.9(2.4) |
Completeness, % | 86(83.4) |
Redundancy | 3.6(2.7) |
Refinement | |
Resolution, Å | 2.35 |
No. reflections | 57,896 |
R work / Rfree | 0.201/0.251 |
No. atoms | |
Protein | 6,299 |
Ligand/ion | 192 |
Water | 178 |
B factors | |
Protein | 58.11 |
Ligand/ion | 61.02 |
Water | 34.53 |
rmsd | |
Bond lengths, Å | 0.005 |
Bond angles, ° | 1.01 |
Highest resolution shell (2.5-2.35Å) is shown in parentheses.
Table 2. Kinetics and thermodynamics of TRIM21:Fc interaction
Protein | k on,´106 M-1·s-1 | k off , s-1 | K d, kinetic, M† | K d, equilibrium, M | DDGbinding, kcal/mol |
WT | 3.5 (±0.3) | 0.46 (±0.01) | 1.3 (±0.15) x10-7 | 1.2 (±0.2) x10-7 ‡ | |
W299A | 3.6 (±0.2) | 1.41 (±0.01) | 4.0 (±0.26) x10-7 | 4.0 (±0.1) x10-7 § | 0.7 (±0.1) |
W299F | 4.2 (±0.3) | 0.47 (±0.01) | 1.1 (±0.11) x10-7 | N.D. | -0.1 (±0.07) |
Y328A | 3.6 (±0.5) | 2.45 (±0.01) | 6.8 (±1.11) x10-7 | N.D. | 1.0 (±0.15) |
Y328F | 3.3 (±0.3) | 0.03 (±0.001) | 9.1 (±0.13) x10-9 | N.D. | -1.6 (±0.07) |
D355A | N.D. | N.D. | N.D. | >5 x10-4 § | >5 |
H368A | 4.3 (±0.3) | 0.13 (±0.02) | 3.0 (±0.7) x10-8 | N.D. | -0.9 (±0.2) |
L370A | 3.7 (±0.2) | 2.98 (±0.01) | 8.1 (±0.5) x10-7 | N.D. | 1.1 (±0.1) |
W381A | N.D. | N.D. | N.D. | 1.1 (±0.2) x10-4 § | 4.0 (±0.1) |
W383A | N.D. | N.D. | N.D. | 1.1 (±0.2) x10-4 § | 4.0 (±0.1) |
W383F | 3.4 (±0.2) | 0.93 (±0.01) | 2.7 (±0.2) x10-7 | N.D. | 0.4 (±0.11) |
Y391A | 4.5 (±0.1) | 0.53 (±0.01) | 1.2 (±0.04) x10-7 | N.D. | -0.05 (±0.08) |
F450A | N.D. | N.D. | N.D. | >5 x10-4 § | >5 |
D452A | 7.0 (±0.4) | 2.6 (±0.01) | 3.7 (±0.15) x10-7 | 8 (±0.1) x10-7 | 0.6 (±0.08) ¶ 1.1 (±0.2) |
DDG = RTln[Kd(mutant)/Kd(wild-type)]. N.D., not determined.
†
The kinetically derived dissociation constant, Kd, kinetic = koff/kon.‡
Measured by isothermal titration calorimetry.§
Measured by fluorescence anisotropy titration.¶
Two values are calculated based on the measured Kd, kinetic and Kd, equilibrium, respectively.SI Data Set
Contacts between TRIM21 and Fc
As calculated by the CCP4 program 'Contact'(1).
Fc ATOMS TRIM21 ATOMS DISTANCE
/1/A/ 253(ILE). / CG2[ C]: /1/B/ 16(ASN). / OD1[ O]: 4.88
/1/B/ 16(ASN). / ND2[ N]: 4.66
/1/A/ 253(ILE). / CG1[ C]: /1/B/ 18(TRP). / CG [ C]: 4.91
/1/A/ 253(ILE). / O [ O]: /1/B/ 18(TRP). / CD1[ C]: 4.57
/1/A/ 253(ILE). / CB [ C]: /1/B/ 18(TRP). / CD1[ C]: 4.88
/1/A/ 253(ILE). / CG1[ C]: /1/B/ 18(TRP). / CD1[ C]: 4.35
/1/A/ 253(ILE). / CG2[ C]: /1/B/ 18(TRP). / CD1[ C]: 4.26
/1/A/ 253(ILE). / O [ O]: /1/B/ 18(TRP). / NE1[ N]: 3.57
/1/A/ 253(ILE). / CB [ C]: /1/B/ 18(TRP). / NE1[ N]: 4.67
/1/A/ 253(ILE). / CG1[ C]: /1/B/ 18(TRP). / NE1[ N]: 4.20
/1/A/ 253(ILE). / CG2[ C]: /1/B/ 18(TRP). / NE1[ N]: 4.33
/1/A/ 253(ILE). / C [ C]: /1/B/ 18(TRP). / NE1[ N]: 4.50
/1/A/ 253(ILE). / O [ O]: /1/B/ 18(TRP). / CE2[ C]: 4.32
/1/A/ 253(ILE). / CG1[ C]: /1/B/ 18(TRP). / CE2[ C]: 4.69
/1/A/ 309(LEU). / CD1[ C]: /1/B/ 18(TRP). / CZ3[ C]: 4.89
/1/A/ 309(LEU). / CD2[ C]: /1/B/ 18(TRP). / CZ3[ C]: 4.23
/1/B/ 18(TRP). / CH2[ C]: 3.89
/1/A/ 253(ILE). / O [ O]: /1/B/ 18(TRP). / CZ2[ C]: 4.46
/1/A/ 310(HIS). / ND1[ N]: /1/B/ 18(TRP). / CZ2[ C]: 4.79
/1/A/ 309(LEU). / CD1[ C]: /1/B/ 32(ASP). / CA [ C]: 4.24
/1/B/ 32(ASP). / CB [ C]: 4.00
/1/B/ 32(ASP). / C [ C]: 4.23
/1/B/ 32(ASP). / O [ O]: 3.42
/1/A/ 309(LEU). / CG [ C]: /1/B/ 32(ASP). / O [ O]: 4.33
/1/A/ 309(LEU). / CD2[ C]: /1/B/ 32(ASP). / O [ O]: 4.11
/1/A/ 253(ILE). / CG2[ C]: /1/B/ 45(ASP). / C [ C]: 4.91
/1/B/ 45(ASP). / O [ O]: 4.22
/1/A/ 254(SER). / N [ N]: /1/B/ 45(ASP). / O [ O]: 4.67
/1/A/ 254(SER). / CA [ C]: /1/B/ 45(ASP). / O [ O]: 4.83
/1/A/ 254(SER). / CB [ C]: /1/B/ 45(ASP). / O [ O]: 4.82
/1/A/ 253(ILE). / CG2[ C]: /1/B/ 46(SER). / N [ N]: 4.85
/1/B/ 46(SER). / CA [ C]: 4.00
/1/A/ 254(SER). / CB [ C]: /1/B/ 46(SER). / OG [ O]: 4.97
/1/A/ 253(ILE). / CB [ C]: /1/B/ 46(SER). / C [ C]: 4.67
/1/A/ 253(ILE). / CG2[ C]: /1/B/ 46(SER). / C [ C]: 3.63
/1/A/ 253(ILE). / N [ N]: /1/B/ 46(SER). / O [ O]: 4.96
/1/A/ 253(ILE). / CA [ C]: /1/B/ 46(SER). / O [ O]: 4.96
/1/A/ 253(ILE). / CB [ C]: /1/B/ 46(SER). / O [ O]: 3.99
/1/A/ 253(ILE). / CG2[ C]: /1/B/ 46(SER). / O [ O]: 3.33
/1/B/ 47(TYR). / N [ N]: 4.32
/1/B/ 47(TYR). / CA [ C]: 4.70
/1/A/ 434(ASN). / O [ O]: /1/B/ 47(TYR). / CZ [ C]: 4.89
/1/A/ 436(TYR). / CB [ C]: /1/B/ 47(TYR). / CZ [ C]: 4.56
/1/A/ 436(TYR). / CG [ C]: /1/B/ 47(TYR). / CZ [ C]: 4.62
/1/A/ 436(TYR). / CD1[ C]: /1/B/ 47(TYR). / CZ [ C]: 4.49
/1/A/ 436(TYR). / CB [ C]: /1/B/ 47(TYR). / OH [ O]: 3.66
/1/A/ 436(TYR). / CG [ C]: /1/B/ 47(TYR). / OH [ O]: 3.41
/1/A/ 436(TYR). / CD2[ C]: /1/B/ 47(TYR). / OH [ O]: 4.20
/1/A/ 436(TYR). / CD1[ C]: /1/B/ 47(TYR). / OH [ O]: 3.18
/1/A/ 436(TYR). / CE1[ C]: /1/B/ 47(TYR). / OH [ O]: 3.78
/1/A/ 436(TYR). / CZ [ C]: /1/B/ 47(TYR). / OH [ O]: 4.49
/1/A/ 436(TYR). / CE2[ C]: /1/B/ 47(TYR). / OH [ O]: 4.69
/1/A/ 434(ASN). / CG [ C]: /1/B/ 47(TYR). / CE2[ C]: 4.63
/1/A/ 434(ASN). / OD1[ O]: /1/B/ 47(TYR). / CE2[ C]: 4.77
/1/A/ 434(ASN). / CB [ C]: /1/B/ 47(TYR). / CE2[ C]: 4.07
/1/A/ 434(ASN). / C [ C]: /1/B/ 47(TYR). / CE2[ C]: 4.78
/1/A/ 434(ASN). / O [ O]: /1/B/ 47(TYR). / CE2[ C]: 3.73
/1/A/ 436(TYR). / CB [ C]: /1/B/ 47(TYR). / CE2[ C]: 4.38
/1/A/ 436(TYR). / CG [ C]: /1/B/ 47(TYR). / CE2[ C]: 4.88
/1/A/ 434(ASN). / CG [ C]: /1/B/ 47(TYR). / CD2[ C]: 4.42
/1/A/ 434(ASN). / OD1[ O]: /1/B/ 47(TYR). / CD2[ C]: 4.61
/1/A/ 434(ASN). / ND2[ N]: /1/B/ 47(TYR). / CD2[ C]: 4.98
/1/A/ 434(ASN). / CB [ C]: /1/B/ 47(TYR). / CD2[ C]: 4.20
/1/A/ 434(ASN). / O [ O]: /1/B/ 47(TYR). / CD2[ C]: 4.14
/1/A/ 434(ASN). / CG [ C]: /1/B/ 49(MET). / CE [ C]: 4.79
/1/A/ 434(ASN). / OD1[ O]: /1/B/ 49(MET). / CE [ C]: 4.02
/1/A/ 433(HIS). / CE1[ C]: /1/B/ 74(ASP). / CB [ C]: 4.96
/1/A/ 433(HIS). / ND1[ N]: /1/B/ 74(ASP). / CG [ C]: 4.86
/1/A/ 433(HIS). / CE1[ C]: /1/B/ 74(ASP). / CG [ C]: 3.63
/1/A/ 433(HIS). / NE2[ N]: /1/B/ 74(ASP). / CG [ C]: 4.00
/1/A/ 434(ASN). / CG [ C]: /1/B/ 74(ASP). / CG [ C]: 4.83
/1/A/ 434(ASN). / OD1[ O]: /1/B/ 74(ASP). / CG [ C]: 4.09
/1/A/ 434(ASN). / ND2[ N]: /1/B/ 74(ASP). / CG [ C]: 4.72
/1/A/ 433(HIS). / CG [ C]: /1/B/ 74(ASP). / OD1[ O]: 4.94
/1/A/ 433(HIS). / ND1[ N]: /1/B/ 74(ASP). / OD1[ O]: 3.86
/1/A/ 433(HIS). / CE1[ C]: /1/B/ 74(ASP). / OD1[ O]: 2.71
/1/A/ 433(HIS). / NE2[ N]: /1/B/ 74(ASP). / OD1[ O]: 3.39
/1/A/ 434(ASN). / CG [ C]: /1/B/ 74(ASP). / OD1[ O]: 4.60
/1/A/ 434(ASN). / OD1[ O]: /1/B/ 74(ASP). / OD1[ O]: 4.06
/1/A/ 434(ASN). / ND2[ N]: /1/B/ 74(ASP). / OD1[ O]: 4.40
/1/A/ 433(HIS). / CD2[ C]: /1/B/ 74(ASP). / OD1[ O]: 4.70
/1/A/ 433(HIS). / CE1[ C]: /1/B/ 74(ASP). / OD2[ O]: 3.86
/1/A/ 433(HIS). / NE2[ N]: /1/B/ 74(ASP). / OD2[ O]: 4.12
/1/A/ 434(ASN). / CG [ C]: /1/B/ 74(ASP). / OD2[ O]: 4.06
/1/A/ 434(ASN). / OD1[ O]: /1/B/ 74(ASP). / OD2[ O]: 3.23
/1/A/ 434(ASN). / ND2[ N]: /1/B/ 74(ASP). / OD2[ O]: 4.07
/1/A/ 436(TYR). / CD2[ C]: /1/B/ 87(HIS). / CA [ C]: 4.99
/1/A/ 436(TYR). / CE1[ C]: /1/B/ 87(HIS). / CA [ C]: 4.66
/1/A/ 436(TYR). / CZ [ C]: /1/B/ 87(HIS). / CA [ C]: 4.44
/1/A/ 436(TYR). / OH [ O]: /1/B/ 87(HIS). / CA [ C]: 4.69
/1/A/ 436(TYR). / CE2[ C]: /1/B/ 87(HIS). / CA [ C]: 4.62
/1/A/ 436(TYR). / CG [ C]: /1/B/ 87(HIS). / CB [ C]: 4.26
/1/A/ 436(TYR). / CD2[ C]: /1/B/ 87(HIS). / CB [ C]: 3.76
/1/A/ 436(TYR). / CD1[ C]: /1/B/ 87(HIS). / CB [ C]: 4.40
/1/A/ 436(TYR). / CE1[ C]: /1/B/ 87(HIS). / CB [ C]: 4.08
/1/A/ 436(TYR). / CZ [ C]: /1/B/ 87(HIS). / CB [ C]: 3.57
/1/A/ 436(TYR). / OH [ O]: /1/B/ 87(HIS). / CB [ C]: 4.01
/1/A/ 436(TYR). / CE2[ C]: /1/B/ 87(HIS). / CB [ C]: 3.40
/1/A/ 436(TYR). / CB [ C]: /1/B/ 87(HIS). / CG [ C]: 4.77
/1/A/ 436(TYR). / CG [ C]: /1/B/ 87(HIS). / CG [ C]: 4.18
/1/A/ 436(TYR). / CD2[ C]: /1/B/ 87(HIS). / CG [ C]: 3.73
/1/A/ 436(TYR). / CD1[ C]: /1/B/ 87(HIS). / CG [ C]: 4.70
/1/A/ 436(TYR). / CE1[ C]: /1/B/ 87(HIS). / CG [ C]: 4.80
/1/A/ 436(TYR). / CZ [ C]: /1/B/ 87(HIS). / CG [ C]: 4.40
/1/A/ 436(TYR). / CE2[ C]: /1/B/ 87(HIS). / CG [ C]: 3.85
/1/A/ 436(TYR). / CB [ C]: /1/B/ 87(HIS). / ND1[ N]: 3.76
/1/A/ 436(TYR). / CG [ C]: /1/B/ 87(HIS). / ND1[ N]: 3.53
/1/A/ 436(TYR). / CD2[ C]: /1/B/ 87(HIS). / ND1[ N]: 3.47
/1/A/ 428(MET). / SD [ S]: /1/B/ 87(HIS). / ND1[ N]: 4.98
/1/A/ 436(TYR). / CD1[ C]: /1/B/ 87(HIS). / ND1[ N]: 4.16
/1/A/ 436(TYR). / CE1[ C]: /1/B/ 87(HIS). / ND1[ N]: 4.64
/1/A/ 436(TYR). / CZ [ C]: /1/B/ 87(HIS). / ND1[ N]: 4.58
/1/A/ 436(TYR). / CE2[ C]: /1/B/ 87(HIS). / ND1[ N]: 4.04
/1/A/ 252(MET). / CG [ C]: /1/B/ 87(HIS). / CE1[ C]: 4.49
/1/A/ 436(TYR). / CB [ C]: /1/B/ 87(HIS). / CE1[ C]: 4.20
/1/A/ 436(TYR). / CG [ C]: /1/B/ 87(HIS). / CE1[ C]: 4.35
/1/A/ 436(TYR). / CD2[ C]: /1/B/ 87(HIS). / CE1[ C]: 4.26
/1/A/ 428(MET). / SD [ S]: /1/B/ 87(HIS). / CE1[ C]: 4.51
/1/A/ 252(MET). / CB [ C]: /1/B/ 87(HIS). / NE2[ N]: 4.95
/1/A/ 252(MET). / CG [ C]: /1/B/ 87(HIS). / NE2[ N]: 3.55
/1/A/ 252(MET). / SD [ S]: /1/B/ 87(HIS). / NE2[ N]: 3.93
/1/A/ 436(TYR). / CD2[ C]: /1/B/ 87(HIS). / NE2[ N]: 4.89
/1/A/ 428(MET). / SD [ S]: /1/B/ 87(HIS). / NE2[ N]: 4.29
/1/A/ 252(MET). / CG [ C]: /1/B/ 87(HIS). / CD2[ C]: 4.31
/1/A/ 252(MET). / SD [ S]: /1/B/ 87(HIS). / CD2[ C]: 4.15
/1/A/ 436(TYR). / CD2[ C]: /1/B/ 87(HIS). / CD2[ C]: 4.65
/1/A/ 428(MET). / SD [ S]: /1/B/ 87(HIS). / CD2[ C]: 4.67
/1/A/ 436(TYR). / CE2[ C]: /1/B/ 87(HIS). / CD2[ C]: 4.83
/1/A/ 436(TYR). / CD1[ C]: /1/B/ 87(HIS). / C [ C]: 4.87
/1/A/ 436(TYR). / CE1[ C]: /1/B/ 87(HIS). / C [ C]: 4.23
/1/A/ 436(TYR). / CZ [ C]: /1/B/ 87(HIS). / C [ C]: 4.20
/1/A/ 436(TYR). / OH [ O]: /1/B/ 87(HIS). / C [ C]: 4.20
/1/A/ 436(TYR). / CE2[ C]: /1/B/ 87(HIS). / C [ C]: 4.82
/1/A/ 436(TYR). / CE1[ C]: /1/B/ 87(HIS). / O [ O]: 5.00
/1/A/ 436(TYR). / CZ [ C]: /1/B/ 87(HIS). / O [ O]: 4.84
/1/A/ 436(TYR). / OH [ O]: /1/B/ 87(HIS). / O [ O]: 4.50
/1/A/ 438(GLN). / NE2[ N]: /1/B/ 87(HIS). / O [ O]: 4.95
/1/A/ 436(TYR). / CG [ C]: /1/B/ 88(PHE). / N [ N]: 4.86
/1/A/ 436(TYR). / CD1[ C]: /1/B/ 88(PHE). / N [ N]: 4.09
/1/A/ 436(TYR). / CE1[ C]: /1/B/ 88(PHE). / N [ N]: 3.56
/1/A/ 436(TYR). / CZ [ C]: /1/B/ 88(PHE). / N [ N]: 3.96
/1/A/ 436(TYR). / OH [ O]: /1/B/ 88(PHE). / N [ N]: 4.18
/1/A/ 436(TYR). / CE2[ C]: /1/B/ 88(PHE). / N [ N]: 4.77
/1/A/ 438(GLN). / NE2[ N]: /1/B/ 88(PHE). / N [ N]: 4.98
/1/A/ 436(TYR). / CD1[ C]: /1/B/ 88(PHE). / CA [ C]: 4.59
/1/A/ 436(TYR). / CE1[ C]: /1/B/ 88(PHE). / CA [ C]: 3.87
/1/A/ 436(TYR). / CZ [ C]: /1/B/ 88(PHE). / CA [ C]: 4.43
/1/A/ 436(TYR). / OH [ O]: /1/B/ 88(PHE). / CA [ C]: 4.39
/1/A/ 438(GLN). / NE2[ N]: /1/B/ 88(PHE). / CA [ C]: 4.54
/1/A/ 436(TYR). / CD1[ C]: /1/B/ 88(PHE). / C [ C]: 4.16
/1/A/ 436(TYR). / CE1[ C]: /1/B/ 88(PHE). / C [ C]: 3.70
/1/A/ 436(TYR). / CZ [ C]: /1/B/ 88(PHE). / C [ C]: 4.68
/1/A/ 436(TYR). / OH [ O]: /1/B/ 88(PHE). / C [ C]: 4.89
/1/A/ 436(TYR). / CG [ C]: /1/B/ 88(PHE). / O [ O]: 4.70
/1/A/ 436(TYR). / CD1[ C]: /1/B/ 88(PHE). / O [ O]: 3.55
/1/A/ 436(TYR). / CE1[ C]: /1/B/ 88(PHE). / O [ O]: 3.53
/1/A/ 436(TYR). / CZ [ C]: /1/B/ 88(PHE). / O [ O]: 4.68
/1/A/ 436(TYR). / CD1[ C]: /1/B/ 89(LEU). / N [ N]: 4.94
/1/A/ 436(TYR). / CE1[ C]: /1/B/ 89(LEU). / N [ N]: 4.38
/1/A/ 438(GLN). / NE2[ N]: /1/B/ 89(LEU). / N [ N]: 4.99
/1/A/ 436(TYR). / CE1[ C]: /1/B/ 89(LEU). / CA [ C]: 4.93
/1/A/ 438(GLN). / CG [ C]: /1/B/ 89(LEU). / CB [ C]: 4.88
/1/A/ 438(GLN). / N [ N]: /1/B/ 89(LEU). / CG [ C]: 4.32
/1/A/ 436(TYR). / CE1[ C]: /1/B/ 89(LEU). / CG [ C]: 4.78
/1/A/ 438(GLN). / CA [ C]: /1/B/ 89(LEU). / CG [ C]: 4.91
/1/A/ 438(GLN). / CB [ C]: /1/B/ 89(LEU). / CG [ C]: 4.76
/1/A/ 438(GLN). / O [ O]: /1/B/ 89(LEU). / CG [ C]: 4.50
/1/A/ 438(GLN). / CG [ C]: /1/B/ 89(LEU). / CG [ C]: 3.89
/1/A/ 438(GLN). / CD [ C]: /1/B/ 89(LEU). / CG [ C]: 4.57
/1/A/ 438(GLN). / N [ N]: /1/B/ 89(LEU). / CD1[ C]: 4.61
/1/A/ 438(GLN). / O [ O]: /1/B/ 89(LEU). / CD1[ C]: 3.90
/1/A/ 438(GLN). / CG [ C]: /1/B/ 89(LEU). / CD1[ C]: 4.03
/1/A/ 438(GLN). / CD [ C]: /1/B/ 89(LEU). / CD1[ C]: 4.88
/1/A/ 438(GLN). / C [ C]: /1/B/ 89(LEU). / CD1[ C]: 4.77
/1/A/ 436(TYR). / O [ O]: /1/B/ 89(LEU). / CD2[ C]: 4.70
/1/A/ 437(THR). / CA [ C]: /1/B/ 89(LEU). / CD2[ C]: 4.19
/1/A/ 437(THR). / CB [ C]: /1/B/ 89(LEU). / CD2[ C]: 4.64
/1/A/ 437(THR). / OG1[ O]: /1/B/ 89(LEU). / CD2[ C]: 3.98
/1/A/ 438(GLN). / N [ N]: /1/B/ 89(LEU). / CD2[ C]: 4.04
/1/A/ 436(TYR). / CD1[ C]: /1/B/ 89(LEU). / CD2[ C]: 4.79
/1/A/ 436(TYR). / CE1[ C]: /1/B/ 89(LEU). / CD2[ C]: 4.72
/1/A/ 437(THR). / C [ C]: /1/B/ 89(LEU). / CD2[ C]: 4.54
/1/A/ 438(GLN). / O [ O]: /1/B/ 89(LEU). / CD2[ C]: 4.77
/1/A/ 438(GLN). / CG [ C]: /1/B/ 89(LEU). / CD2[ C]: 4.74
/1/A/ 433(HIS). / NE2[ N]: /1/B/ 90(LEU). / CG [ C]: 4.85
/1/A/ 434(ASN). / OD1[ O]: /1/B/ 90(LEU). / CG [ C]: 4.83
/1/A/ 433(HIS). / CE1[ C]: /1/B/ 90(LEU). / CD1[ C]: 4.69
/1/A/ 433(HIS). / NE2[ N]: /1/B/ 90(LEU). / CD1[ C]: 3.91
/1/A/ 434(ASN). / OD1[ O]: /1/B/ 90(LEU). / CD1[ C]: 4.26
/1/A/ 433(HIS). / CD2[ C]: /1/B/ 90(LEU). / CD1[ C]: 4.91
/1/A/ 433(HIS). / ND1[ N]: /1/B/ 100(TRP). / CB [ C]: 4.81
/1/A/ 433(HIS). / CE1[ C]: /1/B/ 100(TRP). / CB [ C]: 3.97
/1/A/ 433(HIS). / NE2[ N]: /1/B/ 100(TRP). / CB [ C]: 4.17
/1/A/ 433(HIS). / CG [ C]: /1/B/ 100(TRP). / CG [ C]: 4.51
/1/A/ 433(HIS). / ND1[ N]: /1/B/ 100(TRP). / CG [ C]: 4.17
/1/A/ 433(HIS). / CE1[ C]: /1/B/ 100(TRP). / CG [ C]: 3.54
/1/A/ 433(HIS). / NE2[ N]: /1/B/ 100(TRP). / CG [ C]: 3.46
/1/A/ 433(HIS). / CD2[ C]: /1/B/ 100(TRP). / CG [ C]: 4.11
/1/A/ 433(HIS). / CG [ C]: /1/B/ 100(TRP). / CD1[ C]: 3.93
/1/A/ 433(HIS). / ND1[ N]: /1/B/ 100(TRP). / CD1[ C]: 3.71
/1/A/ 433(HIS). / CE1[ C]: /1/B/ 100(TRP). / CD1[ C]: 3.54
/1/A/ 433(HIS). / NE2[ N]: /1/B/ 100(TRP). / CD1[ C]: 3.62
/1/A/ 433(HIS). / CB [ C]: /1/B/ 100(TRP). / CD1[ C]: 4.72
/1/A/ 433(HIS). / CD2[ C]: /1/B/ 100(TRP). / CD1[ C]: 3.88
/1/A/ 433(HIS). / CG [ C]: /1/B/ 100(TRP). / NE1[ N]: 3.53
/1/A/ 433(HIS). / ND1[ N]: /1/B/ 100(TRP). / NE1[ N]: 3.82
/1/A/ 433(HIS). / CE1[ C]: /1/B/ 100(TRP). / NE1[ N]: 3.84
/1/A/ 433(HIS). / NE2[ N]: /1/B/ 100(TRP). / NE1[ N]: 3.53
/1/A/ 433(HIS). / CB [ C]: /1/B/ 100(TRP). / NE1[ N]: 4.11
/1/A/ 433(HIS). / CD2[ C]: /1/B/ 100(TRP). / NE1[ N]: 3.34
/1/A/ 433(HIS). / CG [ C]: /1/B/ 100(TRP). / CE2[ C]: 3.95
/1/A/ 433(HIS). / ND1[ N]: /1/B/ 100(TRP). / CE2[ C]: 4.36
/1/A/ 433(HIS). / CE1[ C]: /1/B/ 100(TRP). / CE2[ C]: 4.05
/1/A/ 433(HIS). / NE2[ N]: /1/B/ 100(TRP). / CE2[ C]: 3.33
/1/A/ 433(HIS). / CB [ C]: /1/B/ 100(TRP). / CE2[ C]: 4.74
/1/A/ 433(HIS). / CD2[ C]: /1/B/ 100(TRP). / CE2[ C]: 3.27
/1/A/ 433(HIS). / CG [ C]: /1/B/ 100(TRP). / CD2[ C]: 4.50
/1/A/ 433(HIS). / ND1[ N]: /1/B/ 100(TRP). / CD2[ C]: 4.53
/1/A/ 433(HIS). / CE1[ C]: /1/B/ 100(TRP). / CD2[ C]: 3.84
/1/A/ 433(HIS). / NE2[ N]: /1/B/ 100(TRP). / CD2[ C]: 3.23
/1/A/ 433(HIS). / CD2[ C]: /1/B/ 100(TRP). / CD2[ C]: 3.73
/1/A/ 433(HIS). / CE1[ C]: /1/B/ 100(TRP). / CE3[ C]: 4.58
/1/A/ 433(HIS). / NE2[ N]: /1/B/ 100(TRP). / CE3[ C]: 3.75
/1/A/ 433(HIS). / CD2[ C]: /1/B/ 100(TRP). / CE3[ C]: 4.38
/1/A/ 433(HIS). / NE2[ N]: /1/B/ 100(TRP). / CZ3[ C]: 4.23
/1/A/ 433(HIS). / CD2[ C]: /1/B/ 100(TRP). / CZ3[ C]: 4.54
/1/A/ 433(HIS). / NE2[ N]: /1/B/ 100(TRP). / CH2[ C]: 4.35
/1/A/ 433(HIS). / CD2[ C]: /1/B/ 100(TRP). / CH2[ C]: 4.22
/1/A/ 433(HIS). / CG [ C]: /1/B/ 100(TRP). / CZ2[ C]: 4.48
/1/A/ 433(HIS). / CE1[ C]: /1/B/ 100(TRP). / CZ2[ C]: 4.95
/1/A/ 433(HIS). / NE2[ N]: /1/B/ 100(TRP). / CZ2[ C]: 3.96
/1/A/ 433(HIS). / CD2[ C]: /1/B/ 100(TRP). / CZ2[ C]: 3.60
/1/A/ 433(HIS). / ND1[ N]: /1/B/ 101(LEU). / O [ O]: 4.95
/1/A/ 433(HIS). / CE1[ C]: /1/B/ 101(LEU). / O [ O]: 4.93
/1/A/ 433(HIS). / CG [ C]: /1/B/ 102(TRP). / CB [ C]: 4.76
/1/A/ 433(HIS). / ND1[ N]: /1/B/ 102(TRP). / CB [ C]: 4.00
/1/A/ 433(HIS). / CE1[ C]: /1/B/ 102(TRP). / CB [ C]: 4.86
/1/A/ 433(HIS). / CA [ C]: /1/B/ 102(TRP). / CB [ C]: 4.71
/1/A/ 433(HIS). / CB [ C]: /1/B/ 102(TRP). / CB [ C]: 4.71
/1/A/ 433(HIS). / CG [ C]: /1/B/ 102(TRP). / CG [ C]: 4.40
/1/A/ 433(HIS). / ND1[ N]: /1/B/ 102(TRP). / CG [ C]: 4.08
/1/A/ 433(HIS). / N [ N]: /1/B/ 102(TRP). / CG [ C]: 4.81
/1/A/ 433(HIS). / CA [ C]: /1/B/ 102(TRP). / CG [ C]: 4.19
/1/A/ 433(HIS). / CB [ C]: /1/B/ 102(TRP). / CG [ C]: 4.00
/1/A/ 433(HIS). / CG [ C]: /1/B/ 102(TRP). / CD1[ C]: 4.41
/1/A/ 433(HIS). / ND1[ N]: /1/B/ 102(TRP). / CD1[ C]: 4.31
/1/A/ 433(HIS). / CA [ C]: /1/B/ 102(TRP). / CD1[ C]: 4.73
/1/A/ 433(HIS). / CB [ C]: /1/B/ 102(TRP). / CD1[ C]: 4.09
/1/A/ 433(HIS). / CG [ C]: /1/B/ 102(TRP). / NE1[ N]: 4.64
/1/A/ 433(HIS). / ND1[ N]: /1/B/ 102(TRP). / NE1[ N]: 4.93
/1/A/ 433(HIS). / N [ N]: /1/B/ 102(TRP). / NE1[ N]: 4.87
/1/A/ 433(HIS). / CA [ C]: /1/B/ 102(TRP). / NE1[ N]: 4.63
/1/A/ 433(HIS). / CB [ C]: /1/B/ 102(TRP). / NE1[ N]: 3.94
/1/A/ 433(HIS). / CG [ C]: /1/B/ 102(TRP). / CE2[ C]: 4.82
/1/A/ 432(LEU). / C [ C]: /1/B/ 102(TRP). / CE2[ C]: 4.85
/1/A/ 433(HIS). / N [ N]: /1/B/ 102(TRP). / CE2[ C]: 4.00
/1/A/ 433(HIS). / CA [ C]: /1/B/ 102(TRP). / CE2[ C]: 4.05
/1/A/ 433(HIS). / CB [ C]: /1/B/ 102(TRP). / CE2[ C]: 3.80
/1/A/ 433(HIS). / CG [ C]: /1/B/ 102(TRP). / CD2[ C]: 4.65
/1/A/ 433(HIS). / ND1[ N]: /1/B/ 102(TRP). / CD2[ C]: 4.64
/1/A/ 432(LEU). / C [ C]: /1/B/ 102(TRP). / CD2[ C]: 4.55
/1/A/ 432(LEU). / O [ O]: /1/B/ 102(TRP). / CD2[ C]: 4.82
/1/A/ 433(HIS). / N [ N]: /1/B/ 102(TRP). / CD2[ C]: 3.90
/1/A/ 433(HIS). / CA [ C]: /1/B/ 102(TRP). / CD2[ C]: 3.68
/1/A/ 433(HIS). / CB [ C]: /1/B/ 102(TRP). / CD2[ C]: 3.80
/1/A/ 432(LEU). / CA [ C]: /1/B/ 102(TRP). / CE3[ C]: 4.77
/1/A/ 432(LEU). / C [ C]: /1/B/ 102(TRP). / CE3[ C]: 3.82
/1/A/ 432(LEU). / O [ O]: /1/B/ 102(TRP). / CE3[ C]: 3.93
/1/A/ 433(HIS). / N [ N]: /1/B/ 102(TRP). / CE3[ C]: 3.56
/1/A/ 433(HIS). / CA [ C]: /1/B/ 102(TRP). / CE3[ C]: 3.62
/1/A/ 433(HIS). / CB [ C]: /1/B/ 102(TRP). / CE3[ C]: 4.26
/1/A/ 433(HIS). / C [ C]: /1/B/ 102(TRP). / CE3[ C]: 4.89
/1/A/ 431(ALA). / C [ C]: /1/B/ 102(TRP). / CE3[ C]: 4.95
/1/A/ 431(ALA). / O [ O]: /1/B/ 102(TRP). / CE3[ C]: 4.62
/1/A/ 432(LEU). / CA [ C]: /1/B/ 102(TRP). / CZ3[ C]: 3.93
/1/A/ 432(LEU). / C [ C]: /1/B/ 102(TRP). / CZ3[ C]: 3.39
/1/A/ 432(LEU). / O [ O]: /1/B/ 102(TRP). / CZ3[ C]: 3.77
/1/A/ 433(HIS). / N [ N]: /1/B/ 102(TRP). / CZ3[ C]: 3.34
/1/A/ 433(HIS). / CA [ C]: /1/B/ 102(TRP). / CZ3[ C]: 3.93
/1/A/ 433(HIS). / CB [ C]: /1/B/ 102(TRP). / CZ3[ C]: 4.67
/1/A/ 431(ALA). / CA [ C]: /1/B/ 102(TRP). / CZ3[ C]: 4.62
/1/A/ 431(ALA). / C [ C]: /1/B/ 102(TRP). / CZ3[ C]: 3.80
/1/A/ 431(ALA). / O [ O]: /1/B/ 102(TRP). / CZ3[ C]: 3.31
/1/A/ 432(LEU). / N [ N]: /1/B/ 102(TRP). / CZ3[ C]: 4.16
/1/A/ 432(LEU). / CA [ C]: /1/B/ 102(TRP). / CH2[ C]: 4.04
/1/A/ 432(LEU). / C [ C]: /1/B/ 102(TRP). / CH2[ C]: 3.82
/1/A/ 432(LEU). / O [ O]: /1/B/ 102(TRP). / CH2[ C]: 4.57
/1/A/ 433(HIS). / N [ N]: /1/B/ 102(TRP). / CH2[ C]: 3.50
/1/A/ 433(HIS). / CA [ C]: /1/B/ 102(TRP). / CH2[ C]: 4.29
/1/A/ 433(HIS). / CB [ C]: /1/B/ 102(TRP). / CH2[ C]: 4.69
/1/A/ 431(ALA). / C [ C]: /1/B/ 102(TRP). / CH2[ C]: 4.06
/1/A/ 431(ALA). / O [ O]: /1/B/ 102(TRP). / CH2[ C]: 3.22
/1/A/ 432(LEU). / N [ N]: /1/B/ 102(TRP). / CH2[ C]: 4.49
/1/A/ 345(GLU). / OE1[ O]: /1/B/ 102(TRP). / CH2[ C]: 4.05
/1/A/ 345(GLU). / OE2[ O]: /1/B/ 102(TRP). / CH2[ C]: 4.07
/1/A/ 345(GLU). / CB [ C]: /1/B/ 102(TRP). / CH2[ C]: 4.86
/1/A/ 345(GLU). / CG [ C]: /1/B/ 102(TRP). / CH2[ C]: 4.35
/1/A/ 345(GLU). / CD [ C]: /1/B/ 102(TRP). / CH2[ C]: 3.93
/1/A/ 432(LEU). / CA [ C]: /1/B/ 102(TRP). / CZ2[ C]: 4.93
/1/A/ 432(LEU). / C [ C]: /1/B/ 102(TRP). / CZ2[ C]: 4.55
/1/A/ 433(HIS). / N [ N]: /1/B/ 102(TRP). / CZ2[ C]: 3.84
/1/A/ 433(HIS). / CA [ C]: /1/B/ 102(TRP). / CZ2[ C]: 4.35
/1/A/ 433(HIS). / CB [ C]: /1/B/ 102(TRP). / CZ2[ C]: 4.28
/1/A/ 431(ALA). / O [ O]: /1/B/ 102(TRP). / CZ2[ C]: 4.48
/1/A/ 345(GLU). / OE1[ O]: /1/B/ 102(TRP). / CZ2[ C]: 4.04
/1/A/ 345(GLU). / OE2[ O]: /1/B/ 102(TRP). / CZ2[ C]: 3.89
/1/A/ 345(GLU). / CG [ C]: /1/B/ 102(TRP). / CZ2[ C]: 4.89
/1/A/ 345(GLU). / CD [ C]: /1/B/ 102(TRP). / CZ2[ C]: 4.04
/1/A/ 345(GLU). / OE1[ O]: /1/B/ 106(LYS). / CE [ C]: 4.63
/1/B/ 106(LYS). / NZ [ N]: 3.50
/1/A/ 345(GLU). / CD [ C]: /1/B/ 106(LYS). / NZ [ N]: 4.60
/1/A/ 438(GLN). / O [ O]: /1/B/ 112(TYR). / CZ [ C]: 4.56
/1/A/ 439(LYS). / CD [ C]: /1/B/ 112(TYR). / CZ [ C]: 4.43
/1/A/ 440(SER). / N [ N]: /1/B/ 112(TYR). / CZ [ C]: 4.81
/1/A/ 440(SER). / CB [ C]: /1/B/ 112(TYR). / CZ [ C]: 4.88
/1/A/ 438(GLN). / O [ O]: /1/B/ 112(TYR). / OH [ O]: 4.09
/1/A/ 439(LYS). / CB [ C]: /1/B/ 112(TYR). / OH [ O]: 4.32
/1/A/ 439(LYS). / CG [ C]: /1/B/ 112(TYR). / OH [ O]: 4.43
/1/A/ 439(LYS). / CD [ C]: /1/B/ 112(TYR). / OH [ O]: 3.52
/1/A/ 439(LYS). / CE [ C]: /1/B/ 112(TYR). / OH [ O]: 4.58
/1/A/ 439(LYS). / NZ [ N]: /1/B/ 112(TYR). / OH [ O]: 4.99
/1/A/ 438(GLN). / C [ C]: /1/B/ 112(TYR). / OH [ O]: 4.87
/1/A/ 439(LYS). / N [ N]: /1/B/ 112(TYR). / OH [ O]: 4.85
/1/A/ 439(LYS). / CA [ C]: /1/B/ 112(TYR). / OH [ O]: 3.84
/1/A/ 439(LYS). / C [ C]: /1/B/ 112(TYR). / OH [ O]: 4.25
/1/A/ 440(SER). / N [ N]: /1/B/ 112(TYR). / OH [ O]: 3.51
/1/A/ 440(SER). / OG [ O]: /1/B/ 112(TYR). / OH [ O]: 4.67
/1/A/ 440(SER). / CA [ C]: /1/B/ 112(TYR). / OH [ O]: 4.39
/1/A/ 440(SER). / CB [ C]: /1/B/ 112(TYR). / OH [ O]: 3.92
/1/A/ 440(SER). / O [ O]: /1/B/ 112(TYR). / OH [ O]: 4.83
/1/A/ 438(GLN). / O [ O]: /1/B/ 112(TYR). / CE2[ C]: 4.13
/1/A/ 439(LYS). / CD [ C]: /1/B/ 112(TYR). / CE2[ C]: 4.59
/1/A/ 434(ASN). / ND2[ N]: /1/B/ 169(PHE). / N [ N]: 4.75
/1/A/ 311(GLN). / OE1[ O]: /1/B/ 169(PHE). / CA [ C]: 5.00
/1/A/ 311(GLN). / CD [ C]: /1/B/ 169(PHE). / CB [ C]: 4.99
/1/A/ 311(GLN). / OE1[ O]: /1/B/ 169(PHE). / CB [ C]: 4.11
/1/A/ 435(HIS). / CE1[ C]: /1/B/ 169(PHE). / CB [ C]: 4.62
/1/A/ 435(HIS). / NE2[ N]: /1/B/ 169(PHE). / CB [ C]: 4.66
/1/A/ 311(GLN). / OE1[ O]: /1/B/ 169(PHE). / CG [ C]: 4.40
/1/A/ 435(HIS). / CG [ C]: /1/B/ 169(PHE). / CG [ C]: 4.96
/1/A/ 435(HIS). / ND1[ N]: /1/B/ 169(PHE). / CG [ C]: 4.45
/1/A/ 435(HIS). / CE1[ C]: /1/B/ 169(PHE). / CG [ C]: 4.25
/1/A/ 435(HIS). / NE2[ N]: /1/B/ 169(PHE). / CG [ C]: 4.53
/1/A/ 435(HIS). / CD2[ C]: /1/B/ 169(PHE). / CG [ C]: 4.94
/1/A/ 434(ASN). / ND2[ N]: /1/B/ 169(PHE). / CG [ C]: 4.38
/1/A/ 434(ASN). / CA [ C]: /1/B/ 169(PHE). / CG [ C]: 4.56
/1/A/ 434(ASN). / C [ C]: /1/B/ 169(PHE). / CG [ C]: 4.99
/1/A/ 434(ASN). / CG [ C]: /1/B/ 169(PHE). / CD1[ C]: 4.13
/1/A/ 434(ASN). / ND2[ N]: /1/B/ 169(PHE). / CD1[ C]: 3.11
/1/A/ 434(ASN). / N [ N]: /1/B/ 169(PHE). / CD1[ C]: 4.62
/1/A/ 434(ASN). / CA [ C]: /1/B/ 169(PHE). / CD1[ C]: 3.68
/1/A/ 434(ASN). / CB [ C]: /1/B/ 169(PHE). / CD1[ C]: 4.28
/1/A/ 434(ASN). / C [ C]: /1/B/ 169(PHE). / CD1[ C]: 4.34
/1/A/ 434(ASN). / O [ O]: /1/B/ 169(PHE). / CD1[ C]: 4.64
/1/A/ 435(HIS). / ND1[ N]: /1/B/ 169(PHE). / CE1[ C]: 4.99
/1/A/ 434(ASN). / CG [ C]: /1/B/ 169(PHE). / CE1[ C]: 3.74
/1/A/ 434(ASN). / OD1[ O]: /1/B/ 169(PHE). / CE1[ C]: 4.81
/1/A/ 434(ASN). / ND2[ N]: /1/B/ 169(PHE). / CE1[ C]: 3.09
/1/A/ 434(ASN). / N [ N]: /1/B/ 169(PHE). / CE1[ C]: 4.72
/1/A/ 434(ASN). / CA [ C]: /1/B/ 169(PHE). / CE1[ C]: 3.44
/1/A/ 434(ASN). / CB [ C]: /1/B/ 169(PHE). / CE1[ C]: 3.71
/1/A/ 434(ASN). / C [ C]: /1/B/ 169(PHE). / CE1[ C]: 3.84
/1/A/ 434(ASN). / O [ O]: /1/B/ 169(PHE). / CE1[ C]: 3.79
/1/A/ 435(HIS). / N [ N]: /1/B/ 169(PHE). / CE1[ C]: 4.89
/1/A/ 435(HIS). / CG [ C]: /1/B/ 169(PHE). / CZ [ C]: 4.75
/1/A/ 435(HIS). / ND1[ N]: /1/B/ 169(PHE). / CZ [ C]: 4.33
/1/A/ 435(HIS). / CE1[ C]: /1/B/ 169(PHE). / CZ [ C]: 4.92
/1/A/ 253(ILE). / CD1[ C]: /1/B/ 169(PHE). / CZ [ C]: 4.96
/1/A/ 434(ASN). / CG [ C]: /1/B/ 169(PHE). / CZ [ C]: 4.83
/1/A/ 434(ASN). / ND2[ N]: /1/B/ 169(PHE). / CZ [ C]: 4.38
/1/A/ 434(ASN). / CA [ C]: /1/B/ 169(PHE). / CZ [ C]: 4.17
/1/A/ 434(ASN). / CB [ C]: /1/B/ 169(PHE). / CZ [ C]: 4.50
/1/A/ 434(ASN). / C [ C]: /1/B/ 169(PHE). / CZ [ C]: 4.08
/1/A/ 434(ASN). / O [ O]: /1/B/ 169(PHE). / CZ [ C]: 3.75
/1/A/ 435(HIS). / CB [ C]: /1/B/ 169(PHE). / CZ [ C]: 4.87
/1/A/ 311(GLN). / OE1[ O]: /1/B/ 169(PHE). / CE2[ C]: 4.75
/1/A/ 435(HIS). / CG [ C]: /1/B/ 169(PHE). / CE2[ C]: 4.44
/1/A/ 435(HIS). / ND1[ N]: /1/B/ 169(PHE). / CE2[ C]: 3.66
/1/A/ 435(HIS). / CE1[ C]: /1/B/ 169(PHE). / CE2[ C]: 4.05
/1/A/ 435(HIS). / NE2[ N]: /1/B/ 169(PHE). / CE2[ C]: 4.86
/1/A/ 253(ILE). / CB [ C]: /1/B/ 169(PHE). / CE2[ C]: 4.90
/1/A/ 253(ILE). / CG1[ C]: /1/B/ 169(PHE). / CE2[ C]: 4.66
/1/A/ 253(ILE). / CD1[ C]: /1/B/ 169(PHE). / CE2[ C]: 3.95
/1/A/ 434(ASN). / CA [ C]: /1/B/ 169(PHE). / CE2[ C]: 4.99
/1/A/ 434(ASN). / C [ C]: /1/B/ 169(PHE). / CE2[ C]: 4.80
/1/A/ 434(ASN). / O [ O]: /1/B/ 169(PHE). / CE2[ C]: 4.62
/1/A/ 435(HIS). / CB [ C]: /1/B/ 169(PHE). / CE2[ C]: 4.90
/1/A/ 311(GLN). / CD [ C]: /1/B/ 169(PHE). / CD2[ C]: 4.96
/1/A/ 311(GLN). / OE1[ O]: /1/B/ 169(PHE). / CD2[ C]: 3.76
/1/A/ 435(HIS). / CG [ C]: /1/B/ 169(PHE). / CD2[ C]: 4.52
/1/A/ 435(HIS). / ND1[ N]: /1/B/ 169(PHE). / CD2[ C]: 3.70
/1/A/ 435(HIS). / CE1[ C]: /1/B/ 169(PHE). / CD2[ C]: 3.62
/1/A/ 435(HIS). / NE2[ N]: /1/B/ 169(PHE). / CD2[ C]: 4.29
/1/A/ 435(HIS). / CD2[ C]: /1/B/ 169(PHE). / CD2[ C]: 4.79
/1/A/ 253(ILE). / CD1[ C]: /1/B/ 169(PHE). / CD2[ C]: 4.45
/1/A/ 311(GLN). / CG [ C]: /1/B/ 170(ASN). / N [ N]: 4.32
/1/A/ 311(GLN). / CD [ C]: /1/B/ 170(ASN). / N [ N]: 4.38
/1/A/ 311(GLN). / OE1[ O]: /1/B/ 170(ASN). / N [ N]: 4.25
/1/A/ 311(GLN). / CG [ C]: /1/B/ 170(ASN). / CA [ C]: 4.32
/1/A/ 311(GLN). / CD [ C]: /1/B/ 170(ASN). / CA [ C]: 4.59
/1/A/ 311(GLN). / OE1[ O]: /1/B/ 170(ASN). / CA [ C]: 4.84
/1/A/ 311(GLN). / CG [ C]: /1/B/ 170(ASN). / CB [ C]: 4.12
/1/A/ 311(GLN). / CD [ C]: /1/B/ 170(ASN). / CB [ C]: 4.91
/1/A/ 315(ASN). / ND2[ N]: /1/B/ 170(ASN). / CG [ C]: 4.99
/1/B/ 170(ASN). / ND2[ N]: 4.56
/1/A/ 311(GLN). / NE2[ N]: /1/B/ 170(ASN). / C [ C]: 4.13
/1/A/ 315(ASN). / CG [ C]: /1/B/ 170(ASN). / C [ C]: 4.92
/1/A/ 315(ASN). / OD1[ O]: /1/B/ 170(ASN). / C [ C]: 4.54
/1/A/ 315(ASN). / ND2[ N]: /1/B/ 170(ASN). / C [ C]: 4.33
/1/A/ 311(GLN). / CG [ C]: /1/B/ 170(ASN). / C [ C]: 4.00
/1/A/ 311(GLN). / CD [ C]: /1/B/ 170(ASN). / C [ C]: 4.01
/1/A/ 311(GLN). / OE1[ O]: /1/B/ 170(ASN). / C [ C]: 4.50
/1/A/ 311(GLN). / NE2[ N]: /1/B/ 170(ASN). / O [ O]: 4.32
/1/A/ 315(ASN). / CG [ C]: /1/B/ 170(ASN). / O [ O]: 4.07
/1/A/ 315(ASN). / OD1[ O]: /1/B/ 170(ASN). / O [ O]: 3.81
/1/A/ 315(ASN). / ND2[ N]: /1/B/ 170(ASN). / O [ O]: 3.45
/1/A/ 311(GLN). / CG [ C]: /1/B/ 170(ASN). / O [ O]: 4.17
/1/A/ 311(GLN). / CD [ C]: /1/B/ 170(ASN). / O [ O]: 4.42
/1/A/ 311(GLN). / NE2[ N]: /1/B/ 171(ASP). / N [ N]: 3.64
/1/A/ 315(ASN). / OD1[ O]: /1/B/ 171(ASP). / N [ N]: 4.49
/1/A/ 315(ASN). / ND2[ N]: /1/B/ 171(ASP). / N [ N]: 4.84
/1/A/ 311(GLN). / CG [ C]: /1/B/ 171(ASP). / N [ N]: 4.23
/1/A/ 311(GLN). / CD [ C]: /1/B/ 171(ASP). / N [ N]: 3.71
/1/A/ 311(GLN). / OE1[ O]: /1/B/ 171(ASP). / N [ N]: 4.02
/1/A/ 311(GLN). / NE2[ N]: /1/B/ 171(ASP). / CA [ C]: 3.58
/1/A/ 315(ASN). / CG [ C]: /1/B/ 171(ASP). / CA [ C]: 4.57
/1/A/ 315(ASN). / OD1[ O]: /1/B/ 171(ASP). / CA [ C]: 3.67
/1/A/ 315(ASN). / ND2[ N]: /1/B/ 171(ASP). / CA [ C]: 4.64
/1/A/ 311(GLN). / CG [ C]: /1/B/ 171(ASP). / CA [ C]: 4.86
/1/A/ 311(GLN). / CD [ C]: /1/B/ 171(ASP). / CA [ C]: 4.15
/1/A/ 311(GLN). / OE1[ O]: /1/B/ 171(ASP). / CA [ C]: 4.62
/1/A/ 311(GLN). / NE2[ N]: /1/B/ 171(ASP). / CB [ C]: 4.15
/1/A/ 315(ASN). / OD1[ O]: /1/B/ 171(ASP). / CB [ C]: 4.67
/1/A/ 311(GLN). / CD [ C]: /1/B/ 171(ASP). / CB [ C]: 4.79
/1/A/ 311(GLN). / OE1[ O]: /1/B/ 171(ASP). / CB [ C]: 4.96
/1/A/ 435(HIS). / NE2[ N]: /1/B/ 171(ASP). / CB [ C]: 4.86
/1/A/ 311(GLN). / NE2[ N]: /1/B/ 171(ASP). / CG [ C]: 3.85
/1/A/ 311(GLN). / CD [ C]: /1/B/ 171(ASP). / CG [ C]: 4.25
/1/A/ 311(GLN). / OE1[ O]: /1/B/ 171(ASP). / CG [ C]: 4.06
/1/A/ 435(HIS). / CE1[ C]: /1/B/ 171(ASP). / CG [ C]: 4.15
/1/A/ 435(HIS). / NE2[ N]: /1/B/ 171(ASP). / CG [ C]: 3.38
/1/A/ 435(HIS). / CD2[ C]: /1/B/ 171(ASP). / CG [ C]: 4.42
/1/A/ 430(GLU). / O [ O]: /1/B/ 171(ASP). / CG [ C]: 4.44
/1/A/ 311(GLN). / NE2[ N]: /1/B/ 171(ASP). / OD1[ O]: 3.45
/1/A/ 311(GLN). / CD [ C]: /1/B/ 171(ASP). / OD1[ O]: 3.99
/1/A/ 311(GLN). / OE1[ O]: /1/B/ 171(ASP). / OD1[ O]: 3.74
/1/A/ 435(HIS). / CG [ C]: /1/B/ 171(ASP). / OD1[ O]: 4.78
/1/A/ 435(HIS). / ND1[ N]: /1/B/ 171(ASP). / OD1[ O]: 4.56
/1/A/ 435(HIS). / CE1[ C]: /1/B/ 171(ASP). / OD1[ O]: 3.34
/1/A/ 435(HIS). / NE2[ N]: /1/B/ 171(ASP). / OD1[ O]: 2.60
/1/A/ 435(HIS). / CD2[ C]: /1/B/ 171(ASP). / OD1[ O]: 3.73
/1/A/ 430(GLU). / CA [ C]: /1/B/ 171(ASP). / OD1[ O]: 4.92
/1/A/ 430(GLU). / O [ O]: /1/B/ 171(ASP). / OD1[ O]: 3.69
/1/A/ 314(LEU). / CD1[ C]: /1/B/ 171(ASP). / OD1[ O]: 4.45
/1/A/ 430(GLU). / C [ C]: /1/B/ 171(ASP). / OD1[ O]: 4.68
/1/A/ 311(GLN). / NE2[ N]: /1/B/ 171(ASP). / OD2[ O]: 4.60
/1/A/ 311(GLN). / CD [ C]: /1/B/ 171(ASP). / OD2[ O]: 4.67
/1/A/ 311(GLN). / OE1[ O]: /1/B/ 171(ASP). / OD2[ O]: 4.23
/1/A/ 435(HIS). / CE1[ C]: /1/B/ 171(ASP). / OD2[ O]: 4.15
/1/A/ 435(HIS). / NE2[ N]: /1/B/ 171(ASP). / OD2[ O]: 3.39
/1/A/ 435(HIS). / CD2[ C]: /1/B/ 171(ASP). / OD2[ O]: 4.26
/1/A/ 430(GLU). / O [ O]: /1/B/ 171(ASP). / OD2[ O]: 4.86
/1/A/ 311(GLN). / NE2[ N]: /1/B/ 171(ASP). / C [ C]: 4.88
/1/A/ 315(ASN). / CG [ C]: /1/B/ 171(ASP). / C [ C]: 4.72
/1/A/ 315(ASN). / OD1[ O]: /1/B/ 171(ASP). / C [ C]: 3.82
/1/A/ 315(ASN). / ND2[ N]: /1/B/ 171(ASP). / C [ C]: 4.83
/1/A/ 315(ASN). / OD1[ O]: /1/B/ 171(ASP). / O [ O]: 5.00
/1/A/ 315(ASN). / CG [ C]: /1/B/ 172(GLY). / N [ N]: 4.06
/1/A/ 315(ASN). / OD1[ O]: /1/B/ 172(GLY). / N [ N]: 3.11
/1/A/ 315(ASN). / ND2[ N]: /1/B/ 172(GLY). / N [ N]: 4.44
/1/A/ 315(ASN). / CG [ C]: /1/B/ 172(GLY). / CA [ C]: 4.81
/1/A/ 315(ASN). / OD1[ O]: /1/B/ 172(GLY). / CA [ C]: 4.02
/1/A/ 315(ASN). / CG [ C]: /1/B/ 172(GLY). / C [ C]: 4.59
/1/A/ 315(ASN). / OD1[ O]: /1/B/ 172(GLY). / C [ C]: 4.17
/1/A/ 315(ASN). / ND2[ N]: /1/B/ 172(GLY). / C [ C]: 4.74
/1/A/ 315(ASN). / CG [ C]: /1/B/ 173(GLY). / N [ N]: 4.04
/1/A/ 315(ASN). / OD1[ O]: /1/B/ 173(GLY). / N [ N]: 3.83
/1/A/ 315(ASN). / ND2[ N]: /1/B/ 173(GLY). / N [ N]: 3.84
/1/A/ 315(ASN). / CG [ C]: /1/B/ 173(GLY). / CA [ C]: 4.59
/1/A/ 315(ASN). / OD1[ O]: /1/B/ 173(GLY). / CA [ C]: 4.74
/1/A/ 315(ASN). / ND2[ N]: /1/B/ 173(GLY). / CA [ C]: 4.10
1. CCP4 (1994) Acta Crystallogr D 50:760-763.
SI Text
Protein Expression and Purification
Expressed TRIM21 PRYSPRY domain was purified by Ni-NTA resin (Qiagen) and Superdex75 gel filtration (Amersham Pharmacia Biosciences). Serum Fc fragment was obtained from Calbiochem (cat no. 401104). Protein concentrations were measured by using a extinction coefficients (e0.1%) of 1.4 and 1.9 for Fc and TRIM21, respectively.
Rapid Reaction Kinetics
Experiments were carried out in 5 mM ammonium acetate, pH 8 and 50 mM NaCl, the starting conditions for the crystallographic analysis. A range of pH conditions from pH 5 to pH 8 in 5 mM ammonium acetate and 50 mM NaCl were also tested. Stopped-flow experiments were performed at 30°C in an Applied Photophysics (Leatherhead, UK) SX-18MV stopped-flow spectrofluorimeter in 1:1 mixing mode and a 320-nm cut-off filter. In the association experiments 1 mM TRIM21 was mixed against increasing pseudofirst-order concentrations of Fc and the resulting monophasic fluorescence quench was fitted to a single exponential function (Eq 1).
F
= DF exp(-kobst) + Fe, [1]where F is the observed fluorescence, DF is the fluorescence amplitude, kobs is the observed pseudofirst-order rate constant, which is equivalent to the inverse relaxation time (1/t), and Fe is the end-point fluorescence. Although essentially identical results over short timescales were obtained regardless of which protein was in pseudofirst-order excess, unbound TRIM21 causes a significant photobleach over long timescales. Hence we focused on experiments with excess Fc to eliminate complications caused by this. Fitting of the linear concentration dependence of kobs to Eq. 2 yields the bimolecular association rate constant (kon):
k
obs = kon[Fc binding sites] + kreverse. [2]In the dissociation experiments a premixed complex of 1 mM Fc + 2.5 mM TRIM21 was mixed against 25 mM protein A, and the resulting monophasic fluorescence enhancement was fitted to Eq 1. To minimize photobleaching of released TRIM21, monochromator slit-widths were halved and an excitation of wavelength of 295 nm rather than 280 nm was used. Thus the amplitude change for the dissociation experiment will be different from the association experiment.
Titration Calorimetry
Proteins were dialyzed overnight at 4°C into 5 mM ammonium acetate, pH 8 and 50 mM NaCl, quantified as described above, and diluted to the appropriate concentration in the assay buffer before degassing immediately before use. The machine was used according to the manufacturer's instructions with an initial 3-ml injection followed by 27 10-ml injections. After the heats of dilution were subtracted the data were analyzed by using a single class of binding sites model to directly determine the stoichiometry, affinity constant Ka (and thus Kd), and DH, using the manufacturer's software.
Fluorescein Labeling of TRIM21 Mutants
Proteins were labeled with 5' (and 6') carboxyfluorescein succinimidyl ester (Molecular Probes) as described (1).
Fluorescence Anisotropy
Binding was carried out in PBS, pH 8.0 at 25°C with a Hamilton microlab M dispenser. Fluorescence was excited at 480 nm and emission was detected at 530 nm. Five-microliter aliquots of IgG (at varying concentrations) were added to labeled TRIM21 at 1-min intervals incorporating 30 s of stirring and measurement of the fluorescence anisotropy (1). By mixing excess IgG with TRIM21, binding simplifies to a single site model. After correction for dilution, the dissociation constant (Kd) was determined by fitting the change in anisotropy to a single site binding model by using Kaleidagraph (Synergy Software).
Crystallography
Purified TRIM21 was mixed in a 2:1 molar ratio with Fc and purified on a Superdex 200 size exclusion column. A single peak of 100 kDa corresponding to two TRIM21 and one Fc was observed. The complex was concentrated and used in robotic crystal screening trials. Crystals were observed in many conditions, including within the protein sample if left overnight at 4°C but largest crystals were observed in 12% PEG 6K, 0.1 M LiSO4, and 0.1 M sodium citrate, pH 5.6. Crystals were cryo-protected in 25% glycerol and frozen directly in the cro-stream. Several data sets were collected, the best of which had data to 2.35-Å resolution. The data were indexed in MOSFLM and scaled in SCALA (2). Data could be indexed in either P6 or P622 spacegroups and both were tested in the Molecular Replacement program PHASER, using a single-chain of Fc from 1DN2 (3) as a search model. A molecular replacement solution can also be found with Fc in combination with sRFPL1. The top solution in P61 was refined by using REFMAC, and TRIM21 was built into the Fc-phased maps over several rounds. The complete P61 model was refined with tight NCS for the two TRIM21 molecules and the two Fc chains, which when combined with 178 water molecules gave a final Rfree of 25%. As a result of the unusually high solvent content (~70%) there are few crystal contacts.
1. Fernandez-Fernandez MR, Veprintsev DB, Fersht AR (2005) Proc Natl Acad Sci USA 102:4735-4740.
2. CCP4 (1994) Acta Crystallogr D 50:760-763.
3. DeLano WL, Ultsch MH, de Vos AM, Wells JA (2000) Science 287:1279-1283.