Abstract
目的
在中国非综合征型遗传性聋患者人群中,采用PCR-反向点杂交法检测涵盖中国人群常见4个耳聋基因的20种基因突变,分析总结突变数据,探讨该方法在遗传性聋基因检测中的临床实用价值。
方法
分别采用PCR-反向点杂交法和Sanger测序法,对500例非综合征型遗传性聋患者行4个耳聋相关基因(GJB2、GJB3、SLC26A4和mtDNA)共20种突变检测。以Sanger测序法结果为金标准,探讨PCR-反向点杂交法检测耳聋突变的阳性符合率、阴性符合率、总符合率、灵敏度、特异性、阳性预测值和阴性预测值等检测性能指标。
结果
共检测500例样本,在20种基因突变的突变型范围内,使用PCR-反向点杂交法检出147例野生型样本,81例纯合突变型样本,240例杂合突变型样本,32例复合杂合突变型样本,与Sanger测序法结果完全一致。GJB2 c.235delC和SLC26A4 c.919-2 A > G是本研究中最为常见的热点突变,其次是mtDNA m.1555 A > G。对比Sanger测序法,PCR-反向点杂交法检测20个耳聋基因突变位点的灵敏度(阳性符合率)、特异性(阴性符合率)、阳性预测值、阴性预测值、总符合率均为100%,Kappa值为1。
结论
PCR-反向点杂交法用于耳聋基因突变的检测,具有简便、快速、灵敏度高、特异性强等优点,可准确检出中国人群常见4个耳聋基因的20种基因突变的突变型,将来有望用于耳聋基因的临床检测。
Keywords: 遗传性聋, PCR, 基因突变, 分子杂交
Abstract
Objective
To detect 20 common deafness gene mutations in non-syndromic deafness patients in China using PCR-RDB, and analyze and summarize the mutation data to explore the clinical value of this method.
Method
The PCR-RDB and Sanger sequencing were used to detect 20 common mutations of four deafness genes(GJB2, GJB3, SLC26A4 and mtDNA) in 500 patients with non-syndromic hearing loss. The Sanger sequencing was used to compare the sensitivity, specificity, positive predictive value, negative predictive value, and total coincidence rate of the deafness mutation detected by PCR-RDB.
Result
A total of 500 samples were detected. 147 wild-type samples, 81 homozygous mutant samples, 240 heterozygous mutant samples, 32 composite heterozygous mutant samples were detected using the PCR-RDB within the range of 20 gene mutations, which were identical to the Sanger sequencing results. GJB2 c.235delC and SLC26A4 c.919-2 A > G are the most common hotspot mutations in this study, followed by mtDNA m. 1555 A > G. Compared with the Sanger sequencing method, the sensitivity, specificity, positive predictive value, negative predictive value, and total coincidence rate of the real-time fluorescence PCR melting curve method were 100%, and the Kappa value was one.
Conclusion
PCR reverse dot-blot hybridization is a simple, rapid, sensitive and specific method for detecting 20 mutations of 4 common deafness genes in Chinese population, it is expected to be used in clinical detection of deafness genes in the future.
Keywords: hereditary deafness, PCR, gene mutation, molecular hybridization
耳聋是指听觉系统中传音、感音及听觉部分的听神经及各中枢神经发生病变而引起的听功能障碍,造成不同程度的听力减退。先天性聋是人类最常见的出生缺陷之一,每1000个新生儿中就有1个耳聋患儿(WHO,2017)。耳聋可以由基因突变引起,也可由环境因素,或基因与环境二者共同作用而致。其中,遗传性听力损失可分综合征型和非综合征型,综合征型听力损失伴随有其他组织器官的病变,非综合征型听力损失不伴有其他症状,占遗传性聋发病率的70%〔1〕。此外,遗传基因突变(线粒体基因)与致聋药物共同作用还会导致药物性聋。迄今为止,国内外的研究共定位耳聋相关的基因座位165个,其中仅有113个非综合征型基因被克隆(http://hereditaryhearingloss.org,2019年7月)。耳聋基因的突变位点多样,且具有明显的种族及区域差异,在不同国家或不同种族,即便是在同一国家的不同地区,其突变的频率也有较大差异〔2〕。中国人常见遗传性聋的基因主要包括GJB2、SLC26A4和mtDNA 12S rRNA,且每个基因都有多个较常见的突变类型〔3〕。GJB2基因是导致遗传性非综合征型耳聋最常见的基因,约20%的先天性聋患者的致聋原因与该基因有关〔4〕。其次为与大前庭水管综合征密切相关的SLC26A4基因和导致药物中毒性聋的线粒体基因mtDNA A1555G及C1494T突变〔5-6〕。除了上述3个常见耳聋基因,与语后高频听力下降有关的GJB3是中国首个克隆的遗传疾病基因〔7〕。本研究采用PCR-反向点杂交法,针对500例非综合征型遗传性聋患者,对上述4个常见耳聋基因的20种基因突变进行检测,分析检测结果,并与基因测序金标准Sanger测序进行比较,分析总结突变数据,探讨该方法在遗传性聋基因检测中的临床实用价值。
1. 材料与方法
1.1. 研究对象
选取2015-07-2018-11就诊于中南大学湘雅医院耳鼻咽喉头颈外科的非综合征型耳聋患者500例,其中男283例,女217例;年龄(21.030±17.893)岁。基本特征见表 1。
表 1.
500例非综合征型遗传性聋患者基本特征
| 性别 | 例数(%) | x±s | 中位值 | (P25,P75) | (Min,Max) |
| 男 | 283(56.60) | 19.91±17.89 | 16 | (4.00,31.00) | (0.00,69.00) |
| 女 | 217(43.40) | 22.47±18.04 | 21 | (5.00,35.00) | (0.00,75.00) |
| 合计 | 500(100.00) | 21.03±17.98 | 19 | (5.00,33.50) | (0.00,75.00) |
纳入标准:未知病因的非综合征型感音神经性聋患者。排除标准:①合并有其他临床表现的综合征型耳聋患者;②其他由环境因素引起的耳聋。
1.2. 研究方法
1.2.1. 病史采集
所有患者签署知情同意书后,对患者进行全身体检及耳鼻咽喉专科检查,然后采用问卷调查及询问患儿家长等方式采集病史资料,内容包括基本情况、疾病史、家族史、耳部外伤史、耳毒性药物史、母亲分娩史、新生儿期高危因素、全身其他系统情况(心脏病、骨骼发育、视力)、外耳和中耳疾病史,以及既往听力学检查资料等。
1.2.2. 听力检查
根据研究对象的年龄和配合程度进行听力学检查,包括纯音测听(PTA)、听性脑干反应(ABR)、声导抗、耳声发射(DPOAE)、听觉多频稳态诱发反应(ASSR)、行为测听等。纯音测听以听力水平的分贝值(dB)为单位,以频率500、1000、2000、4000 Hz的听阈平均值计算,将听力损失的严重程度按照WHO 1997年标准分成5级:正常(< 25 dB);轻度(26~40 dB);中度(41~60 dB);重度(61~80 dB);极重度(> 80 dB)。
1.2.3. 基因组DNA的提取及质控
本研究经过中南大学湘雅医院医学伦理委员会批准(审批号:201806086),并获得患者的知情同意后抽取患者的外周血,采用全血基因组DNA提取试剂盒和纯化试剂对样本进行核酸提取和纯化,提取后的DNA-18℃保存。
1.2.4. 基因检测方法
采用PCR扩增和反向点杂交技术,检测500例非综合征型遗传性聋患者的4个遗传性聋相关基因20种突变类型(分别为:GJB2基因35delG、167delT、176-19ldel16、235delC、299-300delAT;GJB3基因中538C>T、547G>A;12SrRNA基因1494C > T和1555A > G;SLC26A4基因2168A > G、IVS7-2A > G、281C > T、589G > A、1174A > T、1226G > A、1229C > T、IVS15+5G > A、1975G > C、2027T > A、2162C > T)。首先根据上述20个基因突变特点设计特异引物,引物为制定碱基序列的寡聚核苷酸,其5’端用生物素标记,在基因扩增仪(Hema9600)上扩增获得包含所要检测的各个位点的目的片段,PCR扩增反应程序为50℃ 15 min;95℃ 10min;PCR循环程序(95℃ 60 s、63℃ 30 s、72℃ 30 s,循环35次);72℃ 5 min。根据检测位点碱基差异设计特异探针,探针为指定碱基序列的寡聚核苷酸,其5’端用氨基标记,再将扩增产物与固定在尼龙膜条上的寡核苷酸探针在分子杂交仪(FinePCRCombi-H12Korea)上进行杂交,依据杂交信号的有无,判断该探针是否与PCR产物杂交,从而确定患者的基因型。以检测位点是否出现蓝色斑迹信号来进行判断,信号点的强弱不能提供定量方面的参考。阳性判断值成立的前提为空白对照的结果应为尼龙膜上所有位点均不显色,所有患者样品10个正常对照位点应大部分出现蓝色斑点。膜条上所有正常位点(N)显色,所有突变位点(M)均不显色,表示该样品阴性。膜条上有突变位点(M)显色,则表示该位点阳性。
1.2.5. Sanger基因测序
对于得到的所有阳性结果和阴性结果进一步采用Sanger双脱氧链终止法进行测序。Sanger双脱氧链终止法是目前获取核酸序列最为常用的方法,用双脱氧核苷酸作为链终止试剂通过聚合酶的引物延伸产生一系列大小不同的分子后再进行分离,对产物进行聚丙烯酰胺凝胶电泳及放射自显影,根据电泳条带确定待测分子的核苷酸序列。该方法是目前基因诊断的金标准。在本研究中,耳聋测序反应液分为Ⅰ~Ⅹ组(表 2),其中使用反向引物测序的第Ⅰ、Ⅳ、Ⅵ、Ⅶ、Ⅸ组需要使用峰图查看软件,先将测序结果进行反向互补后再与标准序列进行比对。根据检测位点的峰图判断样品基因型:某个位点只有正常碱基的峰时,判断为此位点未突变;某个位点只有突变碱基的峰时,判断为此位点纯合突变;某个位点既有正常碱基的峰又有突变碱基的峰时,判断为该位点杂合突变。每次测序实验,都设置了阳性质控和阴性(空白)质控。各基因型对应的标准序列见表 2。
表 2.
20种基因型对应的标准序列
| 测序反应液 | 位点(c.) | 标准序列 (大写为突变检测位点) |
| Ⅰ | 35delG | cctgggggGtgtgaacaaac |
| 167delT | tgcaacacccTgcagccag | |
| 176-191del16 | tgcagccagGCTGCAAGAACGTGTGctacgatcac | |
| 235delC | tatgggccCtgcagctgatc | |
| 299-300delAT | cctaccggagacATgagaagaag | |
| Ⅱ | 1494C > T | caccgcccgtcacCctcctc |
| 1555A > G | tatagaggagAcaagtcgtaacatgg | |
| Ⅲ | 538C>T | gctacattgccCgacctaccgagaagaaaa |
| 547G > A | gctacattgcccgacctaccGagaagaaaa | |
| Ⅳ | 2162C > T | attctttttgaCggtccatg |
| 2168A > G | ttgacggtccAtgatgctatact | |
| Ⅴ | IVS7-2A > G | ttttatttcAgacgataattg |
| Ⅵ | 281C > T | gagttagtaCtgggctagtggc |
| Ⅶ | 589G > A | tgctggttGgaattatacagg |
| Ⅷ | 1174A > T | tgggatcagcAacatcttctcaggatt |
| 1226G > A | tgctctttcccGcacggccgtccagg | |
| 1229C>T | tttcccgcaCggccgtccagg | |
| Ⅸ | IVS15+5G > A | agtccacagtaaGtattttatcccta |
| Ⅹ | 1975G > C | ccatagccttGtgcttgactg |
| 2027T > A | gtgagatcacTgcgggtggtaa |
1.2.6. 突变定性统计方法
针对PCR-反向点杂交法检测所涵盖的20种突变进行定性统计,并与Sanger测序法进行比较(表 3)。PCR-反向点杂交法与Sanger测序法临床性能评价主要考虑其的阳性符合率、阴性符合率、总符合率、灵敏度、特异性、阳性预测值、阴性预测值,且用配对(χ2)与Kappa进行检验,计算方法如下:阳性符合率=[a/(a+c)]×100%;阴性符合率=[d/(b+d)]×100%;总符合率=[(a+d)/n]×100%;灵敏度=[a/(a+c)]×100%;特异性=[d/(b+d)]×100%;阳性预测值=[a/(a+b)]×100%;阴性预测值=[d/(c+d)]×100%。配对χ2检验说明的是检出率或阳性率的高低是否相同。Kappa检验说明的是PCR-反向点杂交法与Sanger测序法检测结果的一致性程度。
表 3.
Sanger测序法与PCR-反向点杂交法结果比较方法
| PCR-反向点杂交法 | 对照方法(Sanger测序) | 合计 | |
| 阳性 | 阴性 | ||
| 阳性 | a | b | a+b |
| 阴性 | c | d | c+d |
| 合计 | a+c | b+d | n(a+b+c+d) |
2. 结果
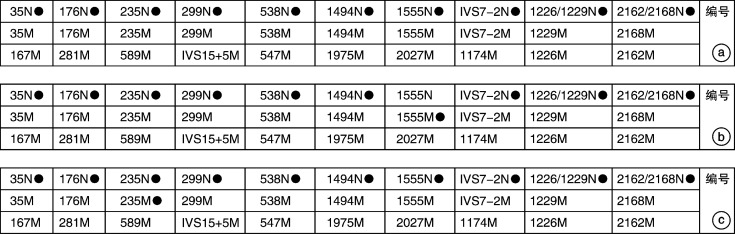
500例患者经病史采集和听力学等相关检查,均确诊为非综合征型感音神经性聋。采用PCR-反向点杂交法检测出147例野生型结果样本,81例纯合突变型样本,240例杂合突变型样本,32例复合杂合突变型样本。PCR-反向点杂交法结果示意见图 1。
图 1.
PCR-反向点杂交法结果示意图
1a:阴性结果(N/N);1b:单突变纯合子(1555M);1c:单突变杂合子(235M/N);“N”为野生型,“M”为突变型。1226M和1229M共用正常对照1226/1229N;2168M和2162M共用正常对照2162/2168N。
使用Sanger测序法检出147例阴性结果样本,81例纯合突变型样本,240例杂合突变型样本,32例复合杂合突变型样本。Sanger测序法与PCR-反向点杂交法检测20种突变位点结果汇总表见表 4,PCR-反向点杂交法覆盖的20个突变位点检测结果与Sanger测序法对比符合率为100%。
表 4.
Sanger测序法与PCR-反向点杂交法检测20种突变位点结果汇总表
| 突变类型 | PCR-反向点杂交法 | Sanger测序法 | 灵敏度 (阳性符合率%) |
特异性 (阴性符合率%) |
总符合率/% | Kappa | P |
| 阴性 | 147 | 147 | / | 100.0 | 100.0 | 1 | < 0.0001 |
| c.35delG | 16 | 16 | 100.0 | 100.0 | 100.0 | 1 | < 0.0001 |
| c.167delT | 12 | 12 | 100.0 | 100.0 | 100.0 | 1 | < 0.0001 |
| c.176_191del16bp | 2 | 2 | 100.0 | 100.0 | 100.0 | 1 | < 0.0001 |
| c.538 C > T | 19 | 19 | 100.0 | 100.0 | 100.0 | 1 | < 0.0001 |
| c.547 G > A | 21 | 21 | 100.0 | 100.0 | 100.0 | 1 | < 0.0001 |
| c.235delC | 73 | 73 | 100.0 | 100.0 | 100.0 | 1 | < 0.0001 |
| c.299_300delAT | 9 | 9 | 100.0 | 100.0 | 100.0 | 1 | < 0.0001 |
| IVS7-2A > G | 46 | 46 | 100.0 | 100.0 | 100.0 | 1 | < 0.0001 |
| c.2162 C > T | 19 | 19 | 100.0 | 100.0 | 100.0 | 1 | < 0.0001 |
| c.2168 A > G | 9 | 9 | 100.0 | 100.0 | 100.0 | 1 | < 0.0001 |
| c.1174 A > T | 13 | 13 | 100.0 | 100.0 | 100.0 | 1 | < 0.0001 |
| c.1226 G > A | 12 | 12 | 100.0 | 100.0 | 100.0 | 1 | < 0.0001 |
| c.1229 C > T | 16 | 16 | 100.0 | 100.0 | 100.0 | 1 | < 0.0001 |
| IVS15+5 G > A | 9 | 9 | 100.0 | 100.0 | 100.0 | 1 | < 0.0001 |
| c.1975 G > C | 15 | 15 | 100.0 | 100.0 | 100.0 | 1 | < 0.0001 |
| c.281C > T | 10 | 10 | 100.0 | 100.0 | 100.0 | 1 | < 0.0001 |
| c.589G > A | 15 | 15 | 100.0 | 100.0 | 100.0 | 1 | < 0.0001 |
| c.2027 T > A | 15 | 15 | 100.0 | 100.0 | 100.0 | 1 | < 0.0001 |
| m.1555 A > G | 37 | 37 | 100.0 | 100.0 | 100.0 | 1 | < 0.0001 |
| m.1494 C > T | 18 | 18 | 100.0 | 100.0 | 100% | 1 | < 0.0001 |
本研究中,GJB2 c.235delC和SLC26A4 IVS7-2A > G是最为常见的热点突变,其次是mtDNA m.1555 A > G(表 4)。统计结果显示,PCR-反向点杂交法检测20个耳聋基因突变位点的灵敏度(阳性符合率)、特异性(阴性符合率)、阳性预测值、阴性预测值及总符合率均为100%。与Sanger测序法比较的配对McNemars精确概率法值由于不存在不一致对数,故统计量缺失。Kappa值为1,95%CI为1.0000~1.0000,P < 0.0001。
3. 讨论
研究表明,耳聋高发致病基因及突变热点存在种族和人群差异〔3〕。流行病学研究结果显示,GJB2基因突变是遗传性聋最常见的原因,在非综合征性耳聋患者中已经发现超过350种GJB2突变类型,这些突变在不同种族人群中的发生频率和分布情况差异很大(http://www.hgmd.cf.ac.uk)。在欧洲、美国、澳大利亚和以色列,50%的耳聋由GJB2突变导致,在其他国家的耳聋人群中也有报道。其中35delG在北欧、南欧及美国的高加索人种中占所有GJB2突变的70%,上述人群35delG的突变携带率为1.3%~2.8%。而在中国人群中,GJB2 c.235delC、c.176-191del16和c.299-300delAT是常见的突变类型,其中c.235delC是携带频率最高的突变位点〔8-9〕。在中国人大前庭水管综合征患者中的检测发现,92.1%的患者存在SLC26A4基因的突变〔10〕。于晓宇等(2018)报道IVS7-2A > G突变是中国人大前庭水管综合征患者中的高发突变,约80%的大前庭水管综合征患者携带此突变。针对SLC26A4基因的IVS7-2A > G突变进行筛查,对于快速发现大前庭水管综合征患者具有重要意义。除了GJB2和SLC26A4基因,线粒体基因12S rRNA是中国耳聋患者最常见的致病基因之一。目前,已发现300多种致病mtDNA点突变和mtDNA重组突变,其中中国耳聋人群主要突变热点是A1555G和C1494T〔6〕。本研究中,GJB2 c.235delC和SLC26A4 IVS7-2A > G是研究人群中最为常见的热点突变,其次是mtDNA m.1555 A > G,与以往研究结果基本一致〔11-12〕。
在实施遗传性聋的整体预防和诊治时,应充分考虑高发致病基因及突变热点在种族和人群中的差异,采用高效、可靠并符合中国人群的耳聋遗传特征的检测技术。目前的耳聋基因检测方法包括检测点突变的Sanger测序、多重连接PCR、荧光PCR法、实时荧光PCR熔解曲线法、微阵列芯片法、飞行时间质谱(MALDI-TOF-MS)和导流杂交法等,以及基于二代测序的目标区域捕获和大规模平行测序基因panel〔13-14〕。这些方法各有优缺点,为早期耳聋基因的发现及鉴定做出了一定的贡献。本研究采用PCR-反向点杂交法,对遗传性聋4个相关基因的20种基因突变进行检测,并采用金标准Sanger测序法进行验证。研究结果显示PCR-反向点杂交法,与作为“金标准”的Sanger测序组对这20个突变位点的检测结果完全一致,诊断试剂的灵敏度、特异度、阳性预测值、阴性预测值、总符合率均为100%,Kappa值为1,95%CI为1.0000~1.0000,配对检验结果不存在配对不一致的例数,表明PCR-反向点杂交法在上述20个耳聋基因突变位点的检测临床有效。与金标准Sanger测序法相比,PCR-反向点杂交技术利用生物素标记的引物进行靶核酸的扩增,扩增产物与固定在尼龙膜上的探针进行杂交,一次杂交反应可以检测多种靶序列。该方法具有快速、简便、高灵敏度和特异性、低成本等特点。PCR-反向点杂交技术有望将来应用于遗传性聋基因检测的临床辅助检测。
References
- 1.Angeli S, Lin X, Liu X Z. Genetics of hearing and deafness. Anat Rec(Hoboken) 2012;295(11):1812–1829. doi: 10.1002/ar.22579. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Tsukada K, Nishio SY, Hattori M, et al. Ethnic-specific spectrum of GJB2 and SLC26A4 mutations: their origin and a literature review. Ann Otol Rhinol Laryngol. 2015;124(Suppl 1):61S–76S. doi: 10.1177/0003489415575060. [DOI] [PubMed] [Google Scholar]
- 3.刘 亚兰, 桑 树山, 刘 学忠. 遗传性聋基因诊断策略进展. 听力学及言语疾病杂志. 2018;26(6):666–672. doi: 10.3969/j.issn.1006-7299.2018.06.025. [DOI] [Google Scholar]
- 4.Dai P, Yu F, Han B, et al. GJB2 mutation spectrum in 2, 063 Chinese patients with nonsyndromic hearing impairment. J Transl Med. 2009;7:26–26. doi: 10.1186/1479-5876-7-26. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.Liu Y, Wang L, Feng Y, et al. A New Genetic Diagnostic for Enlarged Vestibular Aqueduct Based on Next-Generation Sequencing. PLoS One. 2016;11(12):e0168508. doi: 10.1371/journal.pone.0168508. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Shen Z, Zheng J, Chen B, et al. Frequency and spectrum of mitochondrial 12S rRNA variants in 440 Han Chinese hearing impairedpediatric subjects from two otology clinics. J Transl Med. 2011;9:4. doi: 10.1186/1479-5876-9-4. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Xia JH, Liu CY, Tang BS, et al. Mutations in the gene encoding gap junction protein beta-3 associated with autosomal dominant hearing impairment. Nat Genet. 1998;20(4):370–373. doi: 10.1038/3845. [DOI] [PubMed] [Google Scholar]
- 8.Yan D, Tekin D, Bademci G, et al. Spectrum of DNA variants for non-syndromic deafness in a large cohort from multiple continents. Hum Genet. 2016;135(8):953–961. doi: 10.1007/s00439-016-1697-z. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.李 海波, 李 琼, 李 红, et al. 非综合征性聋突变热点的流行病学分析. 临床耳鼻咽喉头颈外科杂志. 2012;26(13):589–594. [PubMed] [Google Scholar]
- 10.王 秋菊, 杜 婉. 大前庭水管综合征的诊断与遗传咨询. 继续教育园地. 2016;24(6):630–635. [Google Scholar]
- 11.Qing J, Zhou Y, Lai R, et al. Prevalence of mutations in GJB2, SLC26A4, and mtDNA in children with severe or profound sensorineural hearing loss in southwestern china. Genet Test Mol Biomarkers. 2015;19(1):52–58. doi: 10.1089/gtmb.2014.0241. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Liu Y, Hu C, Liu C, et al. A rapid improved multiplex ligation detection reaction method for the identification of gene mutations in hereditary hearing loss. PLoS One. 2019;14(4):e0215212. doi: 10.1371/journal.pone.0215212. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Wu H, Feng Y, Jiang L, et al. Application of a New Genetic Deafness Microarray for Detecting Mutations in the Deaf in China. PLoS One. 2016;11(3):e0151909. doi: 10.1371/journal.pone.0151909. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Rudman JR, Mei C, Bressler SE, et al. Precision medicine in hearing loss. J Genet Genomics. 2018;45(2):99–109. doi: 10.1016/j.jgg.2018.02.004. [DOI] [PubMed] [Google Scholar]