Abstract
目的
对3例综合征性聋患儿进行分子遗传学及临床特征分析,明确其致病基因及遗传特征。
方法
对3例患儿及其父母进行病历采集和整理,包括常规检查和听力评估及颞骨CT、头颅MR检查;通过外显子组测序(WES)筛查可能致病的基因变异,并使用Sanger测序对先证者及其父母进行候选阳性变异验证。
结果
3例患儿均为智力正常女性,例2患儿为散发,其余均有耳聋家族史,并符合常染色体显性遗传规律。3例患儿均为双侧极重度感音神经性听力障碍伴眼睛亮蓝色巩膜。其他表型包括眼距增宽(例1)、多发性色素异常沉着(例2)和毛发微黄(例2)、眼睑下垂(例3)。例3患儿影像学检查示双侧前庭扩大、内听道扩大,及双侧内耳畸形。例1患儿母亲仅有左侧轻度听力障碍;例3患儿母亲为双侧听力障碍,伴有单侧亮蓝色巩膜及毛发微黄。WES检出例1、例2、例3患儿分别有不同基因的杂合性变异:PAX3 c.811C>T、MITF c.632T>C,及SOX10 c.1359_1360 insGCCCCACA。例1和例3患儿检出变异均遗传自有听力障碍的母亲,例2患儿可能为自发变异。最终诊断例1患儿为Waardenburg综合征1型(WS1),例1患儿母亲、例2患儿、例3患儿及例3患儿母亲均为WS2。
结论
WS为综合征性聋,临床诊断所依据的主要特征包括常染色体显性遗传和巩膜色素异常。研究表明,同一基因变异导致的WS仍存在表型异质性,其中PAX3变异导致轻度的WS则很可能是外显不全所致,因此,WS的确诊有赖于基因检测结果。例1、例3患儿突变位点为首次在患者中发现并鉴定,扩展了WS的致病性变异谱。
Keywords: 综合征性聋, Waardenburg综合征, 分子遗传学, 临床特征
Abstract
Objective
To analyze the molecular genetics and clinical characteristics of 3 children with syndromic deafness were analyzed to clarify their causative genes and genetic characteristics.
Methods
The medical records of 3 children and their parents were collected and analyzed, including physical examination, hearing evaluation, temporal bone CT, and cranial MRI. Whole-exome sequencing(WES) was used to screen for pathogenic gene variants, and Sanger sequencing was used to verify the candidate positive variants in the probands and their parents.
Results
All 3 patients were female with normal intelligence. Patient 1 and 3 had a family history of deafness, which conformed to the pattern of autosomal dominant inheritance. All three patients had bilateral profound sensorineural hearing impairment with bright-blue sclera. Other phenotypes included hypertelorism(patient 1), multiple dyschromatosis(patient 2), and yellowish hair(patient 2), blepharoptosis(patient 3). Patient 3 had bilateral vestibular enlargement, internal auditory canal enlargement, and bilateral inner ear malformations. Mother of patient 1 had only left mild hearing impairment; mother of patient 3 had bilateral hearing impairment with unilateral bright-blue sclera and yellowish hair. WES detected heterozygous variants, PAX3 c.811C>T, MITF c.632T>C, and SOX10 c.1359_1360 insGCCCCACA, in patient 1, 2, and 3, respectively. The variants in patient 1 and 3 were inherited from their mothers who had hearing impairment, and MITFvariant in patient 2 may be a spontaneous variation. The final diagnoses were that patient 1 with Waardenburg syndrome type 1(WS1), and the mother of patient 1, patient 2, patient 3, and the mother of patient 3 with WS2.
Conclusion
WS is a syndromic deafness, and the main clinical features include autosomal dominant inheritance and scleral pigment abnormalities. However, the findings of this study show that there is still phenotypic heterogeneity in WS even caused by the same gene variant, so it depends on genetic tests to confirm the diagnosis; The gene variant of patient 1 and 2 was never been reported in other patients, which expands the pathogenic variant spectrum of WS.
Keywords: syndromic deafness, Waardenburg syndrome, molecular genetics, clinical features
Waardenburg综合征(Waardenburg syndrome,WS)是一种常染色体显性遗传的综合征型感音神经性听力障碍,由荷兰医师Waardenburg(1951)首次报道。除感音性聋外,WS患者的临床特征还伴有眼、皮肤及附件的色素异常,包括宽鼻根、内眦畸形、虹膜色素异常、眉毛内侧部分多毛症和额白发等多种异质性表型,因此,临床上曾将其命名为“耳聋-白发-眼病综合征”或“听力-色素综合征”。在全球范围内,WS的发病率约为1/42000,且其不完全外显率约为20%[1],占所有先天性听力障碍案例的2%~5%,以及耳聋患者的0.9%~2.8%[2-3]。WS呈较高的遗传异质性和表型异质性,根据其临床特征主要分为4型:WS1~4,其中以WS1和WS2相对常见。目前已证实有7个WS致病基因:PAX3、MITF、SOX10、SNAI2、ENDRB、EDN3和KITLG[3-4],其中PAX3变异多见于WS1和WS3患者[5],MITF和SOX10变异则可见于WS2和WS4患者[6-7]。
本研究通过外显子组测序(whole-exome sequencing,WES)对3个临床疑似WS患儿进行基因检测,分析WS的遗传及表型异质性,明确了其病因,并鉴定了2个在WS患者中首次发现的致病性变异位点。
1. 资料与方法
1.1. 临床资料
纳入2021年3月—2022年3月在我院耳鼻咽喉科门诊、广西都安聋校义诊,及我院行人工耳蜗手术后的患儿。本研究经广西壮族自治区人民医院伦理委员会审批。
1.2. 方法
采集先证者和家系成员的临床资料,进行耳科、眼科、毛发、皮肤、四肢关节、其他各个系统等全面体格检查及智力评估。测量先证者内眦距离(A)、瞳孔距离(B)及外眦距离(C),计算W值。X=[2A-(0.2119C+3.909)]/C,Y=[2A-(0.2497B+3.909)]/B,W=X+Y+(A/B)(内眦异位,W指数>1.95)。临床听力学检测包括行为测听、声导抗、畸变产物耳声发射(DPOAE)、听性脑干反应(ABR),听觉稳态反应(ASSR)等。同时行颞骨CT、头颅MRI检查和内耳水成像等。
1.3. 基因检测
取得患儿家属的同意并签署知情同意书后,对3例患儿进行WES检测,其中,例1患儿为先证者及其父母3人的并行WES检测,例2和例3患儿为单人WES检测。用于验证候选阳性基因变异的Sanger测序均在先证者及其父母中进行。抽取患儿及其父母静脉血2~3 mL,在Chigene(智因东方转化医学研究中心有限公司,北京)进行trio-WES。WES采用The xGen Exome Research Panel v1.0(IDT公司,美国)外显子组捕获芯片用于全外显子文库构建。使用NovaSeq6000系列测序仪(Illumina,美国)进行高通量测序。测序流程及数据生成、清洗和质检使用厂商提供的系统,按建议的标准程序完成。测序平均深度为100X,外显子组DNA序列覆盖度不低于99%。WES数据经智因全谱遗传病精准诊断云平台系统进行自动化的生物信息学分析,生成≤50 bp的插入/缺失(indel)和点突变数据,并利用Chigene自研算法提示多个连续外显子的拷贝数变异。通过云平台整合的基因变异数据库,包括dbSNP、ClinVar、HGMD pro、gnomAD以及OMIM数据库,对所有检出的基因变异进行次等位基因频率(minor allele frequency,MAF)、已报道案例致病性和文献以及变异基因相关疾病的注释。基因变异的致病性根据美国医学遗传学与基因组学学会(American College of Medical Genetics,ACMG)临床实践指南进行分级,分为致病性(pathogenic)、可能致病(likely pathogenic)、致病性不明确(uncertain significance)、可能良性(likely benign)和良性(benign)。候选的基因变异在患儿及其父母3人中进行Sanger测序验证;WES提示外显子拷贝数变异的基因,通过逆转录cDNA的实时荧光定量PCR (RT-PCR)进行外显子拷贝数的检测验证。本研究中提及基因的参考转录本为PAX3:NM_181458,MITF:NM_000248和SOX10:NM_006941。
2. 结果
3例患儿流行病学及临床特征、基因检测结果及诊断见表 1。3个患病家系的家系图及Sanger测序验证结果及临床表现分别见图 1~6。其中,患儿2母亲因样本未能采集到,未进行Sanger测序。其中,例1患儿有眼距增宽:瞳距47 mm,内眦间距36 mm,外眦间距74 mm,W指数=2.75(>1.95),这符合WS1的临床诊断标准。然而,该患儿母亲无眼距异常及其他头面部畸形,且仅有左侧轻度听力下降,其临床特征仅表现轻度WS2。WES检出PAX3c.811C>T(p.Arg271Cys)为实验室已报道致病性变异,根据ACMG临床实践指南,该变异为致病性:PS1+ PM1+ PM2+ PP1+ PP3;PS1:该变异与已确定的致病变异有着相同的氨基酸改变;PM1:错义变异位于已确定的致病变异左右的错义变异且位于致病热点突变区域;PM2:所有正常人群数据库频率为0;PP1:突变与疾病在家系中共分离;PP3:多种统计方法预测出变异对基因(基因产物)有影响。例2患儿检测出MITFc.632T>C(p.Leu211Pro),患儿父亲自诉母亲表型均正常,考虑该突变可能为自发变异,根据ACMG指南分级为可能致病:PS4_Supporting+PM1+PM2+PP3+PP4;PS4_Supporting:显性遗传中2个先证者携带此变异,且符合PM2;PM1:该错义变异位于致病热点区(突变左右10 bp范围内存在3个以上有害突变);PM2:所有正常人群数据库频率为0;PP3:多种统计方法预测出变异对基因(基因产物)有影响;PP4:变异携带者的表型高度符合某种单基因遗传疾病。例3患儿检测出SOX10c.1359_1360ins GCCCCACA为移码突变,导致p.His454Alafs在数据库中报道为致病性或可能致病性变异,但人群中未发现,根据ACMG分级为致病性:PVS1_Strong+PM2+PP1+PP4;PVS1_Strong:丧失功能(loss of function)变异为疾病致病机制,变异发生在3’末端,预计不会引起转录子降解,该区域对蛋白功能有重要影响;PM2:所有正常人群数据库频率为0;PP1:突变与疾病在家系中共分离;PP4:变异携带者的表型、家族史高度符合某种单基因遗传疾病。
表 1.
3例广西WS患儿临床特征和分子诊断结果
| 项目 | 例1患儿 | 例2患儿 | 例3患儿 |
| 性别 | 女 | 女 | 女 |
| 诊断年龄 | 1岁4个月 | 10岁10个月 | 2岁8个月 |
| 民族 | 汉族 | 瑶族 | 汉族 |
| 体检 | 左侧亮蓝色巩膜,眼距增宽 | 双侧亮蓝色巩膜,面部雀斑,毛发微黄 | 右侧亮蓝色巩膜,左上睑下垂 |
| ABR/dB nHL | 双侧>95 | 双侧>98 | 双侧>95 |
| DPOAE | 未引出 | 未引出 | 未引出 |
| ASSR/dB nHL | 双侧均>95 | 双侧均>100 | 左>97,右>95 |
| 影像学检查 | 未见明显异常 | 未见明显异常 | 颞骨CT提示双侧前庭扩大,内听道扩大,头颅MRI提示双侧内耳畸形 |
| 家族史 | 母亲左侧轻度听力障碍 | 无 | 母亲双侧极重度感音神经性聋,右侧亮蓝色巩膜,发色微黄 |
| 报告基因 | PAX3 | MITF | SOX10 |
| 核酸改变 | c.811C>T | c.632T>C | c.1359_1360ins GCCCCACA |
| 氨基酸改变 | p.Arg271Cys | p.Leu211Pro | p.His454Alafs |
| 最终诊断 | WS1 | WS2 | WS2 |
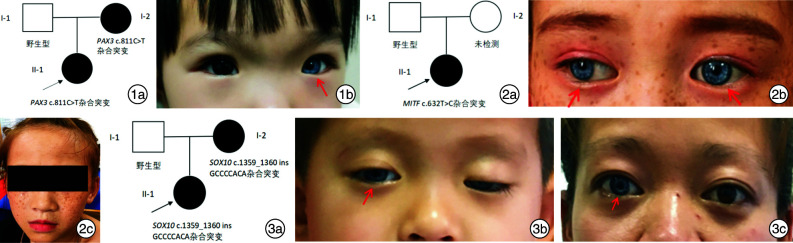
图 1.
例1患儿家系图和临床表现 1a: 家系图; 1b: 先证者1左眼呈现蓝巩膜(红色箭头指示); 图 2 例2患儿家系图及临床表现 2a: 家系图; 2b: 先证者2双眼呈现蓝色巩膜(红色箭头指示); 2c: 先证者2满脸雀斑; 图 3 例3患儿家系图及临床表现 3a: 家系图; 3b: 先证者3右眼呈现蓝色巩膜(红色箭头所示), 左上睑下垂; 3c: 先证者3母亲右眼蓝色巩膜(红色箭头所示)。
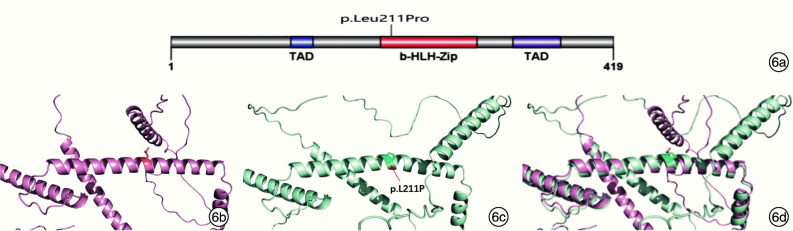
图 6.
MITF蛋白功能结构域及变异体的位置和蛋白结构预测
6a:变异体在结构域的位置;6b:野生型蛋白结构;6c:MITFp.L211P突变的蛋白结构;6d:野生型与突变型蛋白结构组合。
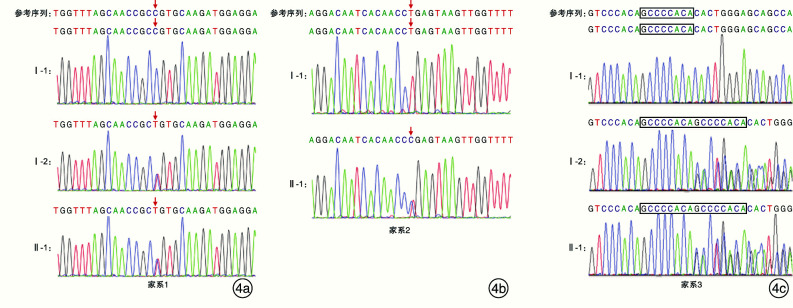
图 4.
Sanger测序验证3例患儿及其父母的检出变异
4a:家系1:PAX3c.811C>T;4b:家系2:MITFc.632T>C;4c:家系3:SOX10c.1359_1360insGCCCCACA
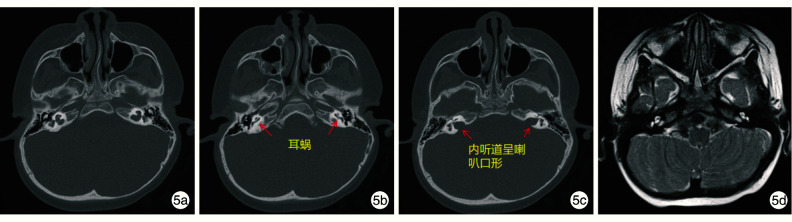
图 5.
例3患儿颞骨CT、MRI
5a:双侧前庭扩大;5b:双侧内耳畸形,耳蜗仅1.5圈;5c:双侧内听道扩大,呈喇叭口形(红箭头所示);5d:双侧耳蜗发育畸形
3. 讨论
WS是由于神经嵴细胞(neural crest cells,NCC)发育异常导致的一种遗传性疾病。人类黑色素细胞、额骨、肢体肌肉和壁内神经节均来源于胚胎期NCC。耳蜗中的黑色素细胞作为耳蜗血管纹中间层的中间细胞,其钾离子通道是耳蜗内电位产生的主要能源,对维持耳蜗内淋巴液K+浓度起着重要的作用,这解释了NCC异常发育所导致的一系列以黑色素细胞功能异常和感音性听力障碍为主要的WS临床特征[8]。四型WS患者主要临床特征是WS1为先天性聋、色素异常和眼距增宽;WS2表型与WS1类似,但WS2患者没有眼距增宽畸形;WS3除包含以上所有表型外,少数患者有骨骼异常,包括肢体肌肉发育不良、骨骼发育不良、肘关节或指关节挛缩;WS4与WS2表型高度重叠,但WS4患者常有先天性巨结肠和胃肠道闭锁。WS主要诊断标准包括:①先天性感音神经性聋;②虹膜色素异色;③头发色素减少;④内眦异位(W指数>1.95即内眦增宽,常见于WS1);⑤常染色体显性遗传。次要诊断标准包括:①遗传性白斑病;②一字眉,眉毛异常浓密;③鼻根宽大;④鼻翼发育不良;⑤早白发(30岁之前头发变白)。WS的诊断需同时满足2个主要诊断标准,或1个主要诊断标准加2个次要诊断标准[9-10]。本研究中,3例患儿均有先天性感音神经性聋和巩膜色素异常的临床表现,符合2个WS的主要诊断。其中,例1患儿有内眦/眼距增宽,符合WS1,但其母亲无头面部及眼部异常,仅有单侧轻度的听力障碍,这可能是外显不全表征,或是该母亲为PAX3变异的体细胞嵌合体,PAX3出现体细胞腺嵌合体这种情况首次报道是在2018年Somashekar等[11]研究的WS家系中患儿遗传自临床无症状的父亲。其余例2、例3患儿及例3患儿的母亲均符合WS2临床特征。
PAX3基因位于2号染色体上,编码转录因子PAX3,其特征是具有高度保守性[12]。PAX3编码的转录因子由N端DNA结合域(包含配对结构域PD和同源结构域HD)、C端转录激活域和八肽基序组成。目前已经报道了约95%的PAX3变异均位于编码配对结构域和同源结构域的2号和6号外显子上,其中2号外显子的突变发生率最高,其次分别是6号和5号外显子[13]。同源结构域HD位于氨基酸残基序列220~277,包含1个DNA结合域,可识别结合各自的DNA序列,与配对结构域PD协同激活调控下游靶基因转录[12]。结构域HD突变通过破坏结构域HD的构象,进而影响结构域折叠的稳定性,导致其PAX3与DNA结合障碍[14-15]。PAX3还可通过与其他蛋白相互作用,完成对下游基因转录的抑制或激活,进而影响下游基因的表达,从而调控黑色素细胞的生长、发育与分化。例如PAX3可与MITF转录起始点上游的顺式作用元件结合,激活下游的MITF转录,也可与SOX10协同上调MITF的表达。SOX10通过与MITF启动子的高度保守序列结合,上调MITF的转录活性[16]。由此可见,PAX3具有促进黑色素细胞发育信号通路的上游激活作用。Song等[2]的研究发现,WS患者的听力损失患病率因基因型而异,其中WS1患者PAX3变异的检出率为52.3%,这进一步表明了PAX3黑色素细胞发育信号通路中的重要调控作用。
MITF基因定位于3号染色体短臂,编码1个小眼畸形相关转录因子(microphthalmia associated transcription factor),是参与多种神经嵴细胞的生长发育,特别是在人体黑色素细胞的发育、增殖、分化及生存过程中,起到关键作用的1个转录因子[17-18]。MITF蛋白由419个氨基酸组成,是一种含有螺旋-环-螺旋碱性亮氨酸拉链结构(bHLH-Zip)的转录因子,该结构域由44个氨基酸组成,具有高度的保守性和特异性DNA结合能力,其结构的完整性对MITF蛋白翻译的准确性发挥着至关重要的作用。Brownstein等[19]鉴定了一个WS家系患者中NM_198159.3:MITF c.935T>C(p.Leu312Pro)杂合变异,该变异位于MITF碱性螺旋环-螺旋结构域中的一个完全保守的残基。这个位置的脯氨酸会破坏螺旋,阻止DNA结合,也可能阻止二聚化。因此,笔者推测例2患儿的相同位点的变异也是脯氨酸破坏螺旋,阻止了DNA结合,进而影响了蛋白结构改变。利用AlphaFold对野生型和突变型蛋白结构进行预测,发现脯氨酸破坏蛋白质螺旋结构(图 6)。据估计,10%~20%的WS2病例是由MITF变异引起[3]。MITF在黑色素细胞的分化中起关键作用。Tachibana等[20]研究发现,MITF能够反式激活酪氨酸酶基因,生成的关键酶如酪氨酸酶和酪氨酸酶相关蛋白在黑色素细胞分化过程中起关键作用;此外,MITF异位表达会使黑色素细胞缺乏,进而导致皮肤、头发和眼睛的色素沉着,以及耳蜗中的听觉功能减退。因此,WS2的色素沉着和听力损失极可能是MITF突变引起的黑色素细胞分化异常的结果。
SOX10基因定位在染色体22q13.1上,有5个外显子,但1、2号外显子不编码蛋白。SOX10蛋白为包含466个氨基酸残基的转录因子,是DNA结合蛋白中具有高活动组分(high mobility group,HMG)超级家族的成员之一[21]。SOX10主要是通过HMG结构域识别并结合靶基因启动子DNA,参与胚胎发育调控和外周神经系统的发育。当SOX10突变发生功能缺陷时,将导致靶基因表达异常使DNA的构象发生改变,从而导致NCC源细胞(血管纹黑色素细胞等)发育异常进而导致耳聋[22]。据统计,在WS2病例中约有15%是由SOX10缺陷造成[23]。有研究者对WS病例进行影像学、统计学分析,发现SOX10变异致病的WS患者发生内耳畸形的概率高于其他基因突变的WS患者,这类患者更容易发生前庭及半规管畸形[23-24]。这与例3患儿检出的SOX10变异与影像学前庭扩大、内耳畸形的表型关联相符,同时进一步证实SOX10对内耳发育的重要性。例3患儿通过右侧人工耳蜗植入术,以及术后2个月的康复训练,现对唤其姓名能做出反应。
本研究展示了WS的遗传异质性和表型异质性,为临床医师尽可能早期诊断WS提供了案例依据。此外,我们的研究还扩展了PAX3、SOX10基因的致病性变异谱。
Funding Statement
国家自然科学基金资助项目(No:82060190);广西科技厅重点研发项目(No:桂科AB17292089);广西医疗卫生适宜技术开发与推广应用项目(No:S2017078)
Footnotes
利益冲突 所有作者均声明不存在利益冲突
References
- 1.Pingault V, Ente D, Dastot-Le MF, et al. Review and update of mutations causing Waardenburg syndrome. Hum Mutat. 2010;31(4):391–406. doi: 10.1002/humu.21211. [DOI] [PubMed] [Google Scholar]
- 2.Song J, Feng Y, Acke FR, et al. Hearing loss in Waardenburg syndrome: a systematic review. Clin Genet. 2016;89(4):416–425. doi: 10.1111/cge.12631. [DOI] [PubMed] [Google Scholar]
- 3.Huang S, Song J, He C, et al. Genetic insights, disease mechanisms, and biological therapeutics for Waardenburg syndrome. Gene Ther. 2022;29(9):479–497. doi: 10.1038/s41434-021-00240-2. [DOI] [PubMed] [Google Scholar]
- 4.李 霞, 赵 声波, 毕 先云, et al. PAX3基因新突变致Ⅰ型Waardenburg综合征家系基因型与表型特征分析. 临床耳鼻咽喉头颈外科杂志. 2021;35(7):621–626. doi: 10.13201/j.issn.2096-7993.2021.07.010. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.Somashekar PH, Upadhyai P, Narayanan DL, et al. Phenotypic diversity and genetic complexity of PAX3-related Waardenburg syndrome. Am J Med Genet A. 2020;182(12):2951–2958. doi: 10.1002/ajmg.a.61893. [DOI] [PubMed] [Google Scholar]
- 6.Wang J, Lu Y, Yan X, et al. Identification of novel MITF mutations in Chinese families with Waardenburg syndrome type Ⅱ. Mol Genet Genom Med. 2021;9(9):e1770. doi: 10.1002/mgg3.1770. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Pingault V, Zerad L, Bertani-Torres W, et al. SOX10:20 years of phenotypic plurality and current understanding of its developmental function. J Med Genet. 2022;59(2):105–114. doi: 10.1136/jmedgenet-2021-108105. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Fleck K, Erhardt G, Luhken G. From single nucleotide substitutions up to chromosomal deletions: genetic pause of leucism-associated disorders in animals. Berl Munch Tierarztl Wochenschr. 2016;129(7/8):269–281. [PubMed] [Google Scholar]
- 9.马 静, 明 澄, 林 垦, et al. Ⅱ型Waardenburg综合征患儿二例基因诊断分析. 中华耳鼻咽喉头颈外科杂志. 2021;56(1):47–54. doi: 10.3760/cma.j.cn115330-20200121-00041. [DOI] [PubMed] [Google Scholar]
- 10.徐 彬, 戴 继任, 毕 静, et al. 新一代测序技术在3例Waardenburg综合征患儿中的应用. 临床耳鼻咽喉头颈外科杂志. 2021;35(10):910–913. doi: 10.13201/j.issn.2096-7993.2021.10.010. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Somashekar PH, Girisha KM, Nampoothiri S, et al. Locus and allelic heterogeneity and phenotypic variability in Waardenburg syndrome. Clin Genet. 2019;95(3):398–402. doi: 10.1111/cge.13468. [DOI] [PubMed] [Google Scholar]
- 12.Boudjadi S, Chatterjee B, Sun W, et al. The expression and function of PAX3 in development and disease. Gene. 2018;666:145–157. doi: 10.1016/j.gene.2018.04.087. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Pingault V, Ente D, Dastot-Le MF, et al. Review and update of mutations causing Waardenburg syndrome. Hum Mutat. 2010;31(4):391–406. doi: 10.1002/humu.21211. [DOI] [PubMed] [Google Scholar]
- 14.Birrane G, Soni A, Ladias JA. Structural basis for DNA recognition by the human PAX3 homeodomain. Biochemistry. 2009;48(6):1148–1155. doi: 10.1021/bi802052y. [DOI] [PubMed] [Google Scholar]
- 15.Chi YI. Homeodomain revisited: a lesson from disease-causing mutations. Hum Genet. 2005;116(6):433–444. doi: 10.1007/s00439-004-1252-1. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Lang D, Epstein JA. Sox10 and Pax3 physically interact to mediate activation of a conserved c-RET enhancer. Hum Mol Genet. 2003;12(8):937–945. doi: 10.1093/hmg/ddg107. [DOI] [PubMed] [Google Scholar]
- 17.Alehabib E, Alinaghi S, Pourfatemi F, et al. Incomplete penetrance of MITF gene c.943C > T mutation in an extended family with Waardenburg syndrome type Ⅱ. Int J Pediatr Otorhinolaryngol. 2020;135:110014. doi: 10.1016/j.ijporl.2020.110014. [DOI] [PubMed] [Google Scholar]
- 18.Grill C, Bergsteinsdottir K, Ogmundsdottir MH, et al. MITF mutations associated with pigment deficiency syndromes and melanoma have different effects on protein function. Hum Mol Genet. 2013;22(21):4357–4367. doi: 10.1093/hmg/ddt285. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Brownstein Z, Gulsuner S, Walsh T, et al. Spectrum of genes for inherited hearing loss in the Israeli Jewish population, including the novel human deafness gene ATOH1. Clin Genet. 2020;98(4):353–364. doi: 10.1111/cge.13817. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Tachibana M, Takeda K, Nobukuni Y, et al. Ectopic expression of MITF, a gene for Waardenburg syndrome type 2, converts fibroblasts to cells with melanocyte characteristics. Nat Genet. 1996;14(1):50–54. doi: 10.1038/ng0996-50. [DOI] [PubMed] [Google Scholar]
- 21.Thongpradit S, Jinawath N, Javed A, et al. Novel SOX10 Mutations in Waardenburg Syndrome: Functional Characterization and Genotype-Phenotype Analysis. Front Genet. 2020;11:589784. doi: 10.3389/fgene.2020.589784. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22.Chaoui A, Watanabe Y, Touraine R, et al. Identification and functional analysis of SOX10 missense mutations in different subtypes of Waardenburg syndrome. Hum Mutat. 2011;32(12):1436–1449. doi: 10.1002/humu.21583. [DOI] [PubMed] [Google Scholar]
- 23.Elmaleh-Berges M, Baumann C, Noel-Petroff N, et al. Spectrum of temporal bone abnormalities in patients with Waardenburg syndrome and SOX10 mutations. AJNR Am J Neuroradiol. 2013;34(6):1257–1263. doi: 10.3174/ajnr.A3367. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.徐 广雨, 郝 青青, 钟 玲玲, et al. SOX10基因突变对Waardenburg综合征患者内耳发育的影响. 中华耳鼻咽喉头颈外科杂志. 2016;51(11):832–837. doi: 10.3760/cma.j.issn.1673-0860.2016.11.006. [DOI] [PubMed] [Google Scholar]