Abstract
分子伴侣和辅助伴侣分子能够促进新合成多肽的组装以及帮助未折叠或错误折叠的蛋白质重新折叠形成正确折叠的蛋白质,从而维持细胞内蛋白系统的稳态。作为热休克蛋白(HSP)70的辅助伴侣分子,HSP40(DNAJ)蛋白家族是目前已知的最大分子伴侣家族,能够通过J结构域与HSP70结合,从而帮助蛋白质折叠。近年研究发现,DNAJ家族蛋白与阿尔茨海默病、帕金森病、亨廷顿病、脊髓小脑性共济失调、进行性神经性腓骨肌萎缩症、脊髓性肌萎缩、远端型遗传性运动神经病变、肢带型肌营养不良、神经元蜡样质脂褐质沉积症和特发性震颤等神经退行性疾病的发生和发展有密切关系,如DNAJA1可有效降解亨廷顿蛋白聚集体;DNAJB1可降解蛋白聚集体ataxin-3;DNAJB2能够抑制亨廷顿蛋白聚集体的形成;DNAJB6能够抑制Aβ 42和α-突触核蛋白的聚集;DNAJC5可以促进TDP-43、τ蛋白和α-突触核蛋白释放到细胞外空间;与特发性震颤相关的DNAJC13的突变可能阻碍核内体蛋白运输。本文就DNAJ蛋白家族在神经退行性疾病中的作用机制进行综述。
Abstract
Molecular chaperones and co-chaperones facilitate the assembly of newly synthesized polypeptides and refolding of unfolded or misfolded proteins, thereby maintaining protein homeostasis in cells. As co-chaperones of the master chaperone heat shock protein (HSP) 70, the HSP40 (DNAJ) proteins are largest chaperone family in eukaryotic cells. They contain a characteristic J-domain which mediates interaction with HSP70, thereby helping protein folding. It is well perceived that protein homeostasis is vital for neuronal health. DNAJ family proteins have been linked to the occurrence and progression of neurodegenerative diseases such as Alzheimer’s disease, Parkinson’s disease, Huntington’s disease, spinocerebellar ataxia, Charcot-Marie-Tooth disease, spinal muscular atrophy, distal hereditary motor neuropathy, limb-girdle type muscular dystrophy, neuronal ceroid lipofuscinosis and essential tremor in recent studies. DNAJA1 effectively degrades huntington aggregates; DNAJB1 can degrade protein aggregates ataxin-3; DNAJB2 can inhibit the formation of huntington aggregates; DNAJB6 can inhibit the aggregation of Aβ 42 and α-synuclein; DNAJC5 can promote the release of TDP-43, τ protein, and α-synuclein into the extracellular space. Mutations in the essential tremor-associated DNAJC13 gene can prevent endosome protein trafficking. This article reviews the mechanism of DNAJ protein family in neurodegenerative diseases.
Keywords: Molecular chaperones, HSP40, DNAJ, Neurodegenerative diseases, Protein homeostasis, Review
脊髓小脑性共济失调(spinocerebellar ataxia,SCA);β淀粉样蛋白(amyloid β-protein,Aβ);突触核蛋白(synuclein,syn);多聚谷氨酰胺(polyglutamine,polyQ);热休克蛋白(heat shock protein,HSP);富含甘氨酸/苯丙氨酸结构域(glycine/phenylalanine rich,G/F);富含半胱氨酸(Cys-rich);腺苷三磷酸(adenosine triphosphate,ATP);进行性神经性腓骨肌萎缩症(Charcot-Marie-Tooth disease,CMT);串联泛素结合模体(ubiquitin-interaction motif,UIM);酪蛋白激酶(casein kinase,CK);脊髓性肌萎缩(spinal muscular atrophy,SMA);远端型遗传性运动神经病变(distal hereditary motor neuropathy,dHMN);D1型肢带型肌营养不良(limb-girdle type muscular dystrophy type D1,LGMDD1);神经元蜡样质脂褐质沉积症(neuronal ceroid lipofuscinosis,NCL);
神经退行性疾病是指神经元的结构和功能不断受到破坏从而导致神经系统产生功能性障碍的一类疾病 [1] 。常见的神经退行性疾病包括亨廷顿病、SCA、阿尔茨海默病、特发性震颤和帕金森病等 [1] 。目前,这些神经退行性疾病的病因并不明确,且几乎无有效的治疗方式,严重影响了老年人的生活质量 [ 1- 2] 。未来二十年,神经退行性疾病将成为仅次于心血管疾病的第二大常见死因 [2] 。
特定脑区的蛋白质聚集体是许多神经退行性疾病的病理标志,通常由错误折叠的相关蛋白组成 [ 3- 5] 。如阿尔茨海默病的特征是细胞外Aβ沉积形成的老年斑和细胞内高磷酸化微管相关蛋白τ蛋白聚集形成的神经原纤维缠结;帕金森病与α-syn在细胞内的沉积有关;亨廷顿病中存在polyQ形式的细胞内聚集体 [ 3- 5] 。在神经系统中,蛋白质质量控制系统对于抵御这些蛋白聚集体对细胞造成的威胁至关重要 [6] 。蛋白质质量控制系统包括泛素-蛋白酶体系、自噬-溶酶体途径和分子伴侣体系 [6] 。分子伴侣体系存在于各类生物体中,其主要功能之一是将错误折叠的蛋白质重折叠形成正常结构 [7] 。异常蛋白质若不能通过分子伴侣重折叠形成正常折叠的蛋白质,将通过泛素-蛋白酶体系或自噬-溶酶体途径被降解,以维持蛋白质系统的稳态 [6] 。
近年来,DNAJ家族基因或蛋白已被广泛证明与神经退行性疾病相关。DNAJ家族基因通过影响突触传递、内吞体运输和线粒体系统等调节蛋白质稳态,从而直接或间接地影响神经发育与神经退行性疾病 [ 5- 6, 8] 。本文主要就DNAJ调控神经退行性疾病进行归纳和分析,进一步阐述了DNAJ的作用机制,为今后探究DNAJ在神经系统疾病中的作用提供思路和参考。
HSP分类及分子伴侣模型
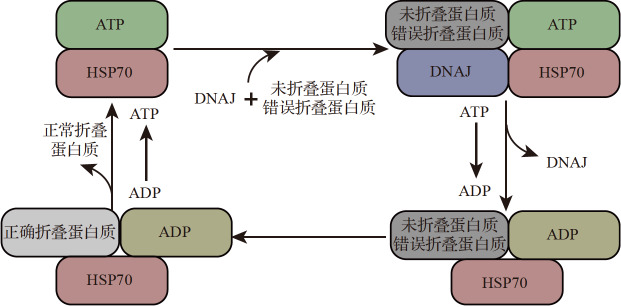
HSP是一类ATP依赖型分子伴侣 [9] 。HSP按相对分子质量分为HSP110、HSP90、HSP70、HSP40(又称为DNAJ)、HSP60和小HSP等,其中DNAJ蛋白由70个氨基酸组成,拥有高度保守的J结构域 [10] 。根据其蛋白结构域,DNAJ家族蛋白可分为三类:DNAJA(Ⅰ型)、DNAJB(Ⅱ型)和DNAJC(Ⅲ型) [8] 。DNAJA和DNAJB蛋白氨基末端含有J结构域 [8] 。DNAJA还包括G/F结构域和Cys-rich结构域以及羧基末端结合域;DNAJB无Cys-rich结构域和羧基末端结合域,G/F结构域较DNAJA更大;DNAJC无G/F结构域,且J结构域的分布无规律 [11] 。DNAJ通过J结构域与HSP70结合,增强HSP70的ATP酶活性,促进HSP70与底物蛋白的结合稳定性。之后DNAJ被释放,在ATP转变成ADP后,底物从HSP70中释放并成为正确折叠的蛋白质( 图1) [5] 。
图 1.
HSP70-DNAJ复合物辅助蛋白质折叠的分子伴侣模型
DNAJ家族蛋白与未折叠蛋白质或错误折叠蛋白质(底物)瞬时结合,将底物传递给HSP70. 通过J结构域的介导,DNAJ与HSP70结合并促进其ATP酶活性,从而使得HSP70与底物进行稳定相互作用. 核苷酸交换后,底物从HSP70中释放出来成正确折叠的蛋白质. HSP:热休克蛋白;ATP:腺苷三磷酸;ADP:腺苷二磷酸.
DNAJA1与亨廷顿病
亨廷顿病是一种常染色体显性遗传疾病,通常在成年期发病。该疾病是由 HTT基因中CAG核苷酸异常重复引起的。在突变携带者中, HTT基因序列编码产生含有polyQ的蛋白,这些蛋白在细胞内异常聚集,导致神经元功能障碍甚至死亡 [12] 。在表达polyQ74亨廷顿蛋白的HEK293细胞中敲除 DNAJA1基因,可导致polyQ74亨廷顿蛋白聚集减少至1/4 [13] 。这可能是由于DNAJA1能与细胞内其他DNAJ蛋白形成异源二聚体,从而产生显性负性作用 [13] 。因此, DNAJA1敲除可能有助于缓解亨廷顿病中因ployQ聚集导致的神经元功能障碍相关症状。
DNAJB1与SCA3
SCA3是一种神经系统常染色体显性遗传疾病,由ataxin-3蛋白中异常扩增的polyQ重复序列导致。polyQ重复次数与疾病的严重程度呈正相关,与发病年龄呈负相关 [14] 。在SCA3患者脑组织标本中发现,DNAJB1存在于polyQ聚集体中;而DNAJB1过表达会降低polyQ蛋白的毒性 [15] 。在SCA3中,DNAJB1被募集到由polyQ蛋白ataxin-3形成的蛋白质聚集体上,进而降解蛋白质聚集体 [4] 。在源自SCA3患者的多能干细胞特异性诱导的神经元中,上调miR-370和miR-543水平可降低DNAJB1表达;抑制miR-370和miR-543可增加DNAJB1表达,促进DNAJB1对polyQ的降解,进而缓解SCA3病情 [4] 。通过靶向miR-370和miR-543来上调DNAJB1表达可能成为治疗SCA3的新方法。
DNAJB2与神经退行性疾病
DNAJB2与CMT
CMT是一种遗传性运动感觉神经病变,以四肢远端进行性肌无力和萎缩、感觉障碍为特征,具有明显的遗传异质性 [16] 。在CMT患者中发现DNAJB2的纯合错义突变c.14A>G,该突变发生于DNAJB2的J结构域中 [16] 。最近一项研究发现DNAJB2的移码突变与CMT有关 [17] 。CMT患者中DNAJB2的突变c.145delG从Val49开始改变其氨基酸序列,并在第25个氨基酸后提前终止;淋巴细胞中DNAJB2 mRNA水平显著降低,蛋白质印迹法显示其皮肤内的TDP-43积累,说明患者体内DNAJB2的缺失显著影响了有毒蛋白质聚集物的去除 [17] 。
DNAJB2与亨廷顿病
将DNAJB2a的转基因小鼠与亨廷顿病小鼠模型R6/2杂交发现,DNAJB2a过表达显著降低亨廷顿蛋白的聚集并增强polyQ蛋白溶解度 [18] 。DNAJB2b与HSP90之间具有直接相互作用,其J结构域和UIM能够与泛素化的底物蛋白结合,参与蛋白的降解 [ 19- 20] 。CK2是一种持续激活型的蛋白激酶,CK2可导致DNAJB2的第二个UIM位点(Ser247和Ser250)磷酸化 [21] 。该磷酸化位点突变、加入CK2特异性抑制剂可降低DNAJB2与泛素化的蛋白结合能力及其分子伴侣活性 [21] 。这表明CK2抑制剂能释放DNAJB2的神经保护潜力,未来可能成为神经退行性疾病的治疗策略之一 [21] 。
DNAJB2与SMA
SMA是一种由脊髓前角及延髓运动神经元变性导致的神经疾病,其临床表现主要为肌肉萎缩、运动功能严重受限等,常发生于儿童期,是造成婴儿死亡的最常见遗传疾病 [22] 。Sanchez等 [23] 对SMA患者的标本进行全基因组关联研究后发现,DNAJB2的前四个外显子(包括整个J结构域)的缺失与SMA的症状存在显著关联。将该突变型质粒表达于HEK293细胞中,发现该突变能够导致DNAJB2b表达降低,并且促进自噬;在突变型DNAJB2b过表达细胞的细胞裂解物中观察到脑源性神经营养因子和τ蛋白水平显著降低,说明该突变是导致SMA原因之一 [23] 。dHMN是SMA的一种,主要表现为远端肌肉进行性萎缩和无力、电生理异常等。DNAJB2经过可变剪切后会形成两种亚型蛋白:相对分子质量为36000的DNAJB2a和42000的DNAJB2b [24] 。研究发现,DNAJB2的可变剪接导致dHMN。在dHMN患者中发现了DNAJB2纯合剪接位点突变(c.352+1G>A),该突变导致5号内含子部分或全部保留,下调DNAJB2蛋白表达 [25] 。Liu等 [26] 报告了一例因 DNAJB2基因c.184C>T纯合子突变而导致的dHMN患者,病理检查结果显示其具有神经疾病和肌肉疾病特征。综上, DNAJB2突变与SMA息息相关。
DNAJB6与神经退行性疾病
DNAJB6与帕金森病
帕金森病是全球第二大神经退行性疾病,1%~3% 60岁以上人群患有此病。帕金森病的主要病理特征包括黑质致密部腹外侧区域多巴胺能神经元丢失,α-syn聚集形成路易小体,Aβ形成淀粉样斑块,τ蛋白形成神经原纤维缠结等 [27] 。Hussein等 [28] 发现,DNAJB6通过与HSP70的相互作用能够抑制Aβ 42聚集。DNAJB6也可抑制α-syn聚集 [29] 。Deshayes等 [29] 研究发现, DNAJB6敲除细胞中检测到更多α-syn的聚集体;加入蛋白酶体抑制剂MG132孵育后,α-syn预制纤维诱导的α-syn聚集显著性减少,这表明DNAJB6可能帮助错误折叠和聚集的α-syn进行降解;Arkan等 [30] 在小鼠模型中证明了该结论,DNAJB6能够降低因α-syn过表达导致的多巴胺能细胞死亡率。这些研究提供了DNAJB6调控帕金森病相关毒性蛋白的策略,并提出预防或治疗帕金森病及相关疾病的潜在方法。
DNAJB6与LGMDD1
LGMDD1是一种常染色体显性遗传病,其特征是骨盆带、大腿、肩胛带和手臂近端进行性无力 [31] 。LGMDD1肌肉病理学特征还包括形成由 RNA结合蛋白组成的内含物,如TDP43和hnRNAPA2B [32] 。Bengoechea等 [32] 使用酵母、细胞培养物和LGMDD1小鼠模型,发现LGMDD1与DNAJB6突变相关。在酵母中,DNAJB6b突变(F89i和P96R)的过表达会增加细胞中TDP43与应激颗粒的共定位。利用CRISPR/CAS9使野生型小鼠DNAJB6-F90I发生突变,对照组HSP70在骨骼肌的Z椎间盘和肌浆之间迅速扩散;而小鼠模型HSP70在肌肉中的扩散速度降低,这可能缘于突变体小鼠中HSP70与DNAJB6亲和力降低。用DNAJ-HSP70复合物的小分子抑制剂JG231治疗LGMDD1小鼠可重新动员HSP70,而缓解小鼠肌无力等症状 [32] 。因此,以DNAJB6-HSP70为靶点的治疗方法可能是治疗LGMDD1的有效措施。
DNAJC5与神经退行性疾病
DNAJC5与NCL
NCL是一类遗传性神经退行性疾病,其临床症状包括进行性精神和运动退化、失明、癫痫发作及死亡,以及异常蛋白在神经元中发生聚集和沉积 [10] 。DNAJC5含有半胱氨酸串结构域,这些半胱氨酸残基棕榈酰化后能靶向高尔基膜 [33] 。在NCL患者的脑组织中,发现DNAJC5的突变p.L116DEL和p.L115R [10] 。实验证明,半胱氨基酸域中的p.L116DEL和p.L115R突变导致DNAJC5形成高分子量且不溶于十二烷基硫酸钠去垢剂的聚集体,这与DNAJC5中半胱氨酸簇的棕榈酰化有关 [34] 。最近,Huang等 [35] 在一例中国年轻女性NCL患者中发现 DNAJC5基因的p.C128Y突变,该患者出现脑萎缩和记忆丧失等症状。虽然目前已发现NCL与 DNAJC5基因突变有关,但 DNAJC5突变与NCL的关系及其具体机制仍需进一步探究。
DNAJC5与帕金森病
在HEK293T细胞系中过表达DNAJC5可降低细胞内τ蛋白水平,增加细胞外τ蛋白释放 [36] 。 DNAJC5敲除小鼠细胞外τ蛋白水平显著降低,说明DNAJC5是τ蛋白释放的关键因素 [36] 。与τ蛋白类似,TDP-43以及α-syn也通过DNAJC5释放到细胞外,这说明DNAJC5可以促进神经退行性蛋白质释放到细胞外空间 [36] 。Fernández-Chacón等 [37] 发现, DNAJC5敲除小鼠的神经肌肉功能和感受器受到损伤,神经肌肉连接点退化,说明 DNAJC5敲除会导致突触功能障碍;未表达转基因α-syn的 DNAJC5敲除小鼠在几个月内死亡,但表达转基因α-syn的DNAJC5缺陷小鼠存活超过15个月;DNAJC5缺失会导致神经元死亡以及神经肌肉接头恶化,但能被α-syn逆转;行为学实验表明,α-syn能够挽救 DNAJC5敲除小鼠的运动障碍缺失且内源性α-syn缺失会加剧这些表型;DNAJC5缺失抑制了SNARE复合体的组装,α-syn可逆转这种抑制。该研究显示,α-syn与DNAJC5、SNARE蛋白在保护神经末梢免受损伤方面起重要作用。
DNAJC13与特发性震颤
特发性震颤是最常见的运动障碍之一,据报道全世界有超过 6000 万人受到影响 [38] 。 特发性震颤患者主要表现为运动障碍,如姿势性和动作性震颤,但也可能有认知障碍和神经精神症状 [38] 。DNAJC13定位于早期内吞体和循环内吞体中,在核周区域也呈点状分布,在囊泡形成和内吞运输中发挥作用 [39] 。对震颤发作的患者进行外显子组测序分析发现, DNAJC13基因的p.N855S突变与特发性震颤之间存在显著关联 [40] 。细胞过表达p.N855S突变体可导致转铁蛋白在核内吞体中异常滞留,表明 DNAJC13突变可能阻碍核内吞体货物运输 [39] 。除p.N855S突变,E1740Q、R1615H、L2120W等 DNAJC13突变都可能与帕金森病风险增加有关 [ 39, 41] 。
结语
迄今,DNAJ的研究揭示了该家族蛋白在蛋白质折叠、跨膜运输、突触调节等方面具有多种功能 [ 3, 14, 16] 。本文主要总结了DNAJ家族在神经退行性疾病中的作用机制。尽管目前对于部分DNAJ蛋白研究较为深入,但DNAJ家族还有许多分子功能未知,如DNAJC4、DNAJC22、DNAJC25等;同时,DNAJ影响神经退行性疾病的具体机制以及自身是如何被调控等问题均有待进一步探究。
DNAJ家族的表达水平受miRNA的调控,如抑制miR-370和miR-543可以上调DNAJB1表达,促进DNAJB1对polyQ的降解,进而缓解SCA3患者的病情 [4] 。发现和研究DNAJ家族的新功能及其调节机制可以为神经退行性疾病病理学机制的研究提供理论依据,同时为开发有效的治疗方法开辟新道路。
COMPETING INTERESTS
所有作者均声明不存在利益冲突
Funding Statement
国家重点研发计划(2016YFA0501000);国家高层次人才计划(588020-D01907/018)
References
- 1.ZHOU J, JANGILI P, SON S, et al. Fluorescent diagnostic probes in neurodegenerative diseases[J/OL] Adv Mater. . 2020;32(51):e2001945. doi: 10.1002/adma.202001945. [DOI] [PubMed] [Google Scholar]
- 2.BROWN D G, WOBST H J. Opportunities and challenges in phenotypic screening for neurodegenerative disease research[J] J Med Chem. . 2020;63(5):1823–1840. doi: 10.1021/acs.jmedchem.9b00797. [DOI] [PubMed] [Google Scholar]
- 3.PEMBERTON S, MADIONA K, PIERI L, et al. HSC70 protein interaction with soluble and fibrillar α-synuclein[J] J Biol Chem. . 2011;286(40):34690–34699. doi: 10.1074/jbc.M111.261321. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.EVERT B O, NALAVADE R, JUNGVERDORBEN J, et al. Upregulation of miR-370 and miR-543 is associated with reduced expression of heat shock protein 40 in spinocerebellar ataxia type 3[J/OL] PLoS One. . 2018;13(8):e0201794. doi: 10.1371/journal.pone.0201794. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.HASEGAWA T, YOSHIDA S, SUGENO N, et al. DNAJ/HSP40 family and parkinson’s disease[J] Front Neurosci. . 2017;11:743. doi: 10.3389/fnins.2017.00743. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.YAN P, REN J, ZHANG W, et al. Protein quality control of cell stemness[J] Cell Regen. . 2020;9(1):22. doi: 10.1186/s13619-020-00064-2. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.QIU X B, SHAO Y M, MIAO S, et al. The diversity of the DNAJ/HSP40 family, the crucial partners for HSP70 chaperones[J] Cell Mol Life Sci. . 2006;63(22):2560–2570. doi: 10.1007/s00018-006-6192-6. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.DIANE A, ABUNADA H, KHATTAB N, et al. Role of the DNAJ/HSP40 family in the pathogenesis of insulin resistance and type 2 diabetes[J] Ageing Res Rev. . 2021;67:101313. doi: 10.1016/j.arr.2021.101313. [DOI] [PubMed] [Google Scholar]
- 9.MOSSER D D, MORIMOTO R I. Molecular chaperones and the stress of oncogenesis[J] Oncogene. . 2004;23(16):2907–2918. doi: 10.1038/sj.onc.1207529. [DOI] [PubMed] [Google Scholar]
- 10.ZAROUCHLIOTI C, PARFITT D A, LI W, et al. DNAJ proteins in neurodegeneration: essential and protective factors[J] Phil Trans R Soc Lond B Boil Sci. . 2018;373(1738):20160534. doi: 10.1098/rstb.2016.0534. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.HARTL F U. Molecular chaperones in cellular protein folding[J] Nature. . 1996;381(6583):571–580. doi: 10.1038/381571a0. [DOI] [PubMed] [Google Scholar]
- 12.BATES G P, DORSEY R, GUSELLA J F, et al. Huntington disease[J] Nat Rev Dis Primers. . 2015;1(1):15005. doi: 10.1038/nrdp.2015.5. [DOI] [PubMed] [Google Scholar]
- 13.RODRÍGUEZ-GONZÁLEZ C, LIN S, ARKAN S, et al. Co-chaperones DNAJA1 and DNAJB6 are critical for regulation of polyglutamine aggregation[J] Sci Rep. . 2020;10(1):8130. doi: 10.1038/s41598-020-65046-5. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.LI X, LIU H, FISCHHABER P L, et al. Toward therapeutic targets for SCA3: insight into the role of Machado-Joseph disease protein ataxin-3 in misfolded proteins clearance[J] Prog Neurobiol. . 2015;132:34–58. doi: 10.1016/j.pneurobio.2015.06.004. [DOI] [PubMed] [Google Scholar]
- 15.CHAI Y, KOPPENHAFER S L, BONINI N M, et al. Analysis of the role of heat shock protein (HSP) molecular chaperones in polyglutamine disease[J] J Neurosci. . 1999;19(23):10338–10347. doi: 10.1523/JNEUROSCI.19-23-10338.1999. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.GESS B, AUER-GRUMBACH M, SCHIRMACHER A, et al. HSJ1-related hereditary neuropathies: novel mutations and extended clinical spectrum[J] Neurology. . 2014;83(19):1726–1732. doi: 10.1212/WNL.0000000000000966. [DOI] [PubMed] [Google Scholar]
- 17.SAVERI P, MAGRI S, MADERNA E, et al. DNAJB2 ‐related Charcot‐Marie‐Tooth disease type 2: pathomechanism insights and phenotypic spectrum widening[J] . Euro J Neurol. . 2022;29(7):2056–2065. doi: 10.1111/ene.15326. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.LABBADIA J, NOVOSELOV S S, BETT J S, et al. Suppression of protein aggregation by chaperone modification of high molecular weight complexes[J] Brain. . 2012;135(4):1180–1196. doi: 10.1093/brain/aws022. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.WESTHOFF B, CHAPPLE J P, VAN DER SPUY J, et al. HSJ1 is a neuronal shuttling factor for the sorting of chaperone clients to the proteasome[J] Curr Biol. . 2005;15(11):1058–1064. doi: 10.1016/j.cub.2005.04.058. [DOI] [PubMed] [Google Scholar]
- 20.SCHNAIDER T, SŐTI C, CHEETHAM M E, et al. Interaction of the human DNAJ homologue, HSJ1b with the 90 kDa heat shock protein, Hsp90[J] Life Sci. . 2000;67(12):1455–1465. doi: 10.1016/S0024-3205(00)00735-9. [DOI] [PubMed] [Google Scholar]
- 21.OTTAVIANI D, MARIN O, ARRIGONI G, et al. Protein kinase CK2 modulates HSJ1 function through phosphorylation of the UIM2 domain[J] Hum Mol Genet. . 2017;26(3):611–623. doi: 10.1093/hmg/ddw420. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22.GROEN E J N, TALBOT K, GILLINGWATER T H. Advances in therapy for spinal muscular atrophy: promises and challenges[J] Nat Rev Neurol. . 2018;14(4):214–224. doi: 10.1038/nrneurol.2018.4. [DOI] [PubMed] [Google Scholar]
- 23.SANCHEZ E, DARVISH H, MESIAS R, et al. Identification of a large DNAJB2 deletion in a family with spinal muscular atrophy and Parkinsonism[J] . Hum Mutat. . 2016;37(11):1180–1189. doi: 10.1002/humu.23055. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.CHAPPLE J P, CHEETHAM M E. The chaperone environment at the cytoplasmic face of the endoplasmic reticulum can modulate rhodopsin processing and inclusion formation[J] J Biol Chem. . 2003;278(21):19087–19094. doi: 10.1074/jbc.M212349200. [DOI] [PubMed] [Google Scholar]
- 25.BLUMEN S C, ASTORD S, ROBIN V, et al. A rare recessive distal hereditary motor neuropathy with HSJ1 chaperone mutation[J] Ann Neurol. . 2012;71(4):509–519. doi: 10.1002/ana.22684. [DOI] [PubMed] [Google Scholar]
- 26.LIU M, XU Y, HONG D, et al. DNAJB2 c.184C>T mutation associated with distal hereditary motor neuropathy with rimmed vacuolar myopathy[J] Clin Neuropathol. . 2022;41(9):226–232. doi: 10.5414/NP301466. [DOI] [PubMed] [Google Scholar]
- 27.GOEDERT M, COMPSTON A. Parkinson’s disease —— the story of an eponym[J] Nat Rev Neurol. . 2018;14(1):57–62. doi: 10.1038/nrneurol.2017.165. [DOI] [PubMed] [Google Scholar]
- 28.HUSSEIN R M, HASHEM R M, RASHED L A. Evaluation of the amyloid beta-GFP fusion protein as a model of amyloid beta peptides-mediated aggregation: a study of DNAJB6 chaperone[J] Front Mol Neurosci. . 2015;8:40. doi: 10.3389/fnmol.2015.00040. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.DESHAYES N, ARKAN S, HANSEN C. The molecular chaperone DNAJB6, but not DNAJB1, suppresses the seeded aggregation of alpha-synuclein in cells[J] Int J Mol Sci. . 2019;20(18):4495. doi: 10.3390/ijms20184495. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 30.ARKAN S, LJUNGBERG M, KIRIK D, et al. DNAJB6 suppresses alpha-synuclein induced pathology in an animal model of Parkinson’s disease[J] Neurobiol Dis. . 2021;158:105477. doi: 10.1016/j.nbd.2021.105477. [DOI] [PubMed] [Google Scholar]
- 31.THOMPSON R, STRAUB V. Limb-girdle muscular dystrophies —— international collaborations for translational research[J] Nat Rev Neurol. . 2016;12(5):294–309. doi: 10.1038/nrneurol.2016.35. [DOI] [PubMed] [Google Scholar]
- 32.BENGOECHEA R, FINDLAY A R, BHADRA A K, et al. Inhibition of DNAJ-HSP70 interaction improves strength in muscular dystrophy[J] J Clin Invest. . 2020;130(8):4470–4485. doi: 10.1172/JCI136167. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 33.GREAVES J, CHAMBERLAIN L H. Dual role of the cysteine-string domain in membrane binding and palmitoylation-dependent sorting of the molecular chaperone cysteine-string protein[J] Mol Biol Cell. . 2006;17(11):4748–4759. doi: 10.1091/mbc.e06-03-0183. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 34.DIEZ-ARDANUY C, GREAVES J, MUNRO K R, et al. A cluster of palmitoylated cysteines are essential for aggregation of cysteine-string protein mutants that cause neuronal ceroid lipofuscinosis[J] Sci Rep. . 2017;7(1):10. doi: 10.1038/s41598-017-00036-8. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 35.HUANG Q, ZHANG Y F, LI L J, et al. Adult-onset neuronal ceroid lipofuscinosis with a novel DNAJC5 mutation exhibits aberrant protein palmitoylation[J] Front Aging Neurosci. . 2022;14:829573. doi: 10.3389/fnagi.2022.829573. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 36.FONTAINE S N, ZHENG D, SABBAGH J J, et al. DNAJ/HSC70 chaperone complexes control the extracellular release of neurodegenerative‐associated proteins[J] EMBO J. . 2016;35(14):1537–1549. doi: 10.15252/embj.201593489. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 37.FERNÁNDEZ-CHACÓN R, WÖLFEL M, NISHIMUNE H, et al. The synaptic vesicle protein CSPα prevents presynaptic degeneration[J] Neuron. . 2004;42(2):237–251. doi: 10.1016/s0896-6273(04)00190-4. [DOI] [PubMed] [Google Scholar]
- 38.WELTON T, CARDOSO F, CARR J A, et al. Essential tremor[J] Nat Rev Dis Primers. . 2021;7(1):83. doi: 10.1038/s41572-021-00314-w. [DOI] [PubMed] [Google Scholar]
- 39.VILARIÑO-GÜELL C, RAJPUT A, MILNERWOOD A J, et al. DNAJC13 mutations in Parkinson disease[J] . Hum Mol Genet. . 2014;23(7):1794–1801. doi: 10.1093/hmg/ddt570. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 40.RAJPUT A, ROSS J P, BERNALES C Q, et al. VPS35 and DNAJC13 disease-causing variants in essential tremor[J] Eur J Hum Genet. . 2015;23(6):887–888. doi: 10.1038/ejhg.2014.164. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 41.ROSS J P, DUPRE N, DAUVILLIERS Y, et al. Analysis of DNAJC13 mutations in French-Canadian/French cohort of Parkinson’s disease[J] . Neurobiol Aging. . 2016;45:212.e13–212.e17. doi: 10.1016/j.neurobiolaging.2016.04.023. [DOI] [PubMed] [Google Scholar]