Abstract
肝脏计算机断层扫描成像(CT)的三维(3D)肝脏和肿瘤分割对于辅助医生的诊断及预后具有非常重要的临床价值。为了准确快速地分割肝脏及肿瘤区域,本文提出了一种基于条件生成对抗网络(cGAN)的肿瘤 3D 条件生成对抗分割网络(T3scGAN),同时采用了一个由粗到细的 3D 自动分割框架对肝脏及肿瘤区域实施精准分割。本文采用 2017 年肝脏和肿瘤分割挑战赛(LiTS)公开数据集中的 130 个病例进行训练、验证和测试 T3scGAN 模型。最终 3D 肝脏区域分割的验证集和测试集的平均戴斯(Dice)系数分别为 0.963 和 0.961,而 3D 肿瘤区域分割的验证集和测试集的平均 Dice 系数分别为 0.819 和 0.796。实验结果表明,提出的 T3scGAN 模型能够有效地分割 3D 肝脏及其肿瘤区域,因此能够更好地辅助医生进行肝脏肿瘤的精准诊断和治疗。
Keywords: 肝脏和肿瘤区域三维自动分割, 条件生成对抗网络, 深度学习, 计算机断层扫描成像
Abstract
The three-dimensional (3D) liver and tumor segmentation of liver computed tomography (CT) has very important clinical value for assisting doctors in diagnosis and prognosis. This paper proposes a tumor 3D conditional generation confrontation segmentation network (T3scGAN) based on conditional generation confrontation network (cGAN), and at the same time, a coarse-to-fine 3D automatic segmentation framework is used to accurately segment liver and tumor area. This paper uses 130 cases in the 2017 Liver and Tumor Segmentation Challenge (LiTS) public data set to train, verify and test the T3scGAN model. Finally, the average Dice coefficients of the validation set and test set segmented in the 3D liver regions were 0.963 and 0.961, respectively, while the average Dice coefficients of the validation set and test set segmented in the 3D tumor regions were 0.819 and 0.796, respectively. Experimental results show that the proposed T3scGAN model can effectively segment the 3D liver and its tumor regions, so it can better assist doctors in the accurate diagnosis and treatment of liver cancer.
Keywords: three-dimensional automatic segmentation of liver and tumor regions, conditional generative adversarial networks, deep learning, computed tomography
引言
肝脏是承担人体代谢功能的重要器官,肝脏一旦出现恶性肿瘤病变将会严重威胁人体生命健康。计算机断层扫描成像(computed tomography,CT)是目前在肝脏病变诊断中普遍采用的常规诊断方式。CT 图像可以反映出肝脏肿瘤的形态、数目、部位、边界等信息,因此基于 CT 影像技术对肝脏肿瘤区域实施有效的分割具有重要的临床价值。但是由于 CT 图像中肝脏与周边脏器的灰度值非常接近,且不同患者个体差异大等原因,导致肝脏与肝脏肿瘤的三维(three dimensional,3D)分割非常困难。传统的肝脏分割方法,有利用形状先验、灰度分布以及边界和区域信息来描述肝脏特征并划定其边界的统计变形模型,也有基于纹理和灰度等级的方法。这些分割方法的效率很低,鲁棒性比较差,而且需要根据经验调节大量的参数。例如,Stawiaski 等[1]使用最小曲面和马尔科夫随机场对肝脏肿瘤进行分割。Smeets 等[2]提出了一种基于水平集的半自动分割方法,并使用该方法对 CT 影像中的肝脏肿瘤及肝脏转移区域进行分割。Li 等[3]提出了一种新的统一水平集模型,该模型通过整合图像的梯度、图像的区域竞争及先验信息对 CT 图像中的肝脏肿瘤进行分割。Li 等[4]提出了一种水平集模型,该模型将似然能量和边缘能量结合在一起对 CT 图像中的肝脏肿瘤区域进行分割。Zhang 等[5]提出了一种交互式的半自动分割方法来分割 CT 图像中的肝脏及肿瘤区域,该方法首先对输入的 CT 图像进行预处理,粗略地分割出肝脏区域,然后在肝脏肿瘤区域设置一些种子点,最后用这些种子点位置的灰度值训练一个支持向量机(support vector machine,SVM)[6],并用一些形态学的后处理方式对 SVM 的分类结果粗略地勾画出肝脏肿瘤区域的边界。近年来,随着深度学习技术的发展,研究人员开发了一系列基于深度学习的分割算法来分割肝脏及肿瘤区域。例如,Li[7]提出了一种卷积分类网络模型,该模型对二维(two dimensional,2D)CT 图像中 17 × 17 大小的图像块进行逐块预测,判断该图像块究竟为病变区域还是其他区域,最终可以分割出肝脏肿瘤。Li 等[8]提出了一种混合密集连接 U 型网络(hybrid densely connected Unet,H-DenseUNet)分割模型,该模型先对肝脏 2D CT 图像中的肝脏及肿瘤区域进行分割,然后将学习到的 2D 图像高维特征用一个 3D 的卷积网络进行融合,最终得到肝脏和肿瘤的 3D 分割结果。Bi 等[9]提出了一种基于卷积残差网络(residual network,Resnet)[10]的多尺度模型,该模型可以在多个尺度上对 CT 图像中的肝脏肿瘤进行识别,从而提高了识别的精度。Gruber 等[11]比较研究了两种肝脏肿瘤分割方案,第一种是直接采用了 1 个 2D U 型网络(Unet)[12]对肝脏肿瘤进行端到端分割;第二种方案则使用了 2 个 2D Unet,其中第一个 Unet 用于分割肝脏区域,然后把分割的肝脏感兴趣区域(region of interest,ROI)乘以输入 CT 图像提取肝脏区域,之后用提取的肝脏区域图像又训练了一个 2D Unet 对肝脏肿瘤进行 2D 分割。通过对比发现,级联的分割网络可以达到更好的肝脏肿瘤分割性能。Dey 等[13]设计了一种级联的肝脏肿瘤分割模型,该模型先用一个 2D 深度卷积分割网络分割出肝脏及较大的肿瘤区域,然后再用一个 3D 深度卷积分割网络检测分割出小肿瘤区域,从而提高了 CT 图像中肝脏肿瘤的分割准确率。Deng 等[14]先用 1 个 3D 密集连接卷积分类网络检测肝脏肿瘤的边缘,然后用 3D 密集连接卷积分类网络输出的肿瘤边缘概率动态调节水平集分割参数,同时根据分类网络检测的肿瘤区域位置初始化水平集肿瘤分割局部窗口的大小,最后分割出肝脏肿瘤;Lu 等[15]先构建了 1 个 3D 卷积网络对肝脏区域进行分割并输出概率图,然后基于概率图用图切法得到最终的肝脏分割结果。
与传统的肝脏肿瘤分割方法相比,基于深度学习的分割方法在分割效率和准确率上都有很大提升,但是也普遍存在以下几个问题:① 直接进行 3D 分割的模型计算量太大,因此大部分 3D 分割模型都是基于将 2D 分割结果拼接成 3D 的分割结果。基于 2D 分割结果再拼接成 3D 分割结果的分割方法虽然可以减少模型的运算量,但是分割的结果却不如直接以 3D 分割模型进行分割的结果。因为 2D 分割模型的优化目标是 ROI 边界曲线,而 3D 分割模型可以优化 ROI 的整个曲面;② 需要大量的预处理和后处理操作。在肝脏肿瘤的 2D 切面中,由于肝脏肿瘤的大小、位置、纹理等信息在不同的病例中差异较大,如果使用端到端的网络很难直接定位到肿瘤区域;③ 需要很多标记数据进行模型的训练。因为肿瘤的分割检测比较困难,提升模型的性能就需要增加模型的复杂度和可训练参数量,如果训练集的数量过少就会导致模型分割性能不足或者过拟合。
本文基于条件对抗生成网络(conditional generative adversarial networks,cGAN)[16]构建了一个新型的肿瘤 3D 分割条件对抗生成网络(tumor 3D segmentation conditional generative adversarial networks,T3scGAN)作为主要的分割模型对 3D 肝脏区域和 3D 肝脏肿瘤区域进行分割,同时使用了一个由粗到细的 3D 分割框架来精确地分割肝脏肿瘤,从而辅助医生实现对肝脏病变及肝脏肿瘤的快速定位与识别,达到精准诊疗的目的。
1. 由粗到细的肝脏肿瘤分割框架
在 CT 影像中,肝脏肿瘤的全自动精准分割对分割算法具有较大的挑战性,主要原因是不同病例的肝脏肿瘤之间位置、形态和纹理特征差异性很大。因此,本文采用了一个由粗到细的 3D 分割框架来精确分割肝脏肿瘤。分割框架流程图如图 1 所示,当输入一个 3D 的 CT 测试病例时,整个分割框架首先进行自动数据预处理,然后用训练好的肝脏分割 T3scGAN 模型对肝脏区域进行自动 3D 分割,得到肝脏区域 3D 分割结果。之后基于肝脏分割的结果结合预处理后的 CT 图像进行自动肝脏 3D ROI 提取,最后用训练好的肝脏肿瘤分割 T3scGAN 模型对肝脏肿瘤区域进行自动 3D 分割。
图 1.

3D coarse-to-fine liver tumors segmentation framework
由粗到细的肝脏肿瘤 3D 分割框架
1.1. 数据预处理
在训练和测试过程中,本文首先将原始 CT 影像的窗宽设置为 300,窗位设置为 100。之后重新采样到 256 × 256 × 128 大小,再将重采样的数据归一化至 0~1 区间作为网络的输入。
1.2. T3scGAN
传统的 3D 分割模型都有一个预定义的损失函数作为优化目标来训练网络,因此损失函数的选择对网络的性能影响较大。cGAN 提供了可训练的损失函数机制[17]。像素点到像素点(pixel to pixel,pix2pix)的图像翻译模型[18]首次应用这种机制完美地解决了图像翻译问题。受 cGAN 和 pix2pix 模型的启发,本文构建了一种新型的 3D 条件对抗网络模型(T3scGAN)来分割肝脏及肿瘤区域,模型结构如图 2 所示。
图 2.

T3scGAN model structure
T3scGAN 模型结构
T3scGAN 包含 1 个生成器网络和 1 个判别器网络。生成器网络是一个端到端的 3D 分割结构,判别器网络是一个多层的分类模型。
1.2.1. 生成器结构
在医学图像分割任务中,Unet 是目前非常流行的深度框架。Unet 受到全卷积神经网络(fully convolutional networks,FCN)模型[19]的启发,将多个尺度下提取的深度特征进行了融合,但不同于 FCN 直接拼接融合的方式,Unet 采用了相同尺度的编码器和解码器结构,将编码器不同尺度下的深度特征和相同尺度的解码器特征相融合,这样就可以将原始图像的细节信息尽可能地保留下来,同时 Unet 在编码器部分采用了最大池化的方法,使得快速下采样的同时又能够保证分割的精度。3D Unet 模型[20]是基于 Unet 的 3D 分割版本模型。
受 3D Unet 结构的启发,T3scGAN 生成器中也使用了一系列的跳跃式连接,从而可以使得一些细节部分得以保留。生成器中对特征的下采样和上采样均使用卷积和转置卷积操作,这样可以更加平滑地融合各个部分的细节信息。但是在 Unet 中直接将编码器部分的低维特征拼接到解码器部分的高维特征进行操作会导致语义代沟[21]。为了缓解这个问题,本文采用了特征深度融合的方式来使得低维形态特征和高维语义特征之间尽可能相似。
Resnet 模型提供了一种非常有效的特征深度融合方式,其最大的特点是将输入的特征图和输出的特征图相加作为最终的输出。对于一个残差块的输入 x 和输出 y 之间的映射关系如式(1)所示:
 |
1 |
其中 F 表示整个残差块的映射,x 和 Wx 代表输入和对应输入的卷积操作, 表示残差块中的激活函数。为了方便反向传播求梯度,一般
表示残差块中的激活函数。为了方便反向传播求梯度,一般 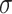 为线性整流单元(rectified linear unit,ReLU)函数。ReLU 函数的表达式如式(2)所示:
为线性整流单元(rectified linear unit,ReLU)函数。ReLU 函数的表达式如式(2)所示:
 |
2 |
ReLU 激活函数仅保留输入特征中大于 0 的部分,并且直接将大于 0 的部分输出,所以在反向传播求梯度的过程中,该激活函数对大于 0 的输入 x 的偏导数恒等于 1,因此使用 ReLU 激活函数可以加快网络的训练。
在 Wx 操作中一般会用一个标准化层来对卷积的输出进行标准化。标准化层的作用非常大,它会将网络的输出一直保持在相同的分布区间,避免梯度消失,同时正则化网络,提高网络的泛化能力。在卷积操作之后常用的标准化方式为批量标准化(batch normalization,BN)[22],该标准化方式的表达式如式(3)所示:
 |
3 |
其中,z 代表 3D 卷积层(3D convolutional layer,Conv3d)输出的特征张量 B × C × H × W × D,B 表示批量样本的数量,C 表示特征张量的通道数,H × W × D 指的是特征张量中的 3D 特征矩阵。但是受限于计算机图形处理器(graphics processing unit,GPU)的显存大小,批量样本的数量只能设置很小,这对于 BN 的方式非常不利,所以本文采用了组标准化(group normalization,GN)[23]的方式来替代 BN。GN 和 BN 的主要区别是 GN 首先将特征张量的通道分为许多组,然后对每一组做归一化。即,将特征张量矩阵的维度由[B,C,H,W,D]改变为[B,C,G,C/G,H,W,D],然后归一化的维度(求均值和方差的维度)为[C/G,H,W,D]。
因此在加入 Resnet 连接的反向传播中,如果对公式(1)求  的偏导数可以得到如式(4)所示:
的偏导数可以得到如式(4)所示:
 |
4 |
所以当网络很深的时候,dF/dx 有可能变为 0,此时 df/dx 恒等于 1,对于整个网络的前向传播仍然有效。所以 T3scGAN 在编码器和解码器之间采用了 3D 残差结构来过渡,即将原始的残差块结构中的操作由 2D 全部替换成了 3D,这样既保证了编码器输出的深度特征充分融合,并且通过增加网络可训练参数来增加网络的鲁棒性,而且也可以避免梯度消失的问题。
T3scGAN 只采用了 2 个尺度下的特征融合,这样做的目的是在保证 3D 分割准确率的条件下,尽可能地减少网络参数和网络计算量,同时避免了下采样尺度过多而导致对尺寸小的肿瘤区域造成不可恢复的细节损失,从而保留更多的细节信息在相等大小输入输出的残差连接中深度融合。在生成器的编码部分,从左到右相邻两个尺度的特征图谱之间的尺寸为正 2 倍的关系,滤波器数量为负 2 倍的关系。对应的在生成器的解码部分,从左到右相邻两个尺度的特征图谱之间的尺寸为负 2 倍的关系,滤波器数量为正 2 倍的关系。在输入层中 T3scGAN 采用了一组卷积操作将输入 CT 映射至特征,同时将原始数据的单通道快速增加为多通道。并且采用了卷积核大小为 7 × 7 × 7,卷积步长为 1 × 1 × 1 的大卷积核来快速地对原始数据进行降维。输出层同样用大卷积核来快速过渡,其主要的作用是与输入层对称,将深度特征的多通道平滑地减少为单通道。并且在输出层的末尾采用双曲正切(hyperbolic tangent,Tanh)激活函数对卷积输出进行激活,因为在输出层的卷积输出特征图中目标和背景的特征相差已经很明显了,而且 Tanh 激活函数是 0 均值输出。在输入和输出层中,为了保证特征的大小不变,因此都在卷积前单独做了 3 × 3 × 3 的特征图 3D 填充(3D padding,Pad3d)。T3scGAN 中采用了边缘复制的方式对输入的特征图进行 Pad3d。在 2 个下采样层中使用了 3 × 3 × 3 的小卷积核有助于平滑有效地提取特征,同时为了将输入特征图下采样 2 倍,卷积的步长为 2 × 2 × 2,并且在卷积后进行了大小为 1 × 1 × 1 的 Pad3d。同样地,在上采样层中 T3scGAN 采用了相同的卷积核大小,卷积步长以及 Pad3d 保证输入和输出的特征图之间的 2 倍关系。不同的是,在上采样层中所有的卷积操作都被替换成了 3D 转置卷积(3D transposed convolution,TConv3d)操作。在残差块中同样采用了 3 × 3 × 3 的小卷积核,同时为了保证输入输出特征图的大小不变,卷积步长为 1 × 1 × 1,而且在卷积后进行了大小为 1 × 1 × 1 的 Pad3d。
如图 3 所示的 T3scGAN 生成器结构中,从左到右依次为图 2 中生成器的 5 个主要部分。卷积滤波器的通道数量由输入层的基本通道数量决定,即相邻的下采样层后一层是前一层的 2 倍。
图 3.

Detailed structure of the modules in the generator of T3scGAN
T3scGAN 生成器模块的详细结构
1.2.2. 判别器结构
在 cGAN 中判别器的主要作用是判断一张图是真实的图像还是生成器生成的结果,即判断生成器生成的结果和真实数据的差异性。因此判别器可以很好地监督生成器的训练,主要是因为它可以从深层的数据分布上比较生成器生成的结果和真实数据之间的差异。所以判别器就等价于是给生成器训练提供了一个可训练的损失函数。同时判别器的输出只是真实/生成(1/0),所以判别器可以用一个二分类的卷积神经网络替代。
为了让网络适应多尺度的输入,本文采用了 pix2pix 中提出的图像块 GAN 技术。该技术的核心是在判别器中,去掉了传统分类网络的特征扁平化操作和全连接层,直接让判别器输出一个单通道的特征图,然后用真实数据得到的特征图和生成数据得到的特征图进行误差计算。同时为了尽可能有效匹配小肿瘤区域的特征,T3scGAN 的判别器都采用了 1 × 1 × 1 的小卷积核和 1 × 1 × 1 的卷积步长保证了特征张量只在通道数量上进行变化,所以判别器最终输出的特征张量维度为[B,1,H,W,D]。为了使得判别器的判别有效,本文采用了 cGAN 的框架,把生成器的输入原始 CT 数据作为判别条件,和生成结果或真实标记在通道维度上进行拼接之后输入判别器网络,所以判别器的输入维度为[B,2,H,W,D]。同时为了保证判别器结构中不会出现“死神经元”现象,所以在判别器网络中卷积层之间使用了泄露的 ReLU(LeakyReLU)激活函数。LeakyReLU 激活函数不同于 ReLU 函数,当输入特征张量中的值小于零时,会有一个泄露值输出,这样就不会导致只要输入的特征张量值小于零时输出始终为 0,其一阶导数也始终为 0,以致于神经元不能更新参数,也就是神经元不学习的情况出现。LeakyReLU 的表达式如式(5)所示:
 |
5 |
本文在判别器中的 LeakyReLU 激活函数的泄露值 ε 均设置为 0.2。
判别器结构如图 4 所示,为了避免判别器判别能力过强而导致生成器的训练崩溃,并且考虑到判别器参数量过多可能会导致判别器过拟合,所以本文采用了一个只有 3 层卷积的判别器。
图 4.
T3scGAN discriminator structure
T3scGAN 判别器结构
1.2.3. 损失函数
对于整个 cGAN 模型而言,优化目标函数如式(6)所示:
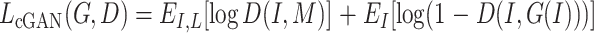 |
6 |
其中,I 是生成器的输入原始数据,M 是真实的标记数据,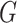 和
和 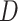 分别代表生成器网络和判别器网络。同时为了增加生成器网络的泛化性,提高其生成能力,T3scGAN 同时使用了 L1 损失函数[24]来优化生成器。L1 损失函数如式(7)所示:
分别代表生成器网络和判别器网络。同时为了增加生成器网络的泛化性,提高其生成能力,T3scGAN 同时使用了 L1 损失函数[24]来优化生成器。L1 损失函数如式(7)所示:
 |
7 |
所以最终的损失函数表达式如式(8)所示:
 |
8 |
其中 λ 为 L1 损失函数的惩罚系数,在本工作中 λ = 10。
2. 实验及评估
2.1. 实验数据
本文采用 2017 肝脏和肿瘤分割挑战赛(liver and tumor segmentation challenge,LiTS)公开数据集中的训练集进行实验。该数据集总共有 131 例标记了 3D 肝脏及 3D 肝脏肿瘤的 CT 病例,所有的 CT 维度均为 512 × 512 × D(D 的范围在 75~750 之间)。本文从中挑选了 130 例 3D 肝脏区域标记清晰的病例作为肝脏分割数据集,然后又从挑选的 130 例肝脏分割数据集中挑选出了 75 例肿瘤标记清晰并且准确的病例作为肝脏肿瘤分割数据集。为了尽量保持肝脏肿瘤的完整性,尤其是体积比较小的肝脏肿瘤,本文采用 256 × 256 × 128 的尺寸作为肝脏 3D 分割的标准尺寸。然后用标记的肝脏区域和原始的 CT 数据点乘提取出肝脏的 3D ROI,并根据最小外接长方体裁剪出 ROI。本文发现裁剪之后的 ROI 维度[H,W,D]大致为 H:185~235、W:135~190、D:20~41,所以将裁剪出来的 ROI 重采样为 256 × 256 × 32 尺寸,作为肝脏肿瘤分割的标准尺寸。本文随机地在肝脏分割数据集中取出 10 个病例作为验证集,然后随机取出 20 个病例作为测试集,剩下的 100 个病例作为训练集。对于肝脏肿瘤分割数据集划分,本文仍然是按照随机的 10∶1∶2 分为训练集、验证集和测试集。在训练过程中,每个迭代周期结束后都在验证集上做一次验证。T3scGAN 在两个数据集上训练和验证的过程记录曲线如图 5 所示,其中蓝色曲线代表生成器的损失曲线,橙色代表判别器的损失曲线,绿色代表验证集的准确率曲线。
图 5.

Training process curve of T3scGAN
T3scGAN 训练过程曲线
从图 5 中可以看出,在两个数据集上训练时,T3scGAN 的训练过程比较稳定,并且收敛的速度也比较快。本文采用戴斯(Dice)系数[25]作为验证集准确率的评估指标。Dice 系数是医学图像分割中常用来评估分割结果和人工标记之间相似度的评估矩阵,Dice 系数(以符号 Dice 表示)的计算公式如式(9)所示:
 |
9 |
其中 yi 和 zi 分别表示真实标记和模型预测输出。当 T3scGAN 训练稳定时,在肝脏数据集和肝脏肿瘤数据集的验证集中 Dice 系数分别为 0.983 和 0.814。为了验证 T3scGAN 的稳定性,在两个数据集上分别对 T3scGAN 进行了 3 折 20 次的交叉验证,最后在肝脏分割数据集和肝脏肿瘤分割数据集上 T3scGAN 的交叉验证 Dice 系数分别为 98.3 ± 0.02 和 82.4 ± 0.03。
本文采用的实验平台为:操作系统 Ubuntu 16.04(开源,美国)、GPU 硬件 GTX 1080Ti(英伟达,美国),可视化软件为 Itk-snap[26-27](宾夕法尼亚大学,美国)。网络固定的超参数为:优化器选用自适应的 Adam[28-29],初始化学习率为 0.000 2,训练的总迭代次数为 200,深度学习框架 Pytorch1.0(Facebook,美国)。
2.2. 肝脏及肿瘤 3D 分割实验评估
CT 图像中肝脏器官的边界清晰,尺寸较大,并且在不同病例之间的纹理相似度较高,所以相对于肿瘤分割,肝脏区域分割比较简单。相对于肝脏区域分割,肝脏肿瘤区域分割更加困难,因为肝脏肿瘤的边界模糊,并且肿瘤的尺寸、位置、纹理等特征差异性大,因此对模型的精准分割是很大的挑战。本文在 2 个数据集上同时对比了 3D Unet 分割模型和 V 型网络(Vnet)分割模型[30],不同模型在肝脏区域及对应的肿瘤区域分割结果对比如图 6 所示。
图 6.
Comparison of liver and corresponding tumor segmentation test results
肝脏及对应肿瘤分割测试对比结果
和肿瘤区域分割相比较,肝脏区域分割的数据输入尺寸较大,受限于 GPU 显存,所以本文将基本通道数量设置为 4,批量的样本数量为 5。肝脏肿瘤分割时,基本通道数量设置为 8,批量的样本数量为 4。
从图 6 的 3D 可视化结果中看到,3 种模型在肝脏分割数据集上的分割性能类似,但是在肝脏肿瘤分割数据集上,3 种模型的分割性能差异较大,T3scGAN 可以达到最好的性能。一些分割结果的 2D 图像细节如图 7 所示,其中红、绿、蓝、黄线分别代表:标记、T3scGAN 的分割结果、3D Unet 分割结果和 Vnet 分割结果。
图 7.
2D slices details
2D 切面细节展示
从图 7 中可以发现,3D Unet 对肝脏肿瘤区域的识别假阳率较高,Vnet 不能够很好地分割出肿瘤的边界。从这两种模型的模型结构上分析,3D Unet 是因为使用了过多的跳跃连接,所以可以在分割中很好地保留细节信息,但是也造成了模型的敏感度很高,所以会将很多相似的非肿瘤小区域预测为肿瘤区域。Vnet 的分割性能要比 3D Unet 好,Vnet 结构中不仅使用了许多跳跃连接,而且网络的参数容量更大,所以数量较少的肿瘤分割训练集会导致 Vnet 训练不充分,无法精确地分割出肿瘤区域的边界。同时 3D Unet 和 Vnet 模型的参数容量较多,3 种模型的网络参数量对比如表 1 所示。
表 1. Comparison of model parameter quantities.
模型参数量对比
| 模型 | 参数量/k | |
| T3scGAN | 肝脏区域:71.5 | 肿瘤区域:279.5 |
| 3D Unet | 16 300 | |
| Vnet | 19 400 | |
从表 1 中可以看出,3D Unet 和 Vnet 模型的参数容量要远大于 T3scGAN 的模型参数容量,所以 3D Unet 和 Vnet 对训练集的数量要求更大。因此在语义信息和空间信息较差,并且数据量较少的肿瘤分割数据集上,3D Unet 和 Vnet 的分割效果都比较差。
本文分别计算了肝脏和肝脏肿瘤测试集中所有样本的 Dice 系数,然后计算了它们的平均值作为评估不同模型性能的指标,评估结果如表 2 所示。
表 2. Comparison of segmentation results of different models.
不同模型的分割结果评估对比
| 分割区域 | 测试样本 数量/例 |
平均 Dice 系数 | ||
| T3scGAN | 3D Unet | Vnet | ||
| 肝脏 3D 分割 | 20 | 0.961 | 0.849 | 0.908 |
| 肝脏肿瘤 3D 分割 | 12 | 0.796 | 0.595 | 0.616 |
从表 2 可以看出,与 3D Unet 和 Vnet 相比,本文提出的 T3scGAN 模型以及由粗到细的肿瘤分割框架对肝脏区域和肝脏肿瘤区域均具有更高的分割精度。
3. 总结与讨论
肝脏及肿瘤区域的全自动精准分割是辅助医生诊断和治疗的重要前提,但是对于一般的深度学习模型[30]而言是一个非常具有挑战性的任务。本文所构建的 T3scGAN 模型具有如下优点:① 模型的复杂度低、参数量少,可以避免网络太深而导致的训练困难和过拟合问题,减少了对训练数据量的要求;② 模型具有很好的监督损失函数机制使得模型得到了充分的训练;③ 模型不仅能优化低维的空间信息(边界、曲面等),而且能保留高维的语义特征(细节、位置等)。本文的下一步工作是将构建的框架和模型应用于更多器官及其肿瘤区域的分割问题,同时基于自动分割结果构建疾病的预测和预后模型。
利益冲突声明:本文全体作者均声明不存在利益冲突。
Funding Statement
国家自然科学基金(U1809205,61771249,91959207,81871352);江苏省自然科学基金(BK20181411);江苏省大气环境与装备技术协同创新中心、江苏省大数据分析技术重点实验室专项课题(2020xtzx005);江苏省“青蓝工程”资助
National Natural Science Foundation of China; Natural Science Foundation of Jiangsu Province of China; Jiangsu Collaborative Innovation Center of Atmospheric Environment and Equipment Technology (CICAEET) and Jiangsu Key Laboratory of Big Data Analysis Technology (B-DAT); Qing Lan Project of Jiangsu Province
References
- 1.Stawiaski J, Decenciere E, Bidault F. Interactive liver tumor segmentation using graph-cuts and watershed//11th International Conference on Medical Image Computing and Computer Assisted Intervention (MICCAI 2008), New York: Springer, 2008.
- 2.Smeets D, Loeckx D, Stijnen B, et al Semi-automatic level set segmentation of liver tumors combining a spiral-scanning technique with supervised fuzzy pixel classification. Med Image Anal. 2010;14(1):13–20. doi: 10.1016/j.media.2009.09.002. [DOI] [PubMed] [Google Scholar]
- 3.Li Bingnan, Chui C K, Chang S, et al A new unified level set method for semi-automatic liver tumor segmentation on contrast-enhanced CT images. Expert Syst Appl. 2012;39(10):9661–9668. doi: 10.1016/j.eswa.2012.02.095. [DOI] [Google Scholar]
- 4.Li C, Wang X, Eberl S, et al A likelihood and local constraint level set model for liver tumor segmentation from CT volumes. IEEE Trans Biomed Eng. 2013;60(10):2967–2977. doi: 10.1109/TBME.2013.2267212. [DOI] [PubMed] [Google Scholar]
- 5.Zhang Xing, Tian Jie, Xiang Dehui, et al. Interactive liver tumor segmentation from CT scans using support vector classification with watershed//2011 Annual International Conference of the IEEE Engineering in Medicine and Biology Society, Boston: IEEE, 2011: 6005-6008.
- 6.Evgeniou T, Pontil M. Support vector machines: theory and applications//Proceedings of the 1999 Advanced Course on Artificial Intelligence, Berlin: Springer, 1999: 249-257.
- 7.Li W Automatic segmentation of liver tumor in CT images with deep convolutional neural networks. Journal of Computer and Communications. 2015;3(11):146. doi: 10.4236/jcc.2015.311023. [DOI] [Google Scholar]
- 8.Li X, Chen H, Qi X, et al H-DenseUNet: hybrid densely connected UNet for liver and tumor segmentation from CT volumes. IEEE Trans Med Imaging. 2018;37(12):2663–2674. doi: 10.1109/TMI.2018.2845918. [DOI] [PubMed] [Google Scholar]
- 9.Bi L, Kim J, Kumar A, et al. Automatic liver lesion detection using cascaded deep residual networks, arXiv preprint, 2017. arXiv: 1704.02703.
- 10.He Kaiming, Zhang Xiangyu, Ren Shaoqing, et al. Deep residual learning for image recognition//2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR), Las Vegas: IEEE, 2016: 770-778.
- 11.Gruber N, Antholzer S, Jaschke W, et al. A joint deep learning approach for automated liver and tumor segmentation, arXiv preprint, 2019. arXiv: 1902.07971.
- 12.Ronneberger O, Fischer P, Brox T. U-net: Convolutional networks for biomedical image segmentation//International Conference on Medical image computing and computer-assisted intervention, Munich: Springer, 2015: 234-241.
- 13.Dey R, Hong Y. Hybrid cascaded neural network for liver lesion segmentation, arXiv preprint, 2019. arXiv: 1909.04797.
- 14.Deng Z, Guo Q, Zhu Z Dynamic regulation of level set parameters using 3D convolutional neural network for liver tumor segmentation. J Healthc Eng. 2019:4321645. doi: 10.1155/2019/4321645. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Lu Fang, Wu Fa, Hu Peijun, et al Automatic 3D liver location and segmentation via convolutional neural network and graph cut. Int J Comput Assist Radiol Surg. 2017;12(2):171–182. doi: 10.1007/s11548-016-1467-3. [DOI] [PubMed] [Google Scholar]
- 16.Goodfellow I, Pouget-Abadie J, Mirza M, et al. Generative adversarial nets//Advances in neural information processing systems, Montreal: NIPS, 2014: 2672-2680.
- 17.Gauthier J Conditional generative adversarial nets for convolutional face generation. Class Project for Stanford CS231N: Convolutional Neural Networks for Visual Recognition, Winter Semester. 2014;2014(5):2. [Google Scholar]
- 18.Isola P, Zhu Junyan, Zhou Tinghui, et al. Image-to-Image translation with conditional adversarial networks//2017 IEEE Conference on Computer Vision and Pattern Recognition (CVPR), Hawaii: IEEE, 2017: 5967-5976.
- 19.Long J, Shelhamer E, Darrell T. Fully convolutional networks for semantic segmentation//2015 IEEE Conference on Computer Vision and Pattern Recognition (CVPR), Boston: IEEE, 2015: 3431-3440.
- 20.Çiçek Ö, Abdulkadir A, Lienkamp S S, et al. 3D U-Net: learning dense volumetric segmentation from sparse annotation//International Conference on Medical Image Computing and Computer-Assisted Intervention, Greece: Springer, 2016: 424-432.
- 21.Zhou Zongwei, Siddiquee M M R, Tajbakhsh N, et al. Unet++: a nested u-net architecture for medical image segmentation// Stoyanov D. et al. Deep Learning in Medical Image Analysis and Multimodal Learning for Clinical Decision Support (DLMIA). Springer, 2018: 3-11.
- 22.Ioffe S, Szegedy C. Batch normalization: accelerating deep network training by reducing internal covariate shift, arXiv preprint, 2015. arXiv: 1502.03167.
- 23.Wu Y, He K. Group normalization//Proceedings of the European Conference on Computer Vision(ECCV), Munich: IEEE, 2018: 3-19.
- 24.Moore R C, DeNero J. L1 and L2 regularization for multiclass hinge loss models//Symposium on Machine Learning in Speech and Language Processing, Bellevue: ISCA, 2013. CiteSeerX. psu: 10.1. 1.296. 5923.
- 25.Tustison N J, Gee J C. Introducing dice, jaccard, and other label overlap measures to ITK, Insight J, 2009. http://hdl.handle.net/10380/3141.
- 26.Yushkevich P A, GAO Yang, Gerig G. ITK-SNAP: an interactive tool for semi-automatic segmentation of multi-modality biomedical images//2016 38th Annual International Conference of the IEEE Engineering in Medicine and Biology Society (EMBC), Florida: IEEE, 2016: 3342-3345.
- 27.Yushkevich P A, Gerig G ITK-SNAP: an intractive medical image segmentation tool to meet the need for expert-guided segmentation of complex medical images. IEEE Pulse. 2017;8(4):54–57. doi: 10.1109/MPUL.2017.2701493. [DOI] [PubMed] [Google Scholar]
- 28.Kingma D P, Ba J. Adam: a method for stochastic optimization, arXiv preprint, 2014. arXiv: 1412.6980.
- 29.LeCun Y, Bengio Y, Hinton G Deep learning. nature. 2015;521(7553):436–444. doi: 10.1038/nature14539. [DOI] [PubMed] [Google Scholar]
- 30.Milletari F, Navab N, Ahmadi S A. V-net: fully convolutional neural networks for volumetric medical image segmentation//2016 Fourth International Conference on 3D Vision (3DV), Stanford: IEEE, 2016: 565-571.





