Abstract
最近,深度学习在医学图像任务中取得了令人瞩目的成果。然而,这种方法通常需要大规模的标注数据,而医学图像的标注成本较高,因此如何从有限的标注数据中进行高效学习是一个难题。目前,常用的两种方法是迁移学习和自监督学习,然而这两种方法在多模态医学图像中的研究却很少,因此本研究提出了一种多模态医学图像对比学习方法。该方法将同一患者不同模态的图像作为正样本,有效增加训练过程中的正样本数量,有助于模型充分学习病灶在不同模态图像上的相似性和差异性,从而提高模型对医学图像的理解能力和诊断准确率。常用的数据增强方法并不适合多模态图像,因此本文提出了一种域自适应反标准化方法,借助目标域的统计信息对源域图像进行转换。本研究以两个不同的多模态医学图像分类任务对本文方法展开验证:在微血管浸润识别任务中,本文方法获得了(74.79 ± 0.74)%的准确率和(78.37 ± 1.94)%的F1分数,相比其它较为熟知的学习方法有所提升;对于脑肿瘤病理分级任务,本文方法也取得了明显的改进。结果表明,本文方法在多模态医学图像数据上取得了良好的结果,可为多模态医学图像的预训练提供一种参考方案。
Keywords: 自监督学习, 多模态医学图像, 疾病诊断, 域自适应反标准化
Abstract
Recently, deep learning has achieved impressive results in medical image tasks. However, this method usually requires large-scale annotated data, and medical images are expensive to annotate, so it is a challenge to learn efficiently from the limited annotated data. Currently, the two commonly used methods are transfer learning and self-supervised learning. However, these two methods have been little studied in multimodal medical images, so this study proposes a contrastive learning method for multimodal medical images. The method takes images of different modalities of the same patient as positive samples, which effectively increases the number of positive samples in the training process and helps the model to fully learn the similarities and differences of lesions on images of different modalities, thus improving the model's understanding of medical images and diagnostic accuracy. The commonly used data augmentation methods are not suitable for multimodal images, so this paper proposes a domain adaptive denormalization method to transform the source domain images with the help of statistical information of the target domain. In this study, the method is validated with two different multimodal medical image classification tasks: in the microvascular infiltration recognition task, the method achieves an accuracy of (74.79 ± 0.74)% and an F1 score of (78.37 ± 1.94)%, which are improved as compared with other conventional learning methods; for the brain tumor pathology grading task, the method also achieves significant improvements. The results show that the method achieves good results on multimodal medical images and can provide a reference solution for pre-training multimodal medical images.
Keywords: Self-supervised learning, Multimodal medical image, Disease diagnosis, Domain adaptive denormalization
0. 引言
最近,深度学习在各种医学图像任务中表现出良好的性能,如病灶分割、疾病诊断和疗效预测[1]。然而,深度学习在医学图像中的应用也面临一些挑战,其中一个主要问题就是缺乏充足的标注数据。医学图像的标注通常需要专业医生才能完成,任务难度较大且成本较高,因此如何从有限的标注数据中学习有代表性的特征成为一个迫切需要解决的问题。
目前,从有限的标注数据集中学习有代表性的特征主要有两种常见的方法:一种方法是在大规模公共数据集上进行有监督的预训练[2-4];另一种方法是在没有标注的数据集上使用自监督学习进行自监督预训练,然后在目标数据集上对预训练的模型进行有监督的微调[5-6]。然而,这些方法在应用于医学图像任务时,存在一些局限性。对于第一种方法,将公共数据集上训练的模型直接迁移到医学图像任务上,似乎没有考虑到由于自然图像和医学图像之间的分布差异而可能导致的模型性能下降的问题。这种方法是次优的,因为医学图像理解通常需要细粒度的视觉特征,这些特征与识别自然图像中物体所需的特征完全不同。例如,Raghu等[7]研究发现,与简单的随机初始化相比,大规模视觉识别挑战赛数据集(ImageNet)预训练对模型性能的提升几乎没有效果。对于第二种方法,虽然自监督学习已经在一些医学影像任务中取得了成果,如心脏磁共振成像(magnetic resonance imaging,MRI)分割[8]、视网膜疾病诊断[9]和新型冠状病毒感染(corona virus disease 2019,COVID-19)诊断[10],但是每种医学图像都有其自身的噪声和缺陷,这可能导致自监督学习方法的性能受到限制。而多模态医学图像可以提供互补信息[11],例如电子计算机断层扫描(computed tomography,CT)可以提供组织密度信息,MRI可以提供组织构造信息等。通过联合使用多模态医学图像进行自监督学习,可以获得更全面和更准确的信息,从而提高对疾病的理解和诊断,因此研究多模态医学图像的自监督学习具有重要的意义。
多模态医学图像是患者在不同的医疗设备或同一设备的不同参数下获得的,可以为医学图像诊断提供更丰富的信息。在这项研究中,本文提出了一个多模态对比学习框架,可以有效地利用每个患者的多模态医学图像来学习共有的特征表达。本文方法是从一个小批量数据中随机选择两个模态图像,如果这两个模态图像属于一个患者,那么就被视为正样本对。如果这两个模态图像不属于同一个患者,那么就被视为负样本对。然后,将这两个模态的图像分别输入到对应特征编码网络中提取图像特征,并通过投影网络将获得的特征映射到一个特征空间。接下来,通过对比损失使正样本对在特征空间中尽可能地接近,而负样本对则尽可能地远离。最后,将预训练好的特征编码网络迁移到目标任务上进行监督微调,以提高模型在目标数据集上的性能。
有研究表明,使用合理的数据增强方法可以改善自监督学习[12]。目前使用的数据增强方法(裁剪、调整大小、旋转等)通常不能很好地考虑多模态医学图像的特点。由于数据集的统计信息包含分类任务的相关信息,因此本文提出了一种域自适应反标准化方法。该方法利用多模态数据集中各模态的统计信息对其它模态的数据进行数据转换,可以有效扩大各模态样本的数据量,从而提高模型对各模态数据的适应能力。具体而言,首先从多模态医学图像数据集中随机选择两种模态作为源域和目标域,然后对源域和目标域的图像进行标准化,最后利用目标域的统计信息对源域进行反标准化,从而得到转换后的图像。域自适应反标准化方法可以合理地利用各个模态图像的统计信息实现有效地数据扩充。
为了公平地比较有监督预训练、对比学习和本文方法,在两个不同的医学图像数据集上进行了对比实验:①基于肝细胞癌(hepatocellular carcinoma,HCC)多期相MRI的微血管浸润(microvascular infiltration,MVI)识别;②基于脑胶质瘤多模态MRI的病理分级。此外,由于两个数据集的数据量较小,本文在多模态对比学习阶段使用域自适应反标准化方法来扩大数据量。综上,通过本文提出的多模态对比学习算法在两个数据集上进行验证,以期获得良好的性能,或可为多模态医学图像的预训练提供一种参考方案。
1. 相关工作
1.1. 对比学习
对比学习作为自监督学习中一个非常重要的部分,被广泛应用于计算机视觉、自然语言处理等领域。在对比学习中,通常会将两个样本输入到模型中,并使用某种度量函数来计算它们之间的相似度或距离。这个度量函数可以是欧式距离、余弦相似度等。如果两个样本属于同一类别,则目标是使它们的相似度尽可能高;如果属于不同类别,则目标是使它们的相似度尽可能低。在计算机视觉领域,基于对比学习的方法已经在大规模公共数据集上取得了最先进的性能。例如,简明对比学习(simple contrastive learning of representations,SimCLR)框架[12]、动量对比学习(momentum contrastive learning,MOCO)框架[13]、自举潜变量学习(bootstrap your own latent learning,BYOL)框架[14]和孪生表示学习(simple siamese representation learning,SimSiam)框架[15]。
最近,对比学习在医学图像任务中已经取得了一些成果。例如,Chaitanya等[16]引入了一个基于混合对比损失的自监督学习分割模型,该模型能够学习局部和全局图像表征。Sowrirajan等[17]使用MOCO在大型X光胸片数据集(chest expert,CheXpert)[18]上进行预训练,然后对模型的分类性能进行评估。He等[10]提出了一种自我迁移对比学习框架(self-supervised learning and transfer learning,Self-Trans),以提高CT影像中COVID-19的分类准确率。Aziz等[19]提出了一种多实例对比学习方法(multi-instance contrastive learning,MICLe),以提高数码相机图像中皮肤病的分类精度。这些工作都表明在医学图像上采用对比学习方法可以提取到有代表性的特征,这些特征有助于模型对医学图像的理解,从而提高模型对疾病的诊断能力。
1.2. 医学多模态学习
与单模态医学图像相比,多模态医学图像通常由不同的医学成像模态组成,例如CT、MRI和正电子发射断层(positron emission tomography,PET)等,这些模态之间存在着丰富的信息交叉和相互依赖关系。研究人员已经开始关注多模态医学图像的应用,例如,Hervella等[9]提出了一个基于自监督多模态重建(self-supervised multimodal reconstruction,SMR)的视网膜计算机辅助诊断(computer-aided diagnosis,CAD)系统,该系统利用互补成像模态之间的多模态重建实现,并取得了比单模态更好的实验结果。Windsor等[20]介绍了一个基于对比学习的多模态医学图像配准框架,该框架可以精确地配准同一患者的不同模态的医学图像。Taleb等[21]提出了一种自监督学习方法,它可以用来匹配无标签的医学图像和遗传数据,以揭示图像和遗传数据之间的关系。这些工作表明,采用多模态数据进行学习可以取得比单模态更好的效果。本文采用对比学习方法在多模态医学图像数据集上进行实验,旨在学习不同模态图像之间病灶的相似性,以获得更全面、准确的信息,从而提高对疾病检测和诊断的能力。
1.3. 数据增强
常用的数据增强方法可以分为两类:一类是标签保持的数据增强方法,例如:对图像进行几何变换,包括翻转、旋转、移动、裁剪、扭曲、缩放等各种操作;或者对图像的颜色进行变换,常见的有添加噪声、模糊、擦除、填充等。然而,这类方法并不是专门为多模态医学图像设计的。另一类是标签扰动的数据增强方法,例如,Zhang等[22]通过将训练集中的图像和标签都按一定比例融合,生成新的数据。Yun等[23]通过随机删除一个图像中的矩形区域,并用另一个图像相同位置的像素值填充的方式进行数据扩充,同时根据像素的比例份额分配标签。虽然标签扰动的数据增强方法可以用于多模态医学图像数据,但是它们改变了数据的分布,可能会引入噪声和误差,从而影响模型的性能和稳定[23]。本研究针对多模态数据专门设计了一种域自适应反标准化方法,该方法能够有效地使用不同模态数据的统计信息对图像进行转换,从而在不改变整个数据分布的情况下实现数据扩展。这是一种新型的多模态数据增强方法,可用于提高多模态医学图像分类的性能。
2. 方法
本文提出的方法主要包括两个阶段,如图1所示。在多模态对比学习阶段,使用提出的多模态对比学习方法对多个模态的图像进行预训练。在监督微调阶段,将训练好的模型迁移到目标数据集上进行有监督微调。接下来,在以下章节详细描述本文方法。
图 1.
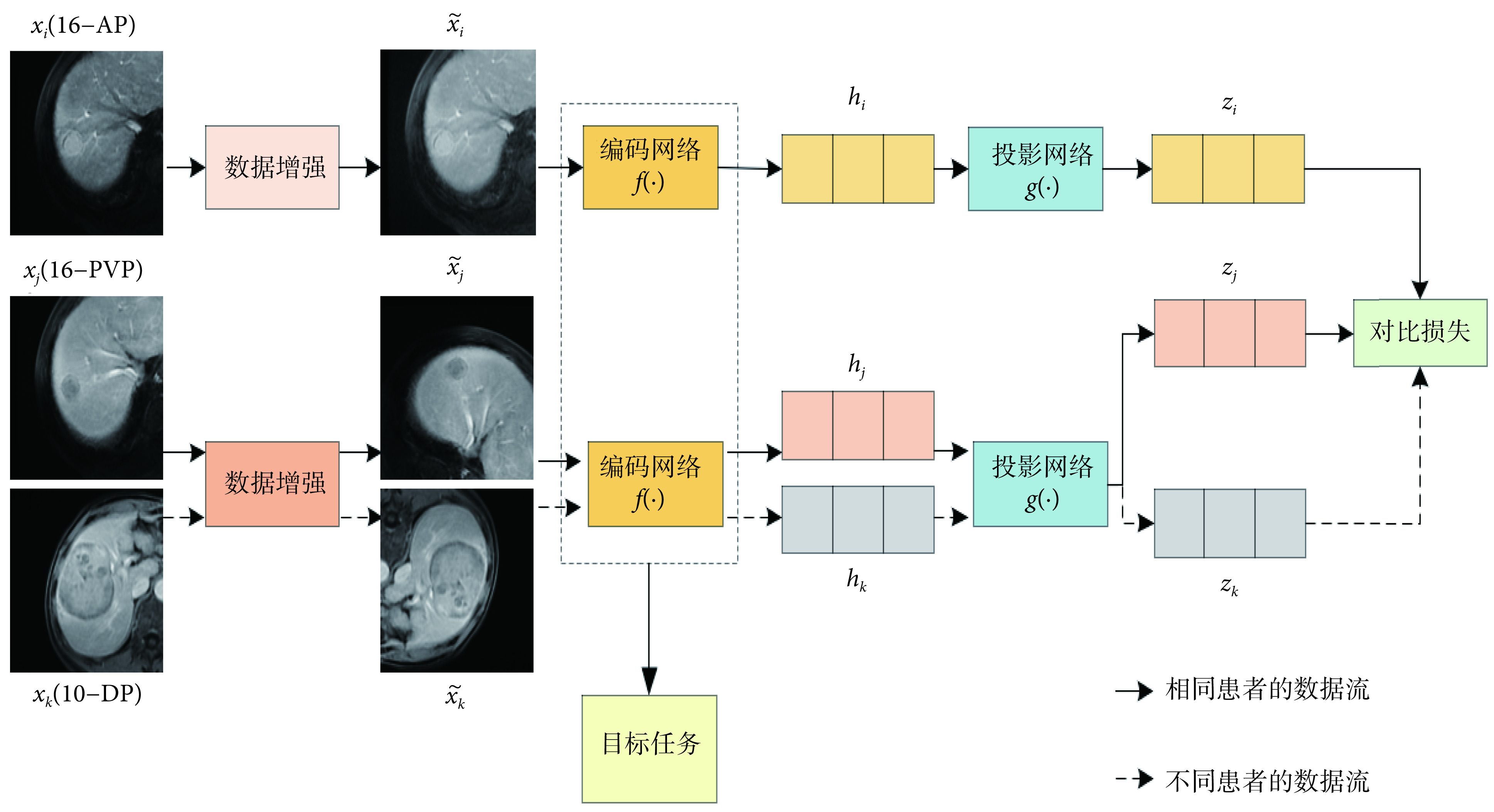
Framework diagram of multimodal contrastive learning method
多模态对比学习方法的框架图
2.1. 多模态对比学习
在对多模态医学图像数据集进行训练时,随机选择一个有N个患者的小批次数据,其中每个患者X={x1, x2, ..., xm}可能包含多个模态医学图像。假设,由于患者的不同,m可能是不同的。在多模态对比学习阶段,从N个患者的小批次数据中随机选择两个模态的图像,如果这两个模态的图像属于同一个患者,如图1中xi和xj分别属于数据集中第16号患者的动脉期(arterial phase,AP)(16-AP)和静脉期(portal venous phase,PVP)(16-PVP),那么xi和xj就构成一个正样本对。如果这两个模态的图像属于不同患者,如图1中xi和xk分别属于数据集中16-AP和第10号患者的延迟期(delayed phase,DP)(10-DP),那么xi和xk就构成一个负样本对。其次,将这两个模态的图像xi和xj(或xk)输入数据增强模块,对它们分别进行随机的图像增强操作,得到增强后的图像 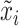 和
和 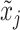 (或
(或  )。然后,将
)。然后,将 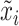 和
和 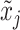 (或
(或 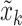 )分别输入对应的特征编码网络f(∙),以提取图像特征hi和hj(或hk)。接着,将这两个特征hi和hj(或hk)分别输入到投影网络g(∙),它们被映射到同一特征空间,并生成两个特征向量zi和zj(或zk)。最后,采用对比损失来缩短zi和zj之间的距离,同时扩大zi和zk之间的距离。通过这种隐式的多模态融合方法使模型学习病灶在不同模态之间的相似性和差异性,有助于更好地理解不同模态之间的关系,从而提高模型对医学图像的理解能力和诊断准确率。
)分别输入对应的特征编码网络f(∙),以提取图像特征hi和hj(或hk)。接着,将这两个特征hi和hj(或hk)分别输入到投影网络g(∙),它们被映射到同一特征空间,并生成两个特征向量zi和zj(或zk)。最后,采用对比损失来缩短zi和zj之间的距离,同时扩大zi和zk之间的距离。通过这种隐式的多模态融合方法使模型学习病灶在不同模态之间的相似性和差异性,有助于更好地理解不同模态之间的关系,从而提高模型对医学图像的理解能力和诊断准确率。
本文提出的多模态对比学习框架由以下几部分组成:
(1)数据增强。数据增强的作用是将输入图像对xi和xj随机转化为增强的图像对  和
和 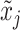 。本研究中使用的数据增强操作包括随机缩放裁剪、随机颜色失真,以及旋转90 º、180 º和270 º。
。本研究中使用的数据增强操作包括随机缩放裁剪、随机颜色失真,以及旋转90 º、180 º和270 º。
(2)编码网络。编码网络的作用是将增强的图像对  和
和 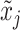 映射成一对特征向量hi和hj。在实验过程中,使用不同结构的网络进行实验,包括通道改组神经网络(shuffle neural network,ShuffleNet)[24]、残差神经网络(residual neural network,ResNet)[25]和带移动窗口的转换器(shifted windows transformer,Swin Transformer)[26],以验证本文方法的有效性。
映射成一对特征向量hi和hj。在实验过程中,使用不同结构的网络进行实验,包括通道改组神经网络(shuffle neural network,ShuffleNet)[24]、残差神经网络(residual neural network,ResNet)[25]和带移动窗口的转换器(shifted windows transformer,Swin Transformer)[26],以验证本文方法的有效性。
(3)投影网络。投影网络的作用是将编码的特征对hi和hj投影到一个向量空间中,然后使用对比损失来评估投影特征对zi和zj的相似度。在实验中,使用两个全连接层来组成投影网络,投影网络的隐藏层和输出层的维度分别为256和128。
(4)对比损失。对比损失的作用是缩短正样本对特征zi和zj之间的距离,同时扩大负样本对zi和zk之间的距离。这样可以使模型学习到肿瘤在不同模态图像之间共同的特征,从而提高模型的表达能力和准确性。本文中对比损失Li, j的定义如式(1)所示:
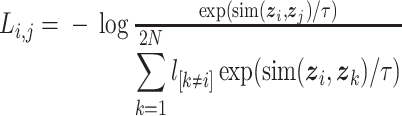 |
1 |
其中,sim(∙, ∙)是两个向量zi和zj之间的余弦相似度; ,当k和i相等时为0,不等时为1;τ是一个温度标量。
,当k和i相等时为0,不等时为1;τ是一个温度标量。
2.2. 监督微调
将多模态医学图像数据集上使用对比学习训练的模型迁移到目标任务上进行监督微调,以提高模型在目标任务上的表现。在实验中,冻结了在多模态对比学习阶段训练的特征编码网络的权重,并在特征编码网络之后添加了一个目标任务的分类网络。该分类网络由两个全连接层组成,中间是批量归一化(batch normalization,BN)和带泄漏修正线性单元(leaky rectified linear unit,Leaky ReLU)激活函数。这两个全连接层的维度为256和2。它们的初始化参数是由恺明(kaiming)[27]初始化方法产生的。
2.3. 域自适应反标准化
在本节中,将仔细介绍域自适应反标准化方法。具体而言,将每一个模态视为一个域,并对每个域的图像进行标准化处理,然后利用其它域的统计信息对当前域的标准化数据进行反标准化处理,从而实现不同域数据之间的适应。
本文使用目标域数据的统计信息来扩展源域数据,对于任何多模态数据集,将任意两个模态作为源域Ds和目标域Dt,分别用M和N表示Ds和Dt的样本数量。整个域自适应反标准化方法的详细描述如下:
步骤(1):分别统计源域Ds中每张图像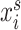 的平均值μs和标准差σs,以及目标域Dt中每张图像
的平均值μs和标准差σs,以及目标域Dt中每张图像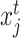 的平均值μt和标准差σt,其定义如式(2)和式(3)所示:
的平均值μt和标准差σt,其定义如式(2)和式(3)所示:
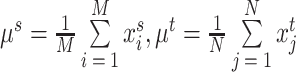 |
2 |
 |
3 |
步骤(2):根据步骤(1)中计算的平均值μs和标准差σs对源域Ds中每张图像 进行标准化处理,得到标准化后的图像
进行标准化处理,得到标准化后的图像 。同样,对目标域Dt中每张图像
。同样,对目标域Dt中每张图像 采取同样的标准化处理,得到标准化后的图像
采取同样的标准化处理,得到标准化后的图像 。标准化的计算如式(4)所示:
。标准化的计算如式(4)所示:
 |
4 |
步骤(3):将源域Ds中每张标准化的图像 乘以目标域的标准差σt,再加上目标域的平均值μt,这样就可以得到反标准化的图像
乘以目标域的标准差σt,再加上目标域的平均值μt,这样就可以得到反标准化的图像 。同样,对目标域Dt中每张标准化的图像
。同样,对目标域Dt中每张标准化的图像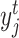 进行同样的反标准化处理,就可以得到反标准化的图像
进行同样的反标准化处理,就可以得到反标准化的图像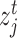 。反标准化的计算如式(5)所示:
。反标准化的计算如式(5)所示:
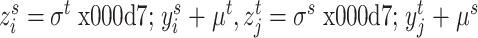 |
5 |
3. 实验数据和设置
3.1. 数据和预处理
本文方法在两个不同的医学图像任务上进行了实验。第一个任务是在三个期相的MRI上诊断HCC的MVI。第二个任务是在四个模态的MRI上对脑肿瘤进行病理分级。
对于MVI识别任务,实验数据集由大连医科大学第一附属医院放射科提供,并且数据集中的所有患者都经组织病理学证实为MVI。该数据集包含了261名HCC患者三种期相的MRI,分别为AP、PVP、DP。其中182名患者为阴性,79名患者为阳性。整个数据集由两位具有10年MRI诊断经验的放射科医生进行注释,并由另一位具有20年经验的资深放射科医生进行校准。该研究经大连医科大学附属第一医院医学伦理委员会审查获得批准,并授权可以使用数据集中所有影像资料数据(批文编号:PJ-KS-KY-2019-167)。
在实验中,将数据集按照8:2的比例随机划分为训练集和测试集。在多模态对比学习和监督微调两个阶段,训练集有1 881张图像,包括567张阳性图像和1 314张阴性图像;测试集有468张图像,包括144张阳性图像和324张阴性图像。对于模型的输入,从每个患者的三个期相MRI中选择肿瘤面积最大的切片及其上下两片。如图2所示,可以看到MVI数据集中阴性和阳性的多期相MRI。
图 2.
Example of multi-phase MRI in MVI dataset
MVI数据集中多期相MRI示例图
对于脑肿瘤病理分级任务,本文采用2019年多模态脑瘤分割挑战赛(multimodal brain tumor segmentation challenge 2019, Brats2019)数据库作为实验数据来源。该数据库共包含626个脑瘤患者,其中训练集有335个患者,验证集有125个患者,测试集有166个患者。由于验证集和测试集没有公开发布,在实验中只使用训练集中335个患者的多模态MRI,其中259名患者为高级别胶质瘤(high-grade glioma,HGG),76名患者为低级别胶质瘤(low-grade glioma,LGG)。每个患者都有四个模态的MRI,分别为T1、T2、液体衰减反转恢复(fluid-attenuated inversion recovery,FLAIR)序列和对比增强T1加权(contrast-enhanced T1-weighted, T1ce)序列。每个模态的MRI有155张切片图像,每张切片图像的大小为240×240。本研究已得到Brats2019数据库创建机构的授权,并可免费使用数据库中所有数据资料。
在实验中,将Brats2019训练集中335个患者以8:2的比例随机划分为训练集和测试集,其中训练集有267个患者,测试集有68个患者。在多模态对比学习和监督微调两个阶段,训练集有3 204张图像,包括2 484张HGG图像和720张LGG图像;测试集有816张图像,包括624张HGG图像和192张LGG图像。对于模型的输入,与MVI一样,选择肿瘤面积最大的切片及其上下两片。如图3所示,可以看到Brats2019数据集中HGG和LGG的多模态MRI。
图 3.

Example of multimodal MRI in Brats2019 dataset
Brats2019数据集中多模态MRI示例图
3.2. 实验环境及参数设置
在实验过程中,所有模型都是使用开源机器学习库PyTorch 1.8.1(Facebook Inc.,美国)实现,这些模型在两块显存为11 G的英伟达显卡(RTX 2080 Ti,NVIDIA Inc.,美国)上并行训练。在多模态对比学习阶段,初始学习率为0.001,批大小为64,训练周期为500个迭代次数(epoch)。在训练过程中,使用自适应边界优化(adaptive bound optimization,adabound[28])方法来优化模型参数,并使用自适应的学习率更新策略:当训练集上的损失在20个epoch中没有减少时,学习率被减小到原来的0.5倍。在监督微调阶段,由于分类网络只有两个全连接层,所以将初始学习率值设定更小,并以较小的epoch进行训练,以防止模型过拟合。具体而言,分类网络初始化的学习率为0.000 3,批大小为64,训练周期为200个epoch,优化器为adabound,还使用了与多模态对比学习阶段相同的学习率自适应更新策略。由于实验数据存在一定程度上的不确定性,因此在课题研究中进行了三次实验并取平均值来提高结果的可靠性。
在多模态对比学习阶段和监督微调阶段都使用了在线数据增强方法,它包括随机缩放裁剪、旋转和颜色失真。具体而言,在两个数据集上进行随机缩放裁剪的裁剪框大小为224×224,对训练集中的所有图像进行90 º、180 º和270 º旋转,还将训练集中的图像以0.8的概率进行颜色转换,其中亮度、对比度和饱和度变为原来的0.4倍,色调变为原来的0.2倍。此外,对数据集中的所有图像都进行标准化处理,即每张图像都进行减均值和除以标准差。在多模态对比学习阶段,不仅使用在线数据增强方法(其中包括缩放裁剪、旋转和颜色失真)对原始数据进行实时变换来扩展训练集,以使模型具有更好的鲁棒性和泛化能力;还使用离线数据增强方法(域自适应反标准化)在模型训练之前,通过对原始数据进行一系列的处理,生成新的虚拟数据集,并将其添加到原始数据集中,从而扩大数据量和多样性。
3.3. 评价标准
本文采用以下五个指标来评估模型的性能:①准确率(accuracy,ACC),表示正确预测的样本占总样本的比例;②受试者工作特征(receiver operating characteristic,ROC)曲线下面积(area under ROC curve,AUC);③精准率(precision, PRE),表示所有预测为正类的样本中实际为正类样本所占的比例;④召回率(recall,REC),表示实际为正类的样本中预测为正类样本所占的比例;⑤F1分数(F1 score, F1)综合考虑PRE和REC,是两者的谐波平均值。
4. 实验结果与分析
本文方法在MVI和Brats2019两个数据集上进行了一系列实验,并与基线模型进行了比较,其中包括随机初始化(random)、迁移学习(transfer)、SimCLR、BYOL、SimSiam、MICLe、Self-Trans和SMR。其中,random和transfer属于监督学习,SimCLR、BYOL、SimSiam、MICLe和Self-Trans属于单模态对比学习,而SMR属于多模态对比学习。同时,在三个不同结构的网络上验证了域自适应反标准化方法的有效性。
4.1. 本文方法的表现
为了验证本文方法的有效性,在MVI数据集上进行了一系列实验,量化实验结果如表1所示。可以看出,本文方法的实验结果不仅比单模态自监督学习方法要好,而且相较于多模态自监督学习方法也有一定的优势。具体而言,和其它对比学习方法相比,本文方法在ACC、AUC、REC和F1上均有所提升。这表明本文提出的多模态对比学习方法可以有效地提高模型分类性能。此外,还可以发现SimCLR、BYOL、SimSiam、MICLe、Self-Trans、SMR以及本文方法都取得了比radom和transfer更好的结果,这说明对比学习的训练策略是有效的,对比学习方法能够从图像中学习有意义的特征表达,从而提高模型在MVI诊断任务上的表现。
表 1. Performance comparison of different pre-training methods on the MVI dataset.
不同预训练方法在MVI数据集上的性能比较
| 算法 | ACC | AUC | REC | PRE | F1 |
| random | (66.03 ± 3.39)% | (68.56 ± 0.88)% | (66.97 ± 6.24% | (64.21 ± 4.66)% | (65.15 ± 2.99)% |
| transfer | (70.51 ± 1.11)% | (72.42 ± 1.02)% | (63.22 ± 3.87)% | (76.27 ± 1.94)% | (68.81 ± 1.99)% |
| SimCLR | (73.40 ± 1.36)% | (77.28 ± 2.26)% | (81.91 ± 0.15)% | (72.04 ± 1.51)% | (76.61 ± 0.91)% |
| BYOL | (72.44 ± 0.65)% | (74.24 ± 2.91)% | (79.95 ± 7.61)% | (72.45 ± 2.45)% | (75.77 ± 4.27)% |
| SimSiam | (70.94 ± 2.43)% | (70.43 ± 2.42)% | (81.68 ± 4.48)% | (69.27 ± 4.78)% | (74.72 ± 2.93)% |
| MICLe | (73.50 ± 2.25)% | (81.13 ± 0.31)% | (81.67 ± 5.88)% | (64.57 ± 2.82)% | (71.97 ± 1.09)% |
| Self-Trans | (71.36 ± 0.74)% | (81.04 ± 2.31)% | (86.91 ± 1.68)% | (66.31 ± 0.79)% | (74.87 ± 0.84)% |
| SMR | (72.86 ± 1.62)% | (82.87 ± 1.83)% | (86.98 ± 1.32)% | (63.97 ± 2.12)% | (73.70 ± 1.08)% |
| 本文算法 | (74.79 ± 0.74)% | (83.60 ± 1.83)% | (87.03 ± 4.25)% | (71.48 ± 1.10)% | (78.37 ± 1.94)% |
为了进一步验证本文方法的有效性,本文还在Brats2019数据集上进行了一系列对比实验,量化实验结果如表2所示。可以看出,与其它方法相比,本文方法在ACC,REC,PRE和F1四个评价指标上均有所提升。实验结果表明,本文方法对HGG患者和LGG患者的分类更加准确,从而说明本文方法的分类结果是稳定可靠的。此外,在Brats2019数据集上,基于对比预训练的方法和基于迁移学习的方法的性能是相当的,而本文方法比前两者有明显优势。这一结果归功于多模态对比学习策略,它将同一患者的多个模态视为正样本,因此可以充分学习各个模态之间的共有特征,从而改善疾病诊断的性能。实验结果表明,本文方法还可以更好地应用于其它多模态医学图像中的疾病诊断任务,并取得有竞争力的结果。
表 2. Performance comparison of different pre-training methods on the Brats2019 dataset.
不同预训练方法在Brats2019数据集上的性能比较
| 算法 | ACC | AUC | REC | PRE | F1 |
| random | (66.42 ± 0.56)% | (69.22 ± 1.57)% | (72.17 ± 5.65)% | (66.11 ± 1.70)% | (68.57 ± 2.11)% |
| transfer | (70.71 ± 0.21)% | (74.89 ± 0.72)% | (79.07 ± 0.52)% | (68.77 ± 1.55)% | (73.35 ± 1.20)% |
| SimCLR | (68.75 ± 0.37)% | (71.23 ± 0.66)% | (77.48 ± 4.00)% | (67.21 ± 1.58)% | (71.64 ± 1.41)% |
| BYOL | (68.99 ± 2.09)% | (75.84 ± 1.04)% | (79.55 ± 3.70)% | (65.58 ± 3.67)% | (72.44 ± 3.65)% |
| SimSiam | (69.49 ± 0.74)% | (74.40 ± 2.63)% | (82.75 ± 5.16)% | (67.00 ± 2.08)% | (73.66 ± 1.97)% |
| MICLe | (67.89 ± 1.40)% | (70.70 ± 0.81)% | (83.15 ± 5.45)% | (65.68 ± 2.10)% | (73.04 ± 2.43)% |
| Self-Trans | (69.73 ± 1.74)% | (72.40 ± 3.34)% | (83.38 ± 5.78)% | (67.59 ± 0.32)% | (74.41 ± 2.37)% |
| SMR | (69.12 ± 1.33)% | (72.71 ± 1.75)% | (79.09 ± 2.24)% | (66.30 ± 4.14)% | (71.79 ± 1.87)% |
| 本文算法 | (73.35 ± 0.78)% | (75.72 ± 6.32)% | (84.52 ± 6.19)% | (70.08 ± 1.98)% | (76.20 ± 1.19)% |
4.2. 多模态对比学习策略消融实验
为了验证多模态对比学习策略的有效性,在MVI数据集上使用不同结构的网络进行了消融实验,包括ShuffleNet、ResNet和Swin Transformer。实验结果如表3所示,ResNet*、ShuffleNet*和Swin Transformer*表示采用多模态对比学习训练策略,而ResNet、ShuffleNet和Swin Transformer表示采用普通的对比学习训练策略。可以看到,在三种不同结构的网络上采用多模态对比学习训练策略取得了明显的提升。具体而言,在MVI数据集上采用多模态对比学习的ResNet*在ACC、AUC、REC、PRE和F1上分别比没有采用多模态对比学习的ResNet有所提升。同样,相较于ShuffleNet和Swin Transformer,采用多模态对比学习策略的ShuffleNet*和Swin Transformer*结果也有所改进。实验结果表明,在多模态医学图像数据集上采用提出的多模态对比学习方法进行预训练有助于提高模型的性能。同时,本文方法具有良好的通用性和可扩展性,可以应用于各种不同结构的网络。
表 3. Results of ablation experiments using multimodal contrast learning for different encoders on the MVI dataset.
在MVI数据集上不同编码网络采用多模态对比学习的消融实验结果
| 编码网络 | ACC | AUC | REC | PRE | F1 |
| ResNet | (73.29 ± 0.98)% | (76.07 ± 2.62)% | (82.11 ± 0.36)% | (71.60 ± 1.30)% | (76.45 ± 0.70)% |
| ResNet* | (75.00 ± 0.64)% | (82.79 ± 2.22)% | (88.37 ± 2.90)% | (72.25 ± 0.89)% | (79.39 ± 0.72)% |
| ShuffleNet | (71.58 ± 3.03)% | (76.82 ± 5.65)% | (70.75 ± 3.82)% | (75.70 ± 3.50)% | (72.97 ± 1.86)% |
| ShuffleNet* | (72.65 ± 0.74)% | (78.24 ± 1.71)% | (83.67 ± 3.20)% | (69.14 ± 4.52)% | (75.43 ± 1.70)% |
| Swin Transformer | (71.15 ± 0.64)% | (75.29 ± 2.21)% | (80.48 ± 1.60)% | (69.15 ± 3.51)% | (73.96 ± 2.48)% |
| Swin Transformer* | (72.44 ± 1.93)% | (77.63 ± 1.32)% | (87.45 ± 2.27)% | (69.37 ± 2.43)% | (77.18 ± 2.19)% |
4.3. 域自适应反标准化方法消融实验
在MVI数据集上验证本文提出的域自适应反标准化方法的消融实验结果如图4所示。ResNet#、ShuffleNet#和Swin Transformer#表示采用域自适应反标准化方法训练,ResNet、ShuffleNet和Swin Transformer表示没有采用域自适应反标准化方法训练。在MVI数据集上使用域自适应反标准化方法训练的ResNet#、ShuffleNet#和Swin Transformer#的损失曲线都低于没有使用的ResNet、ShuffleNet和Swin Transformer的损失曲线。因此,这表明提出的域自适应反标准化方法可以提高模型在对比学习阶段的学习能力。此外,使用ResNet作为特征编码网络的损失收敛最低,其次是ShuffleNet和Swin Transformer。因此,实验结果表明在MVI数据集上使用ResNet可以更好地提取图像特征。
图 4.
Comparison of loss curves using domain adaptive denormalization on the MVI train set
在MVI训练集上使用域自适应反标准化的损失曲线的比较
在MVI数据集上不同编码网络使用域自适应反标准化方法进行的消融实验结果如表4所示。可以看到,采用域自适应反标准化的ResNet#、ShuffleNet#和Swin Transformer#比没有使用域自适应反标准化的ResNet、ShuffleNet和Swin Transformer有一定的提升。实验结果进一步表明在多模态医学图像数据集上采用提出的域自适应反标准化方法对训练集进行扩充,有助于提升模型的性能。
表 4. Experimental results of ablation on MVI dataset with different coding networks using domain adaptive denormalization.
在MVI数据集上不同编码网络使用域自适应反标准化的消融实验结果
| 编码网络 | ACC | AUC | REC | PRE | F1 |
| ResNet | (73.98 ± 1.82)% | (74.83 ± 1.05)% | (78.45 ± 4.13)% | (76.72 ± 0.11)% | (77.54 ± 1.97)% |
| ResNet# | (75.00 ± 0.64)% | (82.79 ± 2.22)% | (88.37 ± 2.90)% | (72.25 ± 0.89)% | (79.39 ± 0.72)% |
| ShuffleNet | (71.93 ± 2.59)% | (77.82 ± 3.27)% | (84.67 ± 3.50)% | (67.12 ± 4.38)% | (70.33 ± 3.19)% |
| ShuffleNet# | (72.65 ± 0.74)% | (78.24 ± 1.71)% | (83.67 ± 3.20)% | (69.14 ± 4.52)% | (75.43 ± 1.70)% |
| Swin Transformer | (70.98 ± 1.61)% | (71.36 ± 0.44)% | (81.36 ± 1.76)% | (74.66 ± 1.90)% | (77.73 ± 0.33)% |
| Swin Transformer# | (72.44 ± 1.93)% | (77.63 ± 1.32)% | (87.45 ± 2.27)% | (69.37 ± 2.43)% | (77.18 ± 2.19)% |
4.4. 编码网络特征维度消融实验
本文探究了多模态对比学习阶段特征编码网络的维度对实验结果的影响。实验中,在MVI数据集上采用ResNet作为特征编码网络,将特征维度分别设置为64、128、256和512进行消融实验。实验结果如表5所示,当特征维度为128时,模型预测的AUC和PRE达到最大值;当特征维度为256时,模型预测的ACC、REC和F1达到最大值,并且REC和F1相比其它维度有明显的优势。综合比较而言,将特诊编码维度设置为256,可以取得更好的实验结果。
表 5. Experimental results on ablation of different feature dimensions in encoded network on the MVI dataset.
在MVI数据集上编码网络中不同特征维度的消融实验结果
| 特征维度 | ACC | AUC | REC | PRE | F1 |
| 64 | (70.59 ± 2.23)% | (77.63 ± 5.17)% | (71.37 ± 3.41)% | (67.48 ± 3.41)% | (69.36 ± 3.35)% |
| 128 | (74.86 ± 0.89)% | (82.88 ± 2.22)% | (75.53 ± 3.40)% | (72.90 ± 1.50)% | (74.18 ± 2.42)% |
| 256 | (75.00 ± 0.64)% | (82.79 ± 2.22)% | (88.37 ± 2.90)% | (72.25 ± 0.89)% | (79.39 ± 0.72)% |
| 512 | (73.36 ± 2.48)% | (78.83 ± 2.55)% | (74.15 ± 8.42)% | (66.49 ± 4.45)% | (70.09 ± 6.82)% |
5. 结论
针对大规模医学图像标注数据难以获取的问题,以及迁移学习和自监督学习在多模态医学图像任务中少有研究的现状,本文提出了一种多模态对比学习方法。该方法将同一患者多模态的医学图像作为正样本,不同患者的多模态医学图像作为负样本,这样可以有效增强训练过程中的正样本数量,使模型尽可能充分地学习多模态医学图像上病灶的相似性,从而提升模型对疾病的分类准确率。此外,由于实验数据规模较小,而传统的数据增强方法并不适用于多模态医学图像,因此本文提出了一种域自适应反标准化方法。该方法采用目标域的统计信息对源域进行数据扩增,从而在不改变整个数据集分布的情况下有效地扩充数据量。最后,在两个数据集(MVI、Brats2019)上进行了广泛的实验,实验结果表明,本文提出的多模态对比学习方法可以在两个数据集上实现良好的性能。该方法为多模态医学图像数据集的预训练提供了一个可行的参考方案,推动了深度学习在多模态医学图像领域的进一步发展。
在未来的工作中,计划将患者的临床数据融入本文提出的多模态对比学习方法中,以高效利用患者的多模态数据进行融合来提升疾病的诊断能力。此外,本文提出的域自适应反标准化方法是一种通用算法,很容易推广到其它的多模态数据集中。
重要声明
利益冲突声明:本文全体作者均声明不存在利益冲突。
作者贡献说明:文含主要负责数据处理、算法设计与实现以及撰写论文,赵莹主要负责数据的收集和整理工作,蔡秀定主要负责算法设计和提出修改意见,付忠良参与了算法设计和指导论文写作,刘爱连和姚宇提供了基金支持和提出修改意见。
伦理声明:本研究通过了大连医科大学附属第一医院伦理委员会的审批(批文编号:PJ-KS-KY-2019-167)
Funding Statement
国家自然科学基金面上项目(61971091);四川省科技计划项目(2021YFG0129);大连市青年科技之星项目支持计划(2022RQ074)
National Natural Science Foundation of China; Sichuan Science and Technology Plan Project; Dalian Young Science and Technology Star Project Support Program
References
- 1.Litjens G, Kooi T, Bejnordi B E, et al A survey on deep learning in medical image analysis. Medical image analysis. 2017;42:60–88. doi: 10.1016/j.media.2017.07.005. [DOI] [PubMed] [Google Scholar]
- 2.Maghdid H S, Asaad A T, Ghafoor K Z, et al. Diagnosing COVID-19 pneumonia from X-ray and CT images using deep learning and transfer learning algorithms. arXiv preprint, 2021, arXiv: 2004.00038.
- 3.Yang Y, Li X, Wang P, et al Multi-Source transfer learning via ensemble approach for initial diagnosis of Alzheimer’s disease. IEEE Journal of Translational Engineering in Health and Medicine. 2020;8:1400310. doi: 10.1109/JTEHM.2020.2984601. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Patrini I, Ruperti M, Moccia S, et al Transfer learning for informative-frame selection in laryngoscopic videos through learned features. Medical & Biological Engineering & Computing. 2020;58(6):1225–1238. doi: 10.1007/s11517-020-02127-7. [DOI] [PubMed] [Google Scholar]
- 5.Zhu J, Li Y, Hu Y, et al Rubik’s cube+: a self-supervised feature learning framework for 3D medical image analysis. Medical Image Analysis. 2020;64:101746. doi: 10.1016/j.media.2020.101746. [DOI] [PubMed] [Google Scholar]
- 6.Li H, Xue F F, Chaitanya K, et al. Imbalance-aware self-supervised learning for 3D radiomic representations//International Conference on Medical Image Computing and Computer-Assisted Intervention. Switzerland: Springer, 2021: 36-46.
- 7.Raghu M, Zhang C, Kleinberg J, et al. Transfusion: understanding transfer learning for medical imaging//International Conference on Neural Information Processing Systems, Vancouver: MIT Press, 2019: 3347-3357.
- 8.Bai W, Chen C, Tarroni G, et al. Self-supervised learning for cardiac MR image segmentation by anatomical position prediction//Medical Image Computing and Computer Assisted Intervention(MICCAI 2019), Shenzhen: Springer, 2019: 541-549.
- 9.Hervella Á S, Rouco J, Novo J, et al Self-supervised multimodal reconstruction pre-training for retinal computer-aided diagnosis. Expert Systems with Applications. 2021;185:115598. doi: 10.1016/j.eswa.2021.115598. [DOI] [Google Scholar]
- 10.He Xuehai, Yang Xingyi, Zhang Shuanghang, et al. Sample-efficient deep learning for COVID-19 diagnosis based on CT scans. arXiv preprint, 2020. DOI: 10.1101/2020.04.13.20063941.
- 11.Li Yi, Zhao Junli, Lv Zhihan, et al Medical image fusion method by deep learning. International Journal of Cognitive Computing in Engineering. 2021;2:21–29. doi: 10.1016/j.ijcce.2020.12.004. [DOI] [Google Scholar]
- 12.Chen T, Kornblith S, Norouzi M, et al. A simple framework for contrastive learning of visual representations//International Conference on Machine Learning, Vienna: ACM, 2020: 1597-1607.
- 13.He K, Fan H, Wu Y, et al. Momentum contrast for unsupervised visual representation learning//IEEE/CVF Conference on Computer Vision and Pattern Recognition, Seattle: IEEE, 2020: 9729-9738.
- 14.Grill J B, Strub F, Altché F, et al Bootstrap your own latent-a new approach to self-supervised learning. Advances in Neural Information Processing Systems. 2020;33:21271–21284. [Google Scholar]
- 15.Chen Xinlei, He Kaiming. Exploring simple siamese representation learning//IEEE/CVF Conference on Computer Vision and Pattern Recognition, Nashville: IEEE, 2021: 15745-15753.
- 16.Chaitanya K, Erdil E, Karani N, et al Contrastive learning of global and local features for medical image segmentation with limited annotations. Advances in Neural Information Processing Systems. 2020;33:12546–12558. [Google Scholar]
- 17.Sowrirajan H, Yang J, Ng A Y, et al. Moco pretraining improves representation and transferability of chest X-ray models// Medical Imaging with Deep Learning, Lubeck: MIDL, 2021: 728-744.
- 18.Irvin J, Rajpurkar P, Ko M, et al. Chexpert: a large chest radiograph dataset with uncertainty labels and expert comparison//AAAI Conference on Artificial Intelligence. 2019, 33(1): 590-597.
- 19.Azizi S, Mustafa B, Ryan F, et al. Big self-supervised models advance medical image classification//IEEE/CVF International Conference on Computer Vision, Montreal: IEEE, 2021: 3478-3488.
- 20.Windsor R, Jamaludin A, Kadir T, et al. Self-supervised multi-modal alignment for whole body medical imaging//Medical Image Computing and Computer Assisted Intervention(MICCAI) 2021, Strasbourg: Springer, 2021: 90-101.
- 21.Taleb A, Lippert C, Klein T, et al. Multimodal self-supervised learning for medical image analysis//Information Processing in Medical Imaging, arXiv preprint, 2020. arXiv: 1912.05396.
- 22.Zhang Hongyi, Cisse M, Dauphin Y N, et al. Mixup: Beyond empirical risk minimization//International Conference on Learning Representations, arXiv preprint, 2018. arXiv: 1710.09412.
- 23.Yun S, Han D, Oh S J, et al. Cutmix: Regularization strategy to train strong classifiers with localizable features//IEEE/CVF International Conference on Computer Vision, Seoul: IEEE, 2019: 6023-6032.
- 24.Zhang X, Zhou X, Lin M, et al. Shufflenet: an extremely efficient convolutional neural network for mobile devices//IEEE Conference on Computer Vision and Pattern Recognition, Salt Lake: IEEE, 2018: 6848-6856.
- 25.He K, Zhang X, Ren S, et al. Deep residual learning for image recognition//IEEE Conference on Computer Vision and Pattern Recognition, Las Vegas: IEEE, 2016: 770-778.
- 26.Liu Z, Lin Y, Cao Y, et al. Swin transformer: hierarchical vision transformer using shifted windows//IEEE/CVF International Conference on Computer Vision, Montreal: IEEE, 2021: 10012-10022.
- 27.He K, Zhang X, Ren S, et al. Delving deep into rectifiers: surpassing human-level performance on imagenet classification//IEEE International Conference on Computer Vision, Santiago: IEEE, 2015: 1026-1034.
- 28.Luo Liangchen, Xiong Yuanhao, Liu Yan, et al. Adaptive gradient methods with dynamic bound of learning rate//International Conference on Learning Representations, arXiv preprint, 2019, arXiv: 1902.09843.




