Abstract
非刚性配准在医学图像分析中有着重要的作用。U-Net被证明是医学图像分析的研究热点,被广泛应用于医学图像配准中,然而现有的基于U-Net及其变体的配准模型在处理复杂形变时,学习能力不足,且没有充分利用多尺度上下文信息,导致配准精度不够理想。针对该问题,本文提出了一种基于可变形卷积和多尺度特征聚焦模块的X线图像非刚性配准算法。该算法首先使用残差可变形卷积替代原U-Net的标准卷积,以增强配准网络对图像几何形变的表达能力;然后使用步长卷积代替下采样操作的池化运算,以缓解连续池化导致的特征丢失问题;此外在编、解码结构的桥接层中引入多尺度特征聚焦模块,以提高网络模型集成全局上下文信息的能力。理论分析和实验结果均表明提出的配准算法能聚焦多尺度上下文信息,能够处理具有复杂形变的医学图像,配准精度有一定提高,适合胸部X线片的非刚性配准。
Keywords: 可变形卷积, 多尺度特征, 非刚性配准, 医学图像
Abstract
Non-rigid registration plays an important role in medical image analysis. U-Net has been proven to be a hot research topic in medical image analysis and is widely used in medical image registration. However, existing registration models based on U-Net and its variants lack sufficient learning ability when dealing with complex deformations, and do not fully utilize multi-scale contextual information, resulting insufficient registration accuracy. To address this issue, a non-rigid registration algorithm for X-ray images based on deformable convolution and multi-scale feature focusing module was proposed. First, it used residual deformable convolution to replace the standard convolution of the original U-Net to enhance the expression ability of registration network for image geometric deformations. Then, stride convolution was used to replace the pooling operation of the downsampling operation to alleviate feature loss caused by continuous pooling. In addition, a multi-scale feature focusing module was introduced to the bridging layer in the encoding and decoding structure to improve the network model’s ability of integrating global contextual information. Theoretical analysis and experimental results both showed that the proposed registration algorithm could focus on multi-scale contextual information, handle medical images with complex deformations, and improve the registration accuracy. It is suitable for non-rigid registration of chest X-ray images.
Keywords: Deformable convolution, Multi-scale feature, Non-rigid registration, Medical image.
0. 引言
胸部X线片是利用X射线对人体胸部扫描所得到的图像,其临床应用广泛,如常被用于检查胸腔内部的肋骨、胸椎、肺组织等处的疾病[1]。直接对比观察不同的胸片图像费时费力,且受医生主观因素的影响。为了减轻医生负担,可利用计算机自动将不同的胸片图像进行对齐,即进行医学图像配准。医学图像配准[2]是医学图像融合、重建等研究的基础,在疾病诊断、临床治疗、手术导航和疗效评估等临床应用中有重要作用。其本质是将不同时间、不同视点或不同传感器采集到的两幅或多幅图像的对应点达到空间位置和解剖位置的对齐。
传统的配准算法通常将变形建模为物理模型,该类方法在执行配准过程中进行迭代优化来搜索空间变换,使已定义的相似性达到最大。Marstal等[3]提出了基于互相关信息的SimpleElastix软件包,它是多种传统配准方法的合集,旨在拓宽医学图像非刚性配准算法的应用。虽然传统算法在配准精度和鲁棒性方面具有较大优势,但其优化过程需要反复迭代,配准速度缓慢。
随着人工智能技术的快速发展,卷积神经网络(convolutional neural networks,CNN)已被应用在医学图像分析的各种任务中,在医学图像配准领域也取得了显著的成果。Sokooti等[4]提出用CNN预测浮动图像与固定图像间的形变场,并利用RegNet模型实现可变形医学图像的配准。de Vos等[5]首先提出了一种无监督端到端的深度学习网络,在手写体数字和心脏核磁共振成像数据集上进行验证,实验结果表明其配准精度与传统配准算法相当,但耗时更短。Balakrishnan等[6]提出了一种称为VoxelMorph的无监督端到端U-Net架构,使用分割掩码作为可用的辅助信息参与配准网络的训练,直接预测网络的变换参数。但当存在较大形变时,Voxelmorph的效果不够理想。Zhu等[7]针对复杂大形变问题,提出了在不同尺度下进行配准,分别得到不同尺度下的形变场并进行融合,实现了从粗到精的图像配准。但是,上述算法均未利用解剖结构的全局背景信息。Mansilla等[8]提出结合U-Net和自编码器结构的AC-RegNet网络模型,将分割掩码作为先验信息,在配准过程中结合全局约束和局部约束,提高了模型的配准精度。针对器官或组织边界不连续的图像配准精度低的问题,Chen等[9]提出了一种图像配准方法,该方法利用图像分割和配准的互补性,实现了图像的联合分割和成对配准,保证了全局不连续和局部平滑的形变场,从而获得更准确和真实的配准结果。现有的医学图像配准算法虽然在配准精度方面取得了一定的提升,但仍然存在以下问题:① 在处理复杂解剖结构差异性较大的图像时,模型的学习能力不足。② 目前的图像配准算法大多数在单一尺度下对图像进行表征,大尺度下的图像表征缺乏图像各异性的细节信息,小尺度下的图像表征过分注意局部细节,忽视全局信息,导致模型求解陷于入局部最优,产生过拟合。
针对上述问题,本文在Mansilla等[8]的基础上提出一种基于可变形卷积和多尺度特征聚焦的X线图像非刚性配准算法,用于胸部X线片图像的配准。首先使用残差可变形卷积模块(residual deformation convolution module,RD)替代原U-Net[10]的标准卷积模块,以增强配准网络对图像复杂几何形变的表达能力。然后使用步长卷积替换下采样操作的池化运算,以缓解连续池化导致的特征丢失问题。其次在编解码结构的桥接层中引入多尺度特征聚焦模块(multiscale feature focusing module,MFF),以提高网络模型集成全局上下文信息的能力。
1. 本文算法
1.1. 算法框架
本文算法所提出的配准网络框架如图1所示,分为上下两个分支,上分支是基于医学图像的配准过程,而下分支是基于分割掩码的辅助约束引导过程。
图 1.
Registration network model
配准网络模型图
具体流程如下:首先,本文在AC-RegNet[8]的基础上,设计了配准网络结构,命名为DM-RegNet。将参考图像和浮动图像输入到DM-RegNet网络得到形变场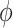 ,然后基于形变场
,然后基于形变场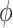 利用空间变换网络(spatial transformer networks,STN)[11]对浮动图像进行矫正,得到配准后的图像。为了提升配准精度,本文参照AC-RegNet[8]也在网络框架下分支增加一个分割掩码辅助约束模块,同时,利用STN[11]对浮动图像的分割掩码进行矫正获得矫正后图像的分割掩码。然后,通过引入两个损失函数迭代更新配准网络的参数,以提高配准网络的精度。两个损失函数分别为:分割掩码的全局约束损失和局部特征约束损失。下分支辅助模块利用分割掩码之间的相似性作为全局约束。同时,利用预先训练好的自编码器计算参考图像分割掩码和矫正后图像分割掩码的潜在特征之间的相似性作为局部特征约束,从而引导网络获得更加合理的形变场。
利用空间变换网络(spatial transformer networks,STN)[11]对浮动图像进行矫正,得到配准后的图像。为了提升配准精度,本文参照AC-RegNet[8]也在网络框架下分支增加一个分割掩码辅助约束模块,同时,利用STN[11]对浮动图像的分割掩码进行矫正获得矫正后图像的分割掩码。然后,通过引入两个损失函数迭代更新配准网络的参数,以提高配准网络的精度。两个损失函数分别为:分割掩码的全局约束损失和局部特征约束损失。下分支辅助模块利用分割掩码之间的相似性作为全局约束。同时,利用预先训练好的自编码器计算参考图像分割掩码和矫正后图像分割掩码的潜在特征之间的相似性作为局部特征约束,从而引导网络获得更加合理的形变场。
1.2. DM-RegNet网络结构
DM-RegNet网络结构如图2所示,配准网络模型使用U型结构。首先,在编码阶段采用连续3次下采样,在下采样中使用残差可变形卷积模块代替U-Net的标准卷积模块,有效地提取输入图像对的特征信息。同时,使用步长为2的3×3卷积层替代下采样的最大池化层。随着下采样操作的执行,特征图的尺寸逐步减半,同时卷积核的感受野不断扩大。下采样结束后,在编、解码结构中间的桥接层引入多尺度特征聚焦模块来捕获不同尺度下的图像特征信息。用反卷积进行上采样,将上采样后的特征与下采样中具有相同分辨率的特征进行跳跃连接,然后再使用两个连续的3×3卷积层进行卷积运算。最后,使用一个1×1卷积层后输出形变场。
图 2.
DM-RegNet registration network
DM-RegNet配准网络
1.3. 残差可变形卷积模块
现有配准算法通常使用固定结构的卷积核来构建配准网络,此类网络模型不能较好地学习到复杂的几何形变。可变形卷积[12]可以通过图像内容自适应调整卷积运算中采样点的位置,以适应目标几何形状的变化。基于此,本文提出使用残差可变形卷积模块替代原U-Net下采样过程中的标准卷积模块,以加强网络模型对目标形态大小差异的关注。该模块的结构如图3所示。可变形卷积可以适应目标几何形状的变化,提高网络对图像相关区域的表征能力。此外,使用残差连接的方式,在加深网络的同时可以缓解过拟合问题。
图 3.
Residuals deformable convolution module
残差可变形卷积模块
标准卷积的运算过程首先是使用规则网格R对输入特征图X进行采样,然后在每个采样点处乘上权值W并求和。以一个3 × 3的卷积运算过程为例,网格 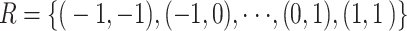 ,任意位置
,任意位置 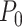 所对应的输出特征图Y上的值可以通过式(1)得到。
所对应的输出特征图Y上的值可以通过式(1)得到。
 |
1 |
其中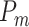 指的是在规则网络
指的是在规则网络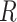 中的位置。
中的位置。
可变形卷积的核心思想是在标准卷积的规则网络采样点上添加偏移量,实现采样网格的自适应变形。具体实现过程是首先使用一个并行连接的卷积层,对输入特征图进行卷积操作,得到一个偏移场的初步估计值。然后对初步估计值进行微调,以便更好地适应输入特征图的形变。其次对微调后的偏移场进行规范化处理,以确保偏移量的大小适中。最后将学习到的偏移量输入原来的卷积层中。通过这个额外的操作,可变形卷积可以根据图像中内容实现对当前位置的自适应采样,而不仅限于规则网络采样。需要注意的是,可变形卷积学习的是特征图中每个位置的偏移量,而不是卷积核的偏移量。在可变形卷积中,规则网络 用偏移量
用偏移量 增广,实现过程如式(2)所示。
增广,实现过程如式(2)所示。
 |
2 |
1.4. 多尺度特征聚焦模块
为了同时获得全局和细节信息,传统方法是在不同的分辨率下提取图像特征,然后将这些特征直接融合得到最终的特征表示。由于不同尺度的特征存在语义鸿沟,直接进行特征融合效果不够理想,因此本文基于注意力机制,引入多尺度特征聚焦模块[13]来有效地提取医学图像的全局上下文信息,该模型能够兼顾全局和局部特征信息,模块中使用较大内核的平均池化层来保留图像的全局信息,而采用较小内核的最大池化层以突出细节信息。此外,使用通道注意力机制来建立通道间的依赖关系,通道注意力机制通过平均池化给每个特征通道生成一个权重值,将得到的权重值进行归一化操作,最后加权到每个通道的特征上,使网络模型自适应聚焦于图像中局部形变较大的区域。该模块的结构如图4所示。具体的实现流程如式(3)所示。
图 4.
Multi-scale feature focusing module
多尺度特征聚焦模块
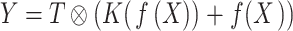 |
3 |
X表示输入的图像或特征图。Y表示输出的图像或特征图。T为通道注意力权重,取值范围在[0, 1]之间,作为权重来细化特征图,具体计算为: ,其中
,其中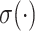 为平均池化运算,将每个通道的二维特征压缩为一个实数,
为平均池化运算,将每个通道的二维特征压缩为一个实数,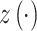 表示归一化操作,
表示归一化操作,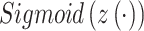 为每个特征通道生成一个权重值。
为每个特征通道生成一个权重值。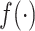 表示1 × 1卷积运算调整输入的特征图通道数。
表示1 × 1卷积运算调整输入的特征图通道数。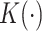 是经四个池化运算后的输出,
是经四个池化运算后的输出,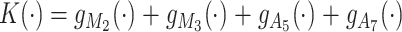 ,其中M2和M3表示最大池化运算,A5和A7表示平均池化运算,下标为各自的池化内核大小,
,其中M2和M3表示最大池化运算,A5和A7表示平均池化运算,下标为各自的池化内核大小,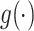 为双线性插值上采样函数。
为双线性插值上采样函数。
1.5. 损失函数
总损失函数包括配准分支参考图像与配准后图像的相似性损失、形变场的平滑约束项、分割掩码的局部和全局约束项。
(1)图像的相似性约束项
本文采用归一化互相关(normalized cross correlation,NCC)作为相似性测度计算两幅图像之间的对齐程度。对于给定的任意一幅参考图像F和一幅配准图像 ,NCC定义为:
,NCC定义为:
 |
4 |
其中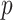 为图像的像素个数,
为图像的像素个数, 为图像中所有像素的集合,
为图像中所有像素的集合, 表示图像的像素位置,
表示图像的像素位置,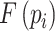 、
、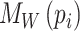 分别表示参考图像和配准后图像上的灰度,
分别表示参考图像和配准后图像上的灰度,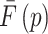 、
、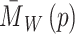 分别表示两幅图像中像素点的平均灰度值。NCC在[0, 1]范围内,其值越接近1,表示两幅图像间灰度值越相关,对齐程度越高。反之,则对齐程度越低。最小化损失函数即最大化图像相似性,因此
分别表示两幅图像中像素点的平均灰度值。NCC在[0, 1]范围内,其值越接近1,表示两幅图像间灰度值越相关,对齐程度越高。反之,则对齐程度越低。最小化损失函数即最大化图像相似性,因此 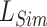 如下所示:
如下所示:
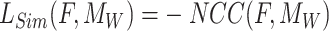 |
5 |
(2)平滑约束项损失
为了使形变场更加平滑,本文在损失函数中添加平滑正则项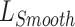 ,其定义如下:
,其定义如下:
 |
6 |
其中p为图像的像素个数, 为图像中所有像素的集合,
为图像中所有像素的集合,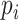 表示图像的像素位置。
表示图像的像素位置。
(3)分割掩模的局部约束项
将解剖先验信息引入到配准网络中,用分割掩码之间的相似度辅助约束与引导网络的训练。局部约束项 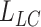 表示矫正后的浮动图像分割掩码
表示矫正后的浮动图像分割掩码 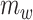 和参考图像分割掩码f之间的相似性。
和参考图像分割掩码f之间的相似性。
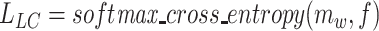 |
7 |
(4)分割掩码的全局约束项
由于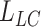 不能保证两幅图像分割掩码之间的解剖结构在全局尺度上的对齐,因此,使用自编码器提取两者的潜在特征,并计算其损失。
不能保证两幅图像分割掩码之间的解剖结构在全局尺度上的对齐,因此,使用自编码器提取两者的潜在特征,并计算其损失。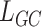 如下所示:
如下所示:
 |
8 |
式中AE( )表示自编码器提取的潜在特征。
(5)总损失函数
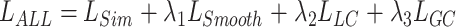 |
9 |
式中  、
、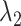 和
和 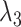 均为超参数,
均为超参数, 为平滑项的权重,
为平滑项的权重,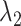 、
、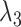 分别为分割掩码局部约束项和全局约束项的权重。
分别为分割掩码局部约束项和全局约束项的权重。
2. 实验与结果分析
2.1. 数据集
为了验证本文算法的配准性能,在3个胸部X线片图像数据集上进行了评估,分别为日本放射技术协会数据集(Japanese Society of Radiological Technology,JSRT)[14]、蒙哥马利县X射线数据集(Montgomery County X-ray,Montgomery)[15]以及深圳医院X射线数据集(Shenzhen Hospital X-ray,Shenzhen)[16]。JSRT数据集是一个公共数据库,共247张胸部X线片图像。Montgomery数据集来自美国马里兰州蒙哥马利县卫生和公共服务部结核病控制项目,包含138张带标注的后前位胸片图像。Shenzhen数据集包含662张不同大小的胸片图像。本文使用与Mansilla等[8]相同的数据预处理方法,首先对Montgomery和Shenzhen数据集进行处理,通过对数据中的最短边填充背景色来调整图像大小,得到相同分辨率的方形图像,然后对三个数据集中所有图像进行仿射对齐,最后将预处理后的所有数据重采样至大小为256×256。
实验环境为:Ubuntu18.04操作系统,编程语言为python,tensorflow1.14框架。硬件设施为:显存11 GB的NVIDIA GeForce 2080TI显卡,内存大小为128 GB。配准模型训练时使用ADM优化器进行网络参数的更新,学习率为0.000 1,批量大小为32。损失函数中超参数的选择通过多次实验得到,其中 取值为0.000 05,
取值为0.000 05,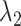 取值为1,
取值为1,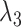 取值为0.1。
取值为0.1。
2.2. 评估指标
本文采用与文献[8]相同的性能评估指标:Dice系数(Dice coefficient,Dice)、豪斯多夫距离(Hausdorff distance,HD)和平均对称表面距离(average symmetric surface distance,ASSD)。Dice系数的取值范围为0到1,Dice值越大,表示结构完全重叠越高;较小的HD值表示两个对象之间的拓扑更紧密;两幅图像的ASSD值越低,配准效果越好。
2.3. 定性分析
为了验证本文所提出DM-RegNet模型的配准性能,利用预处理后的三个数据集进行配准实验,并将本文算法与四种配准算法进行比较。对比算法分别是SimpleElatix[3]、DIRNet[5]、AC-RegNet[8]和SDDIR[9]。其中SimpleElastix是比较流行的图像配准软件包;DIRNet是首个使用无监督模型的非刚性配准算法;SDDIR利用图像分割和配准的互补性,实现了图像的联合分割和成对配准;AC-RegNet是本文所使用的基线模型。
图5表示不同配准算法在JSRT数据集上的两组配准结果。若两种颜色的分割掩码重合程度越高,则表示其对齐效果越好。如图所示,配准之前的浮动图像和参考图像具有较大的形态和空间位置差异,其对齐程度较差。从不同算法在配准之后的轮廓叠加图可以看出对齐程度均优于配准前。其中SimpleElastix算法下的分割掩码叠加图,在整体胸部的左右轮廓上没有得到完全的矫正,在局部较大形变区域如拐角处,其重合度较差。DIRNet算法、SDDIR算法和AC-RegNet算法在整体胸部轮廓上能实现较好的对齐,但仍存在较小范围的变形,其中AC-RegNet采用多个约束引导配准网络的训练,在提升网络性能的同时,也使得其轮廓叠加图优于DIRNet算法和SDDIR算法。相比其他几种算法,无论是在局部较大形变区域还是整体胸部左右轮廓上,本文算法均能够实现最佳的配准效果,其配准图像和参考图像分割掩码的重合度最高。
图 5.

Two sets of registration results on the JSRT dataset
在JSRT数据集上的两组配准结果
2.4. 定量分析
表1显示了不同配准算法在JSRT、Montgomery和Shenzhen三个数据集上配准结果的定量比较。从表1可知,与配准前相比,不同算法配准后的性能在Dice、HD和ASSD指标上均具有显著的优势,其中SimpleElastix[3]、DIRNet算法[5]和SDDIR算法[9]在Dice方面有一定的提升以及在HD和ASSD值上均有所下降,但配准性能提升的幅度不是很大。而AC-RegNet算法[8]使用图像和分割掩码的多损失约束去监督配准网络的训练,分割掩码作为辅助信息约束与引导配准的实现,使得网络模型在配准精度上的提升更为显著,HD和ASSD均有较大幅度降低,且Dice有较大提升,但其配准结果与参考图像仍不能实现完全地对齐。在三个胸部数据集上,本文算法在Dice、HD和ASSD方面均优于其他三种配准算法。这主要得益于本文算法在AC-RegNet[8]网络模型的基础上,使用了多种优化策略如引入残差可变形卷积模块和多尺度特征聚焦模块来训练配准网络,使得生成的配准图像更接近参考图像。
表 1. Quantitative evaluation results.
定量评估结果
| 数据集 | 算法 | Dice | HD | ASSD |
| 注:加粗数字为该数据集下的最优结果 | ||||
| JSRT | 配准前 | 0.764 | 32.19 | 10.13 |
| SimpleElastix[3] | 0.854 | 28.37 | 8.72 | |
| DIRNet[5] | 0.846 | 25.94 | 7.18 | |
| AC-RegNet[8] | 0.942 | 18.21 | 3.45 | |
| SDDIR[9] | 0.921 | 22.36 | 5.69 | |
| 本文算法 | 0.967 | 15.74 | 2.84 | |
| Montgomery | 配准前 | 0.772 | 39.43 | 8.81 |
| SimpleElastix[3] | 0.882 | 37.80 | 6.71 | |
| DIRNet[5] | 0.867 | 26.39 | 5.26 | |
| AC-RegNet[8] | 0.953 | 14.84 | 2.54 | |
| SDDIR[9] | 0.922 | 15.57 | 3.86 | |
| 本文算法 | 0.965 | 10.28 | 1.63 | |
| Shenzhen | 配准前 | 0.785 | 346.24 | 45.12 |
| SimpleElastix[3] | 0.891 | 322.64 | 43.28 | |
| DIRNet[5] | 0.849 | 271.39 | 37.41 | |
| AC-RegNet[8] | 0.934 | 260.47 | 30.65 | |
| SDDIR[9] | 0.916 | 265.44 | 34.35 | |
| 本文算法 | 0.958 | 195.83 | 28.56 | |
2.5. 消融实验
为了验证所提出的残差可变形卷积模块和多尺度特征聚焦模块对配准性能的影响,在JSRT数据集上进行了消融实验研究。本节设置了四种不同的网络模型:将AC-RegNet[8]作为基线模型,命名为Base;在基线模型中单独引入残差可变形卷积模块或多尺度特征聚焦模块,分别命名为Base + RD和Base + MFF;在基线模型中同时引入RD和MFF模块,命名为本文算法。
表2为在JSRT数据集上消融实验的配准精度比较,从表中可知,在基线模型中分别添加RD模块和MFF模块,Dice分别提高了0.007和0.011,HD分别下降了0.86和1.53,ASSD分别下降了0.24和0.36。因此,在基线模型上单独添加优化模块带来性能增益的优先级是,MFF模块对配准精度的提升最为显著,其次是RD模块。这是因为MFF模块结合多尺度特征融合和注意力机制的优势,能有效提升配准网络捕获全局上下文信息的能力,且聚焦于具有较大局部形变的区域,有利于配准精度的提升。当同时添加RD模块和MFF模块后,Dice提升了0.025,且HD与ASSD均有较大的下降,表明当两个优化模块共同作用时,可以更好地增强匹配效果。
表 2. Comparison of registration accuracy of ablation experiments on JSRT datasets.
在JSRT数据集上消融实验的配准精度比较
| 算法 | Dice | HD | ASSD |
| 注:加粗数字为最优结果 | |||
| Base | 0.942 | 18.21 | 3.45 |
| Base + RD | 0.949 | 17.35 | 3.21 |
| Base + MFF | 0.953 | 16.68 | 3.09 |
| 本文算法 | 0.967 | 15.74 | 2.84 |
3. 结论
本文提出一种基于可变形卷积和多尺度特征聚焦模块的 X线图像非刚性配准算法。一方面,本文提出的算法用残差可变形卷积模块代替U-Net中的标准卷积模块,能适应目标几何形状的变化,从而加强网络模型对目标形态大小差异的关注,达到增强网络模型对目标几何形变的建模能力的目的。另一方面,用卷积层替换U-Net模型用于下采样的最大池化层,以缓解连续下采样造成的特征丢失。此外,由于在编、解码结构的桥接层使用多尺度特征聚焦模块,使得网络模型能自适应聚焦于图像中局部形变较大的区域,从而提高获取图像上下文信息的能力。最后,在JSRT、Montgomery和Shenzhen数据集上进行了对比和消融实验。理论分析和实验结果均表明,提出的配准算法能聚焦多尺度上下文信息,能够处理具有复杂形变的医学图像,配准精度有一定提高,适合胸部图像的非刚性配准。
重要声明
利益冲突声明:本文全体作者均声明不存在利益冲突。
作者贡献声明:彭昆负责算法设计与实现、数据处理与分析、论文写作与修改;张桂梅、储珺提供实验指导及论文审阅修订;王杰参与数据集收集及预处理并负责整理实验结果。
Funding Statement
国家自然科学基金项目(62261039)
The National Natural Science Foundation of China
References
- 1.李翠华, 刘玉转, 高昭昇 智能影像辅助诊断在基层医疗机构的应用模式研究. 中国数字医学. 2022;17(5):75–78. doi: 10.3969/j.issn.1673-7571.2022.5.016. [DOI] [Google Scholar]
- 2.Xiao H, Teng X, Liu C, et al A review of deep learning-based three-dimensional medical image registration methods. Quant Imaging Med Surg. 2021;11(12):4895–4916. doi: 10.21037/qims-21-175. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.Marstal K, Berendsen F, Staring M, et al. SimpleElastix: A user-friendly, multi-lingual library for medical image registration// 2016 IEEE Conference on Computer Vision and Pattern Recognition Workshops (CVPRW). Las Vegas: IEEE, 2016: 547-582.
- 4.Sokooti H, Vos B D, Berendsen F, et al. Nonrigid image registration using multi-scale 3D convolutional neural networks// The 20th International Conference on Medical Image Computing and Computer-Assisted Intervention(MICCAI). Quebec: Springer, 2017: 232-239.
- 5.de Vos B D, Berendsen F F, Viergever M A, et al. End-to-end unsupervised deformable image registration with a convolutional neural network// Third International Workshop on Deep Learning in Medical Image Analysis and Multimodal Learning for Clinical Decision Support (DLMIA). Quebec: Springer, 2017: 204-212.
- 6.Balakrishnan G, Zhao A, Sabuncu M R, et al VoxelMorph: A learning framework for deformable medical image registration. IEEE Trans Med Imaging. 2019;38(8):1788–1800. doi: 10.1109/TMI.2019.2897538. [DOI] [PubMed] [Google Scholar]
- 7.Zhu W, Huang Y, Xu D, et al. Test-time training for deformable multi-scale image registration// 2021 IEEE International Conference on Robotics and Automation(ICRA). Xi’an: IEEE, 2021: 13618-13625.
- 8.Mansilla L, Milone D H, Ferrante E Learning deformable registration of medical images with anatomical constraints. Neur Netw. 2020;124:269–279. doi: 10.1016/j.neunet.2020.01.023. [DOI] [PubMed] [Google Scholar]
- 9.Chen X, Xia Y, Ravikumar N, et al. Joint segmentation and discontinuity-preserving deformable registration: Application to cardiac cine-MR images. ArXiv, 2022: 2211.13828.
- 10.Ronneberger O, Fischer P, Brox T. U-net: Convolutional networks for biomedical image segmentation// Medical Image Computing and Computer-Assisted Intervention–MICCAI 2015(MICCAI). Munich: Springer, 2015: 234-241.
- 11.Jaderberg M, Simonyan K, Zisserman A. Spatial transformer networks. Advances in neural information processing systems. ArXiv, 2015: 1506.02025.
- 12.Dai J, Qi H, Xiong Y, et al. Deformable convolutional networks// 2017 IEEE International Conference on Computer Vision (ICCV). Venice: IEEE. 2017: 764-773.
- 13.Chen L C, Papandreou G, Kokkinos I, et al Deeplab: Semantic image segmentation with deep convolutional nets, atrous convolution, and fully connected CRFs. IEEE Trans Pattern Anal Mach Intell. 2017;40(4):834–848. doi: 10.1109/TPAMI.2017.2699184. [DOI] [PubMed] [Google Scholar]
- 14.Shiraishi J, Katsuragawa S, Ikezoe J, et al Development of a digital image database for chest radiographs with and without a lung nodule: receiver operating characteristic analysis of radiologists detection of pulmonary nodules. Am J Roentgenol. 2000;174(1):71–74. doi: 10.2214/ajr.174.1.1740071. [DOI] [PubMed] [Google Scholar]
- 15.Candemir S, Jaeger S, Palaniappan K, et al Lung segmentation in chest radiographs using anatomical atlases with nonrigid registration. IEEE Trans Med Imaging. 2014;33(2):577–590. doi: 10.1109/TMI.2013.2290491. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Jaeger S, Karargyris A, Candemir S , et al Automatic tuberculosis screening using chest radiographs. IEEE Trans Med Imaging. 2014;33(2):233–245. doi: 10.1109/TMI.2013.2284099. [DOI] [PubMed] [Google Scholar]






