Abstract
药物组合的协同作用能够解决单一药物疗法的获得耐药性问题,对于癌症等复杂疾病的治疗具有巨大的潜力。在本项研究中,为了探索不同药物分子间相互作用对于抗癌药物疗效的影响,我们提出了一种基于Transformer的深度学习预测模型——SMILESynergy。首先用药物的文本数据——简化分子线性输入规范(SMILES)表征药物分子,其次通过SMILES Enumeration生成药物分子的异构体进行数据增强,然后利用Transformer中的注意力机制对数据增强后的药物进行编解码,最后连接一个多层感知器(MLP)获得药物的协同作用值。实验结果表明我们的模型在O’Neil数据集的回归分析中均方误差为51.34,分类分析中准确率为0.97,预测性能均优于DeepSynergy和MulinputSynergy模型。SMILESynergy具有更好的预测性能,可辅助研究人员快速筛选最优药物组合以提高癌症治疗效果。
Keywords: 协同作用, 深度学习, 注意力, Transformer
Abstract
The synergistic effect of drug combinations can solve the problem of acquired resistance to single drug therapy and has great potential for the treatment of complex diseases such as cancer. In this study, to explore the impact of interactions between different drug molecules on the effect of anticancer drugs, we proposed a Transformer-based deep learning prediction model—SMILESynergy. First, the drug text data—simplified molecular input line entry system (SMILES) were used to represent the drug molecules, and drug molecule isomers were generated through SMILES Enumeration for data augmentation. Then, the attention mechanism in the Transformer was used to encode and decode the drug molecules after data augmentation, and finally, a multi-layer perceptron (MLP) was connected to obtain the synergy value of the drugs. Experimental results showed that our model had a mean squared error of 51.34 in regression analysis, an accuracy of 0.97 in classification analysis, and better predictive performance than the DeepSynergy and MulinputSynergy models. SMILESynergy offers improved predictive performance to assist researchers in rapidly screening optimal drug combinations to improve cancer treatment outcomes.
Keywords: Synergy, Deep learning, Attention, Transformer
0. 引言
癌症是一种高度异质性的疾病,不同类型的癌症具有不同的遗传变异和表观遗传变异[1],导致不同个体对药物的反应和耐药性[2]也不同,因此选择最优的药物组合来提高治疗效果是癌症治疗的一个重要挑战。传统的化疗药物研究方法需要通过大量的临床试验进行药物筛选和剂量确定,耗费时间和资源,并且可能使患者面临不必要甚至有害的治疗[3]。而且由于癌症的异质性和个体差异性,不同患者对同一种药物的反应和副作用也不同,使得制定治疗方案更加困难。因此利用计算模型来探索高通量筛选[4]实验产生的药物组合数据具有重要意义。
近年来研究人员运用深度学习在抗癌药物协同作用预测的研究中做了大量的工作。如Preuer等[5]提出的DeepSynergy模型,以O’Neil数据集[6]上的药物化合物以及细胞系基因表达信息作为输入,连接一个全连接前馈神经网络(fully forward neural network,FFNN),得到了不错的抗癌药物组合协同作用预测结果。在药物化合物以及细胞系基因表达信息的基础上,Zhang等[7]加入细胞系的拷贝数和基因突变数据作为输入,并提出了基于自编码器(auto encoder)的深度学习模型AuDNNsynergy来预测具有协同作用的药物组合。在文献[7]输入数据的基础上,陈希等[8]通过一维卷积层对输入数据进行降维预处理,进而提出了MulinputSynergy药物预测模型,并在模型中加入残差前馈神经层,最终提升了药物组合预测效果。Sun等[9]在O’Neil数据集的药物数据上引入张量分解方法进行预处理,然后将分解的结果作为特征用于训练深度神经网络(deep neural network,DNN)模型以预测药物对的协同效应。
鉴于近年来深度学习在自然语言处理(natural language process,NLP)领域的发展,以Transformer[10]为代表的预训练语言模型加下游任务的方案在药物化学反应预测[11-12]、药物化学合成[13-14]和药物分子优化[15]等领域得到了广泛应用。Transformer预训练语言模型利用巨大的无标记语料库来学习单词和句子的表征,然后使用相对较小的数据集对预训练模型进行微调以适应下游任务[16-17]。与传统药物化学描述符输入深度学习预测模型不同的是,文献[11-15]将药物的简化分子线性输入规范(simplified molecular input line entry system,SMILES)[18]作为文本数据输入模型。在药物化学反应预测领域,Schwaller等[11]提出了一种基于Transformer结构的Molecular Transformer模型,通过贝叶斯深度学习训练方法预测药物化学反应并得到了较高的准确性。Wang等[12]在Transformer基础上引入BERT半监督模型SMILES-BERT,通过微调使预训练模型应用到不同的分子特性预测任务中。在药物化学合成领域,Tetko等[13]提出了一种使用增强NLP Transformer模型的方法以确定化学反应的可行性,并通过评估合成步骤的预测结果来提高合成规划的效率。Liu等[19]尝试将Transformer模型应用到药物组合协同作用预测领域,提出了一个基于注意力机制的Transformer深度学习模型TranSynergy,提高了协同药物组合预测的性能和可解释性。但是TranSynergy模型的输入数据是O’Neil数据集中两个药物特征与一个细胞系特征组成的三元张量,由于输入数据不是文本形式,所以模型主体部分的Transformer编解码结构没有使用词向量嵌入和位置编码,使得模型失去了理解药物分子结构与药物组合序列中分子距离特征的能力。
为了克服上述局限性,我们提出了一种以药物的文本数据SMILES为输入,以Transformer为预训练模型的药物协同作用预测方法——SMILESynergy。首先将药物分子用SMILES表示,通过SMILES Enumeration对药物组合进行数据增强,预训练模型采用包括词向量嵌入与位置编码的Transformer模型以完成对药物数据的编码,在下游任务中连接一个多层感知器(multilayer perceptron,MLP)对药物组合协同作用的回归与分类任务进行预测。我们在O’Neil和NCI-ALMANAC数据集上对模型性能进行了验证,并与DeepSynergy[5]、MulinputSynergy[8]等模型进行了比较。
1. 数据集
本论文在两个数据集上验证SMILESynergy模型的性能。第一个数据集是默克公司制作的高通量药物组合筛选数据集[6],文中称为O’Neil数据集。该数据集包含583个药物组合作用在39个癌症细胞系上的22 737条实验数据。Preuer等[5]在上述高通量筛选数据集上使用Combenefit[20]批量处理模式计算Loewe Additivity 值量化药物组合的协同作用值,并将它作为回归指标;在分类实验中,我们采用文献[5]中的阈值30进行分类,当药物组合协同作用预测分数大于30时认为具有协同作用,反之无协同作用。
第二个数据集是NCI-ALMANAC数据集[21],包含了5 232个药物对在NCI60的每个细胞系的组合活性,它可以用来发现那些与单一药物相比具有更强生长抑制作用的药物组合。在NCI-ALMANAC数据集中,ComboScore表示药物组合活性:正值表示药物组合具有协同作用,负值表示药物组合无协同作用;PercentGrowth表示单个药物或药物组合的抗癌活性。我们用PercentGrowth和ComboScore分别作为回归指标和分类指标对模型进行训练与测试。
2. 模型
本模型主要由三部分构成,如图1所示。第一部分是SMILES Enumeration生成药物异构体,每种药物不超过5个;第二部分是用于编解码词向量的Transformer模型,包含SMILES文件的词嵌入、位置编码和注意力机制;第三部分由MLP构成,对Transformer编码的指纹文件(fingerprint)进行回归预测,得到协同作用值表示模型的预测结果。
图 1.

Structure of SMILESynergy model
SMILESynergy 模型结构图
2.1. 药物的SMILES表示及SMILES Enumeration数据增强
由于药物的物理化学性质与药物的分子排列组合方式和空间结构有关,并且Transformer模型在自然语言处理领域中通常以文本序列作为输入数据[10],因此我们采用SMILES表征药物。例如结构为C9H11F2N3O4的吉西他滨(GEMCITABINE)的SMILES表示(见图2a)和三维分子结构(见图2b),其中字母如C、N一般代表原子,符号如―、=、#代表化学键。考虑到O’Neil数据集中药物对的数量仅有583个,我们对数据进行增强处理以提高模型的预测性能。SMILES可以表示药物分子不同的异构体,包括构象异构体和位置异构体。我们利用RDKit[22]中EnumerateStereoisomers方法枚举不同的SMILES获得药物分子的异构体来达到数据增强的目的。例如吉西他滨的分子异构体如图2c所示。
图 2.
Gemcitabine
药物吉西他滨
a. 三维分子结构;b. SMILES 表示;c. SMILES异构体
a. three-dimensional molecular structure of gemcitabine; b. SMILES representation; c. SMILES isomers

2.2. 位置编码与注意力机制
当SMILES Enumeration增强后的数据传入Transformer模型时,我们对数据进行词嵌入(Embedding)操作获得药物的词嵌入向量。词嵌入向量表示SMILES序列中的每个药物分子映射到高维向量空间中的向量。为了进一步考虑药物序列中每个分子的位置信息,我们将药物分子的位置信息编码之后添加到词嵌入向量。位置编码表示药物分子在药物序列中的位置或者功能团在药物序列中的位置等,这样模型就可以更好地利用药物的物理化学性质和结构信息预测药物的协同作用。Transformer中的位置编码是由一组三角函数计算而来的,遵循如下公式:
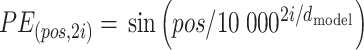 |
1 |
 |
2 |
其中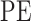 代表位置编码(position encoding),
代表位置编码(position encoding), 表示药物分子在药物序列中的位置,当
表示药物分子在药物序列中的位置,当  为偶数时使用正弦函数进行编码,当
为偶数时使用正弦函数进行编码,当  为奇数时使用余弦函数进行编码。
为奇数时使用余弦函数进行编码。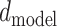 代表词嵌入向量的维度,
代表词嵌入向量的维度,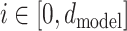 代表位于0到第
代表位于0到第 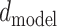 每一维元素都需要对输入序列进行编码。于是根据上述公式,我们可以得到药物序列中第
每一维元素都需要对输入序列进行编码。于是根据上述公式,我们可以得到药物序列中第 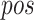 位置药物分子的
位置药物分子的 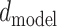 维位置向量。
维位置向量。
注意力机制是对药物分子不同特征之间的相互作用进行编码的模块,它将一个Query和一组Key、Value键值对映射到一个输出,其中Query表示待预测的药物分子的查询向量,用于捕获其在特定任务中的特征表示;Key表示其他药物分子的键向量,用于计算待预测药物与其他药物之间的相似性;Value表示其他药物分子的值向量,用于加权计算其他药物与待预测药物的相似性得分。注意力的计算公式如下:
 |
3 |
 |
4 |
其中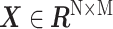 是药物特征矩阵,
是药物特征矩阵,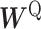 ,
, 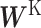 ,
, 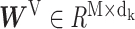 分别对应Query、Key和Value的权重矩阵。
分别对应Query、Key和Value的权重矩阵。 是一个缩放因子,Z是注意力层的输出。
是一个缩放因子,Z是注意力层的输出。
2.3. SMILES Transformer模型架构
我们的预训练模型采用4层Encoder-Decoder的Transformer模型,模型整体架构如图3a所示,输入的药物序列首先经过Embedding变为词向量,并将位置编码添加到词嵌入向量中,随后进入由4个编码器与4个解码器构成的Transformer结构。Transformer编码器和解码器内部细节如图3b所示,Embedding后的药物数据经过位置编码进入到编码器,编码器由多个相同的层组成,每层包括一个多头注意力层和一个全连接网络层。解码器也由多个相同的层组成,每层包括三个子层:多头自注意力子层、编码器-解码器注意力子层和全连接前馈神经网络子层。最终模型输出1 024维的药物fingerprint文件。
图 3.
SMILES Transformer model
SMILES Transformer模型
a. 模型整体架构;b. 模型内部细节
a. overall architecture of the model; b. internal details of the model

我们对ChEMBL24数据集(药物化学和药理学的数据集[23])中861 000个无标签的SMILES随机抽取进行预训练,其架构参数及训练超参数来自文献[12]。在训练中,使用Adam优化器最小化输入SMILES和输出概率之间的交叉熵。训练过程中损失值逐渐下降,同时困惑度也在不断减小,训练了5个epoch后,模型达到收敛状态,困惑度达到了1.0左右,表明模型能够很好地预测出药物组合序列中的每个药物分子。相比原始Transformer[10],SMILES Transformer模型的参数由6 500万下降至400万左右,模型的收敛速度得到了提升。在下游任务中,我们利用SMILES Transformer编码生成药物的1 024维fingerprint数据输入MLP,MLP的迭代次数max_iter设置为1 000,其他默认超参数与Scikit-learn[24]相同。随后将数据集按4∶1随机拆分为训练集和测试集,进行最终的回归训练。SMILESynergy模型的伪代码详见附件1。
3. 结果与讨论
3.1. SMILESynergy在O’Neil数据集的性能评估
我们从回归性能和分类性能两方面对模型进行评估。回归性能分析的主要指标是平均平方误差(mean square error,MSE)、平均绝对误差(mean absolute error ,MAE)、可释方差值(explained variation score,EVS)、皮尔逊相关系数(Pearson)和R²(r-squared)。表1中给出了SMILESynergy模型在39个细胞系中的预测结果。
表 1. SMILESynergy regression performance metrics on O’Neil.
SMILESynergy在O’Neil 数据集上回归性能指标
| 指标 | MAE | MSE | R² | EVS | Pearson |
| 平均值 | 4.39 | 51.34 | 0.90 | 0.90 | 0.95 |
| 最小值 | 2.71 | 16.32 | 0.80 | 0.80 | 0.90 |
| 中位数 | 3.81 | 33.40 | 0.91 | 0.91 | 0.95 |
| 最大值 | 9.01 | 199.60 | 0.96 | 0.96 | 0.98 |
由表1可以看出,MSE的平均值降低到了100以内,在测试的39个细胞系中,各评价指标的中位数都非常接近平均值,说明我们的模型在大多数的细胞系中都具有良好的稳定性。但MSE的最小值与最大值相差约180,为了探究这一原因,根据器官组织分布,我们将细胞系的MSE包点图和箱型图进行了可视化,如图4a所示,可以看出在乳腺(BREAST)、卵巢(OVARY)与肺(LUNG)三个组织中出现了三个异常值;导致MSE相差较大的异常情况主要出现在前列腺组织(PROSTATE)中,而前列腺组织涉及的细胞系只有两个,因此数据平均值产生了较大的偏差。
图 4.
SMILESynergy regression performance analysis on the O’Neil dataset
SMILESynergy在O’Neil数据集上回归性能分析
a. 各器官组织对应细胞系的MSE包点图和箱型图;b. 大肠、前列腺与肺组织的所有细胞系的协同值散点图
a. MSE beeswarm plots and box plots of corresponding cell lines for each organ tissue; b. scatter plots of synergy values of all cell lines for large intestine, prostate and lung tissues

为了进一步分析不同细胞系下预测结果的差异,我们选取图4a中预测效果最好的大肠组织(LARGE_INTESTINE),以及预测效果较差的前列腺组织(PROSTATE)和肺组织(LUNG),对三个组织中所有细胞系对应的药物组合协同作用值进行了可视化,得到散点图4b。从图4b可以看出,前列腺组织的LNCAP细胞系和肺组织的NCIH2122、NCIH23细胞系上的药物组合协同作用值的分布较为分散,相较于其他细胞系有大量的数据分布在―100以下,药物组合之间协同作用值的离散程度比较大,导致模型在训练过程中无法有效学习药物组合数据的特征规律,使得整体的MSE出现了较大范围的波动。
在分类方面,我们采用准确率(accuracy,ACC)、精确率(precision,PREC)、敏感性(sensitivity,SENS)、特异性(specificity,SPEC)、F1分数(F1)、受试者工作特征曲线下的面积(area under curve of receiver operating characteristic,ROC_AUC)来衡量模型的分类性能。
从表2可以看到本文模型的平均准确率达到了0.97,特异性的平均值达到了0.98。同时注意到敏感性出现了一定幅度的波动,敏感性的最大值为0.93,小于特异性的最小值0.97,这说明SMILESynergy在学习预测药物组合数据是否有协同作用的均衡性上有所欠缺。于是我们对O’Neil数据集中各组织药物组合的类别数量分布进行了可视化(数据图见附件2),发现O’Neil数据集中药物组合具有协同作用和无协同作用的数据分布不均衡,无协同作用的药物组合数量远远大于具有协同作用的药物组合数量,导致敏感性与特异性出现了差异。
表 2. SMILESynergy classification performance indicators on O’Neil.
SMILESynergy 在O’Neil 数据集上的分类性能指标
| 指标 | ACC | PREC | SENS | SPEC | F1 | ROC_AUC |
| 平均值 | 0.97 | 0.81 | 0.76 | 0.98 | 0.78 | 0.87 |
| 最小值 | 0.92 | 0.65 | 0.47 | 0.97 | 0.55 | 0.73 |
| 最大值 | 0.98 | 0.93 | 0.93 | 0.99 | 0.90 | 0.95 |
3.2. SMILESynergy 在NCI-ALMANAC数据集的性能评估
我们在NCI-ALMANAC数据集上对本文模型SMILESynergy进一步进行验证,获得了令人满意的结果(详见表3、表4)。在回归方面,MAE和MSE的中位数均小于其平均值,R²、EVS和Pearson的中位数均高于平均值。在分类方面,ACC的中位数与平均值接近,而其他指标的中位数均高于平均值,这表明SMILESynergy在NCI-ALMANAC数据集的大多数细胞系上表现出了良好的稳定性。
表 3. SMILESynergy regression performance metrics on NCI-ALMANAC.
SMILESynergy 在NCI-ALMANAC数据集上的回归性能指标
| 指标 | MAE | MSE | R2 | EVS | Pearson |
| 平均值 | 6.87 | 88.98 | 0.73 | 0.74 | 0.86 |
| 中位数 | 6.55 | 75.39 | 0.74 | 0.74 | 0.86 |
表 4. SMILESynergy classification performance indicators on NCI-ALMANAC.
SMILESynergy在NCI-ALMANAC 数据集上的分类性能指标
| 指标 | ACC | PREC | SENS | SPEC | F1 | ROC_AUC |
| 平均值 | 0.87 | 0.82 | 0.81 | 0.90 | 0.81 | 0.85 |
| 中位数 | 0.87 | 0.83 | 0.81 | 0.91 | 0.81 | 0.86 |
为了探究SMILESynergy在NCI-ALMANAC数据集中的预测性能,我们绘制了全局绝对误差热力图(见附件3)。我们发现不同细胞系下的药物组合数量存在差异,且大部分细胞系下的药物组合的绝对误差集中分布在10以内,这与实验结果中MAE的平均值和中位数皆在10以内相一致。
在HL-60(TB)、SR、MOLT-4和CCRF-CEM这四个细胞系中,绝对误差的波动相对较为明显。我们对这四个细胞系及热图中预测效果较好的HOP-92、SF-268、RXF 393和OVCAR-4四个细胞系数据分布进行可视化分析,如图5所示。结果显示HL-60(TB)、SR、MOLT-4和CCRF-CEM四个细胞系的细胞生长率分布较为分散,使得模型难以学习药物组合数据的特征规律,从而导致整体绝对误差波动较大。
图 5.
Scatter plot of cell growth rate percentage for selected cell lines in the NCI-ALMANAC dataset
NCI-ALMANAC 数据集中部分细胞系的细胞生长率百分比散点图
3.3. 方法对比
为了验证SMILESynergy模型的预测性能,我们在O’Neil数据集上与DeepSynergy模型[5]和MulinputSynergy模型[8]进行实验对比。我们的模型将药物转换成SMILES文件作为输入数据。DeepSynergy计算了药物三种不同类型的化学特征:首先使用jCompoundMapper[25]生成半径为6(ECFP_6)的扩展连接指纹计数,然后利用ChemoPy[26]计算药物的物理化学性质,最后从文献[27]中收集到一组亚结构毒团特征。MulinputSynergy则使用软件alvaDesc计算药物化合物的分子描述符,包括功能组、片段计数和药效团等。
三个模型详细的比较结果列于表5和表6。从表5中可以看出,SMILESynergy模型在预测抗癌药协同作用的回归任务中,相对于MulinputSynergy和DeepSynergy模型,具有更小的MSE和MAE,以及更高的EVS、Pearson和R2指标,这表明SMILESynergy模型能够更准确地预测抗癌药协同作用的强度,并且预测结果与实际值之间的相关性更强。从表6中可以看出,SMILESynergy模型在预测抗癌药协同作用的分类任务中,相对于MulinputSynergy和DeepSynergy模型,具有更高的ACC、PREC、SENS、F1和ROC_AUC指标,这表明SMILESynergy模型不仅能够准确地预测抗癌药协同作用的类别,还能够在预测结果的精度和召回率之间做到更好的平衡。
表 5. Comparison of regression performance metrics of SMILESynergy and other models.
SMILESynergy与其他模型的回归性能指标对比
| 模型 | MSE | MAE | EVS | Pearson | R 2 |
| SMILESynergy | 51.34±0.00 | 4.39±1.55 | 0.89±0.03 | 0.94±0.01 | 0.89±0.03 |
| MulinputSynergy | 176.69±3.09 | 8.77±0.12 | 0.58±0.01 | 0.76±0.00 | 0.57±0.01 |
| DeepSynergy | 197.14±1.63 | 9.48±0.04 | 0.53±0.01 | 0.73±0.00 | 0.53±0.00 |
表 6. Comparison of classification performance metrics of SMILESynergy and other models.
SMILESynergy与其他模型的分类性能指标对比
| 模型 | ACC | PREC | SENS | F1 | ROC_AUC |
| SMILESynergy | 0.97±0.01 | 0.81±0.07 | 0.76±0.10 | 0.78±0.07 | 0.87±0.05 |
| MulinputSynergy | 0.95±0.00 | 0.63±0.02 | 0.58±0.01 | 0.52±0.01 | 0.94±0.00 |
| DeepSynergy | 0.95±0.00 | 0.57±0.01 | 0.53±0.01 | 0.50±0.02 | 0.93±0.00 |
3.4. SMILES Enumeration消融实验
我们将SMILESynergy模型进行消融实验研究来分析SMILES Enumeration模块对抗癌药物协同作用预测的影响,分别评估了无SMILES Enumeration以及加入SMILES Enumeration后的影响,对比结果如表7所示,各指标皆为两模型在各自细胞系上的平均值。由表7可以看出,在加入SMILES Enumeration之后,模型在两个数据集上的回归与分类性能都得到了一定程度的提升。原因可能是经过SMILES Enumeration之后使得模型对药物组合数据的特征进行了更多的训练与学习,从而提升了自身的鲁棒性。
表 7. SMILES Enumeration ablation experiments of regression and classification.
SMILES Enumeration 消融实验回归及分类指标
| 模型 | 数据集 | 回归 | 分类 | |||
| MSE | Pearson | ACC | ROC-AUC | |||
| 无SMILES Enumeration | O’Neil | 244.58 | 0.77 | 0.93 | 0.77 | |
| SMILESynergy | O’Neil | 51.34 | 0.95 | 0.97 | 0.87 | |
| 无SMILES Enumeration | NCI-ALMANAC | 202.64 | 0.55 | 0.67 | 0.58 | |
| SMILESynergy | NCI-ALMANAC | 88.98 | 0.86 | 0.87 | 0.85 | |
4. 结论
我们提出了一种新的抗癌药物协同作用预测模型SMILESynergy。模型以药物的SMILES作为输入,并利用异构体数据进行数据增强,然后采用预训练的Transformer模型编码药物组合数据,最后通过连接MLP完成对药物组合协同作用的预测。在O’Neil数据集上,SMILESynergy优于其他模型,具有更好的预测性能,可帮助研究人员快速筛选最优药物组合,提高癌症治疗效果。数据及工程文件可在https://github.com/unclestrong/SMILESynergy获取。
重要声明
利益冲突声明:本文全体作者均声明不存在利益冲突。
作者贡献声明:张立强主要负责实验方案设计、代码编写与调试、实验结果分析与论文撰写,秦玉芳主要负责算法咨询与建议、实验结果分析、论文撰写与指导,陈明作者为论文提供了资助和支持。
本文附件见本刊网站的电子版本(biomedeng.cn)。
Funding Statement
国家自然科学基金(61702325);江苏现代农业产业关键技术创新(CX(20)2028)
References
- 1.Abeshouse A, Ahn J, Akbani R, et al The molecular taxonomy of primary prostate cancer. Cell. 2015;163(4):1011–1025. doi: 10.1016/j.cell.2015.10.025. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Housman G, Byler S, Heerboth S, et al Drug resistance in cancer: an overview. Cancers. 2014;6(3):1769–1792. doi: 10.3390/cancers6031769. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.Chou T C Theoretical basis, experimental design, and computerized simulation of synergism and antagonism in drug combination studies. Pharmacol Rev. 2006;58(3):621–681. doi: 10.1124/pr.58.3.10. [DOI] [PubMed] [Google Scholar]
- 4.Bajorath J Integration of virtual and high-throughput screening. Nat Rev Drug Discov. 2002;1(11):882–894. doi: 10.1038/nrd941. [DOI] [PubMed] [Google Scholar]
- 5.Preuer K, Lewis R P I, Hochreiter S, et al DeepSynergy: predicting anti-cancer drug synergy with deep learning. Bioinformatics. 2018;34(9):1538–1546. doi: 10.1093/bioinformatics/btx806. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.O’neil J, Benita Y, Feldman I, et al An unbiased oncology compound screen to identify novel combination strategies. Mol Cancer Ther. 2016;15(6):1155–1162. doi: 10.1158/1535-7163.MCT-15-0843. [DOI] [PubMed] [Google Scholar]
- 7.Zhang T, Zhang L, Payne P R O, et al Synergistic drug combination prediction by integrating multiomics data in deep learning models. Methods Mol Biol. 2021;2194:223–238. doi: 10.1007/978-1-0716-0849-4_12. [DOI] [PubMed] [Google Scholar]
- 8.陈希, 秦玉芳, 陈明, 等. 基于多输入神经网络的药物组合协同作用预测. 生物医学工程学杂志, 2020, 37(4): 676-682, 691.
- 9.Sun Z, Huang S, Jiang P, et al DTF: deep tensor factorization for predicting anticancer drug synergy. Bioinformatics. 2020;36(16):4483–4489. doi: 10.1093/bioinformatics/btaa287. [DOI] [PubMed] [Google Scholar]
- 10.Vaswani A, Shazeer N, Parmar N, et al. Attention is all you need// 31st Conference on Neural Information Processing Systems (NIPS 2017). Long Beach: NIPS, 2017: 6000-6010.
- 11.Schwaller P, Laino T, Gaudin T, et al Molecular Transformer: a model for uncertainty-calibrated chemical reaction prediction. ACS Cent Sci. 2019;5(9):1572–1583. doi: 10.1021/acscentsci.9b00576. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Wang S, Guo Y, Wang Y, et al. Smiles-Bert: Large scale unsupervised pre-training for molecular property prediction// BCB '19: Proceedings of the 10th ACM International Conference on Bioinformatics, Computational Biology and Health Informatics. Niagara Falls: Association for Computing Machinery, 2019: 429-436.
- 13.Tetko I V, Karpov P, Van Deursen R, et al State-of-the-art augmented NLP transformer models for direct and single-step retrosynthesis. Nat Commun. 2020;11(1):5575. doi: 10.1038/s41467-020-19266-y. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Honda S, Shi S, Ueda H R. Smiles transformer: pre-trained molecular fingerprint for low data drug discovery. arXiv preprint arXiv, 2019: 1911.04738.
- 15.He J, You H, Sandstrm E, et al Molecular optimization by capturing chemist's intuition using deep neural networks. J Cheminform. 2021;13(1):26. doi: 10.1186/s13321-021-00497-0. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Peters M E, Neumann M, Iyyer M, et al. Deep contextualized word representations// Proceedings of the 2018 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies. New Orleans: Association for Computational Linguistics, 2018: 2227-2237.
- 17.Devlin J, Chang M W, Lee K, et al. Bert: Pre-training of deep bidirectional transformers for language understanding. arXiv preprint arXiv, 2018: 1810.04805.
- 18.Weininger D SMILES, a chemical language and information system. 1. Introduction to methodology and encoding rules. J Chem Inf Comput Sci. 1988;28(1):31–36. [Google Scholar]
- 19.Liu Q, Xie L TranSynergy: Mechanism-driven interpretable deep neural network for the synergistic prediction and pathway deconvolution of drug combinations. PLoS Comput Biol. 2021;17(2):e1008653. doi: 10.1371/journal.pcbi.1008653. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Di Veroli G Y, Fornari C, Wang D, et al Combenefit: an interactive platform for the analysis and visualization of drug combinations. Bioinformatics. 2016;32(18):2866–2868. doi: 10.1093/bioinformatics/btw230. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.Holbeck S L, Camalier R, Crowell J A, et al The national cancer institute ALMANAC: a comprehensive screening resource for the detection of anticancer drug pairs with enhanced therapeutic activity. Cancer Res. 2017;77(13):3564–3576. doi: 10.1158/0008-5472.CAN-17-0489. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22.Landrum G. RDKit: A software suite for cheminformatics, computational chemistry, and predictive modeling. (2013) [2022-09-20]. http: //www.rdkit.org/RDKit_Overview.pdf.
- 23.Gaulton A, Hersey A, Nowotka M, et al The ChEMBL database in 2017. Nucleic Acids Res. 2017;45(D1):D945–D954. doi: 10.1093/nar/gkw1074. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.Pedregosa F, Varoquaux G, Gramfort A, et al Scikit-learn: Machine learning in Python. J Mach Learn Res. 2011;12:2825–2830. [Google Scholar]
- 25.Hinselmann G, Rosenbaum L, Jahn A, et al jCompoundMapper: An open source Java library and command-line tool for chemical fingerprints. J Cheminform. 2011;3(1):3. doi: 10.1186/1758-2946-3-3. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.Cao D S, Xu Q S, Hu Q N, et al ChemoPy: freely available python package for computational biology and chemoinformatics. Bioinformatics. 2013;29(8):1092–1094. doi: 10.1093/bioinformatics/btt105. [DOI] [PubMed] [Google Scholar]
- 27.Singh P K, Negi A, Gupta P K, et al Toxicophore exploration as a screening technology for drug design and discovery: techniques, scope and limitations. Arch Toxicol. 2016;90(8):1785–1802. doi: 10.1007/s00204-015-1587-5. [DOI] [PubMed] [Google Scholar]



