Abstract
近年来,深度学习为癌症预后分析提供了新的方法。对深度学习在癌症预后应用中的相关文献进行归纳总结,可为深入开展癌症预后研究提供借鉴和参考。因此,本文对深度学习在癌症预后模型中的最新研究进展进行了系统综述。首先,明确深度学习癌症预后模型的构建思路及性能评价指标;其次,介绍搭建模型所采用的基本网络结构,对所用数据类型、数据数量、具体网络架构及优缺点进行探讨;然后,验证构建深度学习癌症预后模型的主流方法并对实验结果进行分析;最后,对该领域现阶段面临的挑战及未来研究方向进行总结与展望。深度学习癌症预后模型与以往模型相比,能够更好地提高癌症患者的预后预测能力。未来我们应继续探索深度学习在癌症复发率、治疗方案、药物疗效评估等方面的研究,充分挖掘深度学习在癌症预后模型中的应用价值与潜力,以便建立一个高效精准的癌症预后模型,实现精准医疗的目标。
Keywords: 深度学习, 预后, 癌症预后模型, 精准医疗
Abstract
In recent years, deep learning has provided a new method for cancer prognosis analysis. The literatures related to the application of deep learning in the prognosis of cancer are summarized and their advantages and disadvantages are analyzed, which can be provided for in-depth research. Based on this, this paper systematically reviewed the latest research progress of deep learning in the construction of cancer prognosis model, and made an analysis on the strengths and weaknesses of relevant methods. Firstly, the construction idea and performance evaluation index of deep learning cancer prognosis model were clarified. Secondly, the basic network structure was introduced, and the data type, data amount, and specific network structures and their merits and demerits were discussed. Then, the mainstream method of establishing deep learning cancer prognosis model was verified and the experimental results were analyzed. Finally, the challenges and future research directions in this field were summarized and expected. Compared with the previous models, the deep learning cancer prognosis model can better improve the prognosis prediction ability of cancer patients. In the future, we should continue to explore the research of deep learning in cancer recurrence rate, cancer treatment program and drug efficacy evaluation, and fully explore the application value and potential of deep learning in cancer prognosis model, so as to establish an efficient and accurate cancer prognosis model and realize the goal of precision medicine.
Keywords: deep learning, prognosis, cancer prognosis model, precision medical treatment
引言
深度学习(deep learning,DL)是机器学习(machine learning,ML)的最新领域,通过 ML 中人工神经网络(artificial neural network,ANN)的深度叠加进行学习,根据低层特征组合形成更抽象的高层特征来实现分类或预测[1]。1943 年,McCulloch 等[2]开启了 ANN 的大门,但多层 ANN 反向传播存在梯度消失问题[3]。尽管万能逼近定理为 ANN 提供了强有力的理论支撑,DL 的探索仍几次陷入停滞[4]。直到研究人员发现了能够优化多层神经网络梯度消失问题的线性修正单元,DL 的发展才进入黄金时期[5]。因其自身的独特优势,DL 在医学领域的各个方向发展迅速[6-7]。
癌症预后是指预测癌症患者的可能病程和结局。世界卫生组织 2018 年癌症调查报告显示:全球癌症发病率和死亡率逐年上升,癌症死亡人数高达 960 万[8]。准确的癌症预后分析有助于提高癌症患者的生存机会。随着信息化技术的发展,为辅助临床医生选择合适的治疗方法与评估治疗效果,各种癌症预后模型应运而生。然而由于肿瘤的异质性,不同患者或同一患者不同时间内的预后也有差异,需采用不同的应对措施[9]。因此,探索个体化癌症预后模型具有重要的研究意义。
目前,国内外癌症预后领域已有不少研究成果。常小丽等[10]通过计算机或手工搜索多个数据库的文献,采用荟萃分析方法系统评估不同麻醉方案对癌症患者预后的影响。杨娟等[11]采集 128 例肝内胆管癌患者临床资料,使用 Logistic 回归分析法筛选预后危险因素以便预测患者术后复发风险。这些研究采用统计学方法评估和筛选影响癌症预后的相关因素或者构建癌症预后模型时,能获得的临床资料有限,为充分利用医学大数据中的关键信息以提高预后预测能力,研究者逐渐采用 ML 方法(如 ANN、决策树、随机森林等)构建癌症预后模型,这类方法在一定程度上提高了预测精度[12]。Chen 等[13]结合非小细胞肺癌患者临床资料与基因表达数据,以 3 年生存期为分类标准通过 ANN 建立生存风险模型(ACC 可达 83%)。一些学者根据头颈部癌症患者的放射组学数据,采用 ML 构建治疗效果预测模型,为推测正常组织并发症提供了新方法,有利于改善临床决策及放射治疗工作流程[14-15]。然而大规模医学数据中有效结合基因、蛋白质等组学特征仍存在挑战,医学图像中定量特征需要经验丰富且熟练的医师耗时费力地手工提取,给实际临床应用带来了限制。此外,有文献[16-17]指出手工提取的特征不如 DL 提取到的特征有效。因此,通过 DL 构建精准癌症预后模型逐渐受到研究者的青睐。这类模型旨在辅助医生制定个性化治疗策略以高效治疗癌症患者。
虽然 DL 已在癌症预后分析中取得了一些成果,但有关 DL 癌症预后模型的综述却相对较少。因此,本文主要从以下方面进行综述:① 概括 DL 癌症预后模型的构建思路。② 明确癌症预后模型性能评价指标。③ 以构建 DL 癌症预后模型的网络结构为切入点,归纳近几年 DL 癌症预后模型研究进展及各类模型优缺点,并对主流方法进行实验验证与结果分析。④ 总结并展望目前该领域面临的挑战及未来研究方向,以期为癌症预后研究提供一定的参考。
1. DL 癌症预后模型的构建思路
DL 在癌症预后模型构建中的作用主要为特征提取和特征选择。其建模步骤可概括为:① 确定具体预后角度(复发率、存活率、死亡率、治疗效果等)。② 进行数据采集和数据预处理。③ 将预处理后的数据划分为互不重合的训练集、验证集和测试集。④ 训练集训练模型以确定模型参数,验证集优化网络结构及超参数以确定最优模型。⑤ 测试集检验模型性能。具体流程如图 1 所示。
图 1.
Construction process of deep learning cancer prognosis model
深度学习癌症预后模型构建流程
2. 模型性能评价指标
准确性(accuracy,ACC):所有样本全部预测正确的概率。
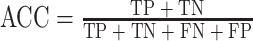 |
1 |
其中,
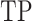 :正类预测为正类的样本数;
:正类预测为正类的样本数;
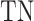 :负类预测为负类的样本数;
:负类预测为负类的样本数;
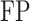 :负类预测为正类的样本数;
:负类预测为正类的样本数;
 :正类预测为负类的样本数。
:正类预测为负类的样本数。
敏感度(sensitivity,SEN):实际为正样本判断为正样本的概率。
 |
2 |
特异度(specificity,SPE):实际为负样本判断为负样本的概率。
 |
3 |
受试者工作特性曲线下面积(the area under the receiver operating characteristic curve,AUC)用于评价二值分类器效果,越接近 1 则分类器效果越好。
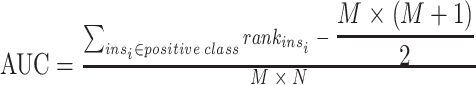 |
4 |
其中,
 :第
:第
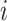 个样本的序号,概率得分从小到大排序,排在第
个样本的序号,概率得分从小到大排序,排在第
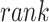 位置;
位置;
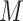 :正样本的个数;
:正样本的个数;
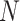 :负样本的个数;
:负样本的个数;
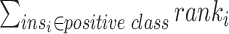 :将正样本的序号相加。
:将正样本的序号相加。
马修斯相关系数(Matthews correlation coefficient,MCC):实际类别和预测类别之间的相关系数,MCC 值越接近 1 则分类器效果越好。
 |
5 |
一致性指数(concordance index,C-index):用于评价生存模型的预测能力,值越接近 1 越好。
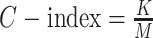 |
6 |
其中,
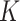 :所有样本互相配对时预测结果与实际相一致的配对数;
:所有样本互相配对时预测结果与实际相一致的配对数;
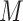 :无法判断是否一致的配对数。
:无法判断是否一致的配对数。
3. DL 癌症预后模型的研究进展
表 1[1, 18-36]按不同神经网络结构归纳了目前 DL 癌症预后模型的相关研究,包括文献作者、癌症类型、数据类型、网络架构、模型性能和预后临床终点。
表 1. Cancer prognosis models based on deep learning.
基于深度学习的癌症预后模型
| 文献 | 癌症类型 | 数据类型 | 网络架构 | 模型性能 | 预后临床终点 |
| 注:DNN:深度神经网络(deep neural networks);CNN:卷积神经网络(convolutional neural network);VGG:视觉几何群网络(visual geometry group network);LSTM:长短时记忆网络(long short-term memory);CT:计算机断层扫描(computed tomography);MRI:磁共振成像(magnetic resonance imaging);PET:正电子发射计算机断层显像(positron emission tomography);RNA:核糖核酸(ribonucleic acid);“—”:文献没有提及相应指标 | |||||
| Sun 等(2018) | 乳腺癌 | 临床、基因、
拷贝数变异 |
DNN | ACC:82.6%;AUC:0.845 | 5 年生存 |
| Lai 等(2020) | 非小细胞肺癌 | 基因、临床数据 | DNN | ACC:75.4%;AUC:0.816 | 5 年生存 |
| Lee 等(2020) | 非小细胞肺癌 | 临床、病理数据 | DNN | C-index:0.731;
AUC:0.768 |
术后无复发生存 |
| Zhu 等(2017) | 肺癌 | 病理图像 | CNN | C-index:0.629 | 总体生存 |
| Mobadersany 等(2018) | 脑胶质瘤 | 组织切片、
基因标志物 |
VGG-19 | C-index:0.754 | 总体生存 |
| Nagpal 等(2018) | 前列腺癌 | 组织切片 | Inception V3 | ACC:70.0% | 无进展间隔、复发 |
| Paul 等(2016) | 非小细胞肺癌 | CT 图像 | CNN_F | ACC:90.0%;AUC:0.935 | 总体生存 |
| Hosny 等(2018) | 非小细胞肺癌 | CT 图像 | 3D-CNN | AUC:0.700 | 2 年总体生存 |
| Diamant 等(2019) | 头颈部鳞状细胞癌 | CT 图像 | CNN | AUC:0.880 | 远处转移 |
| Lao 等(2017) | 脑胶质母细胞瘤 | MRI 图像、
临床数据 |
CNN_S | C-index:0.739 | 1 年、2 年、3 年生存 |
| Han 等(2020) | 高级别胶质瘤 | MRI 图像 | VGG-19 | — | 生存 |
| Tang 等(2020) | 胶质母细胞瘤 | MRI 图像 | CNN | — | 总体生存 |
| Bizzego 等(2018) | 头颈部鳞状细胞癌 | CT、PET 图像 | 3D-CNN | ACC:94.0%;SEN:67.0%;SPE:91.0%;MCC:0.748 | 局部复发 |
| Nie 等(2019) | 神经胶质瘤 | MRI 图像、
人口学特征 |
3D-CNN | ACC:90.7% | 生存 |
| Bychkov 等(2018) | 结直肠癌 | 组织切片 | VGG-16 +LSTM | AUC:0.690 | 5 年疾病特异性生存 |
| Zhang 等(2018) | 乳腺癌 | 基因数据 | Autoencoder | ACC:77.0%;SEN:84.0%;SPE:55.0%;MCC:0.320;AUC:0.740 | 5 年内远处转移 |
| Maggio 等(2018) | 神经母细胞瘤 | 转录组学 | Autoencoder | — | 5 年内的无事件生存、
总体生存 |
| Chaudhary 等(2018) | 肝癌 | RNA、miRNA 测序、
甲基化数据 |
Autoencoder | C-index:0.740 | 生存亚群分类 |
| Wong 等(2019) | 脑胶质母细胞瘤 | 基因数据 | DBN | C-index:0.690 | 生存 |
| Xie 等(2019) | 14 种癌症 | 多组学数据、
临床数据 |
DBN | — | 生存 |
3.1. 基于深度神经网络的癌症预后模型
深度神经网络(deep neural networks,DNN)由输入层、隐藏层和输出层组成[37],层与层之间节点完全连接。通过合并低层特征隐式地提取高层特征,从而进行癌症预后评估。网络结构如图 2 所示。
图 2.
Schematic diagram of DNN structure
DNN 结构示意图
Sun 等[18]提出一种结合多维数据的多模态 DNN 预后模型预测乳腺癌患者五年生存率。为有效整合 1 980 例患者的临床、基因、拷贝数变异数据,分别训练三个独立 DNN 后采用决策级多模态融合构建最终预后模型。其性能(ACC:82.6%;AUC:0.845)比一维预测模型更好。该方法的新颖之处在于集成网络结构的设计和多维数据的融合,验证了融合不同数据类型以提高癌症预后预测性能的有效途径。但获取大量临床资料代价昂贵,在多组学数据不完整时不适用。Lai 等[19]收集 614 例有完整临床资料的非小细胞肺癌患者资料,开发了一种结合基因表达异质性数据和临床数据的双峰 DNN。该模型能够预测患者的 5 年总体生存状态,ACC 可达 75.4%,AUC 为 0.816。其创新点在于采用 6 个独立数据集训练预后模型以提高模型泛化性,并将所用数据严格划分为互不重合的训练集、验证集和测试集。其次,双通道 DNN 能够更好地学习基因和临床数据的特征,提高预后模型精准度。Lee 等[20]纳入 1 320 例非小细胞肺癌患者,根据 8 位独立评审专家收集的 30 项临床和病理资料进行回顾性分析,提出了基于时间约束的 DNN 预后模型来预测患者术后无复发生存率。与传统统计方法相比,该模型(C-index:0.731;AUC:0.768)可以同时进行特征提取、特征选择和生存分析,为 DL 从电子病历中提取特征和进行临床信息学分析提供了一种有效手段。此外,多模态数据有利于癌症预后预测,将放射与病理图像与组学数据加入到该模型的输入特征中有望进一步提高预测性能。
以上研究表明,当医学数据类型为基因组学等高通量数据或专家认可的临床信息时,倾向于使用 DNN 构建 DL 癌症预后模型,但数据源的异质性和多样性对预后模型的影响也不可忽视。
3.2. 基于卷积神经网络的癌症预后模型
卷积神经网络(convolutional neural network,CNN)是一种广泛应用于图像识别和分类的 DL 模型[38]。CNN 基于图像中的肿瘤信息进行癌症预后分析,主要由卷积层、池化层和全连接层组成,结构如图 3 所示。卷积层通过移动卷积核得到覆盖整个图像的特征映射,输入池化层进行特征降维以减少神经元个数,然后将所有的特征图平铺成一维特征向量输入全连接层,最后特征向量通过分类器得到最终预测结果并予以输出。
图 3.
Schematic diagram of CNN structure
CNN 结构示意图
3.2.1. 基于组织病理图像
组织病理图像能够反映患者当前病变情况(如肿瘤良恶性、分化程度、组织病理分级等),随 ML 方法的演进,可挖掘出大量与预后相关的图像特征从而提示潜在的癌症进程。
Zhu 等[21]根据 404 例肺癌患者的病理图像,开发了一种新的用于评估生存风险分层的 DeepConvSurv 模型,该模型由三个卷积层、两个池化层和一个全连接层组成,C-index 值为 0.629,高于两种 Cox 模型(C-index:0.562;C-index:0.556)和随机森林生存模型(C-index:0.511)。DeepConvSurv 是第一个采用 CNN 与病理图像提高生存预测性能的研究,证明了 CNN 模型可以学习预后特征,为采用 DL 技术在生存分析中挖掘更多预后信息奠定了基础。格里森(Gleason)评分是前列腺癌重要的预后指标,Nagpal 等[1]收集 769 名前列腺癌患者 1 226 张组织切片,交由 32 位病理学家注释,通过深度 CNN 提取并选择图像特征,开发了一个基于 DL 的 Gleason 评分模型。该模型诊断 ACC 可达 70%,优于 29 位普通病理专家 61% 的平均准确率,解决了前列腺癌病理特征在显微镜检查中可重复性差的问题,对患者风险分层有更好的区分作用。Mobadersany 等[22]基于 769 例脑胶质瘤患者的 1 061 张组织切片,将 VGG-19 与 Cox 模型相结合,根据组织活检和基因标志物的显微图像预测患者生存。与当前使用的脑胶质瘤临床分类标准相比性能更优(C-index:0.754)。此外,该模型能够可视化与预后密切相关的重要结构(如微血管增生)以便病理学家分级,并且兼顾了不同患者间或患者个体中基因的异质性与遗传信息的丰富性,为进一步提高 DL 癌症预后模型的准确性、客观性和综合性做出了贡献。
上述研究表明:DL 对癌症预后预测有良好的指示作用,能够根据组织病理图像学习到病理专家无法直接定义的特征,还可避免专家因疲劳、主观情绪而遗漏细微病变,显示了融合多种信息且结合 DL 和回归分析方法提升癌症预后模型性能的可行性。但组织病理切片的样本质量要求较高(不得含有伪影、气泡、褶皱等)、制备较复杂,按照像素手动标注数据费时费力,都限制了基于组织病理数据构建 DL 癌症预后模型的普及。
3.2.2. 基于 CT、MRI、PET 图像
CT、MRI、PET 是癌症患者常用的检查方式,借助计算机技术,从这些影像中提取大量描述肿瘤特性的图像特征并进行定量分析,可为临床医生提供有价值的预后信息。
DL 需要比传统 ML 更多的数据来训练模型,但由于患者隐私保护和数据共享限制,CT、MRI、PET 图像中具有明确肿瘤标注信息的数据非常有限。因此,研究人员引入了迁移学习:用其他领域中已训练好的模型权重和新任务的数据对部分网络层进行再训练,实现采用小型医学数据集构建 DL 癌症预后模型的任务[39]。Paul 等[23]挑选 40 名非小细胞肺癌患者肿瘤面积最大的 CT 切片分割感兴趣区域,使用已训练好的 VGG-F、VGG-M、VGG-S 模型从最后一个隐藏层中提取深度特征,将其与强度分布、空间关系、纹理异质性等传统图像特征相结合,采用多种特征选择方法和多个分类器进行实验,最终确定采用 ACC 为 90.0%、AUC 为 0.935 的 VGG-F+贝叶斯、VGG-F+随机森林模型预测患者长短期存活率。但该研究数据太少且没有外部验证集验证模型性能,可靠性有待证实。Lao 等[24]收集 112 例多发性脑胶质母细胞瘤患者的术前多模态 MRI 图像和生存信息,提取 14 303 个传统图像特征和 98 304 个深度特征(由预先训练的 CNN_S 提取),合并后根据筛选出的 150 个图像特征与临床风险因素构建 Cox 模型预测患者生存结局。该模型 C-index 值为 0.739,高于临床风险因素模型(C-index:0.621),在预测患者风险分层方面取得了更好的效果。此外,基于迁移学习提取的深度特征与传统图像特征相结合改善了现有的预后模型性能,证明了深度特征在脑胶质母细胞患者术前护理中的潜力,但该研究仍存在样本量较小的缺陷。Han 等[25]也报告了一种结合 DL 和放射学模型预测高级别胶质瘤患者总体生存期的初步成果。该研究依次计算了 178 例患者的放射特征(348 个)和已训练好的 VGG-19 提取的深度特征(8 192 个),经特征选择后构建 Cox 模型预测长短期生存组。Han 等既进行了特征稳定性的验证,也对比了是否含有正常组织的肿瘤切片对生存预后的影响,但实验中各种特征的筛选较为复杂且描述较为模糊。作为肿瘤生物成像的第一个应用,Bizzego 等[26]采用 CT 和 PET 图像,根据已训练好的三维卷积神经网络(three dimensional convolutional neural network,3D-CNN)预测 298 例头颈部鳞癌患者局部复发。该模型包括两个并行级联的 3D-CNN,可对头颈部肿瘤分期诊断进行预训练,然后通过内部迁移学习对复发任务进行微调以获得深度特征,同时计算传统图像特征,最后合并两种特征训练分类器(SEN:67.0%;SPE:91.0%;ACC:94.0%;MCC:0.748)。该研究证明了在多模态 CT 和 PET 数据集内,混合深度特征和传统图像特征预测患者复发比仅使用一类特征或一种图像模式更为准确。但开发多模态 3D-CNN 是集成 PET 和 CT 图像的第一步,还需要更多实验来验证该方法的鲁棒性。Tang 等[27]提出一个多任务 CNN 预后模型共同完成肿瘤基因型和总体生存任务。该模型从 120 例胶质母细胞瘤患者的术前多模态 MRI 脑影像中提取肿瘤基因型相关特征,并将其用于生存预测。与其他最先进的方法相比,该模型生存预测精度最高。胶质母细胞瘤基因型是反映预后的有力指标,该多任务模型通过学习肿瘤基因型相关特征进行基因型预测,可以显著提高总体生存预测的准确性,并且为术前无创获得肿瘤基因型信息提供了解决思路。但是,以上迁移学习采用的模型均由自然界中彩色图片训练,与医学图像差别较大,可能会影响癌症预后模型性能。
Hosny 等[28]对五家机构共 1 194 例非小细胞肺癌患者的七个 CT 数据集进行了综合分析:以两年生存期为界限,训练 3D-CNN 预测患者死亡率风险。该模型 AUC 值为 0.70,优于基于临床参数建立的随机森林预后模型,同时可通过卷积可视化解释 3D-CNN 捕捉到的特征。该研究证明了 DL 用于预后风险分层的实用性,强调了肿瘤周围组织在患者分层中的重要性,进一步激发了通过 DL 制定个性化癌症治疗方案的前瞻性研究。Nie 等[29]纳入 83 名神经胶质瘤患者的术前多模态 MRI,使用多模态图像块构建多通道 3D-CNN 模型提取特征,结合患者人口学信息训练分类器预测长短生存期。其 ACC(90.7%)高于其他方法,体现了采用多模态 MRI 图像融合多通道 3D-CNN 构建的 DL 癌症预后模型的有效性。同年,Diamant 等[30]收集 300 例来自四家医学机构的头颈部鳞状细胞癌患者的治疗前 CT 图像,训练端到端的 CNN 预后模型评估癌症治疗效果。该模型以患者远处转移为终点,AUC 可达 0.880,融合传统定量特征后 AUC 提高到 0.920。虽然 CNN 不需要提前设计和利用机器学习算法选择特征,它允许算法本身根据预后目标自主学习,但 CNN 学习到的深度特征不能完全代表手工提取的肿瘤定量特征。
上述研究表明,与组织病理图像相比,根据治疗前 CT、MRI、PET 图像构建的 DL 癌症预后模型准确率更高。鉴于迁移学习常用模型中存在医学图像与自然界中彩色图片的差异问题,当构建癌症预后模型的数据集够大时,尽量不选择迁移学习法。在提高预后模型性能方面,深度特征与肿瘤形态学、空间、纹理特征的结合在一定程度上为今后工作指明了方向。但应用于临床实践之前,仍需进行多中心大样本的研究与验证。
3.3. 基于循环神经网络的癌症预后模型
循环神经网络(recurrent neural networks,RNN)由输入层、隐藏层和输出层组成,通过一个重复的隐藏状态(包括当前时间步的信息以及它在前一个时间步的状态)处理顺序输入从而使神经网络像人一样拥有记忆能力,适合处理临床上反映患者病况的时间序列数据,根据这些信息预测癌症预后[40]。但处理长期序列数据时 RNN 存在梯度消失的缺陷,因此长短时记忆网络(long short-term memory,LSTM)应运而生[41]。LSTM 主要由输入门、遗忘门、输出门及存储单元状态构成,可控制长期状态。RNN 和 LSTM 网络结构如图 4 所示。
图 4.
Schematic diagram of RNN and LSTM structure
RNN 和 LSTM 结构示意图
Bychkov 等[31]采用 420 名结直肠癌患者的临床病理数据,结合已训练好的 VGG-16 和 LSTM 构建基于肿瘤组织切片的预后模型预测患者五年生存率。该方法首先将所用切片分割为 38 万个尺寸为 224*224 的图像块,然后通过 VGG-16 进行特征提取并输入一维 LSTM 网络,最后逐块读取特征对图像块进行预测。AUC 可达 0.690,显著高于人类病理专家(AUC:0.580)的视觉评估诊断结果,说明 DL 可从癌症的组织形态中挖掘预后信息。该模型的亮点是 CNN 和 LSTM 两种网络的结合。它不需要像 CNN 一样输入固定尺寸的图像块,也不需要额外步骤合并单个图像的特征;并且 LSTM 可以处理任意序列长度的输入并记住长期依赖关系,能在数字病理工作流程中直接评估结直肠癌肿瘤样本,但其预测精度仍有待提升,需要纳入更多训练数据和外部数据以验证方法的普适性。
3.4. 基于自编码器的癌症预后模型
自编码器(autoencoder,AE)由编码器和解码器两部分构成[42]。编码器通过编码函数将输入压缩成潜在的空间特征,解码器通过解码函数将特征映射到输入空间,使用不同的训练规则令输出值等于输入值。AE 常用来降低特征维数和复杂性,通常只采用编码器得到输入数据的精髓,然后再构造模型学习,这样不仅能减轻 DL 网络的负担,还可以达到一个较好的效果。网络结构如图 5 所示。
图 5.
Schematic diagram of autoencoder structure
Autoencoder 网络结构示意图
Zhang 等[32]使用 AE 和主成分分析提取五个基因数据集的特征,构建 PCA-AE 集成预后模型预测乳腺癌患者远处转移。实验表明:AE 从基因中学习的特征可使模型具有良好的泛化能力;深度特征与传统特征结合后所构建的预后模型性能更优(ACC:76.8%;SEN:84.0%;SPE:55.0%;MCC:0.320;AUC:0.740)。但该模型不易分析,无法评估具体特征的重要性,需加入更多数据提高模型泛化能力。Maggio 等[33]提出一种新型多任务 AE 模型,首次将 498 例神经母细胞瘤的转录组学数据应用于生存分析,同时实现诊断与复发预测。多任务学习在训练时相当于隐式的数据增强,可以在一定程度上防止模型过拟合,提高预测准确率。但如果多个任务间的相关性差异较大,则不利于预后预测。Chaudhary 等[34]第一个填补了采用 DL 整合多组学数据构建癌症预后模型从而预测肝细胞癌患者生存率的研究空白。该模型纳入 360 例患者的 RNA 测序、miRNA 测序及甲基化数据,基于 AE 进行特征选择与优化以区分患者不同生存亚群,但 C-index 仅为 0.740。
以上三篇文献显示了采用组学数据构建 DL 癌症预后模型在预测患者生存方面的潜力。此外,其他预后因素(如年龄、既往史等)信息、多任务学习等技术也应整合到同一 DL 癌症预后模型中以提高精确度。
3.5. 基于深度置信网络的癌症预后模型
深度置信网络(deep belief network,DBN)由受限玻尔兹曼机(restricted Boltzmann machine,RBM)堆叠而成[42]。RBM 由可见层和隐藏层组成,同层神经元相互独立,不同层神经元相互连接且双向对称。DBN 第一层是一个独立的预先训练的 RBM,其隐藏层的输出是第二个 RBM 可见层输入,整个网络逐层训练,最后采用数据标签为监督信号计算网络误差,通过反向传播算法对整个网络中的参数微调。网络结构如图 6 所示。
图 6.
Schematic diagram of RBM and DBN structure
RBM 和 DBN 结构示意图
有研究证明在选择基因方面,DBN 比早期特征选择算法更具优势[43]。Wong 等[35]收集 469 例脑胶质母细胞瘤患者基因数据,利用含有两个隐藏层的 DBN 寻找与治疗耐药性密切相关的基因构建预后模型。实验证明,DBN 具备在多个抽象层次上表征数据的能力,可为患者生存提供非冗余的预后特征,有助于寻找可能成为治疗靶点的特定基因,为医生挑选合适的治疗随访方案提供参考。Xie 等[36]合并临床数据和基因、甲基化等多组学数据,开发了一种融合 DBN 和 Cox 模型进行生存预测的计算工具,该工具在 14 种癌症预后中表现良好,但未详细说明该工具预测每种癌症预后的具体性能。
上述研究表明:DL 可以构建基于微观特征的生存预后模型以进行个体化预后分析。虽然目前肿瘤患者的多组学诊断尚处于起步阶段,但随着基因组芯片与大规模高通量测序技术的飞速发展,结合 DBN 和 Cox 有助于患者预后的精准预测。
3.6. 基于不同网络结构的癌症预后模型对比
前述 3.1~3.5 小节以构建 DL 癌症预后模型的不同网络结构为主要分类标准,在简述网络结构的基础上,分别归纳了 5 种网络结构在癌症预后研究中的应用。为进一步比较与总结,表 2 列举了 DNN、CNN、RNN、AE 以及 DBN 网络架构的提出时间、优缺点、适用医学数据和发展趋势。
表 2. Comparison of cancer prognosis models with different network structures.
不同网络结构的癌症预后模型对比
| 网络结构 | 提出
时间 |
网络架构优点 | 网络架构缺点 | 适用医学数据 | 发展趋势 | |||||
| DNN | 1969 年 | ①可解决复杂问题建模;②可自动学习特征 | ①不能考虑平面结构信息;②时间序列数据不适用;③易过拟合 | ①各种组学数据;②带有特征属性的数据(性别、年龄、癌症分期等) | ①自动设计不同任务 DNN;②降低计算成本,减少耗时;③探索可解释性 | |||||
| CNN | 1989 年 | ①稀疏连接;②参数共享;③平移不变性;④适合处理高维数据 | ①梯度消散;②输入固定输出固定 | ①二维医学图像数据;②三维医学图像数据;③高维数据(基因组学数据等) | ①自动设计不同任务 CNN;②改进卷积核、卷积层通道、卷积层连接等以加速计算;③探索可解释性 | |||||
| RNN | 1982 年 | ①时间维度上的深度模型;②含有记忆单元;③可处理任意长度的输入和输出 | ①梯度消散(RNN 中称作长期依赖问题);②不具备特征学习的能力 | ①临床时间序列数据(序列间隔内心电、药物反映等生命体征情况);②已提取的医学特征 | ①探索可解释性;②寻找更优架构、设计新变体以减少训练时间;③与其他网络或技术结合 | |||||
| AE | 1987 年 | ①可用无标签数据进行模型预训练;②有较强的数据表征能力 | ①易出现过拟合;②梯度消散;③输入数据需平移不变 | ①各种组学数据;②已提取的医学特征 | ①深化网络结构改善性能;②加强约束条件;③改善信息分离的效果 | |||||
| DBN | 2006 年 | ①反映数据本身相似程度;②可用无标签数据训练模型以进行特征识别、数据分类 | ①用于分类时精度没有判别模型高;②输入数据需具有平移不变性 | ①各种组学数据;②二维医学图像数据 | ①改进 DBN 中 RBM 结构,降低计算复杂性;②与先进的分类器相结合 |
4. DL 癌症预后模型的方法验证
现有 DL 癌症预后模型半数以上采用 CNN 构建而成,多数研究倾向于癌症患者生存期的预测。因此,本文采用 TCIA 公共数据库 NSCLC-Radiomics 项目的 lung1 数据集,基于 CT 影像(尺寸:512*512,层厚:3 mm)和 CNN 构建模型预测具有临床意义的非小细胞肺癌患者 3 年生存期。根据患者存活状态和生存时间、CT 图像质量、肿瘤数量、肿瘤病理类型等筛选条件,入组 201 例患者。按患者随机划分互不重合的训练集(150 例)、验证集(21 例)和测试集(30 例)并给定标签[44]。由于数据较少,方法验证采用二维卷积神经网络(two dimensional convolutional neural network,2D-CNN)和迁移学习。参照表 1 总结的 DL 模型与表 2 中适用 CT 影像的网络结构,实验引入预先训练的三种经典 CNN(VGG-19、ResNet50、Inception V3)、LeNet、新型 2D-S_CNN 和新型密集连接 2D-S_CNN 训练并确定最优预后生存模型,使用相同测试集验证模型性能并进行分析与比较。不同模型预测结果如表 3 所示,本文设计的 CNN 网络结构如图 7 所示。由表 3 可知,迁移学习中 Inception V3 性能最好,AUC 可达 0.86,ACC 最高为 78.2%。与采用迁移学习构建的模型相比,本文设计的非小细胞肺癌预后生存模型性能更好,ACC、AUC 可达 79.5%、0.86。这些实验结果证明 DL 在癌症预后模型中的潜力:迁移学习有助于构建小型医学数据集的 DL 癌症预后模型,但预先训练所使用数据与医学数据不一致时,可能会对模型性能产生轻微影响。此外,根据具体任务设计的 DL 模型性能更佳。对比新型 2D-S_CNN 和新型密集连接 2D-S_CNN 预后生存模型实验结果发现:改进 DL 网络结构可小幅度提高癌症预后模型性能,因此将更多先进技术引入 DL 网络是未来癌症预后预测模型的发展趋势之一[45]。
表 3. Comparison of experimental results of survival models with different prognosis in non-small cell lung cancer.
非小细胞肺癌不同预后生存模型实验结果对比
| 预后生存模型
(DL 网络结构) |
模型总参数 | 预先训练 | 性能评价指标 | |||
| ACC | SEN | SPE | AUC | |||
| 经典深度学习网络 | ||||||
| VGG-19 | 20 155 969 | 是 | 73.0% | 71.2% | 76.3% | 0.84 |
| ResNet50 | 24 112 513 | 是 | 72.2% | 68.0% | 80.3% | 0.84 |
| Inception v3 | 23 901 985 | 是 | 78.2% | 81.0% | 73.2% | 0.86 |
| LeNet | 1 139 393 | 否 | 72.5% | 69.0% | 80.0% | 0.83 |
| 本文设计的深度学习网络 | ||||||
| 新型 2D-S_CNN | 287 103 | 否 | 77.2% | 80.5% | 71.6% | 0.84 |
| 新型密集连接 2D-S_CNN | 7 671 529 | 否 | 79.5% | 85.4% | 69.3% | 0.86 |
图 7.
The structure of the CNN network designed in this article
本文设计的 CNN 网络
5. 总结与展望
近几年研究表明:DL 主要用于癌症预后模型构建中的特征选择与特征提取方面,在辅助临床医生选择与制定个体化治疗与随访方案、延长癌症患者存活时间等方面具有重要意义。但现有成果大多是基于 DL 的回顾性研究,如何在临床实践中辅助医生决策尚未进行调查和测试。其展望如图 8 所示。
图 8.
Prospect diagram of deep learning cancer prognosis model
深度学习癌症预后模型展望图
癌症预后角度:生存预测是癌症预后分析的重点领域之一,在避免患者过度治疗、减少医疗费用、满足患者及家属特殊需求、开展临床试验等方面价值极大。目前大部分预后相关研究是预测癌症患者的风险分层与生存结局。此外,研究者应更多探究采用 DL 技术在预测癌症病情发展(缓解率、复发率等)、建议癌症治疗方案,以及预估癌症治疗(手术、放疗、药物等)效果等方面的应用。
预后数据类型:癌症在细胞起源、组织病理形态、临床表现、治疗反应等方面都存在极大的复杂性与异质性。因此,结合各类医学图像特征及衍生信息、潜在基因组、临床资料、专家注释和其他类型数据构建癌症预后模型越来越有价值。然而其他类型数据和 DL 算法仍有待深入研究调查。根据大量且匹配的多类医学数据,通过 DL 将微观特征与宏观特征深度融合,建立多尺度多分支多任务的癌症预后模型是未来发展的必然趋势。但 DL 在多种数据类型中的应用仍取决于数据的可用性以及数据提取和处理技术的发展。
编程语言与搭建平台:目前主要使用的编程语言是 Python 和 Matlab。实现平台包括 Keras、Caffe、Chainer、TensorFlow、Torch 等。这些平台是深度学习的核心技术,能够对模型进行训练和推理,管理癌症预后领域所需的大规模数据和模型,负责底层计算设备的调度和资源申请。未来在完善这些平台的同时,也需要开发更具创新性的新型开源平台。
深度学习网络结构:DL 具有优秀的非线性特征提取能力、高度的运算并行性以及良好的容错性与学习自动性。目前在癌症预后方面取得的进展得益于过去几十年中计算机和开源软件的发展、统计学理论与算法和计算能力的提高[46]。DL 依赖于大量数据的集合,由几千甚至数百万个标记的样本训练而成。然而匮乏的医学数据、共享的限制、低质量的医学标签是 DL 癌症预后模型普及的主要局限性。因此,小型医学数据集目前更多依赖迁移学习算法、轻量型 DL 网络架构或生成合成医学图像的方法。为此,我们要探索不同 DL 网络结构、不同数据集对提高模型准确率与稳健性的影响,探究采用无标注的原始医学数据集构建 DL 癌症预后模型的可行性。由于训练癌症预后模型所需的数据数量取决于应用程序,测试样本数量取决于公认标准,所以模型中所用训练集、验证集、测试集的最佳大小也应进一步摸索。
模型的可解释性:可解释性与复杂性密切相关,DL 癌症预后模型应用于临床的关键一方面在于模型的精准度,另一方面在于模型能否给出令医师充分理解的预测分析。虽然卷积可视化易于理解 DL 提取的深度特征,但这些特征与临床上用以描述肿瘤的高级语义特征有很大差别,难以分析二者的相关性。目前关于 DL 癌症预后模型解释方法的发展还处于相对早期的阶段,可通过深入探究模型并理解如何进行预测,或估计模型中每个输入特征的相关性或贡献来解释。这两类方法在与癌症预后相关的不同建模任务中有相对的灵活性和易适应性,但缺乏足够的工具提供更深入和新颖的理解。一些研究提出根据临床专家的先验知识进行 DL 癌症预后模型的设计,有助于提高 DL 在实验室和临床上的可接受性,产生新的假设并理解癌症状态的潜在机制[47]。但不同癌症类型在特定应用中的先验知识很难获得,并且需要考虑计算机的可读能力。此外,模型设计与训练中有时需要结合临床报告的信息,但临床报告的质量良莠不齐,其解释差别较大,仍需制定医师广泛接受的可用临床报告的参考标准。
模型评估标准:首先应说明所用数据集的纳入和排除标准,明确删除任何一例数据的理由;在模型训练过程中严格定义数据的训练集、验证集和测试集,三者应相互独立。其次,由于 DL 癌症预后模型容易过拟合,需使用来自另一机构的外部测试集对最佳模型进行评估以确定模型是否可以推广。此外,使用图像数据构建癌症预后模型时,需注意不同研究中心、不同医学扫描仪协议下的图像区别,最好在构建预后模型的每一个阶段(训练、验证、测试)均使用多中心医学成像数据,以便提高模型的稳健性和泛化性。由于一些研究已经证明了扫描仪设置(如重建技术和参数)对图像特征的影响,建议后续研究提供这方面的具体信息与处理方法[48]。同时要详细描述为构建癌症预后模型准备的数据,例如研究者将图像裁剪到某一小范围内,或者手动选择了相关图像。这些数据的预处理与注释会影响临床医生对 DL 癌症预后模型的理解。虽然目前研究人员经常将 AUC 值作为不同癌症预后模型的比较指标,但 AUC 值与临床医学关系不大。为了治疗患者,医生们更感兴趣的是将癌症预后模型的预测性能与专家本身进行比较。最后,为证实 DL 癌症预后模型的性能,其算法与程序最好通过 GitHub 等网站上公开发布。
6. 结语
DL 癌症预后模型最终目标是将探索性的解决方案应用到实际的临床预后分析中。随着医疗算法监管审批程序的完善,其临床应用的转化也会加快。在投入临床后,各种相关癌症预后研究仍需根据新的医学数据的增加而持续改进,期望 DL 癌症预后模型的研究能推动精准医疗发展,均衡我国各地医疗资源。
利益冲突声明:本文全体作者均声明不存在利益冲突。
Funding Statement
国家自然科学基金重点项目(81830052);上海市分子影像学重点实验室项目(18DZ2260400);上海市自然科学基金(20ZR1438300)
References
- 1.Nagpal K, Foote D, Liu Yun, et al Development and validation of a deep learning algorithm for improving Gleason scoring of prostate cancer. NPJ Digit Med. 2019;2(1):1–10. doi: 10.1038/s41746-018-0076-7. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.McCulloch W S, Pitts W J B O M B A logical calculus of the ideas immanent in nervous activity. Bull Math Biol. 1990;52(1-2):99–115; discussion 73-97. doi: 10.1016/S0092-8240(05)80006-0. [DOI] [PubMed] [Google Scholar]
- 3.Hameed A A, Karlik B, Salman M S Back-propagation algorithm with variable adaptive momentum. Knowledge-Based Systems. 2016;114:79–87. doi: 10.1016/j.knosys.2016.10.001. [DOI] [Google Scholar]
- 4.Hornik K, Stinchcombe M B, White H Multilayer feedforward networks are universal approximators. Neur Netw. 1989;2(5):359–366. doi: 10.1016/0893-6080(89)90020-8. [DOI] [Google Scholar]
- 5.Jiang X, Pang Y, Li X, et al Deep neural networks with Elastic Rectified Linear Units for object recognition. Neurocomputing. 2018;275:1132–1139. doi: 10.1016/j.neucom.2017.09.056. [DOI] [Google Scholar]
- 6.Greenspan H, Van Ginneken B, Summers R M Guest editorial deep learning in medical imaging: Overview and future promise of an exciting new technique. IEEE Trans Med Imaging. 2016;35(5):1153–1159. doi: 10.1109/TMI.2016.2553401. [DOI] [Google Scholar]
- 7.毛渤淳, 陈圣恺, 谢雨, 等 经典深度学习算法对中文随机对照试验智能判别应用. 中国循证医学杂志. 2019;19(11):1262–1267. [Google Scholar]
- 8.Bray F I, Ferlay J, Soerjomataram I, et al Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA: A Cancer Journal for Clinicians. 2018;68(6):394–424. doi: 10.3322/caac.21492. [DOI] [PubMed] [Google Scholar]
- 9.Dagogo-Jack I, Shaw A T Tumour heterogeneity and resistance to cancer therapies. Nat Rev Clin Oncol. 2018;15(2):81–94. doi: 10.1038/nrclinonc.2017.166. [DOI] [PubMed] [Google Scholar]
- 10.常小丽, 朱丹, 任许利, 等 全身麻醉复合硬膜外麻醉对肿瘤患者预后影响的 Meta 分析. 中国循证医学杂志. 2011;11(8):954–959. [Google Scholar]
- 11.杨娟, 母齐鸣, 谭琴 影响肝内胆管癌手术预后的危险因素 Logistic 回归分析. 江苏大学学报: 医学版. 2019;29(2):161–165. [Google Scholar]
- 12.Jake L, Min W, Deepika G, et al Big data application in biomedical research and health care: A literature review. Biomed Inform Insights. 2016;8:1–10. doi: 10.4137/BII.S31559. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Chen Y C, Ke W C, Chiu H W, et al Risk classification of cancer survival using ANN with gene expression data from multiple laboratories. Comput Biol Med. 2014;48(1):1–7. doi: 10.1016/j.compbiomed.2014.02.006. [DOI] [PubMed] [Google Scholar]
- 14.Giraud P, Giraud P, Gasnier A, et al Radiomics and machine learning for radiotherapy in head and neck cancers. Front Oncol. 2019;9:174–184. doi: 10.3389/fonc.2019.00174. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Lambin P, Leijenaar R T H, Deist T M, et al Radiomics: the bridge between medical imaging and personalized medicine. Nat Rev Clin Oncol. 2017;14(12):749–762. doi: 10.1038/nrclinonc.2017.141. [DOI] [PubMed] [Google Scholar]
- 16.Arimura H, Soufi M, Kamezawa H, et al Radiomics with artificial intelligence for precision medicine in radiation therapy. J Radiat Res. 2019;60(1):150–157. doi: 10.1093/jrr/rry077. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Kontos D, Summers M M D R, Giger M L Special section guest editorial: Radiomics and deep learning. J Med Imaging. 2018;4(4):041301. doi: 10.1117/1.JMI.4.4.041301. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Sun D, Wang M, Li A, et al A multimodal deep neural network for human breast cancer prognosis prediction by integrating multi-dimensional data. IEEE/ACM Trans Comput Biol Bioinform. 2019;16(3):841–850. doi: 10.1109/TCBB.2018.2806438. [DOI] [PubMed] [Google Scholar]
- 19.Lai Y, Chen W, Hsu T, et al Overall survival prediction of non-small cell lung cancer by integrating microarray and clinical data with deep learning. Sci Rep. 2020;10(1):4679. doi: 10.1038/s41598-020-61588-w. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Lee B, Chun S H, Hong J H, et al DeepBTS: Prediction of recurrence-free survival of non-small cell lung cancer using a time-binned deep neural network. Sci Rep. 2020;10(1):1952. doi: 10.1038/s41598-020-58722-z. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.Zhu X, Yao J, Huang J. Deep convolutional neural network for survival analysis with pathological images// IEEE International Conference on Bioinformatics & Biomedicine. Shenzhen: IEEE, 2016: 544-547.
- 22.Mobadersany P, Yousefi S, Amgad M, et al Predicting cancer outcomes from histology and genomics using convolutional networks. Proc Natl Acad Sci U S A. 2018;115(13):2970–2979. doi: 10.1073/pnas.1717139115. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.Paul R, Hawkins S H, Balagurunathan Y, et al Deep feature transfer learning in combination with traditional features predicts survival among patients with lung adenocarcinoma. Tomography. 2016;2(4):388–395. doi: 10.18383/j.tom.2016.00211. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.Lao J, Chen Y, Li Z, et al A deep learning-based radiomics model for prediction of survival in glioblastoma multiforme. Sci Rep. 2017;7(1):10353. doi: 10.1038/s41598-017-10649-8. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.Han W, Qin L, Bay C, et al Deep transfer learning and radiomics feature prediction of survival of patients with high-grade gliomas. Am J Neuroradiol. 2020;41(1):40–48. doi: 10.3174/ajnr.A6365. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.Bizzego A, Bussola N, Salvalai D, et al. Integrating deep and radiomics features in cancer bioimaging// 2019 IEEE Conference on Computational Intelligence in Bioinformatics and Computational Biology (CIBCB). Siena: IEEE, 2019: 1-8.
- 27.Tang Z, Xu Y, Jin L, et al Deep learning of imaging phenotype and genotype for predicting overall survival time of glioblastoma patients. IEEE Trans Med Imaging. 2020;39(6):2100–2109. doi: 10.1109/TMI.2020.2964310. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 28.Hosny A, Parmar C, Coroller T P, et al Deep learning for lung cancer prognostication: A retrospective multi-cohort radiomics study. PLoS Med. 2018;15(11):e1002711. doi: 10.1371/journal.pmed.1002711. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.Nie D, Lu J, Zhang H, et al Multi-channel 3D deep feature learning for survival time prediction of brain tumor patients using multi-modal neuroimages. Sci Rep. 2019;9(1):1103. doi: 10.1038/s41598-018-37387-9. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 30.Diamant A, Chatterjee A, Vallières M, et al Deep learning in head & neck cancer outcome prediction. Sci Rep. 2019;9(1):2764. doi: 10.1038/s41598-019-39206-1. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 31.Bychkov D, Linder N, Turkki R, et al Deep learning based tissue analysis predicts outcome in colorectal cancer. Sci Rep. 2018;8(1):3395. doi: 10.1038/s41598-018-21758-3. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 32.Zhang D, Zou L, Zhou X, et al Integrating feature selection and feature extraction methods with deep learning to predict clinical outcome of breast cancer. IEEE Access. 2018;6:28936–28944. doi: 10.1109/ACCESS.2018.2837654. [DOI] [Google Scholar]
- 33.Maggio V, Chierici M, Jurman G, et al Distillation of the clinical algorithm improves prognosis by multi-task deep learning in high-risk Neuroblastoma. PloS One. 2018;13(12):e0208924. doi: 10.1371/journal.pone.0208924. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 34.Chaudhary K, Poirion O, Lu L, et al Deep learning-based multi-omics integration robustly predicts survival in liver cancer. Clin Cancer Res. 2017;24(6):1248–1259. doi: 10.1158/1078-0432.CCR-17-0853. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 35.Wong K K L, Rostomily R C, Wong S T C Prognostic gene discovery in glioblastoma patients using deep learning. Cancers. 2019;11(1):53–68. doi: 10.3390/cancers11010053. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 36.Xie G, Dong C, Kong Y, et al Group lasso regularized deep learning for cancer prognosis from multi-omics and clinical features. Genes. 2019;10(3):240–255. doi: 10.3390/genes10030240. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 37.Esteva A, Kuprel B, Novoa R A, et al Dermatologist-level classification of skin cancer with deep neural networks. Nature. 2017;542(7639):115–118. doi: 10.1038/nature21056. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 38.Gu J, Wang Z, Kuen J, et al Recent advances in convolutional neural networks. Pattern Recogn. 2018;77:354–377. doi: 10.1016/j.patcog.2017.10.013. [DOI] [Google Scholar]
- 39.Pan S J, Yang Q A survey on transfer learning. IEEE Trans Knowl Data Eng. 2010;22(10):1345–1359. doi: 10.1109/TKDE.2009.191. [DOI] [Google Scholar]
- 40.Lyu H, Lu H, Mou L Learning a transferable change rule from a recurrent neural network for land cover change detection. Remote Sens. 2016;8(6):506–528. doi: 10.3390/rs8060506. [DOI] [Google Scholar]
- 41.Greff K, Srivastava R K, Koutnik J, et al LSTM: A search space odyssey. IEEE Trans Neur Netw. 2017;28(10):2222–2232. doi: 10.1109/TNNLS.2016.2582924. [DOI] [PubMed] [Google Scholar]
- 42.Liu W, Wang Z, Liu X, et al A survey of deep neural network architectures and their applications. Neurocomputing. 2017;234:11–26. doi: 10.1016/j.neucom.2016.12.038. [DOI] [Google Scholar]
- 43.Ibrahim R, Yousri N A, Ismail M A, et al. Multi-level gene/MiRNA feature selection using deep belief nets and active learning// 2014 36th Annual International Conference of the IEEE Engineering in Medicine and Biology Society. Chicago: IEEE, 2014: 3957-3960.
- 44.Bluemke D A, Moy L, Bredella M A, et al Assessing radiology research on artificial intelligence: A brief guide for authors, reviewers, and readers—From the Radiology editorial board . Radiology. 2020;294(3):487–489. doi: 10.1148/radiol.2019192515. [DOI] [PubMed] [Google Scholar]
- 45.Monkam P, Qi S, Ma H, et al Detection and classification of pulmonary nodules using convolutional neural networks: A survey. IEEE Access. 2019;7:78075–78091. doi: 10.1109/ACCESS.2019.2920980. [DOI] [Google Scholar]
- 46.Azuaje F Artificial intelligence for precision oncology: beyond patient stratification. NPJ Precis Oncol. 2019;3(1):6–11. doi: 10.1038/s41698-019-0078-1. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 47.Girardi D, Kung J, Kleiser R, et al Interactive knowledge discovery with the doctor-in-the-loop: a practical example of cerebral aneurysms research. Brain Inform. 2016;3(3):133–143. doi: 10.1007/s40708-016-0038-2. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 48.Yasaka K, Akai H, Mackin D, et al Precision of quantitative computed tomography texture analysis using image filtering: A phantom study for scanner variability. Medicine. 2018;96(21):e6993. doi: 10.1097/MD.0000000000006993. [DOI] [PMC free article] [PubMed] [Google Scholar]










