Abstract
目的
提出一种组织感知的对比度增强网络(T-ACEnet)对CT图像进行增强显示,并验证该结果对于现有器官分割任务的精度提升。
方法
原始CT图像通过映射生成具有肺窗、软组织窗对比度的低动态灰阶图像,监督子网络通过肺部掩膜学习感知肺部、腹腔软组织的最佳窗宽窗位设置。自监督子网络通过极值抑制损失函数保持器官边缘结构信息。增强网络生成的图像被用作分割网络的输入,进行腹部多器官的分割。
结果
T-ACEnet所生成的图像可以在一幅图像中包含更多窗口设置信息,便于医生进行病灶的初步筛查。且T-ACE图像在SSIM、QABF、VIFF、PSNR指标上相较于次优方法分别提升了0.51、0.26、0.10和14.14,MSE则降低了一个数量级。同时,T-ACE图像作为分割网络输入时,相较于原始CT图像,在不改变模型的情况下可以有效提高器官分割精度,5个分割定量指标均得到了提升,最大一项可提高4.16%。
结论
本研究所提出的T-ACEnet可以感知性地增强器官组织对比度,提供更全面、更连续的诊断信息,同时所生成的图像可以显著提高器官分割任务的表现。
Keywords: 计算机断层成像, 深度学习, CT图像可视化, 多器官分割
Abstract
Objective
To propose a tissue- aware contrast enhancement network (T- ACEnet) for CT image enhancement and validate its accuracy in CT image organ segmentation tasks.
Methods
The original CT images were mapped to generate low dynamic grayscale images with lung and soft tissue window contrasts, and the supervised sub-network learned to recognize the optimal window width and level setting of the lung and abdominal soft tissues via the lung mask. The self-supervised sub-network then used the extreme value suppression loss function to preserve more organ edge structure information. The images generated by the T-ACEnet were fed into the segmentation network to segment multiple abdominal organs.
Results
The images obtained by T-ACEnet were capable of providing more window setting information in a single image, which allowed the physicians to conduct preliminary screening of the lesions. Compared with the suboptimal methods, T-ACE images achieved improvements by 0.51, 0.26, 0.10, and 14.14 in SSIM, QABF, VIFF, and PSNR metrics, respectively, with a reduced MSE by an order of magnitude. When T-ACE images were used as input for segmentation networks, the organ segmentation accuracy could be effectively improved without changing the model as compared with the original CT images. All the 5 segmentation quantitative indices were improved, with the maximum improvement of 4.16%.
Conclusion
The T-ACEnet can perceptually improve the contrast of organ tissues and provide more comprehensive and continuous diagnostic information, and the T-ACE images generated using this method can significantly improve the performance of organ segmentation tasks.
Keywords: computed tomography, deep learning, CT image visualization, multi-organ segmentation
CT作为一种非侵入性的医学成像方式可以反映人体的内部特征[1]。CT图像有着超过2000个灰阶的灰度范围,又可称为高动态范围(HDR)图像[2]。而人类仅能分辨720个灰度变化[3],同时医用显示器只能呈现900多个灰度变化,对于CT图像都是远远不足的[4]。为了更好地观察特定组织,临床上往往需要多次切换窗宽窗位[5],其根本原因在于CT图像的HDR属性限制了医生从图像整体进行诊断[6]。但是已有的动态范围压缩方法或是不能同时为多个组织选择合适的对比度[7-11]或是图像表现不一定符合临床专家的诊断习惯[12]。针对不同组织自适应进行对比度增强的技术方法尚不完善,而本研究通过深度学习技术,以医生熟悉的组织对比度指导图像增强过程,从而感知性地为多组织器官同时进行对比度增强。
另一方面,脏器分割是智能化医疗中的关键任务[13]。随着深度学习的兴起,以U-Net为代表[14-17]的深度学习方法取得了比传统方法更优异的性能表现,从而成为了器官分割任务中的主流研究方向[18, 19]。但临床硬件条件始终是限制复杂网络模型部署应用的问题之一。有研究通过直方图均值化等方法增强CT图像进而提高分割精度[20],证明了此类对比度增强方法于图像分割的应用价值。类似地,通过T-ACEnet,CT图像组织间的对比度得到增强从而可以在不改变网络结构的条件下提高分割精度,减少硬件负担,也证明了T-ACE图像的潜在应用价值。
受限于硬件条件和人体视觉极限,医生常使用调整窗宽窗位的方法增强目标器官的对比度以满足下一步的诊断需求。还有研究者使用深度学习技术实现了CT图像自动设置窗宽、窗位[21]。但这种窗口设置方法在增强目标器官的同时往往又会导致其他器官的对比度下降,使得医生所获得的诊断信息不连贯、不系统。针对这一影响CT诊断效率提升的瓶颈以及以往方法的不足,本文提出基于半监督网络结构的组织感知对比度增强网络。T-ACEnet由一个监督子网络和自监督子网络组成,在监督子网络中CT图像经过2次线性映射后被肺部掩膜分割为两部分,并分别作为该子网络输出的指导;在自监督子网络中,一种边界平滑损失函数被用来提高组织边界的纹理细节、视觉效果。T-ACEnet可以在有限动态范围内自适应地为肺部和腹部软组织选取医生熟悉的对比度表现,解决了已有方法中不能多组织增强、不能顺应医生诊断习惯的问题。器官分割实验中,T-ACE图像与原始CT图像使用相同的分割网络模型,分割结果表明在不增加硬件负担的情况下,T-ACE图像相比于原始图像可以有效提高多器官分割任务的精度。T-ACEnet为医疗图像检阅提供了新思路,且T-ACE图像在病灶检测、器官分割等方面都展现了相比原始CT图像更具优势的一面。
1. 材料和方法
图 1说明了整个实验的具体流程。实验主要分为两部分,负责生成T-ACE图像的T-ACEnet模块以及负责CT图像多器官分割任务的分割模块。
图 1.

实验流程示意图
Illustration of the proposed T-ACEnet. A: The supervised sub-network. B: the self-supervised sub-network. C: the segmentation network.
1.1. T-ACEnet结构
图 1A、B分别是T-ACEnet的监督子网络和自监督子网络。监督子网络所使用的标签图像由图 2所示过程生成。原始CT图像经过2次线性窗口映射被调整到两个常见的窗口设置对比度(肺窗、软组织窗),然后通过肺部mask分为肺内、肺外区域。肺部mask来源于UNet网络所生成的肺部分割初步结果,然后由2个具有一到两年工作经验的相关人士进行勘误,尽量减少器官边界处的像素被错误分类的可能性。最终肺内、肺外区域组合为标签图像G。
图 2.

标签图像G的生成过程
Illustration of the label image G generation.
具体来说,窗宽窗位设置可以被视为基于CT值的分段函数。以肺窗Wmin = -1100HU, Wmax = -100HU为例,其映射公式如下:
 |
1 |
其中I(i, j) 表示CT图像中位于(i, j) 位置的像素,Ilung (i, j) 是肺窗映射的结果,DR指映射值的范围,DRsp为该映射范围的起点。在本次实验中,DR = 255,DRsp = 0,监督子网络部分所使用损失函数Lconver为:
 |
2 |
 |
3 |
其中 覆盖整个图像空间域,N表示组织区域的总个数,||·|| 1代表L1范数,Y'是监督子网络的输出。
覆盖整个图像空间域,N表示组织区域的总个数,||·|| 1代表L1范数,Y'是监督子网络的输出。
尽管在生成肺部mask时通过人工手段尽可能地减少了被错误分类的像素,但是其中仍然会残留不少误分类的像素。这些像素因为使用了错误的映射关系,会导致监督子网络的结果中出现不必要的伪影结构。为了消除该伪影并增强组织边界处的细节,一个自监督子网络被提出,使用基于均值滤波方法改进的极值抑制(EVS)损失函数,其表达式为:
 |
4 |
其中y表示来自于Y' 的像素,ei, ej表示器官边界处极值点的坐标,m', n'表示邻域内非极值点的坐标,N2表示邻域内非极值点的总数。这些非极值点也被认为是正确分类的像素,而极值点被认为是误分类的像素。β L表示距离权重,与当前像素点离肺部边缘的距离成反比。
最终T-ACEnet模块所使用的总的损失函数为:
 |
5 |
当前EVS算法处理仍然属于2D层次,极值点的选取局限于图像本身。为了获取图像上下文信息,以更好地保证组织边缘纹理细节,可以将该算法推广至3D层次。具体来说,式(4)中的极值点是在2D范围内进行判断,现将其判断范围扩大,如果像素点在相邻切片所组成的3D范围内仍然为极值点,则进行平滑处理。这种3D处理方法,本文将其称为T-ACE3D。
1.2. T-ACE图像分割实验
图 1C是本文所使用的分割网络结构。它可以被替换为任意的分割网络,以达到更好的实验结果,这里以Nested UNet为基础网络进行实验。为了评估T-ACE图像作为分割预处理的有效性,除了初始输入图像不同外其余条件均一致。首先,所有输入CT图像的值统一映射到[0-255],非增强CT图像通过最大最小归一化映射到该范围。模型输入通道数为1,而输出通道数为14(包括背景、脾脏、右肾、左肾、胆囊、食道、肝脏、胃、主动脉、下腔静脉、门静脉和脾静脉、胰腺、右肾上腺、左肾上腺)。使用学习率为0.001的Adam优化器,batch大小为8,训练epoch数为200以保障网络训练时的损失函数可以到达平台期。分割网络所使用的损失函数为Lovász-Softmax损失函数,其表达式为:
 |
6 |
其中C表示欲分类的总类别数,∆Jc为第c个类的Jaccard损失,m(c) 表示第c个类的像素误差。
1.3. 数据集及实验细节
本实验所使用的数据集来自MICCAI 2015 Multi-atlas Labeling Beyond the Cranial Vault(MLBCV)挑战赛。选择其中30例有器官分类标签的病人数据参与实验,包含13种腹部器官标签数据,具体器官在1.2节中有所介绍。所有数据在门静脉造影阶段重建得到,图像矩阵大小为512×512。在30例数据中,27例数据以8:2的比例被用于网络训练、验证,剩余3例数据被用于网络测试。T-ACEnet的子网络和分割网络均使用相同的Nested Unet网络结构,图 1C中展示了所用到的网络结构,其中VGG模块包含2个卷积层、批量归一化(BN)层以及整流线性单元组合(Relu)。T-ACEnet网络优化器选择Adam优化器,学习率设置为0.001,batch大小为8。
为验证T-ACEnet模型的优越性,5种对比度增强算法包括梯度域图像重建框架[23](GDRF)、自适应TVI-TMO算法[24](ATT)、混合L1-L0层分解方法[25](H3LD)、非均匀量化方法[26](NUQ)和图像尺寸依赖归一化方法[27](NISDN)与T-ACEnet进行对比,并通过结构相似性(SSIM)、峰值信噪比(PSNR)、均方根误差(MSE)、融合图像视觉保真度[28](VIFF)和QABF[29]对6种方法的结果进行测试。
为了比较T-ACE图像与原始CT图像对于分割精度的影响,本文选择了五种基于深度学习的分割方法,分别是作为Baseline的Nested UNet[16]、UNet[14]、结合Transformers和UNet的TransUNet[30]方法、可以捕获全局依赖关系和局部上下文的MissFormer[31]方法和可以克服医学图像放大倍率问题和失真问题的TFCN[32]方法。在实验中,Nested UNet进行2次实验,输入分别为T-ACE图像和原始CT图像,其他4种方法的输入仅为原始CT图像。同时灵敏度、Jaccard相似系数、马修斯系数(MCC)、精确率(Precision)以及Dice度量这五个指标被用于分割结果的量化评估。
2. 结果
2.1. T-ACE图像病灶可视化能力
图 3展示了不同窗宽窗位设置下的胸部CT图像。图 3A为肺窗设置下的胸部CT图像,图 3B为软组织窗设置下的胸部CT图像,可以看到蓝色箭头所指的钙化点在这两幅图像中容易与背景混淆,缺少对比度。图 3C表示为了凸显钙化点使用定制窗宽窗位的胸部CT图像,其窗口为[-700 0]HU,图 3D表示T-ACE图像,在这两幅图像中,钙化点与背景有较为清晰的对比,更易识别。
图 3.
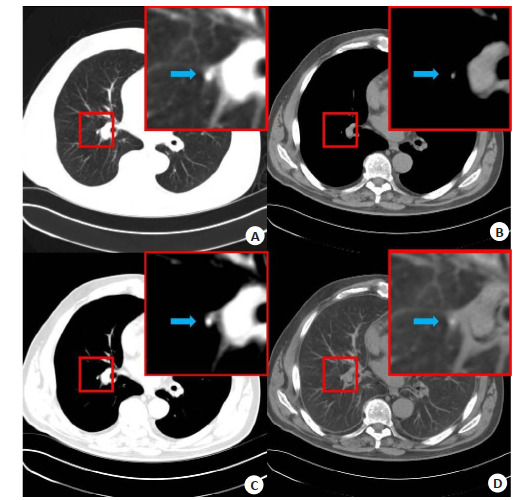
不同窗口设置下的胸部CT图像
Chest CT images with different window settings. A: Chest CT image with the "lung" window setting [-1100 -100] HU. B: Chest CT image with the "soft tissue" window setting [-250 200] HU. C: Chest CT image with a special window setting [-700 0] HU. D: The T-ACE image produced by the presented T-ACEnet method with the window setting [0 255].
图 4展示了不同窗宽窗位设置下的腹部CT图像。图 4A为肺窗设置下的腹部CT图像,图 4B为软组织窗设置下的腹部CT图像,图 4C为T-ACE腹部图像。蓝色指向的肝脏囊肿在图 4A完全无法辨别,而T-ACE图像则有着与图 4B相媲美的软组织病灶可视化能力,同时还提供了一部分肺部的结构细节信息。
图 4.

不同窗口设置下的腹部CT图像
Abdominal CT images with different window settings. A: Chest CT image with the "lung" window setting [-1100 -100] HU. B: Chest CT image with the "soft tissue" window setting [-250 200] HU. C: The T-ACE image produced by the presented T-ACEnet method with the window setting [0 255].
2.2. 对比度增强方法比较
图 5展示了不同对比度增强方法的实验结果。图 5A、B分别是肺窗设置下和软组织窗设置下的胸部CT图像,也可以认为是肺部和肺外部的金标准图像。图 5C~I分别是GDRF、ATT、H3LD、NUQ、NISDN、T-ACEnet以及T-ACE3D方法生成的增强结果。其中,GDRF、H3LD、NUQ和本文所提出的T-ACEnet、T-ACE3D方法在肺部的表现优于ATT和NISDN方法。ATT方法的结果整体呈现过曝光的状态,而NISDN方法则未能成功地显示更多肺部细节。至于肺外区域的视觉效果,T-ACEnet和T-ACE3D明显优于其他竞争方法,肺外结构的细节信息与噪声水平可以与金标准相媲美。
图 5.

不同对比度增强方法的胸部CT图像实验结果。
Chest CT images generated by different methods. A: Chest CT image with the "lung" window setting [-1100 -100] HU. B: Chest CT image with the "soft tissue" window setting [-250 200] HU. The contrast-enhanced images produced by GDRF (C), ATT (D), H3LD (E), NUQ (F), NISDN (G), T-ACEnet (H) and T-ACE3D (I) methods. The contrast-enhanced images are displayed in the same window setting [0 255].
表 1总结了不同方法得到的对比度增强图像的定量测量,其中SSIM、PSNR和MSE的值是肺内和肺外区域分别计算后的累加和,加粗标记的数字为该指标中表现最优的结果。
表 1.
不同对比度增强方法的量化评估
Quantitative assessments of the images generated using different methods
| Item | GDRF | ATT | H3LD | NUQ | NISDN | T-ACEnet | T-ACE3D |
| SSIM | 1.4560 | 1.2729 | 1.4583 | 1.3534 | 1.4367 | 1.9641 | 1.9631 |
| PSNR | 41.1613 | 32.0792 | 46.0644 | 45.6145 | 33.6596 | 60.2038 | 62.4714 |
| MSE | 1425 | 5952 | 2694 | 1442 | 3180 | 134 | 100 |
| QABF | 0.2901 | 0.1996 | 0.4275 | 0.3348 | 0.0542 | 0.6905 | 0.7004 |
| VIFF | 0.0317 | 0.0359 | 0.0606 | 0.0478 | 0.0117 | 0.1623 | 0.1765 |
2.3. T-ACE图像对分割精度的影响
T-ACE图像与原始CT图像作为分割网络输入的定性和定量结果如图 6所示。图 6A~E是5种深度学习方法的分割结果,其输入为原始CT图像;图 6E是输入为T-ACE图像的Nested UNet方法。可以看到,T-ACE图像的分割结果更加接近参考的分割结果。其中胃部组织(青绿色)仅有T-ACE图像可以较为完整地分割标记,肝脏下方的胆囊(淡黄色)仅有UNet和T-ACE可以较为完整地分类。
图 6.

不同分割对比方法在腹部的实验结果。右侧展示了不同颜色与组织间的对应关系
Results of abdominal image segmentation using different segmentation methods and the relationship between colors and tissues (right).
表 2展示了6种分割方法的量化指标评估。每个评价指标的计算仅展示Class 0即所有组织的结果,其中表现最优的结果以粗体突出显示。从表中可以看出,最近几年的方法相比于UNet方法均有一定的提升,其中T-ACE图像作为输入提升最大,在五个指标上相比于表现最差的方法分别提升了3.21%、4.92%、5.89%、5.65% 和3.34%。图 7是对表 2的扩充,图中不仅展示了Class 0的计算结果,更展示了每一个子类的结果。其中T-ACE图像结果以红线表示,可以看到在多数子类上T-ACE图像均位于前列。从折线图可知UNet、Nested UNet和TransUNet在Class 12(右肾上腺)这种小组织上表现极差,而淡绿色的MissFormer和红色的T-ACE图像在所有子类上均保持在一个较高的水平。
表 2.
六种分割方法的量化指标评估
Quantitative evaluation of 6 segmentation methods
| Item | UNet | NestedUNet | TransUNet | MissFormer | TFCN | T-ACE |
| Dice | 0.9209 | 0.9281 | 0.9318 | 0.9440 | 0.9194 | 0.9515 |
| Sensitivity | 0.8755 | 0.9122 | 0.9108 | 0.9069 | 0.9180 | 0.9247 |
| Precision | 0.9712 | 0.9445 | 0.9538 | 0.9842 | 0.9209 | 0.9798 |
| Jaccard | 0.8533 | 0.8658 | 0.8723 | 0.8939 | 0.8509 | 0.9074 |
| MCC | 0.9200 | 0.9262 | 0.9301 | 0.9432 | 0.9171 | 0.9505 |
图 7.
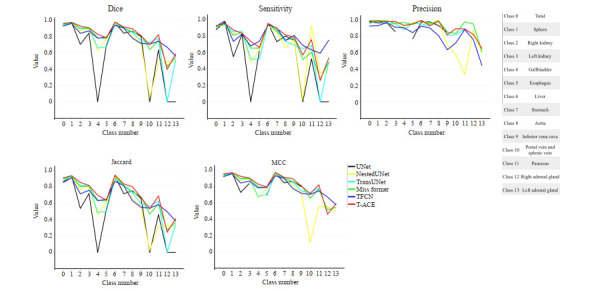
不同方法所得分割结果的定量评估
Quantitative assessment of segmentation results using different methods.
3. 讨论
本文提出的T-ACEnet可以自适应地为器官组织选择恰当的窗口映射关系,从而在一幅CT图像中包含多个窗口设置信息,为医生带来更便捷、更全面的诊断体验。有相关研究表明,类似的技术可用于临床,并且在诊断效率及病灶检测方面不输于传统调窗方法,甚至更优秀[5]。图 3、图 4分别展示了T-ACE图像在胸腔肺部与腹部软组织的病灶检测能力,其中图 3仅有特定的窗宽窗位设置与T-ACE图像可以分辨异常点,图 4的肝脏囊肿则可以在软组织窗图像和T-ACE图像中识别。但是特定的窗宽窗位意味着医生需要手动调整窗口设置,导致诊断效率的降低。而T-ACE图像融合了2种窗口设置信息,相比单一的窗口设置能够在同一时间内提供更多有效诊断信息。综上,T-ACE图像有潜力做到同时诊断肺内、肺外区域,为医生带来更系统、流畅的诊断体验,避免多次切换窗口设置导致的信息丢失。
而不同对比度增强的方法比较证明了本文所提出方法的优越性,T-ACEnet生成图像的肺部细节纹理可以媲美其他同样在肺部表现出色的方法如图 5C、E。至于肺外区域,仅有T-ACE图像(H)、(I)可以媲美金标准。用于评估图像质量的定量指标同样体现了这一点,如表 1所示,T-ACE3D方法取得了5个最优结果,SSIM的最优结果为T-ACEnet,二者相比于其他对比度增强方法在6个定量指标上均有极大的提升。虽然TACE3D方法可以取得更高的定量结果,但相比于2D TACEnet方法并没有很大提升,同时T-ACE3D在训练过程中需要加入图像上下文信息,导致网络训练用时约为T-ACEnet的2倍。考虑到训练时间与实验效果的平衡,本文在其他实验中依然以2D的T-ACEnet实验结果进行展示说明。以T-ACEnet为例,其在SSIM、QABF、VIFF、PSNR指标上相较于次优方法分别提升了0.51、0.26、0.10和14.14,MSE则降低了一个数量级。综合定性、定量实验结果,本文所提出的T-ACEnet和T-ACE3D方法可以获得优于同类方法的对比度增强图像。
为了进一步探寻T-ACE图像的应用场景,我们又看向了器官分割这一智能医疗领域内的关键任务。随着深度学习领域整体的不断进步,基于深度学习的器官分割精度也在随之提升,但是所使用的模型也在不断复杂化,庞大的参数量与医院落后的显卡设备之间的矛盾不可忽视,不能部署到临床实际使用的分割模型其应用场景终归有限。因此,本文验证了对比度增强图像对于分割精度的影响。实验表明,在不改动网络结构的情况下,使用T-ACE图像可以有效提高多器官分割任务的分割精度,减少错误标注的出现。在定量评估中,如表 2所示,以多器官共同评估的情况(Class 0)为例,在不改变模型的情况下,T-ACE图像相比于原始CT图像作为输入的Nested UNet方法,5个分割定量指标Dice、Sensitivity、Precision、Jaccard、MCC分别提升了2.34%、1.25%、3.53%、4.16%、2.43%,提升幅度最大的一项指标(Jaccard系数)提高了4.16%。综合定性、定量结果,本文所提出的T-ACE图像在腹部多组织分割任务中的性能表现要优于其他输入为原始CT图像的深度学习方法,其不仅可以准确地分割大组织,更可以在小组织上保持一定的分割精度,这也为一些小模型继续提高分割性能表现带来了可能。
本研究仍有一些问题有待解决,也是我们未来研究的方向。第一,在覆盖器官区域方面,我们目前的成果只是增强了两个区域(肺部区域和肺外区域)的对比度,没有实现额外的窗宽窗位设置。通过修改损失函数的输入,我们有望得到更多区域的增强显示,比如骨骼区域。第二,我们关注到重建滤波核对于CT图像的影响一样重要,而且和CT图像可视化有着相似的困扰,一个滤波核无法满足所有器官的诊断需要[33]。肺部需要更加锐利的滤波核确保细节,软组织需要更加平滑的滤波核抑制噪声,或许可以在之后研究中加入目前的组织感知网络中,在自适应地为器官选择恰当窗宽窗位的同时为其选择合适的滤波核,最终在一幅CT图像中包含多个滤波核、多个窗口设置,实现“All-in-one”[34]的视觉效果。
Biography
周昊,在读硕士研究生,E-mail: 1029941285@qq.com
Funding Statement
国家自然科学基金(U21A6005,12226004,U1708261);国家重点研发计划(2020YFA0712200)
Supported by National Natural Science Foundation of China (U21A6005, 12226004, U1708261)
Contributor Information
周 昊 (Hao ZHOU), Email: 1029941285@qq.com.
边 兆英 (Zhaoying BIAN), Email: zybian@smu.edu.cn.
References
- 1.Hsieh J. Computed Tomography: Principles, Design, Artifacts, and RecentAdvances. Bellingham, WA: SPIE PRESS; 2015. [Google Scholar]
- 2.Madmad T, Delinte N, De Vleeschouwer C. CNN-based morphological decomposition of X-ray images for details and defects contrast enhancement[C]//2021 IEEE/CVF Conference on Computer Vision and Pattern Recognition Workshops (CVPRW). June 19-25, 2021, Nashville, TN, USA. IEEE, 2021: 2170-80.
- 3.Lin WS, Ghinea G. Progress and opportunities in modelling justnoticeable difference (JND) for multimedia. IEEE Trans Multimed. 2022;24:3706–21. doi: 10.1109/TMM.2021.3106503. [DOI] [Google Scholar]
- 4.Mandell JC, Khurana B, Folio LR, et al. Clinical applications of a CT window blending algorithm: radio (relative attenuation-dependent image overlay) J Digit Imaging. 2017;30(3):358–68. doi: 10.1007/s10278-017-9941-1. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.A, Snoeckx Lesion detection on a combined "All-in-One" window compared to conventional window settings in thoracic oncology chest CT examinations. Diagn Interv Imaging. 2020;101(1):25–33. doi: 10.1016/j.diii.2019.07.009. [DOI] [PubMed] [Google Scholar]
- 6.Abdellatif W, Vasan V, Kay FU, et al. Know your way around acute unenhanced CT during global iodinated contrast crisis: a refresher to ED radiologists. Emerg Radiol. 2022;29(6):1019–31. doi: 10.1007/s10140-022-02085-7. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Lehr JL, Capek P. Histogram equalization of CT images. Radiology. 1985;154(1):163–9. doi: 10.1148/radiology.154.1.3964935. [DOI] [PubMed] [Google Scholar]
- 8.Pizer SM, Amburn EP, Austin JD, et al. Adaptive histogram equalization and its variations. Comput Vis Graph Image Process. 1987;39(3):355–68. doi: 10.1016/S0734-189X(87)80186-X. [DOI] [Google Scholar]
- 9.Fayad LM, Jin YP, Laine AF, et al. Chest CT window settings with multiscale adaptive histogram equalization: pilot study. Radiology. 2002;223(3):845–52. doi: 10.1148/radiol.2233010943. [DOI] [PubMed] [Google Scholar]
- 10.Mi ng, Ze ng. Improving histogram-based image contrast enhancement using gray-level information histogram with application to Xray images. Optik. 2012;123(6):511–20. doi: 10.1016/j.ijleo.2011.05.017. [DOI] [Google Scholar]
- 11.Mary Shyni H, Chitra E. A comparative study of X-ray and ct images in covid-19 detection using image processing and deep learning techniques. Comput Methods Programs Biomed Update. 2022;2:100054. doi: 10.1016/j.cmpbup.2022.100054. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Figueiro Longo MG, Vuylsteke P, Tan CO, et al. "All-in-one"window/level whole-body computed tomography scan-A faster way to evaluate trauma cases. Am J Emerg Med. 2022;62:62–8. doi: 10.1016/j.ajem.2022.09.047. [DOI] [PubMed] [Google Scholar]
- 13.马铭君. 基于深度学习的医学CT图像中器官与病灶分割算法研究[D]. 桂林: 广西师范大学, 2022.
- 14.Ronneberger O, Fischer P, Brox T. U-Net: Convolutional Networks for Biomedical Image Segmentation[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer, 2015: 234-241.
- 15.Çiçek Ö, Abdulkadir A, Lienkamp SS, et al. 3D U-net: learning dense volumetric segmentation from sparse annotation[C]//Medical Image Computing and Computer-Assisted Intervention-MICCAI 2016: 19th International Conference, Athens, Greece, October 17-21, 2016, Proceedings, Part Ⅱ. New York: ACM, 2016: 424-32.
- 16.Zhou ZW, Siddiquee MMR, Tajbakhsh N, et al. UNet++: a nested Unet architecture for medical image segmentation. Deep Learn Med Image Anal Multimodal Learn Clin Decis Support (2018) 2018;11045:3–11. doi: 10.1007/978-3-030-00889-5_1. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Ma MJ, Xia HY, Tan YM, et al. HT-Net: hierarchical contextattention transformer network for medical ct image segmentation. Appl Intell. 2022;52(9):10692–705. doi: 10.1007/s10489-021-03010-0. [DOI] [Google Scholar]
- 18.Wang RS, Lei T, Cui RX, et al. Medical image segmentation using deep learning: a survey. IET Image Process. 2022;16(5):1243–67. doi: 10.1049/ipr2.12419. [DOI] [Google Scholar]
- 19.Aljabri M, AlGhamdi M. A review on the use of deep learning for medical images segmentation. Neurocomputing. 2022;506:311–35. doi: 10.1016/j.neucom.2022.07.070. [DOI] [Google Scholar]
- 20.Islam M, Khan KN, Khan MS. Evaluation of preprocessing techniques for U-net based automated liver segmentation[C]//2021 International Conference on Artificial Intelligence (ICAI). April 5-7, 2021, Islamabad, Pakistan. IEEE, 2021: 187-92.
- 21.Heidarian S, Afshar P, Enshaei N, et al. Wso-caps: diagnosis of lung infection from low and ultra-lowdose CT scans using capsule networks and windowsetting optimization[C]//2021 IEEE International Conference on Autonomous Systems (ICAS). August 11-13, 2021, Montreal, QC, Canada. IEEE, 2021: 1-5.
- 22.Berman M, Triki AR, Blaschko MB. The lovasz-softmax loss: a tractable surrogate for the optimization of the intersection-overunion measure in neural networks[C]//2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition. June 18-23, 2018, Salt Lake City, UT, USA. IEEE, 2018: 4413-21.
- 23.Shibata T, Tanaka M, Okutomi M. Gradient-domain image reconstruction framework with intensity-range and base-structure constraints[C]//2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). June 27-30, 2016, Las Vegas, NV, USA. IEEE, 2016: 2745-53.
- 24.Khan IR, Rahardja S, Khan MM, et al. A tone-mapping technique based on histogram using a sensitivity model of the human visual system. IEEE Trans Ind Electron. 2018;65(4):3469–79. doi: 10.1109/TIE.2017.2760247. [DOI] [Google Scholar]
- 25.Liang ZT, Xu J, Zhang D, et al. A hybrid l1-l0 layer decomposition model for tone mapping[C]//2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition. June 18-23, 2018, Salt Lake City, UT, USA. IEEE, 2018: 4758-66.
- 26.Mehmood A, Khan IR, Dawood H, et al. A non-uniform quantization scheme for visualization of CT images. Math Biosci Eng. 2021;18(4):4311–26. doi: 10.3934/mbe.2021216. [DOI] [PubMed] [Google Scholar]
- 27.Al-Ameen Z, Sulong G, Johar G. Enhancing the contrast of CT medical images by employing a novel image size dependent normalization technique. Int J Biosci Biotechnol. 2012;4(3):63–8. [Google Scholar]
- 28.Yu Han. A new image fusion performance metric based on visual information fidelity. Inf Fusion. 2013;14(2):127–35. doi: 10.1016/j.inffus.2011.08.002. [DOI] [Google Scholar]
- 29.Xydeas CS, Petrović V. Objective image fusion performance measure. Electron Lett. 2000;36(4):308. doi: 10.1049/el:20000267. [DOI] [Google Scholar]
- 30.Chen J, Lu Y, Yu Q, et al. TransUNet: transformers make strong encoders for medical image segmentation"[EB/OL]. 2021: arXiv: 2102.04306. https://arxiv.org/abs/2102.04306".
- 31.Huang XH, Deng ZF, Li DD, et al. MISSFormer: an effective transformer for 2D medical image segmentation. IEEE Trans Med Imaging. 2022;PP(99):1. doi: 10.1109/TMI.2022.3230943. [DOI] [PubMed] [Google Scholar]
- 32.Li ZH, Li DH, Xu CB, et al. TFCNs: A CNN-Transformer Hybrid Network for Medical Image Segmentation[C]//International Conference on Artificial Neural Networks. Cham: Springer, 2022: 781-92.
- 33.Yang S, Kim EY, Ye JC. Continuous conversion of CT kernel using switchable CycleGAN with AdaIN. IEEE Trans Med Imaging. 2021;40(11):3015–29. doi: 10.1109/TMI.2021.3077615. [DOI] [PubMed] [Google Scholar]
- 34.Higashigaito K, Fischer G, Jungblut L, et al. Comparison of detection of trauma-related injuries using combined "all-in-one" fused images and conventionally reconstructed images in acute trauma CT. Eur Radiol. 2022;32(6):3903–11. doi: 10.1007/s00330-021-08473-w. [DOI] [PubMed] [Google Scholar]


