Abstract
目的
探索微RNA(miRNA,miR-)-let-7e-3p在不同宫颈病变中表达情况及临床意义。
方法
收集并整理具有完整临床病理资料的病例,其中正常子宫颈26例、子宫颈高级别鳞状上皮内病变(HSIL)37例、宫颈癌101例;采用实时定量RT-PCR检测临床宫颈组织样本中miR-let-7e-3p的相对表达量,分析miR-let-7e-3p表达水平与宫颈癌的发生及临床不良预后因素间的相关性。将miR-let-7e-3p模拟物转染至宫颈癌细胞株Siha(转染组),采用流式细胞仪检测Siha细胞周期和细胞凋亡变化;CCK-8试剂盒检测Siha细胞增殖变化;Transwell实验检测Siha细胞迁移和侵袭能力变化。
结果
miR-let-7e-3p在正常宫颈、HSIL及宫颈癌组织中的相对表达量分别为1.45±0.24、0.79±0.05、0.46±0.04,正常子宫颈与HSIL、正常子宫颈与宫颈癌、HSIL与宫颈癌相对表达量差异均具有统计学意义(均 P < 0.05);miR-let-7e-3p表达水平与患者宫颈癌临床不良预后因素之间无显著相关性;转染组S期细胞(29.76%±6.60%)高于阴性对照组(13.38%±1.30%, P < 0.05);转染组凋亡率(5.98%±1.38%)高于阴性对照组(3.53%±0.79%, P < 0.05);转染组转染后48、72、96 h吸光度值分别为0.57±0.11、0.65±0.04、0.84±0.14,阴性对照组为0.74±0.05、0.93±0.10、1.47±0.14,细胞增殖均被抑制(均 P < 0.05);转染组与阴性对照组迁移和侵袭能力差异均无统计学意义(均 P>0.05)。
结论
miR-let-7e-3p可能在宫颈癌的发生中发挥抑癌基因的作用,阻滞细胞周期和抑制细胞增殖可能是其重要调控机制。
Abstract
Objective
To investigate the expression of microRNA (miRNA, miR) let-7e-3p in different cervical lesions and its clinical significance.
Methods
The expression of miR-let-7e-3p in the tissues of normal cervix ( n=26), high-grade squamous intraepithelial lesion (HSIL) ( n=37), and cervix carcinoma ( n=101) were detected by reverse transcription and quantitative polymerase chain reaction (RT-qPCR). The correlation of miR-let-7e-3p expression with the clinicopathological parameters of patients with cervical cancer was analyzed. miR-let-7e-3p mimic was transfected into cervical carcinoma Siha cells. The cell cycle and apoptosis were determined by flow cytometry; cell proliferation was determined by CCK-8 kit; and the migration and invasion of cells were determined by Transwell assay.
Results
The relative expression levels of miR-let-7e-3p in normal cervix, HSIL, and cervical carcinoma were 1.45±0.24, 0.79±0.05 and 0.46±0.04, respectively (all P < 0.05). After transfection with miR-let-7e-3p mimic, the S-phase fraction and apoptosis rate of Siha cells were increased significantly compared with control group[(29.76±6.6)% vs (13.38±1.3)%, P < 0.05; (5.98±1.38)% vs (3.53±0.79)%, P < 0.05, respectively]. OD of transfected Siha cells at 48, 72 and 96 h were 0.57±0.11, 0.65±0.04 and 0.84±0.14, which were significantly lower than those of untransfected Siha cells (0.74±0.05, 0.93±0.10 and 1.47±0.14, all P < 0.05). The migration and invasion abilities of transfected Siha cells were not significantly changed (all P>0.05).
Conclusion
The expression of miR-let-7e-3p is down-regulated in cervical neoplasms, which is associated with cell cycle arrest and proliferation inhibition of cervical cancer cells.
Keywords: MicroRNAs; Uterine cervical neoplasms/pathology; Cervical intraepithelial neoplasia/pathology; Genes, tumor suppressor; Cell cycle; Cell proliferation; Gene expression; Reverse transcriptase polymerase chain reaction
宫颈癌是全球女性第四大常见恶性肿瘤,国际癌症研究机构(IARC)2012年癌症统计分析,宫颈癌每年新发约527 000例,同时约265 700例死于宫颈癌,其中80%以上的病例发生在发展中国家 [ 1] 。人乳头瘤病毒疫苗的问世和宫颈癌的筛查大幅度降低了发达国家的宫颈癌发病率和病死率 [ 2] ,但宫颈癌在发展中国家仍然是第二大常见女性恶性肿瘤,严重危害妇女健康。近年有关微RNA(microRNA,miRNA,miR-)与宫颈癌关系的研究日益增多。在宫颈癌患者中,miRNA表达谱存在明显异常,提示miRNA与宫颈癌的发生、发展密切相关 [ 3- 5] 。Lethal-7(let-7)家族是目前研究最多的miRNA家族之一,人类let-7家族有18个高度同源的成员,由于序列相似、5’末端拥有共同的保守序列“TGAGGTA”,所以家族成员在生物体内的功能有很多类似 [ 6] 。已经证实,let-7家族在生长发育、肿瘤发生发展、氧化应激、线粒体凋亡等许多过程中均起到重要调控作用 [ 7- 8] 。let-7家族成员在多种肿瘤组织中表达下降 [ 7- 10] ,但其与宫颈癌的关系研究较少。miR-let-7e是miR-let-7家族重要成员,miR-let-7e-3p(来自3’端臂)表达稳定,本文拟检测不同病理学改变的宫颈组织中miR-let-7e-3p相对表达量,并研究其对宫颈癌细胞株生物学行为的影响,探索其在宫颈癌发生、发展中的作用特点及相关机制,以期为宫颈癌防治提供新的靶标。
宫颈组织标本取自2013年3月至2014年4月在浙江大学医学院附属妇产科医院诊治的不同宫颈病理学改变的164例患者,其中子宫颈组织正常患者26例、子宫颈高级别鳞状上皮内病变(high-grade squamous intraepithelial lesion,HSIL) 患者37例和宫颈癌患者101例。其中,101例宫颈癌患者确诊原发性宫颈鳞状细胞癌,已接受根治性子宫切除和盆腔淋巴结切除,并有完整的临床资料。所有患者在接受手术获取标本前未接受过放射或化学药物等治疗。收集其临床病理参数。标本取出后立即放入液氮,储存于-80 ℃冰箱。宫颈标本收集经过浙江大学医学院附属妇产科医院伦理委员会审查批准,入选病例均签署知情同意书。
人子宫颈鳞癌系Siha细胞购自中国科学院上海生科院细胞资源中心;Siha细胞生长于含体积分数为10%灭活FBS、1×10 5U/L青霉素和100 mg/L链霉素的DMEM完全培养基中,置于37 ℃、5%二氧化碳饱和湿度培养箱内培养。细胞经培养传代,取对数生长期细胞进行实验。
Trizol试剂购自美国Invitrogen公司;FBS购自浙江天杭生物科技股份有限公司;DMEM购自美国Gibco公司;转染试剂Dharma FECT Transfection Reagent购自美国Thermo Scientific公司;荧光定量RT-PCR试剂盒购自日本TaKaRa公司; OPTI-MEM培养基购自美国Gibco公司;细胞周期试剂盒购自中国SAB公司;Annexin V-FITC/PI 凋亡检测试剂盒购自中国SAB公司;基质胶购自美国BD公司;miRNA-let-7e-3p模拟物(以下简称miRNA-let-7e-3p)和阴性对照miRNA购自中国Biomics公司;引物购自中国Biomics公司;Cell Counting Kit-WST-8(CCK-8)试剂盒购自中国上海东仁化学科技公司;ELISA测定仪Thermo Scientific Varioskan Flash购自美国Thermo Scientific 公司;流式细胞仪购自德国MACSQuant Analyzer公司,PCR仪购自美国ABI 7900公司。
用Trizol试剂抽提总RNA,Thermo Nano Drop 2000分光光度计测定RNA的浓度。以0.5 μg RNA进行逆转录,按照试剂盒说明反应分两步:第一步 RNA变性,反应体系:RNA 0.5 μg,去RNA酶DEPC水补足至6.8 μL。反应条件:70 ℃孵育10 min,放置冰上;第二步逆转录,反应体系:5×RT缓冲液(含dNTPs) 2 μL,RT 引物 0.2 μL,RNA酶0.2 μL,逆转录酶 0.8 μL,变性后的RNA 6.8 μL。反应条件:42 ℃孵育60 min,85 ℃灭活5 min后,-20 ℃保存。取1 μL逆转录产物进行荧光定量PCR反应,反应条件:95 ℃ 10 s,随后95 ℃5 s和60 ℃ 30 s共40个循环。采用2 -ΔΔct [ 11] 法计算样本中miR-let-7e-3p的相对表达量。
采用Dharma FECT转染Siha细胞。用胰蛋白酶消化Siha细胞,用完全培养基收集细胞,计数细胞,Siha细胞以3.0×10 5/孔密度接种于六孔板,常规条件培养。过夜后的细胞状态生长良好,融合度达到50%~60%,进行细胞转染。实验分三组,每组均设复孔以平衡实验误差。转染组:miR-let-7e-3p mimic+转染试剂;阴性对照组:miR-let-7e-3p阴性对照+转染试剂;空白对照组:仅加转染试剂。配制转染工作溶液:用去RNA酶的DEPC水溶解miR-let-7e-3p和miR-let-7e-3p阴性对照,分别配制成20 μmol/L的工作溶液。10μL/管分装,-20 ℃冻存备用。取5 μL的miR-let-7e-3p或miR-let-7e-3p阴性对照储存液,分别加入195 μL OPTI-MEM培养基,混匀,配成A液;取5 μL Dharma FECT Transfection Reagent缓慢加入195 μL OPTI-MEM培养基中,混匀,配成B液,室温下孵育5 min;将A液缓慢加入B液中,轻轻吹打混匀,室温下孵育20 min;将DMEM完全培养基1.6 mL缓慢加入AB混合物至终体积2 mL;将2 mL的混合液加入细胞培养板,每孔1 mL,各2个平行复孔。miR-let-7e-3p的终浓度为50 nmol/L。转染24 h更换成新鲜的完全培养基。随后根据实验需要在合适的时间点收集细胞进行下一步实验。
用胰蛋白酶消化Siha细胞,用完全培养基收集细胞,计数细胞,Siha细胞以5000~6000/孔密度接种于96孔板。常规条件培养。过夜后的细胞生长状态良好,融合度达到50%~60%,进行细胞转染。分别于24、48、72、96 h后取出培养箱,每孔加入10 μL CCK-8,37 ℃避光孵育1 h后取出在酶联免疫测定仪上测定波长450 nm处各孔对应的吸光度值,每组设5 个复孔,完成三轮重复实验后汇总,以不同的时间点为横轴,吸光度值为纵轴绘制细胞生长曲线。
上述转染细胞于48 h后,用不含EDTA的胰蛋白酶消化收集10 5个细胞,PBS缓冲液清洗两遍,离心弃上清液。加入1 mL 70%的无水乙醇(PBS稀释)固定-20 ℃过夜。离心细胞,弃去乙醇,加入1 mL PBS放置15 min使细胞再次水化。离心弃上清液,加入1 mL DNA 着色溶液,涡旋振荡5~10 s混匀。室温避光孵育30 min后进行微流式细胞检测,计算细胞周期的百分比。细胞凋亡的检测:上述转染细胞于48 h后取出,用不含EDTA的胰蛋白酶消化收集10 5个细胞,PBS缓冲液清洗两遍,离心弃上清液。取500 μL 1×结合缓冲液重悬细胞。每管加入5 μL Annexin V-FITC和10 μL PI。轻轻涡旋后,室温避光孵育5 min后进行微流式细胞检测,计算细胞凋亡的百分比。
Siha细胞经常规接种培养,按上述转染方法转染miR-let-7e-3p mimic(转染组) 和miR-let-7e-3p阴性对照(阴性对照组),转染后24 h胰蛋白酶消化收集细胞,无血清培养基重悬细胞,计数,配成细胞悬液,细胞密度3×10 5/mL。每个小室上室加入200 μL 细胞悬液,小室下室加入500 μL 含10% FBS的完全培养基,置培养箱继续培养4 h。取出Transwell 小室,PBS 洗三次,用棉签小心擦去上室表面的细胞,倒置晾干,用10%乙醇配置0.1%的结晶紫溶液,将晾干的小室浸入结晶紫溶液,固定染色30 min,PBS 洗三遍,显微镜下观察、拍照,计数,每个小室计数5个视野,取平均数,三次结果用于统计分析。
将冻存于-20℃的基质胶取出放置4 ℃溶化,待1 h后变成液态备用。所用的无菌24孔培养板、Transwell小室、灭菌移液器枪头和Eppendorf管均于4 ℃预冷备用。取基质胶和 OPTI-MEM 以1∶6 比例稀释混匀(冰浴操作),每个小室上室加入100 μL,避免气泡形成,放置在37 ℃培养箱中30 min 待其凝固。使用前吸去小室内析出的液体。其余步骤参照细胞迁移实验。
采用SPSS 19.0统计软件对实验数据进行统计分析,计量资料两样本均数之间比较用单因素方差分析,实验数据用均数±标准差( x ± s)表示,分类变量采用 χ 2检验,连续变量采用 t检验, P<0.05为差异有统计学意义。
正常宫颈、HSIL与宫颈癌组织样本中miR-let-7e-3p的相对表达量分别为1.45±0.24、0.79±0.08、0.46±0.04。经统计学处理,这三组间miR-let-7e-3p的相对表达量差异有统计学意义( P<0.05)。进一步两两比较,正常宫颈与HSIL、正常宫颈与宫颈癌和HSIL与宫颈癌组织之间miR-let-7e-3p的相对表达量差异均有统计学意义(均 P<0.05)。提示miR-let-7e-3p可能在宫颈癌的发生、发展过程中发挥作用。
按照miR-let-7e-3p表达水平从高到低排列,以第75位百分点作为截点,将宫颈癌组织表达miR-let-7e-3p水平分为高和低两个水平。结果发现宫颈癌组织高或低miR-let-7e-3p的表达水平与宫颈癌各临床不良预后因素均无显著相关性,见 表 1。提示miR-let-7e-3p表达下降可能是宫颈癌变发生过程中的早期事件,与宫颈癌进展关系不大。
表1 不同miR-let-7e-3p表达水平宫颈癌患者临床预后不良因素比较(例)
Table 1 Relationship between miR-let-7e-3p expression and clinical prognosis of cervical cancer
|
( n) | |||||
|
因素 |
miR-let-7e-3p |
χ 2值 |
P值 |
||
|
高 |
低 |
||||
|
年龄 |
2.572 |
0.109 |
|||
|
≤35岁 |
7 |
9 |
|||
|
>35岁 |
18 |
67 |
|||
|
FIGO分期 |
2.534 |
0.111 |
|||
|
ⅠB |
17 |
63 |
|||
|
ⅡA |
8 |
13 |
|||
|
淋巴结转移 |
1.955 |
0.162 |
|||
|
阴性 |
25 |
67 |
|||
|
阳性 |
0 |
9 |
|||
|
细胞分化程度 |
1.558 |
0.459 |
|||
|
高分化 |
4 |
13 |
|||
|
中分化 |
15 |
53 |
|||
|
低分化 |
6 |
10 |
|||
|
间质浸润深度 |
0.167 |
0.683 |
|||
|
<2/3 |
16 |
52 |
|||
|
≥2/3 |
9 |
24 |
|||
|
阴道壁累及 |
0.572 |
0.449 |
|||
|
是 |
0 |
1 |
|||
|
否 |
25 |
75 |
|||
|
脉管间隙阳性 |
0.000 |
1.000 |
|||
|
是 |
4 |
13 |
|||
|
否 |
21 |
63 |
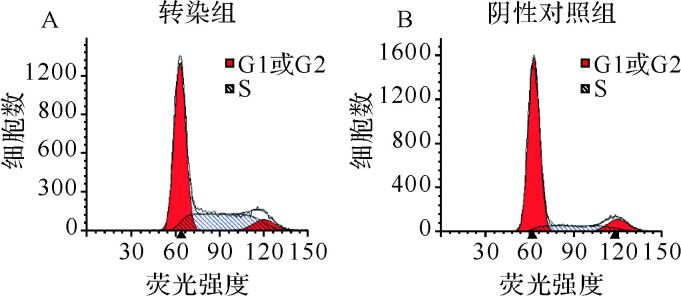
转染组S期细胞显著多于阴性对照组(29.76%±6.60%与13.38%±1.30%, P<0.05),见 图 1。提示miR-let-7e-3p能够诱导细胞周期发生S期阻滞。
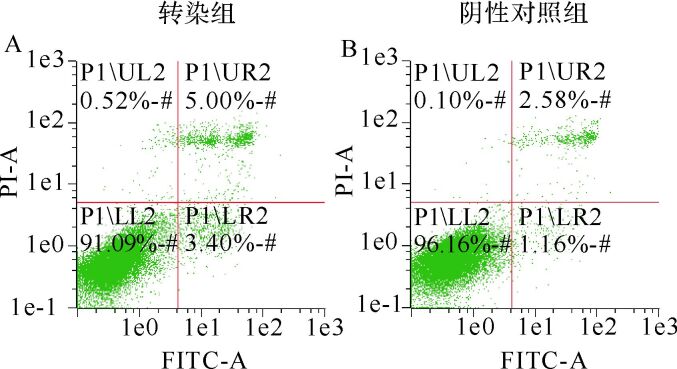
转染组凋亡率高于阴性对照组(5.98%±1.38%与3.53%±0.79%, P<0.05),见 图 2。提示miR-let-7e-3p能够促进肿瘤细胞发生凋亡。
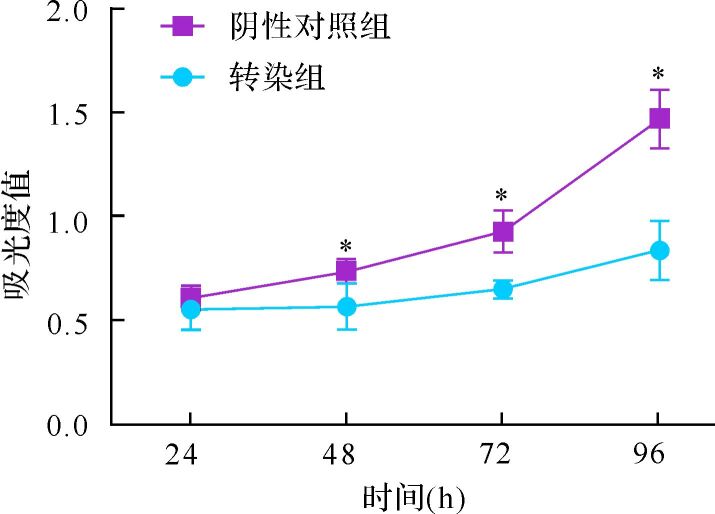
转染组转染后24、48、72、96 h吸光度值分别为0.55±0.09、0.57±0.11、0.65±0.04、0.84±0.14,阴性对照组分别为0.61±0.05、0.74±0.05、0.93±0.10、1.47±0.14。两组除24 h的吸光度值,48、72、96 h时吸光度值差异均具有统计学意义(均 P<0.05),见 图 3。提示miR-let-7e-3p能够抑制肿瘤细胞的增殖,且增殖抑制效应在转染48 h后就开始出现。
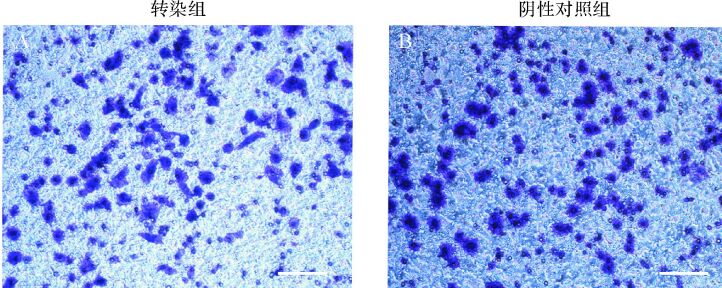
Transwell迁移实验显示,转染组与阴性对照组穿透小室的细胞数量分别为172±39与177±46,见 图 4,差异无统计学意义(P>0.05);Transwell 侵袭实验显示,转染组与阴性对照组细胞穿透小室基质胶的数量分别为218±37与254±47,见 图 5,差异亦无统计学意义(P>0.05)。推测miR-let-7e-3p发挥特定生物学功能需要依赖于组织和/或细胞的特异性。
miRNA是目前生物学界广泛开展的重要研究内容,其表达异常与肿瘤的发生、发展关系密切 [ 12- 13] 。本研究结果提示,在不同宫颈病变组织间,miRNA表达谱存在明显差异,提示miRNA与宫颈癌的发生、发展有关 [ 3- 5] 。 let-7家族是最早发现也是研究最广泛的miRNA家族之一,由Reinhart等 [ 14] 在秀丽隐杆线虫(C. elegans)中发现。let-7在许多肿瘤中表达降低,因而被视为抑癌miRNA [ 7- 10] 。本研究首先比较了miR-let-7e-3p在子宫颈正常组织、HSIL及宫颈癌组织中的表达差异,发现与宫颈正常组织比较,miR-let-7e-3p在HSIL和宫颈癌中表达明显下降,提示miR-let-7可能同样在宫颈癌的发生、发展过程中发挥重要作用。进一步将miR-let-7e-3p表达水平与宫颈癌临床不良预后因素进行相关性分析,发现miR-let-7e-3p与各不良预后因素均无显著相关性,推测miR-let-7e-3p表达下降可能是宫颈癌变发生过程中的早期事件,与宫颈癌进展没有显著相关性。
为了明确在临床标本中观察现象的相关机制,本研究利用宫颈癌细胞株Siha细胞对miR-let-7e-3p进行功能学研究。结果发现,过表达miR-let-7e-3p能够抑制肿瘤细胞的增殖,且增殖抑制效应在转染48 h后就开始出现,证实细胞的生长主要受细胞周期和细胞凋亡两方面的影响;进一步对转染细胞分别进行细胞周期和凋亡检测,结果提示,miR-let-7e-3p能够诱导细胞周期发生S期阻滞,并促进肿瘤细胞发生凋亡。我们在Siha细胞株中的研究结果与其他细胞株一致 [ 15] ,提示上调let-7表达量可以通过改变细胞周期而减慢细胞增殖。
既往研究表明,miR-let-7能够抑制口腔鳞状细胞癌 [ 16] 、前列腺癌 [ 17] 、肺癌 [ 18] 和结直肠癌 [ 19] 等多种肿瘤细胞的迁移和侵袭能力,然而本研究迁移和侵袭实验结果提示,miR-let-7e-3p对宫颈癌细胞的迁移和侵袭能力没有显著影响,符合miR-let-7e-3p与宫颈癌临床不良预后因素相关性分析的结果,推测miR-let-7发挥特定生物学功能需要依赖于组织和/或细胞的特异性。上述功能学实验结果提示,miR-let-7e-3p通过诱导细胞周期阻滞和促进细胞凋亡抑制宫颈癌细胞的增殖能力,从而发挥抑癌作用。
本研究存在如下不足和缺陷:内源性抑癌miRNA相对于小干扰RNA更符合机体内环境 [ 20] ,本研究应用的体外模拟表达不如体内更有说服力。因此,内源性稳定载体的建立以及模型小鼠的建立将是下一步研究的方向。
综上所述,本研究发现,miR-let-7e-3p在宫颈癌中可能发挥抑癌基因作用,并参与宫颈癌的发生过程;miR-let-7e-3p能否成为宫颈癌早期诊断和治疗的新靶点有待进一步体内研究。
Funding Statement
国家自然科学基金(81472421,81272861);浙江省自然科学基金(LQ15H160003);国家重点研发计划“重大慢性非传染性疾病防控研究”重点专项(2016YFC1302900,2016YFC0902900)
References
- 1.TORRE L A, BRAY F, SIEGEL R L, et al. Global cancer statistics, 2012. CA Cancer J Clin. 2015;65(2):87–108. doi: 10.3322/caac.21262. [TORRE L A, BRAY F, SIEGEL R L, et al. Global cancer statistics, 2012[J]. CA Cancer J Clin,2015,65(2):87-108.] [DOI] [PubMed] [Google Scholar]
- 2.ARBYN M, CASTELLSAGUE X, DE S S, et al. Worldwide burden of cervical cancer in 2008. Ann Oncol. 2011;22(12):2675–2686. doi: 10.1093/annonc/mdr015. [ARBYN M, CASTELLSAGUE X, DE S S, et al. Worldwide burden of cervical cancer in 2008[J]. Ann Oncol,2011,22(12):2675-2686.] [DOI] [PubMed] [Google Scholar]
- 3.LI Y, WANG F F, XU J F, et al. Progressive miRNA expression profiles in cervical carcinogenesis and identification of HPV-related target genes for miR-29. J Pathol. 2011;224(4):484–495. doi: 10.1002/path.2873. [LI Y,WANG F F, XU J F, et al. Progressive miRNA expression profiles in cervical carcinogenesis and identification of HPV-related target genes for miR-29[J]. J Pathol,2011, 224(4):484-495.] [DOI] [PubMed] [Google Scholar]
- 4.XU J F, LI Y, WANG F F, et al. Suppressed miR-424 expression via upregulation of target gene Chk1 contributes to the progression of invasive cervical cancer. Oncogene. 2013;32(8):976–987. doi: 10.1038/onc.2012.121. [XU J F, LI Y, WANG F F, et al. Suppressed miR-424 expression via upregulation of target gene Chk1 contributes to the progression of invasive cervical cancer[J]. Oncogene,2013,32(8):976-987.] [DOI] [PubMed] [Google Scholar]
- 5.VILLEGAS R V, JUAREZ M S, PEREZ G O, et al. Heterogeneity of microRNAs expression in invasive cervical cancer cells: over-expression of miR-196a. https://www.ncbi.nlm.nih.gov/pubmed/24817935. Int J Clin Exp Pathol. 2014;7(4):1389–1401. [VILLEGAS R V, JUAREZ M S, PEREZ G O, et al. Heterogeneity of microRNAs expression in invasive cervical cancer cells: over-expression of miR-196a[J]. Int J Clin Exp Pathol,2014,7(4):1389-1401.] [PMC free article] [PubMed] [Google Scholar]
- 6.ROUSH S, SLACK F J. The let-7 family of microRNAs. Trends Cell Biol. 2008;18(10):505–516. doi: 10.1016/j.tcb.2008.07.007. [ROUSH S, SLACK F J. The let-7 family of microRNAs[J]. Trends Cell Biol, 2008,18(10):505-516.] [DOI] [PubMed] [Google Scholar]
- 7.BOYERINAS B, PARK S M, HAU A, et al. The role of let-7 in cell differentiation and cancer. Endocr Relat Cancer. 2010;17(1):F19–36. doi: 10.1677/ERC-09-0184. [BOYERINAS B, PARK S M, HAU A, et al. The role of let-7 in cell differentiation and cancer[J]. Endocr Relat Cancer,2010,17(1):F19-36.] [DOI] [PubMed] [Google Scholar]
- 8.CHIU S C, CHUNG H Y, CHO D Y, et al. Therapeutic potential of microRNA let-7: tumor suppression or impeding normal stemness. http://cn.bing.com/academic/profile?id=2000674381&encoded=0&v=paper_preview&mkt=zh-cn. Cell Transplant. 2014;23(4-5):459–469. doi: 10.3727/096368914X678418. [CHIU S C, CHUNG H Y, CHO D Y, et al. Therapeutic potential of microRNA let-7: tumor suppression or impeding normal stemness[J]. Cell Transplant,2014,23(4-5):459-469.] [DOI] [PubMed] [Google Scholar]
- 9.TAKAMIZAWA J, KONISHI H, YANAGISAWA K, et al. Reduced expression of the let-7 microRNAs in human lung cancers in association with shortened postoperative survival. Cancer Res. 2004;64(11):3753–3756. doi: 10.1158/0008-5472.CAN-04-0637. [TAKAMIZAWA J, KONISHI H, YANAGISAWA K, et al. Reduced expression of the let-7 microRNAs in human lung cancers in association with shortened postoperative survival[J]. Cancer Res, 2004,64(11):3753-3756.] [DOI] [PubMed] [Google Scholar]
- 10.YANAIHARA N, CAPLEN N, BOWMAN E, et al. Unique microRNA molecular profiles in lung cancer diagnosis and prognosis. Cancer Cell. 2006;9(3):189–198. doi: 10.1016/j.ccr.2006.01.025. [YANAIHARA N, CAPLEN N, BOWMAN E, et al. Unique microRNA molecular profiles in lung cancer diagnosis and prognosis[J]. Cancer Cell, 2006,9(3):189-198.] [DOI] [PubMed] [Google Scholar]
- 11.SCHMITTGEN T D, LIVAK K J. Analyzing real-time PCR data by the comparative C T method . Nat Protoc. 2008;3(6):1101–1108. doi: 10.1038/nprot.2008.73. [SCHMITTGEN T D, LIVAK K J. Analyzing real-time PCR data by the comparative C T method[J]. Nat Protoc, 2008, 3(6):1101-1108. ] [DOI] [PubMed] [Google Scholar]
- 12.KONG Y W, FERLAND-MCCOLLOUGH D, JACKSON T J, et al. microRNAs in cancer management. Lancet Oncol. 2012;13(6):e249–2589. doi: 10.1016/S1470-2045(12)70073-6. [KONG Y W, FERLAND-MCCOLLOUGH D, JACKSON T J, et al. microRNAs in cancer management[J]. Lancet Oncol, 2012, 13(6):e249-258.] [DOI] [PubMed] [Google Scholar]
- 13.MO Y Y. MicroRNA regulatory networks and human disease. Cell Mol Life Sci. 2012;69(21):3529–3531. doi: 10.1007/s00018-012-1123-1. [MO Y Y. MicroRNA regulatory networks and human disease[J]. Cell Mol Life Sci, 2012, 69(21):3529-3531.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.REINHART B J, SLACK F J, BASSON M, et al. The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans. Nature. 2000;403(6772):901–906. doi: 10.1038/35002607. [REINHART B J, SLACK F J, BASSON M, et al. The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans[J]. Nature,2000,403(6772):901-906.] [DOI] [PubMed] [Google Scholar]
- 15.JOHNSON C, ESQUELA K A, STEFANI G, et al. The let-7 microRNA represses cell proliferation pathways in human cells. Cancer Res. 2007;67(16):7713–7722. doi: 10.1158/0008-5472.CAN-07-1083. [JOHNSON C, ESQUELA K A, STEFANI G, et al. The let-7 microRNA represses cell proliferation pathways in human cells[J]. Cancer Res,2007,67(16):7713-7722.] [DOI] [PubMed] [Google Scholar]
- 16.CHANG C J, HSU C C, CHANG C H, et al. Let-7d functions as novel regulator of epithelial-mesenchymal transition and chemoresistant property in oral cancer. http://cn.bing.com/academic/profile?id=2130433715&encoded=0&v=paper_preview&mkt=zh-cn. Oncol Rep. 2011;26(4):1003–1010. doi: 10.3892/or.2011.1360. [CHANG C J, HSU C C, CHANG C H, et al. Let-7d functions as novel regulator of epithelial-mesenchymal transition and chemoresistant property in oral cancer[J]. Oncol Rep,2011,26(4):1003-1010.] [DOI] [PubMed] [Google Scholar]
- 17.WANGNER S, NGEZAHAYO A, MURUA E H, et al. Role of miRNA let-7 and its major targets in prostate cancer. http://cn.bing.com/academic/profile?id=2034600438&encoded=0&v=paper_preview&mkt=zh-cn. Biomed Res Int. 2014;2014:376326. doi: 10.1155/2014/376326. [WANGNER S, NGEZAHAYO A, MURUA E H, et al. Role of miRNA let-7 and its major targets in prostate cancer[J]. Biomed Res Int,2014,2014: 376326.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.ZHAO B, HAN H, CHEN J, et al. MicroRNA let-7c inhibits migration and invasion of human non-small cell lung cancer by targeting ITGB3 and MAP4K3. Cancer Lett. 2014;342(1):43–51. doi: 10.1016/j.canlet.2013.08.030. [ZHAO B, HAN H, CHEN J, et al. MicroRNA let-7c inhibits migration and invasion of human non-small cell lung cancer by targeting ITGB3 and MAP4K3[J]. Cancer Lett,2014,342(1):43-51.] [DOI] [PubMed] [Google Scholar]
- 19.HUR K, TOIYAMA Y, SCHETTER A J, et al. Identification of a metastasis-specific MicroRNA signature in human colorectal cancer. J Nat Cancer Inst. 2015;107(3):dju492. doi: 10.1093/jnci/dju492. [HUR K, TOIYAMA Y, SCHETTER A J, et al. Identification of a metastasis-specific MicroRNA signature in human colorectal cancer[J]. J Nat Cancer Inst,2015,107(3):dju492.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.ESQUELA K A, SLACK F J. Oncomirs-microRNAs with a role in cancer. Nat Rev Cancer. 2006;6(4):259–269. doi: 10.1038/nrc1840. [ESQUELA K A, SLACK F J. Oncomirs-microRNAs with a role in cancer[J]. Nat Rev Cancer,2006,6(4):259-269.] [DOI] [PubMed] [Google Scholar]