Abstract
目的
研究成牙本质方向分化下牙髓干细胞(dental pulp stem cells,DPSCs)来源外泌体的特征,比较其与普通培养下DPSCs来源外泌体的微小RNA(microRNA)表达差异,并分析其相关信号传导通路。
方法
(1) 分别采用α-基础伊格尔(氏)培养基(α minimum Eagle’ s medium,α-MEM,Hyclone公司,美国) 和成牙本质方向分化培养基培养DPSCs 21 d,使用茜素红矿化结节染色和碱性磷酸酶染色对两种细胞进行鉴定。分别在两种细胞上清液中提取外泌体,命名为普通培养条件下DPSCs外泌体(dental pulp stem cells-exosomes,DPSCs-Exo)和成牙本质方向分化培养条件下DPSCs外泌体(dental pulp stem cells-odontogenic-exosomes,DPSCs-OD-Exo)。采用透射电镜观察法、纳米粒子示踪分析法和蛋白印迹法观察比较两种外泌体的形态、粒径分布和外泌体标记蛋白表达情况。(2)采用microRNA芯片法分析DPSCs-Exo和DPSCs-OD-Exo的microRNA表达谱差异,选择差异表达最显著的3种micro-RNA进行实时荧光定量聚合酶链式反应(real-time quantitative polymerase chain reaction,real-time PCR)验证。对差异表达的microRNA采用microRNA目标预测数据库及基因信号通路数据库进行分析,预测microRNA在DPSCs成牙本质方向分化中的信号传导途径。
结果
(1) 普通培养条件下的DPSCs呈梭形、多角形等典型的成纤维细胞样形态,成牙本质方向诱导分化21 d后的DPSCs也基本呈梭形、多角形。成牙本质方向诱导分化条件下的细胞茜素红矿化结节染色结果镜下可见大量色暗沉积物形成,碱性磷酸酶染色结果显示细胞颜色深染呈深蓝色,而普通培养条件下的细胞则未见明显染色。两种培养条件下的DPSCs-Exo和DPSCs-OD-Exo形态一致,均呈茶托样,具有双层膜结构。DPSCs-Exo粒径峰值为(114.67±9.07) nm,DPSCs-OD-Exo粒径峰值为(134.00±8.54) nm。DPSCs-OD -Exo的粒径峰值稍大于DPSCs-Exo,差异有统计学意义(t=58.00,P < 0.05)。DPSCs-Exo和DPSCs-OD-Exo均表达外泌体标志蛋白肿瘤易感基因(tumor susceptibility gene,TSG)101蛋白、CD63,符合外泌体特征。(2)microRNA芯片结果显示DPSCs-Exo与DPSCs-OD-Exo的microRNA表达谱存在差异,其中19个增加2倍以上,1个减少64%。real-time PCR验证结果显示,microRNA表达谱中差异表达的microRNA-1246、microRNA-100-5p和microRNA-494-3p在DPSCs-Exo与DPSCs-OD-Exo中存在差异,且差异有统计学意义(P < 0.05)。通过microRNA目标预测数据库及基因信号通路数据库对差异表达的microRNA进行分析,预测差异表达的microRNA可靶向轴抑制蛋白2(axis inhibition protein 2,AXIN2)基因及Wnt/β-catenin信号传导通路。
结论
DPSCs-OD-Exo与DPSCs-Exo均符合外泌体特征,两者的microRNA表达谱存在差异,差异表达的microRNA可能参与调控DPSCs成牙本质方向分化。
Keywords: 牙髓干细胞, 外泌体, 微小RNA
Abstract
Objective
To investigate the characteristics of exosomes derived from dental pulp stem cells (DPSCs) in the direction of odontogenic differentiation, to analyze the differences in microRNA expression profile between exosomes derived from undifferentiated and odontogenic DPSCs, and to analyze their possible signal transduction pathways.
Methods
(1) DPSCs were cultured in α minimum Eagle' s medium (α-MEM), and odontogenic DPSCs were cultured in odontogenic differentiation medium for 21 days, using alizarin red staining and alkaline phosphatase staining to identify the odontogenic differentiation. Exosomes from the cell supernatant were isolated respectively, named as dental pulp stem cells-exosomes (DPSCs-Exo) and dental pulp stem cells-odontogenic-exosomes (DPSCs-OD-Exo). The exosomes were identified by transmission electron microscopy, nanoparticle tracking analysis and Western blot. (2) The microRNA expression profiles of DPSCs-Exo and DPSCs-OD-Exo were investigated by microRNA microarray. To validate the result of the microRNA microarray, real-time quantitative polymerase chain reaction (real-time PCR) assay was applied on 3 most significantly differential expressed microRNA. Pathway analysis was taken to detect enriched pathways associated with the predicted target genes of microRNA.
Results
(1) The DPSCs were isolated and cultured in vitro showed typical fibroblast-like morphology. The odontogenic differentiated DPSCs were spindle-shaped, polygonal, and uniform in size. Odontogenic differentiation group showed a large number of dark deposits in alizarin red staining and the cells were darkly stained in alkaline phosphatase staining, while the cells in normal culture medium group did not show obvious dyeing. The DPSCs-Exo and DPSCs-OD-Exo had the same morphology, both showed bilayer membrane and cup-shape. The peak sizes of DPSCs-Exo and DPSCs-OD-Exo were (114.67±9.07) nm and (134.00±8.54) nm, respectively. The difference between the two was statistically significant. DPSCs-Exo and DPSCs-OD-Exo both expressed the markers of exosomes, tumor susceptibility gene (TSG)101 and CD63. (2) microRNA microarray results showed that the expression profiles of DPSCs-Exo and DPSCs-OD-Exo were different. Nineteen increased by more than two times, and one decreased by 64%. Real-time PCR results showed that the expression levels of microRNA-1246, microRNA-1246-100-5p and microRNA-1246-494-3p in DPSCs-OD-Exo were significantly up-regulated. The difference was statistically significant. microRNA target prediction database and gene signaling pathway database were used to analyze differentially expressed microRNA, and it was predicted that differentially expressed microRNA could target axis inhibition protein 2(AXIN2) gene and Wnt/β-catenin signaling pathway.
Conclusion
DPSCs-OD-Exo and DPSCs-Exo had differences in their microRNA expression profile. Those differentially expressed microRNA may be involved in the regulation of DPSCs odontogenic differentiation.
Keywords: Dental pulp stem cells, Exosomes, microRNA
牙髓干细胞(dental pulp stem cells,DPSCs)的成牙本质方向分化是实现牙髓牙本质再生的关键。成牙本质方向分化条件下培养的DPSCs与普通培养的DPSCs存在微小RNA(microRNA)表达谱差异,提示干细胞内差异表达的microRNA可能是调节DPSCs成牙本质方向分化的重要因素[1]。细胞内的microRNA可能随外泌体的分泌而包裹于外泌体中。外泌体是直径在30~150 nm的囊泡,可由多种类型的细胞分泌,用于介导细胞间的通讯[2]。在脂质膜的包裹下microRNA能稳定存在于外泌体中免于免疫系统的分解,并最终随外泌体以膜融合或直接胞吞的方式进入靶细胞。microRNA以序列互补的方式直接结合RNA,以抑制翻译并促进其靶转录物的降解[3]。Cui等[4]2016年的研究显示, 矿化成骨细胞的外泌体可通过转运microRNA改变靶细胞microRNA表达,进而促进骨髓基质细胞的成骨分化,提示外泌体可通过转运microRNA调控靶细胞的分化方向。Gong等[1]在2012年对DPSCs本身的microRNA表达谱进行了分析,发现成牙本质方向分化培养的DPSCs与普通培养DPSCs的microRNA表达谱存在差异,发现22个microRNA存在差异,这些差异表达microRNA是否存在于外泌体中,目前国内尚未见到报道。
本研究拟分离提取普通培养条件下DPSCs外泌体(dental pulp stem cells-exosomes,DPSCs-Exo)和成牙本质方向分化培养条件下DPSCs外泌体(den-tal pulp stem cells-odontogenic-exosomes,DPSCs-OD-Exo),对两种外泌体特征进行比较,分析microRNA表达谱,进一步对差异表达的microRNA进行靶基因预测,分析分化过程中可能涉及的下游信号传导途径。
1. 资料与方法
1.1. 普通培养和成牙本质方向分化下培养的人DPSCs外泌体的提取与鉴定
1.1.1. 分离培养DPSCs
本研究获得北京大学口腔医院医学伦理委员会批准(批准号:PKUSSIRB-201412020)。收集北京大学口腔医院颌面外科门诊因阻生或正畸需要而拔除的前磨牙或第三磨牙,患者18~25岁,牙齿完整、无龋损、无牙髓或根尖周炎症、无牙周疾病,供者身体健康,自愿签署知情同意书。本研究选用的DPSCs来自18岁男性左下第一前磨牙、23岁男性右下第一前磨牙和21岁女性右下第三恒磨牙。于超净台中对离体牙自根尖至冠部进行反复轻柔冲洗,使用咬骨钳钳碎患牙并取出牙髓组织。将上述组织分别切碎至1 mm3,用3 g/L的Ⅰ型胶原酶(Worthington公司,美国)和4 g/L分离酶(Sigma公司,美国),37 ℃消化30~40 min。将消化后的细胞悬液过70 μm细胞筛,以获得单细胞悬液。将单细胞悬液加入6孔板(Corning公司,美国)中,加入含有15%(体积分数)胎牛血清(fetal bovine serum,FBS)、1%(体积分数)L-谷氨酰胺及1%(体积分数)青-链霉素(Gibco公司,美国)的α-基础伊格尔(氏)培养基(α minimum Eagle ’ s medium,α-MEM,Hyclone公司,美国),置于5%(体积分数)CO2、37 ℃培养。待细胞长满至75%时,将原代细胞按照1 ∶ 3传代,至第3~4代细胞备用。
1.1.2. DPSCs成牙本质方向分化诱导和鉴定
取第3代DPSCs以5×103个细胞/孔接种于24孔板,采用普通细胞培养液培养,待细胞培养至80%汇合时,将细胞分为两组:(1)成牙本质方向分化组:细胞培养液为矿化诱导培养基, 含有15%FBS、10 mmol/L β-甘油磷酸酯、0.2 mmol/L维生素C、100 nmol/L地塞米松和α-MEM;(2)普通培养组:细胞培养液仍为普通培养基。采用5-溴-4-氯-3-吲哚基-磷酸盐(5-bromo-4-chlro-3-indolyl phosphate,BCIP)/四唑硝基蓝(tetranitroblue tetrazolium chloride,NBT)碱性磷酸酶显色试剂盒和茜素红显色试剂盒进行成牙本质方向分化鉴定,将共培养7 d后的细胞培养液吸干净,PBS冲洗3次,95%的乙醇水溶液固定10 min,再用磷酸盐缓冲液(phosphate buffered saline,PBS)洗3次。按照碱性磷酸酶染色试剂盒说明,将盒内BCIP、NBT和显色液按照1 ∶ 1 ∶ 38的体积比进行混合,加入固定后的细胞中,避光放置5~30 min,至显色明显。将茜素红染色试剂加入固定细胞中,避光放置5 min,至显色明显。PBS冲洗3次终止显色反应,进行扫描和镜下拍照。
1.1.3. 提取DPSCs-Exo和DPSCs-OD-Exo
将胎牛血清在4 ℃条件下100 000 ×g超速离心12 h,以去除胎牛血清中的外泌体。取第3代DPSCs细胞以1×107个细胞/孔接种于175 cm2培养瓶中,采用含有15% FBS的普通培养基培养至80%汇合时,弃去上清液,更换为不含外泌体的FBS细胞培养基继续培养2 d,收集上清液。将收集好的上清液转移至50 mL离心管,以下离心步骤均在4 ℃条件下完成:以2 000×g的转速离心20 min,除去上清液中的死细胞;将上清液转移至50 mL洁净离心管,以10 000×g的转速离心30 min,去除上清液中的细胞碎片后收集上清液;加入超速离心机(Optima XPN-10,Backman公司,美国)配套的离心管,100 000×g超速离心70 min,获得外泌体沉淀。离心结束后,弃去上清液,加入2~3 mL PBS重悬沉淀,将相同组的沉淀收集到一个同离心管内,加满PBS, 100 000×g转速离心60 min,获得外泌体沉淀,去除杂质蛋白。离心结束后小心取出离心管,充分弃去上清液,加入50~100 μL PBS重悬,获得外泌体悬液,置于-80 ℃保存。DPSCs-OD-Exo的提取方法类似于DPSCs-Exo, 取第3代DPSCs细胞以1×107个细胞/孔接种于175 cm2培养瓶中,生长至80%汇合后,采用含有15%FBS的成牙本质方向分化培养液进行培养至19 d后,弃上清液,更换为不含外泌体的成牙本质方向分化诱导细胞培养液继续培养2 d后,以除外血清中外泌体的影响, 收集上清液,多步离心法提取外泌体,步骤同DPSCs-Exo组。
1.1.4. 外泌体的鉴定
(1) 透射电镜观察法:将10 μL提取物悬液用50 μL 2%(体积分数)多聚甲醛室温固定30 min,取上述混合液8 μL,滴加至碳涂层铜网格上,风干10 min,再用1%(体积分数)醋酸双氧铀染色两次,每次6 min。将铜网装入透射电镜(JEM-1400,JEOL公司,日本),电压设置为120 kV,观察提取物形态。(2)纳米粒子跟踪分析法(nano-particle tracking analysis,NTA):取10 μL提取物悬液,以1 ∶ 100和过滤后的PBS混合,用NTA确定粒径分布。使用NTA分析仪(NS3000,Malvern公司,英国)记录布朗运动下外泌体的运动轨迹,并通过NTA分析软件进行分析。(3)Western blot检测外泌体标志蛋白:取50 μL提取物悬液,加入20 μL的1%(质量分数))Triton裂解液(Cell Signaling Technology公司,美国),冰上裂解30 min,超声破碎1 min。4 ℃ 12 000 r/min离心30 min提取蛋白,用蛋白浓度测定试剂盒(bicinchoninic acid kit,BCA,Thermo Fisher公司,美国)检测蛋白浓度,记录结果。蛋白在100 ℃中水浴5 min,然后配胶[12% (质量分数)十二烷基硫酸钠-聚丙烯酰胺凝胶(sodium dodecyl sulfate polyacrylamide gel electrophoresis, SDS-PAGE),碧云天公司,中国],取15 μL蛋白上样,蛋白分离2 h(电泳电压为125 V),切胶。1.5 h后将蛋白转移到二氟乙烯(polyvinylidene fluo-ride, PVDF)膜(Millipore Corporation公司,美国)上,转膜电压为100 V,脱脂牛奶封闭1 h,加入一抗CD63(1 ∶ 500,ab68418,Abcam公司,美国)/肿瘤易感基因(tumor susceptibility gene,TSG)101蛋白(1 ∶ 500,14497-1-AP,Proteintech公司,美国)/ 甘油醛-3-磷酸脱氢酶(glyceraldehyde-3-phosphate dehydrogenase,GAPDH,1 ∶ 500,10494-1-AP,Proteintech公司,美国)4 ℃孵育过夜。用三羟甲基氨基甲烷盐酸+聚氧乙烯山梨醇的脂肪酸脂(trometamol hydrochloride+Tween,TBST,华兴博创公司,中国)洗膜3次,每次10 min,用抗兔IgG抗体(1 ∶ 1 000,10285-1-AP,Proteintech公司,美国)孵育1 h,TBST洗膜3次,每次10 min, 应用发光检测试剂盒(Proteintech公司,美国)在凝胶成像系统(Vilber Lourmat公司,美国)中曝光。
1.2. 芯片法分析DPSCs-Exo和DPSCs-OD-Exo的microRNA表达谱差异,查找相关信号通路
1.2.1. DPSCs-Exo和DPSCs-OD-Exo的microRNA芯片分析
采用Agilent人类microRNA微阵列分析DPSCs-Exo和DPSCs-OD-Exo的microRNA表达差异,DPSCs-Exo和DPSCs-OD-Exo送至上海康成生物公司做microRNA芯片实验,芯片信息:Agilent人类microRNA微阵列,靶向2 549个人类的microRNA,数据来源miRBase数据库(v21.0)。
1.2.2. 差异表达的microRNA用实时荧光定量聚合酶链式反应进行验证
选择上述DPSCs-Exo和DPSCs-OD-Exo的microRNA表达谱中差异表达有统计学意义的3种microRNA, 进行实时荧光定量聚合酶链式反应(real-time quantitative polymerase chain reaction,real-time PCR)验证。外泌体RNA的抽提:取出外泌体样品,12 000×g离心10 min,取250 μL样品,转至1.5 mL的离心管中,加入750 μL的TRIzol LS试剂(Thermo Fisher公司,美国)混匀。15~ 30 ℃孵育5 min,加入0.2 mL的氯仿,15~30 ℃孵育2~3 min。4 ℃ 12 000×g离心15 min。将水相转移到新离心管中,并加入500 μL异丙醇,混匀后15~30 ℃孵育10 min后,于4 ℃ 12 000×g离心10 min。移去上清液,加入75%乙醇,清洗RNA沉淀。振荡后,4 ℃ 7 500×g离心5 min。干燥RNA沉淀5~10 min,加入无RNA酶的水反复吹打,55~60 ℃孵育10 min。RNA逆转录:用总RNA加入micro-RNA逆转录试剂、microRNA引物等,无RNA酶的水补足体积至20 μL。反应条件为16 ℃ 30 min,42 ℃ 40 min,85 ℃ 5 min,-20 ℃保存反应产物。micro-RNA逆转录引物见表 1。外泌体microRNA的RT-PCR检测:将cDNA样品中加入2× Master Mix 5 μL(Thermo Fisher公司,美国)、10 μmol的PCR双向引物F和R各0.5 μL,无RNA酶的水补足体积至8 μL,microRNA双向引物见表 2。将10 μL混合液加到PCR板对应的每个孔中,短暂离心混合。反应条件为:95 ℃预变性10 min,95 ℃变性15 s,60 ℃退火1 min,40个循环。溶解曲线分析,95 ℃,15 s,60 ℃,1 min,95 ℃,15 s,60 ℃,15 s。各样品的目的microRNA和内参照(microRNA-191-5p)分别进行real-time PCR反应,数据采用2-ΔΔCT法进行分析。差异表达microRNA相关靶基因及其对应信号通路:采用三种类型的microRNA目标预测数据库,包括miRDB(http://mirdb.org/miRDB/download.html)、miRWalk(http://mirwalk.umm.uni-heidelberg.de/)、miRTarBase(http://mirtarbase.mbc.nctu.edu.tw/php/index.php)预测差异表达的microRNA的靶基因,靶基因确定为这3个数据集的交集。利用GO数据库(http://www.geneontology.org)和KEGG数据库(http://www.genome.jp/kegg/pathway.html)确定潜在的生物学功能和信号通路。
表 1.
microRNA逆转录引物
microRNA reverse transcription primers
| Genes | Reverse transcription primers |
| microRNA-100-5p | 5′GTCGTATCCAGTGCGTGTCGTGGAGTCGGCAATTGCACTGGATACGACCACAAG3′ |
| microRNA-1246 | 5′GTCGTATCCAGTGCGTGTCGTGGAGTCGGCAATTGCACTGGATACGACCCTGCT3′ |
| microRNA-494-3p | 5′GTCGTATCCAGTGCGTGTCGTGGAGTCGGCAATTGCACTGGATACGACGAGGTT3′ |
| microRNA-191-5p | 5′GTCGTATCCAGTGCGTGTCGTGGAGTCGGCAATTGCACTGGATACGACCAGCTG3′ |
表 2.
microRNA双向引物
microRNA bidirectional primers
| Genes | Bidirectional primers | Annealing temperature/℃ | Base pair/bp |
| GSP, gene-specific primer; R, the primer matching GSP. | |||
| microRNA-100-5p | GSP: 5′GCAACCCGTAGATCCGAA3′ R: 5′CAGTGCGTGTCGTGGAGT3′ |
60 | 62 |
| microRNA-1246 | GSP: 5′GGGGAATGGATTTTTGG3′ R: 5′CAGTGCGTGTCGTGGAG3′ |
60 | 63 |
| microRNA-494-3p | GSP: 5′GGGGTGAAACATACACGGGA3′ R: 5′GTGCGTGTCGTGGAGTCG3′ |
60 | 64 |
| microRNA-191-5p | GSP: 5′GGCAACGGAATCCCAAAAG3′ R: 5′GTGCGTGTCGTGGAGTCG3′ |
60 | 63 |
1.3. 统计学分析
使用SPSS 26.0软件进行数据统计分析,每个实验至少重复三次,结果数据表示为均数±标准差,采用配对t检验,P < 0.05为差异具有统计学意义。
2. 结果
2.1. 普通培养和成牙本质方向分化诱导培养DPSCs结果
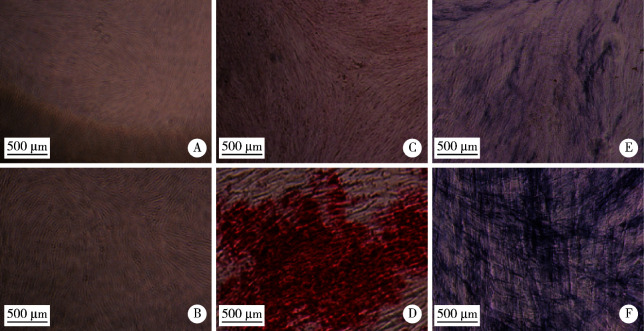
普通培养基条件下DPSCs细胞生长良好,呈梭形、多角形等典型的成纤维细胞样形态,细胞大小基本一致。成牙本质方向诱导分化下的DPSCs也基本呈梭形、多角形,大小一致(图 1A、B)。培养3周后茜素红矿化结节染色结果显示普通培养组未见有明显沉积物(图 1C),而成牙本质方向分化细胞聚集处可见大量色暗沉积物形成(图 1D)。培养3周后碱性磷酸酶染色结果显示成牙本质方向分化组细胞颜色较普通培养组细胞深染,呈深蓝色(图 1E、F)。
图 1.
普通培养和成牙本质方向分化DPSCs形态及分化鉴定结果
Morphology and differentiation identification results of undifferentiated and odontogenic DPSCs
A, B, bright view of undifferentiated and odontogenic DPSCs cultured for 3 weeks; C, D, alizarin red staining results of undifferentiated and odontoge-nic DPSCs; E, F, alkaline phosphatase staining results of undifferentiated and odontogenic DPSCs. DPSCs, dental pulp stem cells.
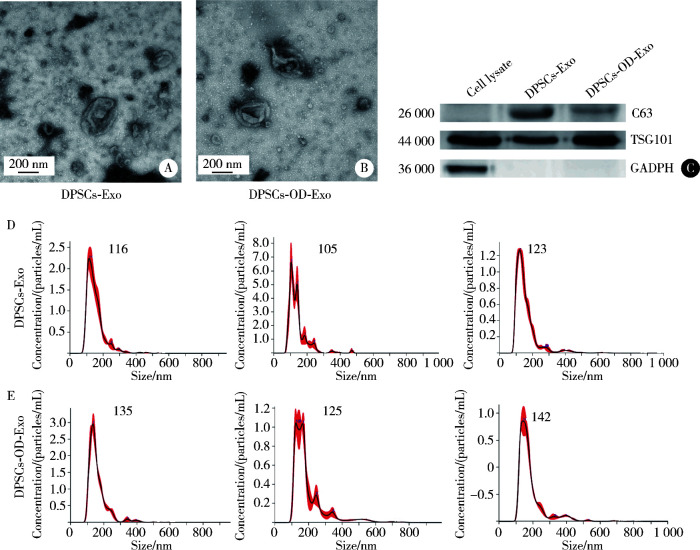
2.2. DPSCs-Exo和DPSCs-OD-Exo的鉴定结果
透射电镜结果显示DPSCs-Exo和DPSCs-OD-Exo外泌体均呈茶托样,具有双层膜结构图(图 2A、B)。Western blot检测DPSCs-Exo和DPSCs-OD-Exo外泌体表达外泌体特征标志蛋白TSG101和CD63,不表达细胞内参蛋白GAPDH(图 2C)。用NTA检测三个不同供者细胞来源的DPSCs-Exo和DPSCs-OD-Exo,结果可见DPSCs-Exo的粒径峰值平均峰值为(114.67±9.07) nm。DPSCs-OD-Exo的粒径峰值稍大,为(134.00±8. 54) nm。采用配对t检验统计方法分析,两者差异有统计学意义(t=58.00,P < 0.05),见图 2D、E。
图 2.
DPSCs-Exo和DPSCs-OD-Exo的鉴定
Identification of DPSCs-Exo and DPSCs-OD-Exo
A, B, DPSCs-Exo and DPSCs-OD-Exo exosomes morphology under transmission electron microscope; C, Western blot detects the expression of exosomal markers in DPSCs-Exo and DPSCs-OD-Exo; D, E, NTA results of DPSCs-Exo and DPSCs-OD-Exo. DPSCs-Exo, dental pulp stem cells-exosomes; DPSCs-OD-Exo, dental pulp stem cells-odontogenic-exosomes; TSG101, tumor susceptibility gene 101; NTA, nanoparti-cletracking analysis.
2.3. microRNA芯片法分析DPSCs-Exo和DPSCs-OD-Exo的microRNA表达谱差异
筛选DPSCs-OD-Exo与DPSCs-Exo表达差异显著的microRNA,其中19个增加2倍以上,1个减少64%(表 3)。
表 3.
DPSCs-OD-Exo和DPSCs-Exo中差异表达显著的microRNA
Differential expressed microRNA in DPSCs-OD-Exo and DPSCs-Exo
| microRNA | Increase/decrease | Fold | P |
| microRNA-100-5p | Increase | 2.16 | < 0.01 |
| microRNA-1246 | Increase | 84.65 | < 0.01 |
| microRNA-1587 | Increase | 2.03 | < 0.01 |
| microRNA-3136-5p | Increase | 2.27 | < 0.01 |
| microRNA-3912-5p | Increase | 2.69 | < 0.01 |
| microRNA-3923 | Increase | 2.38 | < 0.01 |
| microRNA-3945 | Increase | 2.31 | < 0.01 |
| microRNA-4259 | Increase | 2.18 | < 0.01 |
| microRNA-4707-5p | Increase | 2.15 | < 0.01 |
| microRNA-4754 | Increase | 2.00 | < 0.01 |
| microRNA-494-3p | Increase | 2.82 | < 0.01 |
| microRNA-5196-5p | Increase | 2.18 | < 0.01 |
| microRNA-5687 | Increase | 2.37 | < 0.01 |
| microRNA-588 | Increase | 2.25 | < 0.01 |
| microRNA-6133 | Increase | 2.35 | < 0.01 |
| microRNA-6779-5p | Increase | 2.00 | < 0.01 |
| microRNA-6813-5p | Increase | 2.10 | < 0.01 |
| microRNA-6823-5p | Increase | 2.30 | < 0.01 |
| microRNA-6832-5p | Increase | 2.21 | < 0.01 |
| microRNA-1-3p | Decrease | 2.19 | < 0.01 |
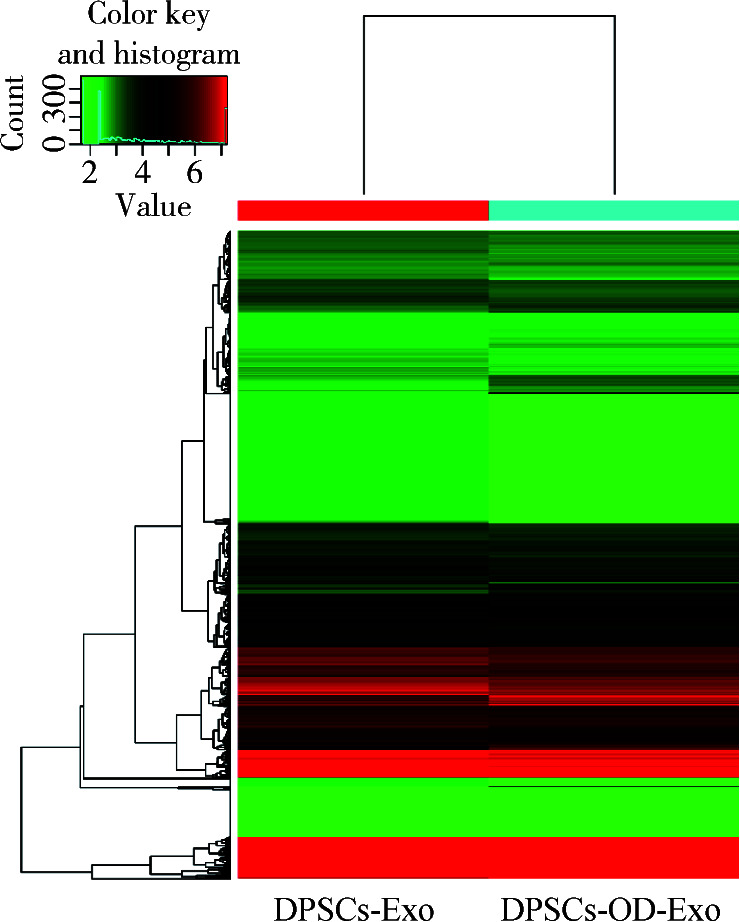
使用microRNA芯片法分析DPSCs-Exo和DPSsC-OD-Ex microRNA的表达谱,以差异表达的microRNA数据生成DPSCs-Exo和DPSCs-OD-Exo microRNA热图(图 3)。
图 3.
DPSCs-Exo和DPSCs-OD-Exo microRNA热图
DPSCs-Exo and DPSCs-OD-Exo microRNA heat map
microRNA levels in DPSCs-OD-Exo significantly changed when compared with that in DPSCs-Exo, the green to red corresponds to the gene expression level from low to high. DPSCs-Exo, dental pulp stem cells-exosomes; DPSCs-OD-Exo, dental pulp stem cells-odontogenic-exosomes.
2.4. 差异表达microRNA的real-time PCR验证结果
选择DPSCs-Exo和DPSCs-OD-Exo芯片结果中差异表达显著的3个microRNA进行real-time PCR验证,包括microRNA-1246、microRNA-100-5p和microRNA-494-3p。以microRNA-191-5p作为内参, DPSCs-OD-Exo的microRNA-1246表达显著升高,是DPSCs-Exo的(7.5±0.2)倍,采用配对t检验统计分析,差异有统计学意义(t =447.59、P=0.00 < 0.05);DPSCs-OD-Exo的microRNA-100-5p也可见升高,是DPSCs-Exo的(3.1±0.3)倍,采用配对t检验统计分析,差异有统计学意义(t=144.76、P=0.00 < 0.05);DPSCs-OD-Exo的microRNA-494-3p表达可见小幅升高,是DPSCs-Exo的(2.5±0.2)倍,采用配对t检验统计分析,差异有统计学意义(t=103.01、P=0.00 < 0.05)。上述结果与芯片结果趋势均一致。
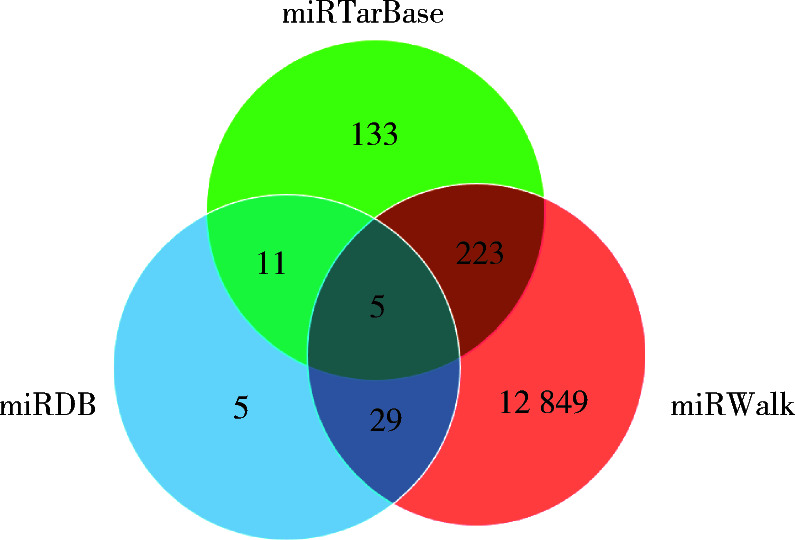
2.5. DPSCs-Exo和DPSCs-OD-Exo差异表达micro-RNA靶基因预测结果
根据上述结果,在miRTarBase、miRDB、miRWalk数据库中分析预测差异表达microRNA的靶基因。以microRNA-1246为例在miRTarBase、miRDB、miRWalk数据库中分析预测靶基因,制作韦恩(Venn)图,三者交集即为microRNA可能靶基因,结果显示3种数据库分析预测靶基因共13 255个,其中三者交集靶基因5个(图 4),分别为轴抑制蛋白2(axis inhibition protein 2,AXIN2)基因、包含G-patch域11蛋白的基因、双特异性酪氨酸(Y)磷酸化调控激酶1A基因、类维生素A相关的孤儿受体α基因以及抑制糖原合成酶激酶3β基因。再利用信号通路数据库对基因分析,结果显示6种差异表达显著的micro-RNA可靶向Wnt/β-catenin信号通路中的9个基因。
图 4.
以microRNA-1246为例利用数据库预测靶基因
microRNA-1246 was used as an example to predict target genes from the database
miRTarBase, miRDB, miRWalk databases were used to predict micro-RNA-1246 target genes to make a Venn diagram.
3. 讨论
本研究按刘敬一等[5]的方法在普通培养和成牙本质方向分化条件下培养DPSCs,分别提取其外泌体,命名为DPSCs-Exo和DPSCs-OD-Exo,结果显示透射电镜下DPSCs-Exo和DPSCs-OD-Exo提取物形态较一致,均呈茶托型和一侧凹陷的半球形,提示不同培养条件下相同来源干细胞提取的外泌体形态相近,这和Narayanan等[6]的结果相似。Nara-yanan等[6]对人间充质干细胞成骨和成脂肪诱导后提取细胞上清液外泌体,透射电镜下显示所有外泌体均为球形。Western blot显示DPSCs-Exo和DPSCs-OD-Exo均表达外泌体特征标志蛋白CD63和TSG101,不表达细胞内参蛋白GAPDH,均符合国际细胞外囊泡学会2014年指导性声明中的外泌体特征[7]。本研究中DPSCs-Exo和DPSCs-OD-Exo提取物粒径峰值均在80~150 nm,符合外泌体粒径大小范围,表明DPSCs在普通培养下和成牙本质方向分化培养下的细胞上清液提取物均符合外泌体特征,但同时也观察到DPSCs-OD-Exo的粒径峰值稍大。Hu等[8]2019年的研究中也提取了两者外泌体,未见两者的粒径比较,但从透射电镜的结果图中可观察到DPSCs-OD-Exo的直径比DPSCs-Exo略大,与本研究结果一致。Narayanan等[6]也发现不同分化谱系细胞来源的外泌体直径存在差异,这可能与外泌体内的内容物不同,如microRNA等不同相关。笔者观察到在成骨谱系细胞外泌体中存在影响成骨的microRNA,如microRNA-34a、microRNA-27a和microRNA-22,而影响脂肪生成的microRNA-143和microRNA-375则出现在成脂谱系细胞的外泌体中,提示外泌体粒径大小可能与外泌体内容物不同相关。
本研究结果发现DPSCs-Exo和DPSCs-OD-Exo的microRNA表达谱存在差异,对于外泌体micro-RNA参与细胞分化阶段的作用,目前研究较少。Wang等[9]对间充质干细胞来源的外泌体成骨分化的作用进行研究,在间充质干细胞成骨分化0 d、早期(3 d)和晚期(21 d)提取外泌体,并进行micro-RNA表达谱分析,发现microRNA-31在成骨后期显著降低,而microRNA-221、microRNA-144在分化后期显著增加,提示外泌体microRNA可能在不同分化阶段发挥不同作用,但仍需进一步研究证实。
本实验中发现microRNA-100、microRNA-494、microRNA-1246在DPSCs-OD-Exo中表达上调,进一步的real-time PCR结果与芯片结果一致。以往研究显示microRNA-100可下调E-钙黏着蛋白,抑制体外的肿瘤发生、细胞运动和侵袭[10]。microRNA-100也可与microRNA-125b协同抑制5种Wnt/β-catenin负调节因子,进而促进Wnt信号转导[11],提示microRNA-100可能通过Wnt等介导细胞黏附、迁移等。Gong等[1]发现DPSCs本身的microRNA表达谱中microRNA-100同样上调,但microRNA-100对牙源性干细胞的作用尚未见到报道。microRNA-494则可调节骨肉瘤细胞的增殖、分化[12]。Zhang等[13]的研究结果显示,microRNA-494可通过调节SIRT3 /TGF-β/SMAD信号传导途径让内皮细胞向间质转化。Nakamura等[14]发现在牙齿上皮分化过程中,microRNA-1的下调诱导细胞内ATP释放至细胞外环境,从而抑制细胞增殖。本研究结果显示DPSCs-Exo和DPSCs-OD-Exo中microRNA-1246差异表达最高。Chai等[15]已证实microRNA-1246在肝癌细胞中通过抑制AXIN2和糖原的表达来激活Wnt /β-catenin途径,从而促进癌细胞自我更新、耐药性、致瘤性和转移合酶激酶3β。Hu等[8]的研究显示microRNA-1246在DPSCs-OD-Exo中下调,与本研究结果不一样,这可能是因为Hu等[8]在第10天提取细胞上清液外泌体,本研究提取DPSCs-OD-Exo的时间为21 d。microRNA-1246是否在后期对牙源性干细胞成牙本质方向分化产生影响,其作用机制仍有待研究。
为进一步研究microRNA在牙源性干细胞成牙本质方向分化中的作用,本研究将表达差异最大的microRNA-1246进行了靶基因预测和相关信号通路的分析。miRTarBase、miRDB、miRWalk数据库中交集的5个靶基因包含AXIN2,它是Wnt/β-catenin信号通路中的相关基因[16]。以往研究显示Wnt/β-catenin信号通路在干细胞分化中可发挥作用。Chen等[17]使用条件转基因小鼠行基因敲除或过表达β-catenin的实验证明,Wnt信号通路是激活牙齿发育、分化所必需的。Wang等[9] 2012年的研究结果证实域结合蛋白1可通过激活Wnt /β-catenin途径促进DPSCs的成牙本质方向分化。Tao等[18]发现生物碱可通过Wnt/β-catenin信号通路在体外诱导间充质干细胞的成牙本质方向分化。以上结果提示microRNA、尤其是microRNA-1246可能通过这些信号传导途径介导牙源性干细胞的成牙本质方向分化,但仍需要进一步研究。
综上所述,本研究结果显示DPSCs-Exo和DPSCs-OD-Exo的microRNA表达谱存在差异,数据库预测结果提示microRNA-1246可能通过AXIN2参与Wnt/β-catenin信号通路从而诱导牙源性干细胞分化,后续仍需要进一步研究证实其参与成牙本质方向分化的作用及机制。
Contributor Information
邹 晓英 (Xiao-ying ZOU), Email: zouxiaoying1125@163.com.
王 晓燕 (Xiao-yan WANG), Email: wangxiaoyanpx@163.com.
References
- 1.Gong Q, Wang R, Jiang H, et al. Alteration of microRNA expression of human dental pulp cells during odontogenic differentiation. JOE. 2012;38(10):1348–1354. doi: 10.1016/j.joen.2012.06.016. [DOI] [PubMed] [Google Scholar]
- 2.Thery C, Zitvogel L, Amigorena S. Exosomes: Composition, biogenesis and function. Nat Rev Immunol. 2002;2(8):569–579. doi: 10.1038/nri855. [DOI] [PubMed] [Google Scholar]
- 3.van Rooij E. The art of microRNA research. Circ Res. 2011;108(2):219–234. doi: 10.1161/CIRCRESAHA.110.227496. [DOI] [PubMed] [Google Scholar]
- 4.Cui Y, Luan J, Li H, et al. Exosomes derived from mineralizing osteoblasts promote ST2 cell osteogenic differentiation by alteration of microRNA expression. Febs Lett. 2016;590(1):185–192. doi: 10.1002/1873-3468.12024. [DOI] [PubMed] [Google Scholar]
- 5.刘 敬一, 邹 晓英, 陈 雪, et al. 脂多糖对人根尖牙乳头干细胞中基质细胞衍生因子1表达的影响. 中华口腔医学杂志. 2015;50(6):346–351. [PubMed] [Google Scholar]
- 6.Narayanan K, Kumar S, Padmanabhan P, et al. Lineage-specific exosomes could override extracellular matrix mediated human mesenchymal stem cell differentiation. Biomaterials. 2018;182(9):312–322. doi: 10.1016/j.biomaterials.2018.08.027. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Lotvall J, Hill AF, Hochberg F, et al. Minimal experimental requirements for definition of extracellular vesicles and their functions: A position statement from the International Society for Extracellular Vesicles. J Extracell Vesicles. 2014;3(3):13–18. doi: 10.3402/jev.v3.26913. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Hu X, Zhong Y, Kong Y, et al. Lineage-specific exosomes promote the odontogenic differentiation of human dental pulp stem cells (DPSCs) through TGFβ1/smads signaling pathway via transfer of microRNAs. Stem Cell Res Ther. 2019;10(1):170–184. doi: 10.1186/s13287-019-1278-x. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Wang J, Liu B, Gu S, et al. Effects of Wnt/β-catenin signalling on proliferation and differentiation of apical papilla stem cells. Cell Prolif. 2012;45(2):121–131. doi: 10.1111/j.1365-2184.2012.00806.x. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Chen D, Sun Y, Yuan Y, et al. miR-100 induces epithelial-mesenchymal transition but suppresses tumorigenesis, migration and invasion. PLoS Genet. 2014;10(2):57–58. doi: 10.1371/journal.pgen.1004177. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Lu Y, Zhao X, Liu Q, et al. lncRNA Mir100HG-derived miR-100 and miR-125b mediate cetuximab resistance via Wnt/β-catenin signaling. Nat Med. 2017;23(11):1331–1341. doi: 10.1038/nm.4424. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Yuan W, Wang D, Liu Y, et al. miR-494 inhibits cell proliferation and metastasis via targeting of CDK6 in osteosarcoma. Mol Med Rep. 2017;16(6):8627–8634. doi: 10.3892/mmr.2017.7709. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Zhang J, Zhu Y, Hu L, et al. miR-494 induces EndMT and promotes the development of HCC (hepatocellular carcinoma) by targeting SIRT3/TGF-β/SMAD signaling pathway. Sci Rep. 2019;9(1):7213–7225. doi: 10.1038/s41598-019-43731-4. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Nakamura T, Iwamoto T, Nakamura HM, et al. Regulation of miR-1 mediated connexin 43 expression and cell proliferation in dental epithelial cells. Front Cell Dev Biol. 2020;8(1):156–157. doi: 10.3389/fcell.2020.00156. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Chai S, Ng KY, Tong M, et al. Octamer 4/microRNA-1246 signaling axis drives Wnt/beta-catenin activation in liver cancer stem cells. Hepatology. 2016;64(6):2062–2076. doi: 10.1002/hep.28821. [DOI] [PubMed] [Google Scholar]
- 16.Katoh M. WNT signaling pathway and stem cell signaling network. Clin Cancer Res. 2007;13(14):4042–4045. doi: 10.1158/1078-0432.CCR-06-2316. [DOI] [PubMed] [Google Scholar]
- 17.Chen J, Lan Y, Baek JA, et al. Wnt/beta-catenin signaling plays an essential role in activation of odontogenic mesenchyme during early tooth development. Dev Biol. 2009;334(1):174–185. doi: 10.1016/j.ydbio.2009.07.015. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Tao K, Xiao D, Weng J, et al. Berberine promotes bone marrow-derived mesenchymal stem cells osteogenic differentiation via canonical Wnt/β-catenin signaling pathway. Toxicol Lett. 2016;240(1):68–80. doi: 10.1016/j.toxlet.2015.10.007. [DOI] [PubMed] [Google Scholar]