Abstract
目的
检测平原大鼠和急进不同海拔高原大鼠血液及组织器官中高原适应性基因(EPAS1、EGLN1和PPARα基因)含量及HIF-2α、PHD2和PPARα蛋白表达量, 初步探讨高原适应性基因及相应表达蛋白在急进高原大鼠体内心、肝、脑、肺、肾组织分布的差异。
方法
实验大鼠分为平原组、急进高原3400 m组、急进高原4300 m组; 急进高原两组到达高原第1、3、5天分别取大鼠血液及组织器官, 检测血常规, 采用实时定量PCR和ELISA方法检测高原适应性基因及相应蛋白表达量, 并对其进行比较分析。
结果
急进高原4300 m组大鼠红细胞、血红蛋白、血细胞比容高于平原组(均 P < 0.05)。与平原组比较, 急进高原两组血液和心、肝、肾组织中EPAS1基因含量明显增加(均 P < 0.05);心、肝、脑、肾组织中EGLN1基因含量增加(均 P < 0.05), 心、肝、肾组织中PPARα基因含量增加(均 P < 0.05)。与平原组比较, HIF-2α蛋白表达量在急进高原两组各组织器官中均增加, 且高海拔处肝、脑、肾组织中增加明显; 心、肝、肾组织中PHD2和PPARα蛋白表达量均增加(均 P < 0.05)。
结论
高原适应性基因的含量和相应蛋白表达量在不同海拔和不同组织中存在一定的差异。提示EPAS1、EGLN1和PPARα基因可作为抗高原缺氧药物靶向标志物。
Abstract
Objective
To detect the expression of the plateau adaptablity gene(EPAS1, EGLN1 and PPARα) and proteins(HIF-2, PHD2 and PPARα) in rats blood, heart, liver, lung and kidney tissue after the rats exposed to high altitude.
Methods
The Wistar rats were randomly divided into plain group(Shanghai, 55 m), acute exposure to high altitude 3400 m group, acute exposure to high altitude 4300 m group. Blood and organs of rats were collected in 1, 3, 5 days after arrival. Real time PCR and ELISA were used to compare the expression of plateau adaptablity gene and related protein between plain group and high altitude exposure groups.
Results
The count of red blood cells, hemoglobin and HCT in high altitude 4300 m were higher than those in plain group. Compared with plain group, the expression of EPAS1 gene in blood, heart, liver and kidney tissue of rats at high altitude increased obviously(all P < 0.05); the expression of EGLN1 in the heart, liver, brain and kidney increased, and PPARα gene in the heart, liver and kidney increased(all P < 0.05). Compared with plain group, the expression of HIF-2 protein increased significantly at high altitudes in the liver, brain and kidney tissues. PHD2 and PPARα increased in the heart, liver and kidney.
Conclusion
Plateau adaptive genes(EPAS1, EGLN1 and PPARα) and protein(HIF-2, PHD2 and PPARα) differed in different altitude and different organizations. They might be used as target markers of plateau hypoxia.
Keywords: Altitude sickness/blood, Altitude, Genes, Rats, Polymerase chain reaction
高海拔地区大气氧分压随着海拔升高而降低,学习、工作、生活在高原地区的人们必将面对高原低氧环境所带来的种种不适。如果机体无法完全适应时就可能导致急、慢性高原病的发生,但并非所有居住于高原的人都将发生高原病。研究报道喜马拉雅山脉的藏族人、安第斯高原的安第斯人已世居高原数千年,均已形成了各自的低氧抵抗策略 [ 1, 2, 3] 。青藏高原的藏族作为高原低氧适应的代表,被公认为能更好地适应高海拔环境,藏族人群有着较高的一氧化氮水平、较大的静息状态下通气量和较强的低氧通气反应、较高的出生体质量以及与海平面居住人群相近的血红蛋白浓度,反映出其对高原环境独特的适应性。近年来,有学者等对比研究了久居西藏人群与居住在低海拔地区人群的基因组模式,发现有30多种基因与高原适应相关 [ 4, 5] 。同时,对藏族高原适应相关基因如内皮型一氧化氮合酶基因、肾素—血管紧张素—醛固酮系统(RAS)相关基因、内皮素-1(ET-1) 基因等研究发现,内皮PAS蛋白1(endothelial PAS domain-containing protein 1,EPAS1)、脯氨酸羟化酶2(prolyl hydroxylase domain-containing protein 2,PHD2)、过氧化物酶体增殖物激活受体(peroxisome proliferator-activated receptors,PPAR)α等参与了高原缺氧适应 [ 4, 6, 7, 8, 9, 10, 11] ,通过EPAS1、EGLN1和PPARα等基因的调节,控制着红细胞生成和其他适应功能 [ 4] 。高原地区藏族、低海拔地区非藏族(汉族或日本人群) EPAS1和EGLN1基因表达明显不同 [ 5, 10] 。EPAS1、EGLN1和PPARα等基因在高原适应性研究中具有重要的作用,同时,这些基因和相关蛋白在重要器官组织中的分布差异性研究将为深入研究高原适应性特点及抗高原缺氧药物生物标志物,对高原相关疾病的防治和抗缺氧药物的研究有重大的科学价值。本研究观察了平原大鼠与急进高原适应大鼠的血液及各器官组织中高原适应性基因含量和相关蛋白的表达差异,为进一步研究急进高原后高原疾病的防治积累基础数据。
ABI 9700型PCR仪和ABI 7300实时定量PCR仪(美国ABI公司),SpectraMax M5酶标仪(美国Molecular Devices公司),PowerPal电泳仪(美国Bio-Rad公司),冷冻高速离心机(德国Sigma公司),UV2800S紫外分光光度计(上海舜玉衡平科学仪器有限公司),AE240电子天平(瑞士梅特勒—托利多公司),超低温冰箱(中国海尔公司),SW-CJ-2FD超净工作台(中国杭州净化工作设备厂),超纯水系统(美国热电科学仪器有限公司),微量移液器(德国Eppendorf公司),液氮罐(四川东亚机电公司),恒温水浴锅(上海比朗仪器有限公司),自动血气采血针(美国BD公司),无酶枪头、PCR八连管(美国Axygen公司)。
PrimeScript TM RT Master Mix Kit、SYBR Premix Ex Taq TMII和TRIzol RNA提取试剂购自大连Takara公司,乙醇、异丙醇和氯仿购自成都金山化学试剂有限公司,DEPC购自生工生物工程(上海)股份有限公司,缺氧诱导因子(hypoxia induced factor, HIF)2α、PDH2和PPARα ELISA试剂盒购自美国RD公司。
70只6~7周龄的SPF级健康雄性Wistar大鼠,体质量(200±10) g,购自上海斯莱克实验动物有限责任公司(动物质量合格证书编号2007000525382)。将大鼠随机分为平原组(上海,海拔55 m, n=10)、高原3400 m组(甘肃碌曲,海拔3400 m, n=30) 和高原4300 m组(青海玛多,海拔4300 m, n=30),进一步将30只大鼠随机分入1 d、3 d、5 d检测( n=10)。
平原大鼠饲养于上海第二军医大学药学院实验动物房,大鼠以标准饲料饲养,饮用自来水。急进高原大鼠采用灭菌垫料、钙塑箱运输,航空运输中采用果冻补水,汽车运输途中饮用纯净水,饲养条件相同,饲料同平原组采用标准饲料,自由进食饮水。急进高原大鼠由上海航空托运至甘肃兰州中川机场后立即由空调箱式货车转运大鼠到达相应的海拔高度的高原,急进过程均在12 h内完成,高原大鼠分别饲养于保温实验室。平原组及急进高原组大鼠分别于到达各实验地点的第1、3、5天后开始取血及组织样品。大鼠眼眶后静脉丛取血1 mL置于1.5 mL离心管中,离心管预先加入20 μL EDTA二钾抗凝剂,血样每300 μL分至不同离心管,迅速保存于-80 ℃冰箱,避免反复冻融。在处死大鼠后完整摘取心、肝、脑、肺、肾组织,用预冷的0.9%氯化钠溶液冲洗,液氮保存以备用。
分别提取各器官约100 mg组织放入研钵中,放入液氮迅速研磨至粉末状,继续加入1 mL TRIzol研磨至澄清液体,转移至1.5 mL的EP管中,4 ℃ 12 000× g离心5 min,上清液转移至另一EP管,加入0.2%氯仿的TRIzol溶液,充分振荡混匀30 s,室温孵育5 min,4 ℃ 12 000× g离心15 min;迅速转移上清液至另一新EP管,按0.5 mL加入0.2%异丙醇的TRIzol溶液,颠倒混匀,室温孵育10 min,4 ℃ 12 000× g离心10 min,弃上清液,沉淀获得RNA;按1 mL 75%乙醇溶液(DEPC处理水配制)/1 mL TRIzol的比例,加入75%乙醇溶液漂洗RNA沉淀,4 ℃ 7 500× g离心5 min,弃上清液;自然干燥RNA沉淀10 min,加入适量DEPC处理水溶解RNA沉淀,于-80 ℃保存备用。逆转录cDNA 10 μL反应体系,37 ℃逆转录15 min,85 ℃热失活反应5 s。以每10 μL体系含RNA 500 ng,将提取液稀释25倍,用紫外分光光度计测定波长260、280 nm处吸光度值,比值为1.8~2.1。
EPAS1、EGLN1、PPARα和β-actin引物由Takara公司设计合成。引物序列见 表 1。实时定量PCR 10 μL反应体系,反应条件为95 ℃预变性30 s,95 ℃反应5 s,60 ℃退火31 s,重复40个循环。每个样品做3个复孔,以β-actin为内参基因,目的基因的相对含量采用2 -ΔΔCT进行计算分析。ΔΔCT=CT值(高原组靶基因-高原组内参)-CT值(平原组靶基因-平原组内参)。
表1 实时定量PCR引物序列
Table 1 Primer sequences of real time PCR
|
基因名称 |
引物序列 |
产物长度(bp) |
|
β-actin |
150 |
|
|
上游引物 |
5′-GGAGATTACTGCCCTGGCTCCTA-3′ |
|
|
下游引物 |
5′-GACTCATCGTACTCCTGCTTGCTG-3′ |
|
|
EPAS1 |
135 |
|
|
上游引物 |
5′-TTCCTGCGAACACATAAACTCCTG-3′ |
|
|
下游引物 |
5′-ATCACCGTCTTGGGTCACCAC-3′ |
|
|
EGLN1 |
109 |
|
|
上游引物 |
5′-AGCCTGCATACGCCACAA-3′ |
|
|
下游引物 |
5′-TCAACCCTCACACCTTTCTCAC-3′ |
|
|
PPARα |
112 |
|
|
上游引物 |
5′-GGCAATGCACTGAACATCGAG-3′ |
|
|
下游引物 |
5′-GCCGAATAGTTCGCCGAAAG-3′ |
|
将96孔板设为12个标准品孔、6个空白孔。在标准品孔内加50 μL,依次稀释;待测样品孔中先加样品稀释液40 μL,再加待测样品10 μL;除空白孔外,每孔加入酶标试剂50 μL,采用摇板机摇匀,37 ℃孵育60 min;洗板5次,拍干;加显色剂A、B各50 μL,37 ℃显色15 min;加终止液50 μL终止反应。进行总蛋白校正,根据标准品的浓度及对应的吸光度值计算标准曲线,在450 nm波长下测各孔吸光度值,即可获得蛋白含量。
所有统计分析均采用SPSS 13.0统计软件完成,计算结果以均数±标准差( x ± s)表示,组间比较用方差分析,检验水准α=0.05, P<0.05表示差异具有统计学意义。
高原4300 m组大鼠红细胞计数、血红蛋白浓度、血细胞比容均高于平原组(均 P<0.05),见 表 2。提示大鼠急进高原后血细胞携氧能力增加。
表2 各组血常规检测结果比较
Table 2 Results of blood indicators in rats at different altitude and exposure time
|
( x ± s, n=10) | |||||
|
组别 |
红细胞计数(×10 12/L) |
血红蛋白浓度(g/L) |
血细胞比容 |
红细胞平均体积(fL) |
红细胞平均血红蛋白浓度(g/L) |
|
平原组 |
7.39±0.09 |
140.6±2.78 |
0.41±0.01 |
57.76±1.34 |
347.17±0.12 |
|
高原4300 m组 | |||||
|
1 d |
7.63±0.12 * |
157.67±4.62 * |
0.45±0.02 * |
58.83±0.93 * |
353.00±0.27 |
|
3 d |
7.95±0.14 * |
160.25±8.02 * |
0.46±0.02 * |
57.27±0.31 |
357.67±0.52 * |
|
5 d |
7.98±0.12 * |
164.00±11.31 * |
0.44±0.02 * |
57.67±0.58 |
348.33±0.31 |
|
与平原组比较, * P<0.05. |
平原组和高原两组不同适应时间(1 d、3 d、7 d)血液及各器官组织EPAS1基因检测结果见 表 3。提示高原两组血液和心、肝、肾组织中EPAS1基因含量均高于平原组(均 P<0.05),而在肺组织这种差异不明显(均 P>0.05)。
表3 各组不同适应时间血液和器官组织中EPAS1基因含量比较
Table 3 Expression of EPAS1 gene in the different tissue of rats at different high alititude and exposure time
|
( x ± s, n=10) | |||||||
|
组别 |
适应时间(d) |
血液 |
心 |
肝 |
脑 |
肺 |
肾 |
|
平原组 |
0 |
1.02±0.16 |
0.89±0.21 |
1.13±0.17 |
0.69±0.21 |
0.99±0.32 |
1.07±0.26 |
|
高原3400 m组 |
1 |
1.63±0.13 * |
0.93±0.11 |
3.94±0.20 * |
0.96±0.32 |
1.24±0.09 |
3.37±0.29 * |
|
3 |
1.85±0.15 * |
1.15±0.21 |
3.01±0.18 * |
0.77±0.14 |
1.01±0.20 |
3.25±0.33 * |
|
|
5 |
1.12±0.02 |
3.72±0.09 * |
1.82±0.12 * |
0.83±0.02 |
1.22±0.04 |
2.13±0.16 * |
|
|
高原4300 m组 |
1 |
1.17±0.01 |
1.20±0.08 |
4.79±0.34 * |
0.32±0.07 * |
1.02±0.10 |
2.36±0.28 * |
|
3 |
3.15±0.28 * |
4.85±0.82 * |
4.18±0.23 * |
0.56±0.22 |
0.94±0.18 |
3.28±0.24 * |
|
|
5 |
3.04±0.21 * |
3.25±0.32 * |
2.14±0.17 * |
0.63±0.05 |
0.81±0.23 |
2.64±0.17 * |
|
|
与平原组比较, * P<0.05. |
与平原组比较,两高原组肝组织EGLN1基因表达均增加(均 P<0.05),高原4300 m组心肌组织EGLN1基因表达均增加(均 P<0.05),两高原组肾组织EGLN1基因表达均减少(均 P<0.05),高原4300 m组脑组织EGLN1基因表达均减少(均 P<0.05),见 表 4。
表4 各组不同适应时间器官组织中EGLN1基因含量的比较
Table 4 Expression of EGLN1 gene in the different tissue of rat at the different high alititude and exposure time
|
( x ± s, n=10) | ||||||
|
组别 |
适应时间(d) |
心 |
肝 |
脑 |
肺 |
肾 |
|
平原组 |
0 |
0.95±0.23 |
0.17±0.22 |
1.01±0.21 |
0.80±0.31 |
1.21±0.33 |
|
高原3400 m组 |
1 |
0.80±0.19 |
0.39±0.12 * |
0.93±0.14 |
0.55±0.06 * |
0.52±0.12 * |
|
3 |
0.78±0.33 |
0.71±0.33 * |
1.19±0.20 |
0.59±0.13 * |
0.60±0.17 * |
|
|
5 |
1.12±0.26 |
0.80±0.16 * |
1.42±0.27 * |
0.93±0.10 |
0.32±0.10 * |
|
|
高原4300 m组 |
1 |
1.46±0.23 * |
0.28±0.09 * |
0.61±0.11 * |
0.81±0.11 |
0.24±0.06 * |
|
3 |
1.31±0.15 * |
0.44±0.24 * |
0.47±0.09 * |
0.83±0.19 |
0.59±0.21 * |
|
|
5 |
1.35±0.27 * |
0.54±0.17 * |
0.77±0.13 * |
0.72±0.13 |
0.36±0.09 * |
|
|
与平原组比较, * P<0.05. |
两高原组心、肝、肾组织中PPARα基因含量均高于平原组(均 P<0.05),而脑、肺组织中PPARα基因无特异表达,见 表 5。
表5 不同海拔不同适应时间大鼠心、肝、肾组织中PPARα基因含量的变化
Table 5 Expression of PPARα gene in the different tissue of rats at different high alititude and exposure time
|
( x ± s, n=10) | ||||
|
组别 |
适应时间(d) |
心 |
肝 |
肾 |
|
平原组 |
0 |
0.91±0.23 |
1.17±0.21 |
0.97±0.25 |
|
高原3400 m组 |
1 |
2.79±0.34 * |
3.81±1.21 * |
4.46±1.21 * |
|
3 |
4.22±1.10 * |
3.29±1.38 * |
2.70±0.17 * |
|
|
5 |
2.36±0.91 * |
3.63±0.89 * |
5.61±0.60 * |
|
|
高原4300 m组 |
1 |
3.95±0.93 * |
7.27±0.93 * |
10.30±2.14 * |
|
3 |
7.13±1.74 * |
10.60±2.05 * |
38.10±9.33 * |
|
|
5 |
7.50±1.48 * |
5.10±1.46 * |
9.21±1.46 * |
|
|
与平原组比较, * P<0.05. |
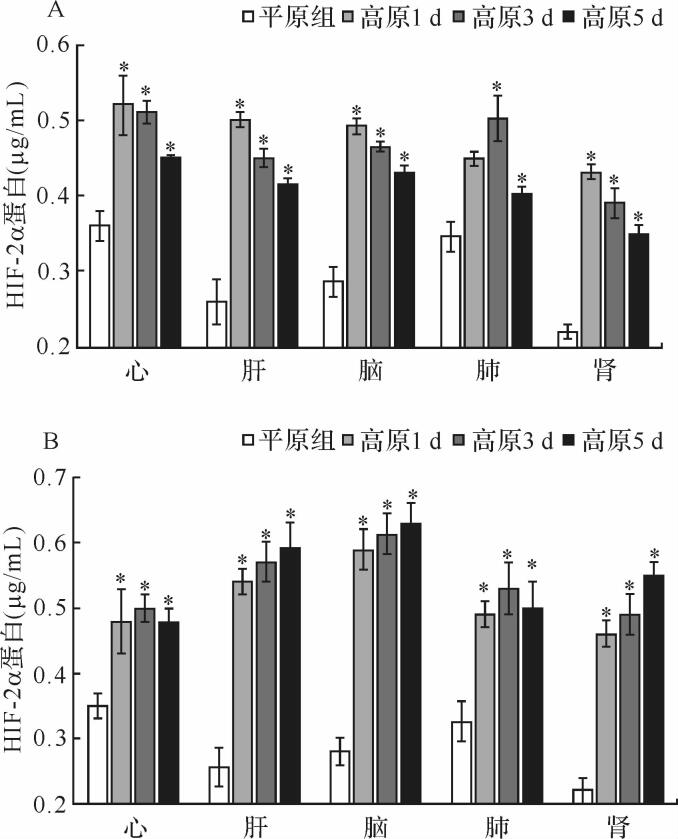
与平原组比较,两高原组心、肝、脑、肺、肾组织中HIF-2α蛋白表达量均增加(均 P<0.05),见 图 1。
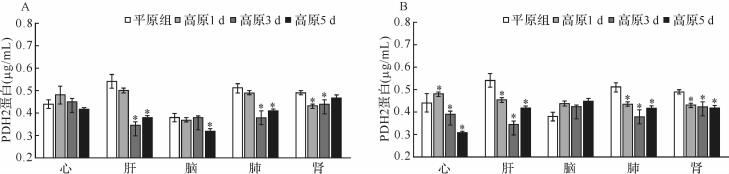
与平原组比较,两高原组肝、肺、肾组织中PHD2蛋白表达的含量均有所降低(均 P<0.05),而心组织中差异不明显,见 图 2。
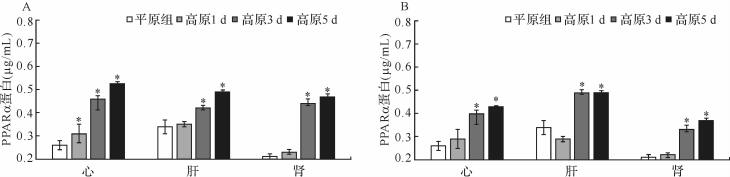
与平原组比较,两高原组心、肝、肾组织PPARα蛋白表达量均增加(均 P<0.05),见 图 3。
本研究观察了平原组大鼠、急进高原3400 m和4300 m并经过不同高原适应时间(1 d、3 d、5 d)大鼠器官组织中EPAS1、EGLN1、PPARα基因含量和PHD2、HIF-2α、PPARα蛋白表达量。结果显示,与平原组比较,EPAS1、EGLN1、PPARα基因含量和PHD2、HIF-2α、PPARα蛋白表达量在大鼠急进不同海拔高原经过不同时间适应后,心、肝、肾器官变化最为显著,提示在心、肝、肾器官中通过EPAS1、EGLN1、PPARα基因调控,PHD2、HIF-2α、PPARα蛋白表达量也发生变化,从而达到调节机体适应高原缺氧的效果。
观察结果表明,与平原组比较,高原大鼠EPAS1基因含量在不同海拔和缺氧时间后有显著差异,海拔4300 m处大鼠肝组织中EPAS1基因含量在急进高原的第1天最高,在第3、5天中基因含量有所减少,表明机体由急性缺氧转为初期习服过程。EPAS1能够促进血管生成并提高了红细胞携带氧气的能力,从而改善细胞的物质代谢,在应激作用下对血红蛋白的生成有促进作用,有利于缓解高原缺氧对动物的影响。本文资料中两高原组大鼠血红蛋白浓度高于平原组亦证实此观点。李雪冰 [ 14] 综述了高原低氧适应与EPAS1/HIF-2α及EGLN1/PHD2的相关性,EPAS1/HIF-2α及邻近的EGLN1/PHD2识别为自然选择的候选基因,提示这些基因变异在藏族高原适应中的关键作用,本观察结果与此一致。在常氧条件下EGLN1/PHD2将HIF-2α泛素化,泛素—蛋白酶被迅速降解,低氧条件下,PHD2羟基化HIF-2α反应受阻,降解途径中断,HIF-2α亚基大量积累 [ 15] 。PHD2蛋白表达量降低,HIF-2α蛋白表达量增加,HIF-2α在调节应答和改变氧气利用过程中起着核心作用,其表达增加以适应缺氧反应,影响着机体发育、代谢和炎症等过程,并证实HIF-2α是高原物种低氧环境的重要基础 [ 16, 17, 18, 19, 20, 21] 。本观察结果与文献报道一致 [ 14, 15, 16, 17, 18, 19, 20, 21] 。
本文资料提示,急进高原大鼠器官组织中EPAS1、EGLN1、PPARα基因含量和PHD2、HIF-2α、PPARP蛋白表达与平原大鼠存在明显差异,其具体调控机制有待于进一步深入研究。
Funding Statement
国家科技重大专项(2008ZXJ09014-010);国家自然科学基金(81401552);全军医学科研"十二五"重点项目(BWS12J012)
References
- 1.WU T, WANG X, WEI C, et al. Hemoglobin levels in Qinghai-Tibet:different effects of gender for Tibetans vs. J Appl Physiol, 2005;98(2):598–604. doi: 10.1152/japplphysiol.01034.2002. [DOI] [PubMed] [Google Scholar]
- 2.BEALL C M. Two routes to functional adaptation:Tibetan and Andean high altitude natives. Proc Natl Acad Sci USA. 2007;104 doi: 10.1073/pnas.0701985104. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.MOORE L G. Human genetic adaptation to high altitude. High Alt Med Biol. 2001;2(2):257–279. doi: 10.1089/152702901750265341. [DOI] [PubMed] [Google Scholar]
- 4.SIMONSON T S, YANG Y Z, HUFF C D, et al. Genetic evidence for high-altitude adaptation in Tibet. Science. 2010;329(5987):72–75. doi: 10.1126/science.1189406. [DOI] [PubMed] [Google Scholar]
- 5.YI X, LIANG Y, HUERTA-SANCHEZ E, et al. Sequencing of 50 human exomes reveals adaptation to high altitude. Science. 2010;329(5987):75–78. doi: 10.1126/science.1190371. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.CHEN Y, GAO Y Q. Practice and application of genomics research in genetic mechanism of high-altitude adaptation in Tibetan. Int J Genet. 2012;35(4):213–216. [Google Scholar]
- 7.WANG B, ZHANG Y B, ZHANG F, et al. On the origin of Tibetans and their genetic basis in adaptating high-altitude environments. PLoS One. 2011;6(2) doi: 10.1371/journal.pone.0017002. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.PENG Y, YANG Z, ZHANG H, et al. Genetic variations in Tibetan populations and high-altitude adaptation at the Himalayas. Mol Biol Evol. 2011;28(2):1075–1081. doi: 10.1093/molbev/msq290. [DOI] [PubMed] [Google Scholar]
- 9.BIGHAM A, BAUCHET M, PINTO D, et al. Identifying signatures of natural selection in Tibetan and Andean populations using dense genome scan data. PLoS Genet. 2010;6(9) doi: 10.1371/journal.pgen.1001116. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.XU S H, LI S L, YANG Y J, et al. Agenome-wide search for signals of high-altitude adaptation in Tibetans. Mol Biol Evol. 2011;28(2):1003–1011. doi: 10.1093/molbev/msq277. [DOI] [PubMed] [Google Scholar]
- 11.WANG B, ZHANG Y B, ZHANG F, et al. On the origin of Tibetans and their genetic basis in adapting high-altitude environments. PLoS One. 2011;6(2) doi: 10.1371/journal.pone.0017002. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.YANG Y Z, WANG Y P, QI Y J, et al. Endothelial PAS domain protein 1 Chr2:46441523(hg18) polymorphism is associated with susceptibility to high altitude pulmonary edema in Han Chinese. Wilderness Environ Med. 2013;24(4):315–320. doi: 10.1016/j.wem.2013.05.006. [DOI] [PubMed] [Google Scholar]
- 13.WEBER R E. High-altitude adaptations in vertebrate hemoglobins. Respir Physiol Neurobiol. 2007;158(2-3):132–142. doi: 10.1016/j.resp.2007.05.001. [DOI] [PubMed] [Google Scholar]
- 14.李 雪冰. 高原低氧适应与EPAS1/HIF-2H及EGLN1/PHD2的相关性. http://www.cnki.com.cn/Article/CJFDTOTAL-YXZS201403006.htm 医学综述. 2014;20(3):401–404. [Google Scholar]
- 15.TAKEDA K, AGUILA H L, PARIKH N S, et al. Regulation of adult erythropoiesis by prolyl hydroxylase domain proteins. Blood. 2008;111(6):3229–3235. doi: 10.1182/blood-2007-09-114561. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.FONG G H, TAKEDA K. Role and regulation of prolyl hydroxylase domain proteins. Cell Death Differ. 2008;15(4):635–641. doi: 10.1038/cdd.2008.10. [DOI] [PubMed] [Google Scholar]
- 17.KAELIN W G JR, RATCLIFFE P J. Oxygen sensing by metazoans:the central role of the HIF hydroxylase pathway. Mol Cell. 2008;30(4):393–402. doi: 10.1016/j.molcel.2008.04.009. [DOI] [PubMed] [Google Scholar]
- 18.HUANG J, ZHAO Q, MOONEY S M, et al. Sequence determinants in hypoxia-inducible factor-1α for hydroxylation by the prolyl hydroxylases PHD1, PHD2, and PHD3. J Bio Chem. 2002;277(42):39792–39800. doi: 10.1074/jbc.M206955200. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.SPEER R E, KARUPPAGOUNDER S S, BASSO M, et al. Hypoxia-inducible factor prolyl hydroxylases as targets for neuroprotection by "antioxidant" metal chelators:from ferroptosis to stroke. Free Radic Bio Med. 2013;62:26–36. doi: 10.1016/j.freeradbiomed.2013.01.026. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.SONG D, LI L S, ARSENAULT P R, et al. Defective Tibetan PHD2 binding to p23 links high altitude adaptation to altered oxygen sensing. J Bio Chem. 2014:289. doi: 10.1074/jbc.M113.541227. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.LI Q, LIU S Y, LIN K Q, et al. Association between six single nucleotide polymorphisms of EGLN1 gene and adaptation to high-altitude hypoxia. Hereditas. 2013;35(8):992–998. doi: 10.3724/sp.j.1005.2013.00992. [DOI] [PubMed] [Google Scholar]