Abstract
目的
比较卒中后认知功能障碍(post-stroke cognitive impairment, PSCI)患者与健康人群的肠道菌群结构差异,并探讨差异菌种与认知功能的相关性。
方法
选取24例患者作为PSCI组,同期匹配23例无心脑血管疾病史的健康人群作为对照组。两组均采集粪便标本,采用简易精神状态检查(Mini-Mental State Examination, MMSE)和蒙特利尔认知评估表(Montreal Cognitive Assessment, MoCA)行认知功能评估。采用16S rRNA基因测序技术进行肠道菌群丰度、多样性和差异性分析,通过RDA/CCA分析差异菌种与认知评分的相关性。
结果
两组一般资料比较及肠道菌群Alpha多样性差异无统计学意义(P>0.05)。组间物种分布比较显示菌群在门和种水平构成存在差异,主要为厚壁菌门、拟杆菌门相对丰度减少和变形菌门相对富集。PSCI组中厚壁菌门的Intestinibacter bartlettii、uncultured bacteriumTyzzerella_3、Fusicatenibacter saccharivorans的相对丰度低于对照组(LDA score>2),且这些细菌与认知功能评分呈正相关,细菌彼此间存在相关性;厚壁菌门的Ruminococcus gnavus、Faecalimonas umbilicata,拟杆菌门的unculturedbacterium Prevotellaceae_NK3B31 group的相对丰度高于对照组(LDA score>2),且这些细菌与认知功能评分呈负相关,细菌彼此间亦存在相关性。
结论
PSCI患者与健康对照比较肠道菌群结构特征存在差异,并且与认知功能存在一定相关性,为PSCI的防治提供了新思路。
Keywords: 肠道菌群, 脑卒中, 认知功能障碍, 简易精神状态检查, 蒙特利尔认知评估表
Abstract
Objective
To identify the differences in the composition of gut microbiota of patients with post-stroke cognitive impairment (PSCI) in comparison with the normal cognition healthy controls (HC), and to study the potential association between gut microbiota and cognition function.
Methods
A total of 24 patients were recruited for the PSCI group, which was matched with 23 healthy subjects with no history of cardiovascular disease recruited over the same period for the control group. Fecal samples were collected for both groups, and Mini-Mental State Examination (MMSE) and Montreal Cognitive Assessment (MoCA) were used to evaluate cognitive functions. The abundance, diversity and group difference of gut bacterial communities were determined with 16S rRNA gene sequencing, and the correlations between differences in bacterial species of the gut microbiota and cognitive function scores were examined with redundancy analysis (RDA)/canonical correspondence analysis (CCA).
Results
There was no significant difference in the general data or the alpha diversity of gut microbiota between the two groups (P>0.05). Inter-group comparison of microbial species composition revealed differences at the phylum and species levels, mainly represented as reduction in the relative abundance of Firmicutes and Bacteroidetes, and the relative enrichment of Proteobacteria. The relative abundance ofIntestinibacter bartlettii, uncultured bacterium Tyzzerella_3, Lactobacillus gasseri, and Fusicatenibacter saccharivorans of phylum Firmicutes in the PSCI patients were significantly reduced in comparison to that of the HC (LDA score>2), and these bacteria were positively correlated with MMSE and MoCA scores. In addition, theRuminococcus gnavus and Faecalimonas umbilicata of phylum Firmicutes and unculturedbacteriumPrevotellaceae_NK3B31 group of phylum Bacteroidetes were significantly enriched in comparison with those of the HC (LDA score>2), and these bacteria were negatively correlated with MMSE and MoCA scores. There were also correlations among these bacteria.
Conclusion
In this study, we observed compositional differences between the gut microbiota of PSCI patients and those of HC, and revealed that the differences were correlated, to some degree, to the cognitive functions, which will provide new perspectives for the clinical diagnosis and treatment of PSCI.
Keywords: Gut microbiota, Stroke, Cognitive impairment, Mini-Mental State Examination (MMSE), Montreal Cognitive Assessment (MoCA)
卒中后认知功能障碍(post-stroke cognitive impairment, PSCI)是脑卒中后常见的并发症之一[1]。我国一项以社区为基础的调查研究显示,PSCI在脑卒中后总的患病率高达80.97%[2]。认知受损不仅导致PSCI患者日常生活能力和社交能力下降,生活质量降低,而且还会影响康复治疗的主动性及依从性,严重妨碍肢体运动功能为主的全面康复[3],增加卒中患者的终身残疾率和死亡率[4]。PSCI发病机制迄今尚不明确,目前临床上仍无有效的防治手段[5]。因此,迫切需要从新的角度探索PSCI的发生机制,为PSCI的防治挖掘新的治疗靶点。近年来,基于“肠道菌群-肠-脑轴”探讨脑卒中的防治逐渐成为新兴热点。该理论认为肠道菌群可以通过免疫、神经内分泌以及迷走神经等“双向脑肠信号”与中枢神经系统进行信息交流,影响宿主的脑功能,进而影响其行为和情绪[6-8]。已有不少研究发现肠道菌群与脑卒中的发生发展及预后密切相关[9-11]。LIU等[12]于2020年首次提出PSCI与肠道菌群及代谢产物的相关性,提示肠道菌群可能在PSCI的发生发展中起重要作用,但目前相关的研究尚少,PSCI患者肠道菌群紊乱特征需要更多的研究验证。基于此,本文拟通过与健康对照组比较,观察PSCI患者肠道菌群的变化特征,以及差异菌种与认知功能的相关性,旨在从肠道菌群角度探索PSCI的发生机制,为PSCI的有效防治提供新的“切入点”。
1. 资料与方法
1.1. 纳入和排除标准
1.1.1. 病例组纳入标准
①符合脑血管病诊断标准[13];②首次卒中,40~80岁,病程≤1月;③发病前无认知障碍,发病后简易精神状态检查(Mini-Mental State Examination, MMSE)和蒙特利尔认知评估表(Montreal Cognitive Assessment MoCA)评定存在认知障碍[14]。④患者居住地点稳定,在成都居住时间>3年,且生活时间>10月/年;⑤饮食结构基本相同,无特殊饮食习惯。⑥体质量指数(bodymass index, BMI)为17.5~30.0 kg/m2;⑦近期没有跨境旅游;⑧1个月内无抗生素、益生元和(或)益生菌使用史;⑨无其它胃肠道、肝脏及胆胰等消化系统疾病和手术史;⑩患者或其家属签署知情同意书。
1.1.2. 对照组纳入标准
同期根据年龄和性别匹配无心脑血管疾病史的健康人群作为对照组:①既往无脑卒中、高血压、糖尿病、冠心病、高脂血症等病史;②颈部动脉超声检测无斑块;③无认知功能障碍;④最近1个月未服用抗生素、益生元和(或)益生菌;⑤无慢性腹泻、便秘病史。
1.1.3. 排除标准
①非动脉粥样硬化导致的脑卒中(如肿瘤、动脉夹层、心源性卒中、或其他TOAST亚型)患者;②既往有脑外伤或其他颅内疾病者;③既往有认知障碍病史,或其他原因(如大量饮酒、滥用药物等)所致的认知障碍;④合并急性或慢性感染性疾病、恶性肿瘤、严重自身免疫性疾病、免疫缺陷综合征、严重精神疾病者;⑤合并炎症性肠病、慢性腹泻、慢性便秘、消化道大出血或肝硬化;⑥合并严重威胁生命的重大疾病(如肝肾功能衰竭、心脏衰竭、呼吸衰竭等);⑦有严重的视力、听力障碍、意识障碍等,无法配合认知功能评估;⑧既往接受过肠道肿瘤手术。
1.2. 一般资料
本研究经医院伦理委员会批准〔批准号:伦审(研)2020年第384号〕,所有研究对象均由患者或其家属签署知情同意书。选取2020年2−12月在我院住院且符合上述标准的PSCI患者作为PSCI组(24例)和健康体检者作为对照组(23例)。收集两组患者一般资料,包括年龄、性别、BMI、受教育年限、烟酒史、饮食结构、排便情况等,两组患者一般资料比较差异无统计学意义(P>0.05)(表1)。
表 1. Analysis of the clinical data of PSCI patients and healthy controls.
PSCI组与对照组临床指标分析结果
| Clinical characteristic | HC group (n=23) | PSCI group (n=24) | P |
| HC: Healthy control; PSCI: Post-stroke cognitive impairment; Dietary habit: Meat means meat (meat, eggs) ratio≥50%, vegetarian means meat (meat, eggs) ratio<50%; Fried/preserved food: Yes indicates the total frequency of fried or preserved food≥3 times per week, otherwise no. | |||
| Age/yr. | 58.13±8.25 | 63.17±12.58 | 0.113 |
| Sex (male/female)/case | 10/13 | 16/8 | 0.055 |
| BMI/(kg/m2) | 23.86±2.10 | 24.73±1.67 | 0.119 |
| Education/yr. | 9.39±2.08 | 9.75±2.38 | 0.586 |
| Smoke (yes/no)/case | 7/16 | 13/11 | 0.100 |
| Alcohol (yes/no)/case | 5/18 | 11/13 | 0.081 |
| Dietary habit (meat/vegetable)/case | 10/13 | 17/7 | 0.058 |
| Staple food proportion
(≥50%/<50%)/case |
8/15
|
11/13
|
0.440
|
| Fried/preserved food (yes/no)/case | 8/15 | 7/17 | 0.680 |
| Defecation
(≥once a day/<once a day)/case |
16/7
|
12/12
|
0.172
|
| Fecal character (soft/hard)/case | 18/5 | 13/11 | 0.081 |
| MMSE/score | 28.83±1.67 | 14.00±2.83 | <0.001 |
| MoCA/score | 29.09±1.20 | 13.25±3.47 | <0.001 |
1.3. 检测方法
所有患者均于入组后48 h内采集粪便标本,并于采集粪便当天再次行认知功能评定。
1.3.1. 认知功能评定
由1名临床经验丰富的康复科高年资主治医师采用MMSE和MoCA进行评定。MMSE采用张明园版本,总分30分,文化程度为初中及以上者≥24分、小学者≥20分、文盲者≥17分为正常。MoCA采用北京版,总分为30分,受教育年限≤12年者总分加1分,以矫正教育程度的偏差。总分≥26分为认知功能正常,<26分存在认知功能损害。
1.3.2. 粪便标本采集
粪便标本采集由1名接受严格无菌操作培训的人员完成。采集新鲜的、中后段、内部的粪便样本,使用干净无菌勺子收集,每份标本4~6 g,放入无菌冻存管中,取样完毕后立即盖上盖子旋紧,并立即保存至-80℃冰箱,保存期间切忌反复冻融。检测时足量干冰低温运送。
1.3.3. 粪便总DNA提取、PCR扩增和测序
采用高通量测序技术对粪便标本进行微生物多样性全长16S rRNA测序。首先按照MN NucleoSpin 96 Soi试剂盒操作流程提取样本总DNA,然后根据16S全长引物27F(5′-AGRGTTTGATYNTGGCTCAG-3′)和1492R(5′-TASGGHTACCTTGTTASGACTT -3′),合成带有Barcode的特异引物,进行PCR扩增。对扩增产物进行纯化、定量和均一化形成SMRT Bell测序文库,对建好的文库先进行文库质检,质检合格的文库用PacBio Sequel测序平台(美国PacBio公司)进行上机测序。PacBio Sequel下机数据为bam格式,通过smrtlink软件(version8.0)导出为环形一致性序列(CCS, Circular Consensus Sequencing)文件。
1.3.4. 生物信息学分析
1.3.4.1. 数据预处理
将 PacBio 下机数据导出为 CCS 文件后,对原始数据主要有如下3个预处理步骤:①CCS识别:使用lima(v1.7.0)软件,通过barcode对CCS进行识别,得到的Raw-CCS序列数据;②CCS过滤:使用cutadapt 1.9.1软件进行引物序列的识别与去除并且进行长度过滤,得到不包含引物序列的Clean-CCS序列;③去除嵌合体:使用UCHIME(version8.1)软件,鉴定并去除嵌合体序列,得到Effective-CCS序列[15]。然后使用百迈客医学微生物多样性分析平台( www.biocloud.net)进行数据分析。
1.3.4.2. OUT聚类
不同序列之间的相似性高于97%则定义为一个操作分类单元(operational taxonomic unit, OTU),每个OTU对应一种代表序列。使用Usearch(version10.0)软件[16]对不同序列在97.0%的相似度水平下进行聚类、获得OTUs。
1.3.4.3. 物种注释及分类学分析
以SILVA为参考数据库使用朴素贝叶斯分类器结合比对的方法对特征序列进行分类学注释,可得到每个特征对应的物种分类信息,进而在各水平统计各样品群落组成,利用QIIME 软件(version 1.8.0)生成不同分类水平上的物种丰度表,再利用R语言工具绘制成样品各分类学水平下的群落结构图。
1.3.4.4. Alpha多样性分析
Alpha多样性反映单个样品物种丰度及物种多样性。有多种衡量指标:Chao1、Ace、Shannon、Simpson等。Chao1和Ace指数衡量物种丰度即物种数量的多少。Shannon和Simpson指数用于衡量物种多样性,受样品群落中物种丰度和物种均匀度的影响。相同物种丰度的情况下,群落中各物种具有越大的均匀度,则认为群落具有越大的多样性,Shannon指数值越大,Simpson指数值越小,说明样品的物种多样性越高[16]。该研究中使用Mothur软件[17]对各个样品的Alpha多样性指数进行评估,比较组间的多样性指数。
1.3.4.5. 组间差异显著性分析
应用LEfSe软件[18]的线性判别分析方法(linear discriminant analysis, LDA)进行组间肠道菌群差异物种(Biomarker)筛选,取LDA>2.0 的菌种,为有统计学意义的差异菌种。
1.3.4.6. 相关性分析
采用RDA(redundancy analysis)/CCA(Canonical Correspondence analysis)[19]分析肠道菌群和环境因子(认知功能评分)之间的相关性。
1.3.5. 一般数据统计学分析
采用SPSS 24.0对临床指标进行统计学分析。计数资料采用χ²检验,计量资料使用Shapiro-Wilk检验(W检验)分析正态性,P>0.05表示正态分布,用F检验分析方差齐性,P>0.05表示方差相等,均满足正态分布且方差相等,采用
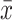 ±s表示,使用两独立样本t检验。P<0.05 为差异有统计学意义。
±s表示,使用两独立样本t检验。P<0.05 为差异有统计学意义。
2. 结果
2.1. 临床指标
PSCI组与对照组一般资料比较差异无统计学意义(P>0.05)。MMSE、MoCA评分PSCI组低于对照组,差异有统计学意义(P<0.001),见表1。
2.2. 肠道菌群结构特征分析结果
2.2.1. Alpha多样性分析
两组间肠道菌群Alpha多样性的Chao1、Ace、Shannon、Simpson指数比较差异均无统计学意义(P>0.05),表明组间肠道菌群Alpha多样性没有差异(表2)。
表 2. Alpha diversity comparison of the PSCI group and the HC group (
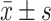 )
.
)
.
两组间Alpha多样性指数比较(
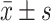 )
)
| Diversity index | HC group (n=23) | PSCI group (n=24) | t | P |
| HC: Healthy control; PSCI: Post-stroke cognitive impairment. | ||||
| Chao1 | 98.12±25.86 | 92.53±27.23 | 0.721 | 0.475 |
| Ace | 100.96±27.95 | 93.37±26.91 | 0.948 | 0.348 |
| Shannon | 3.44±1.10 | 3.34±1.20 | 0.305 | 0.762 |
| Simpson | 0.77±0.18 | 0.75±0.21 | 0.457 | 0.650 |
2.2.2. 肠道菌群构成及丰度对比分析
2.2.2.1. 门水平上的构成比较
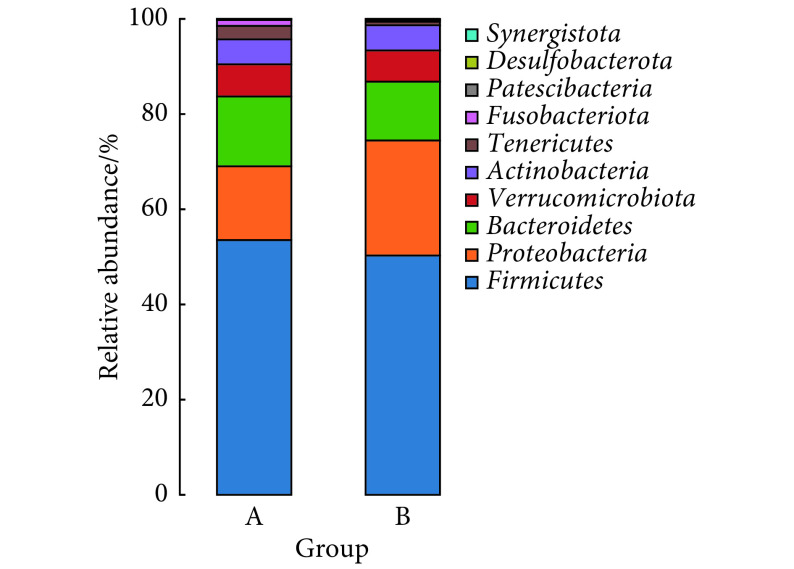
以菌门相对丰度百分比为纵坐标,组别为横坐标作门水平物种分布柱状图(图1)。两组均主要由厚壁菌门(Firmicutes)、变形菌门(Proteobacteria) 、拟杆菌门(Bacteroidetes)、疣微菌门(Verrucomicrobiota)、放线菌门(Actinobacteria)等构成,分别在对照组中依次占53.54%、15.49%、14.69%、6.76%、5.24%;在PSCI组中依次占50.28%、24.17%、12.37%、6.55%、5.31%。与对照组比较,PSCI组厚壁菌门、拟杆菌门有所减少,变形菌门有所增加,疣微菌门和放线菌门变化不明显。
图 1.
Bar chart of the relative abundance of the gut microbiota of the two groups at the phylum level (A: HC group; B: PSCI group)
两组肠道菌群门水平相对丰度分布柱状图(A:对照组;B:PSCI组)
2.2.2.2. 种水平上的构成比较
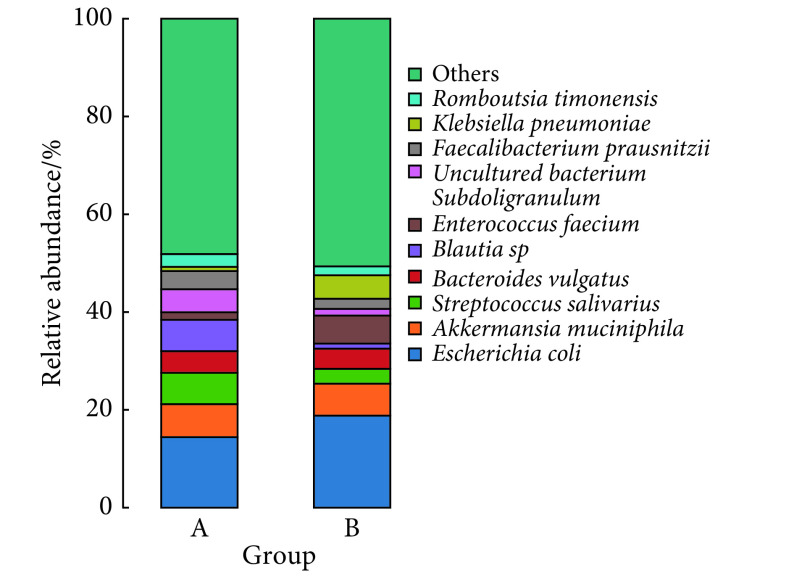
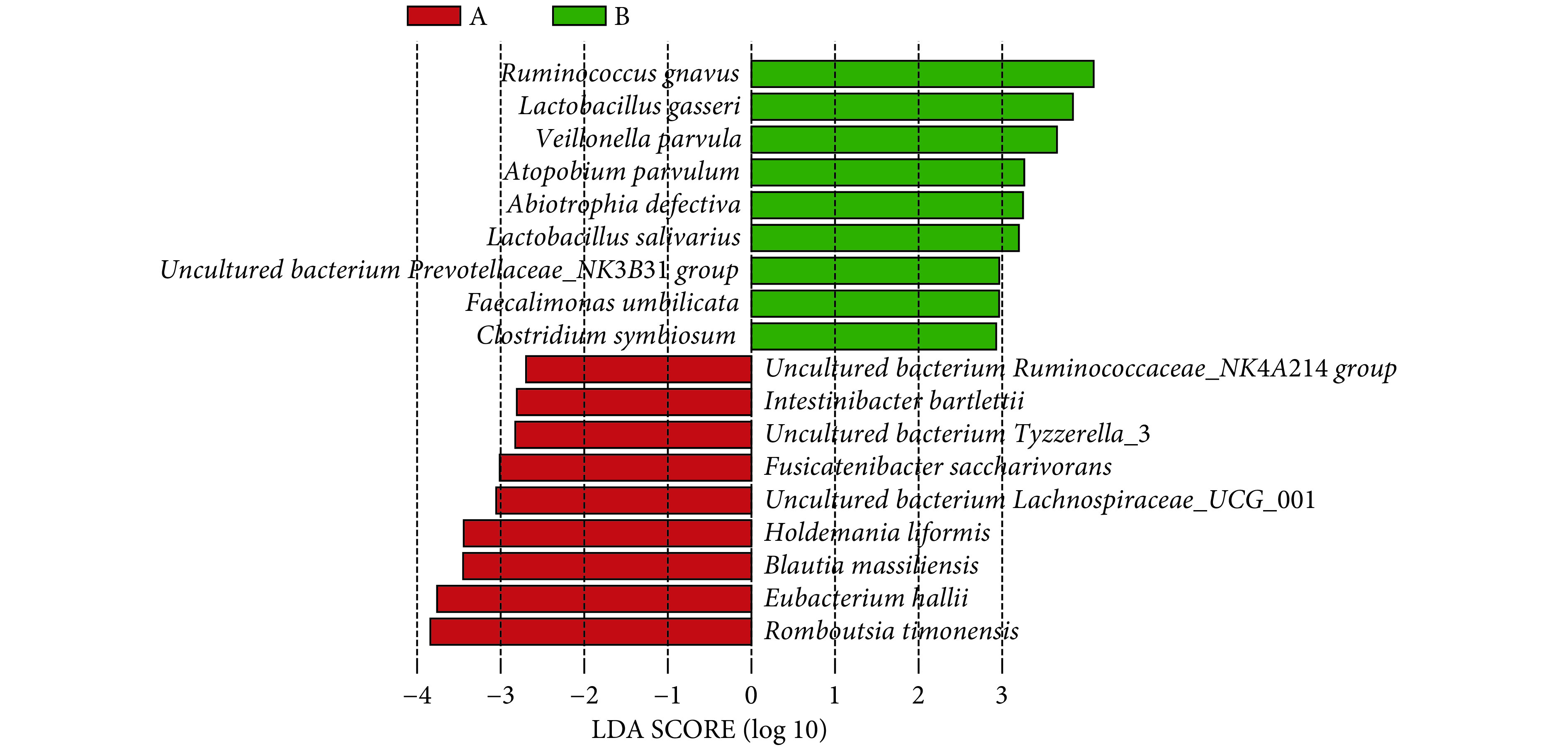
图2为以同样方法制作种水平物种分布的柱状图。图中显示的为两组相对丰度排列前10的菌种,在对照组和PSCI组中分别共占51.86%和49.36%。这些菌种的相对丰度各不相同,相比对照组,PSCI相对丰度改变较大的物种有:变形菌门的大肠杆菌(Escherichia coli);厚壁菌门的唾液链球菌(Streptococcussalivarius)、Blautia sp菌、屎肠球菌(Enterococcusfaecium)、uncultured bacterium Subdoligranulum菌、Faecalibacterium prausnitzii菌和变形菌门的肺炎克雷伯菌(Klebsiella pneumoniae),分别在对照组中依次占14.41%、6.39%、6.44%、1.52%、4.70%、3.72%、0.86%;在PSCI组中依次占18.82%、3.03%、1.04%、5.72%、1.37%、2.07%、4.80%。进一步使用LEfSe分析筛选组间相对丰度差异有统计学意义的菌种(图3和图4),结果显示PSCI组患者中丰度低于对照组的有9个菌种,均属于厚壁菌门(图3和图4中红色标注的菌种);PSCI组患者中丰度高于对照组的有9个菌种,7个属于厚壁菌门,放线菌门和拟杆菌门各含1个(图3和图4中绿色标注的菌种)。
图 2.
Bar chart of the relative abundance of the gut microbiota of the two groups at the species level (A: HC group; B: PSCI group)
两组肠道菌群种水平相对丰度分布柱状图(A:对照组;B:PSCI组)
图 3.
Excavation of microbial species with statistical differences at species level between HC group (A) and PSCI group (B)
对照组(A)和PSCI组(B)差异有统计学意义的微生物种类挖掘
Red indicates that the relative abundance in the HC group was higher than that in the PSCI group. Green indicates that relative abundance in PSCI group was higher than that in the HC group.
图 4.
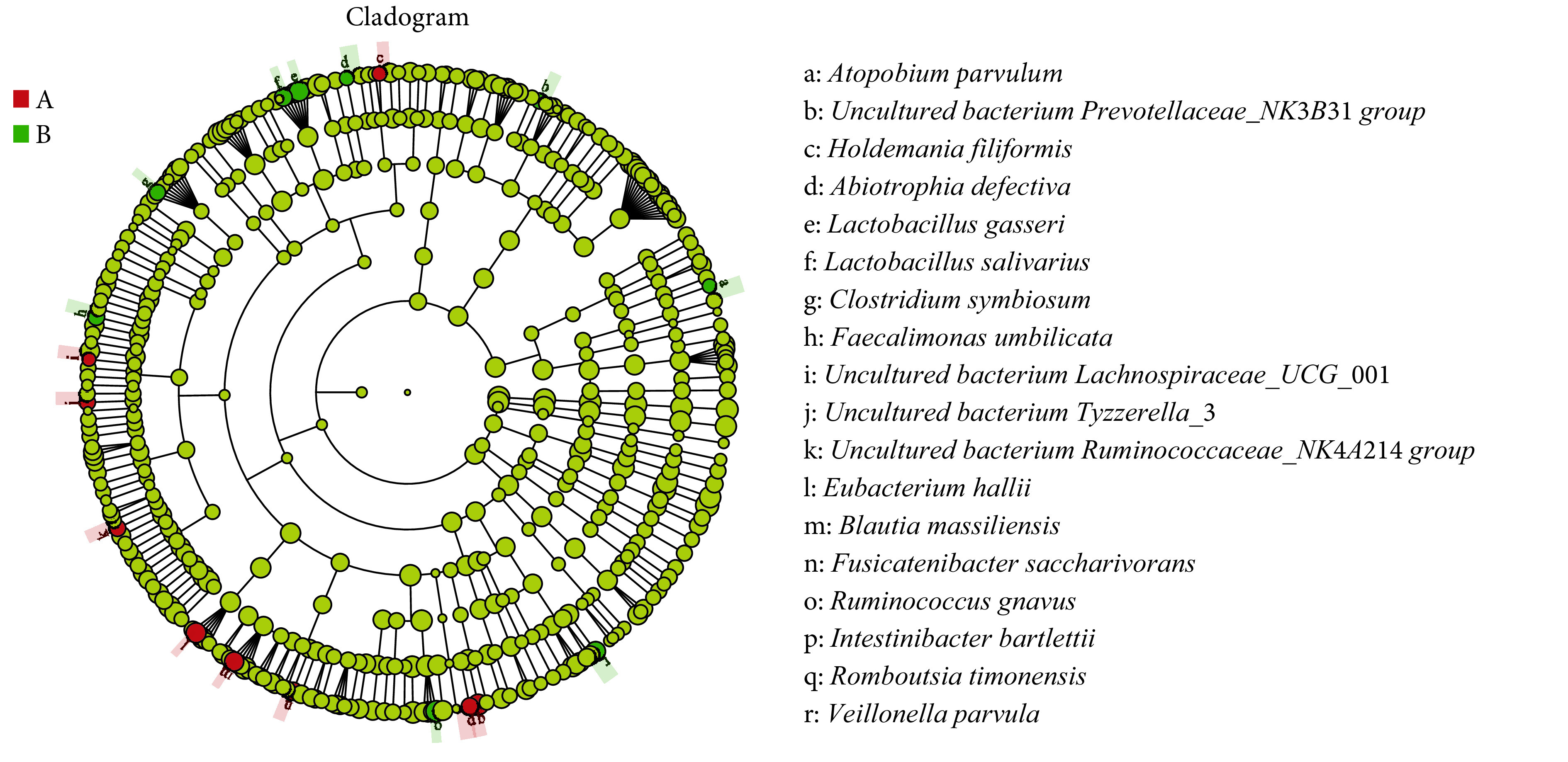
Evolutionary branching of LEfSe analysis at the species level
种水平LEfSe分析进化分枝图
A: HC group; B: PSCI group. The circles radiating from the inside to the outside represent the taxonomic levels from phylum to species. Each small circle at different taxonomic levels represents a taxon at that level, and the diameter of the circle is proportional to the relative abundance. Yellow nodes denote the species with no significant difference between the groups, red nodes denote the species with higher abundance in the HC group than that in the PSCI group, and green nodes denote the species with higher abundance in the PSCI group than that in the HC group.
2.2.3. 差异菌种与认知功能评分的相关性分析
采用RDA/CCA分析种水平差异菌种与认知评分(MMSE和MoCA)的相关性(图5)。 PSCI组中相对丰度低于对照组的菌种:厚壁菌门的Intestinibacter bartlettii菌、uncultured bacterium Tyzzerella_3菌、Fusicatenibactersaccharivorans菌与认知功能评分呈正相关,且这些菌种间呈正相关;另外厚壁菌门的马赛布劳特氏菌(Blautia massiliensis)与认知功能评分呈负相关。PSCI组中相对丰度高于对照组的菌种:厚壁菌门的活泼瘤胃球菌(Ruminococcus gnavus)、Faecalimonas umbilicata菌、拟杆菌门的uncultured bacterium Prevotellaceae_NK3B31 group菌与认知功能呈负相关,且这些菌种间呈正相关;另外厚壁菌门的格氏乳杆菌(Lactobacillusgasseri)、共生梭菌(Clostridium symbiosum)、小韦荣氏球菌(Veillonella parvula)和放线菌门的极小阿托波氏菌(Atopobium parvulum)与认知功能呈正相关,且这些菌种之间也呈正相关。
图 5.
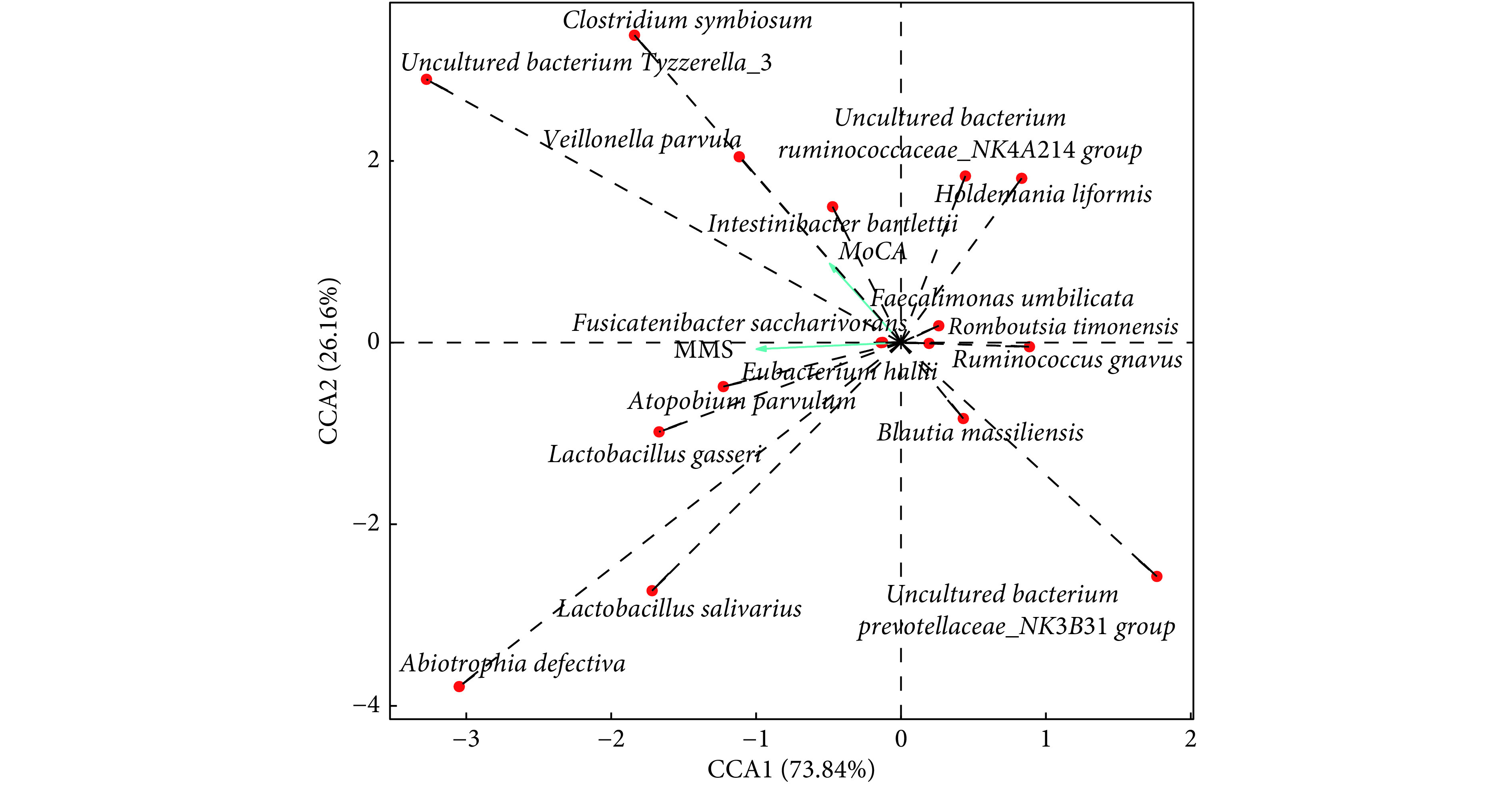
CCA correlation analysis of the gut microbiota and cognition scores at the species level
种水平肠道菌群和认知评分之间相关性CCA分析图
The scales on the horizontal and vertical coordinates are the values generated by the regression analysis of each sample or species with environmental factors. The red dots indicate species with statistical differences between the two groups, and the green arrows represent environmental factors (MoCA and MMSE). The relationship between rays is represented by an included Angle, obtuse representing a negative correlation and acute representing a positive correlation.
3. 讨论
肠道菌群是肠道微生态系统的重要组成部分,参与了代谢、消化、炎症及免疫等多项生理功能的调节[20]。正常生理状态下,肠道菌群处于平衡状态,对人体健康发挥着重要作用;当受到内外环境刺激时,如感染、应激、创伤、药物及理化因素等,平衡可被打破导致肠道菌群失调[21],从而引发各种疾病或加重病情。近年来,肠道菌群及其代谢物与中枢神经系统疾病的关系逐渐成为科学领域的研究热点,并提出肠道菌群与大脑相互作用的生理基础为“肠道菌群-肠-脑轴”[22],即肠道菌群可以通过迷走神经、内分泌、代谢及免疫等途径与中枢神经系统进行信息交流,从而影响大脑功能和行为[6-8];反之,大脑也可以通过神经、内分泌等多种途径影响肠道菌群[10,23]。在此基础上,越来越多的研究提示肠道菌群与脑卒中密切相关,提出肠道菌群不仅与脑卒中的危险因素如高血压、糖尿病、肥胖及动脉粥样硬化等密切相关[24-25],还直接影响着脑卒中的发生、发展及预后[10-11],被认为是脑卒中的一个新的危险因素。随着研究的不断深入,不少研究发现肠道菌群与认知功能密切相关[26]。LIU等[27]发现健康人群、遗忘型轻度认知功能障碍(amnestic mild cognitive impairment, aMCI)和阿尔茨海默病(Alzheimer’s disease, AD)患者的肠道菌群多样性及构成存在差异,其中肠杆菌科从正常人到痴呆逐渐增多,其丰度水平与AD患者认知评分具有相关性,可以用于区分健康人群、aMCI和AD。LEBLHUBER 等[28]通过随机双盲对照研究发现,补充益生菌有助于改善AD患者的认知功能。目前,肠道菌群与PSCI相关性的研究并不多见。2020年才首次报道PSCI患者与卒中后无认知障碍患者比较,肠道菌群多样性下降,菌群构成及相应代谢产物发生变化,且与认知功能障碍程度存在相关性,提示肠道菌群可能在PSCI的发生发展中起重要作用[12]。ZHU等[29]研究发现急性脑卒中患者肠道菌群代谢产物三甲胺-N-氧化物(trimethylamine N-oxide, TMAO)浓度越高,其1年后的MMSE评分越低,提示卒中后急性期血浆TMAO水平对PSCI有较好的预测性。另有研究认为肠道菌群紊乱及其代谢产物可能通过加重缺血性脑损伤[30],或参与动脉粥样硬化的发生发展[31],而缺血性脑损伤和动脉粥样硬化与PSCI密切相关,从而间接参与PSCI的发生、发展[32-33]。由于相关研究尚少,PSCI患者肠道菌群紊乱特征仍不确切,其中的机制和关联需要更多的深入研究。因此,本研究对PSCI患者和健康对照人群的肠道菌群结构、丰度、物种多样性进行对比分析,并探讨肠道菌群改变与PSCI患者认知功能障碍程度的相关性,以期从新的角度探索PSCI的发生机制,为脑卒中后认知功能障碍的防治提供新思路。研究结果发现PSCI患者与健康对照的肠道菌群Alpha多样性分析差异无统计学意义,但肠道菌群在门、种水平构成均存在差异,种水平上两组间具有标志性差异的菌种有18个,其中PSCI组中丰度低于对照组的有9个,高于对照组也有9个。相关分析显示差异菌种与认知功能评分存在一定的相关性。
本研究中Alpha多样性分析提示PSCI组与对照组间肠道菌群多样性并没有显著差异。尽管肠道菌群多样性降低在AD、糖尿病、帕金森病及肝癌等疾病人群中被观察到[27,34-37],但现阶段关于肠道菌群多样性在认知受损患者中变化趋势仍存在争议,如曾秀丽等[38]亦发现轻度认知功能障碍患者与健康对照组比较多样性无明显差异。肠道菌群结构、代谢活性和局部分布的改变都为肠道菌群失衡的范畴[21],由于PSCI患者及健康对照个体间肠道菌群构成及丰度存在明显的异质性,肠道菌群改变可能为某些致病菌增加,而有益菌减少,或者为代谢产物、菌群分布的改变,因此PSCI时其菌群多样性可能不是必然降低,且有研究报道某些疾病好转后其多样性反而降低[39],因此并不是多样性越高就一定代表越健康。
人体肠道菌群主要由厚壁菌门、拟杆菌门组成,其余为变形菌门、放线菌门等[40]。本研究通过对PSCI患者和健康对照的对比分析,在门水平上,两组的优势菌门均为厚壁菌门、变形菌门、拟杆菌门。与对照组比较,PSCI组厚壁菌门有所减少、变形菌门相对富集,这与在重性抑郁症及AD患者中观察到的结果基本一致[34,41]。LIU等[27]研究发现厚壁菌门减少与AD患者认知衰退进展有关;MARIAT等[42]指出厚壁菌门/拟杆菌门比值随着年龄增长而发生变化,从幼年到成年逐渐增大,从成年到老年又逐渐变小,亦提示厚壁菌门减少可能参与从成年到老年的衰老过程。变形菌门在人体的主要菌门中最不稳定[43],其在健康成人肠道中相对丰度较低,富集常被认为是一种肠道菌群紊乱的标志,可引发肠道炎症及慢性炎症免疫应激,具有潜在的疾病诊断价值[44]。与本研究结果类似,其他研究也发现认知损害患者肠道内的变形菌门的相对丰度增加[38,45]。因此,提示肠道菌群门水平结构与丰度的变化可能在PSCI的发病中起着一定的作用。
在种水平上,两组中变形菌门埃希氏菌属下的大肠杆菌丰度均处于最高水平,在对照组和PSCI组中分别共占14.41%和18.82%,而相对丰度排列前10的菌种有2个属于变形菌门,在对照组和PSCI组中分别共占15.27%和23.62%;有6个属于厚壁菌门,在对照组和PSCI组中分别共占25.39%和15.05%。剩余两个分别属于拟杆菌门和疣微菌门,在两组中的占比均较小且相差不大。由此可以看出,对照组优势菌门仍然为厚壁菌门,而PSCI组中厚壁菌门保持较高多样性,相对丰度却有下降,而变形菌门富集。与本研究结果类似,国内外也有研究发现认知损害患者肠道内的变形菌门下的大肠杆菌或志贺氏杆菌的相对丰度增加[37,45]。进一步显著性差异分析发现组间差异菌种有18个,其中PSCI组中相对丰度低于对照组的菌种有9个,均属于厚壁菌门;高于对照组的菌种也有9个,包含7个厚壁菌门的菌种和拟杆菌门、放线菌门各1个菌种,更加反映出PSCI患者与健康对照间肠道菌群的结构差异明显,尤其是厚壁菌门,其丰度和多样性的改变在PSCI发生发展中可能起着关键作用。
种水平差异菌种与认知功能评分的相关性分析发现,厚壁菌门的Intestinibacter bartlettii、unculturedbacterium Tyzzerella_3、Fusicatenibacter saccharivorans与认知功能评分呈正相关,这些细菌在PSCI组患者中的相对丰度均低于对照组,且细菌之间表现为正相关;另外厚壁菌门的Ruminococcus gnavus、Faecalimonasumbilicata、拟杆菌门的uncultured bacteriumPrevotellaceae_NK3B31 group与认知功能呈负相关,这些细菌在PSCI组患者中的相对丰度均高于对照组,且细菌之间表现为正相关。因此,结果提示厚壁菌门一些菌种的减少及一些菌种的增加与PSCI的发病具有紧密的关系,减少的菌种可能是卒中后认知功能的保护因子,而增加的菌种可能是卒中后认知功能的危险因子。另外,拟杆菌门的uncultured bacterium Prevotellaceae_NK3B31 group的增加可能也参与卒中后认知功能的损害。与临床指标相反的是,相关性分析还发现PSCI组中相对丰度低于对照组的菌种——厚壁菌门的Blautiamassiliensis与认知功能评分呈负相关;PSCI组中相对丰度高于对照组的菌种——厚壁菌门的Lactobacillus gasseri、Clostridium symbiosum、Veillonella parvula、放线菌门的Atopobium parvulum与认知功能呈正相关。由于目前相关的研究尚少,与PSCI患者认知功能密切相关的菌种及具体的相关性仍不明确。本研究前面的结果中已发现PSCI患者中厚壁菌门减少,这与之前的在PSCI及AD患者中观察到的结果一致[27,34,42] 。厚壁菌门在人体肠道菌群中占主导地位,即使相对丰度减少,其仍能维持较高的多样性,如文中前面的结果提示。因此,在种水平表现为厚壁菌门某些菌种相对丰度的增加和某些菌种相对丰度的减少,可能是它们的综合作用另加上少数其他菌门下菌种的变化共同参与卒中后认知功能的损害过程。因此,虽然这些相关性看似与临床矛盾,但尚不能完全否认,也不能确定这些相关性在PSCI发病中的作用大小,并给我们提示,PSCI患者肠道菌群紊乱涉及到门、科、属、种等多个水平及多菌类的改变,远远不是单一菌类单一趋势丰度水平发生变化的结果。人体肠道是人体最大和最复杂的微生态系统,单一菌类对疾病的作用是有限的,多水平多菌类丰度的增加或减少以及菌类间的相互作用综合可能才是导致疾病发生发展的原因。总的来说,研究中发现厚壁菌门的Intestinibacter bartlettii、unculturedbacterium Tyzzerella_3、Fusicatenibacter saccharivorans的减少,和厚壁菌门的Ruminococcus gnavus、Faecalimonas umbilicata、拟杆菌门的unculturedbacterium Prevotellaceae_NK3B31 group增加,有可能参与PSCI的发生发展,有望成为PSCI早期筛查和干预的靶点。
综上所述,本研究表明,与健康对照比较,PSCI患者肠道菌群结构特征在门和种水平构成存在差异,主要表现在厚壁菌门的减少和变形菌门的富集,并且组间差异菌种与认知功能存在相关性,有望成为PSCI早期筛查和干预的靶点。尽管本研究存在一定的局限性,如样本量偏小、无干预前后对比等,但是相关的研究工作可为今后PSCI患者肠道菌群的研究和诊治提供一定的参考。下一步的研究中将在现有证据的基础上,科学合理地开展PSCI与肠道菌群的人群或动物实验研究,进一步验证和分析对PSCI发生、发展及预后具有关键影响作用的菌株和代谢产物,为PSCI的早期筛查、康复干预和判断预后提供新的视角。
* * *
利益冲突 所有作者均声明不存在利益冲突
Funding Statement
四川省医学科研课题计划(No. S19025)资助
Contributor Information
荣建 冯 (Rong-jian FENG), Email: 408203364@qq.com.
亚梅 李 (Ya-mei LI), Email: liyamei@med.uestc.edu.cn.
References
- 1.汪凯, 董强, 郁金泰, 等 卒中后认知障碍管理专家共识2021. 中国卒中杂志. 2021;16(4):376–389. doi: 10.3969/j.issn.1673-5765.2021.04.011. [DOI] [Google Scholar]
- 2.QU Y, ZHUO L, LI N, et al Prevalence of post-stroke cognitive impairment in china: A community-based, cross-sectional study. PLoS One. 2015;10(4):e0122864[2021-10-10]. https://doi.org/10.1371/journal.pone.0122864. doi: 10.1371/journal.pone.0122864. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.ROHDE D, GAYNOR E, LARGE M, et al The impact of cognitive impairment on poststroke outcomes: A 5-year follow-up. J Geriatr Psychiatry Neurol. 2019;32(5):275–281. doi: 10.1177/0891988719853044. [DOI] [PubMed] [Google Scholar]
- 4.中国卒中学会, 卒中后认知障碍管理专家委员会 卒中后认知障碍管理专家共识. 中国卒中杂志. 2017;12(6):519–531. doi: 10.3969/j.issn.1673-5765.2017.06.011. [DOI] [Google Scholar]
- 5.BORDET R, IHL R, KORCZYN A D, et al Towards the concept of disease-modifier in post-stroke or vascular cognitive impairment: A consensus report. BMC Med. 2017;15(1):107. doi: 10.1186/s12916-017-0869-6. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.GIAU V V, WU S Y, JAMERLAN A, et al Gut microbiota and their neuroinflammatory implications in Alzheimer’s disease. Nutrients. 2018;10(11):1765. doi: 10.3390/nu10111765. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.WANG Y, KASPER L H The role of microbiome in central nervous system disorders. Brain Behav Immun. 2014;38:1–12. doi: 10.1016/j.bbi.2013.12.015. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.CRUMEYROLLE-ARIAS M, JAGLIN M, BRUNEAU A, et al Absence of the gut microbiota enhances anxiety-like behavior and neuroendocrine response to acute stress in rats. Psychoneuroendocrinology. 2014;42:207–217. doi: 10.1016/j.psyneuen.2014.01.014. [DOI] [PubMed] [Google Scholar]
- 9.LUCZYNSKI P, MCVEY NEUFELD K A, ORIACH C S, et al. Growing up in a bubble: Using germ-free animals to assess the influence of the gut microbiota on brain and behavior. Int J Neuropsychopharmacol, 2016, 19(8): pyw020[2021-10-10]. https://doi.org/10.1093/ijnp/pyw020.
- 10.SINGH V, ROTH S, LLOVERA G, et al Microbiota dysbiosis controls the neuroinflammatory response after stroke. J Neurosci. 2016;36(28):7428–7440. doi: 10.1523/JNEUROSCI.1114-16.2016. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.DURGAN D J, LEE J, MCCULLOUGH L D, et al Examining the role of the microbiota-gut-brain axis in stroke. Stroke. 2019;50(8):2270–2277. doi: 10.1161/STROKEAHA.119.025140. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.LIU Y, KONG C, GONG L, et al The association of post-stroke cognitive impairment and gut microbiota and its corresponding metabolites. J Alzheimers Dis. 2020;73(4):1455–1466. doi: 10.3233/JAD-191066. [DOI] [PubMed] [Google Scholar]
- 13.中华神经内科学会, 中华神经外科学会 各类脑血管疾病的诊断要点. 中华神经科杂志. 1996;29(6):379–380. doi: 10.3760/j.issn:1006-7876.1996.06.006. [DOI] [Google Scholar]
- 14.NASREDDINE Z S, PHILLIPS N A, BÉDIRIAN V, et al The montreal cognitive assessment, MoCA: A brief screening tool for mild cognitive impairment. J Am Geriatr Soc. 2005;53(4):695–699. doi: 10.1111/j.1532-5415.2005.53221.x. [DOI] [PubMed] [Google Scholar]
- 15.EDGAR R C, HAAS B J, CLEMENTE J C, et al UCHIME improves sensitivity and speed of chimera detection. Bioinformatics. 2011;27(16):2194–2200. doi: 10.1093/bioinformatics/btr381. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.EDGAR R C UPARSE: Highly accurate OTU sequences from microbial amplicon reads. Nat Methods. 2013;10(10):996–998. doi: 10.1038/nmeth.2604. [DOI] [PubMed] [Google Scholar]
- 17.SCHLOSS P D, WESTCOTT S L, RYABIN T, et al Introducing mothur: Open-source, platform-independent, community-supported software for describing and comparing microbial communities. Appl Environ Microbiol. 2009;75(23):7537–7541. doi: 10.1128/AEM.01541-09. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.SEGATA N, IZARD J, WALDRON L, et al Metagenomic biomarker discovery and explanation. Genome Biol. 2011;12(6):R60. doi: 10.1186/gb-2011-12-6-r60. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.LINDSTRÖM E S, KAMST-VAN AGTERVELD M P, ZWART G Distribution of typical freshwater bacterial groups is associated with pH, temperature, and lake water retention time. Appl Environ Microbiol. 2005;71(12):8201–8206. doi: 10.1128/AEM.71.12.8201-8206.2005. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.ROMANO K A, VIVAS E I, AMADOR-NOGUEZ D, et al Intestinal microbiota composition modulates choline bioavailability from diet and accumulation of the proatherogenic metabolite trimethylamine-N-oxide. mBio. 2015;6(2):e02481[2021-10-10]. https://doi.org/10.1128/mBio.02481-14. doi: 10.1128/mBio.02481-14. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.李兰娟. 感染微生态学. 北京: 人民卫生出版社, 2012.
- 22.COLLINS S M, SURETTE M, BERCIK P The interplay between the intestinal microbiota and the brain. Nat Rev Microbiol. 2012;10(11):735–742. doi: 10.1038/nrmicro2876. [DOI] [PubMed] [Google Scholar]
- 23.CARABOTTI M, SCIROCCO A, MASELLI M A, et al The gut-brain axis: Interactions between enteric microbiota, central and enteric nervous systems. Ann Gastroenterol. 2015;28(2):203–209. [PMC free article] [PubMed] [Google Scholar]
- 24.PEVSNER-FISCHER M, BLACHER E, TATIROVSKY E, et al The gut microbiome and hypertension. Curr Opin Nephrol Hypertens. 2017;26(1):1–8. doi: 10.1097/MNH.0000000000000293. [DOI] [PubMed] [Google Scholar]
- 25.BAOTHMAN O A, ZAMZAMI M A, TAHER I, et al The role of gut microbiota in the development of obesity and diabetes. Lipids Health Dis. 2016;15:108. doi: 10.1186/s12944-016-0278-4. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.SMITH L K, WISSEL E F Microbes and the mind: How bacteria shape affect, neurological processes, cognition, social relationships, development, and pathology. Perspect Psychol Sci. 2019;14(3):397–418. doi: 10.1177/1745691618809379. [DOI] [PubMed] [Google Scholar]
- 27.LIU P, WU L, PENG G, et al Altered microbiomes distinguish Alzheimer’s disease from amnestic mild cognitive impairment and health in a Chinese cohort. Brain Behav Immun. 2019;80:633–643. doi: 10.1016/j.bbi.2019.05.008. [DOI] [PubMed] [Google Scholar]
- 28.LEBLHUBER F, EGGER M, SCHUETZ B, et al Commentary: Effect of probiotic supplementation on cognitive function and metabolic status in Alzheimer’s disease: A randomized, double-blind and controlled trial. Front Aging Neurosci. 2018;10:54[2021-10-09]. https://doi.org/10.3389/fnagi.2018.00054. doi: 10.3389/fnagi.2018.00054. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.ZHU C, LI G, LV Z, et al Association of plasma trimethylamine-N-oxide levels with post-stroke cognitive impairment: a 1-year longitudinal study. Neurol Sci. 2020;41(1):57–63. doi: 10.1007/s10072-019-04040-w. [DOI] [PubMed] [Google Scholar]
- 30.HOULDEN A, GOLDRICK M, BROUGH D, et al Brain injury induces specific changes in the caecal microbiota of mice via altered autonomic activity and mucoprotein production. Brain Behav Immun. 2016;57:10–20. doi: 10.1016/j.bbi.2016.04.003. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 31.BRANDSMA E, KLOOSTERHUIS N J, KOSTER M, et al A proinflammatory gut microbiota increases systemic inflammation and accelerates atherosclerosis. Circ Res. 2019;124(1):94–100. doi: 10.1161/CIRCRESAHA.118.313234. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 32.GORELICK P B, SCUTERI A, BLACK S E, et al Vascular contributions to cognitive impairment and dementia: A statement for healthcare professionals from the american heart association/american stroke association. Stroke. 2011;42(9):2672–2713. doi: 10.1161/STR.0b013e3182299496. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 33.PANTONI L Cerebral small vessel disease: From pathogenesis and clinical characteristics to therapeutic challenges. Lancet Neurol. 2010;9(7):689–701. doi: 10.1016/S1474-4422(10)70104-6. [DOI] [PubMed] [Google Scholar]
- 34.ANGELUCCI F, CECHOVA K, AMLEROVA J, et al Antibiotics, gut microbiota, and Alzheimer’s disease. J Neuroinflammation. 2019;16(1):108. doi: 10.1186/s12974-019-1494-4. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 35.TILG H, MOSCHEN A R Microbiota and diabetes: An evolving relationship. Gut. 2014;63(9):1513–1521. doi: 10.1136/gutjnl-2014-306928. [DOI] [PubMed] [Google Scholar]
- 36.REN Z, LI A, JIANG J, et al Gut microbiome analysis as a tool towards targeted non-invasive biomarkers for early hepatocellular carcinoma. Gut. 2019;68(6):1014–1023. doi: 10.1136/gutjnl-2017-315084. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 37.PETROV V A, SALTYKOVA I V, ZHUKOVA I A, et al Analysis of gut microbiota in patients with Parkinson’s disease. Bull Exp Biol Med. 2017;162(6):734–737. doi: 10.1007/s10517-017-3700-7. [DOI] [PubMed] [Google Scholar]
- 38.曾秀丽, 谭楚红, 吴齐恒, 等 轻度认知功能障碍患者的肠道菌群特征. 中国神经精神疾病杂志. 2019;45(3):129–134. doi: 10.3969/j.issn.1002-0152.2019.03.001. [DOI] [Google Scholar]
- 39.ZHANG Y, BOBE G, REVEL J S, et al Improvements in metabolic syndrome by xanthohumol derivatives are linked to altered gut microbiota and bile acid metabolism. Mol Nutr Food Res. 2020;64(1):e1900789[2021-10-10]. https://doi.org/10.1002/mnfr.201900789. doi: 10.1002/mnfr.201900789. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 40.HARRIS K, KASSIS A, MAJOR G, et al Is the gut microbiota a new factor contributing to obesity and its metabolic disorders? J Obes. 2012;2012:879151[2021-10-10]. https://doi.org/10.1155/2012/879151. doi: 10.1155/2012/879151. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 41.JIANG H, LING Z, ZHANG Y, et al Altered fecal microbiota composition in patients with major depressive disorder. Brain Behav Immun. 2015;48:186–194. doi: 10.1016/j.bbi.2015.03.016. [DOI] [PubMed] [Google Scholar]
- 42.MARIAT D, FIRMESSE O, LEVENEZ F, et al The Firmicutes/Bacteroidetes ratio of the human microbiota changes with age. BMC Microbiol. 2009;9:123. doi: 10.1186/1471-2180-9-123. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 43.FAITH J J, GURUGE J L, CHARBONNEAU M, et al The long-term stability of the human gut microbiota. Science. 2013;341(6141):1237439. doi: 10.1126/science.1237439. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 44.SHIN N R, WHON T W, BAE J W Proteobacteria: Microbial signature of dysbiosis in gut microbiota. Trends Biotechnol. 2015;33(9):496–503. doi: 10.1016/j.tibtech.2015.06.011. [DOI] [PubMed] [Google Scholar]
- 45.CATTANEO A, CATTANE N, GALLUZZI S, et al Association of brain amyloidosis with pro-inflammatory gut bacterial taxa and peripheral inflammation markers in cognitively impaired elderly. Neurobiol Aging. 2017;49:60–68. doi: 10.1016/j.neurobiolaging.2016.08.019. [DOI] [PubMed] [Google Scholar]