Abstract
目的
使用无监督的方式进行图像分割,作为人工标记的一种替代。
方法
选取了共100张HE染色和巴氏染色切片的全片数字化图像(whole slide image, WSI)数据作为研究和测试的对象,其中乳腺切片70张,肺切片20张,甲状腺切片10张。为了保证数据的多样性,乳腺的切片包含了正常组织、炎症、肿瘤,肺切片取材主要为下叶新生物(包含了炎症和肿瘤),甲状腺为细针穿刺的细胞(均为良性)。每张图像的最大总倍率(原始倍率)均为400倍,文件格式为ndpi。对每张WSI进行人工的标注,每张WSI的标注区域都大于10个视野,标注后的信息将用于有效性的验证。使用基于超像素与全卷积神经网络的算法构建无监督图像分割技术,对没有标记的WSI的任意感兴趣区域(regions of interest, ROI)进行图像分割。与区域邻接图合并的方法进行比较,以欠分割错误差率、边缘召回率和平均交并结果比判定两种方法的分割效果,并比较两种方法的效率。在执行效率的比较中,测试过程包含了超像素的预处理的时间,去掉了加载深度学习引擎的时间。
结果
对WSI任意ROI区域按纹理和颜色对图像实现了无监督的自动分割,乳腺切片、肺切片和甲状腺切片测试的结果差异小,多次测试的结果稳定,但该方法在对炎症和肿瘤的区分中表现一般。其欠分割错误差率、边缘召回率和平均交并结果分别为19.10%、82.06%和45.06%。区域邻接图合并的方法的欠分割错误差率、边缘召回率和平均交并的结果分别为21.52%、78.39%和44.81%。在GPU模式下整个过程平均耗时为0.27 s,在CPU模式下平均耗时为1.30 s,由于区域邻接图合并的方法没有实现GPU模式,在CPU模式下平均耗时为10.5 s。
结论
本方法通过简单的人机交互操作得到理想的像素级标注结果,可以有效降低数字病理切片数据标注的成本,比区域邻接图合并的方法在处理图像纹理的方面表现得更好,处理速度更快。
Keywords: 超像素, 全片数字化图像, 卷积神经网络
Abstract
Objective
To segment images through an unsupervised method as an alternative to manual labeling.
Methods
A total of 100 whole slide image (WSI) data of HE stained and Pap stained slides were selected as the research and test objects, including 70 breast slides, 20 lung slides and 10 thyroid slides. In order to ensure the diversity of data, the breast slides included those of normal tissue, inflammation and tumor, the lung slides were mainly neoplasms in the lower lobe, including those of inflammation and tumor, and the thyroid slides were of cells, all benign, obtained through fine needle aspiration. The maximum total magnification (original magnification) of each image was 400 times, and the file format was NDPI. Each WSI was manually labeled, and the labeled area of each WSI was more than 10 fields of vision. The labeled information was to be used for validity verification. An unsupervised image segmentation technique based on superpixel and fully convolution neural network algorithms was constructed and used to segment any region of interest (ROI) of unlabeled WSI. In comparison with the region adjacency graph merging method, the segmentation effect of the two methods was assessed with the under segmentation error, the boundary recall and the mean Intersection-over-Union, and the efficiency of the two methods was also compared. In the comparison of execution efficiency, the test process included the preprocessing time of superpixel, and excluded the time of loading the deep learning engine.
Results
Unsupervised automatic segmentation was implemented for any ROI region of WSI according to the texture and color. The results of the breast slides, lung slides and thyroid slides showed slight differences, and multiple tests yielded stable results. However, the performance of this method in differentiating inflammation and tumor was average. The under-segmentation error, the boundary recall and the mean Intersection-over-Union were 19.10%, 82.06% and 45.06%, respectively. The under segmentation error, the boundary recall and the mean Intersection-over-Union for the region adjacency graph merging method were 21.52%, 78.39% and 44.81%, respectively. The average time consumption of the whole process was 0.27 s in GPU mode and 1.30 s in CPU mode. The average time consumption of the region adjacency graph merging method was 10.5 s in CPU mode because the method of region adjacency graph merging was not realized in the GPU mode.
Conclusion
This method produced ideal pixel level labeling results through simple human-computer interaction, which could effectively reduce the cost of digital pathology slide data labeling. Compared with the region adjacency graph merging method, this method had better performance in processing image texture and had faster processing speed.
Keywords: Superpixel, Whole slide images, Convolutional neural network
数字图像在现代医学中的应用越来越广泛,全片数字化图像(whole slide image, WSI)已经应用于病理学各个领域[1],医学图像分割作为处理特征匹配、结构分析、运动分析、三维重建等相关任务的基础,同时还可帮助医生区分每个细节,因此在医学图像处理中起关键作用。处理图像分割,如果已经有了特定的大型训练数据集,那么使用有监督的分割方法来获得准确的结果,是一种快速且十分有效的做法,例如使用全卷积网络(fully convolutional networks, FCN)[2]的有监督模型来预测结果。然而,创建训练数据集需要专家手动来描述标签,目前在WSI中常见的标注方式以边界框(如矩形框、圆形框等)标注和多边形标注为主。边界框标注最为简单,但是得到的信息都过于简单,且标注精度偏低,容易选取多余的信息,为后续的处理带来不便。而使用多边形标注的方式,虽然能够得到比较高的精度,但是在操作过程中对人为错误非常敏感,所以非常费时。随着图像的数量和种类的不断增加,再加上WSI中有太多的不规则纹理、色差、外形等,这也进一步让标注训练集数据成为了一项耗时且繁重的工作。相比之下,无监督分割的好处是不需要训练数据集,对于没有见过的数据也可以有效的分割。虽然无监督分割不能分割出全部的类型,但是探索创建一种在数字病理图像中使用的无监督图像分割技术来代替大量费时耗力的人工标注方法是非常有价值的。
在医学图像分割中常见的经典的无监督图像分割有基于k-means的分割、基于分水岭的分割、基于高斯混合模型[3]的分割、基于隐马尔可夫随机场模型[4]和隐马尔可夫树[5]的无监督图像分割等。K-means[6]的分割,实现比较简单,但k值选取困难,通常需要手动去选取;基于分水岭算法的分割常用于灰度图像的分割,会出现过分割现象;基于高斯混合模型的分割精度比较高,但效率较低;而基于隐马尔可夫随机场模型和隐马尔可夫树的无监督图像分割,在一些细节分割上有非常不错的效果,精度比较高,但是在图像边缘的信息挖掘效果不是很好,容易产生方块效应,且效率也不高。为了解决这些问题,本研究根据病理图像的多分辨率、亮度均匀等特点,提出了一种无监督的分割方法对WSI可见区域或者感兴趣区域(regions of interest, ROI)进行分割,该方法先使用了简单的非迭代聚类(simple non-iterative clustering, SNIC)[7]算法进行快速超像素的计算对图线进行预处理,然后将超像素输入到卷积神经网络(convolutional neural network, CNN)[8]中进行区域合并,最后得到分割结果。报道如下。
1. 对象与方法
1.1. 对象
我们选取了共100张HE染色和巴氏染色切片的WSI数据作为研究和测试的对象,其中乳腺切片70张,肺切片20张,甲状腺切片10张。为了保证数据的多样性,乳腺的切片包含了正常组织、炎症、肿瘤,肺取材主要为下叶新生物,包含了炎症和肿瘤,甲状腺为细针穿刺的细胞,均为良性。这些图像均由滨松XR-C12000-02数字切片扫描仪扫描获得,每张图像的最大总倍率(原始倍率)均为400倍,文件格式为ndpi。我们对每张WSI进行了人工的标注,每张WSI的标注区域都大于10个视野,标注后的信息将用于有效性的验证。
1.2. 计算超像素
超像素是一种强大的图像预处理工具,它可以有效消除伪影和噪声,有良好的聚类效果,降低图像分割时的语义冲突。目前常见生成超像素的算法主要分为基于图和基于聚类的方式。这两种类型中,熵率超像素(entropy rate superpixel, ERS)[9]和简单线性迭代聚类(simple linear iterative clustering, SLIC)[10]是分别基于图和基于聚类的经典算法的代表,由于基于图的方法效率比较低,本文选用的是基于聚类的SNIC算法,它是SLIC的一种改进算法,无需多次迭代,内存占用更少,边界与图像贴合更好。
SNIC对像素的颜色相似性和空间邻近性来进行聚类,将图像转到CIELAB颜色空间,并将颜色向量和位置向量结合构成5维空间向量[l, a,b, x,y],接下来需要设置超像素数量K和超像素颜色紧凑度Z,K值用来计算的超像素空间距离的归一化因子S,计算公式:S=(N/K)1/2,其中N表示图像的像素数。使用Z值用来计算超像素颜色距离的归一化因子M,计算公式:M=1/Z。因为WSI有多种不同的倍率,在不同倍率下选择K值和Z值也会有所不同,我们借鉴了BEJNORDI等[11]提出的概念,但区别在于本文将200倍以下的倍率归为一类,200倍以上的倍率归为另一类,第一种K值选择6 000~8 000这个范围,Z的选值为10;第二种K值选择8 000~10 000这个范围,Z的选值为16。
SNIC和SLIC最大的区别在于,SNIC不用迭代去完成超像素的分割,而是使用优先队列在(1)式中求平均更新超像素的中心,并标记标签,计算的范围是相邻的4或8个像素,其中s和m分别表示超像素空间距离和颜色距离的归一化因子,X代表当前像素坐标值,C代表当前像素的颜色值,Dj,k表示从k像素到j像素空间距离和颜色距离的归一化因子的结合。
 |
1 |
1.3. 训练卷积神经网络进行区域合并
超像素计算多用于对图像像素距离和图像颜色这两个特征进行聚类分割,对图像纹理的聚类效果不佳,为了解决这个问题,可以使用CNN来进一步的对图像的纹理进行感知,得到更加精确的分割结果[12]。本文借鉴了KANEZAKI[13]的方法构建了CNN模型,该模型以全连接的形式以任意大小的图像为输入,同时映射之前超像素输出的标签进行学习,最后会得到一个与输入的图像有相同空间维度的输出。如图1,并根据GALLOWAY等[14]的描述,调整了归一化层(batch normalization)激活函数ReLU层的顺序,将批归一化放在激活函数ReLU的前面,使得输入到ReLU的图片接近正态分布N(0,1),让激活函数的非线性特性得到更好的发挥,其构造出来的损失函数会变得更加光滑。模型中增加了Dropout层,设置的丢弃率为0.5,用于防止过拟合。在第二次卷积的激活层后增加了一层最大池化层(Max_pool2d),用于增大卷积层内核的感受野,降低计算复杂度,让模型更加稳定。
图 1.
Convolutional neural network model used for training
用于训练的卷积神经网络模型
本研究选择Adam优化器来训练模型,学习率设置为0.05。训练的迭代次数为64次,使用GPU进行加速训练,损失函数选用交叉熵。
1.4. 方法的结果判定
为了验证本文提出方法的有效性,我们使用欠分割错误差(under-segmentation error, UE)、边缘召回率(boundary recall, BR)[15]和平均交并比mean Intersection-over-Union(mIoU)[16]进行评估,它们的定义如式(2)~(4)所示:
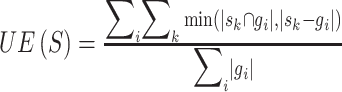 |
2 |
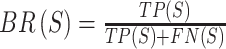 |
3 |
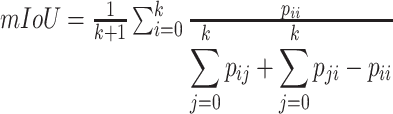 |
4 |
UE主要用来测量算法在分割图像的时候产生的错误,与算法比较的是已知的精确分割的结果,(2)式中,g表示已知的精确分割像素集,s表示超像素分割的像素集,误差值越小越好。BR用来计算落入精确分割边界的超像素的比例,(3)式中,TP(S)表示在精确分割像素边界内落入的超像素边界内的像素数量,FN(S)的情况正好和TP(S)相反,值越大越好。mIoU是一种图像分割的经典指标,其值越大越好,(4)式中,pii、pij、pji分别表示真阳、假阳、假阴的数据,k表示类别个数。
1.5. 方法效率评测
最后的比较测试中,选择应用场景一致的区域邻接图(region adjacency graph, RAG)[17]算法进行比较。测试过程中,统一使用SNIC超像素算法作为预处理。在执行效率的比较中,纳入计算预处理的时间,去掉加载深度学习引擎的时间。
2. 结果
2.1. 算法指标评测结果
对乳腺切片、肺切片和甲状腺切片的数据,本文的方法的UE平均为19.10%,BR平均为82.06%,mIoU平均为45.06%。在测试中,乳腺切片、肺切片和甲状腺切片测试的结果差异都不大,并且经过多次测试结果保持稳定,但该方法在对炎症和肿瘤的区分中表现的一般。如图2。
图 2.

Experimental group of superpixel segmentation, merging and selection
超像素分割、合并、选取的实验图组
A is the original image, from left to right are images with three magnification rates of 12.5, 200, and 400 respectively; B is the superpixel obtained after calculatiion of image A, and is expressed in yellow color; C is the final segmentation result obtained based on RAG; D is the final segmentation result obtained by our proposed method. The magnification of B, C and D is 200 times on the left and 400 times on the right.
2.2. 算法执行对比测试结果
本文的算法,在GPU模式下整个过程平均耗时为0.27 s,在CPU的模式下平均耗时为1.30 s。由于RAG的方法没有实现GPU模式,在CPU模式下其执行效率平均为10.5 s。在对RAG做指标性测试中,它的UE为21.52%,BR为78.39%,mIoU为44.81%,对甲状腺切片的表现结果不稳定。
3. 讨论
本研究发现,作为对照的RAG算法对WSI进行聚类后,存在RAG的区域合并,染色淡的纹理未被区分,且最后的聚类不太理想的情况,主要的原因是RAG通过图像的颜色和距离这两项特征进行聚类,对图像纹理处理不佳。在执行效率上,虽然没有实现RAG算法的GPU模式,但在CPU模式下,本文的算法也比RAG算法快了7倍。另外,本文对CNN的结构进行了改进,增加了Dropout层和最大池化层,调整了归一化和激活函数的顺序,提高了训练速度和模型稳定性,特别是在训练速度上,比原有的网络快了将近3倍。
本文提出的方法,可以在WSI上实现像素级的快速标注,充分利用分割后的标签数据转换为列表,通过简单的人机交互,便可以快速选取列表中的一个或者多个对象,在选取的过程中,通过高亮提示来确定选取的位置,最后为这些选取的对象添加新的特征,完成标注工作,这样就有效的避免了使用多边形工具去手工勾画复杂外形、花费大量时间的问题,也避免了使用矩形框或者圆形框工具去选取时少选或多选、造成标注对象不准确的问题。通过实验证明,此方法在定义的ROI区域或者可见区域内,对于WSI这种支持多种分辨率的图像进行有效的分割是可行的,精度、效率佳,但仍有一定的不足。比如,需要人工设置分割数量,尚未完全做到全自动化,分割速度有延时(达不到“实时”水平),对于区分肿瘤和炎症,表现一般,不过这也在预料之中——对于分类问题,有监督学习是更好的选择。成功的分割有助于后续更高层次的图像识别和理解,利用无监督且与任务无关的方式对图像进行分割,是对大多数有监督学习的一种补充,可以改善有监督且与任务有关方法的性能,加快其计算速度,这一点在SCHUURMANS等[18]的文章中得到较好阐述。
近几年随着硬件设备和人工智能算法的快速发展,很多项目的需求都转向人工智能,以降低对人的依赖,或者减轻人的劳动强度——不管具体出于什么目的,这些需求都要用到大量的数据作为支撑,才能保证应用的正常运行。随着新的医疗设备的不断升级和普及,获得数据的质量和数量也不断提升,而医疗环境格局的限制与老龄化人口的日益增加,加重了医生和相关技术人员的相对短缺,整个医疗行业对人工智能的需求变得非常急迫,同时也带动了医疗数据产业的发展。大部分的数据对于人工智能的算法来说无法直接使用,于是催生大量数据清洗和数据标注[19]的需求。这为数据标注行业带来巨大的市场,同时也带来挑战。挑战主要表现在两个方面,一个是数据安全控制的问题,另一个是数据标注的质量与数量互斥的问题。这些问题同样会出现在医疗领域。对于第一个挑战,可以通过数据脱敏、区块链[20]等技术手段,从预防和反向追溯来确保数据的安全性问题,同时通过人员角色的划分,让角色之间相互制约,避免权限过大造成数据安全问题的发生。对于第二个挑战,可以利用无监督自动分割等技术,让机器自动分割图像,减少人工手动标注的过程,同时将自动切割算法集成到现有的一些标注工具中,提高标注效率;另外这些算法要保证较高的精度,并且具有快速处理数据的能力和稳定性、泛化能力,这些都需要控制在一个可接受的范围内,让标注出来的数据能够真正符合需求。另外有人提出了一种解决标注数据耗时费力的方法,可以将主任务的算法变得不再需要更多训练数据,直接使用原有数据也能达到一个理想的结果,例如CAMPANELLA等[21]提出的基于弱监督深度学习的临床级计算病理学就是这样的思路,但是目前这种方法是建立在需要使用大量计算资源的基础上,并没有实际解决降低经济成本的问题,目前这种方法也只有少数资金充足的机构有能力去实施。
未来,随着医学图像处理和研究中人工智能应用的增加,数据和数据标注的需求会越来越大,图像分割技术在其处理的精度、速度等方面都会有更高的要求,无监督自动分割技术作为图像分割中的一个重要分支,值得进一步研究。
* * *
利益冲突 所有作者均声明不存在利益冲突
Funding Statement
科技部重点研发计划重点专项(No. 2017YFC0113908),北京精鉴病理发展基金会(No. 2019-0007),四川省国际科技合作与交流研发项目(No. 2017HH0070、No.2018HH0037),成都市新型产业技术研究院技术创新项目(No. 2017-CY02-00026-GX),四川大学华西医院临床研究孵化项目(No. 2020HXFH029)和四川大学华西医院学科卓越发展1·3·5 工程项目(No. ZYGD18012)资助
Contributor Information
航宇 秦 (Hang-yu QIN), Email: acproject@qq.com.
骥 包 (Ji BAO), Email: baoji@scu.edu.cn.
References
- 1.叶美华, 盛弘强, 王怡栋, 等 数字病理切片系统可视化数据应用简介. 中华病理学杂志. 2012;41(1):66–68. doi: 10.3760/cma.j.issn.0529-5807.2012.01.020. [DOI] [Google Scholar]
- 2.LONG J, SHELHAMER E, DARRELL T. Fully convolutional networks for semantic segmentation. arXiv, 2015: 1411.4038[2020-11-03]. https://arxiv.org/abs/1411.4038v1.
- 3.向日华, 王润生 一种基于高斯混合模型的距离图像分割算法. 软件学报. 2003;14(7):1250–1257. [Google Scholar]
- 4.SHIRAZI M N, NODA H. A deterministic iterative algorithm for HMRF-textured image segmentation//Proceedings of 1993 International Conference on Neural Networks (IJCNN-93-Nagoya, Japan). Nagoya, Japan: IEEE, 1993: 2189-2194.
- 5.侯彪, 徐婧, 刘凤, 等 基于第二代Bandelet域隐马尔可夫树模型的图像分割. 自动化学报. 2009;35(5):498–504. [Google Scholar]
- 6.霍迎秋, 秦仁波, 邢彩燕, 等 基于CUDA的并行K-means聚类图像分割算法优化. 农业机械学报. 2014;45(11):47–53. doi: 10.6041/j.issn.1000-1298.2014.11.008. [DOI] [Google Scholar]
- 7.ACHANTA R, SUSSTRUNK S. Superpixels and polygons using simple non-iterative clustering//2017 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Honolulu, USA: IEEE, 2017: 4895–4904.
- 8.HADJI I, WILDES R P. What do we understand about convolutional networks? arXiv, 2018: 1803.08834[2020-11-03]. https://arxiv.org/abs/1803.08834.
- 9.TANG Y, ZHAO L, REN L. Different versions of entropy rate superpixel segmentation for hyperspectral image//2019 IEEE 4th International Conference on Signal and Image Processing (ICSIP). Wuxi,China: IEEE, 2019:1050-1054.
- 10.ACHANTA R, SHAJI A, SMITH K, et al SLIC superpixels compared to state-of-the-art superpixel methods. IEEE Transactions Pattern Analysis Machine Intelligence. 2012;34(11):2274–2282. doi: 10.1109/TPAMI.2012.120. [DOI] [PubMed] [Google Scholar]
- 11.BEJNORDI B E, LITJENS G, HERMSEN M, et al. A multi-scale superpixel classification approach to the detection of regions of interest in whole slide histopathology images//Proceedings Volume 9420, Medical Imaging 2015: Digital Pathology. Orlando, USA: SPIE Medical Imaging, 2015: 94200H.
- 12.CIMPOI M, MAJI S, KOKKINOS I, et al. Deep filter banks for texture recognition, description, and segmentation. arXiv, 2015: 1507.02620[2020-11-03]. https://arxiv.org/abs/1507.02620.
- 13.KANEZAKI A. Unsupervised image segmentation by backpropagation//2018 IEEE International Conference on Acoustics, Speech and Signal Processing (ICASSP). Calgary, Canada: IEEE, 2018: 1543–1547.
- 14.GALLOWAY A, GOLUBEVA A, TANAY T, et al. Batch normalization is a cause of adversarial vulnerability. arXiv, 2019: 1905.02161[2020-11-03]. https://arxiv.org/abs/1905.02161.
- 15.罗学刚, 吕俊瑞, 彭真明 超像素分割及评价的最新研究进展. 激光与光电子学进展. 2019;56(9):53-63. [Google Scholar]
- 16.GHORBANZADEH O, BLASCHKE T, GHOLAMNIA K, et al Evaluation of different machine learning methods and deep-learning convolutional neural networks for landslide detection. Remote Sens. 2019;11(2):196[2020-11-03]. https://doi.org/10.3390/rs11020196. [Google Scholar]
- 17.TREMEAU A, COLANTONI P Regions adjacency graph applied to color image segmentation. IEEE Transactions Image Processing. 2000;9(4):735–744. doi: 10.1109/83.841950. [DOI] [PubMed] [Google Scholar]
- 18.SCHUURMANS M, BERMAN M, BLASCHKO M B. Efficient semantic image segmentation with superpixel pooling. arXiv, 2018: 1806.02705[2020-11-03]. https://arxiv.org/abs/1806.02705.
- 19.蔡莉, 王淑婷, 刘俊晖, 等 数据标注研究综述. 软件学报. 2020;31(2):302–320. [Google Scholar]
- 20.薛腾飞, 傅群超, 王枞, 等 基于区块链的医疗数据共享模型研究. 自动化学报. 2017;43(9):1555–1562. [Google Scholar]
- 21.CAMPANELLA G, HANNA M G, GENESLAW L, et al Clinical-grade computational pathology using weakly supervised deep learning on whole slide images: 8. Nat Med. 2019;25(8):1301–1309. doi: 10.1038/s41591-019-0508-1. [DOI] [PMC free article] [PubMed] [Google Scholar]



