Abstract
目的
探讨关键干细胞特性基因在卵巢癌的诊断和治疗中的意义。
方法
加权基因共表达网络分析(WGCNA)方法筛选出关键模块和基因;基因集富集分析(GSEA)和单细胞测序数据分析关键基因上调组高表达的信号通路;pRRophetic预测卵巢癌的化疗药物的敏感性;流式细胞术检测SKOV3细胞和肿瘤干细胞中CD44及CD117的表达情况;实时荧光定量(qRT)-PCR证实关键基因在卵巢癌干细胞中的表达;GeneMANIA分析鉴定出核心基因。
结果
WGCNA结果显示在转录水平上筛选出15个关键基因,其在多种肿瘤中高表达,参与细胞周期、DNA修复、E2靶点和G2M检查点通路,且与化疗药物敏感性有显著的关联性。CD44+CD117+细胞在SKOV3细胞和卵巢癌干细胞中的比例分别为(1.20±0.34)%、(37.17±1.80)%,差异有统计学意义(P<0.05)。qRT-PCR证实WGCNA结果中7个关键基因(BUB1、CDC20、CCNB2、DLGAP5、KIF4A、NEK2、NUSAP1)在卵巢癌干细胞中高表达,并且BUB1可能起着核心的作用。
结论
本研究通过构建基因共表达网络筛选出7个干细胞特性关键基因,尤其是BUB1,可能成为潜在的卵巢癌基因生物标志物。
Keywords: 卵巢癌, 肿瘤干细胞, 关键基因, 加权基因共表达网络分析(WGCNA)
Abstract
Objective
To investigate the significance of stemness-related genes in the diagnosis and treatment of ovarian cancer.
Methods
Key modules and genes were identified with weighted gene co-expression network analysis (WGCNA). The signal pathways of high expression of key genes were analyzed by gene set enrichment analysis (GSEA) and single cell sequencing data. The chemosensitivity of ovarian cancer to chemotherapy drugs was estimated with pRRophetic. Flow cytometry was used to examine the expression of CD44+CD117+in SKOV3 cells and cancer stem cells. The expression of key genes in ovarian cancer stem cells was confirmed by qRT-PCR. The core genes were identified by GeneMANIA analysis.
Results
According to the WGCNA results, 15 key genes were identified at the transcription level, all being highly expressed in many kinds of tumors. They were involved in the cell cycle, DNA repair, E2 target and G2M checkpoint pathway, and had significant correlation with chemosensitivity. The proportion of CD44+ CD117+ cells in SKOV3 cells and ovarian cancer stem cells were (1.20±0.34)% and (37.17±1.80)% respectively, with statistically significant difference (P<0.05). qRT-PCR confirmed that seven key genes (BUB1, CDC20, CCNB2, DLGAP5, KIF4A, NEK2, NUSAP1) in the WGCNA results were highly expressed in ovarian cancer stem cells, and BUB1 might have played a core role.
Conclusion
Seven hub genes, especially BUB1, were identified by constructing gene co-expression network, which may become potential biomarkers of ovarian cancer gene.
Keywords: Ovarian cancer, Cancer stem cell, Hub genes, Weighted gene co-expression network analysis
卵巢癌是最致命的妇科癌症,2018年全球发病295 414例,死亡184 799例[1]。其发病隐匿,复发率高。虽然治疗上有很大进步,但患者的预后仍不理想,其5年生存率不足50%[2]。卵巢癌的发生是多种基因及其产物相互作用的结果,其早期诊断和治疗依旧是个难题。目前已有应用单细胞测序来研究癌症基因组图谱(the cancer genome atlas,TCGA)肿瘤的免疫细胞类型和免疫系统中的干细胞[3],并根据现有的分子靶点和机制确定特定的药物,以开发有助于肿瘤细胞分化和降低高度侵袭性肿瘤的干细胞潜能的创新疗法[3]。加权基因共表达网络分析(weighted gene co-expression network analysis, WGCNA)是一种系统生物学方法,它基于基因变量之间的相关性,构建基因互相作用网络体系,能探究整体生物学过程的变化,同时识别几百个致病基因和治疗靶点[4]。目前,有很多关于运用WGCNA研究各种肿瘤的潜在靶标的报道,但是,未见针对卵巢癌干性的WGCNA。本研究以数据库中的卵巢癌数据构建共表达网络,明确与卵巢癌干性相关的枢纽基因,以期寻找诊断和治疗卵巢癌的潜在生物标志物,并通过Oncomine、富集分析、药物敏感性及细胞实验进行验证。
1. 方法和材料
1.1. 数据下载与处理
从基因表达综合数据库(gene expression omnibus,GEO)下载卵巢癌基因数据集GSE5396(n=174),GSE140082(n=277)( http://www.ncbi.nlm.nih.gov/geo/)。原始数据用R包affy和Bioconductor进行处理和注释( http://www.bioconductor.org)。从TCGA数据库下载RNA测序数据( http://cancergenome.nih.gov/dataportal/data/access),由234个卵巢癌样本组成。R软件包DESeq2对mRNA测序数据进行标准化处理。
1.2. WGCNA鉴定关键模块和基因
WGCNA根据基因集与表型的内在联系,通过识别和加权基因对的连接来构建基因网络[5]。筛选出前19 754个按SD值排序的基因,构建共表达网络,识别与干性指数相关的基因模块。WGCNA是分析相邻系数(
 )的强大工具,反映每对节点之间的连接强度,如下所示:
)的强大工具,反映每对节点之间的连接强度,如下所示:
 |
假设xi和xj代表基因i和j的表达值的向量,
 是两个载体的Pearson相关系数;
是两个载体的Pearson相关系数;
 是
是
 的指数变换,计算基因i和j之间网络连接强度。软阈值β设为3(scale-free R2=0.95),建立一个无标度网络。相关性绝对值较高的基因在共表达网络中属于同一个模块。利用WGCNA方法研究两个连接节点之间的关系及其他相关基因。拓扑重叠度量(topological overlap measure,TOM)显示如下:
的指数变换,计算基因i和j之间网络连接强度。软阈值β设为3(scale-free R2=0.95),建立一个无标度网络。相关性绝对值较高的基因在共表达网络中属于同一个模块。利用WGCNA方法研究两个连接节点之间的关系及其他相关基因。拓扑重叠度量(topological overlap measure,TOM)显示如下:
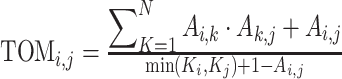 |
TOM是指基因i和j[6]相邻基因的重叠,并利用加权系数矩阵的层次聚类来寻找模块。N : 基因和基因直接相连的节点的邻接系数乘积之和的数量。K : Ki代表基因i单独连接的节点的邻接系数之和,为共同邻接系数的相关性。A:symmetric adjacency matrix A =
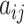 。
。
mRNAsi表示卵巢癌样本关键基因干性指数。通过WGCNA计算module eigengenes(MEs)与mRNAsi的相关性,筛选出显著相关的模块。MEs是每个模块的第一个主要组成部分,是指定模块中所有基因的代表。应用gene significance(GS)评估基因与mRNAsi的相关性,是基因表达与mRNAsi之间线性回归中P值的对数转换(GS=lgP)。模块显著性(MS)相当于模块内的平均基因显著性。在选择与mRNAsi相关的模块后,计算每个关键基因的模块身份(module membership)及其阈值。设置显著性(GS)>0.55和模块身份(MM)>0.8鉴定关键基因。
1.3. 关键基因的验证及功能富集分析
Oncomine评估肿瘤组织和正常组织关键基因的mRNA表达水平( http://www.oncomine.org)。为验证关键基因在蛋白质水平上的作用,采用人类蛋白质图谱对关键基因进行免疫组化研究并下载( http://www.proteinatlas.org)关键基因的免疫组化图片,比较差异。基于KEGG和HALLMARK数据库,利用fgsea包进行gene set enrichment analysis (GSEA)。从CancerSEA下载单细胞RNA测序图谱GSE85534和癌症相关通路的标记物( http://biocc.hrbmu.edu.cn/CancerSEA/)。
1.4. 治疗预测
卵巢癌的主要治疗药物选自GDSC库(the genomics of drug sensitivity in cancer)。R包“pRRophetic”和Wilcoxon检验估计卵巢癌标本的半数最大抑制浓度(IC50)。10倍交叉验证法对预测精度进行评价。使用“combat”和“all Sold Tumors”组织类型消除批次效应后,对所有参数和重复基因表达执行的默认值汇总为中间值。此外,connectivity map(CMap)是一个通过基因表达特征将疾病与小分子药物联系起来的数据库,用于预测潜在的小分子抑制剂和mechanism of actions(MoA)。每种药物都是通过大量的细胞系来概括的。为评价基因图谱的相似性,将基因表达谱数据集分为正调控基因群和负调控基因群,输出连接性分数。预测平均关联指数<−0.2的小分子抑制剂可能为高干性指数卵巢癌的候选药物。
1.5. 细胞株及主要试剂
人卵巢癌细胞株SKOV-3(中国科学院上海细胞库),DMEM培养基(Gibco,美国),RMPI-1640培养基,CD117、CD44抗体(CST,美国),SYBR Premix Ex TaqⅡ荧光染料试剂盒(TaKaRa,日本),人重组胰岛素(Peprotech,美国),B-27(Gibco,美国),肝素钠(Sigma,美国),重组人表皮生长因子(EGF,Peprotech,美国),重组人碱性成纤维细胞生长因(bFGF,Peprotech,美国),总RNA提取试剂盒(TIANGEN,北京),逆转录试剂盒(TaKaRa,日本),SYBR Premix Ex TaqⅡ荧光染料试剂盒(TaKaRa,日本),Real-time PCR引物由成都擎科生物公司合成。
1.6. 肿瘤干细胞(cancer stem cell, CSC)的无血清培养
SKOV-3细胞株的培养基为含有10%胎牛血清的RMPI-1640培养基。CSC为无血清的DMEM/F12培养基中含葡萄糖(6 mg/mL)、胰岛素(2 μg/mL)、B27(与培养基体积比1∶50)、肝素钠(4 μg/mL)、EGF(20 ng/mL)、bFGF(20 ng/mL)。取对数生长期的SKOV-3细胞,胰酶消化5 min,用培养基终止消化,离心后用无血清培养基重悬,密度调整至2×104mL-1培养。为防止细胞贴壁,每隔2 h摇晃1次。隔天更换无血清培养基。每隔7~10 d收集悬浮的克隆球细胞,经离心、轻柔吹打分散、洗涤后,于无血清培养基中连续扩增。
1.7. 流式细胞检测肿瘤干性标志物
收集卵巢癌干细胞和正常肿瘤细胞,用胰酶消化获得单细胞悬液,PBS洗涤细胞2次,调整细胞密度为1×106mL-1。取单细胞悬液100 μL离心去上清,加入PBS稀释的FITC-CD44抗体(1∶500)和PE-CD117抗体(1∶500)100 μL标志细胞,每种荧光抗体设同型对照,室温染色30 min,离心弃上清,加入300 μL PBS,上机检测肿瘤干性标志物CD44和CD117染色阳性率。
1.8. 采用实时荧光定量(qRT)-PCR法检测关键基因表达
引物序列见表1。用Trizol提取对数生长期的肿瘤细胞和悬浮细胞球的mRNA,并按PrimeScript RT Kit说明书操作,逆转录得到cDNA。各取1 µg的cDNA,按照20 µL的反应体系进行PCR扩增,内参为GAPDH,反应条件:98 ℃ 3 min预变性,98 ℃ 10 s,56 ℃ 30 s,72 ℃延伸60 s,总反应40个循环,72 ℃最终延伸10 min。每个样品设置3个复孔,Bio-Rad CFX Manager 3.1测样本的Ct值,并用2−∆∆Ct来表示目的RNA的相对表达量。
表 1. Primer sequence in PCR.
PCR引物序列
| Gene | Sense | Anti-sense |
| AURKB: Aurora kinase B; BUB1: BUB1 mitotic checkpoint serine/threonine kinase; CDC20: Cell division cycle 20; CCNB2:Cyclin B2; CDCA5: Cell division cycle associated 5; CDK1: Cyclin dependent kinase 1; DLGAP5: DLG associated protein 5; EXO1: Exonuclease 1; KIF4A: Kinesin family member 4A; KIF2C: Kinesin family member 2C; NDC80: Nuclear division cycle 80; NEK2: NIMA related kinase 2; NCAPH: Non-SMC condensing Ⅰ complex subunit H; NUSAP1: Nucleolar and spindle associated protein 1; ORC1: Origin recognition complex subunit 1; GAPDH: Glyceraldehyde-3-phosphate dehydrogenase. | ||
| AURKB | 5′-CTGCACCATCCCAACATCCT-3′ | 5′-TGCCAACTCCTCCATGATCG-3′ |
| BUB1 | 5′-TAAACCCACAGGAGCCAGGAC-3′ | 5′-CAAGCCTCAACGCCCAACT-3′ |
| CDC20 | 5′-ATGCGCCAGAGGGTTATCAG-3′ | 5′-AGGATGTCACCAGAGCTTGC-3′ |
| CCNB2 | 5′-CACAGGATACACAGAGAATG-3′ | 5′-CTTGATGGCGATGAATTTAG-3′ |
| CDCA5 | 5′-GGCCAGAGACTTGGAAATGT-3′ | 5′-GGCCAGAGACTTGGAAATGT-3′ |
| CDK1 | 5′-CCCTTTAGCGCGGATCTACC-3′ | 5′-CATGGCTACCACTTGACCTGT-3′ |
| DLGAP5 | 5′-AAGTGGGTCGTTATAGACCTGA-3′ | 5′-TGCTCGAACATCACTCTCGTTAT-3′ |
| EXO1 | 5′-TCTGAGAGGTAGTTAATTTGG-3′ | 5′-TACATCCATCAAATACGAGA-3′ |
| KIF4A | 5′-TCTGTTTCAGGCTGCTTTCA-3′ | 5′-GCCCTGAAATATTTGATTGGAG-3′ |
| KIF2C | 5′-GATGGAAGCCTGCTCTAACG-3′ | 5′-GAGCAGATTCCGCTTTGTTC-3′ |
| NDC80 | 5′-CCTCTCCATGCAGGAGTTAAGA-3′ | 5′-GGTCTCGGGTCCTTGATTTTCT-3′ |
| NEK2 | 5′-TGCTTCGTGAACTGAAACATCC-3′ | 5′-CCAGAGTCAACTGAGTCATCACT-3′ |
| NCAPH | 5′-GTCCTCGAAGACTTTCCTCAGA-3′ | 5′-TGAAATGTCAATACTCCTGCTGG-3′ |
| NUSAP1 | 5′-CTGTGCTTGGGACACAAA-3′ | 5′-TTGTCAACTTGAATGGGGTAATAA-3′ |
| ORC1 | 5′-ACGCAATGAAGTCTCTGGAATGTG-3′ | 5′-ACGGGCACGAAGAGAAGTATGGA-3′ |
| GAPDH | 5′-CATTCAAGACCGGACAGAGG-3' | 5′-ACATACTCAGCACCAGCATCACC-3′ |
1.9. GeneMANIA分析
GeneMANIA( http://www.genemania.org)提供了一个灵活的界面,用于根据基因功能推导假设[7]。GeneMANIA生成一个与查询基因功能相似的基因列表,并构建一个交互式功能关联网络来说明基因和数据集之间的关系。本研究利用该数据库,从物理相互作用、共表达、预测、共定位和遗传相互作用等方面构建了蛋白的基因-基因相互作用网络。
1.10. 统计学方法
用GraphPad Prism8统计软件进行处理,数值以
 表示。两独立样本的比较采用非配对t检验,P<0.05为差异有统计学意义。
表示。两独立样本的比较采用非配对t检验,P<0.05为差异有统计学意义。
2. 结果
2.1. 加权共表达网络的构建及关键模块的识别
采用组平均聚类法对234个样本聚类,并删除8个异常值。通过建立无标度网络筛选出与mRNAsi高度相关的卵巢癌基因模块,并确定加权共表达网络的尺度独立性和平均连接性,选择软阈值为3(无尺度R2=0.95),最终确定模块(图1A~1B,非聚类模块为灰色)。一些基因模块与mRNAsi呈相对负相关,即棕色、品红和绿松石模块(图1C),黄色模块与mRNAsi最为相关(图1C),为关键模块。10个模块之间的关系如图1D~1E所示。
图 1.
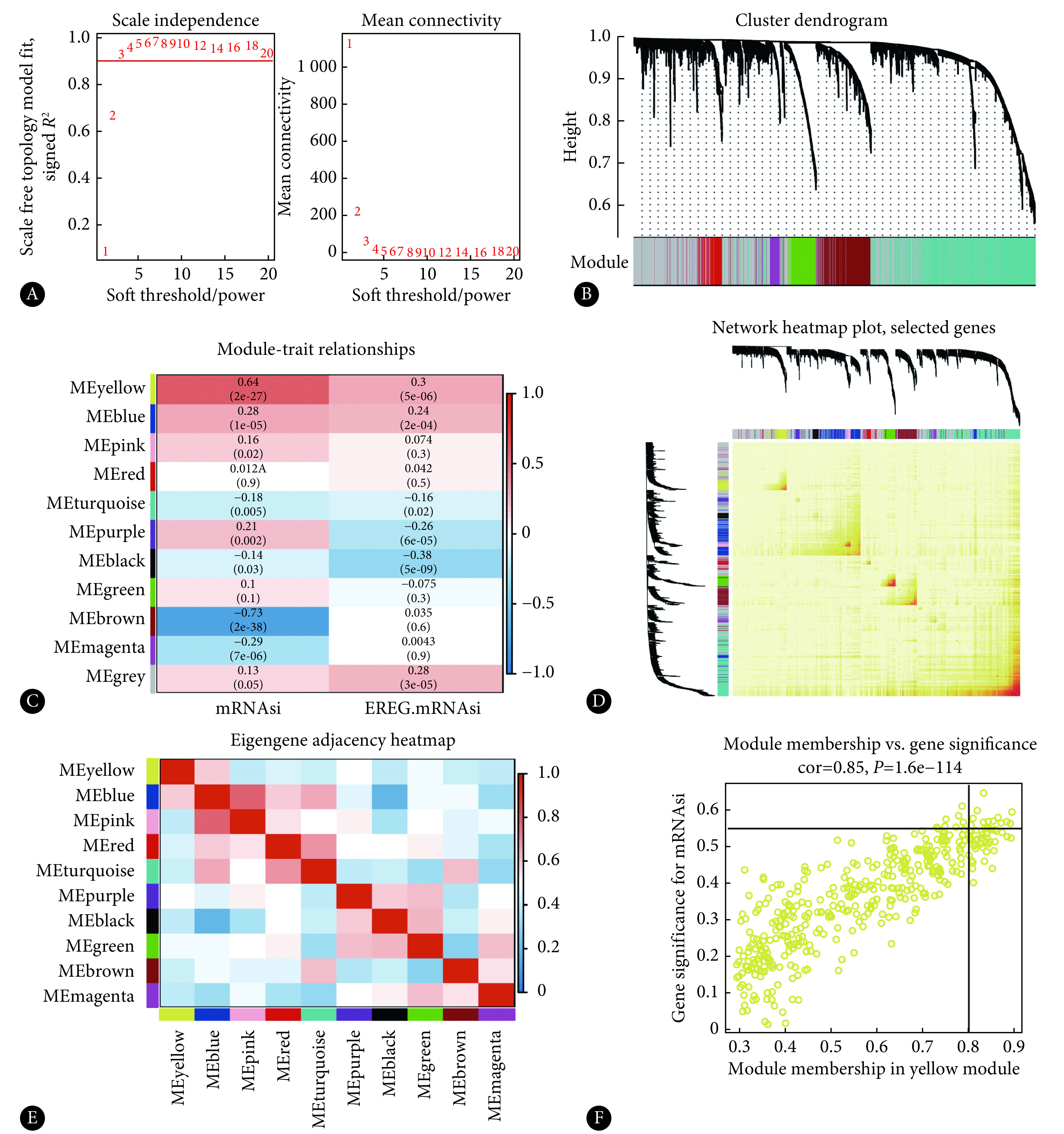
Construction of weighted co-expression modules and identification of significant modules
加权共表达网络的构建及关键模块的识别
A: Analysis of the scale independence and the mean connectivity for different soft-thresholding powers (scale-free R2=0.95); B: Identification of a co-expression module in ovarian cancer and the branches of the cluster dendrogram; C: Heatmap of the correlation between module eigengenes and the mRNAsi of ovarian cancer samples; D: Interaction relationship analysis of the most variable genes and modules they belong to (the brightness of yellow in the middle shows the degree of connectivity of different genes); E: Heatmap plot of the relationship analysis of modules (the color of the grid represents lgPvalue, which means the overlapping degree of modules); F: Scatter plot of module eigengenes in the yellow module.
2.2. 关键基因表达的分析与验证
模块身份(MM)>0.8和基因显著性>0.55为mRNAsi组选择关键基因的阈值。在黄色模型中筛选了15个关键基因(图1F),包括aurora kinase B(AURKB)、mitotic checkpoint serine/threonine kinase(BUB1)、cell division cycle 20(CDC20)、cyclin B2(CCNB2)、cell division cycle associated 5(CDCA5)、cyclin dependent kinase 1(CDK1)、DLG associated protein 5(DLGAP5)、exonuclease 1(EXO1)、kinesin family member 4A(KIF4A)、kinesin family member 2C(KIF2C)、nuclear division cycle 80(NDC80)、NIMA related kinase 2(NEK2)、non-SMC condensin Ⅰ complex subunit H(NCAPH)、nucleolar and spindle associated protein 1(NUSAP1)和origin recognition complex subunit 1(ORC1)。其表达水平在肿瘤分级中有显著差异(图2A)。这些基因在Oncomine数据库中的卵巢癌和其他癌症中过度表达(图2B)。
图 2.

Validation of the expression of hub genes
关键基因的表达验证
A: The trend of mRNAsi (mRNA expression-based stemness index) in different stages of ovarian cancer; B: The mRNA expression patterns of hub genes in overall cancers. The number in the colored cell represents the number of analyses meeting threshold. The color shade was determined by the gene ranks (%). The red cells indicate that the mRNA levels of target genes are higher in tumor tissues than in normal tissues, while blue cells indicate that the mRNA levels of target genes are lower in tumor tissues than in normal tissues.
2.3. 关键基因功能富集分析
为阐明关键基因的生物学功能,对其进行基因集富集分析(gene set enrichment analysis,GSEA)。在基于KEGG和HALLMARK数据库的信号通路,关键基因高表达组中,细胞周期、EF2靶点、G2M检查点和DNA复制的信号通路表达显著提高(图3A)。为评价关键基因在肿瘤细胞中的作用,采用单细胞RNA测序数据集分析关键基因与一些重要的细胞信号传导途径的相关性。4种信号通路(细胞周期、干性、增殖和DNA修复)与关键基因的表达显著相关(图3B)。由此推测,这些关键基因可能与卵巢癌的生长和干性有关。
图 3.

Functional enrichment analysis of hub genes
关键基因的功能富集分析
A: Gene set enrichment analysis (GSEA) based on HALLMARK and KEGG database (NES: Normalized enrichment score; Pval: Pvalue; Padj: P-adjusted); B: Single-cell RNA sequencing dataset showed that the relative expression of hub genes was related to the expression of some critical cell signaling pathways based on the GEO database.
2.4. 关键基因相关治疗策略的评价
通过最大的公开药物基因组学数据库,即癌症药物敏感性基因组学(GDSC),预测了所有样本的化疗反应。将每个患者样本关键基因GSVA分析的干性指数排序,以50%为分界线,分为关键基因高表达组(HIGH)和低表达组(LOW),然后用R包“pRRophetic”和Wilcoxon检验在3个独立数据集中估计卵巢癌样本中HIGH组和LOW组的IC50(图4)。结果表明,在顺铂、多西紫杉醇、阿霉素、依托泊苷、吉西他滨等5种化疗药物中,HIGH组的卵巢癌样品的标准化IC50均较低。此外,CMap和作用机制(MoA)分析被用于预测潜在的卵巢癌小分子抑制剂及其机制(表2)。对20种最重要的小分子抑制剂进行鉴定,发现其潜在的治疗机制主要是多巴胺受体拮抗剂、DNA甲基转移酶抑制剂等。
图 4.

Estimated IC50 for chemotherapeutic drugs in samples of three independent cohorts (Wilcoxon test)
关键基因高表达组和低表达组在3个独立样本库中化疗药物的标准化IC50预测(Wilcoxon检验)
A-E: TGGA dataset (n=234); F-J: GSE53963 (n=174); K-O: GSE140082 (n=277). The pRRophetic analysis results in a negative or positive number when the predicted IC50 is standardized. HIGH: Hub genes high-expression group; LOW: Hub genes low-expression group.
表 2. List of the 20 most significant small-molecule inhibitors according to estimates of CMap analysis and mechanism of actions (MoA).
CMap和作用机制(MoA)分析预测的20种最重要的小分子抑制剂作用机理
| CMap name | Mean | Enrichment | P | MoA |
| RPS2: Ribosomal protein S2; SIRT: Sirtuin activator; TRPV: Transient receptorpotential vanilloid. | ||||
| MG-262 | −0.855 | −0.978 | 0.000 04 | |
| Prochlorperazine | −0.476 | −0.534 | 0.000 08 | Dopamine receptor antagonist |
| Withaferin A | −0.704 | −0.918 | 0.000 1 | |
| MS-275 | −0.879 | −0.989 | 0.000 3 | |
| Emetine | −0.701 | −0.89 | 0.000 3 | RPS2 |
| Metixene | −0.532 | −0.886 | 0.000 38 | Acetylcholine receptor antagonist |
| Etoposide | −0.343 | −0.885 | 0.000 38 | Topoisomerase inhibitor |
| Ciclopirox | −0.606 | −0.881 | 0.000 48 | |
| Mevalolactone | 0.562 | 0.933 | 0.000 52 | |
| Puromycin | −0.643 | −0.859 | 0.000 7 | Protein synthesis inhibitor |
| 5155877 | −0.372 | −0.85 | 0.000 93 | |
| Resveratrol | −0.537 | −0.608 | 0.000 98 | Cytochrome P450 inhibitor, SIRT activator |
| 5182598 | −0.843 | −0.976 | 0.001 27 | |
| Lomustine | −0.409 | −0.833 | 0.001 45 | |
| 5224221 | −0.839 | −0.973 | 0.001 57 | |
| Econazole | −0.382 | −0.829 | 0.001 61 | Bacterial cell wall synthesis inhibitor; Lanosterol demethylase inhibitor; Sterol demethylase inhibitor |
| Molindone | 0.37 | 0.822 | 0.001 67 | Dopamine receptor antagonist |
| Ciclosporin | −0.331 | −0.691 | 0.002 13 | |
| Thioridazine | −0.352 | −0.395 | 0.002 54 | Dopamine receptor antagonist, |
| Disulfiram | −0.446 | −0.731 | 0.002 84 | Aldehyde dehydrogenase inhibitor;
TRPV agonist; DNA methyltransferase inhibitor |
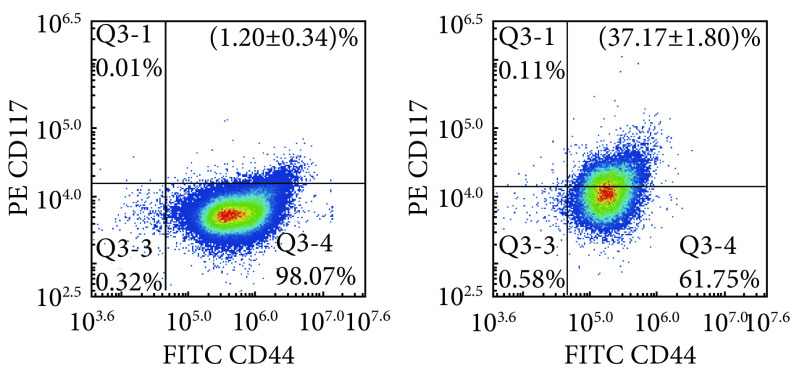
2.5. 人卵巢癌SKOV-3细胞的流式检测结果
流式细胞检测结果(图5)显示,SKOV-3细胞在经过无血清悬浮培养后,卵巢癌细胞的干性标志物被诱导,SKOV-3细胞中CD44+CD117+细胞的比例为(1.20±0.34)%,卵巢癌干细胞中CD44+CD117+细胞的比例为(37.17±1.80)%,差异有统计学意义(P<0.05)。
图 5.
Expression of CD44 and CD117 in SKOV-3 cells (left) and SKOV-3 CSCs (right) detected by flow cytometry (n=3)
流式细胞术检测SKOV-3细胞(左)与其CSC(右)中的CD44、CD117的表达(n=3)
2.6. qRT-PCR验证SKOV-3细胞和SKOV-3干细胞中的关键基因
为了验证WGCNA的发现,分别检测了15个干性基因在SKOV-3细胞和SKOV-3干细胞中的表达水平。qRT-PCR结果(图6)显示BUB1、CDC20、CCNB2、DLGAP5、KIF4A、NEK2、NUSAP1在SKOV-3干细胞组表达明显增加,AURKB、CDCA5、CDK1、NDC80、NCAPH在诱导干性后表达降低,EXO1、KIF2C、ORC1在两组之间的表达无明显差异。
图 6.
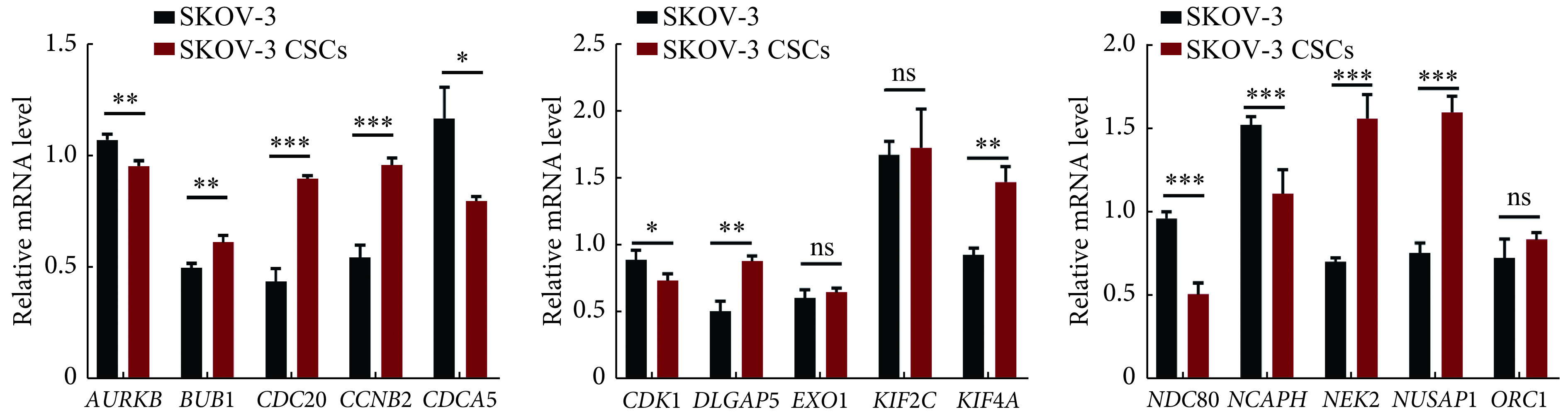
qRT-PCR validation of the expression of hub genes in SKOV-3 and SKOV-3 CSCs
qRT-PCR法验证关键基因在SKOV-3细胞和SKOV-3干细胞中的表达
*P<0.05, **P<0.01, ***P<0.001; ns: No significance.
2.7. GeneMANIA鉴定核心基因
为了发现上游可能的核心基因,利用GeneMANIA数据库构建了7个基因的基因-基因相互作用网络(图7)。7个中心节点与20个节点关联,这些节点代表的基因在物理相互作用、共表达、预测、共定位和遗传相互作用方面密切相关。BUB1与其他6个基因直接或间接地通过不同的作用方式相互关联,如与CDC20在物理作用方面相关,与CCNB2在通路作用上相关,与NUSAP1在遗传作用相关等。
图 7.

GeneMANIA identification of core genes
GeneMANIA鉴定核心基因
GTSE1: G2 and S-phase expressed 1; ARHGAP11A: Rho GTPase activating protein 11A; DEPDC1: DEP domain containing 1; SPATA6L: Spermatogenesis associated 6 like; MKI67: Marker of proliferation ki-67; CENPE: Centromere protein E; CENPF: Centromere protein F; BIRC5: Baculoviral IAP repeat containing 5; KIF11: Kinesin family member 11; ZWINT: ZW10 interacting kinetochore protein; KIF23: Kinesin family member 23; MAD2L1: Mitotic arrest deficient 2 like 1; CCNA2: Cyclin A2; HMMR: Hyaluronan mediated motility receptor; CCNB2: Cyclin B2; BUB1B: BUB1mitotic checkpoint serine/threonine kinase B; BUB1: BUB1 mitotic checkpoint serine/threonine kinase; CDC20: Cell division cycle 20; CCNB2: Cyclin B2; CDCA2: Cell division cycle associated 2; CDK1: Cyclin dependent kinase 1; DLGAP5: DLG associated protein 5; KIF4A: Kinesin family member 4A; KIF2C: Kinesin family member 2C; NDC80: Nuclear division cycle 80; NEK2: NIMA related kinase 2; NUSAP1: Nucleolar and spindle associated protein 1; ORC1: Origin recognition complex subunit 1.
3. 讨论
卵巢癌是一种发病机制不明的复杂疾病。近年来,CSC在肿瘤复发、预后和耐药性中起着重要作用。在此研究中,我们首次运用WGCNA研究卵巢癌干性的关键基因,运用基因富集分析、单细胞测序数据发掘其功能,以及它们对卵巢癌治疗敏感性及机理的影响,最后通过细胞实验验证出7个基因的作用,并发现基因BUB1在其中的核心地位。
WGCNA是一种有效的分析方法,可以构建一个无尺度的共表达网络来识别与mRNAsi显著正相关的关键模块。本研究获得了15个与卵巢癌干性相关的关键基因,并证实了它们在肿瘤组织中的表达水平。某些关键基因已被报道与CSCs的特性有关。BUB1是一种在乳腺癌和其他癌症中过度表达的有丝分裂检查点蛋白,对保持乳腺癌干细胞的潜能是必要的[8]。CDC20被认为是细胞周期检查点的调节器,通过促进核转位和β-catenin的反式激活而增强前列腺CSCs的自我更新能力[9]。NEK2是Nek丝氨酸/苏氨酸激酶家族的一员,先前的研究表明它可以通过Wnt/β-catenin信号途径调节CSC的干细胞样特性[10]。Notch、Wnt/β-catenin和Hedgehog通路被报道可能参与CSC的调控[11-12]。此外,Wnt/β-catenin途径中的CDC20和NEK2以及hedgehog途径中的NUSAP1可能是CSCs的关键基因[13-18]。基于上述证据,这些关键基因可能是卵巢癌干细胞的潜在生物标记物。
本研究用GSEA和单细胞RNA测序数据来探讨上述15个基因与一些细胞信号通路的关系。细胞周期模式被认为与CSCs的形成有关,并且细胞周期机制维持着CSC[15]。例如,CSCs可能通过改变卵巢癌细胞周期检查点和DNA修复网络信号的调节来维持干细胞样特性[16]。在乳腺癌中,G0/1细胞增加了CSC的迁移和乳腺球的形成[17]。此外,细胞周期调节蛋白Cycling D1和CDK4/6在乳腺癌的迁移和干细胞样活性中具有雌激素受体依赖的分化功能[18]。另一方面,CSCs也可以被认为是由氧化磷酸化介导的低代谢活性的慢周期细胞,这是CSCs维持肿瘤复发的原因[19-21]。此外,CSCs可以通过多种DNA修复机制存活并维持其功能[22]。在MALTA等[3]的研究中,他们利用分子特征数据库中致癌特征进一步验证了MYC[23]、E2F3[24],MTOR[25]诱导癌基因去分化的功能。真核细胞延长因子2(E2F)是参与细胞周期调控的转录调节因子家族成员,在多种肿瘤中均有表达[26-28]。E2F可能调节上皮间质转化而影响癌细胞的干细胞特性[29]。此外,已经发现Myc的表达增加可能与CSC样特性和肿瘤发生有关[30]。本研究中,富集分析显示关键基因上调组中的细胞信号通路涉及肿瘤干性的多个方面,即细胞周期、癌基因去分化(MYC、E2F、MOTRC1)、DNA修复(错配修复、DNA修复和蛋白酶体)、代谢改变(氧化磷酸化)、增殖(DNA复制、细胞周期、G2M检查点和有丝分裂纺锤体)等。因此,这些途径可能有助于提高卵巢癌的干细胞特性。
本研究利用肿瘤药物敏感基因(GDSC)数据库预测了治疗反应。在3个独立的样本库中,关键基因的表达水平均与化疗药物敏感性有关。此外,小分子抑制剂和潜在的治疗机制被确定。4种化合物(前氯拉嗪、莫利酮、硫哒嗪、二硫仑)共享多巴胺受体拮抗剂的作用机制。这些分析使我们找到了潜在的小分子抑制剂及MoA,但疗效和机制需进一步实验研究。
本研究中,通过无血清悬浮培养诱导卵巢癌细胞干性标志物的表达,CD44+CD117+细胞的比例明显增加。CD44和CD117是卵巢癌干细胞典型的表面标志物。已有研究报道卵巢癌患者腹水细胞进行悬浮培养后,CD44+CD117+细胞对顺铂和紫杉醇具有耐药性,接种于免疫缺陷的小鼠体内,可使其产生与来源肿瘤病理类型相似的移植瘤[31]。本研究用qRT-PCR进一步验证关键基因和卵巢癌干性之间的联系,发现7个基因(BUB1、CDC20、CCNB2、DLGAP5、KIF4A、NEK2、NUSAP1)在卵巢癌干细胞组表达升高,其中BUB1、CDC20、NEK2、NUSAP1已有证据表明和肿瘤干性明显相关[8, 10, 32-33]。GeneMANIA分析发现BUB1在7个基因中的核心地位。但其中一些从未被报道与CSC有关,值得进一步探讨。
综上所述,本研究建立了一个基因共表达网络并鉴定了7个与卵巢癌干性相关的关键基因,发现BUB1起着核心的作用。这些基因与卵巢癌干性特征和治疗策略有显著相关性。它们可能是卵巢癌干细胞的潜在生物标志物。目前CSC动物模型并不成熟,所以仍需通过进一步的生物学研究来探索这些发现。散点基因之间的关系分析还较为浅显,可通过阻断或沉默实验深入探索这些基因对卵巢癌干性特征及信号通路的影响,或借助临床样本的高通量测序来验证本研究的发现。
Contributor Information
梦 骆 (Meng LUO), Email: luomeng_1017@163.com.
学磊 马 (Xue-lei MA), Email: drmaxuelei@gmail.com.
References
- 1.TORRE L A, TRABERT B, DESANTIS C E, et al Ovarian cancer statistics, 2018. Ca-Cancer J Clin. 2018;68(4):284–296. doi: 10.3322/caac.21456. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.MOUFARRIJ S, DANDAPANI M, ARTHOFER E, et al Epigenetic therapy for ovarian cancer: promise and progress. Clin Epigenetics. 2019;11(1):7. doi: 10.1186/s13148-018-0602-0. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.MALTA T M, SOKOLOV A, GENTLES A J, et al. Machine learning identifies stemness features associated with oncogenic dedifferentiation. Cell, 2018, 173(2): 338-54 e15[2020-11-18]. https://pubmed.ncbi.nlm.nih.gov/29625051/. doi:10.1016/j.cell.2018.03.034.
- 4.PENG Q, LI R, LI Y, et al Prediction of a competing endogenous RNA co-expression network as a prognostic marker in glioblastoma. J Cell Mol Med. 2020;24(22):13346–13355. doi: 10.1111/jcmm.15957. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.LUO Y, SHEN D, CHEN L, et al Identification of 9 key genes and small molecule drugs in clear cell renal cell carcinoma. Aging (Albany NY) 2019;11(16):6029–6052. doi: 10.18632/aging.102161. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.DENG J, KONG W, MOU X, et al Identifying novel candidate biomarkers of RCC based on WGCNA analysis. Per Med. 2018;15(5):381–394. doi: 10.2217/pme-2017-0091. [DOI] [PubMed] [Google Scholar]
- 7.GOU R, ZHU L, ZHENG M, et al. Annexin A8 can serve as potential prognostic biomarker and therapeutic target for ovarian cancer: based on the comprehensive analysis of Annexins. J Transl Med, 2019, 17(1): 275[2020-11-18]. https://pubmed.ncbi.nlm.nih.gov/31474227/. doi: 10.1186/s12967-019-2023-z.
- 8.HAN J Y, HAN Y K, PARK G Y, et al. Bub1 is required for maintaining cancer stem cells in breast cancer cell lines. Sci Rep, 2015, 5: 15993[2020-11-18]. https://pubmed.ncbi.nlm.nih.gov/26522589/. doi: 10.1038/srep15993.
- 9.ZHANG Q, HUANG H, LIU A, et al Cell division cycle 20 (CDC20) drives prostate cancer progression via stabilization of beta-catenin in cancer stem-like cells. Ebiomedicine. 2019;42:397–407. doi: 10.1016/j.ebiom.2019.03.032. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.LIN S, ZHOU S, JIANG S, et al NEK2 regulates stem-like properties and predicts poor prognosis in hepatocellular carcinoma. Oncol Rep. 2016;36(2):853–862. doi: 10.3892/or.2016.4896. [DOI] [PubMed] [Google Scholar]
- 11.BARNAWI R, AL-KHALDI S, MAJED SLEIMAN G, et al Fascin is critical for the maintenance of breast cancer stem cell pool predominantly via the activation of the notch self-renewal pathway. Stem Cells. 2016;34(12):2799–2813. doi: 10.1002/stem.2473. [DOI] [PubMed] [Google Scholar]
- 12.MEDINA V, CALVO M B, DIAZ-PRADO S, et al Hedgehog signalling as a target in cancer stem cells. Clin Transl Oncol. 2009;11(4):199–207. doi: 10.1007/s12094-009-0341-y. [DOI] [PubMed] [Google Scholar]
- 13.LI H, ZHANG W J, YAN M, et al. Nucleolar and spindle associated protein 1 promotes metastasis of cervical carcinoma cells by activating Wnt/beta-catenin signaling. J Exp Clin Canc Res, 2019, 38(1): 33[2020-11-18]. https://pubmed.ncbi.nlm.nih.gov/30678687/. doi: 10.1186/s13046-019-1037-y.
- 14.WU X, XU B, YANG C, et al. Nucleolar and spindle associated protein 1 promotes the aggressiveness of astrocytoma by activating the Hedgehog signaling pathway. J Exp Clin Cancer Res, 2017, 36(1): 127[2020-11-18]. https://pubmed.ncbi.nlm.nih.gov/28899410/. doi: 10.1186/s13046-017-0597-y.
- 15.HAN T, YIN Q, WAN L Cycling for renewal: cell cycle machinery maintains prostate cancer stem-like cells. Ebiomedicine. 2019;42:24–25. doi: 10.1016/j.ebiom.2019.03.074. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.MENG E, MITRA A, TRIPATHI K, et al. ALDH1A1 maintains ovarian cancer stem cell-like properties by altered regulation of cell cycle checkpoint and DNA repair network signaling. PLoS One, 2014, 9(9): e107142[2020-11-18]. https://pubmed.ncbi.nlm.nih.gov/25216266/. doi: 10.1371/journal.pone.0107142.
- 17.LAMB R, LISANTI M P, CLARKE R B, et al Co-ordination of cell cycle, migration and stem cell-like activity in breast cancer. Oncotarget. 2014;5(17):7833–7842. doi: 10.18632/oncotarget.2312. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.LAMB R, LEHN S, ROGERSON L, et al Cell cycle regulators cyclin D1 and CDK4/6 have estrogen receptor-dependent divergent functions in breast cancer migration and stem cell-like activity. Cell Cycle. 2013;12(15):2384–2394. doi: 10.4161/cc.25403. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.LEE K M, GILTNANE J M, BALKO J M, et al. MYC and MCL1 cooperatively promote chemotherapy-resistant breast cancer stem cells via regulation of mitochondrial oxidative phosphorylation. Cell Metab, 2017, 26(4): 633-647e7[2020-11-18]. https://pubmed.ncbi.nlm.nih.gov/28978427/. doi: 10.1016/j.cmet.2017.09.009.
- 20.LAGADINOU E D, SACH A, CALLAHAN K, et al BCL-2 inhibition targets oxidative phosphorylation and selectively eradicates quiescent human leukemia stem cells. Cell Stem Cell. 2013;12(3):329–341. doi: 10.1016/j.stem.2012.12.013. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.FABIAN A, STEGNER S, MIARKA L, et al Metastasis of pancreatic cancer: an uninflamed liver micromilieu controls cell growth and cancer stem cell properties by oxidative phosphorylation in pancreatic ductal epithelial cells. Cancer Lett. 2019;453:95–106. doi: 10.1016/j.canlet.2019.03.039. [DOI] [PubMed] [Google Scholar]
- 22.VITALE I, MANIC G, DE MARIA R, et al DNA damage in stem cells. Mol Cell. 2017;66(3):306–319. doi: 10.1016/j.molcel.2017.04.006. [DOI] [PubMed] [Google Scholar]
- 23.KOOREMAN N G, KIM Y, DE ALMEIDA P E, et al. Autologous iPSC-based vaccines elicit anti-tumor responses in vivo. Cell Stem Cell, 2018, 22(4): 501-513. e7[2020-11-18].https://pubmed.ncbi.nlm.nih.gov/29456158/. doi: 10.1016/j.stem.2018.01.016.
- 24.WILLIAMS L, ZHAO J, MOROZOVA N, et al Chromatin reorganization accompanying cellular dedifferentiation is associated with modifications of histone H3, redistribution of HP1, and activation of E2F-target genes. Dev Dyn. 2003;228(1):113–120. doi: 10.1002/dvdy.10348. [DOI] [PubMed] [Google Scholar]
- 25.BAMODU O A, CHANG H L, ONG J R, et al. Elevated PDK1 expression drives PI3K/AKT/MTOR signaling promotes radiation-resistant and dedifferentiated phenotype of hepatocellular carcinoma. Cells, 2020, 9(3): 746[2020-11-18]. https://pubmed.ncbi.nlm.nih.gov/32197467/. doi: 10.3390/cells9030746.
- 26.DE MEYER T, BIJSMANS I T, VAN DE VIJVER K K, et al E2Fs mediate a fundamental cell-cycle deregulation in high-grade serous ovarian carcinomas. J Pathol. 2009;217(1):14–20. doi: 10.1002/path.2452. [DOI] [PubMed] [Google Scholar]
- 27.KENT L N, LEONE G The broken cycle: E2F dysfunction in cancer. Nat Rev Cancer. 2019;19(6):326–338. doi: 10.1038/s41568-019-0143-7. [DOI] [PubMed] [Google Scholar]
- 28.ZHAN L, ZHANG Y, WANG W, et al E2F1: a promising regulator in ovarian carcinoma. Tumour Biol. 2016;37(3):2823–2831. doi: 10.1007/s13277-015-4770-7. [DOI] [PubMed] [Google Scholar]
- 29.HASS R, VON DER OHE J, UNGEFROREN H. Potential role of MSC/cancer cell fusion and EMT for breast cancer stem cell formation. Cancers (Basel), 2019, 11(10): 1432[2020-11-18]. https://pubmed.ncbi.nlm.nih.gov/31557960/. doi: 10.3390/cancers11101432.
- 30.ZHANG L, LI J, XU H, et al Myc-Miz1 signaling promotes self-renewal of leukemia stem cells by repressing Cebpalpha and Cebpdelta. Blood. 2020;135(14):1133–1145. doi: 10.1182/blood.2019001863. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 31.ZHANG S, BALCH C, CHAN M W, et al Identification and characterization of ovarian cancer-initiating cells from primary human tumors. Cancer Res. 2008;68(11):4311–4320. doi: 10.1158/0008-5472.CAN-08-0364. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 32.ZHAO M, CHEN Z, ZHENG Y, et al Identification of cancer stem cellrelated biomarkers in lung adenocarcinoma by stemness index and weighted correlation network analysis. J Cancer Res Clin Oncol. 2020;146(6):1463–1472. doi: 10.1007/s00432-020-03194-x. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 33.ZHANG X, PAN Y, FU H, et al Nucleolar and spindle associated protein 1 (NUSAP1) inhibits cell proliferation and enhances susceptibility to epirubicin in invasive breast cancer cells by regulating Cyclin D kinase (CDK1) and DLGAP5 expression. Med Sci Monit. 2018;24:8553–8564. doi: 10.12659/MSM.910364. [DOI] [PMC free article] [PubMed] [Google Scholar]