Abstract
目的
研究N6-腺苷酸甲基化(N6-methyladenosine, m6A)结合蛋白YTH结构域蛋白2(YTH domain-containing protein 2,YTHDC2)对人骨髓间充质干细胞(human bone marrow mesenchymal stem cells,hBMSCs)成骨及成脂分化的调控。
方法
通过小干扰RNA(siRNA)体外对hBMSCs进行YTHDC2基因表达的敲降,并进行成骨及成脂诱导分化,以研究YTHDC2敲降后hBMSCs分化表型的改变。利用碱性磷酸酶(alkaline phosphatase,ALP)染色和茜素红染色鉴定成骨活性和钙结节形成,尼罗红染色检测脂滴形成。利用荧光定量PCR(RT-qPCR)检测成骨和成脂相关基因的表达。通过RNA测序(RNA-seq)分析YTHDC2敲降后的转录组变化,探索YTHDC2调控hBMSCs分化的潜在机制。
结果
敲降YTHDC2促进hBMSCs成骨分化中的ALP活性及钙结节形成,并显著上调成骨相关基因表达;同时降低了hBMSCs在成脂分化中的脂滴形成能力,并显著下调成脂相关基因表达。RNA-seq的基因富集分析显示YTHDC2与核糖体功能及mRNA翻译有关信号通路显著相关。
结论
敲降YTHDC2可促进hBMSCs成骨分化,抑制成脂分化。敲降YTHDC2可能造成核糖体功能改变。
Keywords: YTH结构域蛋白2, 人骨髓间充质干细胞, N6-腺苷酸甲基化 , 成骨分化, 成脂分化
Abstract
Objective
To study the regulatory effect of YTH domain-containing protein 2 (YTHDC2), a member of N6-methyladenosine (m6A) readers, on the osteogenic or adipogenic differentiation of human bone marrow mesenchymal stem cells (hBMSCs).
Methods
YTHDC2 expression was knocked down by small interfering RNA (siRNA) in vitro. Osteogenic differentiation and adipogenic differentiation of hBMSCs were induced after YTHDC2 knockdown in order to study the changes in the differentiation phenotype of hBMSCs. Alkaline phosphatase staining (ALP staining) and alizarin red S staining were performed to examine osteogenic activity and calcium-nodular formation. Nile red staining was performed to examine lipid-droplet formation. Real-time quantitative polymerase chain reaction (RT-qPCR) was used to assess the expression of osteogenesis and adipogenesis-related genes. RNA-sequencing was performed to identify the transcriptome changes after YTHDC2 knockdown and to explore the potential regulatory mechanism by which YTHDC2 regulated the diferentiation of hBMSCs.
Results
In this study, we found that siRNA-induced YTHDC2 knockdown resulted in increased ALP activity and calcium-nodular formation of hBMSCs during osteogenic differentiation, and significantly upregulated the expression of osteogenesis-related genes. In addition, the lipid-droplet formation capacity of hBMSCs was decreased during adipogenic differentiation. The expression of adipogenesis-related genes was significantly down-regulated. Gene-set enrichmen analysis of RNA-seq data showed that YTHDC2 was significantly correlated with ribosome function and mRNA-translation-related signaling pathways.
Conclusion
The findings indicate that YTHDC2 knockdown can promote the osteogenic differentiation of hBMSCs and inhibit the adipogenic differentiation. YTHDC2 knockdown may cause changes in ribosome function.
Keywords: YTH domain-containing protein 2, Human bone marrow mesenchymal stem cells, N6-methyladenosine , Osteogenic differentiation, Adipogenic differentiation
骨髓间充质干细胞(bone marrow mesenchymal stem cells,BMSCs)是一种在骨髓中广泛存在的干细胞,它可以多向分化为成骨细胞、软骨细胞、脂肪细胞或造血支持基质[1-2]。BMSCs在成骨和成脂分化方向的命运决定影响着骨骼健康[3]。N6-腺苷酸甲基化(N6-methyladenosine,m6A)修饰是真核生物体内mRNA上最广泛的一种表观遗传修饰。该修饰表现为腺嘌呤六号N原子上的甲基化[4-6]。近年来,研究揭示了m6A修饰在哺乳动物细胞中的一系列生理作用,包括调控mRNA剪切、出核、成熟、翻译、肿瘤代谢以及细胞增殖分化等[7]。m6A修饰需要多种蛋白复合体的参与,包括m6A“写码器”甲基转移酶、“消码器”去甲基化酶和“读码器”m6A结合蛋白。其中,甲基转移酶介导在mRNA上添加m6A修饰,去甲基化酶介导m6A修饰的清除,m6A结合蛋白可以特异性识别m6A修饰[8]。本课题组前期研究发现了m6A甲基转移酶3(methyltransferase-like 3,METTL3)介导的m6A修饰对BMSCs分化的调控[9]。而作为m6A代谢中的重要一环,目前缺乏m6A结合蛋白调控BMSCs分化的研究[10]。
m6A结合蛋白包括YTH N6甲基腺苷RNA结合蛋白1、2、3(YTHDF1、YTHDF2、YTHDF3),YTH结构域蛋白(YTH domain-containing protein)1、2(YTHDC1、YTHDC2)。其中,YTHDC2是YTH家族中最新发现的蛋白。YTHDC2广泛分布于真核生物胞质内,可通过YTH序列特异性识别并结合m6A,从而促进mRNA的翻译并影响mRNA的稳定性[11]。研究表明,YTHDC2对维持减数分裂及生殖细胞的产生至关重要[12-14],并参与多种疾病的发生发展,包括:自闭症、胰腺癌、结肠腺癌、肝癌、肺腺癌和头颈部鳞状细胞癌[15-20]。而YTHDC2如何调控BMSCs的命运决定尚不清楚。本研究的目的是探究YTHDC2对人骨髓间充质干细胞(hBMSCs)成骨和成脂分化的影响及其作用机制。
1. 材料和方法
1.1. 细胞培养
hBMSCs原代细胞系(ATCC® PCS-500-012)购自American Type Culture Collection。hBMSCs具有多向分化潜能。我们将hBMSCs细胞悬浮液转移至培养皿中,采用α-MEM(Gibco)培养基进行培养。培养基中添加10%胎牛血清(Gibco),100 U/mL青霉素(Gibco)及100 µg/mL链霉素(Gibco)。培养条件为37 ℃、体积分数5%CO2,每2 d更换1次培养基,每6 d进行1次传代。每次传代采用胰酶重悬细胞,按1×104/cm2的密度均匀铺在培养皿中。hBMSCs在传代至第3、4代时增殖活性较强,细胞形态为纺锤状,因此本研究采用第4代hBMSCs进行后续实验。
1.2. siRNA转染试验
使用si-YTHDC2(Santa Cruz Biotechnology,sc-91804)及标准对照si-CTRL(Sangon Biotech)对hBMSCs进行转染。转染试剂为50 nmol/L Lipofectamine™ RNAiMAX(Invitrogen)溶于无血清Opti-MEM®I Medium(Invitrogen)。hBMSCs在无抗生素培养基内培养24 h后,加入含si-YTHDC2或si-CTRL的转染试剂,在37 ℃、体积分数5%CO2下孵育12 h换液。转染后48 h,使用YTHDC2成品引物(Santa Cruz Biotechnology,sc-91804-PR)通过荧光定量PCR(RT-qPCR)检测YTHDC2表达(具体方法见1.5),并计算si-YTHDC2转染hBMSCs的敲降效率。
1.3. hBMSCs成骨分化诱导实验
经siRNA转染的hBMSCs在成骨诱导液中培养进行成骨诱导分化。成骨诱导液以α-MEM培养基为溶剂,含100 nmol/L地塞米松(Sigma)、50 µg/mL抗坏血酸(Sigma)、5 mmol/L β-甘油磷酸(Sigma)、10%胎牛血清、100 U/mL青霉素和100 µg/mL链霉素。碱性磷酸酶(ALP)活性是早期成骨的重要标志[21]。细胞在24孔板中成骨诱导7 d后进行ALP染色:PBS洗涤孔板3次,多聚甲醛固定20 min,再用ALP染液(Beyotime Biotechnology)避光孵育20 min使其显色,去除染液后PBS洗涤孔板3次,采集图片。hBMSCs成骨诱导3 、7 d后进行ALP定量:细胞经胰酶消化后使用超声裂解,离心后吸取上清液,采用BCA蛋白检测试剂盒(Beyotime Biotechnology)通过562 nm处吸光度值测定样本蛋白浓度。按ALP检测试剂盒(Beyotime Biotechnology)说明书操作,测定样本520 nm处吸光度值。ALP=(样品吸光度值−空白孔吸光度值)/(标准孔吸光度值−空白孔吸光度值)×酶标准品浓度/样本蛋白浓度。上式中,ALP单位为U/g protein,酶标准品浓度为0.02 mg/mL,样本蛋白浓度单位为g protein/mL。
茜素红染色检测成骨中后期细胞沉积钙盐并形成钙结节的能力[22]。hBMSCs在24孔板中成骨诱导14 d后进行茜素红染色:PBS洗涤3次,多聚甲醛固定20 min,再使用茜素红染液(Solarbio Technology)避光孵育30 min使其显色。显色后用10%氯化十六烷基吡啶处理15 min,在562 nm处测定吸光度,通过对比钙浓度标准曲线进行定量测定。
hBMSCs在成骨诱导3、5、7 d后进行成骨相关基因的RT-qPCR检测,包括distal-less homeobox 5(DLX5)、RUNX family transcription factor 2(RUNX2)、Sp7 transcription factor(SP7)、collagen type Ⅰ alpha 1(COL1A1)、bone gamma-carboxyglutamate protein(BGLAP)和secreted phosphoprotein 1(SPP1)。具体方法见后文1.5。
1.4. hBMSCs成脂分化诱导实验
将hBMSCs转染后在成脂诱导液中进行成脂诱导分化。成脂诱导液以DMEM高糖培养基为溶剂,含1 mmol/L地塞米松、4 mg/mL胰岛素(Sigma)、50 mmol/L3-异丁基-1-甲基黄嘌呤(IBMX)、10%胎牛血清、100 U/mL青霉素和100 µg/mL链霉素。尼罗红染色:细胞在96孔板中成脂诱导28 d后,PBS洗涤孔板3次,多聚甲醛固定20 min,再使用尼罗红荧光染料(Genmed)避光孵育10 min后显色,PBS洗涤3次,于荧光显微镜下观察脂滴,对成脂表型进行鉴定。
hBMSCs在成脂诱导3、5、7 d后进行成脂相关基因的RT-qPCR检测,包括CCAAT enhancer binding protein alpha(CEBPα)、perilipin 1(PLIN1)和lipoprotein lipase(LPL)。具体方法见1.5。
1.5. RT-qPCR检测成骨、成脂分化相关基因表达
使用TRIzol试剂(Invitrogen)提取总RNA,使用PrimeScript RT试剂盒(TaKaRa)和gDNA eraser试剂盒(TaKaRa)制备cDNA。RT-qPCR在SYBR Premix Ex TaqⅡ(TaKaRa)反应体系及Bio-Rad CFX96 real-time PCR仪中进行。扩增反应条件为:95 ℃下30 s,1次循环;95 ℃下变性5 s,60 ℃下退火31 s,40个循环。成骨相关基因引物序列见表1,使用GAPDH作为内参;成脂相关基因序列见表2,使用36B4作为内参。使用LightCycler 96软件(Roche)统计Ct值,以2−ΔΔCt表示基因表达量。
表 1. Primer sequences of hBMSCs osteogenic differentiation related genes.
hBMSCs成骨分化相关基因引物序列
| Gene | Primer sequence (5′-3′) |
| DLX5: Distal-less homeobox 5; RUNX2: RUNX family transcription factor 2; SP7: Sp7 transcription factor; COL1A1: Collagen type Ⅰ alpha 1; BGLAP: Bone gamma-carboxyglutamate protein; SPP1: Secreted phosphoprotein 1; GAPDH: Glyceraldehyde-3-phosphate dehydrogenase. | |
| DLX5 | F: TTCCAAGCTCCGTTCCAGAC |
| R: GAATCGGTAGCTGAAGACTCG | |
| RUNX2 | F: TGGTTACTGTCATGGCGGGTA |
| R: TCTCAGATCGTTGAACCTTGCTA | |
| SP7 | F: CCTCTGCGGGACTCAACAAC |
| R: AGCCCATTAGTGCTTGTAAAGG | |
| COL1A1 | F: GTGCGATGACGTGATCTGTGA |
| R: CGGTGGTTTCTTGGTCGGT | |
| BGLAP | F: CACTCCTCGCCCTATTGGC |
| R: CCCTCCTGCTTGGACACAAAG | |
| SPP1 | F: GCCGCTGTAACCTCTTCGG |
| R: GTCTTCGGCCAATCTGGCTTT | |
| GAPDH | F: ACTGAGGACCAGGTTGTC |
| R: TGCTGTAGCCGTATTCATTG | |
表 2. Primer sequences of hBMSCs adipogenic differentiation related genes.
hBMSCs成脂分化相关基因引物序列
| Gene | Primer sequence (5′-3′) |
| CEBPα: CCAAT enhancer binding protein alpha; PLIN1: Perilipin 1; LPL: Lipoprotein lipase; 36B4: Ribosomal protein lateral stalk subunit P0. | |
| CEBPα | F: TTCACATTGCACAAGGCACT |
| R: GAGGGACCGGAGTTATGACA | |
| PLIN1 | F: TGTGCAATGCCTATGAGAAGG |
| R: AGGGCGGGGATCTTTTCCT | |
| LPL | F: CCCTCTCTTACAAGCCCATCA |
| R: GAGCCAGTCTGGTAGTACATCA | |
| 36B4 | F: TGAGATTCGGGATATGCTGTTGG |
| R: CGGGTCCTAGACCAGTGTTCT | |
1.6. RNA测序(RNA-seq)检验YTHDC2敲降在转录组水平的变化
为了进一步探究YTHDC2影响hBMSCs成骨分化的机制,我们采用YTHDC2敲降的细胞进行了RNA-seq,以检验YTHDC2敲降在转录组水平的变化。siRNA转染48 h后,使用TRIzol试剂提取总RNA,si-CTRL与si-YTHDC2各3个生物学重复。使用Illumina TrueSeq mRNA sample preparation kit制备样本,并在Illumina HiSeq 3000 machine上进行测序。使用STAR_2.6.0a将其映射到人类基因组(UCSChg19)并使用DESeq2软件(1.16.1)进行两个比较组合之间的差异性表达分析,adjusted P value(padj)小于0.05和log2fold change绝对值大于0.5的基因为差异基因。绘制基因差异性表达分析结果图(火山图)(Metascape网站在线工具)。GO分析通过clusterProfiler(3.4.4)软件进行,利用GO数据库实现差异表达基因的功能富集分析。KEGG分析使用clusterProfiler(3.4.4)软件进行,利用KEGG数据库实现差异表达基因的通路富集分析。
1.7. 统计学方法
所有数据均以
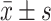 表示。通过非配对双侧t检验进行统计效能检验,P<0.05为差异有统计学意义。
表示。通过非配对双侧t检验进行统计效能检验,P<0.05为差异有统计学意义。
2. 结果
2.1. YTHDC2敲降后成骨分化能力增强
经RT-qPCR验证,si-YTHDC2转染hBMSCs的敲降效率为62%,满足后续实验要求。ALP染色结果显示,成骨诱导第7天,YTHDC2敲降造成hBMSCs的ALP活性显著升高,早期成骨分化活性增强(图1A)。茜素红染色结果显示,成骨诱导14 d后,YTHDC2敲降导致hBMSCs形成钙结节的能力增强(图1B)。成骨诱导第3、5、7天,RT-qPCR结果显示:si-YTHDC2组的DLX5、RUNX2、SP7、COL1A1、BGLAP、SPP1表达均升高(图2)。证明YTHDC2敲降后hBMSCs成骨分化能力显著增强。
图 1.

hBMSCs alkaline phosphatase staining and alizarin red staining results after siRNA transfection
siRNA转染后,hBMSCs的碱性磷酸酶染色及茜素红染色结果
A: Alkaline phosphatase staining; B: Alizarin red staining. n=3. ***P<0.001, vs. si-CTRL group at the same time point.
图 2.

After siRNA transfection, the expression of osteogenic differentiation related genes at day 3, 5 and 7 of osteogenesis induction
siRNA转染后,hBMSCs成骨诱导3、5、7 d,成骨分化相关基因表达趋势
n=3. *P<0.05, **P<0.01,***P<0.001, vs. si-CTRL group at the same time piont.
2.2. YTHDC2敲降后成脂分化诱导能力减弱
成脂诱导28 d后,尼罗红染色显示,在同一细胞密度下,si-YTHDC2组形成的脂滴数量和体积都更小(图3)。RT-qPCR结果显示,成脂诱导第3、5、7天,YTHDC2敲降组成脂相关基因CEBPα、PLIN1、LPL的表达总体呈现下降趋势(图4),证明YTHDC2敲降后成脂分化能力显著减弱。
图 3.
Nile red staining results of hBMSCs after siRNA transfection (scale bar 25 μm)
siRNA转染后hBMSCs尼罗红染色结果(标尺25 μm)
图 4.

After siRNA transfection, the expression of hBMSCs adipogenic differentiation-related genes at day 3, 5 and 7 of adipogenesis induction
siRNA转染后hBMSCs第3、5、7 d,成脂分化相关基因表达趋势
n=3. *P<0.05, **P<0.01, ***P<0.001, vs. si-CTRL group at the same time piont.
2.3. YTHDC2敲降后基因转录组的改变
si-YTHDC2及si-CTRL转染后的RNA-seq的结果如下。基因差异性表达分析结果(图5A)显示,YTHDC2敲降后,162个基因表达上调,230个基因表达下调。GO功能富集分析发现,与YTHDC2相关的生物学功能中,相关性最强的为核糖体,其次为甲型流感、麻疹、单纯疱疹病毒感染,NOD样受体信号通路(图5B)。KEGG通路富集分析显示,7条通路与mRNA翻译相关,其中包括:核糖体组成结构、核糖体功能、核糖体亚基、翻译起始、病毒基因表达、病毒转录和核转录mRNA的分解(图5C)。
图 5.

RNA-seq results after siRNA transfection
siRNA转染后RNA-seq结果
A: Valcano plot; B: GO analysis results; C: KEGG analysis results. padj: Adjusted P value.
3. 讨论
m6A修饰广泛参与机体内骨形成过程的调节,一系列研究证明了m6A调节蛋白在骨形成以及BMSCs分化中具有重要调控作用。研究表明,m6A甲基转移酶METTL3可通过m6A修饰调节NF-κB信号通路,从而抑制骨质疏松[23]。这一过程可被m6A去甲基化酶AlkB同源蛋白5(AlkB homolog 5,ALKBH5)逆转[24]。METTL3的表达水平还可以调控BMSCs的成骨分化[9]。同样,对m6A与BMSCs成脂分化的研究显示,m6A甲基转移酶METTL3可以通过m6A-YTHDF2途径抑制BMSCs成脂向分化[25]。ALKBH5则可以抑制成骨并促使BMSCs分化为脂肪细胞[26]。这些研究结果表明,m6A修饰可调节骨形成过程并广泛参与BMSCs成骨,成脂分化的调控。而本实验通过对m6A结合蛋白YTHDC2在转录水平的敲降,证明了YTHDC2也是调节骨髓间充质干细胞分化的重要蛋白。不仅发现了YTHDC2全新的生物学功能,还进一步扩展了m6A修饰影响BMSCs分化的作用机制。
YTHDC2是调节核糖体功能及mRNA翻译的重要蛋白。一方面,YTHDC2可以与核糖体结合促进翻译起始,提高翻译效率[21,27-28]。另一方面,它具有3'→5'RNA解旋酶活性,并可与核酸外切酶5'-3' exoribonuclease 1(XRN1)相互作用,介导mRNA的降解,降低胞内mRNA丰度[29-32]。GO分析及KEGG分析结果均显示,核糖体相关通路出现了显著改变。这与已阐明的YTHDC2生物学功能相一致。这些信息提示我们,YTHDC2敲降可能造成核糖体功能发生显著改变。核糖体是mRNA翻译过程中重要的组分,协调与介导mRNA翻译起始、延长与终止。核糖体功能的改变可能广泛地影响一系列细胞的生理功能。因而,在hBMSCs的分化中, YTHDC2 有可能通过改变核糖体功能进而影响mRNA的翻译,以此来调控成骨与成脂分化。
本研究首次明确了YTHDC2在hBMSCs分化中具有重要的调控作用,即YTHDC2缺失会促进hBMSCs成骨分化,抑制其成脂分化。通过RNA-seq,本研究进一步探索了YTHDC2敲降对hBMSCs转录组的影响,提示我们YTHDC2可能通过核糖体相关通路发挥作用。本研究局限性在于并未进行体内实验,因此课题组将在后续研究中进行动物模型的构建,并进一步验证具体的分子生物学机制。
* * *
利益冲突 所有作者均声明不存在利益冲突
References
- 1.FENG Q, ZHENG S, ZHENG J. The emerging role of microRNAs in bone remodeling and its therapeutic implications for osteoporosis. Biosci Rep, 2018, 38(3): BSR20180453[2021-04-14]. https://doi.org/10.1042/BSR20180453.
- 2.GARIA-GARCIA A, DE CASTILLEJO C L, MENDEZ-FERRER S BMSCs and hematopoiesis. Immunol Lett. 2015;168(2):129–135. doi: 10.1016/j.imlet.2015.06.020. [DOI] [PubMed] [Google Scholar]
- 3.CICCIU M, BRAMANTI E, CECCHETTI F, et al FEM and Von Mises analyses of different dental implant shapes for masticatory loading distribution. Oral Implantol (Rome) 2014;7(1):1–10. [PMC free article] [PubMed] [Google Scholar]
- 4.KOUZARIDES T Chromatin modifications and their function. Cell. 2007;128(4):693–705. doi: 10.1016/j.cell.2007.02.005. [DOI] [PubMed] [Google Scholar]
- 5.MACHNICKA M A, MILANOWSKA K, OSMAN OGL O U, et al. MODOMICS: a database of RNA modification pathways—2013 update. Nucleic Acids Res, 2013, 41:D262-D267[2021-04-14]. https://doi.org/10.1093/nar/gks1007.
- 6.ZHANG C, FU J, ZHOU Y. A review in research progress concerning m6A methylation and immunoregulation. Front Immunol, 2019, 10: 922[2021-04-14]. https://doi.org/10.3389/fimmu.2019.00922.
- 7.FU Y, DOMINISSINI D, RECHAVI G, et al Gene expression regulation mediated through reversible m6A RNA methylation . Nat Rev Genet. 2014;15(5):293–306. doi: 10.1038/nrg3724. [DOI] [PubMed] [Google Scholar]
- 8.WU H, ZHANG Y Reversing DNA methylation: mechanisms, genomics, and biological functions. Cell. 2014;156(1/2):45–68. doi: 10.1016/j.cell.2013.12.019. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.WU Y, XIE L, WANG M, et al. Mettl3-mediated m6A RNA methylation regulates the fate of bone marrow mesenchymal stem cells and osteoporosis. Nat Commun, 2018, 9(1): 4772[2021-04-14]. https://doi.org/10.1038/s41467-018-06898-4.
- 10.YAO Y, BI Z, WU R, et al METTL3 inhibits BMSC adipogenic differentiation by targeting the JAK1/STAT5/C/EBPβ pathway via an m6A-YTHDF2-dependent manner . FASEB J. 2019;33(6):7529–7544. doi: 10.1096/fj.201802644R. [DOI] [PubMed] [Google Scholar]
- 11.HSU P J, ZHU Y, MA H, et al YTHDC2 is an N6-methyladenosine binding protein that regulates mammalian spermatogenesis . Cell Res. 2017;27(9):1115–1127. doi: 10.1038/cr.2017.99. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.SOH Y Q S, MIKEDIS M M, KOJIMA M, et al. Meioc maintains an extended meiotic prophase I in mice. PLoS Genet, 2017, 13(4): e1006704[2021-04-14]. https://doi.org/10.1371/journal.pgen.1006704.
- 13.BAILEY A S, BATISTA P J, GOLD R S, et al. The conserved RNA helicase YTHDC2 regulates the transition from proliferation to differentiation in the germline. eLife, 2017, 6: e26116[2021-04-14]. https://doi.org/10.7554/eLife.26116.
- 14.JAIN D, PUNO M R, MEYDAN C, et al. ketu mutant mice uncover an essential meiotic function for the ancient RNA helicase YTHDC2. elife, 2018, 7: e30919[2021-04-14]. https://doi.org/ 10.7554/eLife.30919.
- 15.FANALE D, IOVANNA J L, CALVO E L, et al Germline copy number variation in the YTHDC2 gene: does it have a role in finding a novel potential molecular target involved in pancreatic adenocarcinoma susceptibility? . Expert Opin Ther Targets. 2014;18(8):841–850. doi: 10.1517/14728222.2014.920324. [DOI] [PubMed] [Google Scholar]
- 16.XU D, SHAO J, SONG H, et al. The YTH domain family of N6-methyladenosine “readers” in the diagnosis and prognosis of colonic adenocarcinoma. Biomed Res Int, 2020,2020:9502560[2021-04-14]. https://doi.org/10.1155/2020/9502560.
- 17.TANABE A, TANIKAWA K, TSUNETOMI M, et al RNA helicase YTHDC2 promotes cancer metastasis via the enhancement of the efficiency by which HIF-1α mRNA is translated . Cancer Lett. 2016;376(1):34–42. doi: 10.1016/j.canlet.2016.02.022. [DOI] [PubMed] [Google Scholar]
- 18.SUN S, HAN Q, LIANG M, et al Downregulation of m6 A reader YTHDC2 promotes tumor progression and predicts poor prognosis in non-small cell lung cancer . Thorac Cancer. 2020;11(11):3269–3279. doi: 10.1111/1759-7714.13667. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.MA L, CHEN T, ZHANG X, et al. The m6A reader YTHDC2 inhibits lung adenocarcinoma tumorigenesis by suppressing SLC7A11-dependent antioxidant function. Redox Biol, 2021, 38:101801[2021-04-14]. https://doi.org/10.1016/j.redox.2020.101801.
- 20.LI Y, ZHENG J N, WANG E H, et al. The m6A reader protein YTHDC2 is a potential biomarker and associated with immune infiltration in head and neck squamous cell carcinoma. Peer J, 2020, 8: e10385[2021-04-14]. https://doi.org/10.7717/peerj.10385.
- 21.VIMALRAJ S. Alkaline phosphatase: structure, expression and its function in bone mineralization. Gene, 2020, 754: 144855[2021-04-14]. https://doi.org/10.1016/j.gene.2020.144855.
- 22.NAGY A, GERTSENSTEIN M, VINTERSTEN K, et al. Alizarin red staining of post-natal bone in mouse. Cold Spring Harb Protoc, 2009, 2009(3): pdb.prot5171[2021-04-14].https://doi.org/10.1101/pdb.prot5171.
- 23.CHEN X, HUA W, HUANG X, et al. Regulatory role of RNA N6-methyladenosine modification in bone biology and osteoporosis. Front Endocrinol (Lausanne), 2020,1(10):911[2021-04-14]. https://doi.org/10.3389/fendo.2019.00911.
- 24.YU J, SHEN L, LIU Y, et al The m6A methyltransferase METTL3 cooperates with demethylase ALKBH5 to regulate osteogenic differentiation through NF-κB signaling . Mol Cell Biochem. 2020;463(1/2):203–210. doi: 10.1007/s11010-019-03641-5. [DOI] [PubMed] [Google Scholar]
- 25.WU R, GUO G, BI Z, et al m6A methylation modulates adipogenesis through JAK2-STAT3-C/EBP β signaling . Biochim Biophys Acta Gene Regul Mech. 2019;1862(8):796–806. doi: 10.1016/j.bbagrm.2019.06.008. [DOI] [PubMed] [Google Scholar]
- 26.CGEM S, LI J, CAI Z, et al. TRAF4 acts as a fate checkpoint to regulate the adipogenic differentiation of MSCs by activating PKM2. EBio Med, 2020,54:102722[2021-04-14]. https://doi.org/10.1016/j.ebiom.2020.102722.
- 27.DU H, ZHAO Y, HE J, et al. YTHDF2 destabilizes m6A-containing RNA through direct recruitment of the CCR4-NOT deadenylase complex. Nat Commun, 2016, 7: 12626[2021-04-14]. https://doi.org/10.1038/ncomms12626.
- 28.XU C, LIU K, AHMED H, et al Structural basis for the discriminative recognition of N6-methyladenosine RNA by the human YT521-B homology domain family of proteins . J Biol Chem. 2015;290(41):24902–24913. doi: 10.1074/jbc.M115.680389. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.MAO Y, DONG L, LIU X M, et al. m6A in mRNA coding regions promotes translation via the RNA helicase-containing YTHDC2. Nat Commun, 2019, 10(1): 5332[2021-04-14]. https://doi.org/10.1038/s41467-019-13317-9.
- 30.KRETSCHMER J, RAO H, HACKERT P, et al The m6A reader protein YTHDC2 interacts with the small ribosomal subunit and the 5′-3′ exoribonuclease XRN1 . RNA. 2018;24(10):1339–1350. doi: 10.1261/rna.064238.117. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 31.GREGERSEN L H, SCHUELER M, MUNSCHAUER M, et al MOV10 is a 5′ to 3′ RNA helicase contributing to UPF1 mRNA target degradation by translocation along 3′ UTRs. Mol Cell. 2014;54(4):573–585. doi: 10.1016/j.molcel.2014.03.017. [DOI] [PubMed] [Google Scholar]
- 32.NAGARAJAN V K, JONES C I, NEWBURY S F, et al XRN 5′→3′ exoribonucleases: structure, mechanisms and functions. Biochim Biophys Acta. 2013;1829(6/7):590–603. doi: 10.1016/j.bbagrm.2013.03.005. [DOI] [PMC free article] [PubMed] [Google Scholar]



