Abstract
目的
探讨端粒酶新核心亚单位WD40-encoding RNA antisense to p53(WRAP53β)与头颈鳞癌(squamous cell carcinoma of the head and neck, HNSC)的临床、基因组和免疫浸润特征的关系,寻求HNSC的潜在靶向联合治疗方法。
方法
采用TIMER在线模块预测WRAP53β表达与TCGA队列中头颈肿瘤的临床特征、癌基因或免疫浸润之间的关系,TISCH网站分析其在单细胞水平的表达,通过CARE软件分析靶向WRAP53β的小分子抑制剂。体外实验验证中,合成shWRAP53β序列的重组慢病毒颗粒,构建稳定表达shWRAP53β的口腔鳞癌Cal27细胞(shWRAP53β组),并设置两个对照组(shNC组:Cal27细胞加入含非特异性对照序列的慢病毒颗粒,Con组:未处理的Cal27细胞);MTT法检测3组细胞的增殖能力,细胞免疫荧光检测进一步定性检测shWRAP53β组和shNC组细胞中P53蛋白的表达;Western blot检测第18、23、28代shWRAP53β组和shNC组细胞的WRAP53β和DNA损伤蛋白γ-H2AX的表达。最后,收集口腔鳞癌病例样本13例,口腔黏膜炎性病例样本7例,通过免疫组化验证口腔鳞癌临床标本中WRAP53β和γ-H2AX的表达情况。
结果
TIMER分析发现WRAP53β表达在HNSC中较正常组织显著高表达。WRAP53β基因水平与p53通路基因如CCNB1、CCNB2及CDK1呈显著正相关,与HPV阳性的头颈肿瘤炎症因子IFN-γ、IL-23A呈正相关,而与IL-1A、IL-6呈负相关,但与浸润免疫细胞无显著相关。TISCH单细胞测序数据集同样显示该基因在恶性细胞中呈高表达,在免疫细胞中表达水平极低或无表达。CARE评分预测对WRAP53β共抑制效果最强的药物为靶向ATM、CDK1和MDM4的抑制剂。体外细胞实验证实,与对照组相比,shWRAP53β组Cal27细胞的第5天到第7天的增殖能力下降(P<0.05),P53表达降低。Western blot检测示,与对照组相比,shWRAP53β组WRAP53β的表达水平在第18、23、28代均降低(P<0.05),γ-H2AX表达仅在第18、28代降低(P<0.05)。临床标本显示口腔鳞癌组织中γ-H2AX的阳性表达检出率高(12/13),口腔黏膜炎样本未检出WRAP53β阳性表达(0/7)。
结论
WRAP53β在HSNC中呈高表达水平,且与p53通路基因显著相关。ATM、CDK1和MDM4抑制剂可能是WRAP53β的潜在共抑制药物。干扰WRAP53β可能导致DNA损伤降低,从而具有对HNSC的治疗潜力。
Keywords: WRAP53β , TIMER, 生物信息学分析, 免疫浸润, 共抑制
Abstract
Objective
To investigate the association between WD40-encoding RNA antisense to p53 (WRAP53β), a telomerase new core subunit, and the clinical, genomic and immune infiltration characteristics of squamous cell carcinoma of the head and neck (HNSC), and to explore for potential joint targeted therapy of HNSC.
Methods
Tumor IMmune Estimation Resource (TIMER) online modules were adopted to predict the association betweenWRAP53β expression and the clinical features, oncogene, and immune infiltration of HNSC in the Cancer Genome Atlas (TCGA) cohort. Tumor Immune Single-cell Hub (TISCH) was used to analyze WRAP53β expression at the single cell level. Analysis of the small molecule inhibitors potentially targeting WRAP53β was carried out by Computational Analysis of REsistance (CARE). In the in vitro verification experiment, recombinant lentiviral particles with the shWRAP53β sequence were synthesized. Then, the oral squamous cell carcinoma cell line Cal27 (the shWRAP53βgroup) stably expressing shWRAP53β were constructed, and two control groups were set up (the shNC group consisting of Cal27 cells added with lentiviral particles containing non-specific control sequences and the Con group consisting of untreated Cal27 cells). MTT assay was done to examine the proliferation of cells in the three groups. Cellular immunofluorescence assay was done for further qualitative examination of the expression of P53 protein in the cells of the shWRAP53β group and the shNC group. Western blot was done to measure the expression of WRAP53β and γ-H2AX, a DNA damage protein, in the 18th, 23rd and 28th passages of the shWRAP53β group and the shNC group. Finally, specimens of 13 cases of oral squamous cell carcinoma and 7 cases of oral mucosal inflammation were collected, and the expression of WRAP53β and γ-H2AX in the clinical specimens of oral squamous cell carcinoma was verified with immunohistochemistry.
Resluts
TIMER analysis revealed that the expression level of WRAP53β in HNSC tissues was significantly higher than that in normal tissues. There was a significant positive correlation between WRAP53β expression and multiple genes in the p53 pathway, including CCNB1, CCNB2 and CDK1. Although no significant correlation between WRAP53β expression and infiltrating immune cells was found, WRAP53β was significantly positively correlated with the inflammatory factors IFN-γ and IL23A, and negatively correlated with IL-1A and IL-6 in HPV-positive carcinoma of the head and neck. TISCH single cell sequencing datasets also showed higher expression of WRAP53β in malignant cells, and very low or zero expression in immune cells. According to the CARE scores, the most potent WRAP53β co-inhibitory drugs were ATM, CDK1 and MDM4 targeted inhibitors. In vitro cell experiments showed that the proliferation ability of Cal27 cells decreased significantly in the shWRAP53β group as compared with that of the control group between Day 5 and Day 7 (P<0.05). Furthermore, the expression of P53 decreased significantly in the shWRAP53β group. As compared with the control group, the expression of WRAP53β in shWRAP53β group significantly decreased in the 18th, 23rd and 28th passages (P<0.05), while γ-H2AX expression only decreased in the 18th and 28th passages (P<0.05) according to the results of Western blot. Clinical specimens showed rather high positive expression rate of γ-H2AX in oral squamous cell carcinoma tissues (12/13), while the expression of WRAP53β was not detected in oral mucositis samples (0/7).
Conclusions
WRAP53β showed significantly higher expression level in HSNC, and was significantly associated with p53 pathway genes. ATM, CDK1 and MDM4 inhibitors may be potential WRAP53β co-inhibitory agents. RNA interference of WRAP53β expression may cause inhibition of DNA damage, thereby indicating therapeutic potential for HNSC.
Keywords: WRAP53β, Tumor IMmune Estimation Resource (TIMER), Bioinformatics analysis, immune infiltration, Co-inhibition
WD40-encoding RNA antisense to p53(WRAP53β)又名端粒酶卡侯氏体蛋白1(telomerase Cajal body protein 1, TCAB1)和WDR79,是2009年发现的端粒酶核心蛋白[1],其基因位于染色体17p13,并且与TP53基因以相反方向的形式有部分重叠。WRAP53β蛋白在进化中高度保守,蛋白质相对分子质量为65000,属于WD40家族。WD40蛋白家族是个蛋白大家族,涉及细胞凋亡、细胞周期调控、蛋白酶降解及RNA代谢等多个重要生化过程。WRAP53β是卡侯氏体的重要组成部分,没有WRAP53β蛋白,卡侯氏体结构会坍塌[2-5]。运动神经元生存蛋白(survival motor neuron protein, SMN)复合体和小卡体特定的RNA(small Cajal body-special RNA, scaRNA)家族(端粒酶RNA是其中的重要成员)定位于细胞核卡侯氏体内[6-8],参与卡侯氏体内RNA剪接及转录后的修改,在基因的转录调控、调节基因表达中起着重要作用。WRAP53β可通过同SMN复合体、端粒酶和scaRNA直接相互作用,介导三者从胞浆到胞核内卡侯氏体的转移,完成端粒酶在卡侯氏体中的聚集及端粒酶从卡侯氏体到端粒末端的运输,发挥端粒酶维持端粒长度的功能[9-11]。多项研究[12-15]发现在多种来源的肿瘤细胞中均有WRAP53β表达的显著升高,其过表达可促进细胞恶性转化,特异性的WRAP53β基因敲除可诱导肿瘤细胞凋亡,而WRAP53β水平升高与癌症预后不良有关。这些提示WRAP53β在肿瘤形成发展中起着重要作用。阻止WRAP53β蛋白的表达,可能会引起端粒变短、调节肿瘤基因表达、抑制细胞恶性转化等,最终达到治疗肿瘤的目的。WRAP53β也许将成为抗癌疗法的一个极具潜力的新靶标。目前对WRAP53β的了解还非常有限,如果能够揭示它与癌基因、炎症和免疫的相关性,无疑将对头颈肿瘤的联合生物治疗产生积极的推动作用。
近年来,大量研究采用算法从肿瘤转录组数据中推断免疫细胞的组成,由此估计的肿瘤免疫浸润与肿瘤的基因组和转录组变化有关[16-17],为了解肿瘤-转录-免疫相互作用提供了见解。基于前期研究报道,我们猜测WRAP53β与头颈肿瘤的发生发展密切相关,如果能证实这种猜测,或可为头颈肿瘤的联合生物治疗提供新策略。为证实以上猜测,我们首先进行大数据分析预测:利用TIMER软件[18-19]模块来探索WRAP53β表达与癌症基因组图谱TCGA队列头颈肿瘤的相关性,预测WRAP53β与免疫浸润、遗传或临床特征之间的关系,并通过TITCH[20]分析单细胞表达水平,通过CARE软件[21]探索WRAP53β靶向药物的协同作用;然后通过RNA干扰WRAP53β的表达验证其对细胞增殖能力和DNA损伤的作用;最后收集口腔鳞癌患者的标本进行验证。我们期望通过本研究,开发出WRAP53β协同癌基因共抑制的联合抗肿瘤疗法。
1. 材料和方法
1.1. TIMER网站预测WRAP53β基因在人头颈肿瘤中的特性
采用TIMER(Tumor Immune Estimation Resource)网站 https://cistrome.shinyapps.io/TIMER或升级版 http://TIMER.cistrome.org/TIMER2.0中Cancer Exploration模块,使用三大子模块Gene_DE、Gene_Mutation及Gene_Outcome分析WRAP53β在人头颈肿瘤中的表达、突变和生存结局。
1.2. TISCH网站分析WRAP53β在单细胞水平的表达
TISCH网站( http://tisch.comp-genomics.org/home)主要关注单细胞数据集研究免疫微环境。网站一共收集了79个数据集,大约200万个细胞。针对头颈肿瘤的数据集有两个,HNSC_GSE103322和HNSC_GSE139324,我们搜索WRAP53β基因在不同数据集、不同细胞类型的平均表达。
1.3. 预测WRAP53β与TP53基因通路的相互关系
前期实验筛选到肿瘤组和WRAP53β干扰组的差异基因[15]主要富集在p53信号传导通路、丝裂原活化蛋白激酶(mitogen-activated protein kinase, MAPK)信号通路/JAK-STAT信号通路/RNA降解信号通路中。因为WRAP53β是p53的天然反义转录物,因此我们感兴趣的是筛选到的p53信号传导通路中的基因,包括:ATM、ATR、CDK1、CDK6、CCNB1、CCNB2、MDM4、SESN1、SESN3、ZMAT3、FAS、GADD45A、GADD45B、CCND2、BBC3、IGFBP3、SERPINE1和TP53AIP13,进一步分析WRAP53β基因与它们的相关性。同时,由于WRAP53β是端粒酶的重要亚单位之一,本研究进一步分析了它与端粒酶反转录酶(telomerase reverse transcriptase, TERT)及其 RNA 模板(telomerase RNA component, TERC)的相关性。
1.4. WRAP53β与免疫浸润的相关性
肿瘤中的炎症因子高表达,如IL-6、IL-17、IL-22、TNF-α、NF-κB等[22-23],还有一些炎症因子与免疫细胞调控有关,包括IL-10、IL-6、IL-1、IFN-γ[24]等。本研究采用Gene_Corr分析了WRAP53β与12种常见促炎、抑炎因子的相关性,其中促炎因子包括:IL-1α、IL-1β、IL-2、IL-6、IL-17、NF-κB、TNF-α、IFN-γ、IL-23A;抑炎因子包括:IL-10、TGF-β、IL-22。进一步采用Immune Association模块,预测6种主要的浸润免疫细胞(B细胞、CD4+T细胞、CD8+T细胞、中性粒细胞、巨噬细胞和树突细胞)与WRAP53β基因的相关程度。
1.5. CARE软件预测WRAP53β共靶向的协同药物
CARE全称是计算抗性分析(Computational Analysis of REsistance),网址是 http://CARE.dfci.harvard.edu,药物数据库主要包括肿瘤细胞系百科全书(cancer cell line encyclopedi, CCLE),癌症治疗反应门户网站(the cancer therapeutics response portal, CTRP)和癌症基因组项目(cancer genome project, CGP)。CARE是一种针对肿瘤靶向治疗的计算方法,通过细胞系复合筛选推断药物效应的全基因组转录组特征,其输出的基因组评分用于预测药物靶向基因与其他基因的相互作用对复合筛选肿瘤抑制剂功效的影响[21]。CARE在推断基因生物标记物以预测靶向药物治疗和协同药物组合的有效性和可靠性方面已经得到了验证[18]。采用多数据库来搜索WRAP53β的靶向治疗反应。对于每种药物,它的CARE评分可以作为一种良好的反应模式。对于每个基因,CARE评分表明其基因的表达水平或突变情况改变与药物疗效之间的关系:正数表示较高的表达值与药物反应相关(即对药物敏感),而负数表示药物耐药性。通常采用t值(即CARE与靶基因表达的Pearson相关系数)代表CARE评分,通过评估药物靶点与基因的相互作用效应,来预测药物的有效性。本研究也在结果中报告t值。
1.6. 重组shRNA慢病毒敲降WRAP53β表达与检测
根据RNA干扰的设计原则合成针对WRAP53β的RNA干扰的靶点序列及非特异性对照序列,分别合成两条58 nt的寡核苷酸链,即由上海生博公司构建WRAP53β敲低的慢病毒载体(Plvt322),靶向位点为1452~1470(CCAATCAGCGCATCTACTT),无关序列病毒载体shNC(Plvt7)为非特异对照。将寡核苷酸链退火成双链DNA序列,经与载体pMagic7.1连接后转入感受态DH5α细胞,在抗性基因筛选、PCR克隆鉴定后,进一步在HEK293T细胞与病毒包装质粒和膜蛋白质粒共转染,进行病毒包装,测定病毒滴度。取本室保存的对数生长期的口腔鳞癌Cal27细胞接种于6孔板,每孔细胞数为1×105,用含10%小牛血清的DMEM细胞培养液于体积分数5% CO2培养箱中常规培养24 h。待其汇合率30%~50%时,换无血清培养液,并将细胞分为3组。shWRAP53β组细胞加入含shWRAP53β序列的重组慢病毒颗粒,shNC组细胞加入含非特异性对照序列的慢病毒颗粒,Con组为未处理的Cal27细胞。每组加入40 μL滴度为3×108 IU/mL的重组慢病毒,使感染复数(multiplicity of infection, MOI )为100,并同时加入感染增强剂polybrene,使终质量浓度为5 μg/mL。12 h后更换含10%小牛血清的DMEM继续培养。48 h后换含有0.5 mg/L嘌呤霉素的新鲜培养基筛选培养5~7 d,收集shWRAP53β稳定表达的细胞株备用。
1.6.1. 噻唑蓝(MTT)法测定细胞生长曲线
取shWRAP53β组、Con组及shNC组细胞,按1×104/100 μL加入96孔板,分别于1~7 d使用MTT法分析并在酶标检测仪上检测490 nm处的光密度(OD)值。重复3次取平均值描绘生长曲线。
1.6.2. 细胞免疫荧光实验检测各组细胞中P53蛋白的表达
1.6.1实验中Con组及shNC组的细胞生长曲线未见显著差别,表明慢病毒搭载的非特异性对照序列没有产生干扰作用,因此在1.6.2实验中只用shNC组作为对照组细胞。
将shWRAP53β与shNC的Cal27细胞按照每孔2×105接种于盖玻片上,并在体积分数5% CO2培养箱中培养24 h,用1%多聚甲醛固定10 min,0.2% Triton透化后加入10% BSA封闭1 h。分别加入P53一抗(1∶500稀释,Abcam, ab76242)4 ℃孵育过夜,PBS冲洗3次后加入异硫氰酸荧光素(FITC)标记的山羊抗兔二抗37 ℃避光孵育30 min,最后加入DAPI复染细胞核,荧光封片剂封片,于荧光显微镜下观察拍照,实验结果在采集图像时使用固定且相同的曝光时间。
1.6.3. Western blot半定量分析检测WRAP53β和γH2AX蛋白水平
Cal27细胞株经慢病毒处理后稳定筛选至28代,Western blot检测第18、23、28代shWRAP53β组和shNC组细胞的WRAP53β和DNA损伤蛋白的表达。收集细胞后经冰上裂解10~15 min,超声破碎后离心取上清液,采用Bradford法测定蛋白浓度。常规方法进行Western blot检测。分别加入5%牛血清白蛋白稀释的一抗:WRAP53β(1∶4000稀释,Novus, NB100-68252)、DNA损伤标志分子γ-H2AX(1∶1000稀释,Santa Cruz, sc-517348),将内参GAPDH(1∶1000稀释,Santa Cruz, sc-47724)用作内参。4 ℃孵育过夜,洗膜后加入辣根过氧化物酶标记的二抗,采用millipore公司ECL试剂盒于暗房中进行曝光显影。获得蛋白条带后扫描进入计算机,采用Image J图像分析软件对各条带进行分析,以各蛋白条带的灰度值代表蛋白的表达量。将目的蛋白的灰度值除以内参GAPDH的灰度值,进行归一化处理得到各条带的相对灰度值。
1.7. 免疫组织化学检测口腔鳞癌样本WRAP53β和γ-H2AX
经四川大学华西口腔医学院伦理委员会批准(批准号:WCHSIRB-D-2022-095),收集口腔鳞癌病例样本13例,口腔黏膜炎性病例样本7例,免疫组织化学检测WRAP53β(Proteintech, 14761-1-AP,1∶1000)和γ-H2AX (Proteintech, 10856-1-AP,1∶1000)的表达。标本固定后,常规石蜡包埋、切片,采用SP三步法,脱蜡复水,抗原修复,正常山羊血清封闭,分别经一抗、二抗孵育后,DAB室温显色封片观察。阳性细胞核呈现棕黄色或棕褐色。
1.8. 统计学方法
实验所得数据以
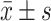 表示。采用重复测量资料方差检验和单因素方差检验进行分析。P<0.05为差异有统计学意义。Spearman’s rho(即Cor值)表征相关性程度水平,越接近于1相关度越高。
表示。采用重复测量资料方差检验和单因素方差检验进行分析。P<0.05为差异有统计学意义。Spearman’s rho(即Cor值)表征相关性程度水平,越接近于1相关度越高。
2. 结果
2.1. WRAP53β在头颈肿瘤表达、突变和生存分析
Gene_DE module分析结果表明,WRAP53β在多种不同肿瘤细胞较正常细胞中显著高表达,且在头颈鳞癌中呈现类似的高表达水平(图1A)。同时,Gene_Mutation module分析WRAP53β基因在头颈肿瘤中并未检测到突变,仅有野生型表达。然而,WRAP53β表达量的高低对5年生存曲线没有显著影响(P=0.37)(图1B),但与肿瘤分期有关。
图 1.

The differential expression of WRAP53βin different tumor types and the survival outcome in HNSC
WRAP53β基因在不同类型肿瘤中的差异表达及其在头颈肿瘤的生存结局
A: The distribution of gene WRAP53β expression in tumors or normal tissues is shown in small red or blue boxes, and its expression in HNSC is shown in large red boxes. *P<0.05, *P<0.01,***P<0.001. HNSC: Squamous cell carcinoma of the head and neck; HPV: Human papillomavirus; TPM: Transcripts per million. B: Kaplan-Meier analysis showed the overall survival rate of HNSC patients with different expression ofWRAP53β(n=522, in which 76 patients were lost to the study and 191 patients died).
在HNSC_GSE103322中有恶性细胞的转录组数据,UMAP图显示HNSC_GSE103322中不同细胞亚群的聚类情况(图2A),其中WRAP53β在恶性肿瘤细胞中均表达最高(图2B),但该基因在两个数据集HNSC_GSE103322和HNSC_GSE139324的免疫细胞散点图中表达水平极低或无表达(图2A~2D)。小提琴图的高度也反映WRAP53β基因在单细胞水平表达量的高低,在恶性细胞中表达最高(图2E红线指示)。
图 2.

Analysis of single cell sequencing clustering and WRAP53β expression
肿瘤细胞的单细胞测序聚类与WRAP53β表达分析
A: UMAP showed the clustering of different cell subsets in HNSC_GSE103322; B: the distribution of WRAP53β expression in Fig A; C: UMAP showed the clustering of different cell subsets inHNSC_GSE139324; D: the distribution of WRAP53β expression in Fig C; E: The expression of WRAP53β in different cell subsets in HNSC_GSE103322 and HNSC_GSE139324.
2.2. WRAP53β基因与p53通路相关基因、 端粒酶亚单位的相关性
采用Gene_Corrmodule模块,选择转录组测序的多个基因验证和WRAP53β的相关性。综合分析Cor值和P值,WRAP53β基因表达在HNSC中与p53通路基因ATM、ATR、BBC3、细胞周期蛋白B1基因(cyclin B1, CCNB1)、CCNB2、周期蛋白依赖性激酶基因(cyclin-dependent kinase 1, CDK1)及MDM4表达呈正相关,与GADD45B呈负相关;与CDK6、SESN1、SESN、ZMAT3、FAS、GADD45A、CCND2、IGFBP3、SERPINE1及TP53AIP1无显著相关性。P<0.05的p53信号传导通路基因和端粒酶亚单位(TERC、TERT)均列入了表1。其中,相关程度Cor值>0.5的基因包括CCNB1、CCNB2及CDK1。同时,TERT与TERC均显示与WRAP53β表达呈正相关。
表 1. The correlation of WRAP53β and p53 in HNSC (Cor value) .
WRAP53β基因与HNSC的p53通路基因之间的相关性(Cor值)
| Targeted gene | ATM | ATR | BBC3 | CCNB1 | CCNB2 | CDK1 | GADD45B | MDM4 | TERC | TERT |
| ATM: Ataxia telangiectasia-mutated gene; ATR: Ataxia telangiectasia and Rad3-related; BBC3: BCL2 binding component 3; CCNB1: Cyclin B1; CCNB2: Cyclin B2; CDK1: Cyclin-dependent kinase 1; GADD45B: Growth arrest and DNA-damage-inducible β; MDM4: Murine double minute 4; TERC: Telomerase RNA component; TERT: Telomerase reverse transcriptase. | ||||||||||
| WRAP53β | 0.17 | 0.213 | 0.243 | 0.526 | 0.532 | 0.512 | −0.117 | 0.178 | 0.155 | 0.229 |
2.3. WRAP53β和免疫浸润的关系
WRAP53β基因与炎症因子的相关性较弱,仅与少数炎性因子的相关性具有统计学意义,但Cor值仅为0.1左右。按照Cor值>0.2统计相关性,在HNSC-HPV+中WRAP53β与IFN-γ、IL-23A呈正相关,与IL-1A、IL-6呈负相关(表2)。在HNSC肿瘤中,WRAP53β基因与B细胞、CD8+ T 细胞、CD4+T细胞、巨噬细胞、中性粒细胞和树突状细胞均没有明显的相关性,Cor 值仅为0.1左右。
表 2. The Spearman correlation analysis of WRAP53β and inflammatory factors (Cor value) .
WRAP53β与差异炎症因子的相关性分析(Cor值)
| Cancer | IFN-γ | IL-1A | IL-22 | IL-23A | IL-6 | TGF-β |
| * Positive correlation, P<0.05; # Negative correlation,P<0.05. IFN-γ: Interferon γ; IL-1A: Interleukin 1A; IL-22: Interleukin 22; IL-23A: Interleukin 23A; IL-6: Interleukin 6; TGF-β: Transforming growth factor β. | ||||||
| HNSC (n=522) | 0.121 | −0.114 | −0.092 | 0.073 | 0.012 | −0.022 |
| HNSC-HPV− (n=422) | 0.031 | −0.228 | −0.114 | −0.017 | 0.133 | 0.117 |
| HNSC-HPV+ (n=98) | 0.244* | −0.214# | −0.151 | 0.302* | −0.294# | −0.120 |
2.4. WRAP53β共抑制药物的筛选预测
经CARE分析评估,CCLE、CGP和CTRP数据库分别筛选得到了7、45和53个靶向基因的抑制剂与WRAP53β基因表达具有相关性,进一步筛选出WRAP53β对CGP数据库包含的ATM、CDK1、MDM4协同抑制剂表现敏感(表3);同时,也对CTRP数据库包含ATM和TERT抑制剂敏感。以t值推断共抑制效果最强的3种药物分别是PHA-793887、CP-466722、58131-57-0。
表 3. The screening of WRAP53β co-inhibitory drugs.
WRAP53β共抑制药物筛选
| Database | Drug | Targeted gene | t | P |
| CGP: Cancer genome project; CTRP: The cancer therapeutics response portal; ATM: Ataxia telangiectasia-mutated gene; CDK1: Cyclin-dependent kinase 1; MDM4: Murine double minute 4; TERT: Telomerase reverse transcriptase. | ||||
| CGP | CP-466722 | ATM | 5.20 | 0.00 |
| AT7519 | CDK1 | 4.02 | 0.00 | |
| PHA-793887 | CDK1 | 5.40 | 0.00 | |
| 58131-57-0 | MDM4 | 4.19 | 0.00 | |
| CTRP | KU-55933 | ATM | 2.58 | 0.01 |
| KU-60019 | ATM | 2.61 | 0.01 | |
| Telomerase inhibitor Ⅸ | TERT | 3.06 | 0.00 | |
2.5. WRAP53β敲低对Cal27细胞增殖能力、P53表达和DNA损伤的影响
MTT结果显示(图3A),ShWRAP53β组较Con组、shNC组的细胞增殖能力显著降低。而对照组shNC与Con组细胞增殖能力无显著差异。细胞免疫荧光检测示(图3B),shWRAP53β组的Cal27细胞中P53的荧光强度显著低于shNC组,表明干扰WRAP53β后P53的表达水平明显下调。Western blot结果显示(图3C),与对照组相比,ShWRAP53β组WRAP53β干扰效应可以随着细胞代数增加而稳定传递。而γ-H2AX表达与对照组相比轻微降低,仅第18代、第28代差异有统计学意义(P<0.05)。
图 3.

The effect of knocking down shWRAP53 on the cell viability, P53 expression and DNA damage of Cal27
WRAP53β敲低对Cal27细胞增殖、P53蛋白表达和DNA损伤的影响
A: MTT showed the cell viability of shWRAP53 Cal27 (n=3); B: The effect of shWRAP53β on P53; C: The effect of shWRAP53β on DNA damage (n=3). *P<0.05, vs. Con group.
2.6. 临床组织标本中WRAP53β和γ-H2AX的表达
免疫组织化学染色结果如图4所示,WRAP53β主要表达于细胞核,在口腔鳞癌中的阳性表达检出率为15.4%(2/13),但未在口腔黏膜炎病理标本中检出(0/7)。γ-H2AX主要表达于细胞核,在口腔鳞癌中的阳性表达检出率为92.3%(12/13),而在炎性标本中为28.6%(2/7)。
图 4.

Immunohistochemistry results of the expressions of WRAP53β and γ-H2AX in oral tissue pathology specimens
免疫组织化学检测口腔组织病理标本中WRAP53β和γ-H2AX的表达情况
3. 讨论
WRAP53β在不同动物中高度保守。WRAP53β在80%的鼻咽癌临床样本中过表达,仅在15%的鼻咽炎样本中表达[15]。口腔鳞癌细胞系中WRAP53β的蛋白表达水平显著高于正常细胞系,同样地,我们进一步发现WRAP53β在13例口腔鳞癌临床病理组织样本中有2例阳性检出,在7例口腔黏膜炎样本中并无阳性发现,本研究中口腔鳞癌WRAP53β检出率较低的原因可能与样本量不足有关,后续将进一步增加样本量进行验证。肺腺癌肿瘤直径大于3.0 cm的WRAP53β过表达率明显高于肿瘤直径小于3.0 cm的患者[20]。并且在多种类型肿瘤中均呈过表达[23-24]。WRAP53β是端粒酶的核心组分,在肿瘤形成发展中起着重要作用。WRAP53β对帮助端粒酶复合体在卡侯体的定位和端粒合成必不可少[1, 12, 25]。阻止WRAP53β蛋白的表达,可能会引起端粒变短、调节肿瘤基因表达、抑制细胞恶性转化等,最终达到治疗肿瘤的目的。端粒酶的活性依赖于WRAP53β,它反过来控制着远端三通连接CR4/5的构象[26]。虽然目前尚无有效的端粒酶抑制剂,但WRAP53β与相关基因如TERT共靶向或许将成为抗癌疗法的一个极有价值的方案,本研究CARE筛选结果也初步支持了这一观点,但进一步的验证工作还有待展开。
WRAP53β是p53的天然反义转录物(natural antisense transcript, NAT)。p53诱导激活后被抑制的关键基因有CCNB1、CCNB2及CDK1等。WRAP53β、CCNB1/2、CDK1等已经被发现或证实为p53-DREAM通路的靶基因[27]。本研究中,TIMER分析结果揭示了它们的关系,CARE预测分析结果也发现CDK1的抑制剂对WRAP53β可能存在协同抑制作用。而 MDM4抑制剂对WRAP53β存在共抑制效应的原因可能与其阻止MDM4对p53的抑制作用有关,该机制仍有待进一步探究。
ATM(ataxia telangectasia mutated)和ATR(ATM and Rad3-related)是DNA损伤反应中重要的效应因子,细胞在其DNA遭到损害时,可以通过ATM/ATR等激酶感受器分子感知DNA的损伤,并将DNA损伤信号传导到下游靶蛋白,通过级联放大反应,启动应急系统,产生细胞周期阻滞,从而完成DNA修复或启动细胞凋亡程序[28]。本研究使用TIMER预测协同基因,CARE筛选敏感药物,均显示ATM抑制剂可能对WRAP53β存在共抑制效应。WRAP53β是DNA双链断裂修复的支架,作为DNA损伤修复的必需调节因子,促进E3连接酶RNF8在损伤位点的积累,促进损伤修复复合物的高效组装,均是以ATM或ATR依赖的方式[7]。WRAP53β以依赖于ATM、ATR、H2AX和MDC1的方式迅速募集到DNA损伤区域,并在DNA断裂和周围区域累积[29]。本研究发现,与对照组细胞相比,shWRAP53β组细胞生长缓慢,P53表达减弱,WRAP53β表达减弱,DNA损伤标志物γ-H2AX的表达仅发生轻微减弱;临床病理组织样本显示口腔鳞癌组织中WRAP53β阳性可检出,γ-H2AX的阳性检出率高。这可能与ATM之外的DNA损伤修复通路有关。WRAP53β通过非同源末端连接和同源重组损害了双链DNA断裂的修复,导致DNA损伤的积累并延迟了G2/M细胞周期阻滞的恢复[30-31]。相关文献也发现,siRNA沉默WRAP53β基因可显著降低 p53 mRNA表达,抑制p53诱导的DNA损伤[32]。
本课题将shWRAP53β组细胞传代至第28代,并分别与第23、第18代细胞总蛋白共同行Western blot检测,结果发现,第28代细胞的WRAP53β表达较第18代显著下降,提示对WRAP53β基因的沉默效应具有持久性及放大性,这种效应可能持续抑制端粒酶活性,也可能导致端粒缩短等级联效应。
细胞因子TNF-α和IFN-γ可增加口腔癌细胞系Ca9-22端粒酶活性,IL-1β可降低其活性。提示长期慢性炎症可能会增加端粒酶活性,从而促进口腔黏膜上皮的恶性转化。但尚没有相关文献表明WRAP53β与免疫浸润和炎症因子水平有关,采用 TIMER分析也未发现它们之间的显著联系,提示炎症因子与免疫细胞可能不是WRAP53β的共抑制靶点。然而,免疫细胞和炎症因子是复杂的网络,能否对WRAP53β进行调控以及如何调控需要更深入的研究。
本研究通过生物信息学技术分析了WRAP53β在头颈鳞癌的临床、基因组和免疫浸润特征,并在细胞水平证实WRAP53β基因沉默能够显著抑制口腔鳞癌细胞的增殖能力和P53的表达。CARE评分结果提示ATM、CDK1和MDM4的抑制剂与WRAP53β可能存在共靶向协同抗肿瘤效应,然而本研究尚未对该预测结果进行实验验证;大数据预测结果的Cor值偏低,其对相关性的说服力也不能令人满意;临床样本过少也可能导致取样误差和暴露偏倚,加强而不是矫正了我们推断中存在的偏见。尽管目前取得了一些研究进展,但头颈肿瘤的联合疗法尚需更多的证据。
* * *
利益冲突 所有作者均声明不存在利益冲突
Funding Statement
四川大学大学生创新创业训练计划项目(No. C2019105015、No. C2021116672)和四川大学华西口腔医院探索与研发项目(No. RD-02-202010)资助
Contributor Information
羽菲 姚 (Yu-fei YAO), Email: feiyuyao@126.com.
琨 王 (Kun WANG), Email: wangk0613@126.com.
References
- 1.VENTEICHER A S, ABREU E B, MENG Z, et al A human telomerase holoenzyme protein required for Cajal body localization and telomere synthesis. Science. 2009;323(5914):644–648. doi: 10.1126/science.1165357. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.LEE J H, LEE Y S, JEONG S A, et al Catalytically active telomerase holoenzyme is assembled in the dense fibrillar component of the nucleolus during S phase. Histochem Cell Biol. 2014;141(2):137–152. doi: 10.1007/s00418-013-1166-x. [DOI] [PubMed] [Google Scholar]
- 3.YUAN X S, CAO L X, HU Y J, et al. Clinical, cellular, and bioinformatic analyses reveal involvement of WRAP53 overexpression in carcinogenesis of lung adenocarcinoma. Tumour Biol, 2017, 39 (3): 1010428317694309[2021-05-11]. https://doi.org/10.1177/1010428317694309.
- 4.MAHMOUDI S, HENRIKSSON S, FARNEBO L, et al. WRAP53 promotes cancer cell survival and is a potential target for cancer therapy. Cell Death Dis, 2011, 2 (1): e114−e114[2021-04-13]. https://doi.org/10.1038/cddis.2010.90.
- 5.GARVIN S, TIEFENBÖCK K, FARNEBO L, et al Nuclear expression of WRAP53β is associated with a positive response to radiotherapy and improved overall survival in patients with head and neck squamous cell carcinoma. Oral Oncol. 2015;51(1):24–30. doi: 10.1016/j.oraloncology.2014.10.003. [DOI] [PubMed] [Google Scholar]
- 6.RASSOOLZADEH H, BÖHM S, HEDSTRöM E, et al. Overexpression of the scaffold WD40 protein WRAP53β enhances the repair of and cell survival from DNA double-strand breaks. Cell Death Dis, 2016, 7 (6): e2267-e2267[2021-04-13]. https://doi.org/10.1038/cddis.2016.172.
- 7.HENRIKSSON S, RASSOOLZADEH H, HEDSTRÖM E, et al The scaffold protein WRAP53β orchestrates the ubiquitin response critical for DNA double-strand break repair. Genes Dev. 2014;28(24):2726–2738. doi: 10.1101/gad.246546.114. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.MAHMOUDI S, HENRIKSSON S, WEIBRECHT I, et al. WRAP53 is essential for Cajalbodyformation and for targeting the survival of motor neuron complex to Cajalbodies. PLoS Biol, 2010, 8 (11): e1000521[2021-05-11]. https://doi.org/10.1371/journal.pbio.1000521.
- 9.陆文昕, 葛奕辰, 肖丽英, 等 WD40编码的P53 RNA反义转录子基因和蛋白及其功能. 国际口腔医学杂志. 2016;43(2):244–248. doi: 10.7518/gjkq.2016.02.028. [DOI] [Google Scholar]
- 10.HENRIKSSON S, FARNEBO M. On the road with WRAP53β: Guardian of Cajal bodies and genome integrity. Front Genet, 2015, 6: 91[2021-05-11]. https://doi.org/10.3389/fgene.2015.00091.
- 11.TAPIA O, BENGOECHEA R, PALANCA A, et al Reorganization of Cajal bodies and nucleolar targeting of coilin in motor neurons of type I spinal muscular atrophy. Histochem Cell Biol. 2012;137(5):657–667. doi: 10.1007/s00418-012-0921-8. [DOI] [PubMed] [Google Scholar]
- 12.TYCOWSKI K T, SHU M D, KUKOYI A, et al A conserved WD40 protein binds the Cajal body localization signal of scaRNP particles. Mol Cell. 2009;34(1):47–57. doi: 10.1016/j.molcel.2009.02.020. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.RAO X, HUANG D, SUI X, et al. Overexpression of WRAP53 is associated with development and progression of esophageal squamous cell carcinoma. PLoS One, 2014, 9 (3): e91670[2021-05-11]. https://doi.org/10.1371/journal.pone.0091670.
- 14.HEDSTRÖM E, PEDERIVA C, FARNEBO J, et al. Downregulation of the cancer susceptibility protein WRAP53β in epithelial ovarian cancer leads to defective DNA repair and poor clinical outcome. Cell Death Dis, 2015, 6 (10): e1892−e1892[2021-04-13]. https://doi.org/10.1038/cddis.2015.250.
- 15.SUN C K, LUO X B, GOU Y P, et al. TCAB1: A potential target for diagnosis and therapy of head and neck carcinomas. Mol Cancer, 2014, 13 (1): 180[2021-05-11]. https://doi.org/10.1186/1476-4598-13-180.
- 16.BECHT E, GIRALDO N A, LACROIX L, et al. Estimating the population abundance of tissue-infiltrating immune and stromal cell populations using gene expression. Genome Biol, 2016, 17 (1): 218[2021-05-11]. https://doi.org/10.1186/s13059-016-1070-5.
- 17.NEWMAN A M, LIU C L, GREEN M R, et al Robust enumeration of cell subsets from tissue expression profiles. Nat Methods. 2015;12(5):453–457. doi: 10.1038/nmeth.3337. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.LI T, FU J, ZENG Z, et al TIMER2.0 for analysis of tumor-infiltrating immune cells. Nucleic Acids Res. 2020;48(W1):W509–W514. doi: 10.1093/nar/gkaa407. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.LI T, FAN J, WANG B, et al. TIMER: A web server for comprehensive analysis of tumor-infiltrating immune cells. Cancer Res, 2017, 77 (21): e108−e110[2021-05-11]. https://doi.org/10.1158/0008-5472.CAN-17-0307.
- 20.SUN D, WANG J, HAN Y, et al TISCH: A comprehensive web resource enabling interactive single-cell transcriptome visualization of tumor microenvironment. Nucl Acids Res. 2020;49(D1):D1420–D1430. doi: 10.1093/nar/gkaa1020. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.JIANG P, LEE W, LI X, et al. Genome-scale signatures of gene interaction from compound screens predict clinical efficacy of targeted cancer therapies. Cell Syst, 2018, 6 (3): 343−354.e5[2021-05-11]. https://doi.org/10.1016/j.cels.2018.01.009.
- 22.YUAN X, CAO L, HU Y, et al. Clinical, cellular, and bioinformatic analyses reveal involvement of WRAP53 overexpression in carcinogenesis of lung adenocarcinoma. Tumour Biol, 2017 , 39(3): 1010428317694309[2021-05-11]. https://doi.org/10.1177/1010428317694309.
- 23.SHAY J, WRIGHT W Telomeres and telomerase: three decades of progress. Nat Rev Genet. 2019;20(5):299–309. doi: 10.1038/s41576-019-0099-1. [DOI] [PubMed] [Google Scholar]
- 24.BINNEWIES M, ROBERTS E W, KERSTEN K, et al. Understanding the tumor immune microenvironment (TIME) for effective therapy. Nat Med, 2018, 24(5): 541-550.
- 25.RAI A, NAIKMASUR V G, SATTUR A Quantification of telomerase activity in normal oral mucosal tissue and oral squamous cell carcinoma. Indian J Med Paediatr Oncol. 2016;37(3):183–188. doi: 10.4103/0971-5851.190350. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.CHEN L, ROAKE C M, FREUND A, et al An activity switch in human telomerase based on RNA conformation and shaped by TCAB1. Cell. 2018;174(1):218–230. doi: 10.1016/j.cell.2018.04.039. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27.ENGELAND K Cell cycle arrest through indirect transcriptional repression by p53: I have a DREAM. Cell Death Differentiation. 2018;25(1):114–132. doi: 10.1038/cdd.2017.172. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 28.CIMPRICH K A, CORTEZ D ATR: An essential regulator of genome integrity. Nat Rev Mol Cell Biol. 2008;9(8):616–627. doi: 10.1038/nrm2450. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.COUCORAVAS C, DHANJAL S, HENRIKSSON S, et al Phosphorylation of the Cajal body protein WRAP53β by ATM promotes its involvement in the DNA damage response. RNA Biol. 2017;14(6):804–813. doi: 10.1080/15476286.2016.1243647. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 30.RASSOOLZADEH H, COUCORAVAS C, FARNEBO M The proximity ligation assay reveals that at DNA double-strand breaks WRAP53β associates with γH2AX and controls interactions between RNF8 and MDC1. Nucleus. 2015;6(5):417–424. doi: 10.1080/19491034.2015.1106675. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 31.SANCHEZ-MORAN I, RODRIGUEZ C, LAPRESA R, et al. Nuclear WRAP53 promotes neuronal survival and functional recovery after stroke. Sci Adv, 2020, 6(41): eabc5702[2021-05-11]. https://doi.org/10.1126/sciadv.abc5702.
- 32.MAHMOUDI S, HENRIKSSON S, CORCORAN M, et al WRAP53, a natural p53 antisense transcript required for p53 induction upon DNA damage . Mol Cell. 2009;33(4):462–471. doi: 10.1016/j.molcel.2009.01.028. [DOI] [PubMed] [Google Scholar]


