Abstract
目的
研究大脑中动脉栓塞(MCAO)大鼠不同造模时程外周血的特异表达基因及特征共表达网络模块。
方法
利用GEO数据库的基因表达谱芯片GSE119121联合R语言分析MCAO造模后不同时间点(0、1、2、3、6及24 h)外周血的差异表达基因。通过STEM工具筛选不同造模时程基因表达模式。利用基因本体(GO)数据库和京都基因与基因组百科全书(KEGG)数据库对差异表达的基因进行功能注释和通路富集。在R语言环境下利用CEMiTool包对基因表达谱矩阵进行共表达网络构建及模块分析,并将模块与不同造模时间点进行富集分析。
结果
与造模0 h相比,造模后1、2、3、6及24 h差异表达基因数分别为163、502、527、550、75。共有38种基因表达模式被富集,其中模式65和模式34分别在2~6 h特异上调或下调, Hp、 Nos2、 P2ry10及 Klf12为两种模式的代表性基因。共表达网络模块分析显示,造模急性期早期(1~6 h)基因状态与模块2正相关,造模1~3 h基因状态与模块3正相关,造模2~6 h基因变化与模块4正相关。基因模块6随着造模时间的迁移,与各时间点从正相关(0~2 h)逐渐转为负相关(3~24 h),模块6主要与病毒应答及固有免疫应答相关,其网络核心节点包括 Mx1、 Mx2、 Rtp4等基因。
结论
本研究初步筛选了缺血性脑卒中急性发病期大鼠外周血的特征基因及动态共表达网络模块,为探究缺血性脑卒中的病理生理变化规律提供了依据。
Abstract
Objective
To identify the time dependent profiles of gene expression and featured co-expression network modules in peripheral blood of rats after middle cerebral artery occlusion (MCAO).
Methods
Microarray GSE119121 from GEO database was analyzed by R language to identify the significantly changed genes in peripheral blood at different time points (0, 1, 2, 3, 6 and 24 h) after MCAO. Gene expression patterns at different time courses were screened by STEM tools. Then, function annotation and pathway enrichment of differentially expressed genes (DEGs) were performed using the Gene Ontology (GO) database and the Kyoto Gene and Genomic Encyclopedia (KEGG) database. Depending on CEMiTool package, gene expression profile matrix was inputted into R to construct the co-expression networks and to analyze modules, and enrichment analysis was conducted to evaluate the correlation between the modules and different time points.
Results
Comparing with gene at 0 h, the numbers of DEGs in peripheral blood at different time points after MCAO were 163 (1 h), 502 (2 h), 527 (3 h), 550 (6 h), and 75 (24 h), respectively. Moreover, a total of 38 gene expression patterns were enriched, and pattern 65 and pattern 34 were specifically up-regulated or down-regulated at 2-6 h. Hp, Nos2, P2ry10, and Klf12 were representative genes of these two models. The co-expression network module analysis showed that the gene status in the early acute phase (1-6 h) was positively correlated with the Module 2. Module 3 and Module 4 was positively correlated with phase phase 1-3 h and 2-6 h, respectively. Noteworthy, Module 6 gradually changed from positive correlation (0-2 h) to negative correlation (3-24 h) with the MCAO time course, and Module 6 was mainly related to viral response and innate immune response. The hub genes of Module 6 included Mx1, Mx2, and Rtp4.
Conclusion
Our study has identified the featured genes and dynamic co-expression network modules in peripheral blood after acute ischemic stroke, which provides a potential basis for judging the onset time of ischemic stroke.
Keywords: Hypoxia-ischemia, brain/blood; Stroke/blood; Gene expression; Gene regulatory networks
脑卒中已成为威胁人类健康的第三大“杀手”,具有高发病率、高致残率和高病死率等特点 [ 1] 。中国是全世界脑卒中高发病率国家之一,国内首发脑卒中患病率每年增加6.5 %,且发病年龄呈年轻化趋势,严重危害患者的健康和生活质量 [ 2] 。缺血性脑卒中是脑卒中的主要类型,通常由脑内动脉栓塞造成局部供血障碍引起,最终导致脑组织缺血坏死。目前,唯一被证明对治疗缺血性脑卒中有效的方法是采用重组组织型溶酶原激活剂(recombinant tissue plasminogen activator, rt-PA)超早期溶栓,通过恢复缺血区脑组织血液供应发挥疗效。然而,rt-PA的有效治疗时间窗仅为发病4.5 h内,极大地限制了其应用 [ 3] 。由于病情诊断、初始发病时间、脑内出血风险等因素的不确定性,临床上rt-PA的合理使用并不理想 [ 4- 5] 。
缺血性脑卒中的进展是一个复杂的动态过程,其病理机制尚未完全明确。除了局部脑组织缺血坏死,缺血性脑损伤还可引起外周血细胞因子及趋化因子等信号快速变化,同时外周血单核细胞、红细胞、血小板等的状态也随着病情进展而发生改变 [ 6- 8] 。因此,通过筛选缺血性脑损伤急性发病期不同时间点外周血的关键基因及分子网络变化,可为探索缺血性脑损伤的病理机制提供线索。基于此,本研究利用生物信息学技术分析大脑中动脉栓塞(middle cerebral artery occlusion, MCAO)大鼠不同造模时程外周血基因表达谱,筛选不同时程特异表达的基因及特征分子共表达网络,进而挖掘关键网络节点,以探究缺血性脑损伤的动态进展过程及病理生理变化的规律。
在美国国立生物技术信息中心(NCBI)平台的基因表达综合数据库(GEO, https://www.ncbi.nlm.nih.gov/geo/)中筛选得到缺血性脑损伤造模后不同时间点外周血基因表达谱芯片GSE119121 [ 9] ,并下载其分组信息及探针表达谱文件“GSE119121_series_matrix.txt.gz”。芯片平台为GPL6247,芯片含大鼠大脑MCAO造模后0、1、2、3、6及24 h外周血细胞基因表达谱,除了6 h为7只大鼠的数据,其余时间点均为8只大鼠的数据。
将芯片样本分成0、1、2、3、6及24 h六个组别,在R3.5.1()环境下,利用limma包 [ 10] 将每个组别分别与0 h进行比较,获取差异基因。筛选条件为|log差异倍数|≥1,校正后的 P值水平0.05。得到5份显著差异表达基因的列表。
合并不同时间点的差异基因,利用时间序列分析软件Short Time-series Expression Miner(STEM, v1.3.11) [ 11] 设置100种表达模式,最大单位变化设为1,将差异基因标准化,0 h基因表达值设为1,计算不同时间点的相对表达值并进行表达模式富集,富集模式设为默认值。彩色模块为显著富集表达模式。
将差异基因列表分别导入DAVID 6.8( https://david.ncifcrf.gov)在线基因功能富集工具,对其进行基因本体和京都基因与基因组百科全书(KEGG)富集分析,得到对应的富集功能与信号通路信息。在R3.5.1环境下,利用GOplot包的GOCircle功能对富集结果进行可视化分析,按 P值取显著性前10位的功能及信号通路。
在R3.5.1环境下,利用CEMiTool包 [ 12] 对基因表达谱矩阵进行共表达网络模块构建及分析。首先,从STRING v11.0(string-db.org)导出该基因表达谱对应的大鼠蛋白互作用关系文件,并作为CEMiTool包进行共表达模块网络分析的背景文件。利用CEMiTool包输出共表达模块网络分析结果,包括样本聚类树(检验输入样本差异程度)、β与 R 2平均连通性图(选定软阈值,以筛选无标度网络模块)、模块富集评分图( P值大于0.05不显示)、模块基因表达折线图(不同组别的模块基因表达量变化可视化)、模块基因共表达及互作用网络图(Degree值越大,节点直径越大,颜色越深)。
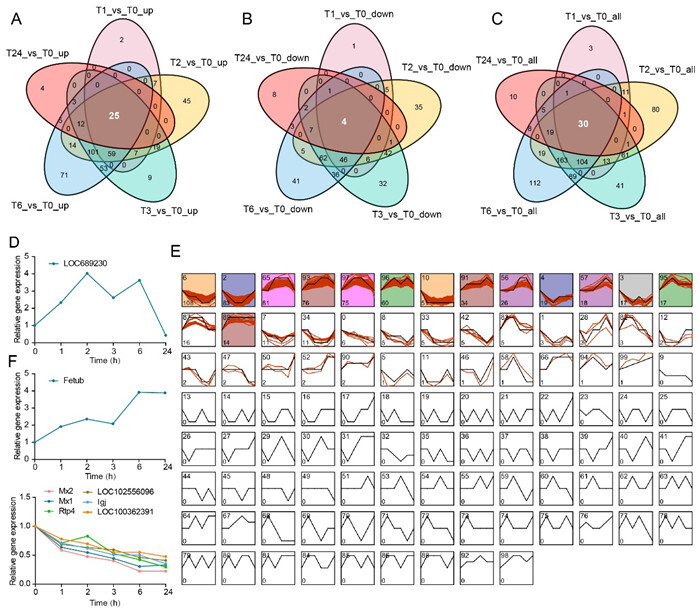
差异基因分析显示,与造模0 h相比,造模后1、2、3、6及24 h差异基因数目分别为163、502、527、550、75,造模后各时间点上调及下调倍数前50位的差异基因如 附表 1~ 10所示。其中,五个时间点共有25个共同上调差异基因( 附图 1A)以及4个共同下调差异基因( 附图 1B)。此外,差异变化基因的交集显示各时间点共有30个差异变化基因( 附图 1C),其中 LOC689230在1~6 h上调,在24 h下调( 附图 1D),提示LOC689230的表达模式可能与脑缺血造模急性期的机体状态相关。
表附表 1 大脑中动脉栓塞模型大鼠造模后1 h外周血上调倍数前50位基因
Table 附表 1 List of the top 50 upregulated genes in peripheral blood at 1 hour of middle cerebral artery occlusion rats
|
上调基因 |
Log差异倍数 |
校正后的 P值 |
|
Fpr1 |
2.37 |
1.74×10 -9 |
|
Il1r2 |
2.29 |
3.88×10 -12 |
|
Mrgprx3 |
2.16 |
3.39×10 -12 |
|
Mmp9 |
2.06 |
1.85×10 -9 |
|
Ifitm6 |
1.89 |
4.06×10 -8 |
|
Olfm4 |
1.78 |
2.24×10 -7 |
|
Amica1 |
1.75 |
2.50×10 -8 |
|
Bmx |
1.69 |
4.32×10 -9 |
|
Mir223 |
1.68 |
1.37×10 -7 |
|
Ldhc |
1.64 |
4.71×10 -10 |
|
LOC679818 |
1.63 |
1.98×10 -8 |
|
Fkbp5 |
1.62 |
1.87×10 -11 |
|
Pram1 |
1.55 |
9.02×10 -10 |
|
Hp |
1.52 |
4.51×10 -8 |
|
Stfa3 |
1.51 |
0.000505 |
|
Cd33 |
1.51 |
8.86×10 -9 |
|
Cd177 |
1.50 |
1.47×10 -6 |
|
Alox5 |
1.50 |
8.04×10 -9 |
|
Ugt8 |
1.49 |
5.65×10 -9 |
|
Dhrs9 |
1.49 |
1.78×10 -9 |
|
Clec5a |
1.46 |
1.21×10 -6 |
|
Mmp25 |
1.45 |
1.24×10 -8 |
|
Nlrp12 |
1.44 |
1.87×10 -9 |
|
Mcemp1 |
1.44 |
1.39×10 -9 |
|
Camp |
1.44 |
2.04×10 -8 |
|
LOC24906 |
1.43 |
4.82×10 -10 |
|
Mir292 |
1.43 |
4.95×10 -7 |
|
Dgat2 |
1.41 |
1.43×10 -8 |
|
Il18rap |
1.40 |
2.64×10 -9 |
|
Cpm |
1.39 |
3.11×10 -7 |
|
Oas1k |
1.38 |
1.32×10 -8 |
|
Pglyrp4 |
1.38 |
4.83×10 -9 |
|
Adgrg3 |
1.37 |
1.48×10 -10 |
|
Tnnc1 |
1.36 |
1.49×10 -6 |
|
Fcar |
1.36 |
1.08×10 -9 |
|
P2ry13 |
1.36 |
1.08×10 -9 |
|
Tcn2 |
1.34 |
2.85×10 -10 |
|
Rab44 |
1.34 |
1.51×10 -10 |
|
Nos2 |
1.33 |
8.04×10 -9 |
|
LOC690020 |
1.33 |
4.47×10 -6 |
|
Hk3 |
1.33 |
9.13×10 -11 |
|
LOC102557597 |
1.32 |
2.23×10 -6 |
|
Lcn2 |
1.32 |
7.49×10 -5 |
|
Aqp9 |
1.30 |
1.41×10 -7 |
|
Rgs1 |
1.28 |
0.001199 |
|
Rbm47 |
1.26 |
3.45×10 -8 |
|
Tarm1 |
1.25 |
1.10×10 -5 |
|
Alox5ap |
1.24 |
1.71×10 -9 |
|
LOC689230 |
1.22 |
5.29×10 -5 |
|
Uaca |
1.22 |
3.15×10 -8 |
表附表 10 大脑中动脉栓塞模型大鼠造模后24 h外周血下调基因
Table 附表 10 List of downregulated genes in peripheral blood at 24 hour of middle cerebral artery occlusion rats
|
下调基因 |
Log差异倍数 |
校正后的 P值 |
|
Mx2 |
-2.15 |
2.53 ×10-11 |
|
Rnase6 |
-1.84 |
1.87 ×10-7 |
|
Rtp4 |
-1.76 |
2.49 ×10-9 |
|
Mx1 |
-1.59 |
2.49 ×10-9 |
|
Igj |
-1.50 |
1.23 ×10-7 |
|
MGC108823 |
-1.34 |
1.38 ×10-6 |
|
LOC102556096 |
-1.29 |
2.92 ×10-6 |
|
Eno3 |
-1.26 |
3.02 ×10-7 |
|
Oas1b |
-1.21 |
7.97 ×10-7 |
|
LOC689230 |
-1.21 |
0.000732 |
|
Irgm |
-1.17 |
1.87 ×10-7 |
|
Ddx60 |
-1.14 |
3.27 ×10-6 |
|
Nrp1 |
-1.13 |
3.21 ×10-7 |
|
Ly6e |
-1.10 |
4.76 ×10-7 |
|
Slc6a12 |
-1.10 |
2.07 ×10-7 |
|
LOC100362391 |
-1.07 |
2.77 ×10-6 |
|
Spn |
-1.07 |
1.87 ×10-7 |
|
Slamf9 |
-1.07 |
4.73 ×10-7 |
|
Snrpd1 |
-1.04 |
0.00011 |
|
RGD1309362 |
-1.03 |
3.31 ×10-7 |
|
Epsti1 |
-1.02 |
1.20 ×10-7 |
|
Mras |
-1.02 |
8.15 ×10-8 |
|
Trat1 |
-1.02 |
3.52 ×10-6 |
|
Snrpf |
-1.01 |
3.87 ×10-5 |
|
Ngp |
-1.00 |
0.003918 |
|
Oas2 |
-1.00 |
8.13 ×10-7 |
图附图 1.
大脑中动脉栓塞模型大鼠造模后外周血差异表达基因及特征基因
A:不同造模时间点上调差异基因交集;B: 不同造模时间点下调差异基因交集;C: 不同造模时间点总差异基因交集;D: LOC689230在各时间点的表达;E:差异基因在不同造模时间点表达模式富集;F: 随发病时程持续上调或下调的基因表达趋势图
表附表 2 大脑中动脉栓塞模型大鼠造模后1 h外周血下调倍数前50位基因
Table 附表 2 List of the top 50 downregulated genes in peripheral blood at 1 hour of middle cerebral artery occlusion rats
|
下调基因 |
Log差异倍数 |
校正后的 P值 |
|
Cd79b |
-1.91 |
3.47×10 -10 |
|
Fcmr |
-1.79 |
3.88×10 -12 |
|
Ebf1 |
-1.79 |
1.59×10 -9 |
|
LOC501110 |
-1.76 |
2.55×10 -8 |
|
Fcer2 |
-1.74 |
1.06×10 -9 |
|
B3gnt5 |
-1.72 |
2.93×10 -9 |
|
Fcrla |
-1.70 |
1.48×10 -10 |
|
Hvcn1 |
-1.67 |
2.85×10 -10 |
|
Igd |
-1.64 |
1.72×10 -10 |
|
Ms4a1 |
-1.61 |
3.76×10 -9 |
|
RT1-Da |
-1.60 |
4.82×10 -10 |
|
Pde6h |
-1.53 |
1.80×10 -7 |
|
Cd79al |
-1.53 |
4.82×10 -10 |
|
Swap70 |
-1.53 |
7.49×10 -12 |
|
Cd19 |
-1.48 |
1.31×10 -10 |
|
Bank1 |
-1.48 |
3.88×10 -12 |
|
Rnase6 |
-1.47 |
8.79×10 -7 |
|
Cd180 |
-1.46 |
5.10×10 -8 |
|
Bcl11a |
-1.40 |
1.13×10 -7 |
|
Btla |
-1.40 |
3.47×10 -10 |
|
Nrp1 |
-1.40 |
1.69×10 -10 |
|
Cd209a |
-1.38 |
1.08×10 -9 |
|
Cd74 |
-1.38 |
3.76×10 -9 |
|
Myo1e |
-1.36 |
1.87×10 -11 |
|
Blnk |
-1.31 |
1.08×10 -9 |
|
Eno3 |
-1.31 |
9.02×10 -10 |
|
Kynu |
-1.27 |
1.87×10 -7 |
|
RGD1560455 |
-1.27 |
7.87×10 -7 |
|
Acsl3 |
-1.26 |
9.07×10 -8 |
|
Rnase4 |
-1.26 |
3.52×10 -7 |
|
Gngt2 |
-1.25 |
1.71×10 -9 |
|
LOC100361706 |
-1.25 |
1.21×10 -9 |
|
Qrfpr |
-1.23 |
3.56×10 -7 |
|
LOC100911716 |
-1.20 |
2.02×10 -8 |
|
Ly86 |
-1.19 |
1.37×10 -7 |
|
Samhd1 |
-1.18 |
1.52×10 -8 |
|
Zfp958 |
-1.17 |
1.29×10 -6 |
|
Trpm6 |
-1.16 |
4.68×10 -8 |
|
Mal |
-1.15 |
1.82×10 -8 |
|
Fchsd2 |
-1.13 |
1.85×10 -9 |
|
Ell3 |
-1.13 |
3.88×10 -8 |
|
Akap17b |
-1.13 |
1.59×10 -9 |
|
Crip1 |
-1.12 |
1.91×10 -6 |
|
Cd22 |
-1.12 |
2.20×10 -7 |
|
Ifi30 |
-1.09 |
3.40×10 -8 |
|
Insig1 |
-1.09 |
2.29×10 -7 |
|
Snrpd1 |
-1.09 |
0.000153 |
|
Cd200 |
-1.09 |
2.65×10 -9 |
|
Plxnb2 |
-1.07 |
2.80×10 -8 |
|
Klf4 |
-1.07 |
2.50×10 -8 |
表附表 3 大脑中动脉栓塞模型大鼠造模后2 h外周血上调倍数前50位基因
Table 附表 3 List of the top 50 upregulated genes in peripheral blood at 2 hour of middle cerebral artery occlusion rats
|
上调基因 |
Log差异倍数 |
校正后的 P值 |
|
Il1r2 |
2.96 |
1.45×10 -14 |
|
Ifitm6 |
2.86 |
1.10×10 -10 |
|
Mrgprx3 |
2.83 |
4.47×10 -14 |
|
Fpr1 |
2.81 |
1.23×10 -11 |
|
Mmp9 |
2.75 |
6.37×10 -12 |
|
Hp |
2.61 |
1.49×10 -12 |
|
Pram1 |
2.48 |
7.24×10 -14 |
|
Olfm4 |
2.48 |
7.55×10 -10 |
|
Mmp25 |
2.48 |
2.57×10 -12 |
|
Cd33 |
2.47 |
1.84×10 -12 |
|
Bmx |
2.45 |
3.97×10 -12 |
|
Cpm |
2.27 |
4.15×10 -11 |
|
Nos2 |
2.26 |
2.63×10 -12 |
|
Mir292 |
2.22 |
3.04×10 -10 |
|
Stfa3 |
2.17 |
6.88×10 -8 |
|
Dgat2 |
2.17 |
7.12×10 -13 |
|
LOC679818 |
2.17 |
8.00×10 -12 |
|
Mcemp1 |
2.16 |
7.24×10 -14 |
|
Alox5 |
2.14 |
6.00×10 -12 |
|
Oas1k |
2.14 |
1.37×10 -11 |
|
Camp |
2.13 |
9.61×10 -11 |
|
Ldhc |
2.13 |
6.86×10 -13 |
|
Rab44 |
2.12 |
3.24×10 -14 |
|
Dhrs9 |
2.11 |
2.57×10 -12 |
|
Ppp1r3b |
2.10 |
3.95×10 -13 |
|
Adgrg6 |
2.10 |
1.84×10 -12 |
|
Amica1 |
2.09 |
9.94×10 -11 |
|
Tas2r126 |
2.08 |
2.32×10 -9 |
|
Tnnc1 |
2.07 |
2.57×10 -10 |
|
Rbm47 |
2.07 |
7.65×10 -13 |
|
Ugt8 |
2.07 |
2.49×10 -11 |
|
Uaca |
2.06 |
3.38×10 -12 |
|
Hk3 |
2.06 |
4.64×10 -13 |
|
LOC690020 |
2.04 |
7.29×10 -8 |
|
Prok2 |
2.02 |
2.39×10 -11 |
|
LOC689230 |
2.01 |
2.92×10 -8 |
|
Tcn2 |
2.00 |
4.59×10 -14 |
|
Nlrp12 |
2.00 |
2.57×10 -12 |
|
LOC24906 |
2.00 |
1.84×10 -12 |
|
Fkbp5 |
1.99 |
3.95×10 -13 |
|
Slc2a3 |
1.98 |
6.82×10 -12 |
|
LOC102557597 |
1.95 |
9.34×10 -11 |
|
Alox5ap |
1.92 |
1.98×10 -13 |
|
Tarm1 |
1.92 |
2.10×10 -9 |
|
Stfa2l2 |
1.91 |
3.96×10 -11 |
|
Clec5a |
1.91 |
2.64×10 -9 |
|
Il18rap |
1.90 |
2.97×10 -11 |
|
Amdhd1 |
1.90 |
1.37×10 -11 |
|
Cd177 |
1.89 |
2.66×10 -8 |
|
Bcr |
1.88 |
1.16×10 -13 |
表附表 4 大脑中动脉栓塞模型大鼠造模后2 h外周血下调倍数前50位基因
Table 附表 4 List of the top 50 downregulated genes in peripheral blood at 2 hour of middle cerebral artery occlusion rats
|
下调基因 |
Log差异倍数 |
校正后的 P值 |
|
Ms4a1 |
-2.54 |
3.24×10 -14 |
|
Ebf1 |
-2.36 |
4.39×10 -13 |
|
Cd79b |
-2.33 |
1.39×10 -12 |
|
B3gnt5 |
-2.30 |
1.39×10 -12 |
|
Hvcn1 |
-2.24 |
4.59×10 -14 |
|
Fcer2 |
-2.22 |
3.74×10 -14 |
|
RGD1560455 |
-2.20 |
8.21×10 -11 |
|
Fcmr |
-2.18 |
3.74×10 -14 |
|
LOC501110 |
-2.16 |
2.57×10 -10 |
|
Igd |
-2.16 |
4.59×10 -14 |
|
Fcrla |
-2.11 |
4.59×10 -14 |
|
Btla |
-2.06 |
5.44×10 -13 |
|
Cd19 |
-2.01 |
5.06×10 -14 |
|
Rnase6 |
-2.00 |
8.35×10 -12 |
|
Cd79al |
-1.97 |
2.97×10 -13 |
|
Nrp1 |
-1.96 |
3.24×10 -14 |
|
Cd180 |
-1.96 |
3.80×10 -11 |
|
Bcl11a |
-1.88 |
2.39×10 -11 |
|
Bank1 |
-1.87 |
3.74×10 -14 |
|
Pde6h |
-1.86 |
2.37×10 -9 |
|
RT1-Da |
-1.82 |
2.83×10 -12 |
|
Eno3 |
-1.80 |
3.69×10 -12 |
|
Fcrl1 |
-1.76 |
2.69×10 -10 |
|
Trpm6 |
-1.73 |
1.93×10 -12 |
|
Blnk |
-1.72 |
2.96×10 -13 |
|
Kynu |
-1.67 |
6.65×10 -10 |
|
Qrfpr |
-1.67 |
1.26×10 -9 |
|
P2ry10 |
-1.65 |
2.68×10 -11 |
|
Insig1 |
-1.63 |
1.50×10 -10 |
|
LOC100361706 |
-1.62 |
1.30×10 -11 |
|
Gpr174 |
-1.58 |
2.47×10 -11 |
|
Cd209a |
-1.57 |
1.56×10 -11 |
|
Immp1l |
-1.57 |
5.86×10 -8 |
|
Cd74 |
-1.56 |
2.41×10 -11 |
|
Ell3 |
-1.54 |
1.63×10 -10 |
|
Ly86 |
-1.54 |
7.50×10 -10 |
|
Slc25a36 |
-1.52 |
3.87×10 -8 |
|
Cmah |
-1.51 |
4.39×10 -13 |
|
Gngt2 |
-1.50 |
2.48×10 -10 |
|
Lax1 |
-1.49 |
8.93×10 -11 |
|
Swap70 |
-1.49 |
1.08×10 -13 |
|
Myo1e |
-1.48 |
6.18×10 -13 |
|
Cd22 |
-1.48 |
1.99×10 -11 |
|
Rhoh |
-1.48 |
1.35×10 -11 |
|
LOC100911716 |
-1.47 |
6.71×10 -9 |
|
Slc9a7 |
-1.47 |
1.64×10 -10 |
|
Fam49a |
-1.44 |
4.35×10 -11 |
|
LOC499229 |
-1.44 |
1.60×10 -10 |
|
Mal |
-1.43 |
1.13×10 -10 |
|
Ccdc50 |
-1.42 |
4.23×10 -12 |
表附表 5 大脑中动脉栓塞模型大鼠造模后3 h外周血上调倍数前50位基因
Table 附表 5 List of the top 50 upregulated genes in peripheral blood at 3 hour of middle cerebral artery occlusion rats
|
上调基因 |
Log差异倍数 |
校正后的 P值 |
|
Hp |
3.22 |
6.28×10 -14 |
|
Plscr1 |
3.14 |
6.49×10 -13 |
|
Tgm1 |
3.05 |
2.66 ×10-12 |
|
Il1r2 |
3.03 |
1.74 ×10-15 |
|
Ifitm6 |
2.94 |
1.14 ×10-11 |
|
Cd33 |
2.81 |
1.56 ×10-13 |
|
Serpinb1a |
2.68 |
1.56 ×10-13 |
|
Nos2 |
2.66 |
4.80 ×10-15 |
|
Fpr1 |
2.57 |
2.26 ×10-11 |
|
Ugt8 |
2.56 |
2.99 ×10-11 |
|
Prok2 |
2.54 |
1.07 ×10-13 |
|
Dgat2 |
2.49 |
2.63 ×10-13 |
|
Bmx |
2.46 |
1.95 ×10-12 |
|
Mmp25 |
2.45 |
2.15 ×10-13 |
|
Dhrs9 |
2.44 |
6.86 ×10-13 |
|
Olfm4 |
2.40 |
2.06 ×10-9 |
|
Pram1 |
2.40 |
2.94 ×10-13 |
|
Cpne8 |
2.39 |
1.81 ×10-11 |
|
Dab2 |
2.33 |
1.51 ×10-10 |
|
Mmp9 |
2.33 |
1.74 ×10-11 |
|
Vcan |
2.32 |
2.27 ×10-11 |
|
Ppp1r3b |
2.32 |
3.68 ×10-13 |
|
Cpm |
2.31 |
1.14 ×10-11 |
|
Mrgprx3 |
2.22 |
3.37 ×10-14 |
|
Rab44 |
2.21 |
3.66 ×10-15 |
|
Tarm1 |
2.20 |
1.80 ×10-10 |
|
P2ry13 |
2.13 |
1.29 ×10-12 |
|
Ampd3 |
2.13 |
5.53 ×10-12 |
|
Csf2rb |
2.12 |
3.54 ×10-14 |
|
Rrm2 |
2.12 |
2.23 ×10-12 |
|
Slc2a3 |
2.12 |
8.29 ×10-12 |
|
Rbm47 |
2.11 |
1.70 ×10-10 |
|
Oas1k |
2.09 |
1.63 ×10-12 |
|
Mcemp1 |
2.07 |
5.12 ×10-13 |
|
Crispld2 |
2.07 |
2.73 ×10-13 |
|
Adgrg6 |
2.06 |
2.15 ×10-13 |
|
Msr1 |
2.05 |
2.36 ×10-11 |
|
Tfec |
2.04 |
1.82 ×10-8 |
|
Slpi |
2.03 |
5.15 ×10-13 |
|
Nlrp12 |
2.01 |
3.38 ×10-12 |
|
Slc5a3 |
2.01 |
1.51 ×10-10 |
|
Il18rap |
1.99 |
4.99 ×10-12 |
|
LOC679818 |
1.99 |
6.06 ×10-11 |
|
Il1rap |
1.98 |
1.87 ×10-11 |
|
Hk2 |
1.98 |
3.71 ×10-12 |
|
Tpd52 |
1.95 |
1.56 ×10-13 |
|
Gk |
1.94 |
1.04 ×10-9 |
|
Upp1 |
1.93 |
1.95 ×10-12 |
|
Mocos |
1.92 |
1.74 ×10-11 |
|
Hk3 |
1.92 |
1.51 ×10-13 |
表附表 6 大脑中动脉栓塞模型大鼠造模后3 h外周血下调倍数前50位基因
Table 附表 6 List of the top 50 downregulated genes in peripheral blood at 3 hour of middle cerebral artery occlusion rats
|
下调基因 |
Log差异倍数 |
校正后的 P值 |
|
Rnase6 |
-2.56 |
3.38 ×10-12 |
|
Ms4a1 |
-2.48 |
7.64 ×10-14 |
|
Ebf1 |
-2.42 |
5.15 ×10-13 |
|
Cd79b |
-2.35 |
1.04 ×10-12 |
|
Fcer2 |
-2.24 |
3.60 ×10-13 |
|
Fcrla |
-2.21 |
7.54 ×10-14 |
|
LOC501110 |
-2.21 |
2.40 ×10-8 |
|
Fcmr |
-2.18 |
1.74 ×10-15 |
|
Nrp1 |
-2.18 |
1.42 ×10-14 |
|
Cd79al |
-2.15 |
1.67 ×10-13 |
|
Cd180 |
-2.14 |
8.93 ×10-11 |
|
Igd |
-2.14 |
1.67 ×10-13 |
|
Btla |
-2.13 |
9.53 ×10-13 |
|
Hvcn1 |
-2.10 |
2.71 ×10-12 |
|
Eno3 |
-2.06 |
1.66 ×10-14 |
|
B3gnt5 |
-2.03 |
6.49 ×10-13 |
|
Cd19 |
-2.00 |
1.64 ×10-12 |
|
RGD1560455 |
-1.98 |
4.79 ×10-8 |
|
Bank1 |
-1.91 |
3.40 ×10-15 |
|
Gngt2 |
-1.87 |
1.64 ×10-12 |
|
Pde6h |
-1.87 |
2.84 ×10-10 |
|
Ly86 |
-1.83 |
8.27 ×10-10 |
|
P2ry10 |
-1.80 |
3.61 ×10-10 |
|
LOC100361706 |
-1.79 |
3.32 ×10-13 |
|
Dnase2b |
-1.76 |
6.94 ×10-10 |
|
Trpm6 |
-1.73 |
4.59 ×10-11 |
|
Heg1 |
-1.72 |
1.67 ×10-13 |
|
Gpr174 |
-1.71 |
4.81 ×10-10 |
|
Blnk |
-1.65 |
6.49 ×10-13 |
|
Cd209a |
-1.65 |
1.30 ×10-11 |
|
Il5ra |
-1.64 |
8.27 ×10-12 |
|
Hmgn3 |
-1.64 |
1.43 ×10-10 |
|
Cd22 |
-1.63 |
1.22 ×10-11 |
|
Xkrx |
-1.62 |
5.78 ×10-9 |
|
Cmah |
-1.60 |
3.38 ×10-12 |
|
Nr4a1 |
-1.60 |
1.91 ×10-10 |
|
Qrfpr |
-1.59 |
3.55 ×10-9 |
|
Slc6a12 |
-1.58 |
6.40 ×10-12 |
|
Mal |
-1.58 |
7.79 ×10-11 |
|
Ccdc50 |
-1.55 |
2.15 ×10-12 |
|
Fam49a |
-1.55 |
8.21 ×10-10 |
|
Spn |
-1.54 |
1.56 ×10-12 |
|
Lax1 |
-1.52 |
6.04 ×10-11 |
|
Klf12 |
-1.52 |
5.20 ×10-12 |
|
Adgre1 |
-1.52 |
2.08 ×10-9 |
|
Fcrl1 |
-1.51 |
5.40 ×10-11 |
|
Cd24 |
-1.50 |
1.90 ×10-10 |
|
Bcl11a |
-1.50 |
2.28 ×10-10 |
|
Trat1 |
-1.49 |
1.23 ×10-8 |
|
Ell3 |
-1.48 |
8.40 ×10-12 |
表附表 7 大脑中动脉栓塞模型大鼠造模后6 h外周血上调倍数前50位基因
Table 附表 7 List of the top 50 upregulated genes in peripheral blood at 6 hour of middle cerebral artery occlusion rats
|
上调基因 |
Log差异倍数 |
校正后的 P值 |
|
Tgm1 |
4.06 |
1.99 ×10-12 |
|
Cpne8 |
3.76 |
6.80 ×10-15 |
|
Ifitm6 |
3.61 |
7.01 ×10-12 |
|
Hp |
3.57 |
1.72 ×10-13 |
|
Serpinb1a |
3.50 |
6.80 ×10-15 |
|
Olfm4 |
3.31 |
4.23 ×10-10 |
|
Il1r2 |
3.31 |
8.33 ×10-15 |
|
Prok2 |
3.17 |
1.01 ×10-13 |
|
Clec4b2 |
3.16 |
5.43 ×10-13 |
|
Adgb |
3.10 |
1.31 ×10-11 |
|
Cd33 |
3.07 |
4.97 ×10-13 |
|
Plscr1 |
2.97 |
3.36 ×10-13 |
|
Wfdc15b |
2.84 |
6.80 ×10-15 |
|
Mmp25 |
2.83 |
1.31 ×10-11 |
|
Ugt8 |
2.83 |
4.91 ×10-12 |
|
Lcn2 |
2.79 |
7.32 ×10-8 |
|
Il1rap |
2.77 |
7.32 ×10-13 |
|
Slpi |
2.75 |
1.01 ×10-13 |
|
Rrm2 |
2.72 |
3.11 ×10-13 |
|
Bmx |
2.70 |
2.49 ×10-12 |
|
Dgat2 |
2.67 |
3.41 ×10-12 |
|
Car4 |
2.67 |
2.43 ×10-10 |
|
Sycp2 |
2.63 |
4.57 ×10-12 |
|
Gk |
2.62 |
7.97 ×10-12 |
|
Tas2r126 |
2.62 |
1.79 ×10-10 |
|
Msr1 |
2.61 |
1.12 ×10-11 |
|
Tarm1 |
2.59 |
1.06 ×10-10 |
|
Nos2 |
2.58 |
3.41 ×10-12 |
|
Fpr1 |
2.57 |
1.97 ×10-10 |
|
Oscar |
2.55 |
6.72 ×10-12 |
|
Slc2a3 |
2.55 |
3.23 ×10-12 |
|
Il22ra2 |
2.54 |
1.43 ×10-12 |
|
Slc5a3 |
2.49 |
4.01 ×10-13 |
|
Ampd3 |
2.48 |
1.55 ×10-11 |
|
Mmp9 |
2.43 |
2.43 ×10-10 |
|
LOC102557597 |
2.42 |
2.62 ×10-10 |
|
Vcan |
2.42 |
1.92 ×10-11 |
|
Pram1 |
2.40 |
2.43 ×10-12 |
|
Ccr1 |
2.40 |
8.43 ×10-12 |
|
Tas2r143 |
2.36 |
5.07 ×10-12 |
|
Nlrp12 |
2.35 |
5.07 ×10-12 |
|
Crispld2 |
2.35 |
4.16 ×10-12 |
|
Tas2r135 |
2.34 |
2.51 ×10-9 |
|
Gca |
2.31 |
7.33 ×10-10 |
|
Cpm |
2.31 |
8.47 ×10-11 |
|
Alox5ap |
2.31 |
2.73 ×10-13 |
|
P2ry13 |
2.29 |
8.30 ×10-13 |
|
Csf2rb |
2.26 |
3.69 ×10-12 |
|
RGD1307182 |
2.26 |
1.72 ×10-11 |
|
Alpk1 |
2.25 |
5.49 ×10-11 |
表附表 8 大脑中动脉栓塞模型大鼠造模后6 h外周血下调倍数前50位基因
Table 附表 8 List of the top 50 downregulated genes in peripheral blood at 6 hour of middle cerebral artery occlusion rats
|
下调基因 |
Log差异倍数 |
校正后的 P值 |
|
Rnase6 |
-2.58 |
7.17 ×10-11 |
|
Eno3 |
-2.23 |
1.38 ×10-13 |
|
Mx2 |
-2.16 |
6.58 ×10-14 |
|
Fcer2 |
-2.07 |
6.49 ×10-12 |
|
Ebf1 |
-1.95 |
1.77 ×10-11 |
|
Ms4a1 |
-1.94 |
4.04 ×10-11 |
|
Cd79al |
-1.93 |
7.32 ×10-13 |
|
Adgre1 |
-1.93 |
3.78 ×10-10 |
|
Dnase2b |
-1.92 |
6.01 ×10-10 |
|
Fcrla |
-1.91 |
2.30 ×10-12 |
|
Cd79b |
-1.89 |
1.16 ×10-10 |
|
Fcmr |
-1.88 |
5.16 ×10-13 |
|
Slc6a12 |
-1.82 |
1.10 ×10-12 |
|
Hvcn1 |
-1.78 |
8.00 ×10-12 |
|
Il5ra |
-1.72 |
6.34 ×10-10 |
|
Nrp1 |
-1.72 |
3.56 ×10-11 |
|
Mx1 |
-1.71 |
1.10 ×10-12 |
|
P2ry10 |
-1.69 |
9.78 ×10-9 |
|
LOC100361706 |
-1.66 |
1.31 ×10-11 |
|
Cd19 |
-1.66 |
1.05 ×10-11 |
|
Btla |
-1.62 |
1.49 ×10-9 |
|
Emr4 |
-1.59 |
2.32 ×10-8 |
|
LOC103692555 |
-1.59 |
9.79 ×10-5 |
|
Ly86 |
-1.58 |
1.77 ×10-9 |
|
Cd180 |
-1.58 |
8.22 ×10-10 |
|
Cd4 |
-1.58 |
3.41 ×10-12 |
|
Igd |
-1.57 |
3.58 ×10-10 |
|
MGC108823 |
-1.55 |
9.90 ×10-8 |
|
Gpr174 |
-1.54 |
2.76 ×10-8 |
|
Cd24 |
-1.53 |
6.19 ×10-10 |
|
LOC501110 |
-1.53 |
1.09 ×10-7 |
|
Bank1 |
-1.52 |
1.58 ×10-11 |
|
Bcl2 |
-1.52 |
9.74 ×10-12 |
|
Camk2d |
-1.50 |
1.00 ×10-10 |
|
Ddx60 |
-1.49 |
1.18 ×10-8 |
|
Blnk |
-1.48 |
6.79 ×10-11 |
|
B3gnt5 |
-1.48 |
5.04 ×10-9 |
|
Myo1e |
-1.47 |
2.36 ×10-11 |
|
Cxcl16 |
-1.47 |
1.73 ×10-9 |
|
Heg1 |
-1.47 |
9.74 ×10-12 |
|
Trpm6 |
-1.47 |
1.32 ×10-9 |
|
Nr4a1 |
-1.46 |
4.71 ×10-10 |
|
Lbh |
-1.45 |
8.22 ×10-10 |
|
Gngt2 |
-1.45 |
4.08 ×10-11 |
|
Gapt |
-1.45 |
1.39 ×10-8 |
|
RGD1309362 |
-1.44 |
7.49 ×10-9 |
|
Cd28 |
-1.44 |
1.26 ×10-8 |
|
Mras |
-1.44 |
1.92 ×10-11 |
|
Ms4a6c |
-1.43 |
5.39 ×10-8 |
|
Atp2b4 |
-1.43 |
4.11 ×10-11 |
表附表 9 大脑中动脉栓塞模型大鼠造模后24 h外周血上调基因
Table 附表 9 List of upregulated genes in peripheral blood at 24 hour of middle cerebral artery occlusion rats
|
上调基因 |
Log差异倍数 |
校正后的 P值 |
|
Fetub |
1.96 |
3.27 ×10-6 |
|
Cd33 |
1.87 |
3.31 ×10-7 |
|
Cpne8 |
1.80 |
1.06 ×10-5 |
|
Clec4b2 |
1.64 |
1.81 ×10-5 |
|
Tas2r126 |
1.55 |
6.15 ×10-6 |
|
Prok2 |
1.52 |
6.54 ×10-6 |
|
LOC679818 |
1.49 |
4.84 ×10-6 |
|
Serpinb1a |
1.47 |
0.000168 |
|
Tarm1 |
1.47 |
6.78 ×10-6 |
|
Oscar |
1.42 |
9.67 ×10-6 |
|
Prg4 |
1.40 |
0.000124 |
|
Adgb |
1.37 |
0.000141 |
|
Bmx |
1.35 |
1.14 ×10-5 |
|
Nlrp12 |
1.32 |
4.02 ×10-6 |
|
Serpinb2 |
1.31 |
1.08 ×10-5 |
|
Wfdc15b |
1.30 |
1.20 ×10-7 |
|
Nek10 |
1.30 |
0.000397 |
|
Car4 |
1.26 |
8.91 ×10-5 |
|
Il22ra2 |
1.26 |
1.29 ×10-5 |
|
Ugt8 |
1.24 |
3.36 ×10-5 |
|
Slc12a2 |
1.23 |
4.73 ×10-7 |
|
Slpi |
1.23 |
2.27 ×10-5 |
|
Il1r2 |
1.23 |
0.00013 |
|
Tfrc |
1.22 |
8.13 ×10-7 |
|
Mir223 |
1.21 |
6.63 ×10-5 |
|
Ampd3 |
1.18 |
6.42 ×10-5 |
|
Slc7a11 |
1.17 |
5.61 ×10-6 |
|
Slc2a3 |
1.17 |
8.30 ×10-5 |
|
Mir292 |
1.16 |
2.30 ×10-5 |
|
Tspan8 |
1.16 |
1.99 ×10-5 |
|
Dgat2 |
1.14 |
0.000201 |
|
Mmp25 |
1.14 |
7.08 ×10-5 |
|
Mcemp1 |
1.14 |
0.000123 |
|
Tas2r143 |
1.13 |
1.59 ×10-5 |
|
Fpr1 |
1.13 |
0.000638 |
|
Il1rap |
1.12 |
6.03 ×10-5 |
|
Bco1 |
1.11 |
2.07 ×10-7 |
|
Siglec8 |
1.10 |
2.60 ×10-5 |
|
Vcan |
1.07 |
5.38 ×10-5 |
|
Gk |
1.06 |
0.000146 |
|
Ccr1 |
1.05 |
0.000726 |
|
Ifitm6 |
1.05 |
0.014119 |
|
Fbxl5 |
1.03 |
1.32 ×10-5 |
|
Sort1 |
1.02 |
6.98 ×10-7 |
|
Fam92a1 |
1.01 |
2.22 ×10-7 |
|
Tns1 |
1.01 |
2.10 ×10-5 |
|
Bst1 |
1.01 |
2.93 ×10-5 |
|
Mir290 |
1.01 |
2.27 ×10-5 |
为进一步分析不同时间点的特征表达基因,研究通过富集分析发现差异基因共存在38种表达模式,其中14种模式为显著富集( 附图 1E)。模式95(17个基因)、58(1个基因)和模式4(19个基因)为造模2 h的特征基因,在2 h上调或下调;模式97和模式83在2~3 h分别特异上调或下调;模式93(76个基因)和模式6(108个基因)在3~6 h特异变化;模式65(81个基因)和模式34(11个基因)分别在2~6 h上调或下调;模式91(34个基因)、57(18个基因)和模式8(5个基因)、42(5个基因)在6 h特异上调及下调;模式50和90为24 h的特异表达模式。值得注意的是,模式28(3个基因)在1 h特异下调而在6 h特异上调;模式46在3 h特异下调,而在24 h特异上调。此外,研究亦发现7个基因随发病时程持续上调或下调,包括 Fetub、 Mx1、 Mx2、 Rtp4、 LOC102556096、 Igj及 LOC100362391 ( 附图 1F、 G)。提示这些基因与MCAO大鼠病程进展密切相关。
不同时间点外周血差异表达基因基因本体的生物进程注释结果显示,MCAO造模后1 h差异基因主要参与细胞对脂多糖的应答(上调)、炎症反应(上调)、免疫应答(下调)以及抗原加工和主要组织相容性复合体Ⅱ介导的外源肽抗原呈递(下调)等生物进程( 附图 2A)。造模后2 h上调排名前二的生物进程为炎症反应及细胞对脂多糖的应答,下调排名前二的为B细胞增殖正调控及对γ干扰素的反应( 附图 2B)。造模3 h上调的生物进程排名前二为细胞对脂多糖的应答及固有免疫应答,下调排名前二的为细胞表面受体信号通路及获得性免疫应答( 附图 2C)。造模后6 h上调排名前二的为细胞对脂多糖的应答及固有免疫应答,下调排名前二的为细胞对细胞因子刺激的反应及获得性免疫应答( 附图 2D)。造模后24 h上调排名前二的为负调控内肽酶活性及负调控炎症反应,下调排名前二的为固有免疫应答及防御应答( 附图 2E)。以上数据提示,炎症反应、固有免疫应答及细胞对脂多糖的应答等生物进程在MCAO早期被激活,而在24 h呈现负调控或未明显激活状态。
图附图 2.
MCAO造模后不同时间点外周血差异基因的基因本体(生物进程)注释
A:造模后1 h;B:造模后2 h;C:造模后3 h;D:造模后6 h;E:造模后24 h. MCAO:大脑中动脉栓塞.

进一步对MCAO造模后不同时间点外周血差异基因进行KEGG通路富集分析。造模后1 h差异基因参与排名前十的通路均为下调趋势,包括结核病、造血细胞系等通路( 附图 3A)。造模后2 h上调排名前二的通路包括利什曼病及金黄色葡萄球菌感染,下调排名前二的通路包括造血细胞系及原发性免疫缺陷( 附图 3B)。造模后3 h上调排名前二的通路包括结核病及利什曼病,下调排名前二的通路为造血细胞系及T细胞受体信号通路( 附图 3C)。造模后6 h上调排名前二的通路包括细胞因子-细胞因子受体相互作用及利什曼病,下调排名前二的通路为造血细胞系及原发性免疫缺陷( 附图 3D)。造模后24 h无显著富集通路。
图附图 3.
MCAO造模后不同时间点外周血差异基因的KEGG通路富集
A:造模后1 h;B:造模后2 h;C:造模后3 h;D:造模后6 h. MCAO:大脑中动脉栓塞.

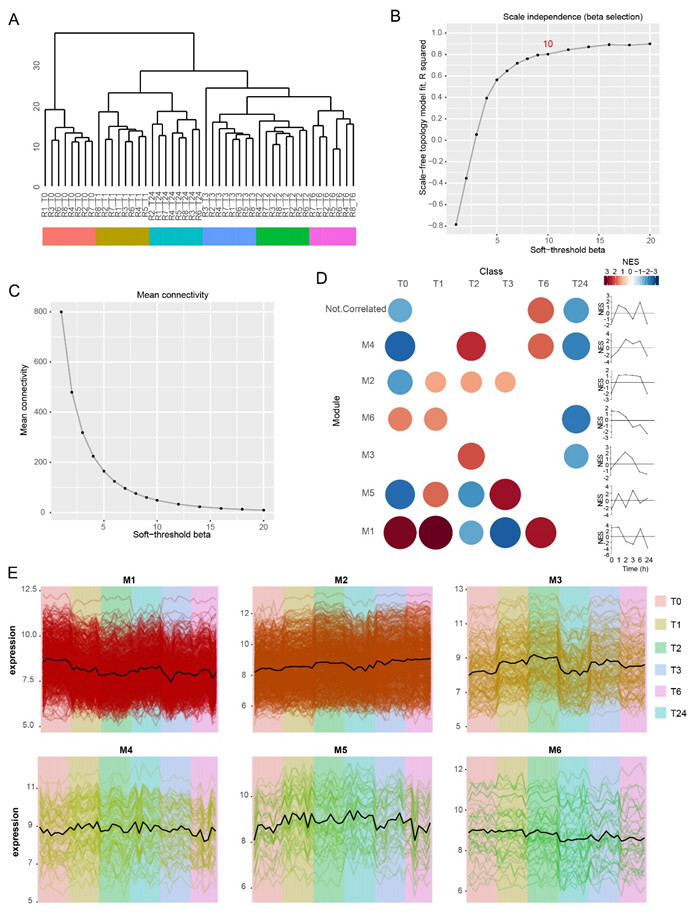
为进一步考察与缺血性脑损伤发病时程密切相关的特征基因,研究通过构建共表达网络分析每个时间点的相关模块。首先对样本进行聚类,发现每个时间点的样本差异较小( 附图 4A)。共表达网络构建的软阈值筛选显示 β值为10( 附图 4B、 C)。共表达网络分析显示,共有六个特征模块被富集,模块与各时间点的相关性以标准化富集分数表示,所展示模块校正后的 P值均小于0.05( 附图 4D)。基因模块富集分析表明,造模急性期早期(1~6 h)基因状态与模块2显著正相关,造模1~3 h基因变化与模块3正相关,造模2~6 h基因状态与模块4密切正相关,提示这些模块可能与缺血性脑损伤的早期发病时程密切相关( 附图 4D)。值得注意的是,随着造模时间的迁移,模块6与各时间点从正相关(0~2 h)逐渐转为负相关(3~24 h),提示该模块可能与疾病进程相关( 附图 4D)。最后,研究进一步呈现了6个模块不同时间点的基因表达模式,结果提示基因的表达高低与模块的富集密切相关( 附图 4E)。
图附图 4.
MCAO造模不同时间点外周血特征共表达模块分析
A:各造模时间点样本聚类,不同颜色的方块代表不同时间点的样本;B、C:软阈值筛选;D:共表达模块在各造模时间点的活跃状态;E:各共表达模块基因在不同造模时间点的表达模式. MCAO:大脑中动脉栓塞.
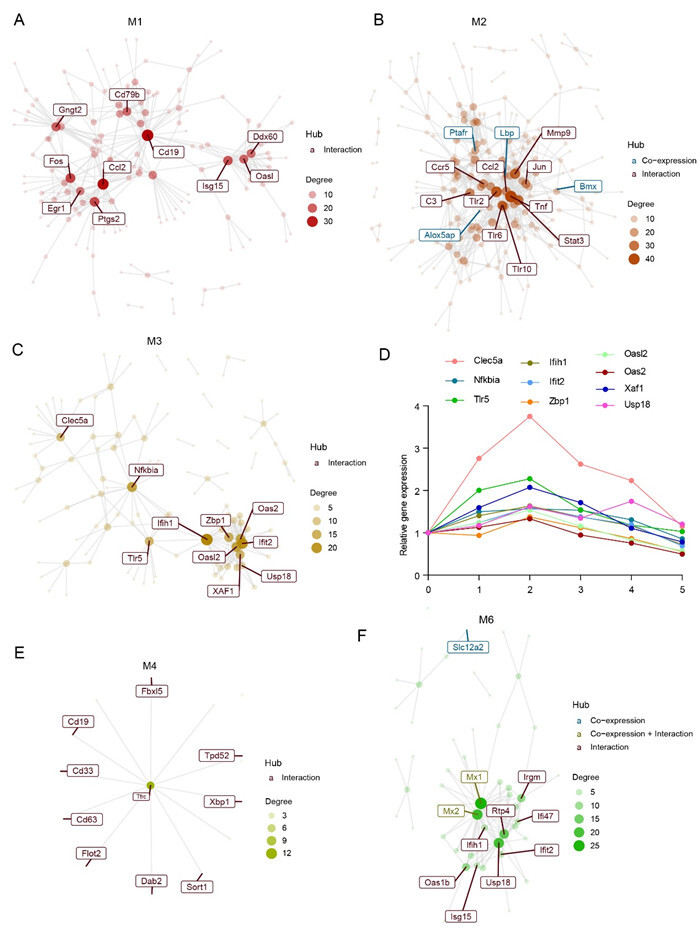
共表达网络构建结果显示:模块1共含696个基因,构建网络后其核心节点为 Gngt2、 Fos、 Egr1、 Ptgs2、 Ccl2、 Cd79b、 Cd19、 Isg15、 Oasl及 Ddx60 ( 附图 5A);模块2共含694个基因,构建网络后其核心节点为 Ptafr、 Alox5ap、 Lbp、 Bmx、 Ccr5、 C3、 Ccl2、 Tlr2、 Tlr6、 Tlr10、 Jun、 Stat3、 Mmp9及 Tnf ( 附图 5B);模块3共含142个基因,构建网络后其核心基因为 Clec5a、 Nfkbia、 Tlr5、 Ifih1、 Ifit2、 Zbp1、 Oasl2、 Oas2、 Xaf1及 Usp18 ( 附图 5C),模块3核心基因在造模后不同时间点的表达趋势如 附图 5D所示;模块4共含76个基因,其网络核心节点为 Cd19、 Cd33、 Cd63、 Flot2、 Fbxl5、 Tfrc、 Dab2、 Tpd52、 Xbp1及 Sort1 ( 附图 5E);模块5共含45个基因,但蛋白-蛋白互作用(PPI)较少,无法呈现网络;模块6共含38个基因,其网络核心节点为 Slc12a2、 Oas1b、 Mx1、 Mx2、 Ifih1、 Isg15、 Rtp4、 Usp18、 Irgm、 Ifi47及 Ifit2 ( 附图 5F)。
图附图 5.
共表达模块蛋白互作用网络构建及核心基因分析
A:模块M1共表达网络;B:模块M2共表达网络;C:模块M3共表达网络;D:模块M3核心基因在造模后不同时间点的表达趋势;E:模块M4共表达网络;F:模块M6共表达网络.
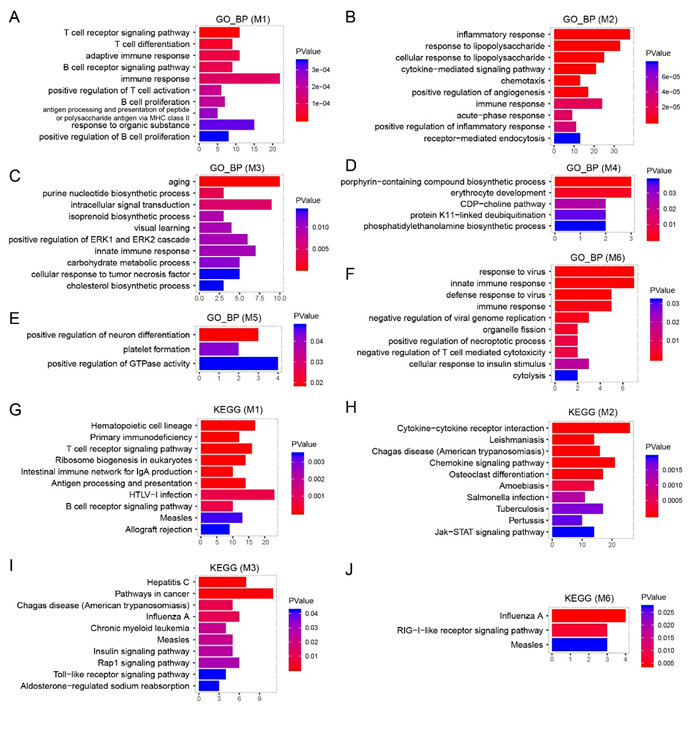
为明确每个特征模块的生物学意义,研究利用基因本体及KEGG对其进行注释。模块1主要参与的生物进程包括T细胞受体信号通路、T细胞分化等( 附图 6A);模块2主要与炎症反应、细胞对脂多糖的应答等相关( 附图 6B);模块3主要与衰老及嘌呤核苷酸生物合成过程等相关( 附图 6C);模块4主要参与含卟啉的化合物合成过程及红细胞发育等生物进程( 附图 6D);模块5主要参与正向调控神经元分化及血小板形成等过程( 附图 6E);模块6主要与病毒应答及固有免疫应答相关( 附图 6F)。KEGG通路富集结果显示,模块1主要与造血细胞系及原发性免疫缺陷相关( 附图 6G);模块2主要参与细胞因子-细胞因子受体互作及利什曼病等通路( 附图 6H);模块3主要与丙型肝炎及肿瘤通路相关( 附图 6I);模块6主要参与甲型流感及维甲酸诱导基因蛋白1样受体信号通路等( 附图 6J)。模块4、5无显著富集通路。
图附图 6.
共表达模块基因本体注释及KEGG通路富集
A:模块M1基因本体(生物进程)富集;B:模块M2基因本体(生物进程)富集;C:模块M3基因本体(生物进程)富集;D:模块M4基因本体(生物进程)富集;E:模块M5基因本体(生物进程)富集;F:模块M6基因本体(生物进程)富集;G:模块M1的KEGG富集;H:模块M2的KEGG富集;I:模块M3的KEGG富集;J:模块M6的KEGG富集.
缺血性脑卒中由于脑内动脉栓塞造成局部供血障碍,导致患者脑组织缺血坏死以及神经功能缺失。研究表明,急性缺血性脑卒中患者在未接受治疗时,大脑每分钟约190万个神经元、140亿个突触及累计长达12 km的神经纤维被破坏 [ 13] 。因此,脑卒中患者的早期治疗尤为关键。目前,采用rt-PA超早期溶栓是唯一被证明治疗缺血性卒中有效的方法。然而,研究显示多数患者对rt-PA的使用并未在最佳时间窗内 [ 4, 14] 。因此,筛选缺血性脑卒中不同发病时期的基因表达特征有助于诠释疾病的病理生理变化规律,为临床判断准确的发病时间提供线索。缺血性脑损伤常伴随全身组织的状态改变,外周血由于取样及检测便捷的优势,常作为疾病的标志物。本研究通过分析缺血性脑损伤急性发病期不同时间点外周血特征基因及共表达网络变化,发现了一些时间特异性表达的基因模块,为脑卒中外周血的病理机制研究提供线索。
缺血性脑卒中的进展动态而多变,反映在外周血的基因变化亦是如此。不同造模时间点基因表达模式富集结果显示,从造模前到急性发病期,外周血基因共产生了38种表达模式。其中,在急性发病期早期(2~6 h),共有92个基因发生特异变化, Hp(编码触珠蛋白)、 Nos2、 P2ry10及 Klf12为变化最为明显的四个基因。触珠蛋白是一种急性期反应血浆蛋白,可在应激反应时保护机体免于氧化损伤。在MCAO造模2~6 h, Hp的表达显著上调,并于6 h达到高峰。研究表明,外周血中触珠蛋白表达上调是急性缺血性脑卒中患者的预后不良因素 [ 15] 。Brea等 [ 16] 通过检测首次发生缺血性脑卒中且发病12 h以内的患者血清触珠蛋白及淀粉样蛋白A,发现两者的表达水平均可较好地用于诊断确认动脉粥样硬化导致的缺血性卒中。然而,将触珠蛋白用于缺血性脑卒中急性发病期早期的诊断尚未见报道。 Nos2可参与脑卒中引起的神经炎症,上调血液中的miR-122可直接抑制 Nos2表达,促进MCAO大鼠的神经功能恢复 [ 17] 。本文资料显示, Nos2在造模后2~6 h存在一过性高表达,提示 Nos2可能参与缺血性脑损伤的进展,但目前将 Nos2表达变化用于判断脑缺血进展时程的研究尚未见报道。 P2ry10及 Klf12在造模后2~6 h一过性下调,24 h又恢复正常水平。 P2ry10是一类G蛋白偶联受体,可被鞘氨醇-1-磷酸及溶血磷脂酸结合激活,目前在脑缺血中的作用未明。 Klf12是抗炎的调控因子,与脑卒中相关研究亦有限。这些急性发病期特异变化的基因可能作为机体异常的感受器,敏感地反映缺血性脑损伤早期的分子生物学状态。
病理状态下基因并非单独失调,而是伴随着整个分子网络的改变。共表达网络分析显示,缺血性脑损伤进展过程基因表达模块是动态且特异的,如造模急性期早期(1~6 h)基因状态与模块2正相关,造模1~3 h基因变化与模块3正相关,造模2~6 h基因状态与模块4正相关。模块2共包含694个基因,主要参与炎症反应、细胞对脂多糖的应答等生物进程。核心基因如 Ptafr、 Alox5ap、 Tlr2、 Tlr6、 Tlr10、 Jun、 Stat3及 Tnf等与上述进程密切相关。 Alox5ap参与白三烯的合成,研究表明 Alox5ap是脑卒中的易感基因,其单核苷酸多态性位点如 SG13S114突变与欧洲人群的中风风险相关 [ 18] ,初步临床试验显示针对Alox5ap的抑制剂可改善哮喘症状 [ 19] ,但是否对脑卒中患者有效尚待研究。此外,研究亦发现基因模块6随着造模时间的迁移,与各时间点从正相关(0~2 h)逐渐转为负相关(3~24 h),模块6主要与病毒应答及固有免疫应答相关,其网络核心节点包括 Mx1、 Mx2、 Rtp4等基因。基因表达模式显示 Mx1、 Mx2、 Rtp4等随着造模时程逐渐下调。 Mx1和 Mx2可被干扰素诱导表达,参与抗病毒免疫应答 [ 20- 21] ,但在缺血性脑损伤中的作用未知。 Rtp4是一类受体转运体蛋白,可参与调控鸦片受体的细胞膜定位,Décaillot等 [ 22] 发现吗啡可促进下丘脑Rtp4的表达。此外,Schoggins等 [ 23] 研究指出Rtp4可被Ⅰ型干扰素上调进而参与抗病毒免疫应答。研究表明,在脑卒中时Ⅰ型干扰素分泌增加,导致小胶质细胞过度活化,并影响脑内神经免疫环境以及血脑屏障完整性,引起白质的小胶质细胞过度增生以及促炎因子释放 [ 24] 。虽然 Mx1、 Mx2、 Rtp4在脑卒中的作用尚未明确,但Ⅰ型干扰素分泌失调提示了这些下游分子参与脑卒中进展的可能性。
综上,本研究分析了缺血性脑卒中大鼠急性发病期的外周血特征基因及共表达网络模块,补充了脑部病灶以外的机体状态变化规律,为后续rt-PA的合理使用提供了潜在标志物。后续的研究仍需要大样本验证其诊断效能,包括扩大大鼠的样本量确定时间特异性表达基因以及收集临床患者血样进行标志物验证。此外,鉴于脑卒中的有效溶栓时间窗极短,目前基因检测技术尚无法实时呈现检测结果,如何提升标志物检测效率将是治疗脑卒中的重要研究方向。根据脑卒中的发病时间进行标志物细分,描绘动态的基因群变化,将为缺血性脑卒中提供一个精准的发病“时钟”,为后续临床决策提供参考依据。
Funding Statement
国家自然科学基金(81802673);浙江省公益性技术研究计划(LQ18H160017);浙江省医药卫生科技计划(2017KY023);杭州市卫生科技计划(2017B56)
References
- 1.MURRAY C J, LOPEZ A D. Measuring the global burden of disease. N Engl J Med. 2013;369(5):448–457. doi: 10.1056/NEJMra1201534. [MURRAY C J, LOPEZ A D. Measuring the global burden of disease[J]. N Engl J Med, 2013, 369(5):448-457.] [DOI] [PubMed] [Google Scholar]
- 2.WANG J, AN Z, LI B, et al. Increasing stroke incidence and prevalence of risk factors in a low-income Chinese population. Neurology. 2015;84(4):374–381. doi: 10.1212/WNL.0000000000001175. [WANG J, AN Z, LI B, et al. Increasing stroke incidence and prevalence of risk factors in a low-income Chinese population[J]. Neurology, 2015, 84(4):374-381.] [DOI] [PubMed] [Google Scholar]
- 3.HACKE W, KASTE M, BLUHMKI E, et al. Thrombolysis with alteplase 3 to 4.5 hours after acute ischemic stroke. N Engl J Med. 2008;359(13):1317–1329. doi: 10.1056/NEJMoa0804656. [HACKE W, KASTE M, BLUHMKI E, et al. Thrombolysis with alteplase 3 to 4.5 hours after acute ischemic stroke[J]. N Engl J Med, 2008, 359(13):1317-1329.] [DOI] [PubMed] [Google Scholar]
- 4.FAIZ K W, SUNDSETH A, THOMMESSEN B, et al. Reasons for low thrombolysis rate in a Norwegian ischemic stroke population. Neurol Sci. 2014;35(12):1977–1982. doi: 10.1007/s10072-014-1876-4. [FAIZ K W, SUNDSETH A, THOMMESSEN B, et al. Reasons for low thrombolysis rate in a Norwegian ischemic stroke population[J]. Neurol Sci, 2014, 35(12):1977-1982.] [DOI] [PubMed] [Google Scholar]
- 5.FINK J N, SELIM M H, KUMAR S, et al. Why are stroke patients excluded from tPA therapy? An analysis of patient eligibility. Neurology. 2001;57(9):1739–1740. doi: 10.1212/WNL.57.9.1739. [FINK J N, SELIM M H, KUMAR S, et al. Why are stroke patients excluded from tPA therapy? An analysis of patient eligibility[J]. Neurology, 2001, 57(9):1739-1740.] [DOI] [PubMed] [Google Scholar]
- 6.MOORE D F, LI H, JEFFRIES N, et al. Using peripheral blood mononuclear cells to determine a gene expression profile of acute ischemic stroke:a pilot investigation. Circulation. 2005;111(2):212–221. doi: 10.1161/01.CIR.0000152105.79665.C6. [MOORE D F, LI H, JEFFRIES N, et al. Using peripheral blood mononuclear cells to determine a gene expression profile of acute ischemic stroke:a pilot investigation[J]. Circulation, 2005, 111(2):212-221.] [DOI] [PubMed] [Google Scholar]
- 7.STAMOVA B, XU H, JICKLING G, et al. Gene expression profiling of blood for the prediction of ischemic stroke. Stroke. 2010;41(10):2171–2177. doi: 10.1161/STROKEAHA.110.588335. [STAMOVA B, XU H, JICKLING G, et al. Gene expression profiling of blood for the prediction of ischemic stroke[J]. Stroke, 2010, 41(10):2171-2177.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.WANG Q, TANG X N, YENARI M A. The inflammatory response in stroke. J Neuroimmunol. 2007;184(1-2):53–68. doi: 10.1016/j.jneuroim.2006.11.014. [WANG Q, TANG X N, YENARI M A. The inflammatory response in stroke[J]. J Neuroimmunol, 2007, 184(1-2):53-68.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.DAGONNIER M, WILSON W J, FAVALORO J M, et al. Hyperacute changes in blood mRNA expression profiles of rats after middle cerebral artery occlusion: Towards a stroke time signature[J/OL]. PLoS One, 2018, 13(11): e0206321. [DOI] [PMC free article] [PubMed]
- 10.SMYTH G K. limma: linear models for microarray data[M]//GENTLEMAN R, VINCENT J, CAREY V J, et al. Bioinformatics and computational biology solutions using R and bioconductor. Springer, 2011: 397-420.
- 11.ERNST J, BAR-JOSEPH Z. STEM:a tool for the analysis of short time series gene expression data. BMC Bioinformatics. 2006;7:191. doi: 10.1186/1471-2105-7-191. [ERNST J, BAR-JOSEPH Z. STEM:a tool for the analysis of short time series gene expression data[J]. BMC Bioinformatics, 2006, 7:191.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.RUSSO P S T, FERREIRA G R, CARDOZO L E, et al. CEMiTool:a Bioconductor package for performing comprehensive modular co-expression analyses. BMC Bioinformatics. 2018;19(1):56. doi: 10.1186/s12859-018-2053-1. [RUSSO P S T, FERREIRA G R, CARDOZO L E, et al. CEMiTool:a Bioconductor package for performing comprehensive modular co-expression analyses[J]. BMC Bioinformatics, 2018, 19(1):56.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.SAVER J L. Time is brain-quantified. Stroke. 2006;37(1):263–266. doi: 10.1161/01.STR.0000196957.55928.ab. [SAVER J L. Time is brain-quantified[J]. Stroke, 2006, 37(1):263-266.] [DOI] [PubMed] [Google Scholar]
- 14.MINNERUP J, WERSCHING H, RINGELSTEIN E B, et al. Impact of the extended thrombolysis time window on the proportion of recombinant tissue-type plasminogen activator-treated stroke patients and on door-to-needle time. Stroke. 2011;42(10):2838–2843. doi: 10.1161/STROKEAHA.111.616565. [MINNERUP J, WERSCHING H, RINGELSTEIN E B, et al. Impact of the extended thrombolysis time window on the proportion of recombinant tissue-type plasminogen activator-treated stroke patients and on door-to-needle time[J]. Stroke, 2011, 42(10):2838-2843.] [DOI] [PubMed] [Google Scholar]
- 15.ZHANG T, XIANG L. Elevated plasma haptoglobin level as a potential marker for poor prognosis in acute cerebral infarction. Eur Neurol. 2018;79(3-4):154–160. doi: 10.1159/000487648. [ZHANG T, XIANG L. Elevated plasma haptoglobin level as a potential marker for poor prognosis in acute cerebral infarction[J]. Eur Neurol, 2018, 79(3-4):154-160.] [DOI] [PubMed] [Google Scholar]
- 16.BREA D, SOBRINO T, BLANCO M, et al. Usefulness of haptoglobin and serum amyloid A proteins as biomarkers for atherothrombotic ischemic stroke diagnosis confirmation. Atherosclerosis. 2009;205(2):561–567. doi: 10.1016/j.atherosclerosis.2008.12.028. [BREA D, SOBRINO T, BLANCO M, et al. Usefulness of haptoglobin and serum amyloid A proteins as biomarkers for atherothrombotic ischemic stroke diagnosis confirmation[J]. Atherosclerosis, 2009, 205(2):561-567.] [DOI] [PubMed] [Google Scholar]
- 17.LIU DA Z, JICKLING G C, ANDERB P, et al. Elevating microRNA-122 in blood improves outcomes after temporary middle cerebral artery occlusion in rats. J Cereb Blood Flow Metab. 2016;36(8):1374–1383. doi: 10.1177/0271678X15610786. [LIU DA Z, JICKLING G C, ANDERB P, et al. Elevating microRNA-122 in blood improves outcomes after temporary middle cerebral artery occlusion in rats[J]. J Cereb Blood Flow Metab, 2016, 36(8):1374-1383.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.LÕHMUSSAAR E, GSCHWENDTNER A, MUELLERJ C, et al. ALOX5AP gene and the PDE4D gene in a central European population of stroke patients. Stroke. 2005;36(4):731–736. doi: 10.1161/01.STR.0000157587.59821.87. [LÕHMUSSAAR E, GSCHWENDTNER A, MUELLERJ C, et al. ALOX5AP gene and the PDE4D gene in a central European population of stroke patients[J]. Stroke, 2005, 36(4):731-736.] [DOI] [PubMed] [Google Scholar]
- 19.EVANS J F, FERGUSON A D, MOSLEYR T, et al. What's all the FLAP about?:5-lipoxygenase-activating protein inhibitors for inflammatory diseases. Trends Pharmacol Sci. 2008;29(2):72–78. doi: 10.1016/j.tips.2007.11.006. [EVANS J F, FERGUSON A D, MOSLEYR T, et al. What's all the FLAP about?:5-lipoxygenase-activating protein inhibitors for inflammatory diseases[J]. Trends Pharmacol Sci, 2008, 29(2):72-78.] [DOI] [PubMed] [Google Scholar]
- 20.JOHANNES L, KAMBADUR R, LEE-HELLMICH H, et al. Antiviral determinants of rat Mx GTPases map to the carboxy-terminal half. J Virol. 1997;71(12):9792–9795. doi: 10.1128/JVI.71.12.9792-9795.1997. [JOHANNES L, KAMBADUR R, LEE-HELLMICH H, et al. Antiviral determinants of rat Mx GTPases map to the carboxy-terminal half[J]. J Virol, 1997, 71(12):9792-9795.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.ARNHEITER H, SKUNTZ S, NOTEBORN M, et al. Transgenic mice with intracellular immunity to influenza virus. http://d.old.wanfangdata.com.cn/OAPaper/oai_pubmedcentral.nih.gov_2698557. Cell. 1990;62(1):51–61. doi: 10.1016/0092-8674(90)90239-b. [ARNHEITER H, SKUNTZ S, NOTEBORN M, et al. Transgenic mice with intracellular immunity to influenza virus[J]. Cell, 1990, 62(1):51-61.] [DOI] [PubMed] [Google Scholar]
- 22.DÉCAILLOT F M, ROZENFELD R, GUPTA A, et al. Cell surface targeting of mu-delta opioid receptor heterodimers by RTP4. Proc Natl Acad Sci U S A. 2008;105(41):16045–16050. doi: 10.1073/pnas.0804106105. [DÉCAILLOT F M, ROZENFELD R, GUPTA A, et al. Cell surface targeting of mu-delta opioid receptor heterodimers by RTP4[J]. Proc Natl Acad Sci U S A, 2008, 105(41):16045-16050.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.SCHOGGINS J W, WILSON S J, PANIS M, et al. A diverse range of gene products are effectors of the type Ⅰ interferon antiviral response. Nature. 2011;472(7344):481–485. doi: 10.1038/nature09907. [SCHOGGINS J W, WILSON S J, PANIS M, et al. A diverse range of gene products are effectors of the type Ⅰ interferon antiviral response[J]. Nature, 2011, 472(7344):481-485.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.MCDONOUGH A, LEE R V, WEINSTEIN J R. Microglial interferon signaling and white matter. Neurochem Res. 2017;42(9):2625–2638. doi: 10.1007/s11064-017-2307-8. [MCDONOUGH A, LEE R V, WEINSTEIN J R. Microglial interferon signaling and white matter[J]. Neurochem Res, 2017, 42(9):2625-2638.] [DOI] [PMC free article] [PubMed] [Google Scholar]


