Abstract
分子模拟作为传统计算机模拟经典技术之一,可以直观地展示和量化分子结构,从微观分子水平解释实验现象。而当模拟体系增大时,计算量也会增加,对模拟系统造成很大的负担。粗粒化分子动力学属于介观尺度分子模拟的一种方法,能够简化分子结构,提高计算效率,所以在模拟药物载体材料等大分子体系时常选用粗粒化分子动力学。本文综述了近年来国内外利用粗粒化分子动力学模拟药物载体的研究成果,为未来药物制剂研究提供参考,以期促使药物研究更快地进入精准药物设计时代。
Keywords: 分子模拟, 粗粒化分子动力学, 药物载体材料
Abstract
As one of the traditional computer simulation techniques, molecular simulation can intuitively display and quantify molecular structure and explain experimental phenomena from the microscopic molecular level. When the simulation system increases, the amount of calculation will also increase, which will cause a great burden on the simulation system. Coarse-grained molecular dynamics is a method of mesoscopic molecular simulation, which can simplify the molecular structure and improve computational efficiency, as a result, coarse-grained molecular dynamics is often used when simulating macromolecular systems such as drug carrier materials. In this article, we reviewed the recent research results of using coarse-grained molecular dynamics to simulate drug carriers, in order to provide a reference for future pharmaceutical preparation research and accelerate the entry of drug research into the era of precision drug design.
Keywords: Molecular simulation, Coarse-grained molecular dynamics, Drug carrier material
0. 引言
药物的直接给药方式生物利用度低,在给药次数增多的同时可能会对机体产生一定的毒副作用,增加肝肾系统的负担[1]。药物载体材料能够有效地控制药物的释放,增加药物稳定性,降低药物毒副作用,实现靶向运输功能[2]。但筛选出合适的药物载体的成本是巨大的,需要考察药辅相容性、载体材料的生物相容性、包封率和靶向性等。计算机模拟技术的发展为筛选和设计药物载体材料提供了新的方法,如量子力学方法、全原子分子模拟和介观尺度分子模拟等方法,合理地利用分子模拟技术能高效地构建和筛选出合适的药物载体材料并进行评价。但是药物与药物载体材料体系复杂,分子量大,全原子模拟和量子力学方法存在时间和空间尺度上的限制,而介观尺度分子模拟中的粗粒化分子动力学(coarse-grained molecular dynamics,CGMD)可以简化复杂的分子系统,降低体系的计算量,计算效率更高,载体材料筛选时的随机性更小,大大提高了药物研发效率。
1. 分子模拟简介
分子模拟作为传统计算机模拟的经典技术之一,能够模拟实验研究不能解决的多体体系的实验情况。分子模拟常用的模拟方法有CGMD、蒙特卡洛法(Monte Carlo,MC)和分子动力学模拟(molecular dynamics,MD)。MC是第一个计算机模拟方法,但是模拟过程中缺少动态信息,局限性较大。MD体系内所有粒子的运动都以牛顿运动方程为基础,常用力场有CHARMM[6]、AMBER[7]和COMPASS[8]力场,适用范围较广。CGMD体系中粗粒化模型自由度较低,但依然是聚合物等大分子的最适合的模拟方法[9]。
2. 粗粒化分子动力学简介
CGMD是介观尺度分子模拟的一种方法,常选用Martini力场[10],Martini力场中分子间相互作用主要分为键势和非键势两种。与MD相比,CGMD能够模拟更长的时间尺度和更大的空间尺度,适用于较大的共聚物和多种生物分子等。CGMD在生物分子配合物领域也有重要作用[11],为多种生物分子提供重要参数,如脂质[12]、DNA[13]、蛋白质[14]等。
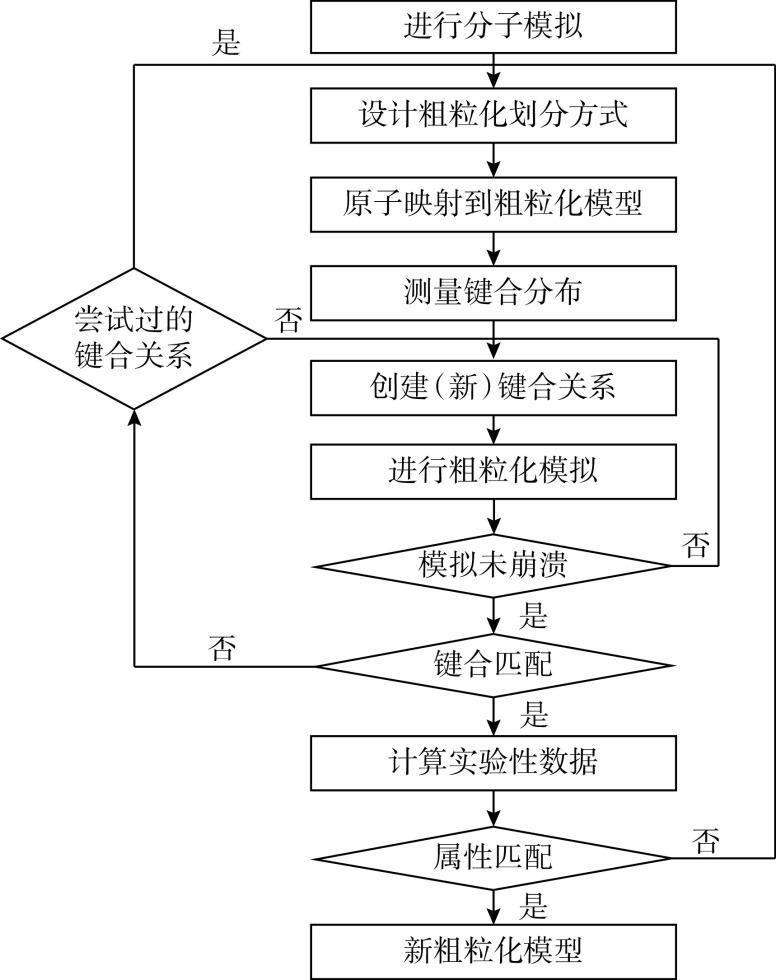
CGMD与耗散粒子动力学(dissipative particle dynamics,DPD)等计算机模拟不同,其粗粒化方式严格按照四合一映射成一个粗粒化(coarse-grained,CG)粒子。CG粒子主要分为四种类型:极性(polar)、非极性(non-polar)、近极性(apolar)、荷电(charged),每种类型的珠子可进一步划分为几种亚型,可以准确描述出分子、原子结构的化学性质,以再现小化合物在极性相和非极性相之间的分配自由能[15]。具体粗粒化过程操作如图1所示。
图 1.
Work flow chart of Martini CG model parameterization
Martini CG模型参数化的工作流程图
CGMD也有一定局限性,Martini力场仅对特定类型的分子组合进行参数化,且粗粒化后的分子体系过于粗糙,无法观察到一些细致的分子运动行为,如氢键的方向性[16],所以未来我们需要对粗粒化力场进行不断优化以提高模拟的准确度和重现性。
3. 粗粒化分子动力学在药物载体材料中的应用
许多实验研究已经证明CGMD可以模拟分子在复杂的人体环境内的运动规律,如Su等[17]在Martini粗粒化力场条件下寻找在水中最稳定的生物分子配合物,利用分子生物力场,研究受体与目标分子之间的作用机制;Liu等[18]通过分析CGMD得到的模拟结果,总结出球形囊泡破碎的机制,并预测其破碎条件;Zhang等[19]结合DPD和CGMD方法来研究血小板流变学的动态结构;Negami等[20]利用CGMD成功重现了蛋白质-配体的结合过程;Chen等[21]利用CGMD研究纳米粒子的表面电荷密度对肺表面活性物质的穿透作用。
上述研究成果表明CGMD不仅可以筛选出与药物最适合的载体材料并模拟出它们的结合过程,还可以考察它们在人体内的释放过程甚至是药物的作用机制,使模拟结果更加准确。
3.1. 纳米粒子设计
近几年,纳米制剂已经成为口服给药系统中的一个研究热门,它可以调节释药速度,增加生物膜的透过性以更深层次地达到病灶,达到靶向给药的效果,提高生物利用度[22]。纳米颗粒已经广泛应用于各种药物制剂,如虾青素[23]、姜黄素[24]、白蛋白[25]等。
纳米颗粒的弹性对其细胞摄取率和血液循环有着重要的影响。Shen等[26]为了研究纳米颗粒的内吞作用,开发了用于弹性纳米颗粒的粗粒度分子动力学模型,将单个脂质分子的头珠作为活性位点,用随机蒙特卡洛过程代表配体-受体键的形成与断裂,以每个尺寸的弹性纳米颗粒包裹效率为参考,弹性纳米颗粒和膜变形引起的能垒作为包裹效率的最关键因素,发现在纳米粒子变形时引起的能量损失导致软纳米颗粒的膜包裹速度比硬纳米颗粒包裹速度慢,与实验观察结果一致,这项研究为我们在设计纳米药物载体时提供了高效的CGMD计算模型。Hossain等[27]利用CGMD技术成功模拟出了磷脂包裹的金纳米颗粒与肺泡单分子层模型的分子水平的相互作用,通过不同的定性和定量分析(如肽聚类分析等)来显示肺表面的金纳米粒子的结构和动力学变化,更好地解释了纳米载体在肺表面的吸附与扩散过程,为设计新的更有效的纳米颗粒载体提供了参考。Souza等[28]使用肺表面活性物质模型评估了抗菌肽BP100对不同聚合物包覆的金纳米颗粒的吸附情况,通过CGMD计算出纳米粒子的吉布斯自由能,并评价纳米粒子在气相和水相中的稳定性,径向分布函数证明该纳米粒子会在水相中释放出抗菌肽BP-100,在其余模拟时间内都保持稳定,研究表明这种药物递送系统可以在体内和体外进行实验测试,是一种新型的肺部疾病治疗方法。
Ozmaian等[29]用CGMD模拟了一种通过控制聚合物涂层表面性能来选择优先与特定类型的纳米离子表面接枝,通过比较不同数量的惰性聚合物链的热波动强度来判断聚合物与胶体的分离情况,这种引入优先与某些类型的聚合物相互作用的纳米颗粒来改变胶体表面特性的机制,原则上也适用于药物输送系统和自组装领域。同样在药物自组装研究领域,Gong等[30]联用CGMD与DPD方法探索Eudragit S100与白头翁皂苷D自组装结肠靶向粉的pH响应性能,先使用CGMD模拟分析白头翁皂苷D与S100自组装性能对载药量的影响,再使用DPD方法模拟不同pH条件下的释放情况。这项研究结合了CGMD与DPD技术互补的特性,使计算机模拟研究更加科学完整,为传统计算机模拟在药物研究领域的多技术联用提供了参考。
3.2. 载药胶束
胶束(micelle)[31]通常由两亲性嵌段共聚物在水溶液中通过疏水作用自组装形成。它既可以避免药物直接与胃肠道表面接触,减少药物对胃肠道的刺激,又可以避免药物被网状内皮系统吞噬,延长药物在体内的作用时间,提高生物利用度。
Seif等[32]利用CGMD对聚氧乙烯-20-十六烷基醚与氟比洛芬在水中自组装胶束进行表征,在模拟体系中,通过最大限度地减少水和碳氢化合物之间的错误链接,保证胶束体系的稳定,模拟结果与实验结果完全吻合,从分子层面上解释了氟比洛芬与胶束的结合方式。之后,又使用CGMD研究了氟比洛芬的药代动力学特征和水合作用等,为设计相关的药物递送系统提供了方向。
Wu等[33]用CGMD研究了控制聚合物混合胶束(polymer mixed micelles,PMMs)空间分布的因素并预测其载药性能,通过GROMACS软件进行模拟,并用分子可视化程序(visual molecular dynamics,VDM)将模拟轨迹可视化,发现药物在PMM内的空间分布取决于药物与胶束材料所形成区域的疏水相容性。因此,我们可以通过药物-PMM相互作用和药物定位研究来调整载药稳定性和药物释放行为,Wu等[34]进一步用CGMD技术对载葛根素聚合物混合胶束(FS/PMMs)的载药行为进行研究,考察不同Pluronic F127与Solutol HS15配比形成的FS/PMMs的自聚集行为,通过计算动力学过程的溶剂可及表面积、回转半径以及能量分布等实验属性数据判断该体系的物理结构稳定性。该研究通过模拟与实验相结合,证明CGMD能够准确地优化复杂的胶束载药体系,是一种有效的药物载体材料研究工具。
Raman等[35]开发了一种适用于聚合物和两亲性大分子系统的粗粒化力场,相比于传统的Martini力场,可极化Martini力场的水具有三个相互作用中心,设置为“POL”珠而不是传统的“P4”珠,并且该Martini粗粒化力场有良好的温度可传递性,适用于不同温度的环境体系。这项研究所开发的Martini-CG模型具有重要的生物学意义,为后续进行多嵌段共聚物和其他更复杂系统的粗粒化模拟提供了理论支持。
还有一种特殊的胶束—反胶束。反胶束是在非水溶剂中自发形成的表面活性剂纳米聚集体。将反胶束作为亲水性药物的载体,可以有效控制药物的释放从而达到全身治疗的水平。Liu等[36]曾用CGMD评估透皮载胰岛素反胶束的渗透机制以改善透皮药物的递送,发现胰岛素的释放速率与反胶束的黏度有关。Liu等的实验为我们以后制备持续有效释药的透皮制剂提供了一种模拟评价方法,为CGMD在反胶束这种药物载体材料中的研究提供了新颖的概念验证数据。
3.3. 水凝胶
水凝胶(hydrogel)[37]是一类极为亲水的三维网络结构凝胶,是在水中具有溶胀而不溶解特性的聚合物材料。由于其高吸水性、良好的生物相容性和对组织较小的刺激性,水凝胶在生物医学、组织工程等领域得到广泛应用。
Salahshoor等[38]在水对多尺度交联水凝胶影响的MD研究的基础上,提出了一种新的水凝胶粗粒化方案,研究发现使用Martini力场将水凝胶的全原子模型映射到CG模型后,CG模型可以更好地表示出系统的刚度。于是,Salahshoor等考察了含水量0%~80%的水凝胶的机械性能,并与全原子模型进行比较来验证结果的准确度。Salahshoor等的研究意味着CGMD的仿真体系进一步完善,能更准确全面地模拟出体系的各种属性。但是CGMD更适合干粉体系,在对水凝胶进行模拟时比较困难,所以在这项实验基础上,我们可以联用DPD等技术进行结构模拟然后使用CGMD对DPD的模拟结果进行评价。联用CGMD与其他计算机模拟技术进一步探究载药体系的不同条件及形态,能更充分地发挥计算机模拟的优势,提高模拟的效率和准确度。
水凝胶作为一种新型智能药物载体[39],有着极具潜力的发展前景,但CGMD在水凝胶载体材料方面的应用较少,是一个潜在的有价值的研究方向。
3.4. 脂质体
脂质体(liposome)[40]是一种将药物包封于类脂质双分子层内而形成的微型泡囊体,可作为疏水性药物的载体。将药物包裹于脂质体内,既可以提高靶向性,又能减少对正常组织的刺激或毒性,提高用药的安全性和治疗效果。
目前,脂质体是药物递送中应用极广泛的载体,尤其是在恶性肿瘤的靶向给药方面极有潜力[41]。Winter等[42]用CGMD模型研究溶血脂结合的脂质体的渗透性增强及其增强机制并考察了释放时间,描述了一种抗癌药物的新型递送系统。在模拟体系中,考虑到CGMD中水的扩散速率是实际情况下水扩散速度的四倍,所以有效时间尺度也是模拟实际时间尺度的四倍,并将水作为探针来考察释放速度,合理推测溶血脂是否能够有效地增强脂质体的渗透性。
由于对脂质体制剂内部结构的认知较少,Larsson等[43]将一个C1磁珠和三个Na珠作为长链的头部,用C1珠作为烷基链将脂质体进行粗粒化建模,用CGMD模拟出脂质复杂的内部环境,通过模拟研究不同生理条件的系统,如改变脂质体组成来模拟消化过程,并与实验中测得的载药能力进行数据对比分析。实验证明CGMD可以高效地进行纳米结构的虚拟筛选和基于脂质体的药物递送系统的相变。Rezaei等[44]合成了不同比例的磷酸胆碱脂质体制剂,有效增加了相关肽的稳定性和血浆半衰期,再用CGMD模拟研究肽-脂质体的相互作用,发现肽的自组装倾向与水分子的接触较少,证明肽是一种两亲性物质,既无法在脂质体的双层膜中停留也无法在内部扩散,解释了脂质体的稳定性与缓释的原因。
CGMD还可以用来模拟药物对细胞的毒性作用,如Li等[45]用CGMD模拟研究脂质体和疏水纳米片之间的相互作用,将纳米片不同的尺寸、方向、亲油性等输入与不同特征相互作用状态的输出对应,模拟药物对细胞结构的破坏和毒性作用。这项实验结果促进了我们对纳米材料细胞毒性在分子水平的理解,揭示了丰富的动力学和热力学细节,并为未来我们将计算机模型与实验相关联的纳米毒理研究提供了帮助。
4. 结论与展望
由于人体内复杂的生理环境和组织结构,极大地限制了临床药物的治疗效果和生物利用度,制备合适的药物载体材料是目前最有潜力的药物制剂解决途径之一。本文系统地阐述了CGMD在药物载体材料研究中的应用。CGMD既可以模拟药物以及载体材料各自的特性,又减少了模拟的计算量,对研究各种药物体系提供了巨大的帮助。通过CGMD模拟和实验结果相互验证,相辅相成,可以不断深入在药物制剂领域中的研究。同时,因为药物组方多、成分复杂,对药物制剂的制备工艺要求越来越高,CGMD模拟可以避免许多不必要的实验错误,进行精确的实验研究,大大减少了实验成本。未来在药学研究领域,我们也将进入精准药物设计时代,因此CGMD在未来药物载体材料研究中有着十分重要的意义。
重要声明
利益冲突申明:本文全体作者均声明不存在利益冲突。
作者贡献说明:张民权负责文献搜集、整理、概括及文章撰写;龚铭城负责文献收集、修改文章;王进负责文献调研、修改文章;陈振华和周良良负责文章选题、指导和审阅文章。
Funding Statement
江西省杰出青年基金(20224ACB216019);江西省自然科学基金青年基金(20202BABL216026);江西省教育厅科技计划研究项目(GJJ201134);江西科技师范大学博士启动基金(2019BSQD015)
Science and Technology Department of JiangXi Province; Science and Technology Department of JiangXi Province; Department of Education of Jiangxi Province; Scientific Research Department of Jiangxi Science and Technology Normal University
Contributor Information
振华 陈 (Zhenhua CHEN), Email: zhenhuachen@jxstnu.edu.cn.
良良 周 (Liangliang ZHOU), Email: zhoulliang@163.com.
References
- 1.Lan Y, Sun Y, Yang T, et al Co-delivery of paclitaxel by a capsaicin prodrug micelle facilitating for combination therapy on breast cancer. Mol Pharm. 2019;16(8):3430–3440. doi: 10.1021/acs.molpharmaceut.9b00209. [DOI] [PubMed] [Google Scholar]
- 2.马骏威, 安娜 冻干注射剂中辅料选择的考虑. 中国药学杂志. 2020;55(7):568–572. [Google Scholar]
- 3.Falin T, Tongtao Y, Ye L, et al Computer simulation studies on the interactions between nanoparticles and cell membrane. Sci China Chem. 2014;57(12):1662–1671. doi: 10.1007/s11426-014-5231-7. [DOI] [Google Scholar]
- 4.Badu S, Prabhakar S, Melnik R Coarse-grained models of RNA nanotubes for large time scale studies in biomedical applications. Biomedicines. 2020;8(7):195. doi: 10.3390/biomedicines8070195. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.Marchetto A, Chaib Z S, Rossi C A, et al CGMD platform: integrated web servers for the preparation, running, and analysis of coarse-grained molecular dynamics simulations. Molecules. 2020;25(24):5934. doi: 10.3390/molecules25245934. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Allen C, Bureau H R, Mcgee T D, et al Benchmarking adaptive steered molecular dynamics (ASMD) on charmm force fields. Chemphyschem. 2022;23(17):e202200175. doi: 10.1002/cphc.202200175. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Vani I, Dans P D, Noy A, et al A refined force field for DNA simulations. Nat Methods. 2016;13(1):55–58. doi: 10.1038/nmeth.3658. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Savin A, Mazo M A The compass force field: validation for carbon nanoribbons. Physica E. 2020;118:113937. doi: 10.1016/j.physe.2019.113937. [DOI] [Google Scholar]
- 9.龚铭城, 周良良, 马欣悦, 等 分子模拟在纳米载药材料中的应用研究进展. 高分子通报. 2022;280(8):21–28. [Google Scholar]
- 10.MacCallum J L, Hu S, Lenz S, et al An implementation of the Martini coarse-grained force field in OpenMM. Biophys J. 2023;122(14):2864–2870. doi: 10.1016/j.bpj.2023.04.007. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Takada S, Kanda R, Tan C, et al Modeling structural dynamics of biomolecular complexes by coarse-grained molecular simulations. Acc Chem Res. 2015;48(12):3026–3035. doi: 10.1021/acs.accounts.5b00338. [DOI] [PubMed] [Google Scholar]
- 12.Moore T C, Iacovella C R, Hartkamp R, et al A coarse-grained model of stratum corneum lipids: free fatty acids and ceramidens. J Phys Chem B. 2016;120(37):9944–9958. doi: 10.1021/acs.jpcb.6b08046. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Bae S, Oh I, Yoo J, et al Effect of DNA flexibility on complex formation of a cationic nanoparticle with double-stranded DNA. ACS Omega. 2021;6(29):18728–18736. doi: 10.1021/acsomega.1c01709. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Majumder A, Straub J E Addressing the excessive aggregation of membrane proteins in the Martini model. J Chemical Theory Comput. 2021;17(4):2513–2521. doi: 10.1021/acs.jctc.0c01253. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Alessandri R, Grunewald F, Marrink S J The Martini model in materials science. Adv Mater. 2021;33(24):2008635. doi: 10.1002/adma.202008635. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.De Jong D H, Singh G, Bennett W F D, et al Improved parameters for the Martini coarse-grained protein force field. J Chem Theory Comput. 2013;9(1):687–697. doi: 10.1021/ct300646g. [DOI] [PubMed] [Google Scholar]
- 17.Su C H, Chen H L, Ju S P, et al Exploring the most stable aptamer/target molecule complex by the stochastic tunnelling-basin hopping-discrete molecular dynamics method. Sci Rep. 2021;11(1):11406. doi: 10.1038/s41598-021-90907-y. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Liu P, Li J, Zhang Y W Breakup of spherical vesicles caused by spontaneous curvature change. Acta Mech Sin. 2012;28(6):1545–1550. doi: 10.1007/s10409-012-0165-9. [DOI] [Google Scholar]
- 19.Zhang P, Zhang N, Deng Y, et al A multiple time stepping algorithm for efficient multiscale modeling of platelets flowing in blood plasma. J Comput Phys. 2015;284:668–686. doi: 10.1016/j.jcp.2015.01.004. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Negami T, Shimizu K, Terada T Coarse-grained molecular dynamics simulations of protein-ligand binding. J Comput Chem. 2014;35(25):1835–1845. doi: 10.1002/jcc.23693. [DOI] [PubMed] [Google Scholar]
- 21.Chen P, Zhang Z, Gu N, et al Effect of the surface charge density of nanoparticles on their translocation across pulmonary surfactant monolayer: a molecular dynamics simulation. Mol Simulat. 2018;44(2):85–93. doi: 10.1080/08927022.2017.1342118. [DOI] [Google Scholar]
- 22.Anjum S, Ishaque S, Fatima H, et al Emerging applications of nanotechnology in healthcare systems: grand challenges and perspectives. Pharmaceuticals. 2021;14(8):707. doi: 10.3390/ph14080707. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.Zhu Y, Gu Z, Liao Y, et al Improved intestinal absorption and oral bioavailability of astaxanthin using poly(ethylene glycol)‐graft‐chitosan nanoparticles: preparation, in vitro evaluation, and pharmacokinetics in rats. J Sci Food Agric. 2022;102(3):1002–1011. doi: 10.1002/jsfa.11435. [DOI] [PubMed] [Google Scholar]
- 24.Del P M, Caballero F I, Meza-toledo J, et al Formulations of curcumin nanoparticles for brain diseases. Biomolecules. 2019;9(2):56. doi: 10.3390/biom9020056. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.Guo H, Tai Z, Liu F, et al Research and application of kupffer cell thresholds for BSA nanoparticles. Molecules. 2023;28(2):880. doi: 10.3390/molecules28020880. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.Shen Z, Ye H, Li Y Understanding receptor-mediated endocytosis of elastic nanoparticles through coarse grained molecular dynamic simulation. Phys Chem Chem Phys. 2018;20(24):16372–16385. doi: 10.1039/C7CP08644J. [DOI] [PubMed] [Google Scholar]
- 27.Hossain S, Gandhi N S, Hughes Z E, et al Computational studies of lipid-wrapped gold nanoparticle transport through model lung surfactant monolayers. J Phys Chem B. 2021;125(5):1392–1401. doi: 10.1021/acs.jpcb.0c09518. [DOI] [PubMed] [Google Scholar]
- 28.Souza F R, Fornasier F, Carvalho A S, et al Polymer-coated gold nanoparticles and polymeric nanoparticles as nanocarrier of the BP100 antimicrobial peptide through a lung surfactant model. J Mol Liq. 2020;314:113661. doi: 10.1016/j.molliq.2020.113661. [DOI] [Google Scholar]
- 29.Ozmaian M, Freitas B A, Coalson R D Controlling the surface properties of binary polymer brush-coated colloids via targeted nanoparticles. J Phys Chem B. 2019;123(1):258–265. doi: 10.1021/acs.jpcb.8b05520. [DOI] [PubMed] [Google Scholar]
- 30.Gong M, Chen Z, Zhou L, et al Application of mesoscale simulation to explore the pH response of Eudragit S100 used as the novel colon-targeted powder of Pulsatilla saponin D. J Nanomater. 2021;2021:9556911. [Google Scholar]
- 31.Bu X, Ji N, Dai L, et al Self-assembled micelles based on amphiphilic biopolymers for delivery of functional ingredients. Trends Food Sci Tech. 2021;114:386–398. doi: 10.1016/j.tifs.2021.06.001. [DOI] [Google Scholar]
- 32.Seif M, Montazeri A Molecular dynamics simulation reveals the reliability of Brij-58 nanomicellar drug delivery systems for flurbiprofen. J Mol Liq. 2022;360:119496. doi: 10.1016/j.molliq.2022.119496. [DOI] [Google Scholar]
- 33.Wu W, Gu Y, Li W, et al Understanding the synergistic correlation between the spatial distribution of drug-loaded mixed micellar systems and in vitro behavior via experimental and computational approaches. Mol Pharm. 2021;18(4):1643–1655. doi: 10.1021/acs.molpharmaceut.0c01095. [DOI] [PubMed] [Google Scholar]
- 34.Wu W, Zou Z, Yang S, et al Coarse-grained molecular dynamic and experimental studies on self-assembly behavior of nonionic F127/HS15 mixed micellar systems. Langmuir. 2020;36(8):2082–2092. doi: 10.1021/acs.langmuir.9b03936. [DOI] [PubMed] [Google Scholar]
- 35.Raman A S, Vishnyakov A, Chiew Y C A coarse-grained model for PCL: conformation, self-assembly of MePEG-b-PCL amphiphilic diblock copolymers. Mol Simulat. 2017;43(2):92–101. doi: 10.1080/08927022.2016.1233550. [DOI] [Google Scholar]
- 36.Liu Y, Zhao F, Dun J, et al Lecithin/isopropyl myristate reverse micelles as transdermal insulin carriers: Experimental evaluation and molecular dynamics simulation. J Drug Deliv Sci Technol. 2020;59:101891. doi: 10.1016/j.jddst.2020.101891. [DOI] [Google Scholar]
- 37.侯萍, 李铭, 马军, 等 天然高分子材料水凝胶的制备及其应用进展. 高分子通报. 2022;280(8):29–36. [Google Scholar]
- 38.Salahshoor H, Rahbar N Multi-scale mechanical and transport properties of a hydrogel. J Mech Behav Biomed Mater. 2014;37:299–306. doi: 10.1016/j.jmbbm.2014.05.028. [DOI] [PubMed] [Google Scholar]
- 39.Wang L, Zhou M, Xu T, et al Multifunctional hydrogel as wound dressing for intelligent wound monitoring. Chem Eng J. 2022;433:134625. doi: 10.1016/j.cej.2022.134625. [DOI] [Google Scholar]
- 40.张曼玉, 楼晨曦, 曹傲能 主动靶向载药脂质体在肿瘤治疗中的研究进展. 生物医学工程学杂志. 2022;39(3):633–638. doi: 10.7507/1001-5515.202110067. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 41.Yang B, Song B P, Shankar S, et al Recent advances in liposome formulations for breast cancer therapeutics. Cell Mol Life Sci. 2021;78(13):5225–5243. doi: 10.1007/s00018-021-03850-6. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 42.Winter N D, Murphy R K J, O’halloran T V, et al Development and modeling of arsenic-trioxide-loaded thermosensitive liposomes for anticancer drug delivery. J Liposome Res. 2011;21(2):106–115. doi: 10.3109/08982104.2010.483597. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 43.Larsson P, Alskar L C, Bergstrom C A S Molecular structuring and phase transition of lipid-based formulations upon water dispersion: a coarse-grained molecular dynamics simulation approach. Mol Pharm. 2017;14(12):4145–4153. doi: 10.1021/acs.molpharmaceut.7b00397. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 44.Rezaei N, Mehrnejad F, Vaezi Z, et al Encapsulation of an endostatin peptide in liposomes: Stability, release, and cytotoxicity study. Colloids Surf B Biointerfaces. 2020;185:110552. doi: 10.1016/j.colsurfb.2019.110552. [DOI] [PubMed] [Google Scholar]
- 45.Li Z, Zhang Y, Ma J, et al Modeling interactions between liposomes and hydrophobic nanosheets. Small. 2019;15(6):1804992. doi: 10.1002/smll.201804992. [DOI] [PubMed] [Google Scholar]