Abstract
目的
采用两样本孟德尔随机化(MR)方法探究1型糖尿病(T1DM)和甲状腺毒症之间的关系。
方法
利用IEU OpenGWAS project网站获取T1DM及甲状腺毒症的全基因组关联研究(GWAS)数据。筛选与T1DM密切相关且独立的单核苷酸多态性(SNPs)作为工具变量(IVs),用MR-PRESSO进行离群值检验并剔除离群值,分别运用逆方差加权法(IVW)、MREgger回归、加权众数法(WM)和加权中位数法(WME)进MR分析,以OR值及95% CI评价T1DM与甲状腺毒症之间是否存在关联。分别采用IVW和MR-Egger法进行Cochran Q和Rücker Q异质性检验,用Egger-intercept法进行多效性检验、逐个剔除检验进行敏感性分析,并计算F值以评估是否存在弱IVs偏倚。
结果
4种MR方法所得结果显示,整体人群中T1DM与甲状腺毒症具有正向的因果关系,且具有显著的统计学意义,IVW、MR-Egger回归、加权模型和WME计算所得OR值和95% CI分别为1.077(95% CI 1.046~1.109)、1.076(95% CI 1.031~1.124)、1.082(95% CI 1.048~1.118)、1.090(95% CI 1.052~1.129)。异质性检验结果分别为P=0.127和P=0.155,即不存在异质性;Egger-intercept结果为P=0.965,即不存在多效性;敏感性分析显示结果稳定;全部F值大于10,提示不存在弱IVs偏倚。
结论
T1DM可能会增加甲状腺毒症的风险。
Keywords: 1型糖尿病, 甲状腺毒症, 孟德尔随机化
Abstract
Objective
To explore the relationship between type 1 diabetes (T1DM) and thyrotoxicosis using two-sample Mendelian randomization (MR) method.
Methods
Based on the data from a large-scale metabolome-based genome-wide association study (GWAS), we investigated the causality between T1DM and thyrotoxicosis using inverse variance-weighted (IVW) method, MR-Egger regression, weighted Mode (WM) method and weighted median (WME) method. Single nucleotide polymorphisms (SNPs) closely related to T1DM were screened as the instrumental variables (IVs). Outlier testing was performed using MR-PRESSON to reject the outliers. Heterogeneity tests, horizontal pleiotropy and sensitivity tests were performed to evaluate the reliability and stability of the results, and F-values were calculated to assess the presence of weak IVs bias.
Results
There was a positive causal effect between T1DM and thyrotoxicosis in the total samples analyzed with the 4 MR methods. The ORs and 95% CIs calculated by IVW, MR-Egger regression, WM and WME methods were 1.077 (95% CI: 1.046-1.109), 1.076 (95% CI: 1.031-1.124), 1.082 (95% CI: 1.048-1.118), and 1.090 (95% CI: 1.052-1.129), respectively. The results of the heterogeneity test were P=0.127 and P=0.155, respectively, suggesting the absence of heterogeneity. Egger-intercept result was P=0.965, indicating the absence of pleiotropy. Leave-one-out analysis showed stable results. All the F values were greater than 10, indicating that there was no weak IVs bias.
Conclusion
T1DM is likely to increase the risk of thyrotoxicosis.
Keywords: type 1 diabetes, thyrotoxicosis, Mendelian randomization
甲状腺毒症是组织和循环中甲状腺激素水平过高的总称,分为与甲亢相关的甲状腺毒症和非甲亢相关的甲状腺毒症。在碘充足地区,非医源性甲状腺毒症最常见的原因是格雷夫斯病(GD),约占80%。目前认为,GD发病风险主要与遗传因素有关,许多与GD风险增加的相关基因与其他自身免疫性疾病(如T1DM)相关基因重叠[1]。在中国,临床甲亢患病率0.78%、亚临床甲亢0.44%、GD患病率0.53%。未经治疗的甲亢甚至亚临床甲亢可导致心房颤动、卒中和其他心血管事件以及骨质疏松症和骨折的发生风险增加[2]。T1DM是一种慢性自身免疫性疾病,以胰岛素缺乏和高血糖为特征[3],T1DM的患病率在全球范围内呈上升趋势,全球患病率为每万人9.5例[4]。许多观察性研究发现,T1DM和甲状腺毒症密切相关,印度的一项横断面研究显示,130名GD患者中,3.1%伴有T1DM[5];英国的一项横断面研究发现,2791例GD患者种1.11%伴有T1DM[6];一项1993年到2010年的前瞻性研究表示,在3209名白种人GD患者中,观察到T1DM明显高于对照组[7]。但是上述研究未能说明二者因果关系,且观察性研究易受到反向因果关系和混杂因素干扰,从而使因果关系推断受限。
孟德尔随机化(MR)分析方法近年被广泛用于流行病学的因果关系研究。MR研究的基本原理是配子形成中亲代等位基因随机分配给子代,相当于对人群进行随机分组,与随机对照试验类似[8]。由于遗传变异出生时即固定并且伴随一生,因此MR研究能最大限度减少反向因果关系[9]。将遗传变异作为工具变量(IVs)对暴露因素与结局的因果关系进行推断,能减少混杂因素的干扰[10]。
以往的T1DM和甲状腺毒症研究大多是基于观察性的研究,容易受混杂因素或者反向因果关系的影响,而MR分析能最大限度避免二者干扰,但目前尚未有T1DM和甲状腺毒症的MR分析。本研究运用两样本MR方法,以单核苷酸多态性(SNPs)作为IVs,研究T1DM和甲状腺毒症之间的因果关系。由于甲状腺激素过多会影响患者血糖控制、损伤胰岛细胞功能以及降低胰岛素敏感性[11],因此明确二者关系,不仅有助于尽早发现并及时治疗甲状腺毒症,减轻甲状腺毒症导致的危害,也有助于糖尿病患者更好地控制血糖。同时,本次研究为T1DM和甲状腺毒症之间的因果关系提供了统计学上的线索,为实验研究和机制探索提供了理论依据。
1. 资料和方法
1.1. 研究设计
本文利用T1DM作为暴露因素,与T1DM显著相关的SNPs作为IVs,结局变量为甲状腺毒症。剔除离群值后,运用两样本MR分析的方法进行因果分析,并进异质性检验和多效性检验,最后对结果的可靠性进行检验。
MR研究中的IVs需满足三个核心假设:(1)IVs与暴露因素高度相关;(2)IVs仅通过暴露因素影响结局,不与结局直接相关;(3)IVs与“暴露-结局”关联的混杂因素无关(图 1)。
图 1.

两样本MR研究示意图
Model of the two-sample Mendelian randomization (MR) analysis.
1.2. 资料来源
通过https://gwas.mrcieu.ac.uk/网站分别获取T1DM和甲状腺毒症的GWAS。其中T1DM数据来源于EBI GWAS数据库,甲状腺毒症数据来源于芬兰FinnGen数据库,以上数据人源均来自欧洲(表 1)。
表 1.
两样本MR研究GWAS数据简要信息
Summary of the GWAS included in this two-sample MR study
| Name | ID | Sample size | No. of SNPs | Race | Race | Race |
| T1DM | ebi-a-GCST010681 | 24 840 | 12 783 129 | European | Male and Female | 2020 |
| Thyrotoxicosis | finn-b-THYROTOXICOSIS | 217 835 | 16 380 464 | European | Male and Female | 2021 |
1.3. IVs的筛选
依据MR的核心假设进行IVs筛选。以欧洲千人全基因组为参照,设置P<5×10-8筛选与T1DM有显著意义的SNPs,以满足关联性假设。使用R4.2.2软件的TwoSampleMR包进行clump计算,设置参数r2=0.001,kb=10 000,以排除连锁不平衡的影响,保证SNPs之间相互独立[12]。从甲状腺毒症的GWAS数据中提取上述与T1DM密切相关的SNPs,对两者进行整理合并。利用MR-PRESSO进行离群值检验以去除水平多效性[13],删除离群值同时剔除与甲状腺毒症直接相关的SNPs (P<5×10-8)[14]。
1.4. 统计学处理
1.4.1. T1DM与甲状腺毒症的因果效应估计
主要采用逆方差加权法(IVW)[15]进行MR分析,用MR-Egger回归[17]、加权众数法(WM)[17]和加权中位数法(WME)[18]进行补充。IVW主要用于因果关系估计,其估计值可视作结局中的IVs在暴露因素中加权线性回归的斜率,且截距项视为0,当所选SNPs都是有效的IVs时,IVW可以提供准确的估计值[15]。MR-Egger法同样以斜率作为因果效应估计值,但是该法考虑截距项的存在,且其截距可用于评估IVs之间的多效性[16]。WM方法根据因果效应的相似性将SNPs聚类为子集,进而估计具有最大数量SNPs的子集的因果效应[17]。WME法在无效IVs高达一半时,仍能提供一致性的因果效应估计值[18]。采用IVW和MR-Egger法对IVs进行Cochran Q和Rücker Q异质性检验[19, 20]。
1.4.2. 可靠性评价
1.4.2.1. 多效性检测
多效性检验目前普遍采用MREgger回归的截距项来表示,当该截距项接近于0时,表示不存在多效性,可认为排他性假设成立[21, 22]。
1.4.2.2. 敏感性分析
采用逐个剔除检验进行敏感性分析[23]。该法逐一剔除SNPs后计算剩下SNPs的结果,若剔除某一SNPs后结果改变很大,说明该SNPs对结果影响很大。
1.4.3. 弱IVs检验
分别计算单个SNPs的F值,对所选IVs进行弱IVs偏倚检验。F=β2 exposure /SE2 exposure,β为暴露的等位基因效应值,SE为暴露的标准误[24]。
上述方法均通过R4.2.2软件的TwoSampleMR包实现,检验水准α=0.05。
2. 结果
2.1. 筛选出的IVs
经过相关性和去除连锁不平衡筛选后,得到44个SNPs,合并整理并删除缺失项后,得到37个SNPs,经MR-PRESSO检验得到5个离群值,删除离群值最终得到32个SNPs (表 2)。
表 2.
MR分析SNPs信息表
Basic information of the single nucleotide polymorphisms (SNPs)
| SNPs | chr | EA | OA | EAF | T1DM | Thyrotoxicosis | F | ||||
| β | SE | P | β | SE | P | ||||||
| SNPs: Single nucleotide polymorphisms; chr: Chromosome; EA: Effect allele; OA: Other allele; EAF: Effect allele frequency; β: Effect allele value; SE: Standard error; F: Statistics for assessing the impact of weak instrumental variables. | |||||||||||
| rs10183097 | 2 | C | T | 0.1362 | 0.205 | 0.032 | 1.82E-10 | -0.009 | 0.032 | 0.79 | 40.651 |
| rs10760335 | 9 | G | A | 0.3205 | 0.136 | 0.024 | 2.43E-08 | 0.038 | 0.025 | 0.129 | 31.185 |
| rs10774624 | 12 | A | G | 0.5037 | -0.256 | 0.024 | 1.34E-25 | -0.045 | 0.024 | 0.057 | 109.734 |
| rs10830227 | 11 | A | G | 0.5741 | 0.158 | 0.023 | 1.02E-11 | 0.026 | 0.023 | 0.268 | 46.1 |
| rs10865468 | 2 | C | G | 0.2527 | -0.162 | 0.028 | 4.66E-09 | 0.016 | 0.025 | 0.531 | 34.373 |
| rs10911399 | 1 | G | A | 0.0455 | -0.371 | 0.064 | 6.75E-09 | 0.114 | 0.07 | 0.107 | 33.549 |
| rs12722495 | 10 | C | T | 0.1122 | -0.315 | 0.041 | 1.27E-14 | -0.062 | 0.047 | 0.185 | 59.418 |
| rs13182737 | 5 | A | G | 0.2553 | 0.147 | 0.026 | 1.49E-08 | -0.037 | 0.025 | 0.148 | 31.995 |
| rs17125653 | 14 | A | T | 0.077 | 0.236 | 0.040 | 4.75E-09 | 0.021 | 0.034 | 0.536 | 34.319 |
| rs1869449 | 2 | A | G | 0.2967 | 0.177 | 0.027 | 4.55E-11 | 0.002 | 0.026 | 0.927 | 43.246 |
| rs19232474 | 2 | G | T | 0.0131 | 0.562 | 0.088 | 1.37E-10 | 0.262 | 0.095 | 0.006 | 41.253 |
| rs194749 | 14 | C | T | 0.2455 | -0.164 | 0.028 | 5.37E-09 | -0.053 | 0.026 | 0.041 | 33.979 |
| rs202520 | 20 | G | A | 0.7222 | -0.157 | 0.026 | 7.97E-10 | -0.004 | 0.024 | 0.875 | 37.756 |
| rs206763 | 6 | A | G | 0.0202 | 0.679 | 0.078 | 2.93E-18 | -0.038 | 0.094 | 0.683 | 76.019 |
| rs2071647 | 12 | A | T | 0.2755 | 0.153 | 0.026 | 3.30E-09 | 0.009 | 0.026 | 0.744 | 34.984 |
| rs2111485 | 2 | G | A | 0.6033 | 0.158 | 0.025 | 1.89E-10 | 0.009 | 0.023 | 0.710 | 40.435 |
| rs2269247 | 1 | T | C | 0.1804 | 0.171 | 0.030 | 7.28E-09 | -0.003 | 0.025 | 0.921 | 33.561 |
| rs231971 | 16 | G | A | 0.1026 | 0.241 | 0.040 | 1.55E-09 | 0.105 | 0.060 | 0.083 | 36.513 |
| rs34296259 | 9 | A | T | 0.0082 | 0.664 | 0.117 | 1.43E-08 | -0.039 | 0.052 | 0.459 | 32.124 |
| rs34536443 | 19 | C | G | 0.0515 | -0.412 | 0.067 | 4.84E-10 | -0.045 | 0.067 | 0.499 | 38.739 |
| rs4566101 | 15 | C | T | 0.2697 | 0.176 | 0.026 | 6.23E-12 | 0.007 | 0.026 | 0.778 | 47.367 |
| rs506770 | 6 | C | G | 0.768 | 1.005 | 0.043 | 3.33E-123 | 0.092 | 0.028 | 0.001 | 556.34 |
| rs55996894 | 14 | C | G | 0.204 | -0.179 | 0.032 | 3.13E-08 | -0.037 | 0.033 | 0.251 | 30.540 |
| rs59680223 | 12 | T | C | 0.0081 | 0.642 | 0.103 | 5.00E-10 | 0.135 | 0.093 | 0.146 | 38.712 |
| rs62410259 | 4 | A | G | 0.077 | -0.380 | 0.053 | 1.02E-12 | -0.038 | 0.043 | 0.371 | 50.722 |
| rs6719660 | 2 | G | A | 0.9354 | 0.292 | 0.052 | 2.52E-08 | -0.024 | 0.059 | 0.690 | 31.010 |
| rs689 | 11 | T | A | 0.7109 | 0.700 | 0.035 | 2.30E-87 | -0.003 | 0.029 | 0.920 | 391.459 |
| rs6909461 | 6 | C | A | 0.2529 | -0.314 | 0.033 | 3.06E-21 | 0.011 | 0.030 | 0.722 | 89.451 |
| rs741172 | 16 | T | C | 0.3205 | -0.203 | 0.026 | 3.11E-15 | -0.029 | 0.026 | 0.249 | 62.153 |
| rs8056814 | 16 | A | G | 0.0793 | 0.264 | 0.042 | 1.99E-10 | 0.094 | 0.042 | 0.0246 | 40.499 |
| rs9273363 | 6 | A | C | 0.3317 | 1.279 | 0.033 | 1.00E-200 | 0.108 | 0.027 | 5.91E-05 | 1465.47 |
| rs9468618 | 6 | T | C | 0.0975 | -0.301 | 0.049 | 7.53E-10 | -0.301 | 0.050 | 0.230 | 37.864 |
2.2. 两样本MR结果
IVW法结果显示,OR值及95% CI均>1,且P值< 0.05,具有显著统计学意义(OR=1.077,95% CI:1.046~ 1.109,P=7.201×10-7),即说明整体人群中T1DM与甲状腺毒症具有正向的因果关系。MR-Egger回归、WM和WME计算的T1DM与甲状腺毒症之间的OR值、95% CI和P值分别为(OR=1.076,95% CI:1.031~1.124,P=0.002)、(OR=1.082,95% CI:1.048~1.118,P=4.375× 10-5)、(OR=1.090,95% CI:1.052~1.129,P=2.003×10-6) (图 2)。MR-Egger回归、WM和WME与IVW所得效应值及区间范围趋于一致,P值均有统计学意义,四种算法得到的因果效应方向一致(图 3),均显示T1DM是甲状腺毒症的危险因素。
图 2.
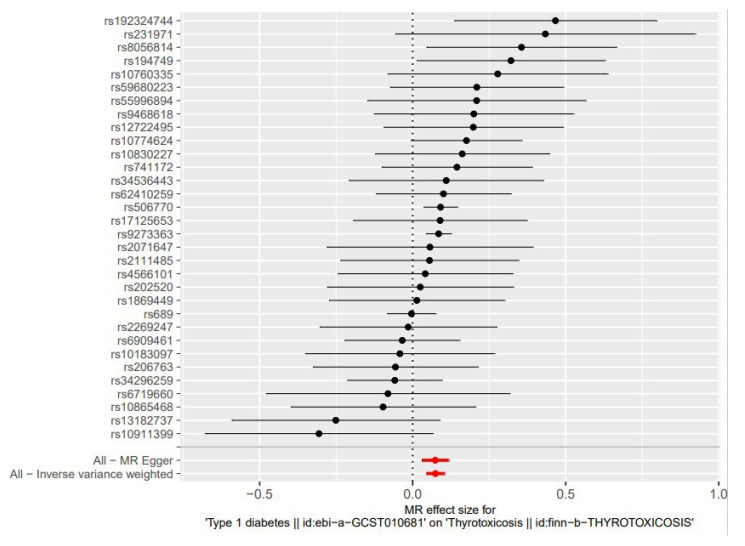
两样本MR结果森林图
Forest plot of the two-sample MR analysis.
图 3.
两样本MR结果散点图
Scatter plot of the two-sample MR analysis.
SNP effect on type 1 diabetes‖id: ebi-a-GCST010681

2.3. 异质性检验
异质性检验中,Cochran Q检验和Rücker Q检验结果分别为P=0.127和P=0.155,表明IVs间不存在异质性。
2.4. 可靠性评价
2.4.1. 多效性检测
MR-Egger回归的截距Eggerintercept=0.0004,P=0.965,表明因果效应分析结果并未受到多效性干扰,可认为排他性假设成立。
2.4.2. 敏感性分析
经逐个剔除检验,依次剔除单个SNPs,余下的31个SNPs分析结果与纳入全部SNPs的IVW分析结果相近,且都在无效线右侧(图 4),说明本次MR分析结果是稳健的。漏斗图显示使用单一SNPs作为IVs时,代表因果关系效应的点大致呈对称分布,表示受潜在偏倚影响的可能性小(图 5)。
图 4.

逐个剔除检验敏感性分析结果
"Leave-one-out" sensitivity analysis.
图 5.

两样本MR结果漏斗图
Funnel plot of the two-sample MR analysis.
2.5. 弱IVs检验
当F>10时,出现弱IVs的可能性比较小[25]。本次32个SNPs的F值范围在30.540~1465.470,无弱IVs偏倚,进一步验证了MR研究的关联性假设(表 2)。
3. 讨论
本次两样本MR分析验证了T1DM与甲状腺毒症之间的关联,IVW法、MR-Egger回归、WM和WME四种方法分析显示,T1DM与甲状腺毒症之间存在因果关系,且因果效应保持稳定。
一项12年全国范围、基于人群(中国台湾青少年和儿童)的回顾性队列研究显示,T1DM中甲状腺毒症的发生率显著高于对照组,发病率比为6.95[26]。一项纳入了761例东亚T1DM患者的前瞻性研究表明,4.1%的患者出现了GD[27]。美国一篇甲亢文章指出,甲状腺毒症的高危人群包括T1DM患者,并建议进行甲状腺毒症筛查[28]。
T1DM引起甲状腺毒症的机制很复杂,目前研究显示与自身免疫和遗传因素密切相关。GD和自身免疫性甲状腺炎是自身免疫性甲状腺疾病(AITD)的两种主要临床表现[29]。T1DM和AITD是两种常见的自身免疫性内分泌疾病,AITD常和其他自身免疫病相关,其中最常见的是T1DM[7, 25]。虽然两种疾病的发病机制存在差异,但流行病学数据显示,T1D和AITD在同一个体和家庭中存在聚集性,这表明这两种疾病具有共同的遗传基础[4]。高达25%的青少年T1DM患者有甲状腺相关抗体,长期随访显示,30%的T1DM患者会发展为AITD[4]。美国糖尿病协会(ADA)也描述了T1DM患者中17%~30%的AITD患病率[29]。目前研究发现,某些HLAII类自身等位基因的携带,与最常见的自身免疫性疾病发病概率的增加存在关联,经遗传学研究已经证实,少数基因同时具有患AITD和T1DM的风险。HLA-dr3是导致AITD和T1DM联合易感性的主要HLA II类等位基因,HLA-dr有独特的氨基酸特性,胰岛肽和甲状腺肽都能与之结合,并诱导甲状腺和胰岛特异性T细胞反应,从而在同一个体中触发T1DM和AITD[4]。除了HLA类基因外、CTLA-4、PTPN22、IL2Ra、VDR、TNF也被认为是潜在的遗传易感性位点[26]。然而不同人群对于基因易感性具有差异,如在阿拉伯和亚洲人群中,HLA易感等位基因与在欧洲和美国白种人人群中发现的不同,部分基因对T1DM和AITD的影响机制亦尚未阐明,未来还需要进一步研究。
由于甲状腺毒症会增强肝糖原异生,增加胰岛素抵抗,使T1DM患者的血糖难以控制稳定,甚至可能导致酮症酸中毒,积极治疗甲状腺毒症, 抑制甲状腺激素过多对T1DM患者糖代谢调节有重要意义。由于早期甲状腺毒症症状缺乏特异性,T1DM患者要及时进行相关检查,以免漏诊、误诊。甲状腺毒症的早发现早治疗,也有助于降低其带来的心血管事件及骨质疏松的发生风险。
本研究具有如下创新性:(1)以大样本GWAS数据为基础,从遗传学角度探讨了T1DM与甲状腺毒症的因果关系,和以往的观察性研究相比不易受混杂因素和反向因果关系干扰;(2)SNPs的筛选比较严格,对结局影响较大的SNPs进行了剔除,并运用了MR-PRESSO删除了离群值;(3)采用了4种MR分析方法,运用2种方法进行异质性检验、3种方法进行了可靠性评价,结果比较稳健。
本次MR研究存在的局限性:(1)纳入对象为欧洲人群,结论外推到其他人群中受限;(2)难以根据GWAS汇总数据按照性别、年龄、既往史进行分层分析;(3)本研究为统计学结果,无法进一步探讨其中机制。后续研究还需在不同人种之间展开,并对不同性别、年龄的人群开展进一步分层研究,以进一步探索T1DM对甲状腺毒症的影响。
综上,本研究采用两样本MR方法,对T1DM和甲状腺毒症之间的因果关系进行探究,结果表明T1DM可能会增加甲状腺毒症的风险。
Biography
占雯婕,在读硕士研究生,E-mail: zwjzyr2021@163.com
Funding Statement
国家重点研发计划项目(2019YFC1709301);广东省名中医传承工作室项目:范冠杰广东省名中医传承工作室(粤中医办函[2021]1号)
Supported by the National Key Research and Development Program of China (2019YFC1709301)
Contributor Information
占 雯婕 (Wenjie ZHAN), Email: zwjzyr2021@163.com.
赵 玲 (Ling ZHAO), Email: nfm16800@163.com.
References
- 1.Sharma A, Stan MN. Thyrotoxicosis: diagnosis and management. Mayo Clin Proc. 2019;94(6):1048–64. doi: 10.1016/j.mayocp.2018.10.011. [DOI] [PubMed] [Google Scholar]
- 2.中华医学会内分泌学分会, 中国医师协会内分泌代谢科医师分会, 中华医学会核医学分会, et al. 中国甲状腺功能亢进症和其他原因所致甲状腺毒症诊治指南. 国际内分泌代谢杂志. 2022;38(8):700–48. [Google Scholar]
- 3.Dimeglio L, Evans-Molina C, Oram R. Type 1 diabetes. Lancet. 2013;391:2449–62. doi: 10.1016/S0140-6736(18)31320-5. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Frommer L, Kahaly GJ. Type 1 diabetes and autoimmune thyroid disease-the genetic link. Front Endocrinol. 2021;12:618213. doi: 10.3389/fendo.2021.618213. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.Sharma H, Sahlot R, Purwar N, et al. Co-existence of type 1 diabetes and other autoimmune ailments in subjects with autoimmune thyroid disorders. Diabetes Metab Syndr Clin Res Rev. 2022;16(2):102405. doi: 10.1016/j.dsx.2022.102405. [DOI] [PubMed] [Google Scholar]
- 6.Boelaert K, Newby PR, Simmonds MJ, et al. Prevalence and relative risk of other autoimmune diseases in subjects with autoimmune thyroid disease. Am J Med. 2010;123(2):183. doi: 10.1016/j.amjmed.2009.06.030. [DOI] [PubMed] [Google Scholar]
- 7.Martina S, Ferrari The association of other autoimmune diseases in patients with Graves' disease (with or without ophthalmopathy): review of the literature and report of a large series. Autoimmun Rev. 2019;18(3):287–92. doi: 10.1016/j.autrev.2018.10.001. [DOI] [PubMed] [Google Scholar]
- 8.Davey Smith G, Ebrahim S. 'Mendelian randomization': can genetic epidemiology contribute to understanding environmental determinants of disease? Int J Epidemiol. 2003;32(1):1–22. doi: 10.1093/ije/dyg070. [DOI] [PubMed] [Google Scholar]
- 9.Pierce BL, Burgess S. Efficient design for Mendelian randomization studies: subsample and 2- sample instrumental variable estimators. Am J Epidemiol. 2013;178(7):1177–84. doi: 10.1093/aje/kwt084. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Burgess S, Butterworth A, Thompson SG. Mendelian randomization analysis with multiple genetic variants using summarized data. Genet Epidemiol. 2013;37(7):658–65. doi: 10.1002/gepi.21758. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.姜 国良, 王 萍. 甲状腺功能亢进与糖代谢紊乱的相关性及对胰岛素敏感性的影响. 疑难病杂志. 2015;14(8):818–20. [Google Scholar]
- 12.Hemani G, Zheng J, Elsworth B, et al. The MR-Base platform supports systematic causal inference across the human phenome. eLife. 2018;7:34408. doi: 10.7554/eLife.34408. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Chen X, Kong JQ, Diao XY, et al. Depression and prostate cancer risk: a Mendelian randomization study. Cancer Med. 2020;9(23):9160–7. doi: 10.1002/cam4.3493. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Hartwig FP, Davies NM, Hemani G, et al. Two-sample Mendelian randomization: avoiding the downsides of a powerful, widely applicable but potentially fallible technique. Int J Epidemiol. 2016;45(6):1717–26. doi: 10.1093/ije/dyx028. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Burgess S, Bowden J, Fall T, et al. Sensitivity analyses for robust causal inference from Mendelian randomization analyses with multiple genetic variants. Epidemiology. 2017;28(1):30–42. doi: 10.1097/EDE.0000000000000559. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Slob EA, Groenen PJ, Thurik AR, et al. A note on the use of Egger regression in Mendelian randomization studies. Int J Epidemiol. 2017;46(6):2094–7. doi: 10.1093/ije/dyx191. [DOI] [PubMed] [Google Scholar]
- 17.Hartwig FP, Davey Smith G, Bowden J. Robust inference in summary data Mendelian randomization via the zero modal pleiotropy assumption. Int J Epidemiol. 2017;46(6):1985–98. doi: 10.1093/ije/dyx102. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Bowden J, Davey Smith G, Haycock PC, et al. Consistent estimation in Mendelian randomization with some invalid instruments using a weightedMedian estimator. GenetEpidemiol. 2016;40(4):304–14. doi: 10.1002/gepi.21965. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Greco M FD, Minelli C, Sheehan NA, et al. Detecting pleiotropy in Mendelian randomisation studies with summary data and a continuous outcome. Stat Med. 2015;34(21):2926–40. doi: 10.1002/sim.6522. [DOI] [PubMed] [Google Scholar]
- 20.Bowden J, Del Greco M F, Minelli C, et al. A framework for the investigation of pleiotropy in two-sample summary data Mendelian randomization. Stat Med. 2017;36(11):1783–802. doi: 10.1002/sim.7221. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.Bowden J, Davey Smith G, Burgess S. Mendelian randomization with invalid instruments: effect estimation and bias detection through Egger regression. Int J Epidemiol. 2015;44(2):512–25. doi: 10.1093/ije/dyv080. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22.Nazarzadeh M, Pinho-Gomes AC, Bidel Z, et al. Plasma lipids and risk of aortic valve stenosis: a Mendelian randomization study. Eur Heart J. 2020;41(40):3913–20. doi: 10.1093/eurheartj/ehaa070. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.Gronau QF, Wagenmakers EJ. Limitations of Bayesian leave- oneout cross-validation for model selection. Comput Brain Behav. 2019;2(1):1–11. doi: 10.1007/s42113-018-0011-7. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.黎 文鸿, 武 丽, 李 紫薇, et al. 弓形虫感染与精神分裂症的两样本孟德尔随机化研究. 现代预防医学. 2022;49(7):1153–8. [Google Scholar]
- 25.Burgess S, Thompson SG. Bias in causal estimates from Mendelian randomization studies with weak instruments. Stat Med. 2011;30(11):1312–23. doi: 10.1002/sim.4197. [DOI] [PubMed] [Google Scholar]
- 26.Lu MC, Chang SC, Huang KY, et al. Higher risk of thyroid disorders in young patients with type 1 diabetes: a 12-year nationwide, population-based, retrospective cohort study. PLoS One. 2016;11(3):e0152168.. doi: 10.1371/journal.pone.0152168. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27.Wang LT, Huang CY, Lin CH, et al. Graves disease is more prevalent than Hashimoto disease in children and adolescents with type 1 diabetes. Front Endocrinol (Lausanne) 2022;13:1083690. doi: 10.3389/fendo.2022.1083690. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 28.McDermott MT. Hyperthyroidism. Ann Intern Med. 2020;172(7):ITC49. doi: 10.7326/AITC202004070. [DOI] [PubMed] [Google Scholar]
- 29.Riquetto ADC, de Noronha RM, Matsuo EM, et al. Thyroid function and autoimmunity in children and adolescents with Type 1 Diabetes Mellitus. Diabetes Res Clin Pract. 2015;110(1):e9–11. doi: 10.1016/j.diabres.2015.07.003. [DOI] [PubMed] [Google Scholar]


