Abstract
目的
探讨脱醇红酒对肝细胞癌(HCC)发生发展的抑制作用,并初步探索其可能的机制。
方法
不同浓度的脱醇红酒(0、5、10、25、50和100 μL/mL)处理人肝癌细胞(Huh7,HepG2和SK-Hep-1)后,采用CCK-8和克隆形成实验分别检测各组细胞的增值活性和克隆形成能力。构建裸鼠皮下瘤模型,分为实验组(脱醇红酒,300 μL/d)和对照组,干预4周后对比两组肿瘤体积。构建化学诱导小鼠肝癌模型,分为实验组(脱醇红酒,300 μL/d)和对照组,干预6周后对比两组肝脏肿瘤数量、最大肿瘤直径和肝体比。脱醇红酒(75 μL/mL)处理48 h后,利用RNA-seq对Huh7细胞转录组测序,基因集富集分析(GSEA)观察基因集及通路的变化;流式细胞术检测脱醇红酒(75 μL/mL)干预后Huh7细胞周期和凋亡变化。
结果
在体外,脱醇红酒呈浓度和时间依赖性地抑制肝癌细胞的增殖活性,呈浓度依赖性地抑制其克隆形成能力。在体内,相比于对照组,脱醇红酒显著抑制实验组裸鼠皮下瘤的体积(P < 0.05),也能显著抑制实验组化学诱导肝癌小鼠的肝脏肿瘤数量(P < 0.001)、最大肿瘤直径(P < 0.05)和肝体比(P < 0.01)。RNA-seq结果显示,脱醇红酒干预后Huh7细胞内有634个基因上调和478个基因下调(|log2FC|≥2,Q≤0.05)。基因集富集分析发现,脱醇红酒能够诱导细胞周期通路相关基因集显著下调(包括E2F Targets、G2M Checkpoint、MYC Targets等)和细胞凋亡通路相关基因集上调;流式细胞检测结果表明,脱醇红酒的干预使Huh7细胞G1期阻滞,同时诱导细胞凋亡。
结论
脱醇红酒对HCC的发生发展具有抑制作用,其机制可能与细胞周期和凋亡相关通路介导的肝癌细胞G1期阻滞和细胞凋亡有关。
Keywords: 脱醇红酒, 肝细胞癌, 多酚, RNA-seq, 细胞周期, 细胞凋亡
Abstract
Objective
To investigate the inhibitory effect of dealcoholized red wine (DRW) on occurrence and progression of hepatocellular carcinoma (HCC) and explore its possible mechanisms.
Methods
Three HCC cell lines (Huh7, HepG2 and SKHep-1) treated with 5, 10, 25, 50 and 100 μL/mL DRW were examined for changes in proliferation and colony formation ability using CCK-8 assay and colony formation assay. A nude mouse model bearing subcutaneous HCC xenograft was used to test the effect of 300 μL/day DRW for 4 weeks on tumor growth. The inhibitory effect of 300 μL/day DRW for 6 weeks on tumor growth was also observed in a mouse model of chemically induced HCC by examining the tumor number, largest tumor diameter and the liver/body ratio. RNA-seq technique was used for transcriptome sequencing of Huh7 cells treated with DRW (75 μL/mL) for 48 h, and gene-set enrichment analysis (GSEA) was performed to identify the changes in genes and pathways. Flow cytometry assay was used to analyze the changes in cell cycle and apoptosis of the cells.
Results
DRW inhibited the proliferation of the HCC cell lines in a concentration-and time-dependent manner, and concentration-dependently inhibited colony formation of the cells. Treatment with DRW significantly reduced the volume of subcutaneous tumor xenograft in the tumor-bearing nude mice (P < 0.05), and lowered the number of tumors (P < 0.001), the largest tumor diameter (P < 0.05) and the liver/body ratio (P < 0.01) in mice with chemically induced HCC. RNA-seq showed that 634 genes were significantly up-regulated and 478 were down-regulated in Huh7 cells after treatment with DRW. Gene-set enrichment analysis revealed that DRW significantly down-regulated cell cycle-related pathways (E2F Targets, G2M Checkpoint and MYC Targets) and up-regulated apoptosis pathways. Flow cytometry assay showed that DRW induced cell cycle arrest in G1 phase and apoptosis of Huh7 cells.
Conclusion
DRW inhibits the occurrence and progression of HCC, and this effect is mediated possibly by inducing cell cycle arrest and apoptosis.
Keywords: dealcoholized red wine, hepatocellular carcinoma, polyphenol, RNA-seq, cell cycle, apoptosis
肝细胞癌(HCC)是最常见的恶性肿瘤之一,是全球癌症相关死亡的第3大原因,其发病率在全球范围内仍呈上升趋势,严重威胁着人类健康[1]。HCC是肝癌中最常见的类型,占所有病例的90%以上。尽管近几十年来,HCC的治疗已取得很大进展,患者总生存期和生活质量有所提高,但在大部分国家5年生存率仍不足30%[2, 3]。因此,需要进一步探索HCC的有效预防和治疗手段。
红葡萄酒是一种由葡萄酿制发酵而成的酒精饮料,含有大量活性化合物,同时含有13%左右被认定为一级致癌物质的酒精[4]。有证据显示,在2020年全球所有新发癌症病例中,有3.5%与过度饮酒和酗酒有关[5],但是有流行病学研究表明,适度饮用红酒与肺癌[6]、前列腺癌[7]、结直肠癌[8]、非霍奇金淋巴瘤[9]等癌症的风险降低有关,这可能与红酒中含有的大量多酚类物质的抑癌作用有关[10]。例如,红酒中提取的白藜芦醇[11]、类黄酮[12]、花青素[13]、没食子酸[14]等均已被证实可通过相应机制发挥对HCC的抑制作用。然而,有研究表明,由于不同多酚之间存在协同作用,相互提高生物利用度和生物效应,多酚混合物的抑癌效果优于单种多酚[15-17]。脱醇红酒作为一种含有多种多酚但不含酒精的饮料,可能具有抑制HCC发生发展的作用,但是尚无研究报道证实。本研究的目的就是探索脱醇红酒对HCC发生发展的影响,并通过RNA-seq探索其可能的机制,为HCC的预防和治疗提供新策略。
1. 材料和方法
1.1. 实验材料
1.1.1. 细胞和动物
所有细胞株包括Huh7,HepG2和SK-Hep-1购自中国科学院细胞库。所有实验动物包括:①BALB/c-Nu小鼠,4周龄,雄性,体质量15 ±2 g,购自湖南斯莱克景达实验动物有限公司(动物许可证号:SCXK(湘)2019-0004);②C57BL/6J小鼠,2周龄,雄性,体质量8±2 g,购自广东斯嘉景达生物科技有限公司(动物许可证号:SCXK(粤)2020-0052)。所有动物实验均由南方医院实验动物伦理委员会审查通过,并遵从动物实验3R原则。
1.1.2. 主要试剂耗材
DMEM、PBS、胰蛋白酶(Gibco);胎牛血清(ThermoFisher);Cell Cycle Staining Kit、Annexin V-FITC/PI kit(Multi Sciences);CCK-8试剂盒(MCE);二已基亚硝胺、四氯化碳(Sigma);橄榄油(阿拉丁);细胞培养皿、枪头、注射器等(NEST)。
1.1.3. 主要仪器
二氧化碳细胞培养箱(Labotect);光学显微镜(Leica);多功能酶标仪(BioTek);生物安全柜、水平台式离心机、-80 ℃超低温冰箱(ThermoFisher);真空冷冻干燥机(LaboGene);InfluxTM流式细胞分选仪(BD Biosciences)。
1.2. 实验方法
1.2.1. 脱醇红酒制备
红葡萄酒产自法国波尔多拉菲罗斯柴德酒庄(传奇波尔多红,Vol 13%)。制备方法:第1阶段:将红酒分装至玻璃容器,密封后放入-80 ℃冰箱过夜使物料冻结;第2阶段:运行真空冷冻干燥机并预冷,待冷阱温度降至-110 ℃时进行下一步;第3阶段:将已冻结红酒迅速转移至冷阱中防止其溶化,放入后旋紧气孔,打开真空泵持续抽真空,在真空状态(< 100 mTorr)下冷冻干燥12 h。脱醇脱水结束后密封冷冻保存。实验时将已脱水脱醇红酒使用含10%血清的DEME溶液溶解至原体积使用,试剂盒检测脱醇后酒精含量 < 0.5%。
1.2.2. 细胞培养
所有细胞系均使用含有10%胎牛血清和1%青霉素/链霉素的DMEM培养基,在37 ℃和5% CO2环境的细胞培养箱中进行培养。
1.2.3. CCK-8增殖实验
取对数生长期细胞,以2000/孔的细胞密度铺板至96孔板,每孔100 μL培养基。孵箱中培养8 h至细胞贴壁后,分为对照组(Control)和实验组(DRW),每组5个重复孔。使用不同浓度脱醇红酒溶液(0、5、10、25、50和100 μL/mL)培养48 h;使用含50 μL/mL脱醇红酒溶液的培养48和96 h。处理结束后,弃掉旧培养基,加入10% CCK-8溶液各100 μL,孵育2 h后使用酶标仪检测450 nm处吸光值(A值)。细胞增殖抑制率(IR%)=(A对照组-A实验组)/A对照组×100%。
1.2.4. 克隆形成实验
取对数生长期细胞,以400个细胞/孔的密度铺板至12孔板培养,待贴壁后加入不同浓度脱醇红酒(0、10、25和50 μL/mL)培养10 d左右。有适当数量克隆形成时,弃掉旧培养基并用PBS洗涤后,加入4%多聚甲醛固定20 min,再次PBS洗涤,随后加入0.4%结晶紫染液染色20 min。染色完成后PBS洗净染液并晾干,使用相机拍照。使用ImageJ软件对克隆形成数量进行统计及分析。
1.2.5. 皮下异种移植肿瘤模型
选择4周龄雄性BALB/c-nu裸鼠18只,饲养于SPF动物房适应环境2 d。将SK-Hep-1细胞以5×106/只的量接种至裸鼠右腹股沟处,接种时于皮下推注100 μL细胞悬液,随机分为对照组(Control)和实验组(DRW),n=9只。实验组每天灌胃给予脱醇红酒300 μL,对照组给予等量生理盐水,连续给药28 d,每周测量肿瘤长径和短径。肿瘤体积=1/2×长径×短径2。
1.2.6. 化学诱导肝癌模型
选择12 d龄雄性C57BL/6J乳鼠,饲养于SPF动物房适应环境2 d。第14 d时腹腔注射二乙基亚硝胺(DEN,25 mg/kg)1次,第4周开始腹腔注射四氯化碳(CCL4,0.5 mL/kg,溶于橄榄油)每周1次至试验结束。第12周时将小鼠随机分为对照组(n= 25)和实验组(DRW,n=12)并开始干预。实验组每天灌胃给予脱醇红酒300 μL,对照组给与等量生理盐水。干预至第18周时,麻醉后采用颈椎脱臼法将小鼠处死,剖腹取出肝脏,测量并记录肝脏肿瘤数量、最大肿瘤直径和肝脏质量。肝体比=肝脏质量/体质量×100%。
1.2.7. RNA-seq
取对数生长期的Huh7细胞接种至6孔板,培养至细胞贴壁后,实验组使用60 μL/mL的脱醇红酒溶液处理,对照组不干预,培养48 h。使用Trizol试剂裂解细胞,以提取总RNA。委托深圳华大基因股份有限公司使用BGISEQ平台进行测序服务,使用DESeq2对实验组和对照组进行差异基因表达分析。相关差异基因列表已公开至“国家生物信息中心国家生物信息中心OMIX多元数据归档库”(ID:OMIX004295,网址: https://ngdc.cncb.ac.cn/omix/)。
1.2.8. 基因集富集分析(GSEA)
参照分子标签数据库(MsigDB)中的HALLMARK类别的基因集,对经过筛选的差异基因(|log2FC|≥2,Q≤0.05)采用GSEA进行基因集富集分析。将与对照组比较出现异常的基因集信号通路按照标准化富集分数(NES)值降序排列,结果中ES为富集分数,NES为标准化后的富集分数,NOM p-val为名义P值,是对ES的统计学分析,FDR p-val为错误发现率。当NES绝对值>1,NOM p-val < 0.05且FDR q-val < 0.25时,其通路下的基因集被认为有意义。
1.2.9. 流式细胞术
选择对数生长期细胞接种至6孔板中,使用不同浓度脱醇红酒溶液(0、75和150 μL/mL)培养48 h。分别使用Cell Cycle Staining试剂盒和Annexin V-FITC/PI试剂盒处理并收集所得的细胞,操作均按照标准化步骤进行,然后通过流式细胞仪进行检测,细胞周期分布和细胞凋亡情况使用FlowJo 7.6软件分析。
1.3. 统计学分析
采用GraphPad Prism 8.0软件进行数据统计分析并绘制图表,采用R 3.6.0进行差异基因筛选,采用GSEA4.3.2软件进行基因集富集分析。计量数据采用均数±标准差表示,每项实验至少重复3次。两组间比较采用独立样本t检验,P < 0.05认为差异具有统计学意义。
2. 结果
2.1. 脱醇红酒抑制肝癌细胞的增殖活性
脱醇红酒以剂量和时间依赖的方式抑制Huh7和HepG2的增殖(图 1)。脱醇红酒对两种细胞的抑制具有剂量效应,随着剂量升高抑制率逐渐增大,对Huh7和HepG2的IC50分别为55.81和23.85 μL/mL。在固定浓度(50 μL/mL)下,脱醇红酒对两种细胞的抑制具有时间效应,时间越久,细胞活力越弱。
图 1.

DRW对肝癌细胞的增殖抑制作用
Inhibitory effect on dealcoholized red wine (DRW) on proliferation of HCC cells. A: Inhibition rate and A valve of Huh7 cells. B: Inhibition rate and OD valve of HepG2 cells. *P < 0.05, **P < 0.01, ***P < 0.001 vs DRW group.
2.2. 脱醇红酒抑制肝癌细胞的克隆形成能力
脱醇红酒以剂量依赖的方式抑制Huh7和SKHep-1细胞的克隆形成能力,剂量越高克隆形成数量越少(图 2)。Huh7细胞中,实验组(10、25和50 μL/mL)克隆形成数量相较于对照组均减少,其中25和50 μL/mL组显著减少(P < 0.001,图 2B);SK-Hep-1细胞中,实验组(10、25和50 μL/mL)克隆形成数量相较于对照组均显著减少(P < 0.001,图 2C)。
图 2.

DRW对肝癌细胞克隆形成数量的影响
Effect of dealcoholized red wine (DRW) on clone formation of HCC cells. A: Gross observation of the colonies. B: Colony number of Huh7 cells. C: Colony number of Sk-Hep-1 cells. ***P < 0.001 vs Control group.
2.3. 脱醇红酒抑制裸鼠异种移植肿瘤的生长
脱醇红酒的干预能够抑制裸鼠皮下荷瘤的生长,脱醇红酒组的肿瘤生长速度显著慢于对照组(图 3)。第4周时两组肿瘤大小(图 3A),结果显示实验组肿瘤的体积(646.2 ± 206.5 mm3)显著小于对照组(360.7 ± 164.3 mm3),差异有统计学意义(P < 0.05,图 3B)。
图 3.

DRW抑制裸鼠皮下荷瘤的生长
Dealcoholized red wine (DRW) inhibits subcutaneous HCC xenograft growth in nude mice. A: General observation of the tumors. B: Tumor growth curve of DRW and Control groups. *P < 0.05 vs DRW group.
2.4. 脱醇红酒抑制小鼠化学诱导原发性肝癌的发生发展
本实验采用DEN联合CCl4诱导小鼠肝癌的发生,造模示意图及两组代表性肝脏肿瘤生长情况(图 4A、B)。该模型结合了DEN诱导的DNA损伤和突变,及CCl4介导的炎症和纤维化,最终导致小鼠肝脏肿瘤发生,与人类HCC的微环境具有一些共同特征[18]。结果显示,脱醇红酒显著抑制小鼠肝脏肿瘤发生和发展,表现为实验组肝脏肿瘤数目(10.8±3.6)显著少于对照组(18.2±5.9,P < 0.001,图 4C),最大肿瘤直径(4.3± 1.00 mm)显著小于对照组(5.1±0.9 mm,P < 0.05,图 4D),肝体比(4.9±0.38)也显著低于Control组(5.45±0.48,P < 0.01,图 4E)。
图 4.

DRW抑制化学诱导小鼠肝癌的发生发展
DRW inhibits occurrence and progression of chemically induced HCC in mice. A: Experimental design of DEN/CCL4-induced mouse model. B: General view of HCC in the two groups. C-E: Tumor number, largest tumor diameter and liver/body ratio in the control group (n=25) and DRW group (n=12). *P < 0.05, **P < 0.01, ***P < 0.001.
2.5. 脱醇红酒对Huh7细胞转录组及通路的影响
对Huh7细胞进行RNA-seq共得到1112个显著差异基因(|log2FC|≥2,Q≤0.05),其中实验组上调478个,下调634个。基因集富集分析结果显示,脱醇红酒干预使细胞周期相关通路(包括E2F TARGETS、G2M CHECKPOINT和MYC TARGETS等基因集)显著下调(表 1),细胞凋亡通路(Apoptosis)显著上调(表 2)。
表 1.
DRW组下调信号通路
Down-regulated signaling pathways in DRW group
| Gene | No. | ES | NES | NOM p-val | FDR q-val |
| ES: Enrichment Score; NES: Normalized Enrichment Score; NOM p-val: Nominal P-value; FDR p-val: False Discovery Rate P-value. | |||||
| HALLMARK_E2F_TARGETS | 200 | -0.72 | -2.95 | < 0.001 | < 0.001 |
| HALLMARK_G2M_CHECKPOINT | 199 | -0.66 | -2.66 | < 0.001 | < 0.001 |
| HALLMARK_MITOTIC_SPINDLE | 198 | -0.49 | -2.03 | < 0.001 | < 0.001 |
| HALLMARK_MYC_TARGETS_V1 | 198 | -0.44 | -1.79 | < 0.001 | 0.001 |
| HALLMARK_MYC_TARGETS_V2 | 57 | -0.44 | -1.51 | 0.014 | 0.017 |
| HALLMARK_SPERMATOGENESIS | 110 | -0.37 | -1.38 | 0.024 | 0.057 |
表 2.
DRW组上调信号通路
Up-regulated signaling pathways in DRW group
| Gene | No. | ES | NES | NOM P-val | FDR q-val |
| ES: Enrichment Score; NES: Normalized Enrichment Score; NOM p-val: Nominal P-value; FDR p-val: False Discovery Rate P-value. | |||||
| HALLMARK_TNFA_SIGNALING_VIA_NFKB | 186 | 0.75 | 3.35 | < 0.001 | < 0.001 |
| HALLMARK_HYPOXIA | 189 | 0.62 | 2.78 | < 0.001 | < 0.001 |
| HALLMARK_EPITHELIAL_MESENCHYMAL_TRANSITION | 171 | 0.59 | 2.68 | < 0.001 | < 0.001 |
| HALLMARK_INFLAMMATORY_RESPONSE | 168 | 0.6 | 2.66 | < 0.001 | < 0.001 |
| HALLMARK_IL6_JAK_STAT3_SIGNALING | 78 | 0.6 | 2.4 | < 0.001 | < 0.001 |
| HALLMARK_APOPTOSIS | 153 | 0.53 | 2.3 | < 0.001 | < 0.001 |
| HALLMARK_IL2_STAT5_SIGNALING | 180 | 0.48 | 2.17 | < 0.001 | < 0.001 |
| HALLMARK_ANGIOGENESIS | 31 | 0.62 | 2.09 | < 0.001 | < 0.001 |
| HALLMARK_INTERFERON_ALPHA_RESPONSE | 93 | 0.51 | 2.08 | < 0.001 | < 0.001 |
| HALLMARK_ALLOGRAFT_REJECTION | 164 | 0.46 | 2.05 | < 0.001 | < 0.001 |
| HALLMARK_ESTROGEN_RESPONSE_EARLY | 184 | 0.46 | 2.05 | < 0.001 | < 0.001 |
| HALLMARK_MYOGENESIS | 164 | 0.47 | 2.05 | < 0.001 | < 0.001 |
| HALLMARK_P53_PATHWAY | 191 | 0.44 | 1.97 | < 0.001 | < 0.001 |
| HALLMARK_INTERFERON_GAMMA_RESPONSE | 178 | 0.43 | 1.95 | < 0.001 | < 0.001 |
| HALLMARK_UV_RESPONSE_UP | 152 | 0.43 | 1.91 | < 0.001 | 0.001 |
| HALLMARK_REACTIVE_OXYGEN_SPECIES_PATHWAY | 49 | 0.49 | 1.8 | < 0.001 | 0.001 |
| HALLMARK_KRAS_SIGNALING_UP | 175 | 0.4 | 1.78 | < 0.001 | 0.001 |
| HALLMARK_GLYCOLYSIS | 193 | 0.38 | 1.73 | < 0.001 | 0.003 |
| HALLMARK_COMPLEMENT | 181 | 0.38 | 1.7 | < 0.001 | 0.003 |
| HALLMARK_TGF_BETA_SIGNALING | 51 | 0.46 | 1.7 | 0.011 | 0.003 |
| HALLMARK_HEDGEHOG_SIGNALING | 35 | 0.49 | 1.64 | 0.01 | 0.004 |
2.6. 脱醇红酒诱导Huh7细胞周期G1期阻滞和细胞凋亡
流式细胞术结果显示,脱醇红酒能够诱导Huh7细胞的G1期阻滞,75 μL/mL和150 μL/mL组的G1期细胞占比([60.5±0.63)%和(63.9±0.58)%]相比于对照组([45.5±0.62)%]显著增加(P < 0.001,图 5C);同时脱醇红酒也能够诱导细胞凋亡,75 μL/mL和150 μL/mL组凋亡率([46.5±1.4)%和(50.6±2.5)%]相比于对照组([2.7± 0.1)%]显著增加(P < 0.001,图 5D)。
图 5.
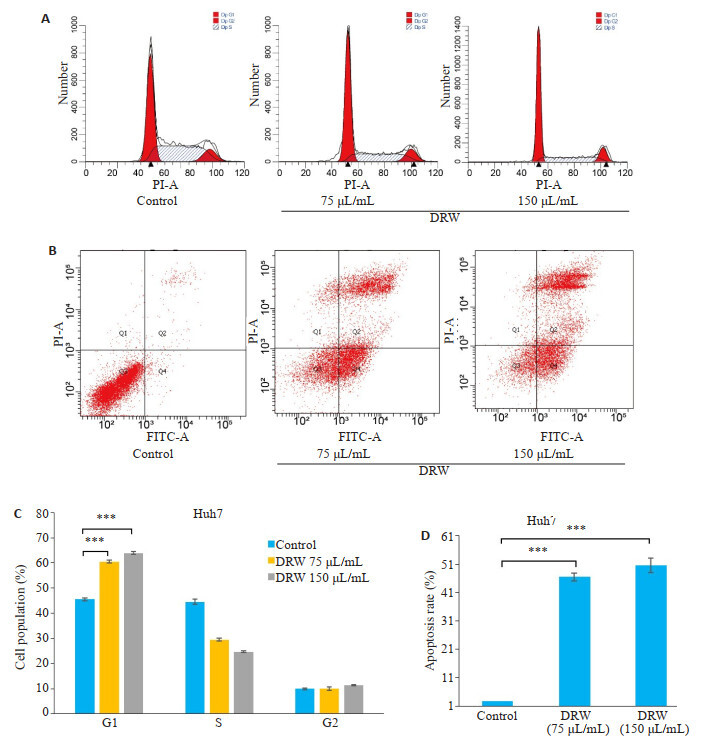
DRW诱导Huh7细胞周期G1期阻滞和细胞凋亡比例增加
DRW induces G1 phase arrest and apoptosis of Huh7 cells. A: Cell cycle distribution in DRW (75 and 150 μL/mL) and control groups. B: Flow cytometric analysis of cell apoptosis in DRW (75 and 150 μL/mL) and control groups. C: Percentage of cell population in G1, S and G2 phases in different groups. D: Cell apoptosis rates in different groups. ***P < 0.001.
3. 讨论
HCC是一个全球性的严重健康问题,迫切需要我们探索新的预防和治疗手段[19]。细胞周期和凋亡失调是所有肿瘤发生发展的关键机制之一,也被认为是HCC预防和治疗的潜在靶点[20, 21]。我们的研究发现,脱醇红酒能够抑制HCC的发生发展,并初步探明其机制涉及脱醇红酒诱导的肝癌细胞周期G1期阻滞和细胞凋亡,这显示了脱醇红酒应用于肝癌预防和治疗的潜在价值。
脱醇红酒的抗癌特性一直受到人们关注,并且在几个靶器官中已被报道,如结肠癌、皮肤癌、神经纤维瘤和骨肉瘤[22-25],我们的研究结果首先发现了脱醇红酒能够抑制HCC的发生发展,进一步扩展了脱醇红酒的应用范围。细胞增殖失控是癌症早期发生和后期进展中的核心要素,被认为是肝癌发病机制的重要基础[26, 27]。因此,寻找能够抑制肝癌细胞增殖的药物是筛选抗肿瘤药物的关键步骤。我们的结果发现,脱醇红酒能够以剂量和浓度依赖的方式抑制多种肝癌细胞的增殖和克隆形成能力,这说明脱醇红酒具备抗HCC的潜力。随后,我们利用裸鼠皮下瘤和化学诱导肝癌小鼠模型对脱醇红酒的抑癌作用做了进一步验证。结果显示,脱醇红酒干预后,裸鼠肿瘤体积显著减小,化学诱导小鼠肝癌的数量和大小也显著低于对照组。这些结果均表明,脱醇红酒具有很好的抑制HCC发生发展的作用。脱醇红酒的抑癌作用有赖于其含有的多酚,既往已有大量研究显示红酒多酚具有抗癌活性[28-30],如白藜芦醇、类黄酮、儿茶素和花青素均已被证实具有抗肝癌作用[12, 31-33]。并且,研究显示多酚相互之间能够通过抑制葡萄糖醛酸化和磷酸化而提高生物利用度,具有协同抗癌作用[34, 35]。在使用脱醇红酒的混合物时,可以更好地观察到葡萄酒多酚的保护作用[36]。脱醇红酒作为富含多酚等活性化合物的混合物,由于酒精脱除,既可避免摄入乙醇对人体健康的不良影响,又可一定程度上保持葡萄酒的风味与化合物性质的稳定,并且制备方法简单成熟[37],更具有进一步开发为抗肿瘤药物的应用价值。
为了进一步探究脱醇红酒抑制HCC发生发展的机制,我们利用RNA-seq对肝癌细胞进行了mRNA测序。GSEA分析发现,脱醇红酒的干预能够显著下调细胞周期相关通路的基因集(包括E2F Targets、G2M Checkpoint和MYC Targets),同时显著上调细胞凋亡相关通路基因(APOPTOSIS)。有研究显示,E2F靶点和G2M检查点相关基因在肝癌组织中高表达,与肝癌患者预后不良相关[38]。E2Fs是一个复杂的转录调节因子家族,主要调控G1-S期细胞周期基因的转录,E2F靶点基因的异常表达会激活致癌E2F的活性,最终导致肿瘤细胞不受控制的增殖[39]。靶向细胞周期,包括影响E2F活性的细胞周期成分,被证明是癌症治疗的关键策略[40]。G2M检查点也是抗肿瘤药物开发的领域之一,许多靶向G2M检查点的小分子抑制剂能够用于治疗癌症[41, 42]。有研究显示红酒多酚共同作用能够通过诱导细胞周期阻滞和促进凋亡的方式协同保护结肠癌[17, 43]。结合我们的结果,提示脱醇红酒可能通过细胞周期和凋亡相关通路基因的介导从而发挥抑癌作用。接下来,我们利用流式细胞术检测验证了脱醇红酒对细胞周期分布和凋亡的影响。结果显示,脱醇红酒能够阻滞细胞周期从G1期到S期的过度,并且诱导细胞凋亡,与以上结果相符。这些结果提示脱醇红酒可能通过细胞周期和凋亡相关通路基因的介导,诱导细胞周期G1期阻滞和凋亡从而抑制HCC的发生发展。需要说明的是,脱醇红酒作为多种活性化合物的混合物,其抑制HCC的机制可能是多方面、多靶点的[44]。我们的结果虽尚未阐明脱醇红酒发挥作用的具体靶点,但为后续研究提供了一定的参考依据。
综上所述,本研究结果表明脱醇红酒能够通过诱导肝癌细胞周期G1期阻滞和细胞凋亡从而抑制HCC的发生发展。这些结果为HCC预防和治疗提供了新思路和潜在手段,后续研究仍需进一步阐明其潜在机制和具体靶点以促进其临床转化。
Biography
兰玉,硕士,E-mail: johnnylan2020@163.com
Funding Statement
国家自然科学基金(U22A20274)
Supported by National Natural Science Foundation of China (U22A20274)
Contributor Information
兰 玉 (Yu LAN), Email: johnnylan2020@163.com.
孙 剑 (Jian SUN), Email: sunjian@smu.edu.cn.
References
- 1.Sung H, Ferlay J, Siegel RL, et al. Global cancer statistics 2020: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CAACancer J Clin. 2021;71(3):209–49. doi: 10.3322/caac.21660. [DOI] [PubMed] [Google Scholar]
- 2.Llovet JM, Montal R, Sia D, et al. Molecular therapies and precision medicine for hepatocellular carcinoma. Nat Rev Clin Oncol. 2018;15(10):599–616. doi: 10.1038/s41571-018-0073-4. [DOI] [PubMed] [Google Scholar]
- 3.Allemani C, Matsuda T, Dicarlo V, et al. Global surveillance of trends in cancer survival 2000-14 (CONCORD-3): analysis of individual records for 37 513 025 patients diagnosed with one of 18 cancers from 322 population-based registries in 71 countries. Lancet. 2018;391(10125):1023–75. doi: 10.1016/S0140-6736(17)33326-3. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Heras-Roger J, Díaz-Romero C, Darias-Martín J. A comprehensive study of red wine properties according to variety. Food Chem. 2016;196:1224–31. doi: 10.1016/j.foodchem.2015.10.085. [DOI] [PubMed] [Google Scholar]
- 5.Rumgay H, Shied K, Charvat H, et al. Global burden of cancer in 2020 attributable to alcohol consumption: a population-based study. Lancet Oncol. 2021;22(8):1071–80. doi: 10.1016/S1470-2045(21)00279-5. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Chao C, Slezak JM, Caan BJ, et al. Alcoholic beverage intake and risk of lung cancer: the California Men's Health Study. Cancer Epidemiol Biomarkers Prev. 2008;17(10):2692–9. doi: 10.1158/1055-9965.EPI-08-0410. [DOI] [PubMed] [Google Scholar]
- 7.Vartolomei MD, Kimura S, Ferro M, et al. The impact of moderate wine consumption on the risk of developing prostate cancer. Clin Epidemiol. 2018;10:431–44. doi: 10.2147/CLEP.S163668. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Zell JA, McEligot AJ, Ziogas A, et al. Differential effects of wine consumption on colorectal cancer outcomes based on family history of the disease. Nutr Cancer. 2007;59(1):36–45. doi: 10.1080/01635580701413926. [DOI] [PubMed] [Google Scholar]
- 9.Chiu BC, Cerhan JR, Gapstur SM, et al. Alcohol consumption and non-Hodgkin lymphoma in a cohort of older women. Br J Cancer. 1999;80(9):1476–82. doi: 10.1038/sj.bjc.6690547. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.He S, Sun CR, Pan YJ. Red wine polyphenols for cancer prevention. Int J Mol Sci. 2008;9(5):842–53. doi: 10.3390/ijms9050842. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Bishayee A, Politis T, Darvesh AS. Resveratrol in the chemoprevention and treatment of hepatocellular carcinoma. Cancer Treat Rev. 2010;36(1):43–53. doi: 10.1016/j.ctrv.2009.10.002. [DOI] [PubMed] [Google Scholar]
- 12.García ER, Gutierrez EA, Fabiana Cristina Silveira Alves de Melo, et al. Flavonoids effects on hepatocellular carcinoma in murine models: a systematic review. Evid Based Complementary Altern Med. 2018;2018:1–23. doi: 10.1155/2018/6328970. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Feng J, Wu LW, Ji J, et al. PKM2 is the target of proanthocyanidin B2 during the inhibition of hepatocellular carcinoma. J Exp Clin Cancer Res. 2019;38(1):1–15. doi: 10.1186/s13046-018-1018-6. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Shi CJ, Zheng YB, Pan FF, et al. Gallic acid suppressed tumorigenesis by an LncRNA MALAT1-wnt/β-catenin axis in hepatocellular carcinoma. Front Pharmacol. 2021;12:708967. doi: 10.3389/fphar.2021.708967. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Niedzwiecki A, Roomi MW, Kalinovsky T, et al. Anticancer efficacy of polyphenols and their combinations. Nutrients. 2016;8(9):552. doi: 10.3390/nu8090552. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Nguyen AV, Martinez M, Stamos MJ, et al. Results of a phase Ⅰ pilot clinical trial examining the effect of plant-derived resveratrol and grape powder on Wnt pathway target gene expression in colonic mucosa and colon cancer. Cancer Manag Res. 2009;1:25–37. doi: 10.2147/CMAR.S4544. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Mazué F, Delmas D, Murillo G, et al. Differential protective effects of red wine polyphenol extracts (RWEs) on colon carcinogenesis. Food Funct. 2014;5(4):663–70. doi: 10.1039/c3fo60417a. [DOI] [PubMed] [Google Scholar]
- 18.Romualdo GR, Leroy K, Costa CJS, et al. In vivo and in vitro models of hepatocellular carcinoma: current strategies for translational modeling. Cancers. 2021;13(21):5583. doi: 10.3390/cancers13215583. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Forner A, Reig M, Bruix J. Hepatocellular carcinoma. Lancet. 2018;391:1301–14. doi: 10.1016/S0140-6736(18)30010-2. [DOI] [PubMed] [Google Scholar]
- 20.Evan GI, Vousden KH. Proliferation, cell cycle and apoptosis in cancer. Nature. 2001;411(6835):342–8. doi: 10.1038/35077213. [DOI] [PubMed] [Google Scholar]
- 21.Liu S, Yang TB, Nan Y, et al. Genetic variants of cell cycle pathway genes predict disease free survival of hepatocellular carcinoma. Cancer Med. 2017;6:1512–1522. doi: 10.1002/cam4.1067. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22.Bastide NM, Naud N, Nassy G, et al. Red wine and pomegranate extracts suppress cured meat promotion of colonic mucin-depleted foci in carcinogen-induced rats. Nutr Cancer. 2017;69(2):289–98. doi: 10.1080/01635581.2017.1263745. [DOI] [PubMed] [Google Scholar]
- 23.Soleas GJ, Grass L, Josephy PD, et al. A comparison of the anticarcinogenic properties of four red wine polyphenols. Clin Biochem. 2002;35(2):119–24. doi: 10.1016/S0009-9120(02)00275-8. [DOI] [PubMed] [Google Scholar]
- 24.Clifford AJ, Ebeler SE, Ebeler JD, et al. Delayed tumor onset in transgenic mice fed an amino acid-based diet supplemented with red wine solids. Am J Clin Nutr. 1996;64(5):748–56. doi: 10.1093/ajcn/64.5.748. [DOI] [PubMed] [Google Scholar]
- 25.Tedesco I, Russo M Bilouos. Dealcoholated red wine induces autophagic and apoptotic cell death in an osteosarcoma cell line. Food Chem Toxicol. 2013;60:377–84. doi: 10.1016/j.fct.2013.07.078. [DOI] [PubMed] [Google Scholar]
- 26.Farber E. Hepatocyte proliferation in stepwise development of experimental liver cell cancer. Digest Dis Sci. 1991;36(7):973–8. doi: 10.1007/BF01297150. [DOI] [PubMed] [Google Scholar]
- 27.Sia D, Viuanueva A, Friedmansl, et al. Liver cancer cell of origin, molecular class, and effects on patient prognosis. Gastroenterology. 2017;152(4):745–61. doi: 10.1053/j.gastro.2016.11.048. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 28.Arranz S, Chiva-Blanch G, Valderas-Martínez P, et al. Wine, beer, alcohol and polyphenols on cardiovascular disease and cancer. Nutrients. 2012;4(7):759–81. doi: 10.3390/nu4070759. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.Osorio-Paz I, Brunauer R, Alavez S. Beer and its non-alcoholic compounds in health and disease. Crit Rev Food Sci Nutr. 2020;60(20):3492–505. doi: 10.1080/10408398.2019.1696278. [DOI] [PubMed] [Google Scholar]
- 30.Chao C. Associations between beer, wine, and liquor consumption and lung cancer risk: a meta-analysis. Cancer Epidemiol Biomarkers Prev. 2007;16(11):2436–47. doi: 10.1158/1055-9965.EPI-07-0386. [DOI] [PubMed] [Google Scholar]
- 31.Gao F, Deng G, Liu WB, et al. Resveratrol suppresses human hepatocellular carcinoma via targeting HGF-c-Met signaling pathway. Oncol Rep. 2017;37(2):1203–11. doi: 10.3892/or.2017.5347. [DOI] [PubMed] [Google Scholar]
- 32.Yang XW, Wang XL, Cao LQ, et al. Green tea polyphenol epigallo-catechin-3-gallate enhances 5-fluorouracil-induced cell growth inhibition of hepatocellular carcinoma cells. Hepatol Res. 2012;42(5):494–501. doi: 10.1111/j.1872-034X.2011.00947.x. [DOI] [PubMed] [Google Scholar]
- 33.Liu GJ, Shi A, Wang NN, et al. Polyphenolic Proanthocyanidin-B2 suppresses proli- feration of liver cancer cells and hepatocellular carcinogenesis through directly binding and inhibiting AKT activity. Redox Biol. 2020;37:101701. doi: 10.1016/j.redox.2020.101701. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 34.De Santi C, Pietrabissa A, Mosca F, et al. Glucuronidation of resveratrol, a natural product present in grape and wine, in the human liver. Xenobiotica. 2000;30(11):1047–54. doi: 10.1080/00498250010002487. [DOI] [PubMed] [Google Scholar]
- 35.De Santi C, Pietrabissa A, Spisni R, et al. Sulphation of resveratrol, a natural compound present in wine, and its inhibition by natural flavonoids. Xenobiotica. 2000;30(9):857–66. doi: 10.1080/004982500433282. [DOI] [PubMed] [Google Scholar]
- 36.Femia AP, Caderni G, Vignali F, et al. Effect of polyphenolic extracts fromred wine and 4-OH-coumaric acidon 1, 2-dimethylhydrazineinducedcolon carcinogenesis in rats. Eur J Nutr. 2005;44(2):79–84. doi: 10.1007/s00394-004-0496-9. [DOI] [PubMed] [Google Scholar]
- 37.徐梦梦. 脱醇过程对红酒品质和抗氧化性能的影响[D]. 杭州: 浙江工商大学.
- 38.Hu WM, Shi YM, Han TQ, et al. A panel of E2F target gene signature predicting the prognosis of hepatocellular carcinoma. Front Genet. 2022;13:879299. doi: 10.3389/fgene.2022.879299. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 39.Kent LN, Leone G. The broken cycle: E2F dysfunction in cancer. Nat Rev Cancer. 2019;19(6):326–38. doi: 10.1038/s41568-019-0143-7. [DOI] [PubMed] [Google Scholar]
- 40.Otto T, Sicinski P. Cell cycle proteins as promising targets in cancer therapy. Nat Rev Cancer. 2017;17(2):93–115. doi: 10.1038/nrc.2016.138. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 41.Barnaba N, LaRocque JR. Targeting cell cycle regulation via the G2- M checkpoint for synthetic lethality in melanoma. Cell Cycle. 2021;20(11):1041–51. doi: 10.1080/15384101.2021.1922806. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 42.Bucher N, Britten CD. G2 checkpoint abrogation and checkpoint kinase-1 targeting in the treatment of cancer. Br J Cancer. 2008;98(3):523–8. doi: 10.1038/sj.bjc.6604208. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 43.Marel AK, Lizard G, Izard JC, et al. Inhibitory effects of transresveratrol analogs molecules on the proliferation and the cell cycle progression of human colon tumoral cells. Mol Nutr Food Res. 2008;52(5):538–48. doi: 10.1002/mnfr.200700185. [DOI] [PubMed] [Google Scholar]
- 44.da Costa DC F, Rangel LP, Quarti J, et al. Bioactive compounds and metabolites from grapes and red wine in breast cancer chemoprevention and therapy. Molecules. 2020;25(15):3531. doi: 10.3390/molecules25153531. [DOI] [PMC free article] [PubMed] [Google Scholar]


