Abstract
目的
探究转化生长因子-β1(TGF-β1)诱导肾纤维化的机制。
方法
将TGF-β1处理和未处理的肾成纤维细胞NRK-49F进行转录组测序,采用DESeq2分析测序结果,以错误发现率低于0.05且log2FC绝对值大于1为标准筛选差异表达的基因,并对差异表达的基因进行基因本体(GO)、京都基因和基因组百科全书(KEGG)通路富集分析。进一步筛选差异表达基因中编码转录因子的基因,并利用单侧输尿管梗阻诱导的小鼠肾纤维化模型和高通量基因表达数据库GSE104954数据集对这些基因在肾纤维化过程中的表达进行验证。
结果
TGF-β1处理6、12和24 h后,分别有552、1209和1028个差异表达基因。GO分析表明,这些差异表达基因在发育、细胞死亡和细胞迁移过程中显著富集。KEGG分析显示,在TGF-β1诱导早期(TGF-β1处理6 h)主要表现为Hippo、TGF-β、Wnt信号通路的变化,而在TGF-β1诱导晚期(TGF-β1处理24 h)主要表现为细胞外基质受体相互作用、局灶黏附和黏附分子连接等相关通路的改变。在TGF-β1处理6 h时291个上调的差异表达基因中,Snai1、Irf8、Bhlhe40、Junb、Arid5a、Vdr、Lef1、Ahr、Foxo1、Myc、Tcf7、Foxc2、Glis1等13个基因编码转录因子。在细胞模型中验证发现,TGF-β1可以诱导其中9个编码转录因子基因(Snai1、Irf8、Bhlhe40、Junb、Arid5a、Vdr、Lef1、Myc、Tcf7)的表达,其余4个基因的表达水平在TGF-β1处理后无显著变化。在动物模型验证中发现,Snai1、Irf8、Bhlhe40、Junb、Arid5a、Myc和Tcf7显著上调,而Vdr显著下调,Lef1无显著变化。在GSE104954数据集中验证发现,IRF8在糖尿病肾病患者和IgA肾病患者的肾小管间质中显著高表达,MYC在糖尿病肾病患者中高表达,而其他7个基因的表达产物与对照组相似。
结论
TGF-β1诱导肾成纤维细胞基因差异表达,Irf8和Myc可能是慢性肾脏病和肾纤维化的潜在靶点。
Keywords: 转录组, 转化生长因子-β1, 成纤维细胞, 肾纤维化, 机制, 小鼠
Abstract
Objective
To explore the mechanism of transforming growth factor-β1 (TGF-β1) induce renal fibrosis.
Methods
Renal fibroblast NRK-49F cells treated with and without TGF-β1 were subjected to RNA-seq analysis. DESeq2 was used for analysis. Differentially expressed genes were screened with the criteria of false discovery rate<0.05 and >1. Gene Ontology (GO) and Kyoto Encyclopedia of Genes and Genomes (KEGG) pathway enrichment analyses were performed for differentially expressed genes. Genes encoding transcription factors were further screened for differential expression genes. Then, the expression of these genes during renal fibrosis was verified using unilateral ureteral obstruction (UUO)-induced mouse renal fibrosis model and a public gene expression dataset (GSE104954).
Results
After TGF-β1 treatment for 6, 12 and 24 h, 552, 1209 and 1028 differentially expressed genes were identified, respectively. GO analysis indicated that these genes were significantly enriched in development, cell death, and cell migration. KEGG pathway analysis showed that in the early stage of TGF-β1 induction (TGF-β1 treatment for 6 h), the changes in Hippo, TGF-β and Wnt signaling pathways were observed, while in the late stage of TGF-β1 induction (TGF-β1 treatment for 24 h), the changes of extracellular matrix-receptor interaction, focal adhesion and adherens junction were mainly enriched. Among the 291 up-regulated differentially expressed genes treated with TGF-β1 for 6 h, 13 genes (Snai1, Irf8, Bhlhe40, Junb, Arid5a, Vdr, Lef1, Ahr, Foxo1, Myc, Tcf7, Foxc2, Glis1) encoded transcription factors. Validation in a cell model showed that TGF-β1 induced expression of 9 transcription factors (encoded by Snai1, Irf8, Bhlhe40, Junb, Arid5a, Vdr, Lef1, Myc, Tcf7), while the expression levels of the other 4 genes did not significantly change after TGF-β1 treatment. Validation results in UUO-induced mouse renal fibrosis model showed that Snai1, Irf8, Bhlhe40, Junb, Arid5a, Myc and Tcf7 were up-regulated after UUO, Vdr was down-regulated and there was no significant change in Lef1. Validation based on the GSE104954 dataset showed that IRF8 was significantly overexpressed in the renal tubulointerstitium of patients with diabetic nephropathy or IgA nephropathy, MYC was highly expressed in diabetic nephropathy, and the expressions of the other 7 genes were not significantly different compared with the control group.
Conclusion
TGF-β1 induces differentially expressed genes in renal fibroblasts, among which Irf8 and Myc were identified as potential targets of chronic kidney disease and renal fibrosis.
Keywords: Transcriptome, Transforming growth factor-β1, Fibroblasts, Renal fibrosis, Mechanism, Mice
肾纤维化是许多进行性肾脏疾病的共同表现,其特征是正常组织结构逐渐被ECM(如胶原、蛋白聚糖和糖蛋白)所取代[1-2],直接原因是间质成纤维细胞增殖并分化为肌成纤维细胞,导致ECM过度合成[3]。Kuppe等[4]通过单细胞转录组测序发现,血小板源生长因子受体α阳性且血小板源生长因子受体β阳性的成纤维细胞/肌成纤维细胞是人肾纤维化瘢痕形成的主要来源。TGF-β1是一种多功能的细胞因子,是促进成纤维细胞分化为促纤维化表型和产生ECM的主要介质[5-6]。通过多种途径靶向TGF-β1可抑制肾纤维化,部分靶向药物已用于临床研究[7-9]。然而,TGF-β1不仅参与纤维化过程,还参与炎症反应、免疫调节等,直接阻断TGF-β1信号通路可能带来诸多副作用,限制了其作为一种抗纤维化治疗方法在临床上的应用[7-9]。因此,了解TGF-β1诱导纤维化的机制和TGF-β1信号通路下游的效应分子将有助于探索预防和抑制肾纤维化的方法。
TGF-β1诱导的正常大鼠肾成纤维细胞NRK-49F是典型的体外肾纤维化细胞模型。Zhou等[10]采用质谱法对TGF-β1处理48 h的NRK-49F细胞和对照细胞的细胞裂解物及条件培养基在蛋白质组水平进行分析。与对照组比较,在NRK-49F细胞的细胞裂解物中鉴定出628种差异表达的蛋白质,在条件培养基中鉴定出62种差异表达的蛋白质。TGF-β1调节的细胞裂解物蛋白表现为核糖体蛋白增加,参与多种代谢途径的蛋白质失调,包括醛脱氢酶3家族成员A1减少、外核苷酸焦磷酸酶/磷酸二酯酶1和肌苷单磷酸脱氢酶2增加;酶联免疫吸附试验结果显示,TGF-β1处理组胶原降解调节因子(丝氨酸蛋白酶抑制剂超家族E成员1增加、基质金属蛋白酶3减少)、胶原交联因子(前胶原-赖氨酸、2-氧葡萄糖酸5-双加氧酶2增加)、信号分子(细胞通信网络因子1、细胞通信网络因子2和Tsukushi增加,细胞通信网络因子3减少)和趋化因子(C-C基序趋化因子配体2、C-C基序趋化因子配体7增加)的分泌异常[10]。然而,由于转录和翻译的调控作用,基因在转录水平与翻译水平的表达可能存在差异。TGF-β1作用于成纤维细胞不同时间后基因的表达可能不同,TGF-β1仅处理48 h可能会丢失一些早期表达的差异基因;此外,由于基因的表达经历先转录后翻译的过程,蛋白质组比转录组提供的诊断信息晚[11]。因此,上述研究在寻找TGF-β1信号通路下游的潜在靶点上存在局限性[10]。本研究使用转录组测序技术检测了TGF-β1处理6、12和24 h的NRK-49F细胞转录组,汇总了表达上调和下调的基因、差异表达基因参与的生物过程和信号通路,并采用qRT-PCR在动物模型和数据库慢性肾脏病患者的转录组数据中进行验证,为更好地理解TGF-β1诱导的肾纤维化机制以及研发新的诊断和治疗方案提供参考。
1. 材料和方法
1.1. 细胞、动物、试剂和仪器
正常大鼠肾成纤维细胞株NRK-49F 购自中国科学院细胞库。8周龄C57B/L6小鼠(体重约20 g)购自扬州大学比较医学中心,动物在通风、室温为24 ℃的动物房中饲养,进行12 h光照、12 h黑暗的循环,所有动物均可自由获得食物和水。动物实验方案通过扬州大学医学院伦理委员会审查(YXYLL-2021-05),符合美国国立卫生研究院发布的《实验动物护理和使用指南》。
DMEM/F12培养基、胎牛血清为赛澳美细胞技术(北京)有限公司产品;青霉素/链霉素为武汉赛维尔生物科技有限公司产品;重组小鼠/大鼠TGF-β1为北京义翘神州科技股份有限公司产品;TRIzol试剂为美国Invitrogen公司产品;戊巴比妥钠为北京方程生物科技有限公司产品;HiScript Q RT SuperMix试剂盒和ChamQ SYBR qPCR Master Mix试剂盒为南京诺唯赞生物科技股份有限公司产品。PCR仪为德国Eppendorf 公司产品;ABI 7500实时荧光定量PCR仪为美国ABI公司产品。
1.2. 细胞培养及处理
NRK-49F细胞在含10%胎牛血清和青霉素/链霉素的DMEM/F12培养基中,置于37 ℃、5%二氧化碳的培养箱中培养。加入终浓度为5 ng/mL的TGF-β1处理6、12和24 h后收集细胞,同期收集未经TGF-β1处理的对照细胞。
1.3. 转录组测序文库构建及测序
转录组测序文库构建及测序由武汉爱基百客生物科技有限公司完成,主要步骤如下:使用TRIzol试剂从细胞中提取总RNA,并使用Qsep1仪器评估所有RNA样品的完整性;取3 μg总RNA,采用MGIEasy mRNA文库制备试剂盒构建RNA文库(具体包括使用Oligo-dT磁珠分离polyA富集的mRNA、mRNA片段化、片段化的mRNA逆转录合成cDNA、末端修复、加碱基A并连接测序接头);文库在Illumina测序平台(Illumina NovaSeq 6000)上使用武汉爱基百客生物科技有限公司的双末端技术进行测序。
1.4. 转录组测序结果的生物信息学分析
1.4.1. 识别差异表达基因
通过Cutadapt 1.11过滤出衔接子和低通量测序片段,然后使用Hisat 2 2.1.0将纯化的测序片段与大鼠参考基因组RN6进行比对[12-13]。将所有样本的比对信息使用Stringtie 2.0.4进行转录本重构。使用RSEM 1.2.6评估转录本的丰度,并将表达值标准化为FPKM(每千个碱基的转录本每百万映射读取的片段数)[14]。差异表达基因用DESeq2进行鉴定,FDR低于0.05且log2FC绝对值大于1的基因作为差异有统计学意义的基因。使用R语言中的ggplot2包绘制火山图展示差异表达基因的分布,使用Venn Diagram包绘制维恩图展示TGF-β1处理6、12和24 h后差异表达的交集基因数,使用pheatmap包对差异表达基因进行层次聚类并绘制热图,展示组间差异和差异基因表达的动态变化。
1.4.2. 差异表达基因的GO和KEGG通路富集分析
使用R语言中的ClusterProfiler包对差异表达基因分别进行GO和KEGG通路富集分析[15-16]。按照FDR低于0.05筛选富集结果。
1.5. 转录因子的筛选
根据美国国家生物技术信息中心在线网站(https://www.ncbi.nlm.nih.gov/)提供的基因信息,在差异表达的基因中筛选编码转录因子的基因。
1.6. 动物模型验证TGF-β1信号通路下游的转录因子
C57B/L6小鼠在造模前禁食8 h,腹腔注射戊巴比妥钠(50 mg/kg)麻醉小鼠,无菌条件下剪开小鼠皮肤及肌层至腹膜后,通过中线切口暴露并游离左侧输尿管,并用4-0丝线在肾盂下方5 mm处结扎两次,建立UUO诱导的肾纤维化小鼠。手术对照组以类似方式处理,但不结扎输尿管。造模7 d后收取小鼠肾组织,去包膜,使用TRIzol试剂提取总RNA。
1.7. 通过数据集GSE104954验证TGF-β1信号通路下游的转录因子
为了进一步证实肾纤维化的潜在靶点,从高通量基因表达数据库中筛选GSE104954数据集,并在数据集中选择健康对照(活体捐献者)、糖尿病肾病患者和IgA肾病患者数据,使用GEO2R工具筛选上述数据集中糖尿病肾病患者和IgA肾病患者肾组织标本分别相对于健康对照肾组织标本间的差异表达基因,筛选条件为FDR低于0.05和log2FC绝对值大于1。使用Prism Graph 8.0软件绘制相关转录因子的表达图谱。
1.8. qRT-PCR检测相关基因的表达
使用HiScript Q RT SuperMix试剂盒将1 µg总RNA进行逆转录,合成cDNA。使用ChamQ SYBR qPCR Master Mix试剂盒进行定量PCR检测。实验中所使用的引物见表1。PCR条件:95 ℃ 30 s,95 ℃ 10 s,60 ℃ 30 s,进行40次扩增。使用ABI 7500实时荧光定量PCR仪进行定量PCR检测,每个基因的表达量表示为β-actin的倍数,每组进行三次独立重复实验。
表1.
定量逆转录聚合酶链反应中所使用的引物
| 基 因 | 上游引物(5-3) | 下游引物(5-3) |
|---|---|---|
| Snai1 | CCACTCGGATGTGAAGAGATAC | AGACTCTTGGTGTTTGTGGAG |
| Irf8 | TGGACATTTCCGAGCCATAC | TCAGCTCCTCGATCTCTGAA |
| Bhlhe40 | CTCCTACCCGAACATCTCAAAC | TCTGCTGCTGCTGATCAATTA |
| Junb | CCTTTCTATCACGACGACTCATAC | CTAAGGTGGGTTTCAGGAGTTT |
| Arid5a | GTTGGGACCACCTGGTAAAG | GCTTTGGGCTTGGGATAGAA |
| Vdr | CTGAACGGAGAGGATTCTGATG | GGATGCTGTAACTGACAAGGT |
| Lef1 | GAGAAAGGAACAGGAGCCTAAA | CTACGACATTCGCCCTCATT |
| Ahr | CAGTGTAGAGCACAAGTCAGAG | GGATAGTGGAAGACGCATAGAAG |
| FoxO1 | CGCCAAACACCAGTCTAAATTC | CTGTCCTGAAGTGTCTGCATAG |
| Myc | AGCTTCGCTAACAGGAACTATG | CTGCTGTTGCTGGTGATAGA |
| Tcf7 | CTCGGACAAACTTCCAGAGTC | GAGCAGATTGAAGGCAGAGTAG |
| FoxC2 | GGACGTGCCCAAGGATAAG | GCTCTTGACCACCACTTTCT |
| Glis1 | GTCTGTGAGAGACTGCTTCATT | GGTAGGGCTTCTTGCTTGAT |
Snai:蜗牛家族转录抑制因子;Irf:干扰素调节因子;Bhlhe40:基本螺旋-环-螺旋家族成员e40;Arid5a:富含AT的交互式含域蛋白5A;Vdr:维生素D受体;Lef:淋巴增强结合因子;Ahr:芳香烃受体;FoxO1:叉头框-O1;Tcf:转录因子;FoxC2:叉头框-C2;Glis:GLIS家族锌指蛋白.
1.9. 统计学方法
采用Prism Graph 8.0软件进行统计分析。计量数据以均数±标准差()表示,两组样本之间比较采用双侧Student’s t检验,P<0.05为差异有统计学意义。
2. 结果
2.1. TGF-β1诱导的体外纤维化模型构建成功
qRT-PCR结果显示,纤维化相关基因Acta2、Col1a1和Col3a1的表达在TGF-β1处理后显著上调(图1),提示TGF-β1诱导的体外纤维化细胞模型构建成功。
图1. TGF-β1处理组与对照组肾纤维化相关基因的mRNA表达.
与对照组比较,*P<0.05,**P<0.01. TGF:转化生长因子;ACTA:α-平滑肌肌动蛋白;COL1A1:Ⅰ型胶原α1;COL3A1:Ⅲ型胶原α1.
2.2. TGF-β1诱导的差异表达基因
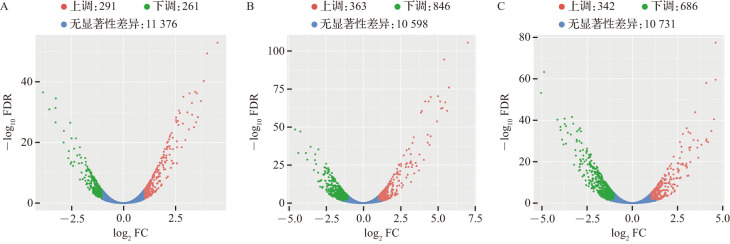
高通量转录组测序结果显示,与对照组比较,TGF-β1处理6 h时有552个基因差异表达,其中291个基因表达上调,261个基因表达下调;TGF-β1处理12 h时有1209个基因差异表达,其中363个基因表达上调,846个基因表达下调;TGF-β1处理24 h时有1028个基因差异表达,其中342个基因表达上调,686个基因表达下调(图2)。将三个时间点的差异表达基因取交集,共有207个基因在TGF-β1处理6、12和24 h时均差异表达,其中89个基因在三个时间点均上调,110个基因在三个时间点均下调(图3)。聚类分析显示两个主要分支,即对照组和TGF-β1处理组(图4)。以上结果表明,TGF-β1处理后成纤维细胞中的基因表达出现变化,且与TGF-β1诱导的时间相关。
图2. TGF-β1处理6、12、24 h后表达变化基因的火山图.
A:TGF-β1处理6 h后差异表达基因;B:TGF-β1处理12 h后差异表达基因;C:TGF-β1处理24 h后差异表达基因. 红色表示上调基因,绿色表示下调基因,蓝色表示使用标准log2 FC绝对值大于1无法区分上调和下调的基因. FDR:错误发现率;FC:倍数变化;TGF:转化生长因子.
图3. TGF-β1处理6、12、24 h后表达变化基因的维恩图.
TGF:转化生长因子.
图4. TGF-β1处理组与对照组基因表达分层聚类和热图.

TGF:转化生长因子.
2.3. 差异表达基因GO分析和KEGG分析结果
GO分析结果显示,前二十个富集条目集中在生物过程上。其中,TGF-β1处理6和12 h差异表达的基因主要富集在组织发育(tissue development)和小管发育(tube development),而TGF-β1处理24 h差异表达的基因主要富集在细胞迁移的调控(regulation of cell migration)。细胞死亡的调控(regulation of cell death)、细胞凋亡的调控(regulation of apoptotic process)和程序性细胞死亡的调控(regulation of programmed cell death)仅出现在TGF-β1处理6 h差异表达的基因中,而细胞对细胞因子刺激的反应(cellular response to cytokine stimulus)、对含氧化合物的反应(response to oxygen-containing compound)和对细胞因子的反应(response to cytokine)仅出现在TGF-β1处理24 h差异表达的基因,见图5。结果提示,TGF-β1作用后,成纤维细胞中与差异表达基因相关的分子过程和生物途径发生改变,且与TGF-β1诱导的时间相关。
图5. 差异表达基因的GO分析结果.

A:TGF-β1处理6 h与同一时间对照组差异表达基因富集的前20条通路;B:TGF-β1处理12 h与同一时间对照组差异表达基因的前20条通路;C:TGF-β1处理24 h与同一时间对照组差异表达基因的前20条通路. TGF:转化生长因子;GO:基因本体. *表示分子功能,其余为生物过程.
KEGG富集分析结果显示,在TGF-β1诱导早期(TGF-β1处理6 h)主要表现为Hippo、TGF-β、Wnt信号通路的变化,而在TGF-β1诱导晚期(TGF-β1处理24 h)主要表现为ECM受体相互作用、局灶黏附和黏附分子连接等相关通路的改变,见图6。结果提示,TGF-β1作用后,成纤维细胞中差异表达基因参与的信号通路发生改变,且与TGF-β1诱导的时间相关。
图6. 差异表达基因的KEGG分析结果.
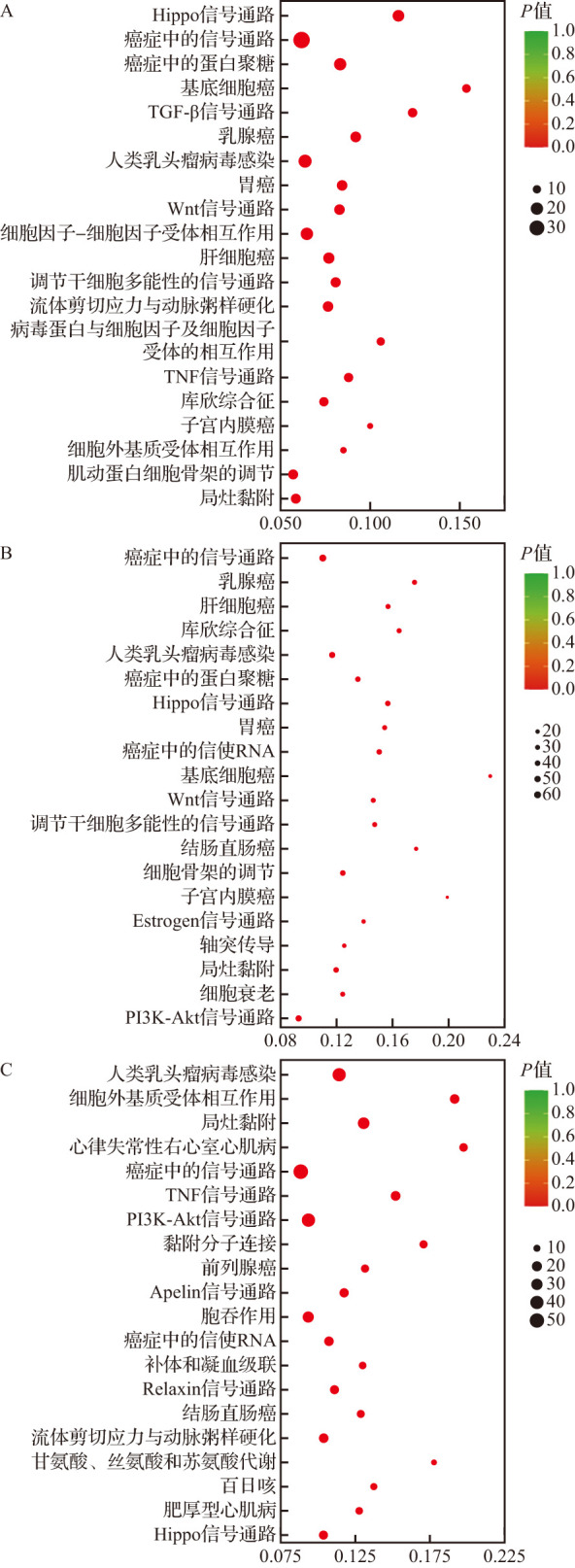
A:TGF-β1处理6 h与同一时间对照组相比上调基因富集的前20条通路;B:TGF-β1处理12 h与同一时间对照组相比上调基因富集的前20条通路;C:TGF-β1处理24 h与同一时间对照组相比上调基因富集的前20条通路. TGF:转化生长因子;TNF:肿瘤坏死因子;PI3K:磷酸肌醇3-激酶;KEGG:京都基因和基因组百科全书.
2.4. 肾间质成纤维细胞中TGF-β1信号通路下游的转录因子
对TGF-β1处理6 h时差异表达基因中编码转录因子的基因进行筛选发现,影响上述生物过程和信号通路的基因主要集中在上调的基因中。在TGF-β1处理6 h时291个上调的差异表达基因中,Snai1、Irf8、Bhlhe40、Junb、Arid5a、Vdr、Lef1、Ahr、Foxo1、Myc、Tcf7、Foxc2、Glis1这13个基因可以编码转录因子,其中部分转录因子在TGF-β1处理12、24 h时较对照组也上调(图7)。qRT-PCR验证发现,在细胞模型中,TGF-β1可以诱导其中9个编码转录因子基因(Snai1、Irf8、Bhlhe40、Junb、Arid5a、Vdr、Lef1、Myc、Tcf7)的表达(图8),其余4个基因的表达水平在TGF-β1处理后没有显著变化。在动物肾纤维化模型中,上述9个编码转录因子基因中Snai1、Irf8、Bhlhe40、Junb、Arid5a、Myc和Tcf7这7个编码转录因子基因显著上调,而Vdr显著下调,Lef1没有变化,见图9。
图7. 热图显示13个转录因子的表达.

数值表示为log2FC值. FC:倍数变化.
图8. 九种转录因子基因在细胞模型中的表达水平.

图9. 九种转录因子基因在肾纤维化动物模型中的表达水平.
A:造模后3 d;B:造模后7 d. 每组至少包括5只小鼠,每个实验至少重复三次;与对照组比较,*P<0.05,**P<0.01;Snai:蜗牛家族转录抑制因子;Irf:干扰素调节因子;Bhlhe40:基本螺旋-环-螺旋家族成员e40;Arid5a:富含AT的交互式含域蛋白5A;Vdr:维生素D受体;Lef:淋巴增强结合因子;Tcf:转录因子.
数据集GSE104954验证结果显示,IRF8在糖尿病肾病患者(1.149±0.119)和IgA肾病患者(1.077±0.098)的肾小管间质中相比健康对照(1.000±0.093)显著高表达(均P<0.01),MYC在糖尿病肾病患者(1.100±0.075)中相比健康对照(1.000±0.088)高表达(P<0.01),而其他7个基因的表达产物与对照组比较差异无统计学意义。以上结果提示,Irf8和Myc可能是肾纤维化的潜在干预靶点。
3. 讨论
肾纤维化是糖尿病、高血压、免疫复合物沉积等引发的慢性肾脏病的共同病理特征,表现为ECM合成与降解的失调[1-2]。TGF-β1等细胞因子诱导间质成纤维细胞增殖并分化为肌成纤维细胞,导致ECM过度合成,是肾纤维化发生的直接原因[3]。深入了解TGF-β1诱导纤维化的机制和TGF-β1信号通路下游的效应分子将有助于探索预防和抑制肾纤维化的方法。相比蛋白质组,转录组的变化更为提前,因此可以为诊断提供更早期的信息。本研究采用转录组测序技术检测了TGF-β1处理6、12和24 h后NRK-49F细胞和对照细胞的基因表达谱,对差异表达的基因进行了全面分析,为进一步了解TGF-β1诱导肾纤维化的机制提供了信息。
本文资料显示,TGF-β1处理6、12和24 h后,分别有552、1209和1028个差异表达基因。GO分析表明,这些差异表达基因在发育、细胞死亡和细胞迁移过程中显著富集,而Zhou等[10]的蛋白质组研究显示差异表达蛋白主要富集在翻译、氧化还原过程、对药物的反应等生物过程,进一步证明了TGF-β1处理的成纤维细胞在转录和翻译水平的差异。KEGG分析显示,在TGF-β1诱导的早期主要表现为Hippo、TGF-β、Wnt信号通路的变化,与之前的研究报道TGF-β信号通路与Hippo、Wnt信号通路相互交叉共同影响纤维化一致[17-18];而在TGF-β1诱导的晚期主要表现为ECM受体相互作用、局灶黏附和黏附分子连接等相关通路的改变,这可能是因为胶原等细胞外基质蛋白开始合成。
为了探索影响以上生物过程和信号通路的转录因子,本研究在TGF-β1处理6 h时291个上调的差异表达基因中筛选到13个编码转录因子的基因(Snai1、Irf8、Bhlhe40、Junb、Arid5a、Vdr、Lef1、Ahr、Foxo1、Myc、Tcf7、Foxc2、Glis1),GO分析显示Snai1、Junb、Arid5a、Vdr、Lef1、Ahr、Foxo1、Myc、Tcf7和Foxc2参与发育过程,Snai1、Junb、Vdr、 Lef1、Ahr、Foxo1、Myc、Tcf7和Foxc2与细胞凋亡相关,Snai1、Lef1、Myc和Foxc2参与细胞迁移;KEGG分析显示Lef1、Myc和Tcf7参与Hippo和Wnt信号通路,Myc参与TGF-β信号通路,Snai1、Lef1和Tcf7参与黏附分子连接。在细胞模型中验证发现,TGF-β1可以诱导其中9个编码转录因子基因(Snai1、Irf8、Bhlhe40、Junb、Arid5a、Vdr、Lef1、Myc、Tcf7)的表达,其余4个基因(Ahr、Foxo1、Foxc2、Glis1)的表达水平在TGF-β1处理后无显著变化。在动物模型中进一步验证发现,Snai1、Irf8、Bhlhe40、Junb、Arid5a、Myc和Tcf7显著上调,Vdr显著下调,Lef1无显著变化。在GSE104954数据集中验证发现,IRF8在糖尿病肾病患者和IgA肾病患者的肾小管间质中显著高表达,MYC在糖尿病肾病中高表达,而其他7个基因的表达产物与对照组相比无显著差异。
文献检索显示,Snai1[19-20]、Junb[21]和Lef1[22-23]在肾纤维化动物模型中显著上调,且能够诱导肾小管上皮细胞-间质细胞转化并促进肾纤维化。但在肾间质成纤维细胞中,TGF-β1是否能诱导Snai1、Junb和Lef1的表达及其功能尚不清楚。Zhang等[24]通过生物信息学预测发现Bhlhe40可能在SARS-CoV-2诱导的急性肾损伤到慢性肾脏病的转变中发挥重要作用。Martínez-Arias等[25]发现Vdr的水平与肾纤维化程度成负相关。Irf8是干扰素调节因子家族的转录因子,其特异性结合Ⅰ型Ifn和Ifn诱导的MHC Ⅰ类基因的上游调节区[26-27]。GO分析显示,Irf8参与多细胞生物过程的正向调节、对含氧复合物和细胞因子刺激的反应。KEGG信号通路分析显示,Irf8与百日咳相关。关于Irf8的研究多集中在免疫细胞,既可以作为免疫细胞的转录激活因子,也可以作为抑制因子[28-29]。Irf8是多发性硬化的易感基因,Ototake等[30]发现多发性硬化症患者单核细胞和巨噬细胞中Irf8下调可能加重纤维化表型。而Yoshida等[31]的研究显示,Irf8在抗原提呈细胞(如巨噬细胞、树突状细胞和小胶质细胞)中表达,Irf8敲除能够抑制小鼠实验性自身免疫性脑脊髓炎(一种用于研究多发性硬化症的动物模型);Irf8一方面通过增强抗原提呈细胞中αvβ8整合素的表达,激活TGF-β信号,导致Th17细胞分化,另一方面通过刺激IL-12和IL-23的产生、抑制IL-27的产生,有利于Th1细胞和Th17细胞生长和维持的细胞因子环境,从而促进疾病的发生和发展。Fu等[32]发现Irf8在肾移植后发生间质纤维化和小管萎缩的肾组织中表达增加。Guo等[33]在数据集GSE30122和GSE30529中分析了糖尿病肾病患者的差异表达基因,结果显示Irf8在糖尿病肾病组显著上调。总之,Irf8可能是肾纤维化的潜在治疗靶点,但其具体作用机制还须进一步研究。人MYC是一种原癌基因,其编码蛋白与MYC相关因子X形成异源二聚体,该异源二聚体与DNA的启动子结合,并调节特定靶基因的转录,许多人类肿瘤中会出现该基因的异常扩增[34]。因其在细胞周期进程、细胞凋亡和细胞转化中的作用,近年来越来越多的研究发现该蛋白参与纤维化进程。据报道,Myc在肾纤维化过程中显著上调,并通过直接结合成纤维细胞的整合素αv的启动子促进其表达,进而激活TGF-β1信号通路,促进纤维化的发生[35]。本文资料进一步证实TGF-β1也能反向诱导成纤维细胞中Myc的表达,TGF-β1、Myc和整合素αv在肾脏成纤维细胞中可能以正反馈的环路发挥促纤维化的作用。
综上所述,本研究对TGF-β1诱导的NRK-49F细胞肾纤维化模型的转录组测序结合细胞、动物、患者数据库层面的验证,提出Irf8和Myc可能是肾纤维化的潜在干预靶点。今后将进一步探索Irf8和Myc在肾纤维化进程中的具体作用和机制,以期为肾纤维化的临床诊治提供理论依据。
Acknowledgments
研究得到国家自然科学基金(32100911)、江苏省高等学校自然科学研究项目(20KJB310002)、2019年江苏省双创博士项目、2020年扬州市“绿扬金凤计划”支持
Acknowledgments
This work was supported by the National Natural Science Foundation of China (32100911), Natural Science Research Project for Universities of Jiangsu Province (20KJB310002), 2019 Double-Innovation Doctor Program of Jiangsu Province, 2020 Lyuyang Jinfeng Project of Yangzhou City.
[缩略语]
细胞外基质(extracellular matrix,ECM);转化生长因子(transforming growth factor,TGF);聚合酶链反应(polymerase chain reaction,PCR);定量逆转录PCR(quantitative reverse transcription PCR,qRT-PCR);信使RNA(messenger RNA,mRNA);错误发现率(false discovery rate,FDR);倍数变化(fold change,FC);基因本体(Gene Ontology,GO);京都基因和基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG);单侧输尿管梗阻(unilateral ureteral obstruction,UUO);蜗牛家族转录抑制因子(Snail family transcriptional repressor,Snai);干扰素调节因子(interferon regulatory factor,Irf);基本螺旋-环-螺旋家族成员e40(basic helix-loop-helix family member e40,Bhlhe40);富含AT的交互式含域蛋白5A(AT-rich interactive domain 5A,Arid5a);维生素D受体(vitamin D receptor,Vdr);淋巴增强结合因子(lymphoid enhancer binding factor,Lef);芳香烃受体(aryl hydrocarbon receptor,Ahr);叉头框-O1(forkhead box O1,FoxO1);转录因子(transcription factor,Tcf);叉头框-C2(forkhead box C2,FoxC2);GLIS家族锌指蛋白(GLIS family zinc finger,Glis);辅助性T细胞(helper T cell,Th细胞)
利益冲突声明
所有作者均声明不存在利益冲突
Conflict of Interests
The authors declare that there is no conflict of interests
参考文献(References)
- 1.HUMPHREYS B D. Mechanisms of renal fibrosis[J]. Annu Rev Physiol, 2018, 80: 309-326. 10.1146/annurev-physiol-022516-034227 [DOI] [PubMed] [Google Scholar]
- 2.FENG X, ZHANG J, YANG R, et al. The CaMKⅡ inhibitory peptide AIP alleviates renal fibrosis through the TGF-β/Smad and RAF/ERK pathways[J]. J Phar-macol Exp Ther, 2023, 386(3): 310-322. 10.1124/jpet.123.001621 [DOI] [PubMed] [Google Scholar]
- 3.YUAN Q, TAN R J, LIU Y. Myofibroblast in kidney fibrosis: origin, activation, and regulation[J]. Adv Exp Med Biol, 2019, 1165: 253-283. 10.1007/978-981-13-8871-2_12 [DOI] [PubMed] [Google Scholar]
- 4.KUPPE C, IBRAHIM M M, KRANZ J, et al. Decoding myofibroblast origins in human kidney fibrosis[J]. Nature, 2021, 589(7841): 281-286. 10.1038/s41586-020-2941-1 [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.MA T T, MENG X M. TGF-β/Smad and renal fibrosis[J]. Adv Exp Med Biol, 2019, 1165: 347-364. 10.1007/978-981-13-8871-2_16 [DOI] [PubMed] [Google Scholar]
- 6.MENG X M, NIKOLIC-PATERSON D J, LAN H Y. TGF-β: the master regulator of fibrosis[J]. Nat Rev Nephrol, 2016, 12(6): 325-338. 10.1038/nrneph.2016.48 [DOI] [PubMed] [Google Scholar]
- 7.NASTASE M V, ZENG-BROUWERS J, WYGRECKA M, et al. Targeting renal fibrosis: mechanisms and drug delivery systems[J]. Adv Drug Deliv Rev, 2018, 129: 295-307. 10.1016/j.addr.2017.12.019 [DOI] [PubMed] [Google Scholar]
- 8.ISAKA Y. Targeting TGF-β signaling in kidney fibrosis[J]. Int J Mol Sci, 2018, 19(9): 2532. 10.3390/ijms19092532 [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.LI X, DING Z, WU Z, et al. Targeting the TGF-β signaling pathway for fibrosis therapy: a patent review (2015—2020)[J]. Expert Opin Ther Pat, 2021, 31(8): 723-743. 10.1080/13543776.2021.1896705 [DOI] [PubMed] [Google Scholar]
- 10.ZHOU S, YIN X, MAYR M, et al. Proteomic landscape of TGF-β1-induced fibrogenesis in renal fibroblasts[J]. Sci Rep, 2020, 10(1): 19054. 10.1038/s41598-020-75989-4 [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.ASLAM B, BASIT M, NISAR M A, et al. Proteomics: technologies and their applications[J]. J Chromatogr Sci, 2017, 55(2): 182-196. 10.1093/chromsci/bmw167 [DOI] [PubMed] [Google Scholar]
- 12.MCCOMBIE W R, MCPHERSON J D, MARDIS E R. Next-generation sequencing technologies[J]. Cold Spring Harb Perspect Med, 2019, 9(11): a036798. 10.1101/cshperspect.a036798 [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.SU W L, LIU N, MEI L, et al. Global transcriptomic profile analysis of genes involved in lignin biosynthesis and accumulation induced by boron deficiency in poplar roots[J]. Biomolecules, 2019, 9(4): 156. 10.3390/biom9040156 [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.LI B, DEWEY C N. RSEM: accurate transcript quan-tification from RNA-Seq data with or without a reference genome[J]. BMC Bioinformatics, 2011, 12: 323. 10.1186/1471-2105-12-323 [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.YU G, WANG L G, HAN Y, et al. ClusterProfiler: an R package for comparing biological themes among gene clusters[J]. Omics, 2012, 16(5): 284-287. 10.1089/omi.2011.0118 [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.ALTERMANN E, KLAENHAMMER T R. Pathway-Voyager: pathway mapping using the Kyoto Encyclopedia of Genes and Genomes (KEGG) database[J]. BMC Genomics, 2005, 6: 60. 10.1186/1471-2164-6-60 [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.PATEL S, TANG J, OVERSTREET J M, et al. Rac-GTPase promotes fibrotic TGF-β1 signaling and chronic kidney disease via EGFR, p53, and Hippo/YAP/TAZ pathways[J]. FASEB J, 2019, 33(9): 9797-9810. 10.1096/fj.201802489rr [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.PIERSMA B, BANK R A, BOERSEMA M. Signaling in fibrosis: TGF-β, Wnt, and YAP/TAZ converge[J]. Front Med (Lausanne), 2015, 2: 59. 10.3389/fmed.2015.00059 [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.GRANDE M T, SÁNCHEZ-LAORDEN B, LÓPEZ-BLAU C, et al. Snail1-induced partial epithelial-to-mesenchymal transition drives renal fibrosis in mice and can be targeted to reverse established disease[J]. Nat Med, 2015, 21(9): 989-997. 10.1038/nm.3901 [DOI] [PubMed] [Google Scholar]
- 20.LI S, YU L, HE A, et al. Klotho inhibits unilateral ureteral obstruction-induced endothelial-to-mesenchymal transition via TGF-β1/Smad2/Snail1 signaling in mice[J]. Front Pharmacol, 2019, 10: 348. 10.3389/fphar.2019.00348 [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.YAO F, WANG X, CUI Z K, et al. ETS2 promotes epithelial-to-mesenchymal transition in renal fibrosis by targeting JUNB transcription[J]. Lab Invest, 2020, 100(3): 438-453. 10.1038/s41374-019-0331-9 [DOI] [PubMed] [Google Scholar]
- 22.XIAO Z, CHEN C, MENG T, et al. Resveratrol attenuates renal injury and fibrosis by inhibiting trans-forming growth factor-β pathway on matrix metallopro-teinase 7[J]. Exp Biol Med (Maywood), 2016, 241(2): 140-146. 10.1177/1535370215598401 [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.JIANG M Q, WANG L, CAO A L, et al. HuangQi decoction improves renal tubulointerstitial fibrosis in mice by inhibiting the up-regulation of Wnt/β-catenin signaling pathway[J]. Cell Physiol Biochem, 2015, 36(2): 655-669. 10.1159/000430128 [DOI] [PubMed] [Google Scholar]
- 24.ZHANG W, LIU L, XIAO X, et al. Identification of common molecular signatures of SARS-CoV-2 infection and its influence on acute kidney injury and chronic kidney disease[J]. Front Immunol, 2023, 14: 961642. 10.3389/fimmu.2023.961642 [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.MARTÍNEZ-ARIAS L, PANIZO S, ALONSO-MONTES C, et al. Effects of calcitriol and paricalcitol on renal fibrosis in CKD[J]. Nephrol Dial Transplant, 2021, 36(5): 793-803. 10.1093/ndt/gfaa373 [DOI] [PubMed] [Google Scholar]
- 26.TAMURA T, OZATO K. ICSBP/IRF-8: its regulatory roles in the development of myeloid cells[J]. J Interferon Cytokine Res, 2002, 22(1): 145-152. 10.1089/107999002753452755 [DOI] [PubMed] [Google Scholar]
- 27.LEVI B Z, HASHMUELI S, GLEIT-KIELMANOWICZ M, et al. ICSBP/IRF-8 transactivation: a tale of protein-protein interaction[J]. J Interferon Cytokine Res, 2002, 22(1): 153-160. 10.1089/107999002753452764 [DOI] [PubMed] [Google Scholar]
- 28.SALEM S, LANGLAIS D, LEFEBVRE F, et al. Functional characterization of the human dendritic cell immunodeficiency associated with the IRF8 (K108E) mutation[J]. Blood, 2014, 124(12): 1894-1904. 10.1182/blood-2014-04-570879 [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.SALEM S, SALEM D, GROS P. Role of IRF8 in immune cells functions, protection against infections, and susceptibility to inflammatory diseases[J]. Hum Genet, 2020, 139(6-7): 707-721. 10.1007/s00439-020-02154-2 [DOI] [PubMed] [Google Scholar]
- 30.OTOTAKE Y, YAMAGUCHI Y, ASAMI M, et al. Downregulated IRF8 in monocytes and macrophages of patients with systemic sclerosis may aggravate the fibrotic phenotype[J]. J Invest Dermatol, 2021, 141(8): 1954-1963. 10.1016/j.jid.2021.02.015 [DOI] [PubMed] [Google Scholar]
- 31.YOSHIDA Y, YOSHIMI R, YOSHII H, et al. The transcription factor IRF8 activates integrin-mediated TGF-β signaling and promotes neuroinflammation[J]. Immunity, 2014, 40(2): 187-198. 10.1016/j.immuni.2013.11.022 [DOI] [PMC free article] [PubMed] [Google Scholar]
- 32.FU Q, LIAO M, FENG C, et al. Profiling of mRNA of interstitial fibrosis and tubular atrophy with subclinical inflammation in recipients after kidney transplantation[J]. Aging (Albany NY), 2019, 11(14): 5215-5231. 10.18632/aging.102115 [DOI] [PMC free article] [PubMed] [Google Scholar]
- 33.GUO M, DAI Y, JIANG L, et al. Bioinformatics analysis of the mechanisms of diabetic nephropathy via novel biomarkers and competing endogenous RNA network[J]. Front Endocrinol (Lausanne), 2022, 13: 934022. 10.3389/fendo.2022.934022 [DOI] [PMC free article] [PubMed] [Google Scholar]
- 34.GAO F Y, LI X T, XU K, et al. c-MYC mediates the crosstalk between breast cancer cells and tumor microenvironment[J]. Cell Commun Signal, 2023, 21(1): 28. 10.1186/s12964-023-01043-1 [DOI] [PMC free article] [PubMed] [Google Scholar]
- 35.SHEN Y, MIAO N, WANG B, et al. c-Myc promotes renal fibrosis by inducing integrin αv-mediated trans-forming growth factor-β signaling[J]. Kidney Int, 2017, 92(4): 888-899. 10.1016/j.kint.2017.03.006 [DOI] [PubMed] [Google Scholar]