Abstract
目的
探讨循环肿瘤DNA(ctDNA)检测在嵌合抗原受体T细胞(CAR-T细胞)治疗难治复发弥漫大B细胞淋巴瘤(R/R DLBCL)中的预后预测价值,为CAR-T细胞治疗失败患者的预防和后续治疗提供一定指导。
方法
纳入2017年12月至2022年3月在浙江大学医学院附属第一医院接受CAR-T细胞治疗的48例R/R DLBCL患者。对患者治疗前外周血进行187个淋巴瘤相关基因集的ctDNA检测。将患者分为完全缓解(CR)和未达完全缓解(nonCR)两组。使用卡方检验和t检验比较组间临床特征的差异,使用Log-rank检验比较组间生存差异。
结果
CAR-T细胞治疗R/R DLBCL nonCR患者中,突变频率最高的10个基因由高到低依次为TP53(41%)、TTN(36%)、BCR(27%)、KMT2D(27%)、IGLL5(23%)、KMT2C(23%)、MYD88(23%)、BTG2(18%)、MUC16(18%)、SGK1(18%)。Kaplan-Meier生存分析结果表明相较于ctDNA突变基因数≤10的患者,ctDNA突变基因数>10的患者总生存(OS)(1年OS率:0对73.8%,P<0.001)和无进展生存(PFS)较差(1年PFS率:0对51.8%, P=0.011)。治疗前MUC16突变阳性的患者OS更好(2年OS率:56.8%对26.7%,P=0.046),而BTG2突变阳性的患者OS较差(1年OS率:0对72.5%,P=0.005)。
结论
ctDNA检测可以作为评估CAR-T细胞治疗R/R DLBCL患者疗效的工具,治疗前的基因突变负荷、MUC16以及BTG2的突变具有潜在的预后预测价值。
Keywords: 弥漫大B细胞淋巴瘤, 嵌合抗原受体T细胞, 预后, 循环肿瘤DNA
Abstract
Objective
To explore the prognostic value of circulating tumor DNA(ctDNA)testing in patients with refractory/relapsed diffuse large B-cell lymphoma(R/R DLBCL)undergoing chimeric antigen receptor T-cell(CAR-T)therapy, and to guide the prevention and subsequent treatment of CAR-T-cell therapy failure.
Methods
In this study, 48 patients with R/R DLBCL who received CAR-T-cell therapy at the First Affiliated Hospital of Zhejiang University School of Medicine between December 2017 and March 2022 were included. Furthermore, ctDNA testing of 187 lymphoma-related gene sets was performed on peripheral blood samples obtained before treatment. The patients were divided into complete remission and noncomplete remission groups. The chi-square test and t-test were used to compare group differences, and the Log-rank test was used to compare the differences in survival.
Results
Among the patients who did not achieve complete remission after CAR-T-cell therapy for R/R DLBCL, the top ten genes with the highest mutation frequencies were TP53(41%), TTN(36%), BCR(27%), KMT2D(27%), IGLL5(23%), KMT2C(23%), MYD88(23%), BTG2(18%), MUC16(18%), and SGK1(18%). Kaplan-Meier survival analysis revealed that patients with ctDNA mutation genes >10 had poorer overall survival(OS)rate(1-year OS rate: 0 vs 73.8%, P<0.001)and progression-free survival(PFS)rate(1-year PFS rate: 0 vs 51.8%, P=0.011)compared with patients with ctDNA mutation genes ≤10. Moreover, patients with MUC16 mutation positivity before treatment had better OS(2-year OS rate: 56.8% vs 26.7%, P=0.046), whereas patients with BTG2 mutation positivity had poorer OS(1-year OS rate: 0 vs 72.5%, P=0.005).
Conclusion
ctDNA detection can serve as a tool for evaluating the efficacy of CAR-T-cell therapy in patients with R/R DLBCL. The pretreatment gene mutation burden, mutations in MUC16 and BTG2 have potential prognostic value.
Keywords: Diffuse large B-cell lymphoma, Chimeric antigen receptor T-cell, Prognosis, ctDNA
CAR-T细胞疗法在治疗难治复发弥漫大B细胞淋巴瘤(R/R DLBCL)方面取得了巨大突破[1]–[4]。迄今为止,美国食品药品管理局(FDA)已分别于2017年8月、2017年10月、2021年2月批准了三种CAR-T细胞疗法用于DLBCL的治疗,分别为axicabtagene ciloleucel、tisagenelecleucel、lisocabtagene maraleucel。CAR-T细胞治疗通常是R/R DLBCL患者的为数不多的治疗选择。但是仍有近半的患者不能达到完全缓解(CR),这部分患者生存差,因此亟需寻找预测CAR-T细胞治疗R/R DLBCL 患者新的生物标志物,从而及早干预预后欠佳患者,改善生存。
循环肿瘤DNA(ctDNA)从细胞凋亡和(或)坏死的肿瘤细胞中释放,是淋巴瘤的一种新兴生物标志物[5]–[9]。从血液中提取ctDNA的便利性促进了肿瘤突变的鉴定和序列监测。与组织活检相比,外周血ctDNA检测显示出如下明显优势:无创或微创,可反复取材、收集、处理并且分析报告时间短;同时ctDNA能克服肿瘤空间异质性,可相对全面实时地反映患者的肿瘤分子特征;此外,ctDNA还可增加癌症驱动基因突变检出率[10]–[11]。目前ctDNA监测在CAR-T细胞治疗中的应用主要是通过免疫球蛋白重链(IgH)-VDJ、IgH-DJ和免疫球蛋白Kappa轻链或免疫球蛋白lambda轻链区域追踪克隆以评估微小残留[12]–[13],然而,这种监测方法忽略了其他重要的遗传变异。探索更好的覆盖率和预后生物标志物的替代方法是必要的。
因此,本研究通过对接受CAR-T细胞治疗的DLBCL患者外周血进行187个淋巴瘤相关基因集ctDNA的检测,评估ctDNA监测在CAR-T细胞治疗R/R DLBCL患者预后中的应用价值,以预测患者的疾病转归并指导CAR-T细胞治疗失败患者的后续治疗。
病例与方法
一、患者纳入
本研究共收集48例2017年12月至2022年3月在本中心接受CAR-T细胞治疗的R/R DLBCL患者的治疗前血清样本进行187个淋巴瘤相关基因集的ctDNA检测。本研究通过浙江大学医学院附属第一医院伦理委员会批准(浙大一院伦审2023研第79号),所有患者均签署了样本获取知情同意书。
二、外周血血清的分离和制备
抽取患者外周血10 ml至EDTA抗凝管。使用PBS缓冲液以1∶1比例稀释样本,并用吸管混匀。按1∶2比例先将Ficoll溶液加入离心管,然后再用吸管将血样本沿管壁缓慢加至分离液上面,避免两者混合。在密度梯度离心机中进行离心,400 ×g离心25 min,加速度为4级,减速度为0级。离心结束后,得到上层血清,放至−80 °C冰箱保存,用于后续ctDNA测序。
三、ctDNA靶向文库制备
使用VAHTS Serum/Plasma Circulating DNA Kit(南京诺唯赞生物科技有限公司)从血清中提取DNA。利用VAHTS Universal DNA Library Prep Kit for lllumina V3(南京诺唯赞生物科技有限公司)建库。使用 Oubit 荧光定量(赛默飞世尔科技有限公司)和安捷伦 2200 TapeStation 系统质检。使用NextSeq 500(illumina)测序。
四、生物信息分析
原始图像数据文件,经过碱基识别分析转化为原始测序序列,结果以FASTQ文件格式存储,采取fastp软件对转化得到的FASTQ格式原始数据进行过滤去接头。使用BWA、samtools、PICARD、GATK等工具对过滤后的FASTQ格式文件进行比对、排序、标记重复、局部重比、质量值校验后得到校正后bam文件。使用samtools和VarScan2对比对校正后的bam文件进行变异检测,生成原始的vcf文件。使用ANNOVAR软件[14]对体细胞突变进行注释,添加相关字段。根据本地数据库和过滤条件对检出变异进行过滤,等级分类后汇总最终结果用于后续分析。使用maftools工具对后续肿瘤突变数据进行分析。根据突变对蛋白质结构的影响将突变分为:错义突变(missense mutation)、移码缺失(frameshift deletion),移码插入(frameshift insertion)、非移码缺失(nonframeshift deletion)、非移码插入(nonframeshift insertion)、无义突变(stopgain)、终止密码子缺失(stoplost)、启动密码子缺失(startloss)以及剪切突变(splicing)。
五、随访
采用电话或查阅患者病历的方式进行随访,随访截止时间为2022年9月。
六、统计学处理
研究的主要终点为CAR-T细胞治疗后R/R DLBCL患者的无进展生存(PFS)和总生存(OS)。PFS期被定义为从接受CAR-T细胞回输至出现肿瘤进展或死亡的时间间隔;OS期被定义为从接受CAR-T细胞回输至因任何原因引起死亡的时间间隔。采用Kaplan-Meier法绘制生存曲线,采用Log-rank检验比较组间的生存事件差异。使用卡方检验或Fisher精确检验对分类变量进行差异分析。所有统计分析均采用R软件(version 4.0.3)进行,P<0.05为差异具有统计学意义。
结果
一、患者临床特征
本研究回顾性分析了自2017年12月至2022年3月在本中心接受CAR-T细胞治疗的48例R/R DLBCL患者,其中26例达到CR,14例达到部分缓解(PR),8例疾病稳定(SD)或疾病进展(PD)。患者的基线特征如表1所示,CR组和未达CR(nonCR)组间的年龄、性别、分型、末次化疗间隔时间、化疗线数、是否经过移植、细胞因子释放综合征(CRS)等级、Ann Arbor分期、IPI评分以及基线乳酸脱氢酶和β2微球蛋白水平差异均无统计学意义。
表1. CAR-T细胞治疗后CR组与nonCR组弥漫大B细胞淋巴瘤患者临床特征比较.
| 指标 | 总数(48例) | CR组(26例) | nonCR组(22例) | P值 |
| 性别[例(%)] | 0.90 | |||
| 女 | 19(40) | 11(42) | 8(36) | |
| 男 | 29(60) | 15(58) | 14(64) | |
| 年龄[岁,M(Q1,Q3)] | 59.5(46.75, 65.25) | 60.5(49, 65.75) | 54.5(43.25, 65) | 0.40 |
| 分型 | 0.23 | |||
| GCB | 9(19) | 6(23) | 3(14) | |
| nonGCB | 31(65) | 14(54) | 17(77) | |
| NA | 8(16) | 6(23) | 2(9) | |
| 末次化疗间隔时间[月,M(Q1,Q3)] | 54(36, 102) | 91.5(41.25, 108) | 49(33, 92) | 0.16 |
| 化疗线数[例(%)] | 0.48 | |||
| ≤2线 | 15(33) | 7(29) | 8(36) | |
| >2线 | 33(67) | 19(71) | 14(64) | |
| 是否移植[例(%)] | 1.00 | |||
| 否 | 43(90) | 23(88) | 20(91) | |
| 是 | 5(10) | 3(12) | 2(9) | |
| 乳酸脱氢酶[U/L,M(Q1,Q3)] | 286.00(225.75, 409.50) | 279.50(229.75, 412.50) | 297.50(231.00, 361.25) | 0.90 |
| β2微球蛋白[µg/L,M(Q1,Q3)] | 2 595.0(1 952.5, 3 575.0) | 2 809.5(2 037.5, 3 575.0) | 2 460.0(1 977.5, 3 190.0) | 0.82 |
| Ann Arbor分期[例(%)] | 0.56 | |||
| Ⅰ/Ⅱ期 | 7(14) | 5(19) | 2(9) | |
| Ⅲ/Ⅳ期 | 41(86) | 21(81) | 20(91) | |
| IPI评分[例(%)] | 0.32 | |||
| 0~1分 | 8(16) | 6(23) | 2(9) | |
| 2~3分 | 32(67) | 17(65) | 15(68) | |
| 4~5分 | 8(17) | 3(12) | 5(23) | |
| CRS等级[例(%)] | 0.28 | |||
| 0~1级 | 28(59) | 17(66) | 11(50) | |
| 2~3级 | 20(41) | 9(35) | 11(50) |
注 CR:完全缓解;nonCR:未达完全缓解;CRS:细胞因子释放综合征;GCB:生发中心来源;nonGCB:非生发中心来源;IPI:淋巴瘤的国际预后指数
二、CAR-T细胞治疗DLBCL患者基因突变图谱
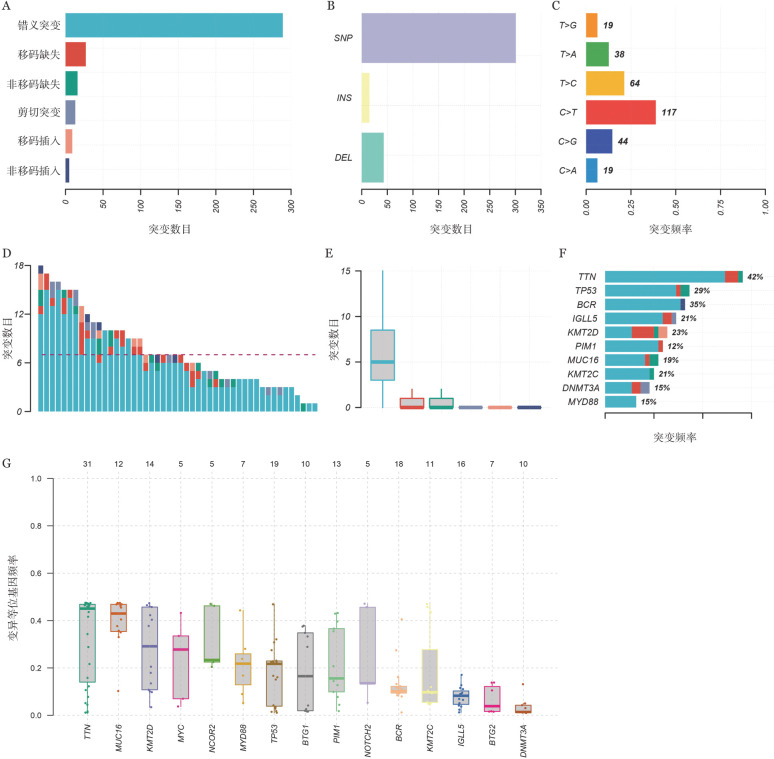
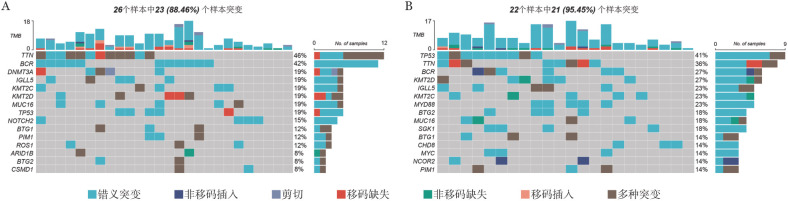
对突变信息进行注释和过滤后,48个样本以错义突变为主(图1A、B),其中又以C>T和T>C突变为主(图1C),中位ctDNA突变数目为7个(图1D),错义突变的中位ctDNA最高(图1E)。如图1F所示,突变频率最高的10个基因由高到低依次为TTN(42%)、BCR(35%)、TP53(29%)、KMT2D(23%)、IGLL5(21%)、KMT2C(21%)、MUC16(19%)、DNMT3A(15%)、MYD88(15%)、PIM1(12%)。突变频率最高的15个基因的变异等位基因频率(VAF)分布如图1G所示。不同疗效组患者基因突变图谱如图2所示,CR组突变频率最高的10个基因由高到低依次为TTN(46%)、BCR(42%)、IGLL5(19%)、KMT2C(19%)、KMT2D(19%)、MUC16(19%)、TP53(19%)、NOTCH(15%)、BTG1(12%)、PIM1(12%);在nonCR组中,突变频率最高的10个基因由高到低依次为TP53(41%)、TTN(36%)、BCR(27%)、KMT2D(27%)、IGLL5(23%)、KMT2C(23%)、MYD88(23%)、BTG2(18%)、MUC16(18%)、SGK1(18%)。
图1. 48例难治复发弥漫大B细胞淋巴瘤患者治疗前的血清样本的循环肿瘤DNA(ctDNA)突变景观图 A 6种主要突变数目柱状图; B 3种突变类型柱状图,SNP:点突变;DEL:缺失突变;INS:插入突变; C 6种SNV突变频谱; D 每个样本ctDNA突变数目柱状图; E 所有样本6种主要突变数目箱型图,从左往右分别代表错义突变、移码缺失、非移码缺失、剪切突变、移码插入、非移码插入; F 突变频率前10基因的突变频率; G 突变频率最高的15个基因的变异等位基因频率分布.
图2. 完全缓解组(A)与未达完全缓解组(B)循环肿瘤DNA突变瀑布图.
将CR组和nonCR组患者治疗前的特定基因分配到每个通路上,评估了10条与癌症研究相关的典型信号通路,包括细胞周期、Hippo、Myc、Notch、Nrf2、PI3K、RTK-RAS、TGFb、p53和Wnt通路,结果表明CR组受影响频率最高的三条信号通路由高到低依次为RTK-RAS(38.5%)、TP53(19.2%)、NOTCH(19.2%)信号通路,而nonCR组受影响频率最高的三条信号通路由高到低依次为TP53(45.5%)、RTK-RAS(36.4%)、NOTCH(31.8%)信号通路(图3)。
图3. 完全缓解组(A)与未达完全缓解组(B)致癌生物途径的富集.
三、CAR-T细胞治疗DLBCL患者基因突变的互斥性与共现性
Oncodrive函数结果表明NCOR2、MYD88和BCR可能是接受CAR-T细胞疗法的R/R DLBCL患者的癌症驱动基因(图4A)。随后用pfamDomains函数来注释和统计导致氨基酸改变的突变所在pfam结构域,结果表明出现突变频率最高的5个pfam结构域由高到低依次为FN3、P53、RhoGAP_Bcr、Pkinase以及I-set(图4B)。此外,通过somaticInteractions函数对基线ctDNA突变频率最高的15个基因两两之间进行成对的Fisher精确检验分析突变的互斥性和共现性,共发现4对共现性的基因对(P<0.05),未发现互斥性基因对。其中,MYD88与BTG2以及IGLL5呈共现性,BCR与MYC呈共现性,而BTG1与PIM1呈共现性,具体结果如图4C所示。
图4. 循环肿瘤DNA(ctDNA)突变的互斥性与共现性 A 基线时由 oncodrive 鉴定的疾病相关驱动基因(FDR<0.05),图中点的大小以及基因名后的数字展示的是基因内突变cluster的数量,X轴为cluster内的突变数量或者占突变总数的比例,Y轴是-log10(FDR); B Pfam蛋白结构域和基因数散点图,圆点代表pfam结构域,对应X轴为该结构域中出现的突变数量,Y轴以及圆点的大小为影响的基因数,参数top用来选择标记突变数最多的pfam domain数量; C 基线时中互斥和共存的ctDNA对显示为三角矩阵。绿色表示共现趋势,而黄色表示排它性趋势.
四、ctDNA在CAR-T细胞治疗DLBCL预后中的作用
根据每例患者检测到的突变基因的数目将患者分为≤10个和>10个突变基因的两组,治疗前突变基因>10个的患者OS(1年OS率:0对73.8%,P<0.001)(图5A)和PFS(1年PFS率:0对51.8%,P=0.011)(图5B)更差。对治疗前突变频率大于10%的突变基因进行Kaplan-Meier生存分析,结果表明治疗前MUC16突变阳性的患者OS更好(2年OS率:56.8%对26.7%,P=0.046)(图5C),而BTG2突变阳性的患者OS较差(1年OS率:0对72.5%,P=0.005)(图5D)。
图5. 循环肿瘤DNA(ctDNA)对CAR-T细胞治疗弥漫大B细胞淋巴瘤患者预后的预测作用 A 治疗前不同ctDNA突变基因数目组的总生存曲线; B 治疗前不同ctDNA突变基因数目组的无进展生存曲线; C 治疗前不同MUC16状态组的总生存曲线; D 治疗前不同BTG2状态组的总生存曲线.
讨论
尽管在大量的R/R DLBCL患者的临床实践中,CAR-T细胞疗法展现了前所未有的有效性,但仍有近半的患者不能缓解或缓解后出现复发,这部分患者往往后续可选治疗方案有限,生存期短[15]–[16]。因此,提前预测CAR-T细胞治疗疗效欠佳的患者以制定早期干预方案至关重要。
癌症基因组测序计划加深了对不同癌种基因组突变谱的理解,发现了很多新的癌症驱动基因突变以及疗效预测标志物。然而,对于接受CAR-T细胞治疗的R/R DLBCL患者的基因突变谱及其分子生物学的研究仍然有限。R/R DLBCL研究进展受到的最主要限制是DLBCL组织标本的获取困难,穿刺活检样本往往难以满足检测要求,并且深层组织部位的DLBCL样本难以获得。因此,在临床上迫切需要寻找一种能够替代穿刺组织活检的标本,全面实时地反映DLBCL患者肿瘤分子特征。ctDNA的预后预测作用已经得到很好的证实,治疗前和治疗中的浓度都能很好地评估CAR-T细胞治疗的预后[13],[17]。我们前期的一项8例动态ctDNA监测的研究结果表明了类似的结果,治疗前ctDNA的阳性突变基因数目与接受CAR-T细胞治疗的DLBCL患者的长期预后有关[18]。本研究共入组了48例接受CAR-T细胞治疗的R/R DLBCL患者,对治疗前外周血进行187个淋巴瘤相关基因集的捕获和测序,绘制了接受CAR-T细胞治疗的难治复发DLBCL患者的基因突变图谱,并分析了不同疗效组基因突变图谱的差异、癌症相关通路的差异以及其在预后预测中的作用。进一步证实了治疗前ctDNA的阳性突变基因数目能很好地预测CAR-T治疗DLBCL患者的临床转归。
此外,研究发现治疗前MUC16(CA125)以及BTG2两个基因突变对CAR-T细胞治疗DLBCL患者的预后具有预测作用。MUC16是一种Ⅰ型跨膜黏蛋白,由C末端结构域、串联重复区和细胞外N末端三部分组成,已被广泛用作卵巢癌的生物标志物,其表达与疾病进展有关[19]。多项研究表明,MUC16在多种癌症类型中过度表达,并且,其过表达与多种恶性肿瘤预后较差有关[20]–[23]。一项大样本胃癌基因组测序结果表明:MUC16突变可能与更好的预后以及免疫反应和细胞周期信号通路有关[24]。另一项对黑色素瘤的基因组研究同样显示了类似的结果:MUC16突变可能会影响免疫相关途径和肿瘤浸润免疫细胞亚群,从而改善黑色素瘤患者的预后[25]。目前尚未见MUC16在CAR-T细胞治疗中的报道。本研究结果表明治疗前MUC16突变阳性的患者相较于MUC16野生型患者预后更好。这提示MUC16可能是CAR-T细胞治疗DLBCL的潜在生物标志物。BTG/TOB基因家族是一个抗增殖基因家族,被发现在调节细胞周期、凋亡和分化等方面都发挥着重要作用。BTG2作为该家族第一个被发现的基因,已被证实在淋巴恶性肿瘤和实体瘤中发挥肿瘤抑制因子的作用[26]。之前多项全基因组分析结果研究表明,在B细胞恶性肿瘤中经常观察到BTG2的遗传畸变[27]–[28],这表明BTG2的突变可能导致其肿瘤抑制因子作用减弱从而导致肿瘤的发生发展。在本研究中12%的DLBCL患者发生了BTG2的突变,并且BTG2突变阳性的患者OS较差。这提示BTG2突变可能可以作为CAR-T细胞治疗DLBCL患者预后差的标志。
总体而言,本研究探索了ctDNA在CAR-T细胞治疗R/R DLBCL患者中作为预后生物标志物的可行性。治疗前的基因突变个数、MUC16以及BTG2突变状态在CAR-T细胞疗法中具有潜在的预后价值,但本研究的样本量仍较为有限,研究结果需要在更大的独立队列中进行验证。
Funding Statement
基金项目:国家自然科学基金(82130003、82270234);深圳三明医药工程(SZSM202111004)
Fund program: National Natural Science Foundation of China(82130003, 82270234); Sanming Project of Medicine in Shenzhen(SZSM202111004)
Footnotes
利益冲突 所有作者声明无利益冲突
作者贡献声明 周凌辉:研究设计,数据分析,文章起草;冯友琴:样本收集;胡永仙、黄河:研究设计,文章审核
References
- 1.Kochenderfer JN, Dudley ME, Kassim SH, et al. Chemotherapy-refractory diffuse large B-cell lymphoma and indolent B-cell malignancies can be effectively treated with autologous T cells expressing an anti-CD19 chimeric antigen receptor[J] J Clin Oncol. 2015;33(6):540–549. doi: 10.1200/JCO.2014.56.2025. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Kochenderfer JN, Somerville R, Lu T, et al. Lymphoma Remissions Caused by Anti-CD19 Chimeric Antigen Receptor T Cells Are Associated With High Serum Interleukin-15 Levels[J] J Clin Oncol. 2017;35(16):1803–1813. doi: 10.1200/JCO.2016.71.3024. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.Locke FL, Neelapu SS, Bartlett NL, et al. Phase 1 Results of ZUMA-1: A Multicenter Study of KTE-C19 Anti-CD19 CAR T Cell Therapy in Refractory Aggressive Lymphoma[J] Mol Ther. 2017;25(1):285–295. doi: 10.1016/j.ymthe.2016.10.020. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Neelapu SS, Locke FL, Bartlett NL, et al. Axicabtagene Ciloleucel CAR T-Cell Therapy in Refractory Large B-Cell Lymphoma[J] N Engl J Med. 2017;377(26):2531–2544. doi: 10.1056/NEJMoa1707447. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.Kurtz DM, Green MR, Bratman SV, et al. Noninvasive monitoring of diffuse large B-cell lymphoma by immunoglobulin high-throughput sequencing[J] Blood. 2015;125(24):3679–3687. doi: 10.1182/blood-2015-03-635169. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Roschewski M, Dunleavy K, Pittaluga S, et al. Circulating tumour DNA and CT monitoring in patients with untreated diffuse large B-cell lymphoma: a correlative biomarker study[J] Lancet Oncol. 2015;16(5):541–549. doi: 10.1016/S1470-2045(15)70106-3. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Scherer F, Kurtz DM, Newman AM, et al. Distinct biological subtypes and patterns of genome evolution in lymphoma revealed by circulating tumor DNA[J] Sci Transl Med. 2016;8(364):364ra155. doi: 10.1126/scitranslmed.aai8545. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Rossi D, Diop F, Spaccarotella E, et al. Diffuse large B-cell lymphoma genotyping on the liquid biopsy[J] Blood. 2017;129(14):1947–1957. doi: 10.1182/blood-2016-05-719641. [DOI] [PubMed] [Google Scholar]
- 9.宋 妮妮, 盛 立霞, 欧阳 桂芳. 循环肿瘤DNA在B细胞非霍奇金淋巴瘤中的研究进展[J] 白血病·淋巴瘤. 2019;(5):306–309. doi: 10.3760/cma.j.issn.1009-9921.2019.05.013. [DOI] [Google Scholar]
- 10.Diaz LA, Jr, Bardelli A. Liquid biopsies: genotyping circulating tumor DNA[J] J Clin Oncol. 2014;32(6):579–586. doi: 10.1200/JCO.2012.45.2011. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Crowley E, Di Nicolantonio F, Loupakis F, et al. Liquid biopsy: monitoring cancer-genetics in the blood[J] Nat Rev Clin Oncol. 2013;10(8):472–484. doi: 10.1038/nrclinonc.2013.110. [DOI] [PubMed] [Google Scholar]
- 12.Hossain NM, Dahiya S, Le R, et al. Circulating tumor DNA assessment in patients with diffuse large B-cell lymphoma following CAR T-cell therapy[J] Leuk Lymphoma. 2019;60(2):503–506. doi: 10.1080/10428194.2018.1474463. [DOI] [PubMed] [Google Scholar]
- 13.Frank MJ, Hossain NM, Bukhari A, et al. Monitoring of Circulating Tumor DNA Improves Early Relapse Detection After Axicabtagene Ciloleucel Infusion in Large B-Cell Lymphoma: Results of a Prospective Multi-Institutional Trial[J] J Clin Oncol. 2021;39(27):3034–3043. doi: 10.1200/JCO.21.00377. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Wang K, Li M, Hakonarson H. ANNOVAR: functional annotation of genetic variants from high-throughput sequencing data[J] Nucleic Acids Res. 2010;38(16):e164. doi: 10.1093/nar/gkq603. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.张 琪, 肖 毅. CD19嵌合抗原受体T细胞治疗弥漫大B细胞淋巴瘤的复发机制及应对策略[J] 中华血液学杂志. 2022;43(9):788–792. doi: 10.3760/cma.j.issn.0253-2727.2022.09.014. [DOI] [Google Scholar]
- 16.肖 霞, 江 嫣雨, 曹 雅青, et al. CD19CAR-T细胞治疗B细胞淋巴瘤22例疗效及安全性[J] 中华血液学杂志. 2019;40(4):276–280. doi: 10.3760/cma.j.issn.0253-2727.2019.04.003. [DOI] [Google Scholar]
- 17.Sworder BJ, Kurtz DM, Alig SK, et al. Determinants of resistance to engineered T cell therapies targeting CD19 in large B cell lymphomas[J] Cancer Cell. 2023;41(1):210–225.e5. doi: 10.1016/j.ccell.2022.12.005. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Zhou L, Zhao H, Shao Y, et al. Serial surveillance by circulating tumor DNA profiling after chimeric antigen receptor T therapy for the guidance of r/r diffuse large B cell lymphoma precise treatment[J] J Cancer. 2021;12(18):5423–5431. doi: 10.7150/jca.60390. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Felder M, Kapur A, Gonzalez-Bosquet J, et al. MUC16 (CA125): tumor biomarker to cancer therapy, a work in progress[J] Mol Cancer. 2014;13:129. doi: 10.1186/1476-4598-13-129. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Shimizu A, Hirono S, Tani M, et al. Coexpression of MUC16 and mesothelin is related to the invasion process in pancreatic ductal adenocarcinoma[J] Cancer Sci. 2012;103(4):739–746. doi: 10.1111/j.1349-7006.2012.02214.x. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.Chen SH, Hung WC, Wang P, et al. Mesothelin binding to CA125/MUC16 promotes pancreatic cancer cell motility and invasion via MMP-7 activation[J] Sci Rep. 2013;3:1870. doi: 10.1038/srep01870. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22.Higashi M, Yamada N, Yokoyama S, et al. Pathobiological implications of MUC16/CA125 expression in intrahepatic cholangiocarcinoma-mass forming type[J] Pathobiology. 2012;79(2):101–106. doi: 10.1159/000335164. [DOI] [PubMed] [Google Scholar]
- 23.Liang C, Qin Y, Zhang B, et al. Oncogenic KRAS Targets MUC16/CA125 in Pancreatic Ductal Adenocarcinoma[J] Mol Cancer Res. 2017;15(2):201–212. doi: 10.1158/1541-7786.MCR-16-0296. [DOI] [PubMed] [Google Scholar]
- 24.Li X, Pasche B, Zhang W, et al. Association of MUC16 Mutation With Tumor Mutation Load and Outcomes in Patients With Gastric Cancer[J] JAMA Oncol. 2018;4(12):1691–1698. doi: 10.1001/jamaoncol.2018.2805. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.Wang Z, Hou H, Zhang H, et al. Effect of MUC16 mutations on tumor mutation burden and its potential prognostic significance for cutaneous melanoma[J] Am J Transl Res. 2022;14(2):849–862. [PMC free article] [PubMed] [Google Scholar]
- 26.Kim SH, Jung IR, Hwang SS. Emerging role of anti-proliferative protein BTG1 and BTG2[J] BMB Rep. 2022;55(8):380–388. doi: 10.5483/BMBRep.2022.55.8.092. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27.Lohr JG, Stojanov P, Lawrence MS, et al. Discovery and prioritization of somatic mutations in diffuse large B-cell lymphoma (DLBCL) by whole-exome sequencing[J] Proc Natl Acad Sci U S A. 2012;109(10):3879–3884. doi: 10.1073/pnas.1121343109. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 28.Morin RD, Mendez-Lago M, Mungall AJ, et al. Frequent mutation of histone-modifying genes in non-Hodgkin lymphoma[J] Nature. 2011;476(7360):298–303. doi: 10.1038/nature10351. [DOI] [PMC free article] [PubMed] [Google Scholar]