Abstract
目的
基于Luminex液相芯片检测系统构建粪便人源性蛋白诊断体系,并评估其对结直肠肿瘤的早期诊断价值。
方法
收集2021年1月~2023年1月蚌埠医学院第一附属医院收治的结直肠癌(CRC)患者70例为CRC组、结直肠腺瘤(CRA)患者42例为CRA组,以及同期健康对照(HC)受试者38例为HC组。收集各组受试者粪便,使用Tris-Hcl缓冲液提取粪便总蛋白,通过Luminex液相芯片检测技术分析3组受试者粪便中基质金属蛋白酶-9(MMP-9)、人源性视黄醇结合蛋白4(RBP4)、甲壳素酶-3样蛋白1(CHI3L1)和补体成分3a(C3a)的水平差异;收集受试者外周血,分离血清检测癌胚抗原(CEA)和糖类抗原19-9(CA19-9)的水平;使用受试者工作曲线(ROC)分析MMP-9、RBP4、CHI3L1和C3a联合以及CEA和CA19-9联合对CRC、CRA、CRC和CRA的诊断效能,并比较两种组合诊断效能的差异。
结果
CRC组患者粪便中MMP-9、RBP4、CHI3L1和C3a水平均显著高于HC组(P < 0.05),CRC组粪便中MMP-9和CHI3L1水平显著高于CRA组(P < 0.05),但是RBP4和C3a在CRC和CRA组间并无显著性差异(P > 0.05);另外,CRC患者外周血中CEA和CA19-9水平均显著高于HC组和CRA组(P < 0.05),但是CRA组和HC组之间无显著性差异(P > 0.05)。ROC曲线分析显示,MMP-9、RBP4、CHI3L1和C3a联合诊断CRC的灵敏度为91.4%,特异度为100.0%,联合诊断CRA的灵敏度为81.0%,特异度为89.5%,联合诊断CRC和CRA的灵敏度为83.9%,特异度为97.4%。Z检验比较分析显示,粪便蛋白MMP-9、RBP4、CHI3L1和C3a联合诊断CRC,CRA以及CRA和CRC的效能显著优于外周血肿瘤标志物CEA和CA19-9(P < 0.05)。
结论
Luminex液相芯片检测粪便人源性蛋白RBP4、MMP-9、CHI3L1、C3a对结直肠肿瘤早期诊断具有价值且优于外周血肿瘤标志物CEA和CA19-9,具有较高的临床应用推广意义。
Keywords: 结直肠肿瘤, 早期诊断, 粪便蛋白, Luminex液相芯片技术
Abstract
Objective
To construct a stool-based human protein diagnostic system using the Luminex liquid chip system for early diagnosis of colorectal tumors.
Methods
From January, 2021 to January, 2023, 70 patients with colorectal cancer (CRC), 42 patients with colorectal adenoma (CRA), and 38 healthy individuals were recruited from our hospital for detecting fecal protein levels of matrix metalloproteinase-9 (MMP-9), retinol-binding protein 4 (RBP4), chitinase-3-like protein 1 (CHI3L1), and complement component 3a (C3a) using Luminex liquid chip technology and serum levels of carcinoembryonic antigen (CEA) and carbohydrate antigen 19-9 (CA19-9) using chemiluminescence assay. Receiver-operating characteristic (ROC) curve analysis was used for assessing the diagnostic efficacy of the combination of MMP-9, RBP4, CHI3L1 and C3a and the combination of CEA and CA19-9 for colorectal tumors.
Results
The fecal contents of MMP-9, RBP4, CHI3L1, and C3a were significantly higher in CRC patients than in healthy individuals (P < 0.05). Fecal MMP-9 and CHI3L1 levels were significantly higher in CRC than in CRA patients (P < 0.05), but RBP4 and C3a levels did not differ significantly (P>0.05). CRC patients had significantly higher serum CEA and CA19-9 levels than healthy individuals and CRA patients (P < 0.05), but the differences were not significant between the latter two groups (P>0.05). ROC analysis showed that the sensitivity and specificity of the combination of MMP-9, RBP4, CHI3L1, and C3a was 91.4% and 100.0%, for diagnosing CRC, 81.0% and 89.5% for diagnosing CRA, and 83.9% and 97.4% for a combined diagnosis of CRC and CRA, respectively. Z-test analysis indicated that fecal MMP-9, RBP4, CHI3L1, and C3a contents had a greater diagnostic efficacy than serum tumor markers CEA and CA19-9 for a combined diagnosis of colorectal tumors (P < 0.05).
Conclusion
The Luminex liquid chip detection system for detecting decal RBP4, MMP-9, CHI3L1, and C3a provides an effective means for early diagnosis of colorectal tumors with a greater diagnostic efficacy than serum CEA and CA19-9 levels.
Keywords: colorectal cancer, early diagnosis, fecal protein, Luminex liquid chip technology
结直肠癌(CRC)在我国的发病率和死亡率均居第4位,由于早期诊断率低导致其病死率高于全球平均水平[1-3]。在CRC的诊断方式中,准确性高的结肠镜检查因有创而难以成为筛查手段,而外周血肿瘤标志物的诊断效能不足,目前仍然缺乏适用于早期筛查的方法[4]。粪便检测具有无创、便利等优势有望成为CRC的早筛途径之一,而当前基于粪便标本的早筛检查多为基因检测,却忽略了粪便蛋白的价值。相比较粪便蛋白检测,粪便中基因检测等分子诊断标志物在结直肠癌的早期诊断中展现出更多的优势,且近年来多个基于粪便的结直肠癌早筛早诊基因检测应用于临床,本研究采用蛋白检测优势在于蛋白质类分子是CRC相关脱落细胞在粪便中稳定存在的主要分子形式也最能直接反应身体状况,可精准和全面预测疾病进展情况[5, 6]。临床常用的粪便隐血试验即是一种粪便蛋白检测,但其对CRC的早期诊断存在敏感性高而特异性不理想的弊端,多个粪便蛋白的联合检测有望提高诊断效能。新近,我们采用蛋白质组学筛查结合ELISA验证的方式鉴定出多个具有CRC早期诊断价值的粪便蛋白[7],但如何向临床转化应用还需解决粪便蛋白的多指标联合检测以及多样本同时检测等技术问题。为解决以上问题,本研究引入Luminex液相芯片检测技术,其不仅具有高通量特性,还可实现单次上机检测384样本中的80个指标,极具临床应用价值[8-10]。本研究基于前期蛋白质组学鉴定出的4种CRC粪便差异蛋白(MMP-9、RBP4、CHI3L1和C3a),以CRC、结直肠腺瘤(CRA)和健康对照(HC)的粪便样本为研究对象,尝试采用Luminex检测技术分析粪便差异蛋白的联合检测对于结直肠肿瘤(CRC、CRA)的诊断价值,以期望为CRC的早筛提供高通量、安全、无创的新方法。
1. 资料和方法
1.1. 研究对象
本研究纳入2021年1月~2023年1月于蚌埠医学院第一附属医院就诊的结直肠癌患者列为CRC组,结直肠腺瘤患者列为CRA组,另纳入健康受试者列为HC组。入选标准:所有受试者均接受电子结肠镜检查,CRC和结直肠腺瘤的诊断和鉴别诊断依据活组织病理学检查,CRC的临床分期参照AJCC推荐的《结直肠癌TNM分期标准·第八版》;当前未接受其他临床或流行病学研究;患者或监护人签署知情同意书。排除标准:CRC组患者合并其它组织起源的恶性肿瘤,腺瘤组和对照组合并任何组织起源的恶性肿瘤;除分组疾病外,合并消化系统急慢性疾病;合并活动性感染、严重肝肾功能障碍、血液病等已知可能影响患者肿瘤标志物表达的疾病;研究者认为患者有可能混淆研究结果或者存在其他额外风险的病史;入选前30 d内已经接受任何研究治疗。剔除标准:受试者出现不可耐受等不良事件,研究者认为会危及受试者安全;受试者撤回知情同意书;违反研究方案者(不符合入选/排除标准,未按规定随访);全部退出受试者的信息及退出的原因均被记录存档。
依据以上标准,纳入CRC患者70例,CRA患者42例,另纳入健康受试者38例。本研究经蚌埠医学院第一附属医院医学伦理委员会批准,伦理号为伦科批字[2021]第221号。
1.2. 研究方法
1.2.1. 标本采集
所有的受试者在手术前或内镜检查前留取粪便2~4 g,置于粪便收集器中,并在1 h内转移至-80 ℃冰箱中保存。使用肝素钠抗凝管采集受试者空腹静脉血3~5 mL,离心(2000 g,5 min)分离血浆,分装后冻存至-80 ℃冰箱中。
1.2.2. 粪便蛋白提取方法
取新鲜粪便中间部位,每份粪便称取1 g,置于10 mL Tris-Hcl缓冲液(20 mmol/L Tris-HCl,1%Triton X-100,pH值7.5)中,含蛋白酶抑制剂混合物(Solarbio)。采用涡旋震荡仪充分混匀,于冰上静置裂解细胞20 min后,使用低温离心机(2500 g,5 min)离心去除粪渣,将粪便上清液转移至新的离心管中,并重复斡旋震荡、裂解和离心步骤,直至无明显粪渣。使用低温超速离心机(13 000 g,30 min)离心收集含蛋白上清液,分装后冻存于-80 ℃冰箱,待检测。
1.2.3. Luminex技术检测粪便蛋白(RBP4、MMP-9、CHI3L1、C3a)表达
(1) 标本处理和预实验:将含粪便蛋白的上清液从-80 ℃冰箱取出于冰上解冻,涡旋振荡混匀后再次离心(10 000 g,5 min)除去样品中的固体成分。首先选择3例CRC,3例CRA及3例HC粪便蛋白标本开展预实验,根据试剂盒说明书进行检测,依据预实验结果确定粪便上清液最佳稀释比例。(2)试剂准备和标准品稀释:试剂盒(Thermo Fisher Scientific)于室温下平衡后,进行Wash buffer(10×)、磁珠微球Beads(50×)、Detection Antibody(50×)以及标准品的稀释。标准品使用Universal Assay Buffer进行4倍梯度稀释,C3a标准品浓度梯度为0、2.18、34.86、139.45、557.81、2231.25、8925.00 pg/mL;RBP4标准品浓度梯度为0、11.06、44.22、176.88、2830.08、11320.31、45281.25 pg/mL;MMP-9标准品浓度梯度为0、0.92、3.66、14.65、58.59、937.50和3750.00 pg/mL;CHI3L1标准品浓度梯度为0、32.45、129.78、519.14、2076.56、8306.25、132 900.00 pg/mL。(3)实验步骤:将预混的Beads加入96孔板中,50 μL/孔,使用1×Wash buffer洗涤,96孔板全程置于磁性分离板中,确保Beads被吸附在96孔板中。向96孔板中分别加入标准品和样本(50 μL,原液),空白对照加入等体积Universal Assay Buffer,于摇床上(500 r/min,4 ℃)孵育过夜。使用1× Wash buffer洗板3次,加入Detection Antibody抗体(25 μL),置于摇床上(500 r/min,室温)孵育30 min。重复洗板步骤后加入50 μL SA-PE,置于摇床上(500 r/min,室温)孵育30 min。重复洗板步骤后每孔中加入120 μL Reading buffer孵育30 min。(4)数据检测与分析:使用Luminex 200仪器读取各孔平均荧光强度(MFI)值,标准曲线采用五参数非线性回归的方式拟合,并根据标准曲线计算样品浓度。
1.2.4. 外周血CEA和CA19-9水平检测
采用雅培ARCHITECT i2000SR免疫测定分析仪及其配套试剂盒定量测定外周血CEA和CA19-9水平,严格按照仪器和项目标准操作程序(SOP)文件进行操作。
1.3. 统计学方法
采用SPSS22.0软件分析数据,正态分布的计量资料以均数±标准差表示,组间差异比较使用单因素方差分析;非正态分布的计量资料以中位数(四分位数)[M (Q1,Q3)]表示,组间差异比较使用秩和检验;计数资料采用百分数(%)表示,组间差异比较使用χ2检验。以活组织病理学检查结果为金标准,诊断价值分析采用ROC曲线,诊断效能的组间比较采用Z检验;利用ROC曲线下面积计算样本量,计算采用Medcalc 13.0软件,设定具有诊断效能的最小ROC曲线下面积为0.80,α=0.05,β=0.10,阴性组与阳性组的比例为1[11],计算结果显示阳性组(CRC、CRA)和阴性组(HC)各需17例可满足本研究需求。P < 0.05(双侧)认为差异有统计学意义。
2. 结果
2.1. 基线资料
本文共纳入CRC患者70例(男性43例,女性27例),CRA患者42例(男性30例,女性12例),HC受试者38例(男性18例,女性20例),3组受试者的性别比例、年龄和体质量指数(BMI)差异无统计学意义(P>0.05,表 1)。
表 1.
基线资料
Demographic data of the participants
| Clinical parameters | HC (n=38) | CRA (n=42) | CRC (n=70) | χ2/F | P |
| HC: Health control; CRA: Colorectal adenomas; CRC: Colorectal cancer. | |||||
| Gender (Male) | 18 (47.37%) | 30 (71.43%) | 43 (61.43%) | 4.872 | 0.088 |
| Age (year) | 60.18±1.91 | 64.76±2.35 | 64.31±1.36 | 1.652 | 0.195 |
| BMI (kg/m2) | 20.94±0.27 | 21.05±0.36 | 20.33±0.26 | 1.901 | 0.153 |
2.2. CRC组、CRA组和HC组粪便中MMP-9、RBP4、CHI3L1和C3a蛋白水平比较
使用Luminex技术分析MMP-9、RBP4、CHI3L1和C3a在CRC组、CRA组和HC组受试者粪便中的表达水平。结果显示,CRC组患者粪便中MMP-9水平显著高于CRA组和HC组(P < 0.05),且CRA组显著高于HC组(图 1A,P < 0.05);RBP4在CRC组和CRA组粪便中的水平均显著高于HC组(P < 0.05),而CRC组和CRA组之间并差异无统计学意义(P>0.05,图 1B);CHI3L1在CRC组和CRA组均显著高于HC组(P < 0.05),且CRC组显著高于CRA组(P < 0.05,图 1C);C3a在CRC组和CRA组粪便中均显著高于HC组(P < 0.05),但是CRC组和CRA组之间差异无统计学意义(P>0.05,图 1D)。
图 1.
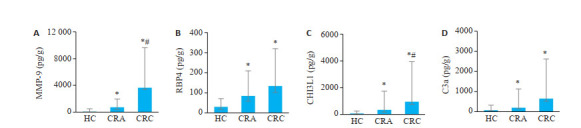
Luminex技术检测CRC组、CRA组和HC组受试者粪便中MMP-9、RBP4、CHI3L1和C3a蛋白水平
Protein levels of MMP-9 (A), RBP4 (B), CHI3L1 (C) and C3a (D) in fecal specimens of CRC patients, CRA patients and healthy individuals (HC) detected by Luminex technology. *P < 0.05 vs HC group; #P < 0.05 vs CRA group.
2.3. CRC组、CRA组和HC组受试者外周血肿瘤标志物CEA和CA19-9的水平比较
CRC患者外周血中CEA和CA19-9水平均显著高于HC组(P < 0.05),而CRA组和HC组之间均差异无统计学意义(P>0.05,图 2)。
图 2.
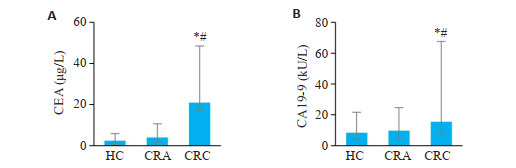
CRC组、CRA组和HC组受试者外周血肿瘤标志物CEA和CA19-9的水平
Serum levels of tumor markers CEA (A) and CA19-9 (B) in peripheral blood of subjects in CRC patients, CRA patients and healthy individuals. *P < 0.05 vs HC group; #P < 0.05 vs CRA group.
2.4. ROC曲线分析MMP-9、RBP4、CHI3L1和C3a联合对CRC、CRA的诊断效能
使用ROC曲线分析结果显示(图 3和表 2),MMP-9、RBP4、CHI3L1和C3a联合诊断CRC的灵敏度为91.4%,特异度为100.0%,AUC曲线下面积为96.8%;MMP-9、RBP4、CHI3L1和C3a联合诊断CRA的灵敏度为81.0%,特异度为89.5%,AUC曲线下面积为89.2%;MMP-9、RBP4、CHI3L1和C3a联合诊断CRC和CRA的灵敏度为83.9%,特异度为97.4%,AUC曲线下面积为93.7%。
图 3.
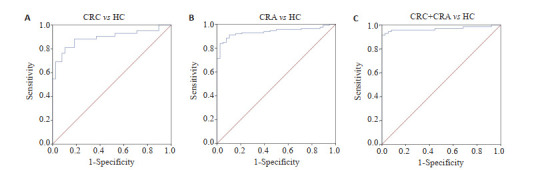
MMP-9、RBP4、CHI3L1和C3a诊断CRC和CRA的ROC曲线
ROC curves of MMP-9, RBP4, CHI3L1 and C3a for diagnosing CRC and CRA. A: ROC curves of MMP-9, RBP4, CHI3L1 and C3a for diagnosing CRC. B: ROC curves of MMP-9, RBP4, CHI3L1 and C3a for diagnosing CRA. C: ROC curves of MMP-9, RBP4, CHI3L1 and C3a for a combined diagnosis of CRC and CRA.
表 2.
MMP-9、RBP4、CHI3L1和C3a联合对CRA和CRC的诊断效能
Diagnostic efficacy of MMP-9, RBP4, CHI3L1 and C3a for CRA and CRC
| Factor | Sensitivity (%) | Specificity (%) | 95% CI | AUC (%) | P |
| HC: Health control; CRA: Colorectal adenomas; CRC: Colorectal cancer. | |||||
| CRC vs HC | 91.4 | 100.0 | 0.934-1.000 | 96.8 | < 0.001 |
| CRA vs HC | 81.0 | 89.5 | 0.816-0.967 | 89.2 | < 0.001 |
| CRC+CRA vs HC | 83.9 | 97.4 | 0.898-0.976 | 93.7 | < 0.001 |
2.5. ROC曲线分析CEA和CA19-9联合对CRC、CRA的诊断效能
如图 4和表 3,ROC曲线分析CEA和CA19-9联合对CRC、CRA的诊断效能结果显示,CEA和CA19-9联合诊断CRC的灵敏度为77.1%,特异度为97.4%,AUC曲线下面积为85.1%;CEA和CA19-9联合诊断CRA的灵敏度为50.0%,特异度为92.1%,AUC曲线下面积为69.2%;CEA和CA19-9联合诊断CRA和CRC的灵敏度为61.6%,特异度为100.0%,AUC曲线下面积为78.9%。
图 4.
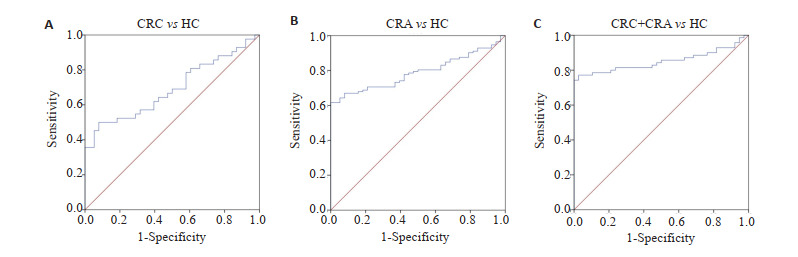
CEA和CA19-9诊断CRC和CRA的ROC曲线
ROC curves of CEA and CA19-9 for diagnosing CRC and CRA. A: ROC curves of CEA and CA19-9 for diagnosing CRC. B: ROC curves of CEA and CA19-9 for diagnosing CRA. C: ROC curves of CEA and CA19-9 for a combined diagnosis of CRC and CRA.
表 3.
CEA和CA19-9联合对CRA和CRC的诊断效能
Diagnostic efficacy of CEA and CA19-9 for CRA and CRC
| Factor | Sensitivity (%) | Specificity (%) | 95% CI | AUC (%) | P |
| HC: Health control; CRA: Colorectal adenomas; CRC: Colorectal cancer. | |||||
| CRC vs HC | 77.1 | 97.4 | 0.777-0.925 | 85.1 | < 0.001 |
| CRA vs HC | 50.0 | 92.1 | 0.575-0.808 | 69.2 | 0.003 |
| CRC+CRA vs HC | 61.6 | 100.0 | 0.720-0.859 | 78.9 | < 0.001 |
2.6. 粪便蛋白组合与外周血肿瘤标志物诊断效能比较
通过Z检验比较分析显示,粪便蛋白MMP-9、RBP4、CHI3L1和C3a联合诊断CRC,CRA,CRA和CRC的效能显著优于外周血肿瘤标志物CEA和CA19-9(表 4,P < 0.05)。
表 4.
粪便蛋白和外周血血清标志物对CRA和CRC诊断效能比较
Comparison of fecal protein and peripheral blood serum markers for diagnosis of CRA and CRC
| Factor | Z | P | AUC difference | 95% CI |
| HC: Health control; CRA: Colorectal adenomas; CRC: Colorectal cancer. | ||||
| CRC vs HC | 2.711 | 0.007 | 0.118 | 0.033-0.203 |
| CRA vs HC | 2.535 | 0.011 | 0.200 | 0.045-0.354 |
| CRC+CRA vs HC | 3.426 | 0.001 | 0.147 | 0.063-0.232 |
3. 讨论
本研究基于Luminex液相芯片技术检测发现人源性蛋白MMP-9、RBP4、CHI3L1、C3a在CRC和CRA粪便中显著升高,并证实MMP-9、RBP4、CHI3L1和C3a联合诊断CRC和CRA的效能优于传统外周血肿瘤标志物CEA和CA19-9,创新性的将高通量和高灵敏性的Luminex液相芯片技术应用于粪便蛋白检测,也为结直肠肿瘤早筛提供了一个新的诊断Panel。
正常肠黏膜上皮以原位凋亡的形式处于持续更新状态,极少产生脱落细胞[12]。CRC病程中,因恶变肠上皮细胞黏附性低、肿瘤生长损伤毗邻细胞等因素,粪便中的脱落细胞显著增加并携带肿瘤相关标志物[13]。早在1998年,Loktionov等[14]学者即在CRC患者粪便中分离出完整的肿瘤脱落细胞,诊断CRC的敏感性约为97%,特异性接近100%。但由于受到肠道内细菌、胆汁酸及细胞毒素等的分解作用,从粪便中分离充足的脱落细胞用于临床诊断仍较为困难。虽然粪便脱落细胞并不适用于CRC早筛,但粪便检测具有无创、取材便利、定位准确等优势,这为CRC的早期筛查提供了一个重要途径[15]。蛋白类分子是CRC相关脱落细胞在粪便中的重要存在形式,临床上粪便隐血试验即是检测粪便中的血红蛋白、转铁蛋白等[16]。近年来诸多学者投入到粪便蛋白对于CRC诊断价值的研究中,先后发现CRC患者粪便中CEA、抗胰蛋白酶、补体C3、乳铁蛋白、肿瘤M2型丙酮酸激酶等蛋白水平显著升高,并具有一定早期诊断价值[17]。我们既往使用宏蛋白质组学在CRC患者粪便中筛查出97个差异蛋白,进一步分析构建了CRC早筛诊断Panel,且开展了一项多中心研究,使用ELISA方法验证了该诊断Panel对CRC早筛的效能[7]。尽管有一些研究已经报告了粪便蛋白在CRC筛查中应用,但是所检测人群来自美国和荷兰,均存在地域、饮食习惯和种族的差异[18, 19]。而本研究中,我们筛查了本医院所在皖北地区结直肠肿瘤患者粪便中蛋白的差异水平,所得结果存在地域和种族差异。
在本研究中,我们创新性的使用Luminex技术验证了4种(MMP-9、RBP4、CHI3L1和C3a)粪便蛋白在CRC和CRA粪便中水平显著高于对照组,且均与CRC进展密切相关,这提示MMP-9、RBP4、CHI3L1和C3a可能用于临床CRC的早期筛查,并具有鉴别CRC和CRA的临床价值。进一步,我们使用ROC工作曲线分析了四种蛋白联合对CRC,CRA的鉴别诊断价值,证实粪便蛋白MMP-9、RBP4、CHI3L1和C3a联合诊断CRC、CRA以及CRA和CRC的效能均显著优于外周血肿瘤标志物CEA和CA19-9。
Luminex液相芯片技术是继人类基因组计划完成后逐渐发展起来的新型生物分子检测技术。近十年来,Luminex公司对液相芯片技术不断推陈出新,使其与传统检验方法相比具有高通量、高灵敏度、样本量小、成本低,适用于多样本和多指标分析等优点。相对于质谱检测技术,Luminex可根据检测目的灵活设计检测谱且适用于多种微量标本的检测,包括泪水、脑脊液、蛋白提取物等[20],在对样本的要求上优于质谱分析技术。目前,Luminex技术被诸多学者尝试用于肿瘤的早期诊断中。Vrzalova等[21]通过其检测血液中催乳素、骨桥蛋白、胰岛素样生长因子Ò等标记物用以早期诊断卵巢癌。Arellano等检测人唾液中IL-8、IL-1β等细胞因子诊断口腔癌[22]。Nolen等[23]应用液相芯片技术监测人血液中细胞因子、激素、黏附分子等55种生物标记监测进展期乳腺癌对新辅助化疗方法的治疗效果。此外,Luminex对多种蛋白质分子的综合监测和评价,比单纯监测单个或多个分子更能反映疾病发生发展的整体变化。本研究中,我们创新性的将Luminex技术应用于人类粪便提取上清液中4种蛋白质分子的检测,发现该技术较ELISA和免疫印记等蛋白质定量检测技术具有操作步骤简单,成本低,灵敏度高等特点,提示Luminex有望成为CRC粪便蛋白肿瘤标志物早筛的理想检测技术。
虽然粪便肿瘤标志物检测显示出对于CRC的早筛潜力,但当前的研究多聚焦于单个粪便肿瘤标志物对于CRC的诊断,但肿瘤的发生和发展过程中伴有复杂的细胞、代谢等生物学行为并可能在粪便中出现多种异常蛋白均有可能提示肿瘤的发生或进展[24]。因此,单一指标的检测显然不能满足CRC早筛对于高通量的要求。本研究中通过对蛋白质组学数据的分析和文献回顾,确定了四种差异粪便蛋白并用于CRC的诊断评估。MMP-9是参与降解细胞外基质的关键酶,可使肿瘤细胞之间的黏附减弱,破坏阻止癌细胞侵袭和转移的屏障,从而为癌细胞向远处扩散和转移提供足够的空间,促进肿瘤的生长、浸润和转移[25]。有研究显示,尿液和血液中MMP-9联合CEA和CA19-9诊断CRC的性能显著高于CEA和CA19-9,可作为潜在的CRC辅助诊断标志物[26]。RBP4属于脂质卡林家族,是视黄醇的特异性载体。Matthew等的研究发现血清高水平RBP4与CRA的发生显著相关;另外血清RBP4通过与受体STRA6结合促进CRC异种移植小鼠模型中肿瘤的发生[27, 28];更重要的是,Komor等[29]的研究发现粪便中RBP4蛋白可作为无创检测进展高风险结直肠腺瘤的生物标志物,这与我们的研究相一致。CHI3L1又称为YKL-40,是哺乳动物几丁质酶蛋白家族的成员,虽然其生物学功能并不完全清楚,但是CHI3L1在细胞外基质重塑、降解以及细胞增殖和分化过程中扮演重要角色。在结肠癌组织中CHI3L1表达水平增高,与慢性炎症、癌症发生和发展密切相关[30],另有研究证明CHI3L1血清水平与结直肠癌合并其他实体瘤患者的不良临床结局有关,可作为CRC预后不良的潜在标志物[31, 32]。C3a是C3活化后水解的产物,Habermann等[33]利用表面增强激光解析离子化-飞行时间质谱技术和ELISA技术检测发现,CRC患者血清C3a水平高于正常人群,且其诊断CRC的敏感性为96.8%,特异性为96.2%,CRA患者有86.1%的血清水平升高,证明提示C3a在CRC和CRA患者血清中的水平明显高于健康人,表明C3a水平的量化可以优化现有的结直肠癌筛查方法。既往研究多关注MMP-9、RBP4、CHI3L1和C3a在患者外周中的差异水平,但是其在粪便中的表达水平及其与结直肠肿瘤进展的关系尚未不清楚。本研究使用Luminex技术在粪便中检测到了MMP-9、RBP4、CHI3L1和C3a,并证实这四种蛋白质在CRC、CRA和HC粪便中存在差异,为CRC无创筛查方法的研发提供了可能。
本研究的创新性和临床价值如下:人体粪便隐藏着大量疾病和健康信息,本研究借助于Luminex技术高通量检测技术,从粪便脱落人源性蛋白对CRC的诊断价值分析着手,不仅有望为CRC的临床诊断提供新方法,且有助于从排泄物角度拓展对CRC的疾病认识。本研究的局限性如下:尽管前期我们基于ELISA技术开展了多中心研究验证了粪便蛋白对CRC的诊断价值,但是本研究使用Luminex检测技术仅纳入单中心150例受试者粪便,可能存在抽样误差,后续仍需要扩大样本数并进行多中心验证;另外,CRC患者粪便中还存在多种差异粪便蛋白,能否具有诊断潜力仍需要进一步验证。
综上所述,Luminex液相芯片检测粪便人源性蛋白RBP4、MMP-9、CHI3L1、C3a对结直肠肿瘤早期诊断具有较高的临床推广价值,与此同时Luminex液相芯片检测技术将在CRC筛查和病情监测中有着广阔的应用前景。
Biography
李静,博士,副教授/副主任技师,E-mail: sdlj13409@163.com
Funding Statement
安徽省高校协同创新项目(GXXT-2020-020);安徽省卫生健康科研项目(AHWJ2022a019,AHWJ2022b088);安徽省教育厅优秀青年基金项目(2022AH030138);蚌埠市科技计划项目(2022hm01)
Contributor Information
李 静 (Jing LI), Email: sdlj13409@163.com.
胡 建国 (Jianguo HU), Email: jghu9200@bbmc.edu.cn.
References
- 1.Ferlay J, Colombet M, Soerjomataram I, et al. Estimating the global cancer incidence and mortality in 2018: GLOBOCAN sources and methods. Int J Cancer. 2019;144(8):1941–53. doi: 10.1002/ijc.31937. [DOI] [PubMed] [Google Scholar]
- 2.Bray F, Ferlay J, Soerjomataram I, et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA Cancer J Clin. 2018;68(6):394–424. doi: 10.3322/caac.21492. [DOI] [PubMed] [Google Scholar]
- 3.Chen ZS, Song WQ, Shu XO, et al. Novel insights into genetic susceptibility for colorectal cancer from transcriptome-wide association and functional investigation. J Natl Cancer Inst. 2023:djad178. doi: 10.1093/jnci/djad178. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Bhardwaj M, Weigl K, Tikk K, et al. Multiplex quantitation of 270 plasma protein markers to identify a signature for early detection of colorectal cancer. Eur J Cancer. 2020;127:30–40. doi: 10.1016/j.ejca.2019.11.021. [DOI] [PubMed] [Google Scholar]
- 5.Baxter BA, Parker KD, Nosler MJ, et al. Metabolite profile comparisons between ascending and descending colon tissue in healthy adults. World J Gastroenterol. 2020;26(3):335–52. doi: 10.3748/wjg.v26.i3.335. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Aghagolzadeh P, Radpour R. New trends in molecular and cellular biomarker discovery for colorectal cancer. World J Gastroenterol. 2016;22(25):5678–93. doi: 10.3748/wjg.v22.i25.5678. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Zhang H, Zuo L, Li J, et al. Construction of a fecal immune-related protein-based biomarker panel for colorectal cancer diagnosis: a multicenter study. Front Immunol. 2023;14:1126217. doi: 10.3389/fimmu.2023.1126217. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Graham H, Chandler DJ, Dunbar SA. The genesis and evolution of bead-based multiplexing. Methods. 2019;158:2–11. doi: 10.1016/j.ymeth.2019.01.007. [DOI] [PubMed] [Google Scholar]
- 9.Cook DB, McLucas BC, Montoya LA, et al. Multiplexing protein and gene level measurements on a single Luminex platform. Methods. 2019;158:27–32. doi: 10.1016/j.ymeth.2019.01.018. [DOI] [PubMed] [Google Scholar]
- 10.Luna-Marco C, de Marañon AM, Hermo-Argibay A, et al. Effects of GLP-1 receptor agonists on mitochondrial function, inflammatory markers and leukocyte-endothelium interactions in type 2 diabetes. Redox Biol. 2023;66:102849. doi: 10.1016/j.redox.2023.102849. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Kats L, Goldman Y, Kahn A. Automatic detection of image sharpening in maxillofacial radiology. BMC Oral Heath. 2021;21(1):411. doi: 10.1186/s12903-021-01777-9. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Yu L, Zhang MM, Hou JG. Molecular and cellular pathways in colorectal cancer: apoptosis, autophagy and inflammation as key players. Scand J Gastroenterol. 2022;57(11):1279–90. doi: 10.1080/00365521.2022.2088247. [DOI] [PubMed] [Google Scholar]
- 13.金 红, 庞 丽莹, 李 华洋, et al. 联合检测粪便中ITGA4和SFRP2基因甲基化在大肠肿瘤诊断和预后中的价值. 南方医科大学学报. 2021;41(6):891–7. [Google Scholar]
- 14.Loktionov A, O'Neill IK, Silvester KR, et al. Quantitation of DNA from exfoliated colonocytes isolated from human stool surface as a novel noninvasive screening test for colorectal cancer. Clin Cancer Res. 1998;4(2):337–42. [PubMed] [Google Scholar]
- 15.赵 丽中, 马 东旺, 张 锡朋. 国内外大肠癌筛查现状及无创筛查方法进展. 天津医药. 2019;47(6):589–93. [Google Scholar]
- 16.Jayasinghe M, Prathiraja O, Caldera D, et al. Colon cancer screening methods: 2023 update. Cureus. 2023;15(4):e37509. doi: 10.7759/cureus.37509. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Niedermaier T, Balavarca Y, Brenner H. Stage-specific sensitivity of fecal immunochemical tests for detecting colorectal cancer: systematic review and meta-analysis. Am J Gastroenterol. 2020;115(1):56–69. doi: 10.14309/ajg.0000000000000465. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Li H, Vanarsa K, Zhang T, et al. Comprehensive aptamer-based screen of 1317 proteins uncovers improved stool protein markers of colorectal cancer. J Gastroenterol. 2021;56(7):659–72. doi: 10.1007/s00535-021-01795-y. [DOI] [PubMed] [Google Scholar]
- 19.de Klaver W, Wisse PHA, van Wifferen F, et al. Clinical validation of a multitarget fecal immunochemical test for colorectal cancer screening: a diagnostic test accuracy study. Ann Intern Med. 2021;174(9):1224–31. doi: 10.7326/M20-8270. [DOI] [PubMed] [Google Scholar]
- 20.Koel-Simmelink MJA, Vennegoor A, Killestein J, et al. The impact of pre-analytical variables on the stability of neurofilament proteins in CSF, determined by a novel validated SinglePlex Luminex assay and ELISA. J Immunol Meth. 2014;402(1/2):43–9. doi: 10.1016/j.jim.2013.11.008. [DOI] [PubMed] [Google Scholar]
- 21.Vrzalova J, Prazakova M, Novotny Z, et al. Test of ovarian cancer multiplex xMAP technology panel. Anticancer Res. 2009;29(2):573–6. [PubMed] [Google Scholar]
- 22.Arellano-Garcia ME, Hu S, Wang J, et al. Multiplexed immunobead-based assay for detection of oral cancer protein biomarkers in saliva. Oral Dis. 2008;14(8):705–12. doi: 10.1111/j.1601-0825.2008.01488.x. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.Nolen BM, Marks JR, Ta'san S, et al. Serum biomarker profiles and response to neoadjuvant chemotherapy for locally advanced breast cancer. Breast Cancer Res. 2008;10(3):1–9. doi: 10.1186/bcr2096. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.Sveen A, Kopetz S, Lothe RA. Biomarker-guided therapy for colorectal cancer: strength in complexity. Nat Rev Clin Oncol. 2020;17(1):11–32. doi: 10.1038/s41571-019-0241-1. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.Barabás L, Hritz I, István G, et al. The behavior of MMP-2, MMP-7, MMP-9, and their inhibitors TIMP-1 and TIMP-2 in adenoma-colorectal cancer sequence. Dig Dis. 2021;39(3):217–24. doi: 10.1159/000511765. [DOI] [PubMed] [Google Scholar]
- 26.魏 文彬, 张 朋军. CEA、CA19-9和基质金属蛋白酶9对于早期结直肠癌诊断价值的评价. 标记免疫分析与临床. 2023;30(2):195–9. [Google Scholar]
- 27.Karunanithi S, Levi L, DeVecchio J, et al. RBP4-STRA6 pathway drives cancer stem cell maintenance and mediates high-fat diet-induced colon carcinogenesis. Stem Cell Rep. 2017;9(2):438–50. doi: 10.1016/j.stemcr.2017.06.002. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 28.Fei WQ, Chen L, Chen JX, et al. RBP4 and THBS2 are serum biomarkers for diagnosis of colorectal cancer. Oncotarget. 2017;8(54):92254–64. doi: 10.18632/oncotarget.21173. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.Komor MA, Bosch LJ, Coupé VM, et al. Proteins in stool as biomarkers for non-invasive detection of colorectal adenomas with high risk of progression. J Pathol. 2020;250(3):288–98. doi: 10.1002/path.5369. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 30.Lehtomäki K, Mustonen H, Kellokumpu-Lehtinen PL, et al. Lead time and prognostic role of serum CEA, CA19-9, IL-6, CRP, and YKL-40 after adjuvant chemotherapy in colorectal cancer. Cancers. 2021;13(15):3892. doi: 10.3390/cancers13153892. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 31.Abd El-Fattah AA, Sadik NAH, Shaker OG, et al. Single nucleotide polymorphism in SMAD7 and CHI3L1 and colorectal cancer risk. Mediators Inflamm. 2018;2018:9853192. doi: 10.1155/2018/9853192. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 32.Cintin C, Johansen JS, Christensen IJ, et al. Serum YKL-40 and colorectal cancer. Br J Cancer. 1999;79(9/10):1494–9. doi: 10.1038/sj.bjc.6690238. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 33.Habermann JK, Roblick UJ, Luke BT, et al. Increased serum levels of complement C3a anaphylatoxin indicate the presence of colorectal tumors. Gastroenterology. 2006;131(4):1020–9. doi: 10.1053/j.gastro.2006.07.011. [DOI] [PMC free article] [PubMed] [Google Scholar]


