Resumen
Introducción.
El comportamiento de la resistencia antimicrobiana es fundamental en el mejoramiento y ajuste de los programas de optimización de uso de antimicrobianos, la implementación de las guías terapéuticas y las precauciones que limitan la transmisión cruzada de bacterias resistentes entre pacientes. Desde el inicio del 2020, la pandemia del SARS-CoV-2 desafió profundamente al sistema de salud y, según algunos reportes, aumentó las tasas de resistencia antimicrobiana.
Objetivo.
Describir el comportamiento de la resistencia antimicrobiana en los microrganismos más frecuentes en veinte hospitales colombianos durante el periodo 2018-2021.
Materiales y métodos.
Se trata de un estudio descriptivo basado en la información microbiológica reportada por veinte instituciones de salud de nivel III y IV, entre enero de 2018 y diciembre de 2021, en doce ciudades de Colombia, las cuales hacen parte del "Grupo para el estudio de la resistencia nosocomial en Colombia", liderado por la Universidad El Bosque. La identificación de género y especie de los microorganismos más frecuentes, junto con su perfil de resistencia frente a antibióticos marcadores, se determinaron mediante el análisis de los datos vía WHONET.
Resultados.
En general, los 10 microorganismos más frecuentes analizados a lo largo de los 4 años no presentaron cambios estadísticamente significativos en sus perfiles de resistencia durante los cuatro años del periodo evaluado, de 2018 a 2021. En contraste, Pseudomonas aeruginosa aumentó su resistencia frente a piperacilinatazobactam y carbapenémicos, lo cual fue estadísticamente significativo.
Conclusiones.
Los cambios en la resistencia antimicrobiana en estos años no han sido estadísticamente significativos, excepto para P. aeruginosa, bacteria que mostró un incremento en las tasas de resistencia a piperacilina-tazobactam y carbapenémicos.
Palabras clave: farmacorresistencia microbiana, monitoreo epidemiológico, Colombia
Abstract
Introduction.
Antimicrobial resistance surveillance is a fundamental tool for the development, improvement, and adjustment of antimicrobial stewardship programs, therapeutic guidelines, and universal precautions to limit the cross-transmission of resistant bacteria between patients. Since the beginning of 2020, the SARS-CoV-2 pandemic profoundly challenged the health system and, according to some reports, increased the rates of antimicrobial resistance.
Objective.
To describe the behavior of antimicrobial resistance of the most frequent bacterial pathogens in twenty Colombian hospitals from January 2018 to December 2021.
Materials and methods.
We conducted a descriptive study based on the microbiological information recorded from January 2018 to December 2021 in twenty levels III and IV health institutions in twelve Colombian cities. We identified the species of the ten most frequent bacteria along with their resistance profile to the antibiotic markers after analyzing the data through WHONET.
Results.
We found no statistically significant changes in most pathogens' resistance profiles from January 2018 to December 2021. Only Pseudomonas aeruginosa had a statistically significant increase in its resistance profile, particularly to piperacillin/ tazobactam and carbapenems.
Conclusions.
The changes in antimicrobial resistance in these four years were not statistically significant except for P. aeruginosa to piperacillin/tazobactam and carbapenems.
Keywords: Drug resistance, microbial; epidemiological monitoring; Colombia
La vigilancia epidemiológica activa, en conjunto con actividades de prevención y control de infecciones, han demostrado ser medidas muy efectivas en la reducción y contención de la resistencia antimicrobiana 1,2. Por otro lado, los programas de optimización de antimicrobianos basados en datos de sensibilidad y resistencia microbianas son fundamentales para implementar las guías de antimicrobianos a nivel hospitalario, junto con la vigilancia y el seguimiento de los patrones de resistencia a nivel local, nacional, regional y global; además, permiten analizar el comportamiento de los agentes patógenos multirresistentes, así como ejercer una vigilancia microbiológica eficaz 3,4. Por todo lo anterior, el tener sistemas que garanticen una vigilancia de la resistencia antimicrobiana es esencial para implementar los programas de optimización de uso de antimicrobianos en cualquier hospital 4.
El uso indiscriminado de los antibióticos ha sido una de las causas principales de la diseminación y el aumento de la resistencia antimicrobiana a nivel mundial 5. Programas como el SENTRY Antimicrobial Surveillance y el Study for Monitoring Antimicrobial Resistance Trends (SMART) han reportado un aumento global en la resistencia antimicrobiana en los últimos años, especialmente para el grupo de agentes patógenos conocidos como ESKAPE (Enterococcus faecium, Staphylococcus aureus, Klebsiella pneumoniae, Acinetobacter baumannii, Pseudomonas aeruginosa y Enterobacter sp.) 6,7. Asimismo, Cantón et al. reportaron un aumento en la resistencia a las cefalosporinas de tercera generación en Escherichia coli, del 4,6 al 10,4 % entre el 2005 y el 2008, principalmente debido a la expansión del grupo clonal ST131, portador de β-lactamasas de espectro extendido (BLEE) CTX-M-15 8. Para el caso de K. pneumoniae, también se encontró un aumento de la resistencia a dichas cefalosporinas: del 21,7 % en el periodo 1997-2000 al 36,1 % en el periodo 2013-2016 8.
Por otro lado, se ha reportado un incremento en la resistencia a los carbapenémicos, especialmente en K. pneumoniae, la cual pasó del 0,6 en 1997-2000 al 2,9 % en 2013-2016, por carbapenemasas diferentes a KPC. Esta tasa es más notoria en Latinoamérica, ya que se ha reportado un incremento del 0,8 al 6,4 % durante el mismo periodo 6. Finalmente, para P. aeruginosa, se ha reportado un incremento de aislamientos multirresistentes, especialmente en Latinoamérica, donde pasó al 41,1 % en los últimos 20 años 9.
El aumento reciente de la resistencia antimicrobiana se ha asociado con la aparición de la COVID-19, especialmente en unidades de cuidados intensivos 10,11. En varios estudios se informó un aumento de la resistencia antimicrobiana durante los primeros 18 meses de la pandemia, siendo A. baumannii resistente a los carbapenémicos, S. aureus resistente a la meticilina (SARM), Enterococcus spp. resistente a la vancomicina, y K. pneumoniae y P. aeruginosa-MDR fueron los principales responsables de este incremento 8,12,13.
Durante la pandemia del SARS-CoV-2, las instituciones de salud fueron sometidas a una gran presión, demandándose grandes recursos del sistema de salud y obligando a muchas a reducir o, incluso, desarticular los planes o iniciativas de programas de optimización de uso de antimicrobianos 14. El personal de salud tuvo que ser reubicado para cubrir la emergencia, se aumentaron rápidamente las camas de cuidados intensivos, las áreas hospitalarias se dividieron en áreas COVID y áreas no-COVID, hubo escasez de personal entrenado e, igualmente, se reportó un aumento en el uso de antibióticos 15.
En paralelo con la infección por el SARS-CoV-2, se reportaron brotes por microorganismos multirresistentes, en gran parte, debido a la poca observancia de las normas de prevención y control de infecciones 16-18. Si bien los factores mencionados anteriormente contribuyeron al aumento de la resistencia antimicrobiana, también se observó que la disminución del número de procedimientos no esenciales, el distanciamiento social y el aumento en las medidas de limpieza y desinfección, así como un aumento general de la higiene de las manos, pudieron contribuir a su disminución 14.
Superada la fase crítica de la pandemia, es fundamental evaluar el comportamiento epidemiológico de la resistencia antimicrobiana en los principales agentes patógenos bacterianos y determinar si ocurrieron cambios importantes en sus patrones de resistencia a los antimicrobianos con respecto al periodo prepandémico. En este estudio, se describen la epidemiología bacteriana y el perfil de resistencia en instituciones de salud de Colombia pertenecientes al "Grupo para el estudio de la resistencia nosocomial en Colombia", durante los años 2018 a 2021.
Materiales y métodos
Diseño del estudio
Se trata de un estudio descriptivo realizado con la información de los aislamientos recopilados vía WHONET (versión 5.6) 19, en veinte instituciones de salud de nivel III y IV de doce ciudades colombianas, pertenecientes al "Grupo para el estudio de la resistencia nosocomial en Colombia" durante el periodo de enero de 2018 a diciembre de 2021.
Se hizo una búsqueda por frecuencia de aislamientos, tipos de muestras clínicas incidentes en estos aislamientos y perfiles de resistencia para los agentes patógenos más frecuentes, en particular, E. coli, K. pneumoniae, P. aeruginosa y S. aureus, frente a antibióticos seleccionados utilizados como marcadores de resistencia; estos últimos incluyeron ceftriaxona, cefotaxime, ceftazidime, cefepime, piperacilina-tazobactam, ertapenem, imipenem, meropenem, amikacina, ciprofloxacina, oxacilina y clindamicina.
Este análisis se llevó a cabo en todos los aislamientos reportados en las salas de hospitalización general y en unidades de cuidados intensivos de adultos.
Población de estudio
Se seleccionaron veinte instituciones de salud de nivel III y IV en doce ciudades de Colombia que contaran con información completa de los cuatro años analizados (2018-2021). La información sobre todos los aislamientos de cada institución se obtuvo directamente de los sistemas automáticos de los laboratorios de microbiología, se utilizó para evaluar la sensibilidad antimicrobiana y fue recopilada por medio del software epidemiológico WHONET (versión 5.6) de la Organización Mundial de la Salud; posteriormente, se convirtió mediante el sistema de conversión de datos BacLink (versión 2.0) 19.
Estos datos fueron analizados por el Grupo de Resistencia Antimicrobiana y Epidemiologia Hospitalaria de la Universidad El Bosque, en Bogotá, Colombia. La información de los diferentes laboratorios de microbiología de las instituciones de salud se estandarizó mediante diccionarios para generar conglomerados según el tipo de muestra clínica. De igual manera, los puntos de corte para los diferentes antibióticos se establecieron según las directrices de la CLSI 2021 20.
Análisis de la información
Se tomó en cuenta el primer aislamiento de cada paciente de las salas de hospitalización general y de las unidades de cuidados intensivos, según la recomendación para realizar este tipo de análisis 21. Se determinaron los porcentajes de frecuencia de aislamientos, tipos de muestra más frecuentes y resistencia por año. Se utilizó la prueba estadística no paramétrica de Kruskal-Wallis para hacer comparaciones de grupos independientes y determinar diferencias en las proporciones de resistencias por año. Los valores de p menores de 0,05 se consideraron estadísticamente significativos.
Resultados
Aislamientos de salas de hospitalización general
Durante el periodo de análisis, se reportaron 61.184 aislamientos. Los diez microorganismos más frecuentes fueron, en orden descendente: E. coli, K. pneumoniae, S. aureus, P. aeruginosa, Enterococcus faecalis, Proteus mirabilis, S. epidermidis, Enterobacter cloacae complex, Candida albicans y Serratia marcescens (cuadro 1). Estos microorganismos representaron el 77 % de todos los aislamientos en los cuatro periodos.
Cuadro 1. Distribución y tendencia de la incidencia de los 10 microorganismos más frecuentes en salas generales en 20 instituciones de nivel III y IV durante el periodo 2018-2021 en Colombia (N=61.184).
| Microorganismo | 2018 | 2019 | 2020 | 2021 | ||||
|---|---|---|---|---|---|---|---|---|
| n | (%) | n | (%) | n | (%) | n | (%) | |
| Escherichia coli | 4084 | 26 | 4.568 | 28 | 3.749 | 26 | 3.814 | 26 |
| Klebsiella pneumoniae | 1.791 | 11 | 1932 | 12 | 1.829 | 13 | 1.845 | 13 |
| Staphylococcus aureus | 1.719 | 11 | 1.655 | 10 | 1.398 | 10 | 1.421 | 10 |
| Pseudomonas aeruginosa | 1.258 | 8 | 1.377 | 8 | 1.128 | 8 | 1.333 | 9 |
| Enterococcus faecalis | 831 | 5 | 836 | 5 | 780 | 5 | 711 | 5 |
| Proteus mirabilis | 637 | 4 | 824 | 5 | 693 | 5 | 706 | 5 |
| Staphylococcus epidermidis | 610 | 4 | 600 | 4 | 612 | 4 | 534 | 4 |
| Enterobacter cloacae complex | 476 | 3 | 446 | 3 | 500 | 3 | 492 | 3 |
| Candida albicans | 354 | 2 | 286 | 2 | 318 | 2 | 256 | 2 |
| Serratia marcescens | 310 | 2 | 310 | 2 | 284 | 2 | 326 | 2 |
| Otros microorganismos | 3.608 | 23 | 3.484 | 21 | 3.258 | 22 | 3.201 | 22 |
| Total | 15.678 | 16.318 | 14.549 | 14.639 | ||||
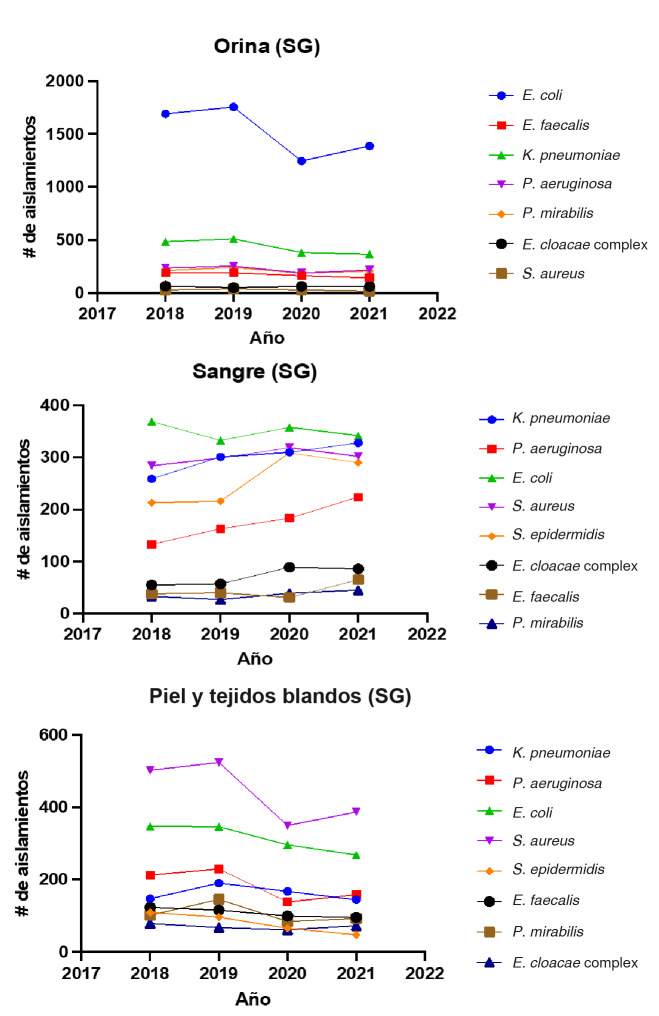
Durante el periodo 2018-2019, las muestras más frecuentes fueron las de orina, seguidas por las de piel y tejidos blandos, y en tercer lugar, las de sangre. Durante 2020 y 2021, la orina siguió siendo la muestra más frecuente, pero la sangre pasó a ocupar el segundo lugar y, la piel y los tejidos blandos, el tercero. En el cuadro 2 se muestran las muestras más frecuentes en los cuatro años del estudio. En las muestras de orina, E. coli se aisló en el 56 % de los casos, mientras que, para piel y tejidos blandos, S. aureus representó el 29 % de los aislamientos, seguido por E. coli con el 21 %. En sangre, E. coli representó el 22 % y S. aureus el 19 %. Llama la atención que S. epidermidis se aisló en el 16 % de las muestras de sangre, lo cual podría estar asociado con contaminación en la toma de la muestra.
Cuadro 2. Distribución por tipo de muestra más frecuente en salas de hospitalización general en 20 instituciones de nivel III y IV, durante el periodo 2018-2021 en Colombia (N=61.184).
| Muestra | 2018 | 2019 | 2020 | 2021 | ||||
|---|---|---|---|---|---|---|---|---|
| n | (%) | n | (%) | n | (%) | n | (%) | |
| Orina | 3.003 | 19,2 | 3.094 | 19,0 | 2.386 | 16,4 | 2.424 | 16,6 |
| Piel y tejidos blandos | 1.653 | 10,5 | 1.756 | 10,8 | 1.289 | 8,9 | 1.310 | 8,9 |
| Sangre | 1.415 | 9,0 | 1.490 | 9,1 | 1.700 | 11,7 | 1.744 | 11,9 |
| Total | 15.678 | 16.318 | 14.549 | 14.639 | ||||
En la figura 1 se muestran las tendencias del número de aislamientos bacterianos por los principales tipos de muestra aislados en salas generales durante los años 2018 a 2021.
Figura 1. Tendencia del número de aislamientos bacterianos según los principales tipos de muestra aislados en salas de hospitalización general, durante los años 2018 a 2021.
SG: salas de hospitalización general
Aislamientos de la unidad de cuidados intensivos
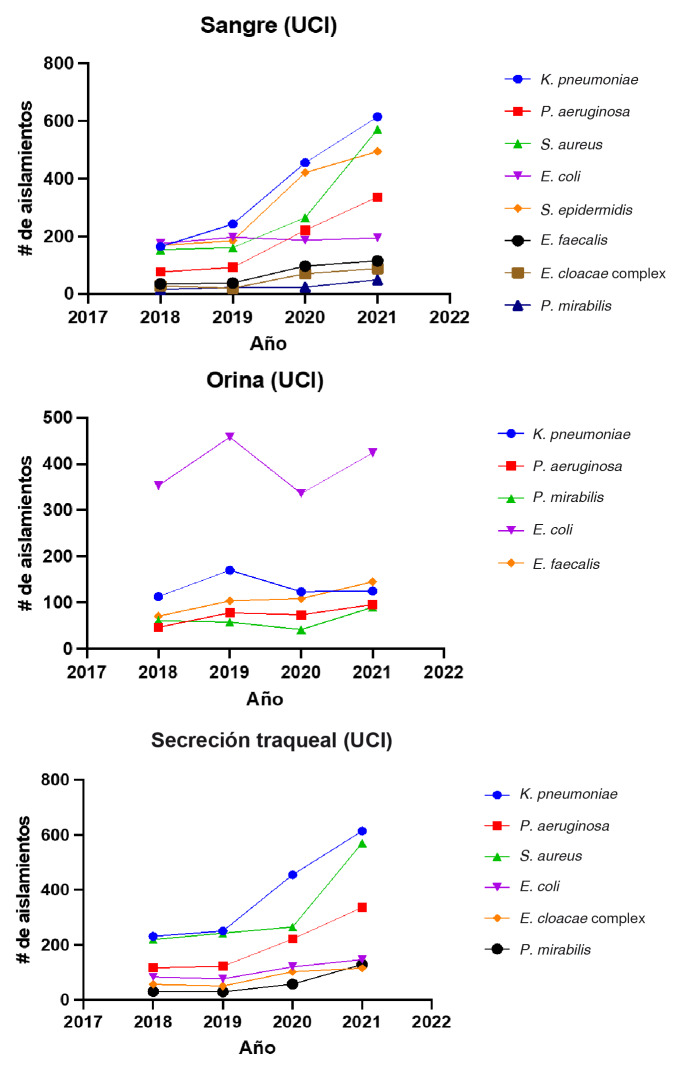
Durante el periodo de análisis de los datos (2018-2021), se reportaron 32.523 aislamientos en la unidad de cuidados intensivos de adultos. Los diez microorganismos más frecuentes en los cuatro años fueron, en orden descendente: E. coli, K. pneumoniae, S. aureus, P. aeruginosa, S. epidermidis, E. faecalis, C. albicans, P. mirabilis, E. cloacae complex y S. marcescens (cuadro 3). Es de particular interés que se observó un incremento en el número de microorganismos aislados durante el periodo 2020-2021, especialmente de K. pneumoniae, que llegó a ser el primer microorganismo aislado en las unidades de cuidados intensivos por encima de E. coli, el cual ocupaba el primer lugar entre 2018 y 2019. En las unidades de cuidados intensivos de adultos, los diez microorganismos más frecuentemente aislados correspondieron al 74 % de todos los aislamientos en los cuatro periodos; las muestras más frecuentes durante el 2018 fueron, en orden descendente, sangre, secreción traqueal y orina. En el período 2020-2021, se incrementó considerablemente el número de cultivos positivos, y las secreciones traqueales pasaron a ser la muestra más frecuente (cuadro 4).
Cuadro 3. Distribución y tendencia de la incidencia de los 10 microorganismos más frecuentes en unidades de cuidados intensivos en 20 instituciones de nivel III y IV, durante el periodo 2018-021 en Colombia (N=32.523).
| Microorganismo | 2018 | 2019 | 2020 | 2021 | ||||
|---|---|---|---|---|---|---|---|---|
| n | (%) | n | (%) | n | (%) | n | (%) | |
| Escherichia coli | 989 | 17 | 1.193 | 17 | 1.097 | 13 | 1.320 | 12 |
| Klebsiella pneumoniae | 897 | 15 | 1.151 | 16 | 1.574 | 18 | 1.993 | 18 |
| Staphylococcus aureus | 616 | 10 | 684 | 10 | 720 | 8 | 1.025 | 9 |
| Pseudomonas aeruginosa | 500 | 8 | 656 | 9 | 792 | 9 | 1.193 | 11 |
| Staphylococcus epidermidis | 291 | 5 | 321 | 5 | 565 | 7 | 636 | 6 |
| Enterococcus faecalis | 255 | 4 | 268 | 4 | 376 | 4 | 495 | 5 |
| Candida albicans | 254 | 4 | 286 | 4 | 417 | 5 | 408 | 4 |
| Proteus mirabilis | 198 | 3 | 240 | 3 | 278 | 3 | 417 | 4 |
| Enterobacter cloacae complex | 186 | 3 | 171 | 2 | 306 | 4 | 377 | 3 |
| Serratia marcescens | 184 | 3 | 188 | 3 | 264 | 3 | 331 | 3 |
| Otros microorganismos | 1.554 | 26 | 1.841 | 26 | 2.257 | 26 | 2.759 | 25 |
| Total | 5.924 | 6.999 | 8.646 | 10.954 | ||||
Cuadro 4. Distribución por tipo de muestra más frecuente en unidades de cuidados intensivos en 20 instituciones de nivel III y IV, durante el periodo 20182021 en Colombia (N=32.523).
| Muestra | 2018 | 2019 | 2020 | 2021 | ||||
|---|---|---|---|---|---|---|---|---|
| n | (%) | n | (%) | n | (%) | n | (%) | |
| Sangre | 863 | 22,6 | 1.043 | 14,9 | 1.572 | 18,2 | 1.890 | 17,2 |
| Traqueal | 821 | 21,5 | 823 | 11,7 | 1.317 | 15,2 | 2.048 | 18,7 |
| Orina | 750 | 19,6 | 1.004 | 14,3 | 845 | 9,8 | 1.056 | 9,6 |
| Total | 5.924 | 6.999 | 8.646 | 10.954 | ||||
Los microorganismos frecuentes en sangre fueron, en orden descendente: E. coli, K. pneumoniae, S. aureus y S. epidermidis; mientras que, en orina, E. coli se aisló en más del 40 % de los cultivos. En secreciones traqueales, el microorganismo aislado con mayor frecuencia fue K. pneumoniae, seguido por S. aureus y P. aeruginosa. En la figura 2 se muestran las tendencias del número de aislamientos bacterianos según los principales tipos de muestra aislados en la unidad de cuidados intensivos durante los años 2018 a 2021.
Figura 2. Tendencia del número de aislamientos bacterianos según los principales tipos de muestra aislados en unidades de cuidados intensivos adultos, durante los años 2018 a 2021.
UCI: unidad de cuidados intensivos
Es relevante que, en el año 2020, se observó un aumento en el número de aislamientos de K. pneumoniae en secreción traqueal: pasó de 231 aislamientos a 614 entre los años 2018 y 2020. Asimismo, se observó un aumento de S. aureus en el mismo tipo de muestra, pues en el 2021, su número fue de 571 aislamientos frente a 219 en el 2018. En sangre, S. epidermidis tuvo un incremento al pasar de 421 a 494 aislamientos en el 2020-2021 (figura 2).
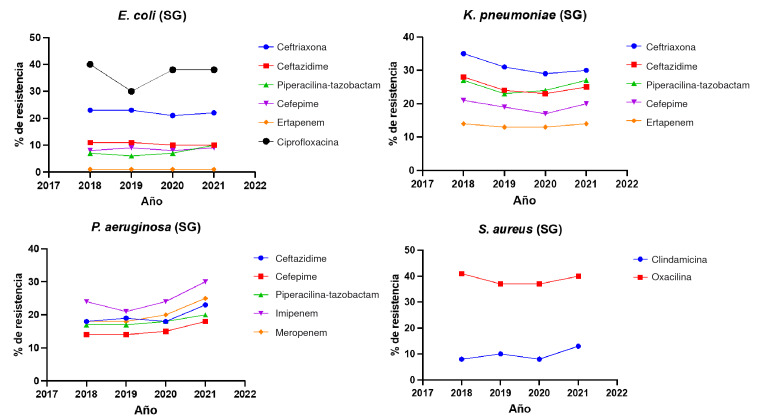
Resistencia de Escherichia coli y Klebsiella pneumoniae en salas de hospitalización general
La resistencia de E. coli ante las cefalosporinas de tercera generación se mantuvo estable durante los cuatro años. La resistencia a la ceftriaxona fue del 22,2 % (21-23 %), como media, similar al comportamiento estable ante el ceftazidime, con una media del 10,5 % (10-11 %). En contraste, la mayor resistencia se presentó ante la ciprofloxacina, con el 36,5 % (30-40 %) (cuadro 5) (figura 3). Por otra parte, la resistencia de K. pneumoniae ante la ceftriaxona y el ceftazidime, disminuyó levemente entre el 2018 y el 2021, sin lograr la significancia estadística. En el caso de la ceftriaxona, fue del 31,2 % (29-35 %) y, con el ceftazidime, del 25 % (23-28 %) (cuadro 5) (figura 3).
Cuadro 5. Porcentaje de resistencia de los principales microorganismos aislados en salas de hospitalización general y unidades de cuidados intensivos durante 2018-2021.
| Microorganismo Antibiótico* | Salas de hospitalización general | Unidades de cuidados intensivos de adultos | ||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Resistencia (%) | P** | Resistencia (%) | P** | |||||||
| 2018 | 2019 | 2020 | 2021 | 2018 | 2019 | 2020 | 2021 | |||
| Escherichia coli | ||||||||||
| Ceftriaxona | 23 | 23 | 21 | 22 | NS | 20 | 24 | 21 | 25 | NS |
| Ceftazidime | 11 | 11 | 10 | 10 | NS | 12 | 13 | 10 | 11 | NS |
| Piperacilina-tazobactam | 7 | 6 | 7 | 10 | NS | 8 | 9 | 11 | 14 | NS |
| Cefepime | 8 | 9 | 8 | 9 | NS | 9 | 9 | 8 | 10 | NS |
| Ertapenem | 1 | 1 | 1 | 1 | NS | 1 | 1 | 1 | 2 | NS |
| Imipenem | 1 | 1 | 1 | 1 | NS | 2 | 2 | 3 | 3 | NS |
| Meropenem | 1 | 1 | 1 | 1 | NS | 1 | 2 | 2 | 2 | NS |
| Ciprofloxacina | 40 | 30 | 38 | 38 | NS | 34 | 37 | 38 | 39 | NS |
| Amikacina | 0 | 0 | 1 | 1 | NS | 0 | 1 | 1 | 1 | NS |
| Klebsiella pneumoniae | ||||||||||
| Ceftriaxona | 35 | 31 | 29 | 30 | NS | 32 | 35 | 27 | 27 | NS |
| Ceftazidime | 28 | 24 | 23 | 25 | NS | 25 | 27 | 23 | 22 | NS |
| Piperacilina-tazobactam | 27 | 23 | 24 | 27 | NS | 28 | 30 | 25 | 26 | NS |
| Cefepime | 21 | 19 | 17 | 20 | NS | 20 | 22 | 17 | 19 | NS |
| Ertapenem | 14 | 13 | 13 | 14 | NS | 16 | 21 | 17 | 15 | NS |
| Imipenem | 17 | 14 | 14 | 14 | NS | 18 | 22 | 20 | 18 | NS |
| Meropenem | 15 | 14 | 14 | 14 | NS | 17 | 22 | 19 | 18 | NS |
| Amikacina | 3 | 3 | 4 | 3 | NS | 0 | 6 | 3 | 2 | NS |
| Pseudomonas aeruginosa | ||||||||||
| Ceftazidime | 18 | 19 | 18 | 23 | NS | 21 | 22 | 17 | 25 | NS |
| Cefepime | 14 | 14 | 15 | 18 | NS | 19 | 17 | 14 | 21 | NS |
| Piperacilina-tazobactam | 17 | 17 | 18 | 20 | NS | 21 | 22 | 18 | 25 | <0,05 |
| Imipenem | 24 | 21 | 24 | 30 | <0,05 | 27 | 30 | 26 | 34 | <0,05 |
| Meropenem | 18 | 18 | 20 | 25 | <0,05 | 25 | 25 | 20 | 28 | <0,05 |
| Amikacina | 10 | 12 | 10 | 15 | NS | 10 | 14 | 10 | 14 | NS |
| Staphylococcus aureus | ||||||||||
| Clindamicina | 8 | 10 | 8 | 13 | NS | 8 | 7 | 8 | 10 | NS |
| Oxacilina | 41 | 37 | 37 | 40 | NS | 28 | 28 | 26 | 26 | NS |
NS: no estadísticamente significativo
* La resistencia se determinó utilizando un grupo de antibióticos marcadores.
** Test de Kruskal-Wallis para comparación de grupos independientes
Figura 3. Tendencia del porcentaje de resistencia a antibióticos marcadores en salas de hospitalización general, durante el periodo 2018 a 2021.
SG: salas de hospitalización general
Para otros antibióticos como cefepime y piperacilinatazobactam, la resistencia en cepas de E. coli fue, como media, del 8,5 % y del 7,5 %, respectivamente y, aunque en el 2021 se observó un aumento en la resistencia de E. coli a estos antibióticos, no fue estadísticamente significativo. Para K. pneumoniae, la resistencia a estos dos antimicrobianos fue, como media, del 19,3 % y del 25,2 %, respectivamente (cuadro 5).
En el caso de los carbapenémicos, la resistencia de E. coli fue baja (alrededor del 1 %), en contraste con K. pneumoniae, cuya resistencia a ertapenem se consideró grande: 13 % (13-14 %). Sin embargo, al analizar el comportamiento de la resistencia a los carbapenémicos durante el periodo en estudio (2018-2021), tanto en E. coli como en K. pneumoniae, no se observaron cambios estadísticamente significativos (cuadro 5).
Resistencia de Pseudomonas aeruginosa a TZP, cefepime y carbapenémicos en salas de hospitalización general
La resistencia de P. aeruginosa frente a ceftazidime y cefepime, se mantuvo estable (19 % y 14 %, respectivamente). En contraste, se observó un aumento estadísticamente significativo durante el 2021 con el imipenem y el meropenem, la cual pasó del 24 % al 30 % y del 18 % al 25 %, respectivamente (cuadro 5) (figura 3).
Resistencia de Staphylococcus aureus a oxacilina y clindamicina en salas de hospitalización general
No se observaron cambios significativos en la resistencia de S. aureus ante la clindamicina y la oxacilina. La resistencia a la clindamicina fue, como media, del 10 % (8-13 %). La resistencia a la oxacilina se mantuvo estable, en el 39 % (37-41 %) en promedio (cuadro 5) (figura 3).
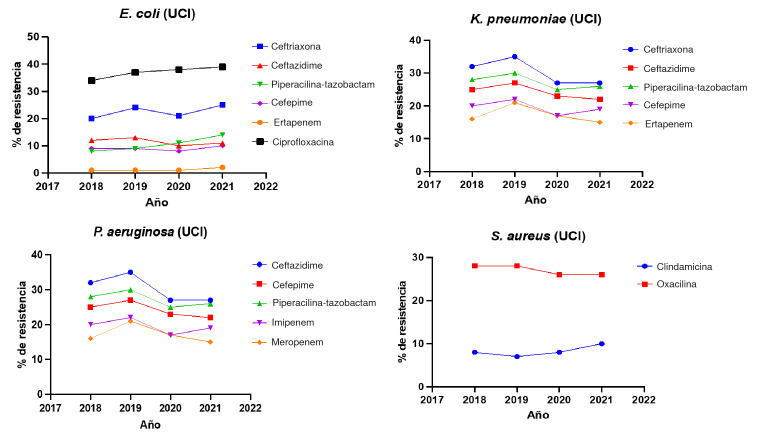
Resistencia de Escherichia coli y Klebsiella pneumoniae en cuidados intensivos
La resistencia de E. coli a la ceftriaxona fue estable, con una media del 22,5 % (20-25 %), similar al comportamiento estable de ceftazidime, con una media del 11,5 % (10-13 %) (cuadro 5). La resistencia a la ciprofloxacina en la unidad de cuidados intensivos para adultos aumentó en el período de estudio, aunque sin significancia estadística, fue del 37 % (34-39 %) (cuadro 5) (figura 4). Por otro lado, en K. pneumoniae la resistencia a ceftriaxona y ceftazidime disminuyó levemente entre el 2018 y el 2021, sin alcanzar la significancia estadística. La resistencia a la ceftriaxona en K. pneumoniae fue del 30,2 % (27-35 %) y, para ceftazidime, del 24,2 % (22-27 %) (cuadro 5) (figura 4).
Figura 4. Tendencia del porcentaje de resistencia a antibióticos marcadores en las unidades de cuidados intensivos de adultos, durante el periodo 2018-2021.
UCI: unidad de cuidados intensivos
Para el cefepime y la piperacilinatazobactam, la resistencia media de E. coli fue del 9 % y el 10,5 %, respectivamente; aunque en el año 2021 se observó un aumento de la resistencia a estos antibióticos, no fue estadísticamente significativo. Para K. pneumoniae, la resistencia a estos dos antimicrobianos tuvo una media del 19,5 % y el 27,2 %, respectivamente (cuadro 5).
Al igual que en las salas de hospitalización general, la resistencia de E. coli a los carbapenémicos en las salas de en cuidados intensivos fue baja, (alrededor del 1 %); en contraste, y similar a lo observado en las salas de hospitalización general, K. pneumoniae presentó gran resistencia al ertapenem, con el 18 % (15-21 %). Sin embargo, al analizar el comportamiento de la resistencia a los carbapenémicos tanto en E. coli como en K. pneumoniae durante el periodo en estudio (2018-2021), no se observaron cambios estadísticamente significativos (cuadro 5). Finalmente, la resistencia de E. coli frente a la ciprofloxacina fue la más grande entre todos los antibióticos tamizados; se situó en el 37 % (34-39 %), como media.
Resistencia de Pseudomonas aeruginosa a piperacilina-tazobactam, cefepime y carbapenémicos en cuidados intensivos
En la unidad de cuidados intensivos de adultos, el ceftazidime y el cefepime tuvieron un comportamiento estable. Fue diferente con la piperacilina-tazobactam, que presentó un aumento significativo: la resistencia pasó de 21 % al 25 %. De igual manera, la resistencia de P. aeruginosa a imipenem y meropenem, aumentó significativamente: pasó de 27 % a 34 % y de 25 % a 28 %, respectivamente (cuadro 5) (figura 4).
Resistencia de Staphylococcus aureus aureus a oxacilina y clindamicina en la unidad de cuidados intensivos de adultos
No se observaron cambios significativos en la resistencia de S. aureus a la clindamicina y la oxacilina. Fue similar con la clindamicina, con el 8 % (710 %) en promedio, en la unidad de cuidados intensivos disminuyó del 28 % en 2018 al 26 % en 2022, pero sin alcanzar significancia estadística (cuadro 5) (figura 4).
Discusión
En este análisis se reportan el comportamiento de las frecuencias, el tipo de muestra más frecuente y el porcentaje de resistencia contra los antimicrobianos en los patógenos más frecuentes provenientes de pacientes hospitalizados en las salas de hospitalización general o en la unidad de cuidados intensivos de adultos, durante el periodo 2018-2021, incluyendo el inicio de la pandemia del SARS-CoV-2, la cual representó un inmenso desafío para el sistema de salud y los programas de optimización de uso de antimicrobianos 22.
De acuerdo con el análisis de estos datos, no se observaron cambios en el orden de frecuencia de los diez primeros microorganismos que se aislaron en el período de estudio, pero sí en el número de aislamientos, el cual disminuyó en las salas de hospitalización general, pero contrastó con un aumento de los aislamientos en la unidad de cuidados intensivos de adultos, particularmente durante el periodo 2020-2021. Esto podría explicarse por la suspensión de procedimientos no esenciales y otros factores, que disminuyeron la hospitalización en salas de hospitalización general durante los primeros meses de la pandemia, así como aumentó el número de adultos con diagnóstico de COVID-19 que ingresaron a la unidad de cuidados intensivos durante la emergencia 14.
Además, entre el 2018 y el 2019, E. coli fue la principal causa de infecciones en la unidad de cuidados intensivos, pero, entre el 2020 y el 2021, K. pneumoniae pasó al primer lugar 23. Estos hallazgos concuerdan con lo reportado en otros países, donde K. pneumoniae se convirtió en el principal agente etiológico en la unidad de cuidados intensivos de adultos debido al aumento de neumonías asociadas con el respirador en pacientes con la COVID-19 24,25. Maes et al. reportaron que hasta el 80 % de los adultos con COVID-19 que ingresó a cuidados intensivos, requirió asistencia respiratoria mecánica invasiva, lo cual se sabe que es un factor de riesgo importante para desarrollar una neumonía asociada al respirador 26. En el presente estudio, en las veinte instituciones de salud, las muestras de origen respiratorio pasaron a ocupar el primer lugar en número de aislamientos durante el año 2021 y el 30 % de estas muestras mostraron la presencia K. pneumoniae.
Briceño et al. (2010) encontraron que las bacterias Gram negativas más frecuentes en la unidad de cuidados intensivos de adultos, durante el periodo 2006-2008 fueron, en orden descendente: E. coli (26 %), K. pneumoniae (9 %), P. aeruginosa (7 %), E. cloacae (3 %) y A. baumannii (1 %) 27. En en el presente análisis, se observó un cambio en la distribución de los microorganismos más frecuentes, tanto en salas de hospitalización general como en la unidad de cuidados intensivos de adultos.
En comparación, en un reporte del Instituto Nacional de Salud que incluyó datos del periodo 2012-2014, A. baumannii estaba entre los 10 principales microorganismos causantes de infecciones en la unidad de cuidados intensivos para adultos y S. aureus ocupaba un cuarto lugar 23. En los cuatro años que incluyen este informe, S. aureus pasó a ocupar el tercer lugar y A. baumannii no se ubicó dentro de los diez primeros microorganismos aislados en la misma unidad. La salida de A. baumannii de este grupo puede deberse a la implementación de programas de desinfección y limpieza más estricta en las instituciones participantes, ya que este agente patógeno es un gran colonizador en el ambiente hospitalario 28.
Con la vigilancia del Instituto Nacional de Salud del 2012 al 2014, se encontró que la resistencia de E. coli a las cefalosporinas de tercera generación era del 23,6 % en la unidad de cuidados intensivos de adultos y del 22,5 % en salas de hospitalización general. Además, en otro estudio sobre infecciones asociadas con la atención en salud, que incluía aislamientos recolectados del 2012 al 2018, se reportó una resistencia a dichas cefalosporinas en un rango de 14 al 31 %, con un promedio del 22,7 % en la unidad de cuidados intensivos y del 24 % en salas de hospitalización general 23,29. Comparativamente, el presente análisis mostró que la resistencia de E. coli a las cefalosporinas de tercera generación durante el periodo 2018-2021 se ubicó en el 22,2 % en salas de hospitalización general y en el 22,5 % en la unidad de cuidados intensivos de adultos, mientras que, para este mismo agente patógeno, la resistencia a carbapenémicos no sobrepasó el 1,5 % en ambos lugares.
Según los reportes del Instituto Nacional de Salud, K. pneumoniae mostró una resistencia a las cefalosporinas de tercera generación y de cuarta generación, del 38,3 % en salas de hospitalización general y de cerca del 30 % en la unidad de cuidados intensivos de adultos 29. En el presente estudio, se observó que la resistencia de K. pneumoniae a las de tercera generación, presentó valores del 30,2 % en salas de hospitalización general y del 31,2 % en la unidad de cuidados intensivos; esto fue similar a lo reportado previamente en las unidades de cuidados intensivos de adultos, pero con una menor tasa de resistencia en las salas de hospitalización general. Frente a la resistencia a carbapenémicos, el Instituto Nacional de Salud reportó el 14,9 % en salas de hospitalización general y el 15,6% en la unidad de cuidados intensivos de adultos, cifras que fueron ligeramente mayores en el presente estudio, del 16 y del 17,2 %, respectivamente.
Para el periodo 2012-2014, Ovalle et al. informaron tasas de resistencia de P. aeruginosa a carbapenémicos que oscilaban entre el 25,2 y el 28,7 % en las unidades de cuidados intensivos, y entre el 19,5 y el 23,3 %, en las salas de hospitalización general 23. En el presente estudio, se encontraron rangos superiores: la resistencia de P. aeruginosa fue del 29,2 % contra imipenem y del 24,5 % contra meropenem en las unidades de cuidados intensivos; y en las salas de hospitalización general, fue de 24,7 y 20,2 %, respectivamente, observándose un aumento estadísticamente significativo en ambos lugares.
En cuanto a S. aureus, su resistencia a la oxacilina reportada previamente se ubicó en el 38,5 % en las salas de hospitalización general y en el 31,3 % las unidades de cuidados intensivos de adultos 23; en el presente análisis fue ligeramente menor, sin significancia estadística: el 37,7 y el 27 %, respectivamente.
Por otro lado, si se comparan nuestros resultados con los del informe de vigilancia del Instituto Nacional de Salud por Whonet de la resistencia antimicrobiana en infecciones asociadas con la atención en salud, se encuentra que, mientras E. coli fue el microorganismo más frecuente en las unidades de cuidados intensivos en el 2019 30, a partir del 2020 y del 2021, K. pneumoniae pasó a ocupar ese lugar 31,32, por encima de E. coli, similar a lo reportado en nuestro estudio. Sin embargo, durante el 2021, P. aeruginosa desplazó a E. coli del segundo puesto en las unidades de cuidado intensivo 32. En comparación, E. coli siguió siendo el segundo microorganismo más frecuente en las unidades de cuidados intensivos en nuestro estudio y P. aeruginosa se ubicó en el tercer puesto. Para el período 2020-2021, el Instituto Nacional de Salud reportó una tendencia al aumento en la resistencia a cefalosporinas de tercera generación en E. coli y K. pneumoniae, en ambos lugares 31,32.
En nuestro estudio, la resistencia de E. coli a las cefalosporinas de tercera generación se mantuvo estable durante los cuatro años del estudio, con una leve disminución para K. pneumoniae. En cuanto a la resistencia a los carbapenémicos, los datos del Instituto Nacional de Salud sobre el período 2020-2021 mostraron que se mantuvo estable en E. coli y K. pneumoniae31,32, igualmente, similar a los hallazgos en nuestro estudio. En general, las cifras de resistencia a las cefalosporinas de tercera generación fueron más altas en los reportes del Instituto Nacional de Salud, pero muy parecidas a las de nuestro estudio para los carbapenémicos 30-32. Los datos del Instituto Nacional de Salud sobre los años 2019-2021 incluyen información de 338 entidades de salud, en promedio; se observan porcentajes de resistencia más altos en algunas regiones, lo que podría elevar el promedio nacional y explicar las diferencias en los datos globales.
La pandemia de COVID-19 en Colombia se inició en marzo de 2020 y el 25 de agosto de 2022 se dio por terminada la emergencia sanitaria; en ese momento, se habían reportado 6'299.595 casos confirmados, 141.519 fallecidos y 6'122.457 recuperados de COVID-19 33. En el consenso colombiano de atención, diagnóstico y manejo de la infección por SARS-CoV-2/COVID-19 en establecimientos de atención de la salud, se recomendó iniciar tratamiento antibiótico empírico en los pacientes con sospecha de neumonía bacteriana leve o moderada, acorde con guías nacionales o guías institucionales, como diagnóstico diferencial o de coinfección SARS-CoV-2 y COVID-19 34. Esto pudo haber impactado por aumento de la resistencia observada en algunos microorganismos debido, más no exclusivamente, al uso inapropiado de los antimicrobianos, a un incremento en su uso o a ambos factores 35.
En los estudios internacionales, se ha encontrado que, al comienzo de la pandemia, entre el 75 y el 95 % de los pacientes con COVID-19 recibió antibióticos a pesar de que menos del 15 % presentaba coinfección bacteriana 36,37. Además, el incremento del uso empírico de antibióticos en pacientes admitidos con COVID-19 grave, estuvo probablemente asociado con la neumonía asociada con el respirador y con bacteriemia, las cuales fueron reportadas como las infecciones más frecuentes 24. En contraste, en otros estudios se reportó que los pacientes infectados con el SARS-CoV-2 tenían tasas bajas de coinfecciones con bacterias y hongos 38.
Como se mencionó anteriormente, en estos estudios la disminución reportada de infecciones bacterianas y fúngicas pudo estar asociada con la suspensión de procedimientos no esenciales, sobre todo en los primeros meses de la pandemia, lo cual llevó a una disminución general del uso de los antibióticos 39. Infortunadamente, los estudios reportados sobre SARS-CoV-2, infecciones bacterianas secundarias y uso de antimicrobianos, no tienen datos estandarizados; utilizan diferentes definiciones, mediciones y poblaciones no comparables, lo que hace difícil establecer relaciones con certeza.
Con base en datos de los institutos nacionales de salud latinoamericanos, Thomas et al. reportaron un incremento en el número de enterobacterias productoras de carbapenemasas, y se observó un aumento de más del doble durante el período 2020-2021 comparado con el 2018-2019 40. Para este reporte de Thomas et al., se recolectaron indistintamente los primeros aislamientos de cada paciente o todos los aislamientos resistentes a carbapenémicos, según el tipo de estudio realizado en cada país 40. Sin embargo, esta recolección no es aleatoria y las instituciones envían solo los aislamientos resistentes, lo cual puede mostrar una resistencia que no refleja la realidad nacional. En nuestro estudio, no se logró evidenciar un aumento significativo en la resistencia basado solo en datos de WHONET.
En este análisis, llama la atención el aumento de S. epidermidis aislado del torrente sanguíneo, que paso del 18 % en 2018 y 2019 al 26 o al 27 % en 2020-2021. Staphylococcus epidermidis puede ser un contaminante en la toma de hemocultivos, pero se ha reportado su aumento global en unidades de cuidados intensivos de adultos durante la pandemia. En México, Martínez et al. encontraron que la sepsis era el segundo tipo de infección en la unidad de cuidados intensivos, y que Staphylococcus coagulasa negativo fue la causa en el 40 % de los casos, en el período de marzo a junio de 2020 24. Si bien no se puede establecer ninguna asociación, la toma de hemocultivos en pacientes críticos con catéteres centrales, podría haber influido en los datos que presentamos aquí.
Finalmente, el que no se haya encontrado un aumento en la resistencia antimicrobiana, a pesar del aparente incremento en el uso empírico de antibióticos, podría deberse a varios factores asociados con el manejo de estos pacientes, como son el incremento en las medidas de control de infecciones implementadas para proteger al personal de salud, las cuales incluyen el lavado de manos, el uso de elementos de protección personal, y mayor desinfección y limpieza en el ambiente hospitalario 8. Es importante continuar la vigilancia epidemiológica local en los hospitales, para entender las dinámicas de la resistencia a los antimicrobianos, las cuales son de especial relevancia en los programas de optimización de uso de antimicrobianos y en la implementación de medidas para prevenir su transmisión.
Agradecimientos
Agradecemos a Alexandra Osorio, por el soporte técnico en el análisis de los datos. Igualmente, a las instituciones participantes y a nuestros colaboradores en: Clínica General del Norte (Barranquilla), Hospital Universitario San Ignacio (Bogotá), Fundación Santa Fe de Bogotá (Bogotá), Fundación Clínica SHAIO (Bogotá), Hospital Central de la Policía Nacional de Colombia (Bogotá), Clínica Juan N. Corpas (Bogotá), Clínica de Occidente (Cali), DIME Clínica Neurocardiovascular (Cali), Clínica Imbanaco Grupo Quirónsalud (Cali), Hospital Universitario Erasmo Meoz (Cúcuta), Hospital Manuel Uribe Ángel (Envigado), Fundación Cardiovascular de Colombia (Floridablanca), Hospital Federico Lleras Acosta (Ibagué), Hospital Universitario Departamental de Nariño (Pasto), Hospital Universitario San Jorge de Pereira (Pereira), Clínica San Marcel (Manizales), Hospital General de Medellín (Medellín), Clínica Las Américas (Medellín), Hospital Universitario Hernando Moncaleano Perdomo (Neiva) y Clínica San Francisco (Tuluá).
Citación: De La Cadena E, Pallares CJ, García-Betancur JC, Porras JA, Villegas MV. Actualización sobre la resistencia antimicrobiana en instituciones de salud de nivel III y IV en Colombia entre enero del 2018 y diciembre del 2021. Biomédica. 2023;43:457-73. https://doi.org/10.7705/biomedica.7065
Contribución de los autores:
Elsa De La Cadena: análisis e interpretación de los datos y redacción del manuscrito
Christian J. Pallares: diseño del estudio y análisis estadístico
Juan Carlos García-Betancur: análisis de los datos y redacción del manuscrito
Jessica A. Porras: recolección y análisis de datos
María Virginia Villegas: diseño del estudio, redacción del manuscrito y supervisión del estudio
Financiación:
El sostenimiento del "Grupo para el estudio de la resistencia nosocomial en Colombia" es posible gracias al apoyo de Pfizer Inc. y MSD Colombia, quienes no tuvieron influencia alguna en la redacción de este manuscrito.
Referencias
- 1.Haustein T, Gastmeier P, Holmes A, Lucet JC, Shannon RP, Pittet D, et al. Use of benchmarking and public reporting for infection control in four high-income countries. Lancet Infect Dis. 2011;(11):471–481. doi: 10.1016/S1473-3099(10)70315-7. [DOI] [PubMed] [Google Scholar]
- 2.World Health Organization . Guidelines for the prevention and control of carbapenemresistant Enterobacteriaceae, Acinetobacter baumannii and Pseudomonas aeruginosa in health care facilities. Geneva: World Health Organization; 2017. [PubMed] [Google Scholar]
- 3.Lan P, Jiang Y, Zhou J, Yu Y. A global perspective on the convergence of hypervirulence and carbapenem resistance in Klebsiella pneumoniae. J Glob Antimicrob Resist. 2021;(25):26–34. doi: 10.1016/j.jgar.2021.02.020. [DOI] [PubMed] [Google Scholar]
- 4.Critchley IA, Karlowsky JA. Optimal use of antibiotic resistance surveillance systems. Clin Microbiol Infect. 2004;(10):502–511. doi: 10.1111/j.1469-0691.2004.00911.x. [DOI] [PubMed] [Google Scholar]
- 5.Harbarth S, Balkhy HH, Goossens H, Jarlier V, Kluytmans J, Laxminarayan R, et al. Antimicrobial resistance: One world, one fight! Antimicrob Resist Infect Control. 2015;(4):1–15. doi: 10.1186/s13756-015-0091-2. [DOI] [Google Scholar]
- 6.Castanheira M, Deshpande LM, Mendes RE, Canton R, Sader HS, Jones RN. Variations in the occurrence of resistance phenotypes and carbapenemase genes among Enterobacteriaceae isolates in 20 years of the SENTRY antimicrobial surveillance program. Open Forum Infect Dis. 2019;(6) 1:S23–S33. doi: 10.1093/ofid/ofy347. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Paterson DL, Rossi F, Baquero F, Hsueh PR, Woods GL, Satishchandran V, et al. In vitro susceptibilities of aerobic and facultative Gram-negative bacilli isolated from patients with intra-abdominal infections worldwide: The 2003 Study for Monitoring Antimicrobial Resistance Trends (SMART) Int J Antimicrob Agents. 2005;(55):965–973. doi: 10.1093/jac/dki117. [DOI] [PubMed] [Google Scholar]
- 8.Canton R, Gijon D, Ruiz-Garbajosa P. Antimicrobial resistance in ICUs: An update in the light of the COVID-19 pandemic. Curr Opin Crit Care. 2020;(26):433–441. doi: 10.1097/MCC.0000000000000755. [DOI] [PubMed] [Google Scholar]
- 9.Shortridge D, Gales AC, Streit JM, Huband MD, Tsakris A, Jones RN. Geographic and temporal patterns of antimicrobial resistance in pseudomonas aeruginosa over 20 years from the SENTRY Antimicrobial Surveillance Program, 1997-2016. Open Forum Infect Dis. 2019;6(1):S63–S68. doi: 10.1093/ofid/ofy343. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Segala FV, Bavaro DF, Di Gennaro F, Salvati F, Marotta C, Saracino A, et al. Impact of sars-cov-2 epidemic on antimicrobial resistance: A literature review. Viruses. 2021;(13):1–15. doi: 10.3390/v13112110. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Jeon K, Jeong S, Lee N, Park MJ, Song W, Kim HS, et al. Impact of COVID-19 on antimicrobial consumption and spread of multidrug-resistance in bacterial infections. Antibiotics. 2022;(11):1–12. doi: 10.3390/antibiotics11040535. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Kariyawasam RM, Julien DA, Jelinski DC, Larose SL, Rennert-May E, Conly JM, et al. Antimicrobial resistance (AMR) in COVID-19 patients: A systematic review and metaanalysis (November 2019-June 2021) Antimicrob Resist Infect Control. 2022;(11) doi: 10.1186/s13756-022-01085-z. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Hughes S, Troise O, Donaldson H, Mughal N, Moore LSP. Bacterial and fungal coinfection among hospitalised patients with COVID-19. Clin Microbiol Infect. 2020;(26):1395–1399. doi: 10.1016/j.cmi.2020.06.025. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.van Duin D, Barlow G, Nathwani D. The impact of the COVID-19 pandemic on antimicrobial resistance: A debate. JAC Antimicrob Resist. 2020;(2):4–5. doi: 10.1093/jacamr/dlaa053. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Bork JT, Leekha S, Claeys K, Seung H, Tripoli M, Amoroso A, et al. Change in hospital antibiotic use and acquisition of multidrug-resistant gram-negative organisms after the onset of coronavirus disease 2019. Infect Control Hosp Epidemiol. 2021;(42):1115–1117. doi: 10.1017/ice.2020.1360. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Russo A, Gavaruzzi F, Ceccarelli G, Borrazzo C, Oliva A, Alessandri F, et al. Multidrugresistant Acinetobacter baumannii infections in COVID-19 patients hospitalized in intensive care unit. Infection. 2022;(50):83–92. doi: 10.1007/s15010-021-01643-4. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Khojasteh S, Jafarzdeh J, Hosseini SA, Haghani I, Turki H, Aghaei Gharehbolagh S, et al. Candida auris and COVID-19: A health threatening combination. Curr Med Mycol. 2022;(8):44–50. doi: 10.18502/cmm.8.3.11211. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Patel A, Emerick M, Cabunoc MK, Williams MH, Preas MA, Schrank G, et al. Rapid spread and control of multidrug-resistant Gram-negative bacteria in COVID-19 patient care units. Emerg Infect Dis. 2021;(27):1234–1237. doi: 10.3201/eid2704.204036. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Stelling JM, O’Brien TF. Surveillance of antimicrobial resistance: The WHONET program. Clin Infect Dis. 1997;24(1):S157–S168. doi: 10.1093/clinids/24.supplement_1.s157. [DOI] [PubMed] [Google Scholar]
- 20.Clinical and Laboratory Standards Institute (CLSI) Performance standards for antimicrobial susceptibility testing. 32. Wayne, PA: Clinical and Laboratory Standards Institute; 2022. [Google Scholar]
- 21.Clinical and Laboratory Standards Institute (CLSI) Analysis and presentation of cumulative antimicrobial susceptibility test data; Approved guideline -Fourth edition. CLSI document M39-A4. Wayne, PA: Clinical and Laboratory Standards Institute; 2014. [Google Scholar]
- 22.Plante JA, Mitchell BM, Plante KS, Debbink K, Weaver SC, Menachery VD. The variant gambit: COVID-19’s next move. Cell Host Microbe. 2021;(29):508–515. doi: 10.1016/j.chom.2021.02.020. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.Ovalle MV, Saavedra SY, González MN, Hidalgo AM, Duarte C, Beltrán M. Resultados de la vigilancia nacional de la resistencia antimicrobiana de enterobacterias y bacilos Gram negativos no fermentadores en infecciones asociadas a la atención de salud, Colombia, 2012-2014. Biomédica. 2017;(37):473–485. doi: 10.7705/biomedica.v37i4.3432. [DOI] [PubMed] [Google Scholar]
- 24.Martínez-Guerra BA, González-Lara MF, de León-Cividanes NA, Tamez-Torres KM, Román- Montes CM, Rajme-López S, et al. Antimicrobial resistance patterns and antibiotic use during hospital conversion in the COVID-19 pandemic. Antibiotics. 2021;(10):1–9. doi: 10.3390/antibiotics10020182. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.Nowak MD, Sordillo EM, Gitman MR, Paniz Mondolfi AE. Coinfection in SARS-CoV-2 infected patients: Where are influenza virus and rhinovirus/enterovirus? J Med Virol. 2020;(92):1699–1700. doi: 10.1002/jmv.25953. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.Maes M, Higginson E, Pereira-Dias J, Curran MD, Parmar S, Khokhar F, et al. Ventilatorassociated pneumonia in critically ill patients with COVID-19. Crit Care. 2021;(25):1–11. doi: 10.1186/s13054-021-03460-5. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27.Briceño DF, Correa A, Valencia C, Torres JA, Pacheco R, Montealegre MC, et al. Antimicrobial resistance of Gram negative bacilli isolated from terciary-care hospitals in Colombia. Biomédica. 2010;(30):371–381. doi: 10.7705/biomedica.v30i3.271. [DOI] [PubMed] [Google Scholar]
- 28.Fournier PE, Richet H. The epidemiology and control of Acinetobacter baumannii in health care facilities. Clin Infect Dis. 2006;(42):692–699. doi: 10.1086/500202. [DOI] [PubMed] [Google Scholar]
- 29.Ovalle MV, Saavedra SY, Rodríguez MK. Informe de resultados de la vigilancia por laboratorio de resistencia antimicrobiana en infecciones asociadas a la atención en salud. Bogotá, D.C.: Instituto Nacional de Salud;; 2018. [Google Scholar]
- 30.Ovalle MV, Duarte C, Zambrano C. Informe de resultados de la vigilancia por laboratorio de resistencia antimicrobiana en infecciones asociadas a la atención en salud. Bogotá, D.C.: Instituto Nacional de Salud;; 2019. [Google Scholar]
- 31.Ovalle MV, Duarte C, Leal AL, Zambrano C. Informe de resultados de la vigilancia por laboratorio de resistencia antimicrobiana en infecciones asociadas a la atención en salud. Bogotá, D.C.: Instituto Nacional de Salud;; 2020. [Google Scholar]
- 32.Ovalle MV, Duarte C, Flórez AC, Leal AL, Zambrano C. Informe de resultados de la vigilancia por laboratorio de resistencia antimicrobiana en infecciones asociadas a la atención en salud. Bogotá, D.C.: Instituto Nacional de Salud;; [Google Scholar]
- 33.Organización Panamericana de la Salud . Reporte de situación COVID-19 Colombia No. 285 - 25 de agosto 2022. [20 de agosto del 2023]. Disponible en: https://www.paho.org/es/reportes-situacion-covid-19-colombia . [Google Scholar]
- 34.Saavedra Trujillo CH. Consenso colombiano de atención, diagnóstico y manejo de la infección por SARS-CoV-2/COVID-19 en establecimientos de atención de la salud. Recomendaciones basadas en consenso de expertos e informadas en la evidencia. Infectio. 2020;(24):50–60. doi: 10.22354/in.v24i3.889. [DOI] [Google Scholar]
- 35.Rawson TM, Ming D, Ahmad R, Moore LSP, Holmes AH. Antimicrobial use, drug-resistant infections and COVID-19. Nat Rev Microbiol. 2020;(18):409–410. doi: 10.1038/s41579-020-0395-y. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 36.Chen N, Zhou M, Dong X, Qu J, Gong F, Han Y, et al. Epidemiological and clinical characteristics of 99 cases of 2019 novel coronavirus pneumonia in Wuhan, China: A descriptive study. Lancet. 2020;(395):507–513. doi: 10.1016/S0140-6736(20)30211-7. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 37.Zhou F, Yu T, Du R, Fan G, Liu Y, Liu Z, et al. Clinical course and risk factors for mortality of adult inpatients with COVID-19 in Wuhan, China: A retrospective cohort study. Lancet. 2020;(395):1054–1062. doi: 10.1016/S0140-6736(20)30566-3. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 38.Rawson TM, Moore LSP, Zhu N, Ranganathan N, Skolimowska K, Gilchrist M, et al. Bacterial and fungal coinfection in individuals with coronavirus: A rapid review to support COVID-19 antimicrobial prescribing. Clin Infect Dis. 2020;(71):2459–2468. doi: 10.1093/cid/ciaa530. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 39.Rawson TM, Moore LSP, Castro-Sánchez E, Charani E, Davies F, Satta G, et al. COVID-19 and the potential long-term impact on antimicrobial resistance. J Antimicrob Chemother. 2020;(75):1681–1684. doi: 10.1093/jac/dkaa194. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 40.Thomas GR, Corso A, Pasterán F, Shal J, Sosa A, Pillonetto M, et al. Increased detection of carbapenemase-producing Enterobacterales bacteria in Latin America and the Caribbean during the COVID-19 pandemic. Emerg Infect Dis. 2022;(28):1–8. doi: 10.3201/eid2811.220415. [DOI] [PMC free article] [PubMed] [Google Scholar]