Abstract
目的
通过生物信息学方法预测早幼粒细胞白血病蛋白与TAK1结合蛋白的相互作用,并通过免疫共沉淀方法进行实验验证。
方法
使用Rosetta软件,采用比较建模的方法,构建TAB1蛋白的三维模型;在PDB数据库中检索PML蛋白二级结构,并解析其晶体结构和三维结构。Zdock3.0.2软件进行PML与TAB1的蛋白-蛋白对接,并提取最佳构象进行对接模型的分子结构分析。α-MMC处理的M1炎性巨噬细胞,利用免疫共沉淀技术检测两种蛋白的相互作用。
结果
以PML的6IMQ为对接部位时,PML蛋白与TAB1蛋白能形成3个盐桥、6个氢键和6个疏水作用;以PML的5YUF为对接部位时,PML蛋白与TAB1蛋白能形成1个氢键、3个静电相互作用和9个疏水作用,两种对接模式皆能形成良好的分子对接和相互作用;在α-MMC处理4 h后,其PML-IP组的细胞裂解沉淀液中分别能检测到显著的PML和TAB1阳性蛋白条带。
结论
PML蛋白与TAB1蛋白能发生较强的相互作用。
Keywords: TAB1蛋白, PML蛋白, 生物信息学, 蛋白-蛋白对接, 免疫共沉淀
Abstract
Objective
To analyze the interaction between PML protein and TAB1 protein using bioinformatic approaches and experimentally verify the results.
Methods
Using Rosetta software, a 3D model of TAB1 protein was constructed through a comparative modeling approach; the secondary structure of PML protein was retrieved in the PDB database and its crystal structure and 3D structure were resolved. Zdock 3.0.2 software was used to perform protein-protein docking of PML and TAB1, and the best conformation was extracted for molecular structure analysis of the docking model. The interaction between the two proteins was detected using immunoprecipitation in α-MMC-treated M1 inflammatory macrophages.
Results
When 6IMQ of PML was used as the docking site, PML protein formed 3 salt bridges, 6 hydrogen bonds and 6 hydrophobic interactions with TAB1 proteins; when 5YUF of PML was used as the docking site, PML protein formed 1 hydrogen bond, 3 electrostatic interactions and 9 hydrophobic interactions with TAB1 proteins, and both of the docking modes formed good molecular docking and interactions. In the M1 inflammatory macrophages treated with α-MMC for 4 h, positive protein bands of PML and TAB1 were detected in the cell lysates in PML-IP group.
Conclusion
PML protein can interact strongly with TAB1 protein.
Keywords: TAB1 protein, PML protein, bioinformatics, protein-protein docking, co-immunoprecipitation
转化生长因子活化蛋白激酶1(TAK1)是TLR受体信号通路的一个重要信号蛋白[1, 2]。当细菌或病毒感染激活该信号通路时,TAK1结合蛋白(TAB1)与TAK1结合形成复合物能通过促进TAK1自我磷酸化而激活下游的MAPK信号通路和NF-κB信号通路,引发TNF-α、IL-6和IL-8等炎性细胞因子的表达,导致炎症反应[3, 4]。在前期研究α-苦瓜素(α-MMC)的抗炎机制时发现,α-MMC能通过抑制TAK1蛋白而抑制LPS诱导的这种炎症反应[5],进一步的蛋白组学分析发现,TAB1蛋白也能被抑制,而早幼粒细胞白血病蛋白(PML)则出现显著性升高。PML是急性早幼粒细胞白血病(APL)的一种肿瘤抑制蛋白,在生长抑制、诱导细胞凋亡和细胞衰老方面具有关键功能[6],并且PML也具有抗病毒感染功能,在Toll样受体信号途径中发挥作用[7]。我们的组学结果提示,α-MMC可能通过激活PML而抑制TAB1,进而抑制了TAK1及其下游的MAPK和NF-κB炎性信号通路。那么,PML是否能跟TAB1发生互作是该信号转导发生的关键环节。经国内外文献查阅,我们并没有发现任何关于PML与TAB1蛋白相互作用的报道,因此,本研究拟首先通过生物信息学预测二者是否有相互作用,再通过免疫共沉淀(Co-IP)实验验证这种互作关系。由于PML蛋白是近些年的一个研究热点,PML与TAB1蛋白互作并抑制Toll样炎性信号通路的发现,有望为探明一条新的抗炎信号通路奠定基础。
1. 材料和方法
1.1. 材料
α-MMC,由四川大学生命科学学院的孟延发教授赠予,使用MacroCap SP和Superdex75柱层析技术,从苦瓜籽中提取纯化而得,其蛋白质样品纯度大于95%;佛波酯(PMA);细菌脂多糖(LPS);RPMI 1640培养基;Anti-PML和Anti-TAB1(abcam);Protein A+G agarose、RIPA lysis buffer、PMSF以及BCA蛋白定量检测试剂盒(碧云天生物技术有限公司);ECL(武汉聚能译通生物公司);HRP标记山羊抗兔二抗(武汉三鹰生物技术有限公司);HRP AffiniPure Mouse Anti-Rabbit IgG(Light Chain)(Abbkine);人单核细胞THP-1细胞系(中国科学院典型文化保护委员会细胞库)。
1.2. 方法
1.2.1. 构建TAB1蛋白模型
1.2.1.1. 解析TAB1蛋白一级结构
在PSST-2.0(http://pranag.physics.iisc.ernet.in/psst/)蛋白质数据库序列搜索工具中检索出TAB1蛋白一级结构即氨基酸排列顺序相关的数据[8]。
1.2.1.2. 解析TAB1蛋白二级结构
根据靶标蛋白TAB1蛋白的一级序列,采用第三代测序技术和多重序列比对方法(MSA法),找到高相似蛋白质,并建立同源数据库;用这些蛋白质中含有的可取代氨基酸信息,使用PSIPRED软件预测TAB1蛋白二级结构[9, 10]。
1.2.1.3. 解析TAB1蛋白三级结构
使用Rosetta软件,采用比较建模的方法,构建完整的包含C-端结构域的TAB1蛋白的三维模型[11, 12];通过序列比对,匹配出对应的模板蛋白。
1.2.2. 构建PML蛋白模型
在PDB数据库中检索PML蛋白[13],并对目前已报道在PDB数据库中的晶体结构进行收集。选取位于PML蛋白RBCC结构域中的晶体结构6IMQ以及5YUF进行三维结构模拟。
1.2.3. 蛋白-蛋白对接
利用Zdock3.0.2软件进行蛋白-蛋白对接。对接前,先设定两个蛋白的关键氨基酸结合位点,设定靶标蛋白TAB1的关键氨基酸结合位点位为Y481、V482、F484、F487、Y488、W491、H495;设定配体蛋白PML的关键氨基酸结合位点,PDB ID为6IMQ的晶体结构设定K160为作用位,PDB ID为5YUF的晶体结构设定K65为作用位点。每次对接将产生10个对接结果,然后挑选对接能量最好的构象进行结构提取。
1.2.4. 极化炎性巨噬细胞(M1型)模型构建及α-MMC处理
培养条件为含有10%热灭活胎牛血清、100 U/mL青霉素和100 µg/mL链霉素的完全RPMI 1640培养基,在37 ℃的含5% CO2的湿空气中。将约1×106 THP-1细胞接入6孔板中,加入PMA(30 ng/mL)培养72 h,换液彻底洗掉漂浮细胞,加入新鲜培养基培养24 h,再以终浓度为20 ng/mL的LPS加入到以上六孔板培养细胞中,在10%胎牛血清的完全RPMI 1640培养基培养48 h,诱导其成为极化炎性巨噬细胞(M1型)。用以终浓度为0.5 μg/mL的α-MMC分别处理M1巨噬细胞0、1、2、4 h。之后,每孔加入RIPA裂解液,在冰上裂解30 min并超声3 min,然后在4 ℃ 12 000 r/min条件下离心10 min,转移到新的EP管中待用。
1.2.5. PML与TAB1蛋白免疫印迹分析
使用BCA法测定上清蛋白的浓度,根据裂解液的体积加入5×SDS loading bufer,100 ℃煮沸5 min。蛋白质样品(20 μg)在10% SDS聚丙烯酰胺凝胶和5% SDS浓缩凝胶上分离。将样品电转移到PVDF上并用5% milk/TBST封闭。洗涤后,用1% BSA/PBST按推荐的稀释比稀释一抗同转移PVDF放入杂交袋,4 ℃冰箱过夜。使用一抗为兔抗人PML单克隆抗体和兔抗人TAB1单克隆抗体,稀释比例分别为1∶1000。再后分别将PVDF膜放入用5% milk/PBST稀释的辣根过氧化物酶标记的羊抗兔IgG(H+L)多克隆抗体(1∶5000稀释)在室温下置于摇床上孵育1 h。之后采用增效鲁米诺试剂和增强型化学发光试剂(ECL)作为显色底物,使用化学发光凝胶成像仪进行显色,用兔抗人β-actin抗体作为内参对照。
1.2.6. PML与TAB1蛋白的免疫共沉淀分析
(1) 样本处理 取α-MMC处理4 h的上清样本,加入100 µL Protein A+G琼脂糖珠子,4 ℃旋转1 h,去除非特异性结合蛋白,留50 µL作为input阳性对照组,取2 µL测定浓度,其余上清备用;(2)抗体与珠子结合 取3 μg抗体,阴性对照组用相应量的正常IgG,按照抗体说明书稀释抗体,分别加入100 µL Protein A+G琼脂糖珠子,4 ℃旋转2 h,然后1000 g,4 ℃离心5 min;(3)免疫印迹分析 取细胞裂解液分别加入到偶联抗体或正常IgG的蛋白A+G琼脂糖珠子中,4 ℃旋转过夜,使蛋白与抗体结合;1000 g 4 ℃离心5 min,去除上清;用PBS洗4次,每次1 mL;尽量去除上清,加入100 µL 1×loading buffer煮沸5 min;1000 g离心5 min,取上清作为PML-IP实验组样品进行免疫印迹检测,免疫印迹的操作方法同1.2.5。由于TAB1分子大小与IgG重链大小相近,故本实验检测TAB1的二抗选用辣根过氧化物酶标记的小鼠抗兔IgG的轻链。显色底物使用增强型鲁米诺试剂和氧化试剂。
2. 结果
2.1. 靶标蛋白TAB1信息
人TAB1蛋白由504个氨基酸组成,蛋白的序列信息见表 1,其主要功能是能募集其他蛋白如TAK1蛋白形成复合物。
表 1.
靶标蛋白的序列信息
Sequence information of the target protein
| Protein | Protein sequence information |
| TAB1 | > NP_006107.1 TGF-beta-activated kinase 1 and MAP3K7-binding protein 1 isoform alpha [Homo sapiens] MAAQRRSLLQSEQQPSWTDDLPLCHLSGVGSASNRSYSADGKGTESHPPEDSWLKFRSENNCFLYGVFNGY DGNRVTNFVAQRLSAELLLGQLNAEHAEADVRRVLLQAFDVVERSFLESIDDALAEKASLQSQLPEGVPQH QLPPQYQKILERLKTLEREISGGAMAVVAVLLNNKLYVANVGTNRALLCKSTVDGLQVTQLNVDHTTENED ELFRLSQLGLDAGKIKQVGIICGQESTRRIGDYKVKYGYTDIDLLSAAKSKPIIAEPEIHGAQPLDGVTGFLVL MSEGLYKALEAAHGPGQANQEIAAMIDTEFAKQTSLDAVAQAVVDRVKRIHSDTFASGGERARFCPRHEDM TLLVRNFGYPLGEMSQPTPSPAPAAGGRVYPVSVPYSSAQSTSKTSVTLSLVMPSQGQMVNGAHSASTLDEA TPTLTNQSPTLTLQSTNTHTQSSSSSSDGGLFRSRPAHSLPPGEDGRVEPYVDFAEFYRLWSVDHGEQSVVTAP |
2.1.1. 靶标蛋白TAB1的二级结构预测
从TAB1二级结构(图 1)可看出,靶标蛋白TAB1的C-端柔性较大,具有一段α螺旋区特征性序列,即FxxFxxxWxxxH。
图 1.

靶标蛋白TAB1的二级结构预测
Secondary structure prediction of the target protein TAB1.
2.1.2. 靶标蛋白TAB1的三维空间结构建立
使用Rosetta软件,共预测出5个候选的三维模型,将这5个候选模型的三维结构与前面预测的二级结构做映射,对比结果发现预测的model2.pdb三维结构与预测的二级结构大致相符合,TAB1蛋白的三维结构模型如图 2所示。
图 2.
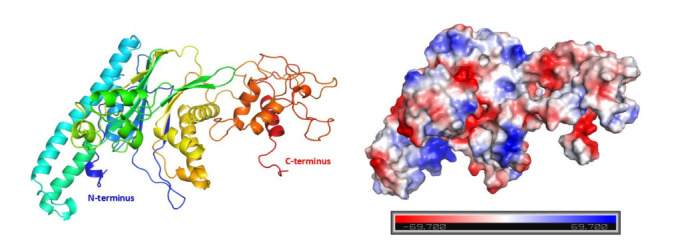
TAB1蛋白的三维结构模型
Three-dimensional structure model of TAB1 protein.
2.2. 配体蛋白PML信息
PML基因位于15号染色体,全长53KB,含有9个外显子。PML蛋白具有富含半胱氨酸的N末端,即1个环指结构域、2个锌指样B盒(B-box)和1个卷曲螺旋结构域构成的“RBCC”结构,是PML发挥功能的关键结构区域。PML“RBCC”结构域上的两个晶体结构6IMQ以及5YUF(图 3)为PML蛋白的120~168位氨基酸和51~96位氨基酸残基片段,其Resolution值分别为2.06 Å,1.60 Å。
图 3.
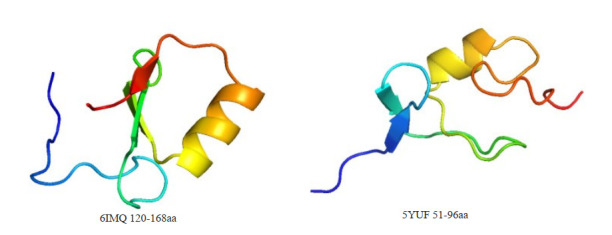
配体蛋白PML的三维结构模型
Three-dimensional structure model of the ligand protein PML.
2.3. 蛋白-蛋白对接
2.3.1. 配体蛋白PML(6IMQ)与靶标蛋白TAB1的蛋白-蛋白对接
对接能量值前10位的pose分别(表 2),其聚类分析(图 4)。
表 2.
蛋白-蛋白对接结果的能量值
Energy value of the protein-protein docking results
| No. | Conformation ID | Energy |
| pose1 | complex_1 | 1322.214 |
| pose2 | complex_2 | 1282.055 |
| pose3 | complex_3 | 1216.260 |
| pose4 | complex_4 | 1204.665 |
| pose5 | complex_5 | 1188.547 |
| pose6 | complex_6 | 1175.859 |
| pose7 | complex_7 | 1171.583 |
| pose8 | complex_8 | 1157.472 |
| pose9 | complex_9 | 1156.579 |
| pose10 | complex_10 | 1151.923 |
图 4.
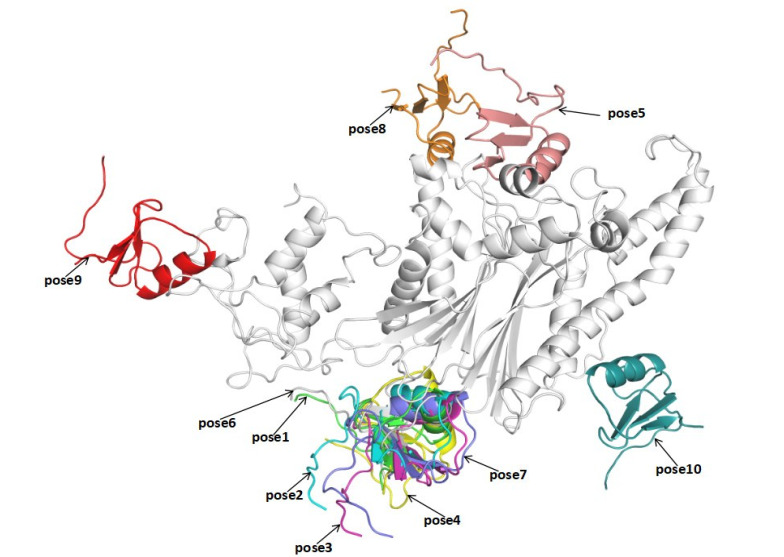
靶标蛋白TAB1与配体蛋白PML(6IMQ)结合位置聚类分析图
Cluster analysis of the binding sites of the target protein TAB1 and ligand protein PML (6IMQ).
聚类分析显示,靶标蛋白TAB1(灰色)与配体蛋白PML(不同颜色)在多个位置都有良好的结合,但大多数的结合位于TAB1蛋白的C-端,如pose1-4和pose6-7皆在该位置与TAB1聚合(图 4)。
选取对接能量值最高的pose1对接结果构象进行结构提取用于相互作用分析,其结合模式分析(图 5),相互作用的关键氨基酸及作用化学键列表(表 3)。从图 5和表 3中可以看出,配体蛋白PML主要结合在靶标蛋白TAB1的C端,二者间有3个盐桥、6个氢键和6个疏水力相互作用。
图 5.
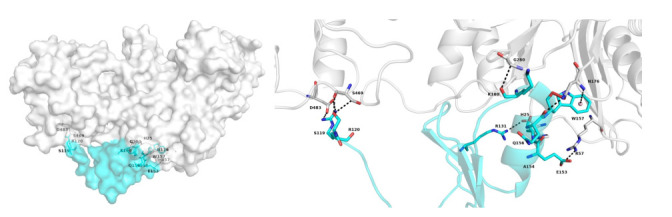
靶标蛋白TAB1与配体蛋白PML(6IMQ)相互作用图(pose1)
Interaction map of the target protein TAB1 and the ligand protein PML (6IMQ) (pose1).
表 3.
靶标蛋白TAB1与配体蛋白PML(6IMQ)相互作用模式
Interaction mode between the target protein TAB1 and the ligand protein PML(6IMQ)
| Interaction pair of TAB1-PML | Distance (Å) | Category | Types |
| ARG57:NH2-GLU153:OE1 | 3.63 | Hydrogen Bond; Electrostatic | Salt Bridge |
| ASP483:OD1-ARG120:NH1 | 2.46 | Hydrogen Bond; Electrostatic | Salt Bridge |
| ASP483:OD2-ARG120:NH2 | 2.89 | Hydrogen Bond; Electrostatic | Salt Bridge |
| ASN176:ND2-GLN156:O | 3.02 | Hydrogen Bond | Conventional Hydrogen Bond |
| SER469:O-SER119:OG | 2.44 | Hydrogen Bond | Conventional Hydrogen Bond |
| HIS25:O-ARG131:NH2 | 3.20 | Hydrogen Bond | Conventional Hydrogen Bond |
| GLY280:CA-LYS160:O | 3.76 | Hydrogen Bond | Carbon Hydrogen Bond |
| SER469:CB-SER119:OG | 3.55 | Hydrogen Bond | Carbon Hydrogen Bond |
| ASN176:N-TRP157 | 3.76 | Hydrogen Bond | Pi-Donor Hydrogen Bond |
| PHE365:CA-PHE158 | 3.84 | Hydrophobic | Pi-Sigma |
| PHE63-TRP157 | 4.16 | Hydrophobic | Pi-Pi T-shaped |
| PHE63-TRP157 | 4.57 | Hydrophobic | Pi-Pi T-shaped |
| LEU26-ALA154 | 4.29 | Hydrophobic | Alkyl |
| HIS25-ALA154 | 5.01 | Hydrophobic | Pi-Alkyl |
| LEU26-PHE158 | 4.57 | Hydrophobic | Pi-Alkyl |
2.3.2. 配体蛋白PML(5YUF)与靶标蛋白TAB1的蛋白-蛋白对接
设定5YUF晶体结构的K65为PML的关键氨基酸结合位点与TAB1蛋白模型进行蛋白-蛋白对接,选取对接能量值前2位的结合构象(pose1为937.271,pose2为934.068)进行结构模拟作图,其中,灰色结构部分为TAB1,其他颜色部分为PML(图 6)。
图 6.
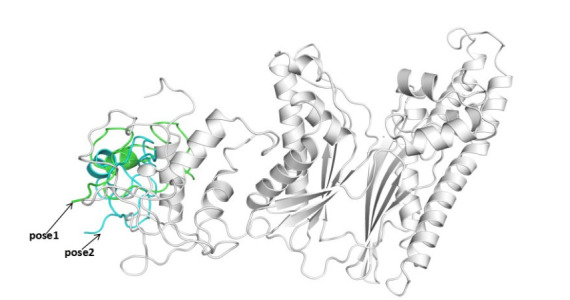
靶标蛋白TAB1与配体蛋白PML(5YUF)结合位置聚类分析图
Cluster analysis of the binding sites of the target protein TAB1 and the ligand protein PML(5YUF).
选取对接能量值最高的pose1对接结果构象进行结构提取用于相互作用分析,其结合模式分析(图 7),相互作用的关键氨基酸及作用化学键列表(表 4)。从图 7和表 4中可以看出,配体蛋白PML主要结合在靶标蛋白TAB1的C端,二者间形成了1个氢键、3个静电和9个疏水相互作用。
图 7.
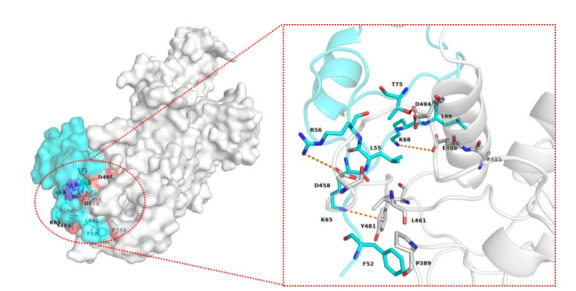
靶标蛋白TAB1与配体蛋白PML(5YUF)相互作用图(pose1)
Interaction between the target protein TAB1 and the ligand protein PML (5YUF) (pose1).
表 4.
靶标蛋白TAB1与配体蛋白PML(5YUF)相互作用模式
Interaction mode between the target protein TAB1 and the ligand protein PML(5YUF)
| Interaction pair of TAB1-PML | Distance (Å) | Category | Types |
| ASP458:OD2-ARG56:NH2 | 4.52 | Electrostatic | Attractive Charge |
| GLU486:OE1-LYS68:NZ | 5.34 | Electrostatic | Attractive Charge |
| ASP494:OD1-THR75:OG1 | 2.57 | Hydrogen Bond | Conventional Hydrogen Bond |
| TYR481-LYS65:NZ | 4.65 | Electrostatic | Pi-Cation |
| LEU461-LEU55 | 5.03 | Hydrophobic | Alkyl |
| VAL482-PRO67 | 5.16 | Hydrophobic | Alkyl |
| ALA485-LEU69 | 5.47 | Hydrophobic | Alkyl |
| ARG489-LEU69 | 4.07 | Hydrophobic | Alkyl |
| LEU490-PRO67 | 5.08 | Hydrophobic | Alkyl |
| TYR481-CYS66 | 5.40 | Hydrophobic | Pi-Alkyl |
| PRO389-PHE52 | 4.97 | Hydrophobic | Pi-Alkyl |
| LEU461-PHE54 | 4.79 | Hydrophobic | Pi-Alkyl |
| VAL493-HIS74 | 4.97 | Hydrophobic | Pi-Alkyl |
2.4. PML与TAB1蛋白免疫印迹分析
PML蛋白与TAB1蛋白的免疫印迹检测结果显示,随着α-MMC处理时间的延长,PML蛋白的浓度逐渐增强,而TAB1蛋白浓度有减弱趋势,在α-MMC处理4 h时表现明显(图 8)。
图 8.
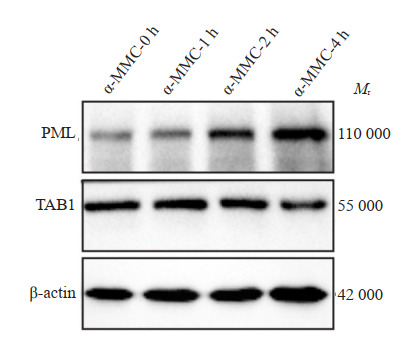
PML蛋白与TAB1蛋白的免疫印迹检测结果
Western blotting of PML protein and TAB1 protein in α-MMC-treated M1 inflammatory macrophages.
2.5. 免疫共沉淀实验验证
PML蛋白与TAB1蛋白的免疫共沉淀WB检测结果显示,当使用抗PML作为检测抗体时,在PML-input组和IgG组相应的蛋白相对分子质量处有单一深染的条带,PML-IP组则显示两条浓密的条带,分别与input组和IgG组形成的条带齐平(图 9A);当使用抗TAB1作为检测抗体时,PML-IP组也显示出两条浓密的条带,分别与TAB1-input组和IgG(轻链)组形成的条带齐平(图 9B)。分别选取图 9A和图 9B的相应条带进行免疫共沉淀的Co-IP拼图,显示出PML蛋白与TAB1蛋白有良好的共沉淀效果(图 9C)。
图 9.
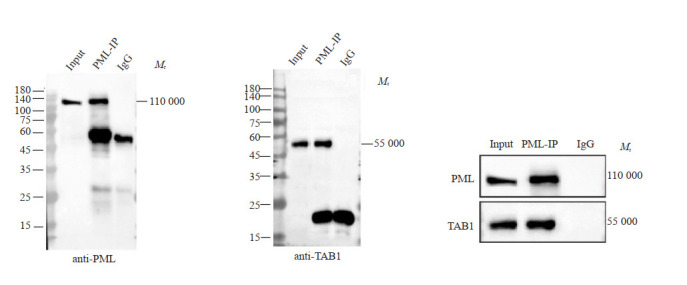
PML蛋白与TAB1蛋白的免疫共沉淀检测结果
Immunoprecipitation results of PML protein and TAB1 protein in α-MMC-treated M1 inflammatory macrophages.
3. 讨论
α-MMC是来自于苦瓜的天然药物,具有抗肿瘤、抗病毒和免疫调节等多种药理效应[14, 15]。我们的研究也发现,α-MMC能通过抑制TLR4受体信号通路的TAK1信号蛋白而抑制下游的NF-κB和MAPK炎性信号通路,从而抑制LPS诱导的炎性细胞因子风暴和炎症[7]。在深入探讨α-MMC抑制TAK1蛋白发生机制时发现,α-MMC能显著升高PML蛋白,同时TAK1结合蛋白TAB1也被显著抑制。PML是在细胞生命过程中发挥多种重要功能的蛋白,包括肿瘤抑制、转录调节、细胞凋亡和衰老、DNA损伤反应和病毒防御等[16-18]。PML蛋白也能参与信号转导,在TGF-β受体信号通路中,细胞质PML能介导SARA蛋白与Smad2/3之间的信号[19, 20]。因此,推测PML蛋白也可能为α-MMC抑制TAB1/TAK1的中间信号转导蛋白。本研究通过生物信息学模拟,观察到了PML蛋白与TAB1的互作,并经过经典的免疫共沉淀实验验证,实验结果证实了以上设想。
生物信息学模拟实验中,通过使用Rostta软件,采用比较建模的方法,构建完整的包含C-端结构域的TAB1蛋白三维模型[21],又通过检索PDB数据库获取PML蛋白信息,并解析其晶体结构和三维结构,然后使用Zdock3.0.2软件对PML与TAB1进行了蛋白-蛋白分子对接模拟,并提取最佳构象进行分子对接的结构分析。结果显示,两个蛋白之间分别形成了3个盐桥、5个氢键和6个疏水相互作用力(以PML的6IMQ为对接部位)以及1个氢键、3个静电相互作用力和9个疏水作用力(以PML的5YUF为对接部位),两种对接模式皆显示出了良好的分子对接和强力的相互作用。Rosetta软件是目前流行的结构分析软件[22],其原理是依据于X射线晶体结构数据,以基于贝叶斯概率理论的能量函数,进行生物分子构象相关的能量分析,从而识别和分析生物分子模型的特征。而Zdock3.0.2蛋白-蛋白对接软件原理是基于快速傅里叶转化相关性技术的刚性蛋白对接算法,用来搜索两种蛋白质之间通过平移和旋转而得到的空间中所有可能的相互结合模式,并使用能量评分函数评估每个结合模型[23]。
在成功预测PML与TAB1的蛋白互作后,我们进行了经典的免疫共沉淀技术验证实验。首先使用免疫印迹技术,观察α-MMC作用后PML蛋白和TAB1蛋白相互作用的有效时间点。结果显示,在给予α-MMC处理M1炎性巨噬细胞后,PML蛋白的表达随着给药时间延长(0、1、2和4 h)而增高,而TAB1蛋白在给药4 h后出现了明显的蛋白量下降。因此,选用4 h作用时间点,进行后续的免疫共沉淀实验。共沉淀后的PML-IP组能检测到显著的PML和TAB1互作阳性蛋白条带,input阳性组和IgG阴性组也都显示出了良好对照效果。实验中使用的M1巨噬细胞模型,是以单核细胞THP-1细胞系为原型细胞,通过PMA诱导为巨噬细胞后再使用LPS极化为炎性巨噬细胞[24, 25]。
在TGFβR信号通路中,PML介导了SARA蛋白与Smad2/3之间的信号,PML发挥的作用是磷酸化Smad2/3蛋白而传递激活信号,而我们的实验结果显示,PML对TAB1的作用并不是磷酸化激活,而是抑制作用。这种抑制作用的机制尚不清楚,我们推测PML发挥的是可能泛素化连接酶作用。PML蛋白有多种功能,其结构具有1个环指结构域、2个锌指样B盒和1个卷曲螺旋结构域,PML的环指结构域具有E3泛素化酶活性,亦即,PML为一种单环指E3泛素化连接酶[26, 27]。泛素化修饰引起靶蛋白降解是信号通路中负性调节的常见模式[28, 29]。PML是否是通过泛素化修饰TAB1蛋白导致其被降解,有待后续进一步研究。本研究通过生物信息学预测以及实验验证,首次揭示了PML与TAB1蛋白的直接相互作用,为探讨PML介导的抗炎信号转导作用奠定了基础。
Biography
成佳聪,在读硕士研究生,E-mail: 2864128160@qq.com
Funding Statement
四川省科技厅重点研发项目(2018SZ0016,2019YFS0307,2021YFS0053);成都医学院自然科学基金(CYZYB22-04);成都医学院检验医学院自然科学基金(JYZK202201)
Contributor Information
成 佳聪 (Jiacong CHENG), Email: 2864128160@qq.com.
沈 富兵 (Fubing SHEN), Email: shenfubing@163.com.
References
- 1.Rodrigues M, Petrova T, Tibbs B, et al. TAK1 protein kinase activity is required for TLR signalling and cytokine production in myeloid cells. Biochem J. 2022;479(17):1891–907. doi: 10.1042/BCJ20220314. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Szili D, Bankó Z, Tóth EA, et al. TGFβ activated kinase 1 (TAK1) at the crossroad of B cell receptor and Toll-like receptor 9 signaling pathways in human B cells. PLoS One. 2014;9(5):e96381. doi: 10.1371/journal.pone.0096381. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.Bao SY, Sun QX, Yao CL. The interaction of TAK1 and TAB1 enhances LPS-induced cytokine release via modulating NF-κB activation (Larimichthys crocea) Fish Shellfish Immunol. 2018;74:450–8. doi: 10.1016/j.fsi.2018.01.005. [DOI] [PubMed] [Google Scholar]
- 4.Xu YR, Lei CQ. TAK1-TABs complex: a central signalosome in inflammatory responses. Front Immunol. 2021;11:608976. doi: 10.3389/fimmu.2020.608976. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.Peng KJ, Deng NH, Meng Y, et al. Alpha-momorcharin inhibits proinflammatory cytokine expression by M1 macrophages but not anti-inflammatory cytokine expression by M2 macrophages. J Inflamm Res. 2022;15:4853–72. doi: 10.2147/JIR.S372306. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Hsu KS, Kao HY. Correction to: PML: regulation and multifaceted function beyond tumor suppression. Cell Biosci. 2018;8:18. doi: 10.1186/s13578-018-0213-7. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Lunardi A, Gaboli M, Giorgio M, et al. A role for PML in innate immunity. Genes Cancer. 2011;2(1):10–9. doi: 10.1177/1947601911402682. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Ananthalakshmi P, Samayamohan K, Chokalingam C, et al. Psst-2.0. Appl-Bioinformatics. 2005;4(2):141–5. doi: 10.2165/00822942-200504020-00008. [DOI] [PubMed] [Google Scholar]
- 9.Buchan DWA, Jones DT. The PSIPRED protein analysis workbench: 20 years on. Nucleic Acids Res. 2019;47(W1):W402–7. doi: 10.1093/nar/gkz297. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Elaheh KA, Ozra TM, Amirhossein S, et al. A systematic review on popularity, application and characteristics of protein secondary structure prediction tools. Curr Drug Discov Technol. 2019;16(2):159–72. doi: 10.2174/1570163815666180227162157. [DOI] [PubMed] [Google Scholar]
- 11.Nerli S, Sgourakis NG. Cs-Rosetta. Methods Enzymol. 2019;614:321–62. doi: 10.1016/bs.mie.2018.07.005. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Alford RF, Gray JJ. Membrane protein engineering with rosetta[M]//Computational Design of Membrane Proteins. New York: Humana, 2021: 43-57.
- 13.Velankar S, Burley SK, Kurisu G, et al. The protein data bank archive[M]//Structural Proteomics. New York: Humana, 2021: 3-21.
- 14.Fan X, He LL, Meng Y, et al. α-MMC and MAP30, two ribosome-inactivating proteins extracted from Momordica charantia, induce cell cycle arrest and apoptosis in A549 human lung carcinoma cells. Mol Med Rep. 2015;11(5):3553–8. doi: 10.3892/mmr.2015.3176. [DOI] [PubMed] [Google Scholar]
- 15.Deng NH, Li MH, Shen D, et al. LRP1 receptor-mediated immuno-suppression of α-MMC on monocytes. Int Immunopharmacol. 2019;70:80–7. doi: 10.1016/j.intimp.2019.01.036. [DOI] [PubMed] [Google Scholar]
- 16.Meng X, Chen Y, Macip S, et al. PML-Ⅱ regulates ERK and AKT signal activation and IFNα-induced cell death. Cell Commun Signal. 2021;19(1):70. doi: 10.1186/s12964-021-00756-5. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Ivanschitz L, De Thé H, Le Bras M. PML, SUMOylation, and senescence. Front Oncol. 2013;3:171. doi: 10.3389/fonc.2013.00171. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Datta N, Islam S, Chatterjee U, et al. Promyelocytic Leukemia (PML) gene regulation: implication towards curbing oncogenesis. Cell Death Dis. 2019;10(9):656. doi: 10.1038/s41419-019-1889-2. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Wu D, Huang D, Li LL, et al. TGF-β1-PML SUMOylation-peptidyl-prolyl cis-trans isomerase NIMA-interacting 1 (Pin1) form a positive feedback loop to regulate cardiac fibrosis. J Cell Physiol. 2019;234(5):6263–73. doi: 10.1002/jcp.27357. [DOI] [PubMed] [Google Scholar]
- 20.El-Asmi F, Chelbi-Alix MK. Les isoformes de PML et la réponse au TGF-Β. médecine. 2020;36(1):50–6. doi: 10.1051/medsci/2019269. [DOI] [PubMed] [Google Scholar]
- 21.Schoeder CT, Schmitz S, Adolf-Bryfogle J, et al. Modeling immunity with Rosetta: methods for antibody and antigen design. Biochemistry. 2021;60(11):825–46. doi: 10.1021/acs.biochem.0c00912. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22.Leman JK, Weitzner BD, Lewis SM, et al. Macromolecular mo-deling and design in Rosetta: recent methods and frameworks. Nat Methods. 2020;17(7):665–80. doi: 10.1038/s41592-020-0848-2. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.Pierce BG, Hourai Y, Weng ZP. Accelerating protein docking in ZDOCK using an advanced 3D convolution library. PLoS One. 2011;6(9):e24657. doi: 10.1371/journal.pone.0024657. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.Baxter EW, Graham AE, Re NA, et al. Standardized protocols for differentiation of THP-1 cells to macrophages with distinct M (IFNγ+ LPS), M(IL-4) and M(IL-10) phenotypes. J Immunol Methods. 2020;478:112721. doi: 10.1016/j.jim.2019.112721. [DOI] [PubMed] [Google Scholar]
- 25.Liu MT, Lu J, Yang YL, et al. Alliin alleviates LPS-induced pyroptosis via pro-moting mitophagy in THP-1 macrophages and mice. Food Chem Toxicol. 2022;160:112811. doi: 10.1016/j.fct.2022.112811. [DOI] [PubMed] [Google Scholar]
- 26.Greta F, Mariam S, Elise R, et al. Restriction factors in human retrovirus infections and the unprecedented case of CⅡTA as link of intrinsic and adaptive immunity against HTLV-1. Retrovirology. 2019;16(1):34. doi: 10.1186/s12977-019-0498-6. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27.Qian H, Chen L. TRIM proteins in fibrosis. Biomed Pharmacother. 2021;144:112340. doi: 10.1016/j.biopha.2021.112340. [DOI] [PubMed] [Google Scholar]
- 28.Rape M. Ubiquitylation at the crossroads of development and disease. Nat Rev Mol Cell Biol. 2018;19(1):59–70. doi: 10.1038/nrm.2017.83. [DOI] [PubMed] [Google Scholar]
- 29.Song L, Luo ZQ. Post-translational regulation of ubiquitin signaling. J Cell Biol. 2019;218(6):1776–86. doi: 10.1083/jcb.201902074. [DOI] [PMC free article] [PubMed] [Google Scholar]


