Abstract
耐药是癌症治疗面临的主要难题,明确肿瘤耐药机制和寻找新的药物靶点有重要的临床意义。多组学主要包括基因组学、表观基因组学、转录组学、蛋白质组学、代谢组学、影像组学等。近年来,肿瘤耐药研究从单组学到多组学联合,取得了飞速的发展,显著加快了肿瘤新的药物靶点的发现。
Keywords: 肿瘤耐药, 基因组学, 表观基因组学, 转录组学, 蛋白质组学, 代谢组学, 影像组学
Abstract
Drug resistance is the main obstacle in the treatment of many cancers. It is of great clinical significance to study the mechanism of drug resistance and find new targets. Multi-omics mainly includes genomics, epigenomics, transcriptomics, proteomics, metabolomics, and radiomics. In recent years, the research of tumor resistance has made rapid development, which has significantly accelerated the discovery of new targets.
Keywords: tumor resistance, genomics, epigenomics, transcriptomics, proteomics, metabolomics, radiomics
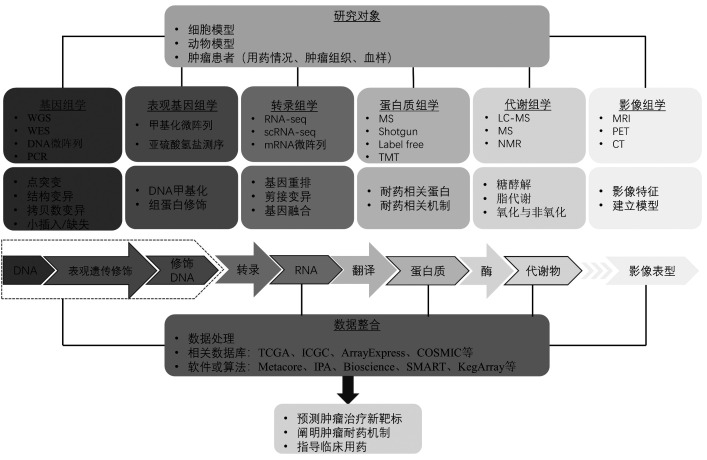
癌症的发病率和病死率逐年增加,目前癌症治疗的主要手段是手术治疗、放射治疗和化学药物治疗(以下简称化疗)。免疫治疗、分子靶向治疗、内分泌治疗是近年来出现的新型疗法。无论是哪种疗法,一旦出现耐药,都会对癌症患者产生严重影响。遗传信息是决定患者疗效的重要因素,它的任一环节发生变异均有可能导致耐药。基因组学是耐药机制研究的基础,是对全部基因进行表征和定量的研究,以期在DNA层面发现与耐药相关的基因突变。转录组学是在RNA水平寻找由基因融合、基因重排、剪接变异等导致的耐药。表观基因组学是从遗传物质的修饰角度研究耐药机制。蛋白质组学和代谢组学分别以蛋白质、代谢物为研究对象,反映耐药过程中其在生物体内的动态变化。影像组学是采用自动化算法从影像的感兴趣区内提取大量的特征信息进行定量分析,从而提供对诊断、治疗、预后有价值的信息。近年来,单组学在耐药领域得到广泛应用,但任一单组学都不足以系统地阐释耐药机制。多组学联合具有多元性、关联性、海量性、系统性等优势,有利于系统阐明肿瘤的耐药机制,推动精准治疗的发展。因此,多组学联合是肿瘤耐药领域发展的必然趋势(图1)。
图1.
组学技术在肿瘤耐药领域的应用和联合
Figure 1 Application and combination of omics technology in the field of tumor resistance
1. 基因组学
基因组学在肿瘤耐药领域被广泛应用,主要是通过测序技术对生物体内所有基因进行表征和定量,研究其对生命活动的影响。基因组学的测序技术主要包括全外显子测序(whole exon sequencing,WES)、全基因组测序(whole genome sequencing,WGS)、DNA微阵列技术。基因组学开创了组学时代的先河,更是多组学的根基。
目前从基因组学角度探索肿瘤耐药机制主要有以下几方面:1)点突变,是目前肿瘤耐药领域中发现的主要生物标志物,例如使用间充质向上皮转化因子(mesenchymal to epithelial transition factor,MET)酪氨酸激酶抑制剂(tyrosine kinase inhibitors,TKIs)的肺癌患者发生MET 14号外显子突变后,将对抑制剂产生耐药,TKIs根据结合区域不同分为I、II、III 3型,研究[1]发现携带D1228和Y1230点突变的患者对I型TKIs耐药,而携带L1195和F1200点突变的患者对II型TKIs耐药。针对不同的点突变类型给予不同类型的TKIs有助于提高疗效。2)结构变异,主要指染色体结构变异,变异类型有重复、倒位、易位,统称为染色体不稳定性,目前关于其与肿瘤耐药的研究较少。3)拷贝数变异,影响肿瘤耐药的拷贝数变异标志物较多,例如最近利用WGS发现在结直肠癌中酪氨酸激酶受体2扩增导致患者对靶向表皮生长因子受体(epidermal growth factor receptor,EGFR)和人类EGFR2(也称HER2)的药物耐药[2]。4)小插入/缺失,如肺癌患者发生EGFR20号外显子插入突变,则对EGFR靶向用药疗效不佳,但目前还没有更好的药物。人第10号染色体缺失的磷酸酶及张力蛋白同源的基因(phosphatase and tensin homolog deleted on chromosome ten,PTEN)、p53、细胞周期依赖性激酶抑制因子2A(cyclin dependent kinase inhibitor 2A,CDKN2A)等抑癌基因两个等位基因的缺失或失活,会使抑癌作用降低,从而促进肿瘤耐药。另外,种系突变也与肿瘤耐药相关,利用WES发现发生铁蛋白4(tektin 4,TEKT4)种系变异的乳腺癌患者会调节微管蛋白稳定性,拮抗紫杉醇的作用,引发耐药[3]。
随着高通量测序技术的深入发展,基因组学在临床上应用更加广泛,测序深度加深,准确性提高,费用降低,因此在多组学联用中,基因组学发挥了至关重要的作用,为多组学研究提供了系统性的基因全貌。
2. 表观基因组学
表观基因组学是指基于非基因序列改变所致基因表达水平的变化,包括DNA甲基化、组蛋白修饰、染色体重塑和非编码RNA调控等,主要通过调控基因转录或翻译过程,影响其功能和特性。其中DNA甲基化和组蛋白修饰对肿瘤耐药的影响最为重要。表观遗传修饰易受环境和药物的影响,具有动态性,其主要技术手段是靶向甲基化微阵列和亚硫酸氢盐测序。
DNA甲基化在DNA调控中起重要作用,位于启动子区或第1外显子区域的CpG岛的甲基化状态对基因表达的影响最为显著。甲基化的实现依靠甲基转移酶,抑制该酶活性可以降低甲基化水平。5-氮杂替丁和地西他滨属于甲基转移酶抑制剂,也有通过其他机制抑制DNA甲基化的药物,例如新型氧化表观遗传调节剂——RRx-001,在肿瘤低氧的环境中可催化亚硝酸盐生成一氧化氮,与超氧化物形成过氧亚硝酸盐,对肿瘤造成高氧和代谢压力,抑制甲基转移酶从而抑制整体的高甲基化水平,恢复抑癌基因的表达,并且不会对正常细胞造成影响,实现耐药逆转[4]。抑癌基因启动子区域发生高甲基化,则该基因表达被抑制。卵巢癌中肿瘤抑制因子对接蛋白2(docking protein 2,DOK2)、miR193a因启动子区域高甲基化沉默,使用卡铂治疗无效,而联用瓜地西滨则可降低甲基化水平,使DOK2、miR193a上调,铂类药物复敏[5]。同理,若高甲基化发生在原癌基因启动子区域,则原癌基因表达受到抑制,有助于提高药物敏感性。骨形态发生蛋白4(bone morphogenetic protein 4,BMP4)是胃癌原癌基因的一种,当启动子区域甲基化水平降低时,BMP4高表达,患者对顺铂耐药[6]。总的来说,抑癌基因CpG岛的高甲基化和原癌基因CpG岛的低甲基化均不利于肿瘤的化疗,但目前还没有针对原癌基因低甲基化的甲基化增强剂,因此,这也为药物研发提供了新思路。
组蛋白修饰主要通过甲基化、乙酰化、磷酸化、泛素化等修饰方式调节基因的转录活性。与肿瘤耐药相关的组蛋白甲基化修饰主要有H3K9me3、H3K27me3和H3K4me3修饰。例如赖氨酸脱甲基酶6B可导致RTK信号拮抗剂4(sprouty RTK signaling antagonist 4,SPRY4)和胰岛素样生长因子结合蛋白5(insulin like growth factor binding protein 5,IGFBP5)基因启动子区域的H3K4me3脱甲基,从而下调这2个基因的表达,负调控干扰素介导的信号通路[7]。另外赖氨酸脱甲基酶5A和5B[8]也与耐药相关。
3. 转录组学
转录组是指生物体内所有转录产物的总和,主要涉及的RNA有mRNA、ncRNA、miRNA、 lncRNA、rRNA和tRNA,转录组学研究依靠RNA测序(RNA sequencing,RNA-seq)技术,其具有低背景噪音、定量、精确的优点[9]。
目前测序技术已经发展到单细胞测序(single-cell RNA sequencing,scRNA-seq)。 scRNA-seq可以从单细胞层面更加精确地揭示肿瘤内与肿瘤间的异质性。Hong等[10]利用scRNA-seq和活细胞成像,在接受内分泌治疗的乳腺癌患者中发现耐药组和敏感组的可塑性细胞和前适应细胞存在显著的异质性,推测其是导致内分泌治疗抵抗的主要原因。scRNA-seq技术还可识别出对药物耐药的细胞亚群,Ho等[11]在黑色素瘤中利用scRNA-seq识别出对靶向BRAF抑制剂耐药的细胞亚群,发现耐药细胞群里多巴色素互变异构酶(dopachrome tautomerase,DCT)高表达,可作为新型抗性标志物。虽然scRNA-seq推动了研究的深入发展,但也存在很多亟待解决的问题,比如转录本丢失率高、全长转录本以及低转录本难以检测等。
在转录水平,基因重排、剪接变异、基因融合等会导致耐药的发生。基因重排改变基因与启动子之间的距离,从而影响基因的转录活性;而基因融合是指2个及以上的基因在同一套调控序列下以首尾相连的方式表达;剪接变异在肿瘤耐药中较为常见,是指在原始RNA剪接形成成熟RNA的过程中发生错误,导致成熟RNA中含有内含子或者缺失外显子。研究[12]报道部分接受雄激素剥夺疗法的前列腺癌患者出现耐药,其机制可能与跨膜丝氨酸蛋白酶 2(transmembrane serine protease 2,TMPRSS2)与成红细胞病毒E26致癌物(E26 oncogene,ERG)融合基因及形成雄激素受体非依赖性剪接变体7(androgen receptor variant7,AR-V7)有关。另有研究[13]报道非小细胞肺癌患者存在神经营养受体酪氨酸激酶1(neurotrophic receptor tyrosine kinase 1,NTRK1)和ROS原癌基因1(ROS proto-oncogene 1,ROS1)重排,会对靶向药物产生耐药。Heining等[14]发现晚期胰腺导管腺癌KRAS野生型患者对小分子TKIs耐药与神经调节蛋白1(neuregulin 1,NRG1)融合有关。
然而,由于分析技术、测序深度、时间、经济成本等原因,目前临床测序工作还未纳入RNA-seq。相信随着技术的成熟与发展,RNA-seq在临床工作中将得到广泛应用。
4. 蛋白质组学
蛋白质组学可以提供整体蛋白质丰度、翻译后修饰(post-translational modifications,PTMs)、蛋白质功能、蛋白质相互作用和蛋白质位置等信息,其中PTMs主要包括糖基化、乙酰化和磷酸化等。蛋白质组学的技术体系可分为定性、定量、修饰技术3类。定性技术为鸟枪法(shotgun),目的是鉴定蛋白质种类,常用于免疫沉淀和免疫共沉淀产物的鉴定;定量技术是比较不同样本中相同蛋白质的表达量,一种技术是非标记(label free)定量蛋白质组学,用于大队列分析,另一种是标记定量蛋白质组学,较前者更灵敏、准确[15];修饰技术用来确定修饰位点,比较不同样本中修饰程度的变化。蛋白质组学已经发展至微型化,微型蛋白质组学技术仅需要25 µg的多肽即可分析出蛋白质谱,可排除非目的样本的干扰。另外,机器学习算法可将细胞或组织中获得的蛋白质量化,再结合数字病理学、机器建模、基因组学等,有助于生物标志物的发现[16]。
蛋白质作为生命活动的主要承担者,与表型直接相关。近年来利用蛋白质组学已经发现了许多与耐药相关的蛋白质分子及其机制。在乳腺癌中经液质联用质谱(liquid chromatography-mass spectrometry,LC-MS)/质谱(mass spectrometry,MS)检测后发现成纤维细胞生长因子2(fibroblast growth factor 2,FGF2)与乳腺癌雌激素耐药有关,再经磷酸化蛋白质组学分析,发现FGF2信号通过FGF受体激活ERK1/2,从而下调Bcl样因子,上调细胞周期蛋白D1(cyclin D1),影响细胞周期而导致雌激素耐药[17]。已经报道的与肿瘤耐药相关的蛋白质有多药耐药相关蛋白、ATP结合盒式蛋白、谷胱甘肽转移酶、拓扑异构酶、P-糖蛋白等,近年来通过蛋白质组学又发现了许多新的与耐药相关的蛋白质,例如对乳腺癌患者的肿瘤标本进行蛋白质组学分析,发现黏蛋白与患者耐药相关[18];在对铂类药物耐药的卵巢癌患者中发现ENOA、EFTU、G3P、GRP75、APOA1、PRDX2和ANXA等蛋白质可作为耐药相关的生物标志物[19];而在铂类敏感的卵巢癌中发现癌症/睾丸抗原家族45(cancer/testis antigen family 45,CT45)高表达,磷酸化蛋白质组学和蛋白质相互作用实验表明CT45可直接调节蛋白磷酸酶4(protein phosphatase 4,PP4)活性,PP4表达升高导致DNA损伤增加,使得患者对铂类药物敏感性增加[20]。在不同肿瘤中运用蛋白质组学发现的与耐药相关的诸多蛋白质分子,它们既可作为预测药物疗效的生物标志物,也可作为药物研发的靶标,并且合并使用相关蛋白质抑制剂可提高化疗患者的药物敏感性。
5. 代谢组学
代谢物作为肿瘤进程中的动态标志物,可反映肿瘤发生发展的过程。主要检测手段是LC-MS、MS及磁共振(magnetic resonance,MR)。代谢组学与耐药主要通过糖酵解、脂代谢、氧化和非氧化过程等产生联系。
代谢组学可以从代谢产物水平区分患者对铂类是否耐药,避免无意义的治疗。Poschner等[21]利用LC-MS表征卵巢癌发生铂类耐药时类固醇、活性雌激素、硫酸化或醛酸化葡萄糖的水平,发现这些代谢产物在卡铂敏感细胞中高表达。代谢组学也可揭示多条与肿瘤耐药相关的代谢途径。例如结直肠癌的巨噬细胞可诱导产生腐胺,从而通过JNK-caspase-3通路对5-FU产生耐药性;而抑制鸟氨酸脱羧酶可抑制巨噬细胞产生腐胺,从而缓解耐药[22]。Gebregiworgis等[23]利用HSQC MR技术研究发现:胰腺癌患者对吉西他滨耐药与葡萄糖代谢重编程和嘧啶合成途径上调相关,原本用于合成葡萄糖的碳元素参与了嘧啶的合成,进一步合成核苷酸;核苷酸浓度上升抑制吉西他滨疗效。
抗代谢药物的上市在一定程度上缓解了化疗耐药的现状。JAK/STAT3可以调节脂肪酸β氧化(fatty acid β-oxidation,FAO)中的关键酶肉碱棕榈酰转移酶(carnitine palmitoyltransferase 1B,CPT1B)的表达,从而调节脂质代谢。对乳腺癌的研究[24]表明:JAK/STAT3-CPT1B-FAO通路会促进干细胞的自我更新能力和化疗耐药,派克昔林是FAO抑制剂,合并使用能逆转耐药。在对非小细胞肺癌的研究[25]中,顺铂耐药细胞比敏感细胞对营养剥夺更敏感,若在顺铂耐药细胞中添加谷氨酰胺则会通过提高细胞内的核苷酸浓度而恢复营养剥夺导致的细胞死亡。因此,顺铂耐药患者合用靶向核苷代谢的药物如5-FU,可以提高疗效。Ricci等[26]发现在卵巢癌人源肿瘤异种移植模型中,耐药组与敏感组中的糖酵解相关基因表达没有差异,但是耐药组在糖酵解、三羧酸循环、尿素循环这几个代谢环节中存在失调,且耗氧率和线粒体修复高于敏感组,给予二甲双胍处理后能改善代谢从而逆转耐药。
代谢组学在肿瘤耐药基因方面的研究已经有了长足的进步,但仍然存在缺点和不足,尤其在研究过程中如何克服食物和环境对代谢产物的影响还是亟待解决的难题。虽然代谢组学是在基因组学和转录组学之后产生的,但由于具有价格低廉、检测范围广泛等特点,必将成为基因组学和转录组学这两大经典组学的重要补充。
6. 影像组学
影像组学是将影像内包含的所有信息提取出来然后进行综合分析,并进一步采用多样化的统计分析和数据挖掘方法从大批量信息中提取和剥离出起作用的关键信息,最终用于疾病的辅助诊断、分类或分级、肿瘤预测、治疗方案选择等。主要步骤是收集图像、分割感兴趣区、提取特征、统计分析、分类预测等。数据是否可重现是影像组学面临的主要问题,厂家仪器、重建参数或扫描时间点不同都有可能影响模型的适用性。
Yang等[27]通过提取新辅助放化疗敏感或耐药的直肠腺癌患者的影像学特征,发现两组患者在间质液压和缺氧两方面的影像学特征存在差异,因此可以根据表观扩散系数这一影像学指标建立分类模型,来预测患者的新辅助放化疗疗效。同样,在新辅助化疗开始前可以根据乳腺癌晚期患者MRI质构分析构建模型来预测化疗反应[28-29]。在脑胶质瘤中也可利用MRI建模判定肿瘤对免疫检查点抑制剂的反应[30]。
实体瘤在基因、蛋白质、细胞、组织、器官等不同水平均有时间和空间的异质性,这限制了基于活检的分子检测手段的使用,相比之下,影像组学具有非侵入性和普遍性,在临床应用方面有巨大的潜力。近年来,研究者多将影像中提取的数据与患者的其他数据整合起来,利用多种生物学工具建立模型,实现诊断、预判患者疗效等目的。影像组学还可利用多项技术在影像资料中挖掘定量的影像学特征,以利于提高诊断和疗效预测的准确性。随着影像组学的发展,将数字图像转化为可挖掘数据在临床上将成为常态。
7. 多组学联合在肿瘤耐药领域的应用
肿瘤发生发展机制十分复杂,耐药机制更为错综纷繁,任一单组学都无法系统地揭示耐药机制,而多组学联合有助于从系统和整体角度深入理解耐药发生发展的机制,发现更多用于诊断、预后和治疗的生物标志物,促进新型药物的开发,推动肿瘤耐药研究领域的发展。
多组学联合的优势主要体现在精度、广度、深度3个方面。1)精度优势体现在缩小了生物标志物的筛选范围,提高了结果的准确性。例如研究者收集对TKIs耐药患者的胸腔积液,进行定量蛋白质组学分析,将其结果与基因表达谱进行交互分析,从两组学数据交集中寻找生物标志物可缩小筛选范围,结果发现钙黏蛋白3(cadherin-3)既可以在耐药组织中过表达,促进上皮间质转化过程,从而导致耐药[31],还可以调节肿瘤生长和细胞迁移[32]。Briffa等[33]先整合DNA拷贝数信息和基因表达数据得到候选基因,再结合反向蛋白质阵列数据验证确定tribbles同源物1(tribbles pseudokinase 1,TRIB1)扩增、程序性细胞死亡6因子(programmed cell death 6,PDCD6)过表达与结直肠癌化疗耐药相关,该研究联合基因组学、转录组学和蛋白质组学,提高了结果的精准性。2)广度优势体现在多组学涵盖生命活动的各个环节,包括DNA复制、DNA表观修饰、RNA转录、蛋白质翻译、代谢过程、最终表型等,还体现在挖掘信息的全面性。在头颈部鳞状细胞癌患者中,利用靶向DNA测序、转录组微阵列、磷酸化蛋白质谱分析等技术,发现患者对EGFR抑制剂耐药相关的原因有17号染色体非整倍性、TP53拷贝数增加、p53过表达、PI3K-AKT-mTOR通路活性增加[34]。影像学技术和基因测序都是广泛应用于临床的技术手段,影像学技术的非侵入性也使患者易于接受,因此将两组学结合有很大的可行性,在临床上有较大的发展前景。有研究者[35]在肝癌患者中利用DNA微阵列技术和CT技术研究耐药机制,结果发现:CT影像特征与使用阿霉素的患者基因表达情况相对应,可根据已知耐药基因对应的影像特征预判患者对阿霉素的反应。利用影像组学非侵入的特点,联合基因组学有望实现对患者用药后基因情况的全程监测,增加多组学监测的时间广度。3)深度优势体现在对肿瘤耐药机制的深入挖掘。聚ADP核糖聚合酶(poly ADP-ribose polymerase,PARP)抑制剂可用于治疗同源重组缺陷型癌症,但耐药成为其在临床广泛应用的主要障碍。Gogola等[36]首先利用短发夹RNA文库在乳腺癌细胞系和类器官中筛选功能缺失型突变,发现聚ADP-核糖水解酶(poly ADP-ribose glycohydrolase,Parg)缺失与PARP抑制剂耐药相关。为了进一步验证此相关,作者在乳腺癌移植瘤小鼠模型中进行RNA-seq和DNA-seq,获得基因表达数据和拷贝数变异信息,通过交集获得82个候选因子,与体外筛选一致,Parg在候选基因首位;最后利用组学信息发现抑制Parg会使PARP下游信号恢复,ADP-核糖聚合物形成,从而募集DNA修复因子,促进耐药。
Li等[37]利用多组学探索了胃癌患者对于新辅助化疗(5-FU+奥沙利铂)耐药的机制,首先对成对胃癌临床标本进行WGS、WES和RNA-seq,数据经MANTIS分析发现耐药患者的微卫星不稳定性增加,突变负荷增加;MuSiC2分析得到显著突变基因,其中C10oef71突变明显,耐药性增加,进行反相蛋白质阵列,发现该基因主要影响细胞周期;GISTIC2.0分析发现耐药患者MYC、MDM2扩增,且经GSEA分析发现这两个基因分别参与MYC通路和DNA修复通路;经HotNet2分析蛋白质相互作用网络发现耐药患者中胰岛素受体底物1蛋白水平升高,该蛋白质主要参与PI3K/AKT通路;TIMER分析发现耐药患者的肿瘤微环境发生重塑。该研究涉及基因组学、转录组学、蛋白质组学及多个生物信息学分析软件或算法,全方位探讨了胃癌患者的化疗耐药机制。
在进行多组学实验设计时应明确组学联合分析目标,从而确定各个组学层面的数据收集标准和分析方法。例如在转录组学中,应在经济允许范围内根据目标优化测序样本量和测序深度。目标若为发现更多差异基因,则应增加样本量;目标若为发现新的转录本,则应加深测序深度。其他组学与之类似[38]。多组学的核心是各组学信息的关联,信息流动是其本质。研究思路多为交集和验证,不同组学数据取交集往往能发现与肿瘤耐药相关的某一表型及相关因子,可进一步分析其内在逻辑,或者确定引起肿瘤耐药的基因或其他变量,再利用其他组学进行验证并识别下游通路和相互作用的因子。各组学之间重叠不广泛,没有很好的相关性,是目前多组学面临的关键问题,有2种潜在的解决方法:一是着眼于基因、表观遗传修饰基因、转录因子、蛋白质、代谢物等因子,利用多组学信息寻找其相关性,ENCODE数据库已经实现将基因表达与DNA序列变异、表观遗传变异和染色质可及性相关联;二是在已有的通路或者相互作用网络里整合多组学数据,发现新的与肿瘤耐药相关的因子,该过程是无偏分析,数据量足够大,在计算和统计上具有挑战性[39]。另外由于组织样本具有固有异质性,多组学技术测得的数据存在一定误差,多采用批量测定的方法以减少技术误差[40],因此高通量单细胞测序对于提高多组学数据精度有重要意义。目前在基因组学、转录组学、表观遗传组学已有相应的单细胞测序技术,蛋白质组学和代谢组学的单细胞测序技术指日可待。
随着高通量技术的发展,多组学的分析方法不断进步,随之衍生出大量的生物医学数据。这些数据庞大、复杂且分散,尚缺乏有效的标准化的分析方法,导致诸多信息浪费。有研究者整合药物特征(包括化学结构、靶标、作用通路、蛋白质功能域、相互作用网络)和细胞系特征(包括基因表达、突变图谱、拷贝数等),在可获取的开放性多组学基础上建立机器学习模型,用来评估结果并作出合理的生物学解释[41]。各个组学的数据库有TCGA、ICGC、ArrayExpress、COSMIC等,整合多组学的生信分析软件有Metacore、IPA、Bioscience、SMART、KegArray[42]。我们可以从数据库中获得已有的组学数据,通过各种方法整合分析,得出新的肿瘤耐药观点,基于系统生物学方法建立模型,帮助提高抗癌药物疗效,克服癌症的化疗或免疫疗法的抗性。数据持续可用是多组学联合的最大优势。多组学的未来发展方向除了优化技术外,还应发展全新的分析和统计方法。
8. 展 望
单一组学的分析思路已经相当成熟,且获得的结果明确;而多组学是整合单一组学的结果,目的是系统解答生物学问题,阐述功能与机制。组学技术可以表征肿瘤细胞的生物标志物,从而推进个体化治疗。随着测序等各项技术的不断优化,组学领域迎来前所未有的机遇,影像学资料的引入、单细胞测序分析、宏基因组学的深入为多组学带来了新数据,利用人工自能算法等新技术进行高维度、多组学的数据整合分析可提高模型的精度,另外也可运用新型算法在已有数据中挖掘新信息、新规律。然而,组学技术在癌症领域的应用仍具有挑战性,虽然我们从患者样本中获得了大量的组学数据,但是有意义或者高质量的却很少,且各数据集之间存在异质性。此外,肿瘤是动态发展的,组学技术只能反映肿瘤某一时间的情况,监测动态变化耗费巨大。相信随着各项技术的发展与成熟,成本逐渐下降,多组学的应用将愈加广泛,在未来多组学将成为化疗耐药的先导应用,推进精准医学和肿瘤药物研发的进展。
基金资助
国家自然科学基金(81974512)。
This work was supported by the National Natural Science Foundation of China (81974512).
利益冲突声明
作者声称无任何利益冲突。
原文网址
http://xbyxb.csu.edu.cn/xbwk/fileup/PDF/202106620.pdf
参考文献
- 1. Fujino T, Kobayashi Y, Suda K, et al. Sensitivity and resistance of MET exon 14 mutations in lung cancer to eight MET tyrosine kinase inhibitors in vitro[J]. J Thorac Oncol, 2019, 14(10): 1753-1765. [DOI] [PubMed] [Google Scholar]
- 2. Owen DR, Wong HL, Bonakdar M, et al. Molecular characterization of ERBB2-amplified colorectal cancer identifies potential mechanisms of resistance to targeted therapies: a report of two instructive cases[J]. Cold Spring Harb Mol Case Stud, 2018, 4(2): a002535. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3. Zaborsky N, Gassner FJ, Höpner JP, et al. Exome sequencing of the TCL1 mouse model for CLL reveals genetic heterogeneity and dynamics during disease development[J]. Leukemia, 2019, 33(4): 957-968. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4. Das DS, Ray A, Das A, et al. A novel hypoxia-selective epigenetic agent RRx-001 triggers apoptosis and overcomes drug resistance in multiple myeloma cells[J]. Leukemia, 2016, 30(11): 2187-2197. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5. Fang F, Cardenas H, Huang H, et al. Genomic and epigenomic signatures in ovarian cancer associated with resensitization to platinum drugs[J]. Cancer Res, 2018, 78(3): 631-644. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6. Ivanova T, Zouridis H, Wu YH, et al. Integrated epigenomics identifies BMP4 as a modulator of cisplatin sensitivity in gastric cancer[J]. Gut, 2013, 62(1): 22-33. [DOI] [PubMed] [Google Scholar]
- 7. Emran AA, Marzese DM, Menon DR, et al. Commonly integrated epigenetic modifications of differentially expressed genes lead to adaptive resistance in cancer[J]. Epigenomics, 2019, 11(7): 732-737. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8. Hinohara K, Wu HJ, Vigneau S, et al. KDM5 histone demethylase activity links cellular transcriptomic heterogeneity to therapeutic resistance[J]. Cancer Cell, 2018, 34(6): 939-953. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9. Orgaz JL, Crosas-Molist E, Sadok A, et al. Myosin II reactivation and cytoskeletal remodeling as a hallmark and a vulnerability in melanoma therapy resistance[J]. Cancer Cell, 2020, 37(1): 85-103. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10. Hong SP, Chan TE, Lombardo Y, et al. Single-cell transcriptomics reveals multi-step adaptations to endocrine therapy[J]. Nat Commun, 2019, 10(1): 3840. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11. Ho YJ, Anaparthy N, Molik D, et al. Single-cell RNA-seq analysis identifies markers of resistance to targeted BRAF inhibitors in melanoma cell populations[J]. Genome Res, 2018, 28(9): 1353-1363. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12. Beltran H, Wyatt AW, Chedgy EC, et al. Impact of therapy on genomics and transcriptomics in high-risk prostate cancer treated with neoadjuvant docetaxel and androgen deprivation therapy[J]. Clin Cancer Res, 2017, 23(22): 6802-6811. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13. Chong CR, Bahcall M, Capelletti M, et al. Identification of existing drugs that effectively target NTRK1 and ROS1 rearrangements in lung cancer[J]. Clin Cancer Res, 2017, 23(1): 204-213. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14. Heining C, Horak P, Uhrig S, et al. NRG1 fusions in KRAS wild-type pancreatic cancer[J]. Cancer Discov, 2018, 8(9): 1087-1095. [DOI] [PubMed] [Google Scholar]
- 15. Li HH, Meng FQ, Jiang L, et al. Comparison of LC-MS/MS-based targeted proteomics and conventional analytical methods for monitoring breast cancer resistance protein expression[J]. Life Sci, 2019, 231: 116548. [DOI] [PubMed] [Google Scholar]
- 16. Gil J, Betancourt LH, Pla I, et al. Clinical protein science in translational medicine targeting malignant melanoma[J]. Cell Biol Toxicol, 2019, 35(4): 293-332. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17. Cheng FX, Zhao JF, Hanker AB, et al. Transcriptome-and proteome-oriented identification of dysregulated eIF4G, STAT3, and Hippo pathways altered by PIK3CA H1047R in HER2/ER-positive breast cancer[J]. Breast Cancer Res Treat, 2016, 160(3): 457-474. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18. Satpathy S, Jaehnig EJ, Krug K, et al. Microscaled proteogenomic methods for precision oncology[J]. Nat Commun, 2020, 11(1): 532. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19. Cruz IN, Coley HM, Kramer HB, et al. Proteomics analysis of ovarian cancer cell lines and tissues reveals drug resistance-associated proteins[J]. Cancer Genomics Proteomics, 2017, 14(1): 35-51. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20. Coscia F, Lengyel E, Duraiswamy J, et al. Multi-level proteomics identifies CT45 as a chemosensitivity mediator and immunotherapy target in ovarian cancer[J]. Cell, 2018, 175(1): 159-170. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21. Poschner S, Wackerlig J, Castillo-Tong DC, et al. Metabolism of estrogens: turnover differs between platinum-sensitive and - resistant high-grade serous ovarian cancer cells[J]. Cancers (Basel), 2020, 12(2): E279. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22. Zhang X, Chen YJ, Hao Li, et al. Macrophages induce resistance to 5-fluorouracil chemotherapy in colorectal cancer through the release of putrescine[J]. Cancer Lett, 2016, 381(2): 305-313. [DOI] [PubMed] [Google Scholar]
- 23. Gebregiworgis T, Bhinderwala F, Purohit V, et al. Insights into gemcitabine resistance and the potential for therapeutic monitoring[J]. Metabolomics, 2018, 14(12): 156. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24. Wang TY, Fahrmann JF, Lee H, et al. JAK/STAT3-regulated fatty acid β-oxidation is critical for breast cancer stem cell self-renewal and chemoresistance[J]. Cell Metab, 2018, 27(6): 1357. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25. Obrist F, Michels J, Durand S, et al. Metabolic vulnerability of cisplatin-resistant cancers[J]. EMBO J, 2018, 37(14): e98597. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26. Ricci F, Brunelli L, Affatato R, et al. Overcoming platinum-acquired resistance in ovarian cancer patient-derived xenografts[J]. Ther Adv Med Oncol, 2019, 11: 1758835919839543. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27. Yang C, Jiang ZK, Liu LH, et al. Pre-treatment ADC image-based random forest classifier for identifying resistant rectal adenocarcinoma to neoadjuvant chemoradiotherapy[J]. Int J Colorectal Dis, 2020, 35(1): 101-107. [DOI] [PubMed] [Google Scholar]
- 28. Sala E, Mema E, Himoto Y, et al. Unravelling tumour heterogeneity using next-generation imaging: radiomics, radiogenomics, and habitat imaging[J]. Clin Radiol, 2017, 72(1): 3-10. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29. Teruel JR, Heldahl MG, Goa PE, et al. Dynamic contrast-enhanced MRI texture analysis for pretreatment prediction of clinical and pathological response to neoadjuvant chemotherapy in patients with locally advanced breast cancer[J]. NMR Biomed, 2014, 27(8): 887-896. [DOI] [PubMed] [Google Scholar]
- 30. Aslan K, Turco V, Blobner J, et al. Heterogeneity of response to immune checkpoint blockade in hypermutated experimental gliomas[J]. Nat Commun, 2020, 11(1): 931. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 31. Hsiao TF, Wang CL, Wu YC, et al. Integrative omics analysis reveals soluble cadherin-3 as a survival predictor and an early monitoring marker of EGFR tyrosine kinase inhibitor therapy in lung cancer[J]. Clin Cancer Res, 2020, 26(13): 3220-3229. [DOI] [PubMed] [Google Scholar]
- 32. Siret C, Dobric A, Martirosyan A, et al. Cadherin-1 and cadherin-3 cooperation determines the aggressiveness of pancreatic ductal adenocarcinoma[J]. Br J Cancer, 2018, 118(4): 546-557. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 33. Briffa R, Um I, Faratian D, et al. Multi-scale genomic, transcriptomic and proteomic analysis of colorectal cancer cell lines to identify novel biomarkers[J]. PLoS One, 2015, 10(12): e0144708. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 34. Niehr F, Eder T, Pilz T, et al. Multilayered omics-based analysis of a head and neck cancer model of cisplatin resistance reveals intratumoral heterogeneity and treatment-induced clonal selection[J]. Clin Cancer Res, 2018, 24(1): 158-168. [DOI] [PubMed] [Google Scholar]
- 35. Kuo MD, Gollub J, Sirlin CB, et al. Radiogenomic analysis to identify imaging phenotypes associated with drug response gene expression programs in hepatocellular carcinoma[J]. J Vasc Interv Radiol, 2007, 18(7): 821-831. [DOI] [PubMed] [Google Scholar]
- 36. Gogola E, Duarte AA, de Ruiter JR, et al. Selective loss of PARG restores PARylation and counteracts PARP inhibitor-mediated synthetic lethality[J]. Cancer Cell, 2019, 35(6): 950-952. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 37. Li Z, Gao X, Peng X, et al. Multi-omics characterization of molecular features of gastric cancer correlated with response to neoadjuvant chemotherapy[J]. Sci Adv, 2020, 6(9): eaay4211. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 38. Hasin Y, Seldin M, Lusis A. Multi-omics approaches to disease[J]. Genome Biol, 2017, 18(1): 83. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 39. Olivier M, Asmis R, Hawkins GA, et al. The need for multi-omics biomarker signatures in precision medicine[J]. Int J Mol Sci, 2019, 20(19): E4781. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 40. Yimit A, Adebali O, Sancar A, et al. Differential damage and repair of DNA-adducts induced by anti-cancer drug cisplatin across mouse organs[J]. Nat Commun, 2019, 10(1): 1-11. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 41. Celebi R, Bear Don’t Walk O 4th, Movva R, et al. In-silico prediction of synergistic anti-cancer drug combinations using multi-omics data[J]. Sci Rep, 2019, 9(1): 8949. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 42. Deyati A, Younesi E, Hofmann-Apitius M, et al. Challenges and opportunities for oncology biomarker discovery[J]. Drug Discov Today, 2013, 18(13/14): 614-624. [DOI] [PubMed] [Google Scholar]