Abstract
针对医学图像分割中U型网络(U-Net)及其变体下采样过程中单尺度信息丢失、模型参数量较大的问题,本文提出了一种基于像素编码和空间注意力的多尺度医学图像分割方法。首先,通过重新设计变换器(Transformer)结构输入策略,提出了像素编码模块,使模型能够从多尺度图像特征中提取全局语义信息,获取更丰富的特征信息,同时在Transformer模块中引入可变形卷积,加快收敛速度的同时提升模块性能。其次,引入空间注意力模块并加入残差连接,使模型能够重点关注融合后特征图的前景信息。最后,通过消融实验实现网络轻量化并提升分割精度,加快模型收敛。本文所提算法在国际计算机医学图像辅助协会官方公开多器官分割公共数据集——突触(Synapse)数据库中得到令人满意的结果,戴斯相似性系数(DSC)和95%豪斯多夫距离系数(HD95)分别为77.65和18.34。实验结果表明,本文算法能够提高多器官分割结果,有望完善多尺度医学图像分割算法的空白,并为专业医师提供辅助诊断。
Keywords: 医学图像分割, U型网络, 变换器, 多尺度语义信息, 注意力模块
Abstract
In response to the issues of single-scale information loss and large model parameter size during the sampling process in U-Net and its variants for medical image segmentation, this paper proposes a multi-scale medical image segmentation method based on pixel encoding and spatial attention. Firstly, by redesigning the input strategy of the Transformer structure, a pixel encoding module is introduced to enable the model to extract global semantic information from multi-scale image features, obtaining richer feature information. Additionally, deformable convolutions are incorporated into the Transformer module to accelerate convergence speed and improve module performance. Secondly, a spatial attention module with residual connections is introduced to allow the model to focus on the foreground information of the fused feature maps. Finally, through ablation experiments, the network is lightweighted to enhance segmentation accuracy and accelerate model convergence. The proposed algorithm achieves satisfactory results on the Synapse dataset, an official public dataset for multi-organ segmentation provided by the International Conference on Medical Image Computing and Computer Assisted Intervention (MICCAI), with Dice similarity coefficient (DSC) and 95% Hausdorff distance (HD95) scores of 77.65 and 18.34, respectively. The experimental results demonstrate that the proposed algorithm can enhance multi-organ segmentation performance, potentially filling the gap in multi-scale medical image segmentation algorithms, and providing assistance for professional physicians in diagnosis.
Keywords: Medical image segmentation, U-Net, Transformer, Multi-scale semantic information, Attention module
0. 引言
医学图像分割是计算机视觉领域中的一项关键任务。近年来,随着医学成像设备快速发展、普及,成像技术在临床中得到广泛应用。作为医学图像可视化的关键,医学图像分割能够从原始医学图像中提取重要信息,凸显病变组织,进一步提供给医师进行后续的诊断以及治疗。然而,医学图像信息量大、数量多,人工对其进行标记的效率低下且准确率较低。因此,使用机器代替人工对医学图像进行处理成为机器学习领域研究的主流之一[1]。
早期的医学图像分割方法,主要是基于专家知识,以手工设计的特征进行分割。这些方法根据图像的灰度值、目标的纹理和边界等特征,采用阈值分析[2-4]、聚类检测[5-6]、边缘检测[7-9]、基于区域的分割[10-11]等方法来进行分割。例如,Masood等[12]首先对图像进行平滑滤波,然后利用C均值与聚类中心的相似性度量函数来优化每个像素的分类。然而,这种人为设计特征的方法需要大量的先验知识,且只能在特定的数据集上生效,分割性能不稳定、普适性较差。
近年来,深度神经网络凭借其优异的性能在图像处理任务中得到了非常广泛的应用。大多数的医学图像分割模型依赖于卷积神经网络(convolutional neural network,CNN)架构。Long等[13]首先将线性层全部替换为卷积层,提出了一种全卷积网络(fully convolutional network,FCN),开创了语义分割的先河。Ronneberger等[14]将压缩路径和扩张路径对称化,提出了U型网络(U-Net)结构,该网络结构简单、参数量小、收敛速度快,在医学图像分割任务上表现出色,成为医学分割任务的基准模型。在U-Net的基准上,Zhou等[15]在编码器和解码器之间引入密集跳跃连接,弥补下采样过程中信息的丢失,提出了巢穴U-Net(U-Net++)。Qin等[16]进一步增大网络规模,将U-Net每一层的卷积操作都更换为一个小型U-Net网络,提出了一种二维U-Net(U2Net)。Oktay等[17]引入注意力机制,加强模型的空间感知能力,提出了注意力U-Net(attention U-Net,Atten-UNet)。Jin等[18]引入残差和注意力相结合的方式提出了残差Atten-UNet(residual Atten-UNet,RA-UNet),在多器官分类中取得良好成绩。后续,仍有多项研究进行了跳跃连接或者注意力等方面的改进,但由于CNN仍存在固有的局限性:卷积操作的感受野有限,使得它只能从图像中捕获局部信息,导致了其对全局信息以及空间细节的忽视,在纹理、形状和大小等特征方面的学习能力较为有限。
变换器(Transformer)是为了提取语言中的全局语义信息而提出的大语义网络模型[19],具有优秀的全局语义提取能力以及空间自适应聚合信息的能力,受到研究人员广泛关注。Dosovitskiy等[20]通过位置编码以及补丁嵌入,将图像转化为序列,从而将Transformer引入到计算机视觉领域,提出了视觉Transformer(vision Transformer,ViT)。受ViT模型的启发,Chen等[21]将U-Net的下采样模块替换为Transformer模块,并跳跃连接来自残差网络(residual net,ResNet)中提取出的初步特征,提出了Transformer与U-Net相结合的网络(Transformer U-Net,TransUNet)结构模型。Wang等[22]则在TransUNet的基础上进一步对跳跃连接进行思考以及修改,使模型可以学到更多的特征。但以上算法依然存在不足:①尽管引入跳跃连接,下采样的过程本质上仍然是单尺度特征图的提取过程,特征信息容易丢失,导致分割精度下降;②上采样模块使用了简单的卷积,学习能力较差;③使用了大量的Transformer模块,参数较多,计算量大。
针对上述问题,本文提出一种基于像素编码和空间注意力的多尺度医学图像分割网络。该网络以TransUNet原始框架作为出发点,通过重新设计输入策略将单尺度下采样优化为多尺度特征提取,同时对网络进行轻量化调整,减小网络参数,加快模型收敛,以期解决单信息尺度特征丢失、网络模型大、难以收敛等问题,有望完善多尺度医学图像分割算法的空白,今后可为专业医师提供辅助诊断。
1. 算法描述
本文以TransUNet模型为基础,结合特征金字塔结构、可变形注意力以及空间注意力等思想,提出了一种基于像素编码和空间注意力的多尺度医学图像分割方法,模型的整体架构如图1所示。其网络模型主要包括四个模块,即:骨干网络、像素编码模块、上采样模块、分割头。不同尺寸大小的灰度图片在预处理阶段统一进行彩色化、裁剪以及反转等处理,最后统一输出通道数为3,尺寸大小为224 × 224的图片,并送入骨干网络提取特征。骨干网络,采用在计算机视觉与模式识别协会官方公开的大型可视化图像网(ImageNet)数据集上进行预训练的50层ResNet(ResNet50)模型[23-24],该网络模型设置了5个瓶颈层,可以逐层提取输入图像的语义特征信息,并同时输出不同分辨率的原始特征。像素编码模块,首先将来自骨干网络的不同分辨率特征进行补丁嵌入、拼接,经过位置编码后输入到Transformer编码器中进行全局语义信息的提取,再还原为多尺度特征,为上采样过程提供信息补偿。上采样模块,首先将来自骨干网络和像素编码模块的特征按通道进行拼接为补偿特征,与上采样过程中的特征进行融合,然后通过空间注意力模块和卷积等方式,逐渐恢复特征图中的空间细节信息。分割头,则通过1×1卷积从特征图中恢复分割后的图像。
图 1.

Network structure
网络结构图
1.1. 像素编码模块
由于医学图像复杂,待分割的目标区域通常形状不规则,边界模糊,而单一尺度的特征提取容易丢失信息,影响分割精度。原始TransUNet在特征提取时,只是将骨干网络输出的多尺度特征中的最小分辨率特征送入Transformer编码器模块进行全局以及远程语义信息提取。因此,本文引入了特征金字塔结构,提出了像素编码模块,将骨干网络输出的不同分辨率的特征统一送入Transformer编码器模块进行全局信息的提取,最终得到与骨干网络输出的相同分辨率的特征金字塔。本文还将Transformer的自注意力替换为可变形注意力,在减小网络参数的同时提高性能。本研究像素编码模块具体细节详细介绍如下文所示。
1.1.1. 补丁拼接
Dosovitskiy等[20]提出的ViT网络的输入只能为一种尺度。针对多尺度输入,Yang等[25]通过卷积上采样等方式将不同分辨率特征转化为相同尺度,再通过相加的方式合并,最终成功将多尺度特征输入到Transformer模块中。但这种方法不可避免地会引入噪声,特征相加的过程也会丢失特征信息。本文结合补丁嵌入的基本方式提出了一种多尺度特征图输入策略,不需要上采样引入噪声的同时修改特征融合方式,将特征相加改为特征拼接,最大限度保留原有特征信息,具体操作如图2所示。
图 2.
Patch concatenation
补丁拼接
根据Dosovitskiy等[20]提出的ViT网络,本文首先对输入的多尺度特征进行卷积,通过卷积将多尺度特征通道数进行统一,然后分别进行补丁嵌入操作,将不同分辨率的特征图(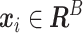 ;其中,xi为输入的多尺度特征,B为批量大小,C为通道数,H和W分别为对应特征图的高和宽,)统一重塑为一个平坦的二维补丁序列(
;其中,xi为输入的多尺度特征,B为批量大小,C为通道数,H和W分别为对应特征图的高和宽,)统一重塑为一个平坦的二维补丁序列(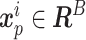 ,其中,xip为输入的二维补丁序列, N为补丁数量,
,其中,xip为输入的二维补丁序列, N为补丁数量,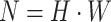 )。接着在补丁数量维度对所有的二维平坦补丁序列进行拼接操作,得到一个整体序列,即为输入序列。
)。接着在补丁数量维度对所有的二维平坦补丁序列进行拼接操作,得到一个整体序列,即为输入序列。
1.1.2. 可变形注意力
Transformer模块参数量大,难以收敛。其主要原因在以下两点:① Transformer在初始化过程中,对所有特征图像素平均分配注意力权重,这导致模型需要较长时间去学习需要关注的位置,且位置大多数情况下为稀疏矩阵。② Transformer在计算注意力权重时,其模块中的每个查询(query)都会和所有的键值(key)进行点积运算,即计算复杂度与特征图中像素点的数量呈平方关系,因此难以处理高分辨率特征。因此,为了解决上述问题,本文引入了可变形注意力[26],可变形注意力只关注参考点周围的小部分关键采样点,而不考虑整体的特征图大小,其结构如图3所示。
图 3.
Illustration of the deformable attention module
可变形注意力模块结构图
如图3所示,输入特征图中每个像素点对应的query通过线性层获得对应采样点数量的偏移量,将当前像素点坐标与偏移量结合确定采样点坐标,再通过双线性插值法获得采样点坐标的key。通过可变形注意力使得query仅与采样点位置的key进行点积运算,计算复杂度减小,参数量减少。
其次,每个query通过线性层以及激活函数获得注意力权重,即在初始化阶段针对采样点赋予不同权重,为模型加入先验条件,加快模型收敛。为了使注意力权重范围保持在[0, 1],激活函数采用柔性最大(softmax)激活函数。
给定输入特征图x、像素的二维坐标Pq以及对应的查询Zq,可变形注意力(以符号y表示)的计算,如式(1)所示:
 |
1 |
其中,m表示注意力头,M表示多头注意力的总头数,k表示采样点,K表示总采样点数,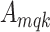 和
和 分别表示第k个采样点的注意力权重和采样偏移量,Wm表示权重。
分别表示第k个采样点的注意力权重和采样偏移量,Wm表示权重。
1.1.3. Transformer模块数量的缩减
Transformer模块的大量堆积同样是造成参数量大的原因之一。根据Wang等[27]的研究,Transformer编码器中距离输入更近的模块更重要。因此,为了进一步轻量化模型,减少参数量,本文对Transformer模块的堆积数量进行了消融实验,并最后确定使用7个Transformer块(n = 7),如图1所示。消融实验,具体细节请见后文。
1.1.4. 后处理
为了获得与输入相同分辨率的多尺度特征,本文对Transformer输出的二维补丁序列进行后处理。首先对补丁序列采用分裂函数还原为多尺度特征,并将原图像的1/16、1/8、1/4分辨率特征图保留。为了恢复空间顺序,对1/32分辨率特征图进行重塑处理。本文使用1 × 1卷积将重塑后的特征通道数缩减为类别数,然后对该特征进行逐步双线性上采样,用于预测最终的分割结果。
1.2. 空间注意力模块
编码器过程中,多层次特征经过通道维度拼接后,通过跳跃连接接入到解码器过程中,本文引入卷积空间注意力模块(spatial attention module,SAM)[28],使模型重点关注空间信息,从而有效提升对纹理、边界等区域的分割精度。空间注意力模块整体结构如图4所示。
图 4.

Illustration of spatial attention module
空间注意力结构示意图
如图4所示,本文在空间注意力模块中加入跳跃连接来防止特征丢失。假定输入特征图F尺度为H × W × C,分别经过最大池化层和平均池化层后得到两个H × W × 1的特征向量,将两个向量在通道维度进行拼接,获得H × W×2的特征向量,通过卷积核为7 × 7、步长为1的卷积层进行降维,使用S型生长曲线(sigmoid)激活函数将其转化为权重向量F1,并与原特征图F相乘得到加权特征图F2,F2再与原特征图F相加获得输出的特征图F3。
1.3. 损失函数
本文训练损失函数采用图像分割领域广泛使用的交叉熵损失(cross entropy loss)[29]函数。为了解决电子计算机断层扫描(computed tomography,CT)图像类别不平衡的问题,本文引入戴斯损失(Dice loss)函数。因此,本文结合Dice loss函数和cross entropy loss函数来共同训练整体模型。
(1)Dice loss函数,其主要是根据分类结果在正类别以及负类别的占比来进行损失函数的计算。根据分类结果是在正类别还是在负类别,可以将其分为真阳性(true positive,TP)、真阴性(true negative,TN)、假阳性(false positive,FP)、假阴性(false negative,FN)。TP表示正类别中正确分类样本的数量;TN表示负类别中正确分类样本的数量;FP表示正类别中错误分类样本的数量;FN是负类别中错误分类样本的数量。Dice loss函数(以符号LDice表示)计算公式如式(2)所示:
 |
2 |
其中,c为某个特定器官类别; 、
、 、
、 分别为对应的真阳性率、假阴性率、假阳性率;C为器官总类别数;α和β为加权权重,均设置为0.5。
分别为对应的真阳性率、假阴性率、假阳性率;C为器官总类别数;α和β为加权权重,均设置为0.5。
(2)cross entropy loss函数,以符号Lcrossentropy表示,其计算公式如式(3)所示:
 |
3 |
其中, 为样本c的真实值;
为样本c的真实值; 为样本c预测的输出值;c为某个特定器官类别;C为器官总类别数。
为样本c预测的输出值;c为某个特定器官类别;C为器官总类别数。
因此,最终的损失函数,以符号Ltotal表示,其计算公式如式(4)所示:
 |
4 |
2. 实验
2.1. 实验环境及数据集
本文使用的医学分割图像数据集为突触(Synapse)多器官医学分割公开数据集。该数据集来自医学图像计算与计算机辅助干预国际会议(International Conference on Medical Image Computing and Computer Assisted Intervention,MICCAI)发起的多图谱标记超越颅穹窿挑战(multi-atlas labeling beyond the cranial vault challenge,MALBCV),由30名受试者的8个器官的腹部CT图像组成,包含30例样本共3 779张腹部临床CT医学图像。数据集中每个轴片文件包含85~198张512 × 512像素的切片。本实验采取与TransUNet相同的数据集处理策略,将18例样本划分为训练集,将12例样本划分为训练集。为了加快模型收敛,提升训练速度,本文实验在训练过程中统一把所有训练集图像的分辨率缩放为224 × 224。
在训练设置方面,本文使用动量梯度下降优化算法对整体模型进行优化,优化器的初始学习率设置为0.01,动量设置为0.6,权重衰减设置为1 × 10−4,批量大小设置为8,总共训练150轮次。为了确保实验的公平性,所有实验都在Windows 操作系统(Microsoft Inc.,美国)上进行,基于PyTorch深度学习框架(Meta Inc.,美国)来实现,图形处理器均为RTX 3060(NVIDIA,美国),编程语言为 Python 3.6(Centrum Wiskunde & Informatica,荷兰)。
2.2. 评价指标
为了验证本文算法的分割性能,本文使用戴斯相似系数(Dice similarity coefficient,DSC)和95%豪斯多夫距离(95% Hausdorff Distance,HD95)两个指标来评估模型在8个腹部器官上的表现。
(1)DSC,为分割预测体素与真值(ground truth)体素之间的重叠,以符号DSC表示,其定义如式(5)所示:
 |
5 |
其中,Y和P分别表示所有体素的真值输出和实际输出。
(2)HD95,通常用作基于边界的度量指标,定义为分割预测体素的边界与真值体素边界之间的最大95%距离,以符号HD95表示,其定义如式(6)所示:
 |
6 |
其中, 是预测体素与真值体素之间的最大95%距离,
是预测体素与真值体素之间的最大95%距离, 是真值体素与预测体素之间的最大95%距离。
是真值体素与预测体素之间的最大95%距离。
由上述指标的定义以及表达式可以看出,DSC更侧重分割掩膜的内部填充,而HD95则对分割后的边界更加敏感。
2.3. 实验结果及分析
为了验证本文所提出的改进算法的有效性,本文将分割结果与之前的六种最先进模型进行比较:① V型网络(V-Net)[30];② 基于自适应阈值的多器官分割网络(domain adaptive relational reasoning,DARR)[31];③ U-Net[14];④ Atten-UNet[17];⑤ ViT[20];⑥ TransUNet[21]。不同网络模型在Synapse数据集的分割结果对比以及每种器官的DSC指标系数如表1所示。其中,DSC指标以及HD95指标均为所有器官对应指标结果的平均值;向上的箭头“ ”表示DSC指标数值越大,对应的分割效果越好;向下的箭头“
”表示DSC指标数值越大,对应的分割效果越好;向下的箭头“ ”表示HD95指标数值越小,对应的分割效果越好。
”表示HD95指标数值越小,对应的分割效果越好。
表 1. Comparison of segmentation results of different network models on the Synapse dataset and Dice score for each organ.
Comparison of segmentation results of different network models on the Synapse dataset and Dice score for each organ
不同网络模型在Synapse数据集的分割结果对比以及每种器官的DSC指标系数
| 模型 | DSC↑ | HD95↓ | DSC指标系数 | |||||||
| 主动脉 | 胆囊 | 左肾 | 右肾 | 肝 | 胰腺 | 脾 | 胃 | |||
| R50 U-Net[14] | 74.68 | 36.87 | 87.74 | 63.66 | 80.60 | 78.19 | 93.74 | 56.90 | 85.87 | 74.16 |
| R50 Atten-UNet[17] | 75.57 | 36.97 | 55.92 | 63.91 | 79.20 | 72.71 | 93.56 | 49.37 | 87.19 | 74.95 |
| R50 ViT[20] | 71.29 | 32.87 | 73.73 | 55.13 | 75.80 | 72.20 | 91.51 | 45.99 | 81.99 | 73.95 |
| TransUNet[21] | 77.48 | 31.69 | 87.23 | 63.13 | 81.87 | 77.02 | 94.08 | 55.86 | 85.08 | 75.62 |
| V-Net[30] | 68.81 | — | 75.34 | 51.87 | 77.10 | 80.75 | 87.84 | 40.05 | 80.56 | 56.89 |
| DARR[31] | 69.77 | — | 74.74 | 53.77 | 72.31 | 73.24 | 94.08 | 54.18 | 89.90 | 45.96 |
| 本文算法 | 77.65 | 18.34 | 87.36 | 60.32 | 81.29 | 79.54 | 93.47 | 60.82 | 88.30 | 70.10 |
由于本文所使用的编码器为ResNet50与Transformer混合编码器,出于公平考虑,本文将U-Net、Atten-UNet的原始编码器替换为在ImageNet数据集上预训练的ResNet50,并将其写作R50 U-Net、R50 Atten-UNet表示,加粗部分表示最优指标。表1中数据均采用作者的官方模型,并在相同的环境中训练得到的。
由表1可以看到,与基准模型TransUNet相比,本文算法在DSC指标上提高了0.13,在HD95指标上降低了13.35,表明本文提出的改进方案有效。同时,在多器官分割Synapse数据集中,本文算法在DSC、HD95两个指标中得分均为最优,其中HD95指标降幅较大,说明本文模型提出的像素编码模块是有效的,该模块将骨干网络的多尺度特征进行编码后统一送入Transformer模块进行全局语义信息的提取,从而增强模型的全局注意力,加强对CT医学图像的边界感知,从而有效地提取图像特征,保留更多的边缘信息。
其次,由于不同器官在CT图像中的体积占比不同,其分割结果受到较大影响,例如:胆囊以及胰腺相比于其它器官体积较小,其分割结果相对较差。从单个器官的分割结果来看,本文模型对于小体积的器官学习能力更强,与基准模型TransUNet相比,胰腺分割的DSC指标提升了4.96,胆囊以及其他相对较大器官的分割结果也均在中上水平,侧面表明本文所提出的改进对模型泛化能力的提升。
最后,将Transformer与传统CNN相结合,即得到表1中的R50 ViT模型,其性能优于V-Net和DARR,但仍低于基于CNN的R50 U-Net和R50 Atten-UNet,但在与U-Net结构融合且加入本文所提出的像素编码模块后,达到了新的高度。这表明,本文算法具有强大的学习高级语义特征和低级语义细节的能力,进一步表现了本文算法相对于传统卷积方法的优势。
3. 消融实验
为了验证所提出的模块对分割效果的有效性,本文对所提的4个改进点:像素编码模块、可变形注意力、空间注意力模块以及Transformer数量模块进行了模块消融实验,结果如表2所示。
表 2. Ablation study on different network architectures.
Ablation study on different network architectures
不同模块组合的消融实验
| 模块组合 | DSC↑ | HD95↓ | |||
| 像素编码模块 | 可变形注意力 | 空间注意力模块 | Transformer模块数量缩减 | ||
| — | — | — | — | 77.48 | 31.69 |
| √ | — | — | — | 77.53 | 27.32 |
| √ | √ | — | — | 77.58 | 24.56 |
| √ | √ | √ | — | 77.72 | 23.08 |
| √ | √ | √ | √ | 77.65 | 18.34 |
表2中,第1行是TransUNet基准模型,第2行是仅加入像素编码模块,第3~5行是分别在第2行基础上增加可变形注意力、空间注意力模块以及削减Transformer模块数量后的网络模型。由表2内数据可以看出,随着模块组合的减少,HD95指标呈现上升趋势。在削减Transformer模块数量后,DSC指标出现一定程度的下降,但相比于近32%的参数量的削减以及近42%的HD95指标的提升,DSC指标仅下降0.07,在可接受范围内。这表明不同模块的合理组合有利于提升医学图像分割的性能,也说明本文重点提出的像素编码模块对多器官CT图像分割是有效的。
此外,为了研究Transformer模块对模型学习能力的影响,本文对Transformer模块进行了消融实验,结果如表3所示。表3中,第1行为原始TransUNet网络对应的参数量及性能指标,第2行为加入像素编码模块及空间注意力模块后对应的参数量及指标,未改变自注意力的情况下,参数量相较于原来网络有所增加,性能指标得到提升,侧面证明本文所提出模块的有效性。第3行为将自注意力替换为可变形注意力后的参数量及性能指标, 在Transformer模块数量均为12个情况下,参数量相比于自注意力时的算法减少了30%,且性能有所提升。
表 3. Comparison of parameters of different network models and segmentation results.
Comparison of parameters of different network models and segmentation results
不同网络模型参数量及分割结果对比
| 模型 | Transformer模块数量 | DSC↑ | HD95↓ | 参数量/MB |
| TransUNet | 12 | 77.48 | 31.69 | 401 |
| 本文算法(自注意力) | 12 | 77.56 | 27.32 | 410 |
| 本文算法(可变形注意力) | 12 | 77.72 | 23.08 | 287 |
| 11 | 77.42 | 19.60 | 284 | |
| 10 | 75.32 | 27.10 | 281 | |
| 9 | 77.19 | 23.15 | 278 | |
| 8 | 75.66 | 27.57 | 275 | |
| 7 | 77.65 | 18.34 | 273 | |
| 6 | 76.99 | 20.61 | 270 |
从第3行往下,为可变形注意力条件下,Transformer模块数量对性能指标及参数量的影响。根据Wang等[27]的研究,Transformer编码器中距离输入更近的模块更重要,因此使用7个Transformer块便能较好地学习到特征,参数量也在适合范围内。
从定量的角度看,本文提出的像素编码模块能够明显提升分割效果,能够把全局信息和局部信息有效地结合起来提取更多特征,使得器官图像更容易分割。此外,多层次的图像特征可弥补骨干网络卷积过程中的信息丢失,减小信息丢失带来的影响。
4. 结论
本文提出一种基于像素编码和空间注意力的多尺度医学图像分割方法。在原始TransUNet的基础上提出了4个改进点:像素编码模块、可变形注意力、空间注意力模块和网络轻量化。
(1)像素编码模块能对骨干网络输出的多尺度信息进行全局语义信息提取,同时输出相同分辨率的多尺度特征,在有效解决单尺度特征下采样信息丢失问题的同时还能为后续上采样特征提供信息补偿。
(2)在传统Transformer编码器基础上引入了可变形注意力,通过学习自适应偏移量来减小计算复杂度,加快收敛速度同时提升模块性能。
(3)在解码器过程中引入空间注意力,增强模型对分割边界的敏感程度,提高分割性能。
(4)最后,本文缩减了模块数量,实现网络轻量化。本文通过两个评价指标评估了算法的有效性,并通过消融实验证明了所提出模块的有效性。在Synapse数据集上的实验结果表明,与其他主流的多器官CT图像分割模型相比,本文提出的医学图像分割算法能够得到更精确的分割结果,填补了多尺度特征全局语义信息提取方向的空白。在未来的工作中,本课题组将对网络进行上采样方面的优化,使用更精细的优化算法替代简单的卷积,并应用于其他二维医学影像分割任务。
重要声明
利益冲突声明:本文全体作者均声明不存在利益冲突。
作者贡献声明:万雨龙负责算法设计与实现、数据处理与分析、论文写作与修改;周冬明提供实验指导及论文审阅修订;刘宜松和王长城参与实验结果的整理;白崇斌负责数据集的收集。
Funding Statement
国家自然科学基金项目(62066047,61966037)
National Natural Science Foundation of China
References
- 1.徐光宪, 冯春, 马飞 基于UNet的医学图像分割综述. 计算机科学与探索. 2023;17(8):1776–1792. [Google Scholar]
- 2.Pun T A new method for grey-level picture thresholding using the entropy of the histogram. Signal Processing. 1980;2(3):223–237. doi: 10.1016/0165-1684(80)90020-1. [DOI] [Google Scholar]
- 3.Yen J C, Chang F J, Chang S A new criterion for automatic multilevel thresholding. IEEE Transactions on Image Processing. 1995;4(3):370–378. doi: 10.1109/83.366472. [DOI] [PubMed] [Google Scholar]
- 4.Batenburg K J, Sijbers J Adaptive thresholding of tomograms by projection distance minimization. Pattern Recognition. 2009;42(10):2297–2305. doi: 10.1016/j.patcog.2008.11.027. [DOI] [Google Scholar]
- 5.Cheng Y Mean shift, mode seeking, and clustering. IEEE Transactions on Pattern Analysis and Machine Intelligence. 1995;17(8):790–799. doi: 10.1109/34.400568. [DOI] [Google Scholar]
- 6.Salvador S, Chan P. Determining the number of clusters/segments in hierarchical clustering/segmentation algorithms//16th IEEE International Conference on Tools with Artificial Intelligence, IEEE, 2004: 576-584.
- 7.徐国雄, 张骁, 胡进贤, 等 基于阈值分割和轮廓提取的图像边缘检测算法. 计算机技术与发展. 2015;25(12):64–67,71. [Google Scholar]
- 8.Khan J F, Bhuiyan S M A, Adhami R R Image segmentation and shape analysis for road-sign detection. IEEE Transactions on Intelligent Transportation Systems. 2010;12(1):83–96. [Google Scholar]
- 9.岳欣华,邓彩霞,张兆茹 BP神经网络与形态学融合的边缘检测算法. 哈尔滨理工大学学报. 2021;26(5):83–90. [Google Scholar]
- 10.Tremeau A, Borel N A region growing and merging algorithm to color segmentation. Pattern Recognition. 1997;30(7):1191–1203. doi: 10.1016/S0031-3203(96)00147-1. [DOI] [Google Scholar]
- 11.Bhargavi K, Jyothi S A survey on threshold based segmentation technique in image processing. International Journal of Innovative Research and Development. 2014;3(12):234–239. [Google Scholar]
- 12.Masood A, Al-Jumaily A A Fuzzy C mean thresholding based level set for automated segmentation of skin lesions. Journal of Signal and Information Processing. 2013;4(3):66. doi: 10.4236/jsip.2013.43B012. [DOI] [Google Scholar]
- 13.Long J, Shelhamer E, Darrell T. Fully convolutional networks for semantic segmentation //Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition, 2015: 3431-3440.
- 14.Ronneberger O, Fischer P, Brox T. U-net: convolutional networks for biomedical image segmentation//Medical Image Computing and Computer-Assisted Intervention–MICCAI 2015: 18th International Conference, Munich, Germany: Springer International Publishing, 2015: 234-241.
- 15.Zhou Z, Rahman Siddiquee M M, Tajbakhsh N, et al. Unet++: a nested U-net architecture for medical image segmentation//Deep Learning in Medical Image Analysis and Multimodal Learning for Clinical Decision Support: 4th International Workshop and 8th International Workshop, Granada, Spain: Springer International Publishing, 2018: 3-11.
- 16.Qin X, Zhang Z, Huang C, et al U2-Net: going deeper with nested U-structure for salient object detection. Pattern Recognition. 2020;106:107404. doi: 10.1016/j.patcog.2020.107404. [DOI] [Google Scholar]
- 17.Oktay O, Schlemper J, Folgoc L L, et al. Attention U-net: learning where to look for the pancreas. arXiv preprint, 2018, arXiv: 1804.03999.
- 18.Jin Q, Meng Z, Sun C, et al RA-UNet: a hybrid deep attention-aware network to extract liver and tumor in CT scans. Frontiers in Bioengineering and Biotechnology. 2020;8:605132. doi: 10.3389/fbioe.2020.605132. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Vaswani A, Shazeer N, Parmar N, et al. Attention is all you need//Proceedings of the 31st International Conference on Neural Information Processing Systems, 2017: 6000-6010.
- 20.Dosovitskiy A, Beyer L, Kolesnikov A, et al. An image is worth 16×16 words: transformers for image recognition at scale. arXiv preprint, 2020, arXiv: 2010.11929.
- 21.Chen J, Lu Y, Yu Q, et al. TransUNet: Transformers make strong encoders for medical image segmentation. arXiv preprint, 2021, arXiv: 2102.04306.
- 22.Wang H, Cao P, Wang J, et al. Uctransnet: rethinking the skip connections in u-net from a channel-wise perspective with transformer//Proceedings of the AAAI Conference on Artificial Intelligence, 2022, 36(3): 2441-2449.
- 23.Krizhevsky A, Sutskever I, Hinton G E ImageNet classification with deep convolutional neural networks. Communications of the ACM. 2017;60(6):84–90. doi: 10.1145/3065386. [DOI] [Google Scholar]
- 24.He K, Zhang X, Ren S, et al. Deep residual learning for image recognition//Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition, 2016: 770-778.
- 25.Yang H, Zhou D, Cao J, et al RainFormer: a pyramid transformer for single image deraining. The Journal of Supercomputing. 2023;79(6):6115–6140. doi: 10.1007/s11227-022-04895-5. [DOI] [Google Scholar]
- 26.Zhu X, Su W, Lu L, et al. Deformable DETR: deformable transformers for end-to-end object detection. arXiv preprint, 2020, arXiv: 2010.04159.
- 27.Wang W, Tu Z. Rethinking the value of transformer components. arXiv preprint, 2020, arXiv: 2011.03803.
- 28.尹稳, 周冬明, 范腾, 等 基于密集空洞空间金字塔池化和注意力机制的皮肤病灶图像分割方法. 生物医学工程学杂志. 2022;39(6):1108–1116. doi: 10.7507/1001-5515.202208015. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.陈英, 张伟, 林洪平, 等 医学图像分割算法的损失函数综述. 生物医学工程学杂志. 2023;40(2):392–400. doi: 10.7507/1001-5515.202206038. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 30.Milletari F, Navab N, Ahmadi S A. V-net: fully convolutional neural networks for volumetric medical image segmentation//2016 fourth international conference on 3D vision (3DV). IEEE, 2016: 565-571.
- 31.Fu S, Lu Y, Wang Y, et al. Domain adaptive relational reasoning for 3D multi-organ segmentation//Medical Image Computing and Computer Assisted Intervention–MICCAI 2020: 23rd International Conference, Lima, Peru: Springer International Publishing, 2020: 656-666.




