Abstract
目的
探讨类风湿关节炎(RA)的潜在发病机制,挖掘可以作为RA早期诊断的生物标志物,并探究其可能的免疫调控机制。
方法
依托GEO数据库多个基因组数据,利用limma包、RRA方法、批次矫正方法与clusterProfiler包筛选差异表达基因并预测其作用功能;通过STRING在线数据库得到蛋白质互作网络,利用Cytoscape3.8.0软件与GeneMANIA数据库筛选差异表达关键基因并预测其互作机制;构建ROC工作曲线验证关键基因诊断模型的信效度,并通过机器学习方法筛选疾病特征免疫细胞;使用corrplot包分析特征免疫细胞与疾病关键基因的相关性;使用GSEA富集方法探究关键基因生物学功能和相关性;通过构建胶原诱导性关节炎(CIA)大鼠模型探究具有较高诊断价值的差异基因在RA滑膜组织中的表达情况。
结果
得到与RA密切相关核心关键基因9个,分别为:CD3G、CD8A、SYK、LCK、IL2RG、STAT1、CCR5、ITGB2、ITGAL,富集分析结果表明差异基因可能主要通过细胞因子相关通路调控滑膜炎症;ROC工作曲线分析表明9个核心基因的预测模型信效度较高,其中STAT1预测模型的AUC值最高(0.909);相关性分析结果显示,CD3G、ITGAL、LCK、CD8A和STAT1等5个核心基因与疾病特征免疫细胞相关性较强,其中STAT1与M1型巨噬细胞的相关性联系最强(R=0.68,P<0.001);CIA大鼠膝关节的HE、番红-O固绿染色结果表明关节炎大鼠造模成功且药物治疗有效,免疫荧光结果表明STAT1和磷酸化的STAT1在CIA大鼠踝关节滑膜组织中表达高于正常组和治疗组;免疫印迹实验结果表明,STAT1蛋白表达量在滑膜成纤维细胞的细胞核(P=0.0002)与细胞质(P<0.0001)中存在差异,且经过治疗,细胞核内p-STAT1与STAT1的蛋白表达量显著降低(P<0.0001)。
结论
CD3G、CD8A、SYK、LCK、IL2RG、STAT1、CCR5、ITGB2与ITGAL等9个基因可作为RA的早期诊断生物标志物,CD3G/CD8A/LCK-γδ T细胞、ITGAL-Tfh细胞、STAT1-M1型巨噬细胞等基因-免疫细胞途径可能与RA的发生发展密切相关。
Keywords: 类风湿关节炎, 生物标志物, 免疫调控, 生物信息学, 机器学习
Abstract
Objective
To identify the biomarkers for early rheumatoid arthritis (RA) diagnosis and explore the possible immune regulatory mechanisms.
Methods
The differentially expressed genesin RA were screened and functionally annotated using the limma, RRA, batch correction, and clusterProfiler. The protein-protein interaction network was retrieved from the STRING database, and Cytoscape 3.8.0 and GeneMANIA were used to select the key genes and predicting their interaction mechanisms. ROC curves was used to validate the accuracy of diagnostic models based on the key genes. The disease-specific immune cells were selected via machine learning, and their correlation with the key genes were analyzed using Corrplot package. Biological functions of the key genes were explored using GSEA method. The expression of STAT1 was investigated in the synovial tissue of rats with collagen-induced arthritis (CIA).
Results
We identified 9 core key genes in RA (CD3G, CD8A, SYK, LCK, IL2RG, STAT1, CCR5, ITGB2, and ITGAL), which regulate synovial inflammation primarily through cytokines-related pathways. ROC curve analysis showed a high predictive accuracy of the 9 core genes, among which STAT1 had the highest AUC (0.909). Correlation analysis revealed strong correlations of CD3G, ITGAL, LCK, CD8A, and STAT1 with disease-specific immune cells, and STAT1 showed the strongest correlation with M1-type macrophages (R=0.68, P=2.9e-08). The synovial tissues of the ankle joints of CIA rats showed high expressions of STAT1 and p-STAT1 with significant differential expression of STAT1 between the nucleus and the cytoplasm of the synovial fibroblasts. The protein expressions of p-STAT1 and STAT1 in the cell nuclei were significantly reduced after treatment.
Conclusion
CD3G, CD8A, SYK, LCK, IL2RG, STAT1, CCR5, ITGB2, and ITGAL may serve as biomarkers for early diagnosis of RA. Gene-immune cell pathways such as CD3G/CD8A/LCK-γδ T cells, ITGAL-Tfh cells, and STAT1-M1-type macrophages may be closely related with the development of RA.
Keywords: rheumatoid arthritis, biomarkers, immune regulatory, bionformatic, machine learning
类风湿关节炎(RA)是一种发病机制复杂的进行性肌肉骨骼损伤疾病,遗传因素、环境因素和微生物群组的交互作用诱导机体固有免疫的异常活化是诱发RA的主要原因[1-3]。RA多发病于中老年人群体,以女性患者居多,近年来其全球患病率和流行率呈现上升趋势,且其流行趋势出现年轻化[4, 5]。尽管提倡疾病的早发现、早诊断、早治疗有助于患者预后,但由于RA的高度异质性,部分患者发病前期的生化指标和体征并不符合RA的诊断金标准,在确诊时疾病往往已经进展为中后期,严重降低了患者的生命质量[6, 7]。因此探究疾病新型生物标志物对于RA的临床前期诊断有重要意义。此外,针对RA免疫系统异常活化的特点,目前临床常用治疗方案多以具有免疫抑制效果的药物为主,但长期服药引起的非靶组织的毒副作用和药物耐受限制了传统抗风湿药物的应用[8, 9]。因此,为了进一步推进RA的精准诊疗,探究潜在生物标志物在RA中的免疫调控机制是有必要的。
基因表达综合(GEO)数据库是国内外常用的疾病基因分析公共数据库,其涵盖了多种疾病的不同类型的基因组数据,可以用于探索病因未明疾病的表观遗传改变并筛选新型疾病诊断和预后生物标志物[10]。本研究基于GEO数据库多个数据集的基因组数据,筛选得到了与RA发生发展密切相关的9个核心基因,通过ROC工作曲线预测了核心基因作为生物标志物的信效度,并初步探究了核心基因与疾病特征免疫细胞之间的关系。随后通过构建胶原诱导性关节炎(CIA)大鼠模型验证了相关性较强的核心基因在RA滑膜组织中的表达情况。本研究通过整合分析多个数据集的基因组数据,从多方面探究了RA发生发展过程中具有重要生物学意义的潜在生物标志物的表达和诊断价值,并结合疾病特征免疫细胞进一步探讨了相关的生物标志物在RA发病过程中的免疫调控过程,对挖掘RA精准诊疗新靶点具有一定意义。
1. 材料和方法
1.1. 数据资料来源
从GEO数据库中筛选含有滑膜组织健康对照组和类风湿关节炎患者组的训练集和验证集数据,共纳入5个训练集:GSE1919,GSE12021,GSE55235,GSE55457与GSE77298;一个验证集:GSE89408。
1.2. 实验动物
5~7周龄雄性SD大鼠25只,体质量180±20 g,饲养于蚌埠医科大学科研中心SPF级动物房,给予充足饮食和饮水,适应性饲养1周后建立CIA大鼠模型。所有动物实验经蚌埠医科大学实验动物管理和伦理委员会批准(伦动科批号[2021]第192号)。
1.3. 主要试剂
牛Ⅱ型胶原(Chondrex)、弗氏不完全佐剂 (Chondrex);甲氨蝶呤(国药准字H31020644,上海上药信谊药厂有限公司);4%多聚甲醛通用型组织固定液(Biosharp);鼠抗信号转导和转录激活因子1(STAT1,Proteintech);TRIzol(Ambion);细胞核蛋白与细胞浆蛋白抽提试剂盒(碧云天)。
1.4. 差异表达基因筛选
1.4.1. 差异表达基因获得
对单个数据集的基因表达矩阵进行差异分析,利用Robust Rank Aggreg(RRA)方法筛选在多个数据集均表现差异的基因。此外,为剔除数据集间的批次效应,将多个数据集合并后使用Batch Normalization方法进行批次矫正,对批次矫正后的数据集进行差异分析。最后将两种方法得到的差异表达基因取交集,得到共差异表达基因。以上两种差异分析筛选标准均为:|logFC|>1,adj.P.Val<0.05。
1.4.2. GO和KEGG功能富集分析
对共差异表达基因进行GO功能富集分析和KEGG通路富集分析。并对GO富集分析和KEGG富集分析结果中各条目相关性统计学意义排列的前5个条目进行可视化展示。
1.4.3. 免疫细胞浸润分析
为量化浸润性免疫细胞在RA中的相对比例,利用CIBERSORT方法分析矫正合并后的共差异表达基因在每个样品中的表达矩阵,以P<0.05过滤免疫浸润结果。计算样品中每种免疫细胞类型的百分比,并以柱形图显示。使用pheatmap包绘制22种免疫细胞的聚类热图,使用corrplot软件包绘制22种免疫细胞的相关性热图。
1.4.4. Hub基因筛选
从STRING在线数据库(https://string-db.org/)检索关键基因编码蛋白间可能的潜在相互作用,同时构建蛋白质互作网络。随后将蛋白互作网络导入Cytoscape3.8.0软件,利用cytohubba插件中的10种计算程序(MCC, DMNC, MNC, Degree, BottleNeck, EcCentricity, Closeness, Radiality, Betweenness, Stress)对基因的表达情况进行打分,将每种打分分数排名前50的基因视为有效打分基因,同时满足10种有效打分的基因认为是蛋白互作网络的关键基因。随后从GEO数据库中获取含有正常组与RA患病早期组和晚期组滑膜组织的基因表达矩阵(GSE89408),用于验证关键基因在病程早期和晚期在滑膜组织中的表达情况。
1.4.5. 疾病特征免疫细胞筛选
对每种免疫细胞的含量在正常组和RA组间循环进行差异分析,将P<0.05的免疫细胞视为差异免疫细胞。同时利用glmnet包对免疫细胞浸润结果构建LASSO回归模型,利用交叉验证和LASSO回归模型筛选特征免疫细胞。将上述实验得到的差异免疫细胞和特征免疫细胞取交集,得到疾病核心免疫细胞。
1.4.6. 核心基因与疾病核心免疫细胞相关性分析
将核心基因的表达量与核心免疫细胞的含量进行相关性检验,设置过滤条件为相关系数(R)的绝对值大于0.4,相关性检验的P<0.001,满足条件说明核心基因的表达与免疫细胞的含量是具有较高相关性的。然后利用corrplot包绘制相关性热图和单个基因与核心免疫细胞散点图,若得到的散点图相关系数大于0,则说明两者间存在正调控关系,反之为负调控关系。
1.4.7. 单基因ssGSEA分析
将关键基因表达分为高表达和低表达两组,将基因按照在RA组和对照组中的差异表达程度排序,引用org.Hs.eg.db人类数据库转化ID和logFoldChange方法进行差异表达分析。对关键基因的KEGG通路进行GSEA富集分析,富集通路过滤条件为:GSSize≤200&GSSize≥10,P<0.05。为了得到单基因在疾病中的富集通路功能差异,从GSEA分子特征数据库(www.gsea-msigdb.org/gsea/msigdb)中下载标志基因集文件,计算ssGSEA功能富集评分并对得分结果进行差异分析,绘制各功能通路在RA组与对照组间差异。
1.5. 胶原诱导性关节炎(CIA)大鼠模型的构建及干预
根据参考文献和本实验室沿用的方法构建CIA大鼠模型[11, 12]。将等量的弗氏不完全佐剂和牛II型胶原(2 mg/mL)在冰上充分混匀,直至完全乳化得到造模所需的乳化剂。为保证动物成模,采取两次加强免疫方法造模。初次免疫时在SD大鼠尾根部皮内注射200 μL乳化剂,1周后加强免疫,在SD大鼠尾根部皮内注射100 μL乳化剂。加强免疫5~8 d后,SD大鼠均出现关节红肿和活动受限,通过关节炎评分(AI)、足爪红肿状态和足跖厚度证明成模。AI>4分时认为模型构建成功,开始给药。
采用随机数法将造模成功的大鼠分为3组,对照组(vehicle)、模型组(CIA组)和甲氨蝶呤组(MTX组),MTX组每两周经口灌胃给药1次,给药浓度为2 mg/kg,对照组和模型组大鼠经口灌胃2 mL的PBS溶液,连续给药4周。1.6 组织病理学染色与免疫荧光染色
大鼠脱颈处死,取下肢骨组织后用4%多聚甲醛固定,慢脱钙处理后脱水透明、浸蜡包埋切片,厚度为5 μm。使用苏木精伊红(HE)染色和番红O-固绿染色观察大鼠膝关节组织炎性细胞浸润、血管翳形成、滑膜衬里层变化和关节软骨破环情况;使用免疫荧光染色观察大鼠踝关节滑膜组织中目标蛋白表达水平。
1.7. 滑膜组织细胞胞质与胞核蛋白提取
收集对照组、模型组和MTX治疗组大鼠滑膜组织的滑膜成纤维细胞至1.5 mL EP管中,使用预冷的PBS洗2次,加入200 L预冷的细胞浆蛋白抽提试剂A (pH 7.9 10 mmol/L HEPES、10 mmol/L KCl、0.1 mmol/L EDTA、1 mmol/L dithiothreitol、0.4% Igepal CA-630、5 mol/L leupeptin、2 mol/L pepstatin A、1 mol/L aprotinin和1 mmol/L phenylmethyl sulfonyl fluoride)悬浮细胞,冰上放置15 min,4℃ 12000 r/min离心5 min,上清液为细胞质含胞浆蛋白-70 ℃保存备用,沉淀物为细胞核。随后将沉淀物中残余的上清液吸净,加入50 L预冷的细胞核蛋白抽提试剂 B(pH 7.9 20 mmol/L HEPES、0.4 mol/L NaCl、1 mmol/L EDTA、1 mmol/L二硫苏糖醇和1 mmol/L苯甲基磺酰氯),4℃涡旋30 min,12 000 r/min离心10 min,随后立即吸取上清液至预冷的EP管中,即得到细胞核蛋白。
1.8. Western blotting
使用上述提取的细胞核蛋白和细胞质蛋白进行免疫印迹实验。首先使用BCA蛋白定量法测定蛋白质浓度。调整蛋白上样量使其符合实验要求。120 V电泳1.5 h,200 mA转膜2 h。使用5%脱脂牛奶封闭2 h,随后将条带置于STAT1抗体(Abcam)与p-STAT1抗体(Abcam)中4 ℃摇床孵育过夜。使用TBST冲洗后,二抗室温封闭2 h,随后用超敏ECL化学发光底物在凝胶成像系统曝光,收集可用图像并用EXCEL做统计学分析。
1.9. 统计学分析
使用R软件处理实验数据,多组间数据使用单因素方差分析,两组间数据采用Wilcoxon秩和检验,P<0.05认为差异有统计学意义,所有实验均独立重复3次。
2. 结果
2.1. 差异表达基因筛选
利用R软件筛选得到不同数据集的差异表达基因(图1)。基于RRA方法获得差异表达基因共454个,其中上调基因292个,下调基因162个。利用批次标准化方法得到差异基因349个。对通过两种方法得到的差异表达基因取交集,共得到338个共差异表达基因,以供后续分析。
图1.
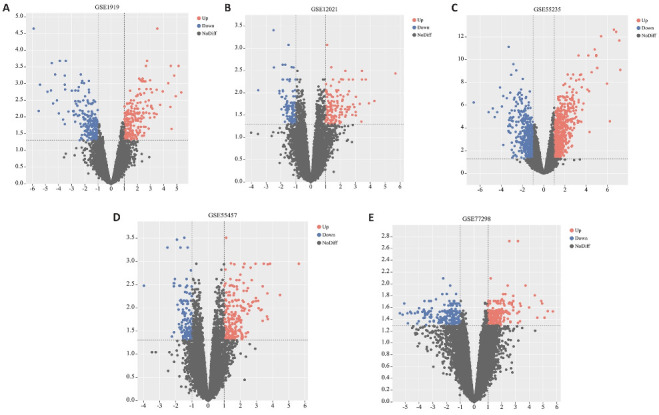
不同数据集的差异基因表达情况
Fig.1 Differential gene expressions in different datasets of RA. A-E: RA-related differentially expressed genes in the GSE1919, GSE12021, GSE55235, GSE55457 and GSE77298 datasets.
2.2. 交集基因的GO和KEGG富集分析结果
GO富集分析结果表明,共差异表达基因在BP中主要富集在白细胞的细胞间粘附、单核细胞分化、淋巴细胞分化、白细胞迁移、调节白细胞的细胞间粘附性;在CC层面主要集中于细胞质膜外侧、凝集素包裹的内泡膜、凝集素包裹的内吞囊泡、凝集素包裹的囊泡膜、膜筏;在MF层面主要富集于趋化因子受体结合、趋化因子活性、免疫受体活性、G蛋白偶联受体结合、细胞因子活性(图2A)。KEGG富集分析结果表明,关键基因主要富集于趋化因子信号通路、细胞因子-细胞因子受体信号通路、类风湿关节炎信号通路、NF-κB信号通路、Th1和Th2免疫细胞分化(图2B)。
图2.

差异表达基因的GO和KEGG富集分析
Fig.2 GO and KEGG enrichment analysis of the differentially expressed genes (DEGs). A: Circle diagram for GO enrichment analysis of the DEGs. B: Sankey diagrams for KEGG enrichment analysis of the DEGs.
2.3. 核心基因筛选和表达水平分析
通过Cytoscape软件共筛选得到9个且均在RA中存在差异表达核心基因(图3A、B),分别为CD3G、CD8A、脾酪氨酸激酶(SYK)、淋巴细胞特异性蛋白酪氨酸激酶(LCK)、白细胞介素2受体γ(IL2RG)、信号传导及转录激活蛋白1(STAT1)、趋化因子受体5(CCR5)、整合素亚基β2(ITGB2)、整合素亚基αL(ITGAL)。
图3.

差异表达核心基因筛选及表达情况
Fig.3 Screening of the differentially expressed core genes and their expressions. A: Results of the core gene screening. B: Complex Heatmap of the core genes. C-K: Expression of the core genes in control, early RA and advanced RA in the validation dataset (C: SYK; D: STAT1; E: LCK; F: IL2RG; G: ITGB2; H: ITGAL; I: CD3G; J: CCR5; K: CD8A).
选择含有正常对照组、RA早期患者和晚期患者滑膜组织样本的数据集分析核心基因的表达情况。相较于正常组的低表达,9个核心基因在RA发病早期的滑膜组织中表达均升高(P<0.001);与RA早期相比,ITGB2和ITGBAL两个核心基因的中位值降低,差异具有统计学意义(P<0.05),但仍高于正常组的表达水平,其余7个基因的表达水平在RA早晚期的滑膜组织中没有统计学差异(P>0.05,图3C~K)。
2.4. 核心基因功能预测和ROC工作曲线结果
将通过GeneMANIA数据库预测得到的基因功能结果根据FDR值进行排序,发现核心基因可能通过细胞因子或其表面受体通路调控细胞异常激活和炎症反应发挥作用(图4A)。
图4.

核心基因功能预测和ROC工作曲线结果
Fig.4 Core gene function prediction and receiver operating characteristic (ROC) curve results. A:Analysis of GeneMANIA results for core genes. B-J: Area under ROC for the core genes (B: CD8A; C: CD3G; D: IL2RG; E: ITGB2; F: ITGAL; G: LCK; H: SYK; I: CCR5; J: STAT1).
ROC曲线分析表明,9个核心基因的AUC值均大于0.65,其中,ITGB2预测模型的AUC值为0.667;ITGAL、LCK、CD8A、CD3G、SYK、CCR5和IL2RG等7个预测模型的AUC值介于0.70~0.85(图4B~I);STAT1预测模型的AUC值为0.909(图4J)。
2.5. 免疫细胞浸润分析与疾病特征免疫细胞筛选
CIBERSORT分析结果显示,多种免疫细胞在RA中存在差异表达(图5A)。其中RA组的记忆B细胞、CD8 T细胞、活化CD4 T细胞、滤泡性辅助性T细胞、γδ T细胞、M0型巨噬细胞和M1型巨噬细胞的表达数量高于正常组(P<0.01),而正常组的静息CD4 T细胞、浆细胞、调节性T细胞、活化的NK细胞、单核细胞、静息树突状细胞、静息肥大细胞和嗜酸性粒细胞的表达数量高于RA组(P<0.01,图5B)。此外,免疫细胞相关性热图结果表明,多对免疫细胞在RA的发生发展中具有协同或拮抗作用(图5C)。LASSO回归分析共得到16个与RA发生发展密切相关的免疫细胞。与上文中筛选的差异免疫细胞取交集后共得到11个疾病特征免疫细胞(图5D)。分别为:浆细胞、静息CD4 T细胞、滤泡性辅助性T细胞、γδ T细胞、单核细胞、M0型巨噬细胞、M1型巨噬细胞、静息树突状细胞、静息肥大细胞、活化的肥大细胞和嗜酸性粒细胞。
图5.

疾病特征免疫细胞表达及筛选
Fig.5 Disease-specific immune cell expression and screening. A: Immune cell infiltration in RA. B: Differential expression of immune cells in RA. C: Correlation Heatmap of the immune cells. D: Intersections of disease-specific cells obtained by screening with Lasso regression and CIBERSORT methods.
2.6. 核心基因与疾病特征免疫细胞的相关性分析
9个核心基因均与疾病特征免疫细胞存在不同程度的相关性(图6A)。其中CD3G、ITGAL、LCK、CD8A和STAT1等5个核心基因与相关的疾病特征免疫细胞相关性较强(图6B~F),CD3G(R=0.54,P<0.001)、CD8A(R=0.65,P<0.001)与LCK(R=0.59,P<0.001)和γδ T细胞存在正相关,ITGAL与Tfh细胞存在正相关(R=0.56,P<0.001),STAT1与M1型巨噬细胞存在正相关,且其相关性系数最高(R=0.68,P<0.001)。
图6.

核心基因与疾病特征免疫细胞相关性分析
Fig.6 Correlation analysis of the core genes with the disease-specific immune cells. A: Heatmap of correlations between core genes and disease-specific immune cells. B-F: Correlation analysis of the core genes with disease-specific immune cells (B: CD8A with γδ T cell; C: CD3G with γδ T cell; D: ITGAL with Tfh cell; E: LCK with γδ T cell; F: STAT1 with macrophages M1).
2.7. STAT1在各组大鼠中的表达情况
GSEA富集分析结果展示了STAT1表达升高情况下可能介导的5条生物学过程,分别为同种异体移植排斥,移植物抗宿主病,产生IgA的肠道免疫网络,原发性免疫缺陷和病毒蛋白与细胞因子及细胞因子受体的相互作用(图7A)。
图7.

STAT1在CIA大鼠踝关节中的表达情况
Fig.7 Expression of STAT1 in ankle joints of CIA rats. A: GSEA analysis of biological processes potentially mediated by upregulated STAT1. B: HE staining of the knee joints of CIA rats. C: Safranine O-Fast Green staining of the knee joints. D: STAT1 expression in the synovial tissue of CIA rats.
膝关节HE结果表明,CIA组大鼠与正常组大鼠相比滑膜组织增生明显,骨质侵蚀程度较重,可见大量炎性细胞浸润和血管翳产生,经MTX治疗后,镜下炎细胞浸润程度明显降低且关节表面光滑无糜烂(图7B);膝关节番红-O固绿结果显示,CIA组大鼠软骨面和软骨下骨组织界限不清晰,软骨细胞减少,而经MTX处理的大鼠的软骨表面光滑且软骨细胞数量明显增加,软骨潮线清晰、骨组织无明显破坏(图7C)。
免疫荧光结果表明,CIA组大鼠的踝关节滑膜组织中STAT1表达水平明显高于正常组,而经过MTX治疗后,STAT1的表达水平明显降低(图7D)。
2.8. STAT1磷酸化和细胞核迁移
CIA组大鼠踝关节滑膜组织中p-STAT1的表达水平显著增高,而MTX组大鼠踝关节滑膜组织中p-STAT1的表达水平下调(图8A)。Western blotting实验结果表明CIA组大鼠滑膜组织细胞核内的STAT1与p-STAT1的蛋白表达量高于细胞质,且显著高于对照组大鼠细胞质与细胞核中STAT1(Cytosolo:P=0.0002;Nuclear:P<0.0001)与p-STAT1(Cytosolo:P=0.0023;Nuclear:P<0.0001)蛋白的表达水平,而经MTX治疗后发现,细胞核内p-STAT1的蛋白表达量明显减少(P<0.0001,图8B~D)。
图8.

p-STAT1在CIA大鼠滑膜组织和滑膜成纤维细胞中的表达情况
Fig.8 Expression of p-STAT1 in the synovial tissue and fibroblast-like synoviocytes of CIA rats. A: p-STAT1 expression in the synovial tissue of CIA rats. B: Immunoblotting showing expressions of STAT1/p-STAT1 in the cytoplasm and nucleus of the synovial tissue cells in different groups. C, D: Western blotting of p-STAT and STAT1 proteins in cytoplasm and nuclei of synovial fibroblasts in the joint tissue of CIA rats. *P<0.05,**P<0.01,***P<0.001, ****P<0.0001.
3. 讨论
借助计算机生物信息学的方法可以探究不同基因间的互作过程和其调控疾病的可能发生机制[13]。RA作为一种高度异质性疾病,具体发病机制尚不明晰[1, 14]。临床常通过患者远端小关节的病理变化和血清类风湿因子、C-反应蛋白和红细胞沉降率等指标判断是否发病,但传统生化指标不适用于部分血清学检验阴性的RA患者[15, 16]。因此本研究依托生信分析探究RA的潜在生物标志物,初步探究有益于RA临床早期诊断的潜在标志物。
本研究通过整合GEO数据库中5个RA患者滑膜组织数据集的基因组数据,初步筛选得到338个与RA发病密切相关的共差异表达基因。KEGG和GO富集分析表明差异基因可能通过激活细胞内外质膜表面受体发挥相关功能,主要调控炎性细胞因子及其受体通路以及免疫细胞的增殖分化。随后进一步筛选与RA发生发展密切相关的核心基因,发现CD3G、CD8A、SYK、LCK、IL2RG、STAT1、CCR5、ITGB2和ITGAL等9个核心基因在疾病差异基因拓扑网络作为枢纽节点与其他基因关联紧密且节点较多。此外,我们发现9个核心基因在RA患者的滑膜组织中表达水平均升高,且CD3G、CD8A、SYK、LCK、IL2RG、STAT1和CCR5这7个核心基因在RA早期和晚期的滑膜组织中表达水平差异不具有统计学意义(P>0.05),这提示以上7个核心基因在RA早期可能作为疾病的诊断标志物。ROC曲线结果结果提示9个核心基因用于RA早期诊断具有一定的意义,其中STAT1作为RA预测模型的AUC值最高(0.909),提示其作为RA发病的生物诊断标志物具有较高信效度。
RA作为一种自身免疫性疾病,免疫细胞的异常活化、侵袭与互作在疾病发展中发挥重要作用[17, 18]。在既往的一些研究中,CD3G、CD8A、ITGAL、LCK和STAT1已被证明与RA的发生发展关系密切[19-21],但大多研究仅通过观察其蛋白质或mRNA的表达水平说明这些基因在RA的病程进展中有一定的价值,而并未与疾病相关免疫细胞相联系进而说明其在RA中的可能调控机制。本研究筛选得到16种疾病特征免疫细胞,初步探究了差异表达核心基因与疾病特征免疫细胞的潜在相关性。结果表明CD3G、CD8A、ITGAL、LCK和STAT1这5个疾病差异表达核心基因主要与不同亚型的T淋巴细胞和M1型巨噬细胞存在密切关联。
γδ T细胞是具有γ链和δ链组成的异二聚体T细胞受体复合物(TCR)的一类T淋巴细胞亚群[22, 23]。研究表明,γδ T细胞在RA患者滑液中高表达,与滑膜炎症的严重程度和疾病活动度呈正相关[24, 25]。本研究表明,CD3G、CD8A与LCK基因与γδ T细胞密切相关,CD3G、CD8A与LCK表达水平的增加可能与γδ T细胞的异常生物学行为有关。尽管在过去的研究中这3个基因作为RA的生物标志均被提及[20, 21, 26],但尚未有研究明确表明其与γδ T细胞之间的潜在互作关系,因此在后续的研究中将CD3G、CD8A与LCK作为调控γδ T细胞的潜在靶点在RA的靶向治疗和明晰其发病机制方面可能具有一定意义。Tfh细胞主要通过分泌炎性介质或介导B淋巴细胞的增殖分化促进RA的疾病进展[27]。本研究发现ITGAL与Tfh细胞的水平呈现正相关,提示ITGAL的表达水平与Tfh细胞的增殖分化可能存在一定关系。研究表明ITGAL(CD11A)与自身免疫性疾病中CD4+T细胞的过度活化密切相关[26, 28],但关于ITGAL调控Tfh细胞在RA中的具体作用机制仍需进一步探究。M1型促炎巨噬细胞可以通过分泌多种炎性因子或激活滑膜细胞异常生物学行为加重滑膜炎症[29, 30]。研究表明STAT1的表达增加能够促进巨噬细胞向M1型促炎表型极化[31],本研究发现STAT1与M1型巨噬细胞的水平存在正相关,同时STAT1可能参与Th1与Th2免疫细胞的增殖分化。GSEA富集分析结果提示STAT1可能通过肠道免疫网络和细胞因子及其受体的激活这两种生物学过程发挥免疫调控作用,而这两种信号通路已被证明在RA的发生发展中具有重要作用[32-34]。
为进一步验证生信分析结果的可信性,本研究选择AUC值最大且与疾病特征免疫细胞联系最为紧密的基因(STAT1)进行动物实验。本研究结果表明,在CIA大鼠中,踝关节滑膜组织的STAT1与p-STAT1的表达明显高于对照组;p-STAT1在CIA大鼠踝关节滑膜成纤维细胞细胞核中的表达量高于细胞质,而经过MTX治疗后,细胞核内p-STAT1的表达量显著降低,这提示STAT1在RA中通过磷酸化以及核迁移介导信号通路的激活,发挥转录因子的作用。以上结果说明STAT1作为RA的潜在生物标志物具有重要意义,其可能通过介导M1型巨噬细胞炎性活化进而调节细胞因子及其受体通路或肠道免疫网络途径在RA的病程进展中发挥重要作用,但具体调控机制还需进一步探讨。
JAK-STAT信号通路的激活与RA微环境中炎症介质的释放、破骨细胞的分化以及骨质的破坏密切相关,针对这一通路所研发并应用于临床的靶点阻滞剂也较成熟[35, 36]。目前临床应用的阻滞剂主要以靶向抑制JAK1/2/3为主,目前报道的靶向STAT3的一些药物,如Stattic、OPB-111077和HL-237等也已处于临床试验阶段,但是关于靶向STAT1的药物研究较少,针对于临床STAT1特异性靶向药也被认为有较大的探索空间[37]。研究表明,STAT1能够驱动趋化因子诱导白细胞募集到关节炎,加重疾病活动度[38];抑制STAT1能够促进Th1细胞分化,缓解关节炎症状[39]。因此,结合本研究结果和文献报道,我们推测STAT1可以作为RA临床潜在治疗靶点。
综上所述,本研究结果表明CD3G、CD8A、SYK、LCK、IL2RG、STAT1、CCR5、ITGB2与ITGAL这9个基因可能作为RA的潜在生物标志物用于疾病早期诊断,且CD3G/CD8A/LCK‑γδ T细胞、ITGAL-Tfh细胞、STAT1-M1型巨噬细胞等基因-免疫细胞途径在RA的发生发展中具有重要作用。本研究不足之处在于本研究缺乏对调控机制的深入探究。在未来的实验中,本团队将依托动物实验、细胞实验以及大型公共数据库进一步探究本研究中获得的多个具有重要意义的潜在生物标志物在RA中的具体调控机制及其与免疫细胞相互作用的生物学过程。
基金资助
国家级大学生创新训练项目(202310367024,202310367027)
参考文献
- 1. Alivernini S, Firestein GS, McInnes IB. The pathogenesis of rheumatoid arthritis[J]. Immunity, 2022, 55(12): 2255-70. [DOI] [PubMed] [Google Scholar]
- 2. Xu Q, Ni JJ, Han BX, et al. Causal relationship between gut microbiota and autoimmune diseases: a two-sample Mendelian randomization study[J]. Front Immunol, 2021, 12: 746998. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3. Kronzer VL, Davis JM. Etiologies of rheumatoid arthritis: update on mucosal, genetic, and cellular pathogenesis[J]. Curr Rheumatol Rep, 2021, 23(4): 21. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4. Chiang PH, Ju PC, Chiang YC, et al. Correspondence on ‘Incidence trend of five common musculoskeletal disorders from 1990 to 2017 at the global, regional and national level: results from the global burden of disease study 2017'[J]. Ann Rheum Dis, 2023, 82(2): e46. [DOI] [PubMed] [Google Scholar]
- 5. Guan SY, Zheng JX, Sam NB, et al. Global burden and risk factors of musculoskeletal disorders among adolescents and young adults in 204 countries and territories, 1990-2019[J]. Autoimmun Rev, 2023, 22(8): 103361. [DOI] [PubMed] [Google Scholar]
- 6. Lenti MV, Rossi CM, Melazzini F, et al. Seronegative autoimmune diseases: a challenging diagnosis[J]. Autoimmun Rev, 2022, 21(9): 103143. [DOI] [PubMed] [Google Scholar]
- 7. Tidblad L, Westerlind H, Delcoigne B, et al. Comorbidities and treatment patterns in early rheumatoid arthritis: a nationwide Swedish study[J]. RMD Open, 2022, 8(2): e002700. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8. Ben Mrid R, Bouchmaa N, Ainani H, et al. Anti-rheumatoid drugs advancements: new insights into the molecular treatment of rheumatoid arthritis[J]. Biomed Pharmacother, 2022, 151: 113126. [DOI] [PubMed] [Google Scholar]
- 9. Tan Y, Buch MH. 'Difficult to treat' rheumatoid arthritis: current position and considerations for next steps[J]. RMD Open, 2022, 8(2): e002387. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10. Clough E, Barrett T. The gene expression omnibus database[J]. Methods Mol Biol, 2016, 1418: 93-110. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11. Zhao T, Xie Z, Xi Y, et al. How to model rheumatoid arthritis in animals: from rodents to non-human Primates[J]. Front Immunol, 2022, 13: 887460. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12. Zhang X, Zhang X, Wang X, et al. Efficient delivery of triptolide plus a miR-30-5p inhibitor through the use of near infrared laser responsive or CADY modified MSNs for efficacy in rheumatoid arthritis therapeutics[J]. Front Bioeng Biotechnol, 2020, 8: 170. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13. Rincón-Riveros A, Morales D, Rodríguez JA, et al. Bioinformatic tools for the analysis and prediction of ncRNA interactions[J]. Int J Mol Sci, 2021, 22(21): 11397. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14. Jang S, Kwon EJ, Lee JJ. Rheumatoid arthritis: pathogenic roles of diverse immune cells[J]. Int J Mol Sci, 2022, 23(2): 905. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15. Yu R, Zhang J, Zhuo Y, et al. Identification of diagnostic signatures and immune cell infiltration characteristics in rheumatoid arthritis by integrating bioinformatic analysis and machine-learning strategies[J]. Front Immunol, 2021, 12: 724934. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16. Cheng Q, Chen X, Wu H, et al. Three hematologic/immune system-specific expressed genes are considered as the potential biomarkers for the diagnosis of early rheumatoid arthritis through bioinformatics analysis[J]. J Transl Med, 2021, 19(1): 18. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17. Firestein GS, McInnes IB. Immunopathogenesis of rheumatoid arthritis[J]. Immunity, 2017, 46(2): 183-96. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18. Wu D, Luo Y, Li T, et al. Systemic complications of rheumatoid arthritis: focus on pathogenesis and treatment[J]. Front Immunol, 2022, 13: 1051082. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19. Matsumoto H, Fujita Y, Onizawa M, et al. Increased CEACAM1 expression on peripheral blood neutrophils in patients with rheumatoid arthritis[J]. Front Immunol, 2022, 13: 978435. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20. Li Z, Xu M, Li R, et al. Identification of biomarkers associated with synovitis in rheumatoid arthritis by bioinformatics analyses[J]. Biosci Rep, 2020, 40(9): BSR20201713. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21. Zhou S, Lu H, Xiong M. Identifying immune cell infiltration and effective diagnostic biomarkers in rheumatoid arthritis by bioinformatics analysis[J]. Front Immunol, 2021, 12: 726747. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22. Vantourout P, Hayday A. Six-of-the-best: unique contributions of γδ T cells to immunology[J]. Nat Rev Immunol, 2013, 13: 88-100. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23. Hayday AC. γδ T cell update: adaptate orchestrators of immune surveillance[J]. J Immunol, 2019, 203(2): 311-20. [DOI] [PubMed] [Google Scholar]
- 24. Mo WX, Yin SS, Chen H, et al. Chemotaxis of Vδ2 T cells to the joints contributes to the pathogenesis of rheumatoid arthritis[J]. Ann Rheum Dis, 2017, 76(12): 2075-84. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25. Jacobs MR, Haynes BF. Increase in TCR gamma delta T lymphocytes in synovia from rheumatoid arthritis patients with active synovitis[J]. J Clin Immunol, 1992, 12(2): 130-8. [DOI] [PubMed] [Google Scholar]
- 26. Pascual-García S, Martínez-Peinado P, López-Jaén AB, et al. Analysis of novel immunological biomarkers related to rheumatoid arthritis disease severity[J]. Int J Mol Sci, 2023, 24(15): 12351. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27. Lu J, Wu J, Xia XL, et al. Follicular helper T cells: potential therapeutic targets in rheumatoid arthritis[J]. Cell Mol Life Sci, 2021, 78(12): 5095-106. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 28. Zhao S, Wang Y, Liang Y, et al. MicroRNA-126 regulates DNA methylation in CD4+ T cells and contributes to systemic lupus erythematosus by targeting DNA methyltransferase 1[J]. Arthritis Rheum, 2011, 63(5): 1376-86. [DOI] [PubMed] [Google Scholar]
- 29. Cutolo M, Campitiello R, Gotelli E, et al. The role of M1/M2 macrophage polarization in rheumatoid arthritis synovitis[J]. Front Immunol, 2022, 13: 867260. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 30. Tardito S, Martinelli G, Soldano S, et al. Macrophage M1/M2 polarization and rheumatoid arthritis: a systematic review[J]. Autoimmun Rev, 2019, 18(11): 102397. [DOI] [PubMed] [Google Scholar]
- 31. Ye Q, Luo F, Yan T. Transcription factor KLF4 regulated STAT1 to promote M1 polarization of macrophages in rheumatoid arthritis[J]. Aging: Albany NY, 2022, 14(14): 5669-80. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 32. Guo DG, Lv JH, Chen X, et al. Study of miRNA interactome in active rheumatoid arthritis patients reveals key pathogenic roles of dysbiosis in the infection-immune network[J]. Rheumatology: Oxford, 2021, 60(3): 1512-22. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 33. Zaiss MM, Wu HJ J, Mauro D, et al. The gut-joint axis in rheumatoid arthritis[J]. Nat Rev Rheumatol, 2021, 17: 224-37. [DOI] [PubMed] [Google Scholar]
- 34. Kondo N, Kuroda T, Kobayashi D. Cytokine networks in the pathogenesis of rheumatoid arthritis[J]. Int J Mol Sci, 2021, 22(20): 10922. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 35. Simon LS, Taylor PC, Choy EH, et al. The Jak/STAT pathway: a focus on pain in rheumatoid arthritis[J]. Semin Arthritis Rheum, 2021, 51(1): 278-84. [DOI] [PubMed] [Google Scholar]
- 36. Banerjee S, Biehl A, Gadina M, et al. JAK-STAT signaling as a target for inflammatory and autoimmune diseases: current and future prospects[J]. Drugs, 2017, 77(5): 521-46. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 37. Hu L, Liu RJ, Zhang LL. Advance in bone destruction participated by JAK/STAT in rheumatoid arthritis and therapeutic effect of JAK/STAT inhibitors[J]. Int Immunopharmacol, 2022, 111: 109095. [DOI] [PubMed] [Google Scholar]
- 38. Karonitsch T, Saferding V, Kieler M, et al. Amino acids fueling fibroblast-like synoviocyte activation and arthritis by regulating chemokine expression and leukocyte migration[J]. Arthritis Rheumatol, 2024, 76(4): 531-40. [DOI] [PubMed] [Google Scholar]
- 39. Mai YP, Yu XT, Gao T, et al. Autoantigenic peptide and immunomodulator codelivery system for rheumatoid arthritis treatment by reestablishing immune tolerance[J]. ACS Appl Mater Interfaces, 2024: Online ahead of print. [DOI] [PubMed] [Google Scholar]


