RESUME
Le spectre des maladies génétiques en Tunisie résulte de l'effet fondateur, de la dérive génétique, de la pression de sélection, et de la consanguinité. Celle-ci représente une déviation de la panmixie, caractérisée par un choix matrimonial non aléatoire, potentiellement influencé par des facteurs socio-culturels, économiques ou autres. Cela éloigne la structure génétique de l'équilibre de Hardy-Weinberg, augmentant les génotypes homozygotes et diminuant les hétérozygotes, ce qui élève la fréquence des maladies autosomiques récessives. Comme d'autres populations arabes, la Tunisie présente des taux de consanguinité élevés variant géographiquement. Les maladies autosomiques récessives représentent 60% des maladies signalées en Tunisie, avec une consanguinité potentielle dans 80% des familles touchées par une maladie spécifique. Dans les populations endogames, la consanguinité amplifie le risque de maladie autosomique récessive. Cependant, elle n’affecte pas la probabilité de maladies autosomiques dominantes, mais influence leur phénotype. La consanguinité est également un facteur majeur dans l'homozygotie des variants délétères conduisant à l'expression de comorbidités. Au niveau génomique, les individus consanguins héritent des mutations homozygotes et des régions génomiques adjacentes appelées régions d’homozygotie (ROHs). Les ROHs courtes et longues indiquent respectivement une consanguinité lointaine et récente. La distribution de ces segments dans le génome est très irrégulièrement avec certaines régions relativement courtes présentant des excès de ROHs, appelées îlots de ROHs. Dans cette revue, nous discutons de l'impact de la consanguinité sur la santé de la population, la dynamique, et la structure du génome, présentant la Tunisie comme modèle.
ABSTRACT
The genetic disease spectrum in Tunisia arises from the founder effect, genetic drift, selection, and consanguinity. The latter represents a deviation from panmixia, characterized by a non-random matrimonial choice that may be subject to several rules, such as socio-cultural, economic, or other factors. This shifts the genetic structure away from the Hardy-Weinberg equilibrium, increasing homozygous genotypes and decreasing heterozygotes, thus raising the frequency of autosomal recessive diseases. Similar to other Arab populations, Tunisia displays high consanguinity rates that vary geographically. Approximately 60% of reported diseases in Tunisia are autosomal recessive, with consanguinity possibly occurring in 80% of families for a specific disease. In inbred populations, consanguinity amplifies autosomal recessive disease risk, yet it does not influence autosomal dominant disease likelihood but rather impacts its phenotype. Consanguinity is also suggested to be a major factor in the homozygosity of deleterious variants leading to comorbid expression. At the genome level, inbred individuals inherit homozygous mutations and adjacent genomic regions known as runs of homozygosity (ROHs). Short ROHs indicate distant inbreeding, while long ROHs refer to recent inbreeding. ROHs are distributed rather irregularly across the genome, with certain short regions featuring an excess of ROH, known as ROH islands. In this review, we discuss consanguinity's impact on population health and genome dynamics, using Tunisia as a model.
Introduction
Le spectre des maladies génétiques en Tunisie est la résultante de plusieurs facteurs : l’effet fondateur, la dérive génétique, la pression de sélection et la consanguinité (1, 2, 3).
Ce dernier, constitue un écart à la panmixie, une situation d’équilibre où les unions se font au hasard et les structures génétiques sont définies par une probabilité d’occurrence correspondant théoriquement à un tirage aléatoire des gamètes à partir d’une urne commune à la population des géniteurs de composition donnée.
Cet écart à la panmixie est représenté par un choix matrimonial non aléatoire qui peut être soumis à plusieurs règles comme des motivations socio-culturelles, économiques ou autres.
Ainsi, les tirages des gamètes à une urne beaucoup plus restreinte sont limités et correspondent à une même catégorie donnée pour les deux géniteurs comme c’est le cas de l’endogamie en général.
Ce tirage non aléatoire peut aussi se faire en cas d’homogamie, c'est-à-dire que les individus s’accouplent selon un certain degré de ressemblance, comme une tare génétique.
Dans ces deux situations (endogamie et homogamie), le choix effectué par les individus reproducteurs modifie les modalités de rencontre des gamètes par rapport à la panmixie.
Il en découle une structure génétique de la population différente d’une population en équilibre de Hardy Weinberg.
Bien que la structure allélique reste invariante, la structure génotypique en est modifiée car les probabilités de formation des zygotes sont modifiées.
Par rapport à une situation d’équilibre, une augmentation des génotypes homozygotes contre un appauvrissement des hétérozygotes sera notée.
En génétique médicale, ceci se manifeste par une augmentation de la fréquence des maladies autosomiques récessives.
En Tunisie, les mariages consanguins sont fréquents et ont été rapportés dans 38% de la population en 1999 (4) et dans 28%, donc presque 1/3 de la population deux décennies après (5).
Dans les spectres rapportés de 2011 et 2021, 60% des maladies rapportées sont autosomiques récessives (3, 6) et pour une maladie donnée, la consanguinité peut être présente dans 80 % des familles.
A l’échelle du génome, l’individu consanguin n’hérite pas seulement la mutation à l’état homozygote, mais toute une région génomique autour de la mutation, communément appelée région d’homozygotie et longuement étudiée grâce aux techniques de génotypage et de séquençage à haut débit (7, 8).
La taille et la distribution des ROHs permettent d’avoir une idée sur la démographie de la population : Les ROHs courtes sont indicateurs d’un lien de parenté ancien et donc observées chez des individus non consanguins, tant disque les ROHs longues sont observées chez des individus appartenant à des populations avec des taux forts de consanguinité et reflètent une parenté proche (8 , 9).
Dans cette revue, nous allons revenir sur les impacts de la consanguinité sur la santé de la population et sur la dynamique du génome et sa structure en prenant le cas de la Tunisie comme exemple.
Généralités sur la consanguinité
Définition
Le terme consanguinité dérive de latin «cum » qui veut dire « avec » et « sanguis », sang.
La consanguinité est un cas d’écart à la panmixie et fait référence aux mariages entre individus apparentés partageant au moins un ancêtre en commun.
En génétique clinique, un mariage entre apparentés est défini comme étant une union entre cousins de second degré ou plus avec un coefficient de consanguinité F supérieur ou égal à 0,0156 (10).
En génétique des populations, la consanguinité peut référer aux unions maritales d’individus avec au moins un ancêtre en commun, comme ceux ayant lieu dans les isolats, les petites villes, les tribus et les communautés.
Différents types de mariages entre apparentés
Le mariage entre cousins fournit d'excellentes opportunités pour la transmission des valeurs culturelles et la pérennité de la culture.
C’est pour cela que les unions consanguines sont généralement considérées comme plus stables que les mariages entre personnes non apparentées (11).
Dans la plupart des sociétés arabes, les mariages entre cousins parallèles sont considérés comme importants pour unir les membres d'un même groupe de descendance et maintenir la transmission de la culture au sein de la même famille.
Ces considérations peuvent être particulièrement importantes dans des conditions de changement social et d'insécurité politique ou socioéconomique (11).
Union entre cousins germain (1C)
C’est le type d’union entre apparentés le plus fréquent (12).
Dans ce cas, les 2 individus possèdent un grandparent en commun et leur progéniture est homozygote à 1/16 de tous les loci, soit un coefficient de consanguinité Fped = 1/16 = 0,0625 (Figure 1 et Tableau 1) (10).
Figure 1. Types de mariage entre apparenté .
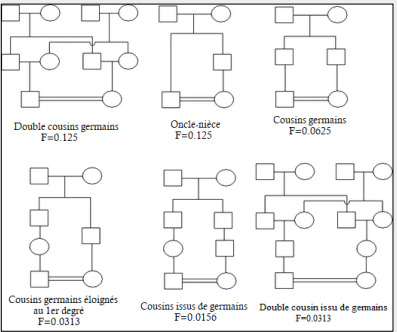
Tableau 1 : Les mariages consanguins .
|
Type de relation |
Degré de parenté |
Fped |
|
Oncle – Nièce / Tante - Neveux |
2ème degré |
1/8=0,125 |
|
Double cousins germains |
2ème degré |
1/8=0,125 |
|
Cousins germains |
3ème degré |
1/16=0,0625 |
|
Cousins germains éloignés au 1er degré |
4ème degré |
1/32=0,0313 |
|
Double cousin issu de germains |
4ème degré |
1/32=0,0313 |
|
Cousins issus de germains |
5ème degré |
1/64=0,0156 |
|
Cousins issus de germains éloignés au 1er degré |
6ème degré |
1/128=0,0078 |
|
Double petits cousins |
6ème degré |
1/128=0,0078 |
|
Petits cousins |
7ème degré |
1/256=0,0039 |
Union entre double cousins germains
Les enfants issus d’un mariage entre double cousins germains ont un coefficient de consanguinité Fped= 1/8 = 0,125, qui est l’équivalent de Fped pour un mariage entre Oncle-Nièce ou Tante-Neveux.
Ce dernier type de mariage est interdit par l’Islam et donc absent chez les populations Arabes mais peut se trouver ailleurs comme chez les Indiens ou les Juifs (Figure 1 et Tableau 1) (11 ).
Union entre cousins issus de germains (2C)
Les cousins issus de germains sont deux individus qui ont un arrière grand-parent en commun. Le coefficient de consanguinité Fped de leur descendance est de 1/64 = 0,0156 (Figure 1 et Tableau 1) (11 ).
Union entre cousins inégaux (1C1R)
Dans ce cas l’union se fait entre un individu et le cousin de son père ou de sa mère.
La descendance a un coefficient de consanguinité Fped= 1/32 = 0,0313. (Figure 1 et Tableau 1) (11).
Mesure de la consanguinité
Le taux de consanguinité
Le taux de consanguinité est calculé comme étant le rapport du nombre des unions consanguines par le nombre total des unions.
Le coefficient de parenté
Le coefficient de parenté de deux individus A et B est calculé comme étant la probabilité que deux gènes tirés au hasard au même locus, l’un chez A et l’autre chez B, soient identiques par descendance.
Le coefficient de consanguinité
Le coefficient de consanguinité d’un individu I, noté FI, est égal à la probabilité pour que les deux gènes qu’il possède à un locus donné soient identiques par descendance.
Il est généralement établi après examen de généalogies par la méthode des chemins (13) par la formule suivante :
FI = (1/2)i + j (1/2 + FA/2)
Avec i et j le nombre de méioses sur les deux branches de l’ancêtre A aux parents de I, autrement dit, i et j sont les nombres de générations qui séparent l’ancêtre A des parents de I, sachant que la probabilité de transmission d’un allèle d’une génération à une autre est de ½ pour les loci autosomiques.
Cette méthode a permis d’établir les valeurs classiques comme 1/8 pour les mariages entre doubles cousins germains, 1/16 pour les unions entre cousins germains, 1/32 pour les unions consanguines entre cousins inégaux et 1/64 dans le cas de mariages entre cousins issus de germains.
Le coefficient de consanguinité F d’un individu est égale au taux de parenté de ses parents.
D’autres valeurs de F peuvent être trouvées dans le tableau 1
Au niveau de la population, il est possible de calculer le coefficient de consanguinité moyen qui est égale à la moyenne pondérée des différents coefficients de consanguinité individuels (Fi) par les fréquences des différents types de croisements entre apparentés (fi).
Ce coefficient moyens a été désigné par Bernstein comme coefficient α et introduit pour mesurer l’homogénéité génétique d’une population (14) :
α = Σ fi Fi
Coefficient de Consanguinité FROH
De point de vue génotypique, une région d’homozygotie ou Runs Of Homozygosity (ROH) est un segment d’ADN ininterrompu sans hétérozygotie à l'état diploïde (7).
Ainsi, l’homozygotie causée par deux segments génomiques identiques par descendance (identical by descent ou IBD) est appelée autozygotie (15).
On définit alors le coeffeicient FROH comme le coefficient de consanguinité F estimé à partir de la ROH et qui est défini comme la proportion du génome qui a le statut homozygote identique par descendance (IBD), ou la probabilité qu'un locus échantillonné au hasard dans le génome soit autozygote (16) .
FROH est considéré comme la méthode la plus puissante pour détecter les effets de la consanguinité parmi plusieurs estimations alternatives de la consanguinité (15).
En effet, ce coefficient prédit avec plus de précision le niveau réel d'autozygotie du génome et peut être estimé pour tout individu disposant de données génotypiques, même si aucune information sur son pedigree n'est disponible (17).
Il peut être déterminé par la formule suivante:
FROH j = (∑k length ROH k)/L
Avec ROH k : le kème ROH dans le génome de l'individu j et L : la longueur du génome couverte par le panel de consensus final.
Coefficient de Consanguinité FHOM ou FIS
Le coefficient FHOM est une mesure de la déviation des proportions de Hardy-Weinberg illustrant la perte d’hétérozygotie (18).
Il est calculé selon la formule suivante :
FHOM = (O-E)/(L-E)
Où O est le nombre de SNPs homozygotes observés, E est le nombre de SNPs homozygotes théorique ou attendu selon l’hypothèse de Hardy-Weinberg, et L est le nombre de SNPs autosomiques génotypés (19 ).
Contrairement à FROH, qui révèle le niveau d'homozygotie indépendamment des fréquences alléliques, le FHOM est influencé par ces fréquences alléliques et par conséquent par l'échantillonnage (20).
FHOM n’est pas influencé uniquement par la consanguinité.
La pression de la sélection naturelle et l'effet Wahlund (excès d’homozygotie par rapport à l’équilibre de HardyWeinberg) peuvent aussi entrainer l’augmentation des valeurs de ce coefficient (21).
Motivations des mariages consanguins
Motivations historique et religieuse
Bien que les religions monothéistes comme le Judaïsme, le Christianisme et l’Islam condamnent les unions incestueuses, quelques minorités de Juifs Marocains pratiquent encore les unions entre oncle et nièce (22).
Contrairement aux idées reçues, l’Islam n’encourage ni interdit les unions consanguines.
Chez la plupart des musulmans, les unions entre cousins germains sont une pratique matrimoniale préférentielle (23).
Certaines règles de l’Islam concernant l’héritage renforcent également la tendance traditionnelle aux mariages consanguins (24 ).
Motivations d’ordre culturel et socio-économique
Les raisons conduisant au choix matriomonial endogame sont aussi liées à des impératifs socioculturels ou politico-économiques (25).
Ces unions servent d’éviter la dispersion du patrimoine familial «N’offrons pas notre bien à autrui» comme le dit l’adage Tunisien.
Ces alliances sont alors considérées comme garant pour conserver l’esprit de la généalogie et aussi pour maintenir les liens familiaux en diminuant les divorces et les ruptures et avoir également la sécurité, le réconfort et la tranquillité et éviter la peur de l’étranger (23).
La stratégie du choix du conjoint est dictée par les coutumes et les règles de vie au sein du clan et toute déviation induirait une sorte de reproche et du blâme de la part de la famille voire une sanction de celle-ci (26).
Motivations géographiques
L’environnement social et géographique joue un rôle très important dans le choix matrimonial et donc dans la circulation de flux de gènes au sein des populations humaines.
Une étude faite en Tunisie a mis en évidence une corrélation positive entre le choix du conjoint et le milieu de résidence (26 ).
Il a été démontré également qu’en milieu rural où la propriété de terrains agricoles et le travail de la terre sont déterminants, le taux de la consanguinité est important, atteignant 25,4% (26) alors qu’en milieu urbain, siège de modernité et d’industrialisation, le choix du conjoint semble être plus libre et moins dicté par la famille (26).
Prévalences et distribution géographique des mariages consanguins en Tunisie
Consanguinité en Afrique du Nord
Les fréquences des mariages consanguins les plus élevées ont été enregistrées en Afrique du Nord, au MoyenOrient, en Asie centrale et en Asie du Sud.
En effet, entre 20 et plus de 50 % des unions au sein de ces populations ont été rapportées être entre cousins issus de germains ou plus proches (10).
En Afrique du Nord, les mariages consanguins semblent être une norme profondément enracinée (2).
Au Maroc, les mariages consanguins représentent 19,9 à 25,4% de tous les unions (27, 28 ).
En Algérie, une étude des registres d'état civil nationaux a estimé que 22,6% de tous les mariages de la population étudiée étaient entre apparentés (29), mais une enquête nationale plus récente a trouvé une valeur plus élevée de 39% (30 ).
Les taux d’endogamie sont également élevés en Égypte, avec une estimation initiale de 23,2 à 35,3 % (31, 32), variant selon les régions.
La fréquence des mariages consanguins varie également selon les groupes ethniques et religieux (33).
Les données sur la prévalence des unions familiales sont rares tant en Libye qu'en Mauritanie, bien que l'on pense que les taux soient élevés.
En Libye, une enquête sur les données des cliniques familiales a montré que 37,6% de tous les mariages sont intrafamiliaux dans la ville de Benghazi (34), et une étude en Mauritanie a estimé un taux de 47,2% (35).
Consanguinité en Tunisie
La population Tunisienne, comme d’autres populations arabes, s’identifie comme une population endogame avec un taux élevé de consanguinité et une distribution différente selon les régions du pays (36) ainsi nous observons que le taux de mariage consanguin augmente selon un gradient Nord-Sud.
Il a été également rapporté qu’en Tunisie 87% des mariages ont lieu entre des personnes partageant la même origine géographique (endogamie géographique) et 29,8 % des mariages se font entre des individus apparentés (consanguins) (37) (Tableau 2).
Les mariages consanguins en Tunisie est une pratique matrimoniale préférable et répandue notamment dans les régions rurales (26 , 37, 38).
L’endogamie fait référence à l’endogamie géographique, c'est-à-dire que le couple est originaire de la même région géographique.
Quelques études ont rapporté la prévalence de telles unions en Tunisie montrant des taux élevés dépassant les 70 %. En effet, elle varie entre 71,48 % dans la région de Nabeul (39) à 98,16 % au Sud Tunisien (37).
Tableau 2 : Profils d’endogamie géographique et de consanguinité dans la population Tunisienne selon des études précédentes .
|
Population d’étude |
Taux de consanguinité (%) |
Taux de cousins germains (%) |
Moyenne de coefficient de consanguinité |
Taux d’endogamie géographique (%) |
Référence |
|
Tunisie (régions urbaines) |
16,30 |
- |
- |
- |
(71) |
|
Tunisie (régions rurales) |
25,36 |
- |
- |
- |
(71) |
|
Tunisie |
38,02 |
18,18 |
- |
87,75 |
(4) |
|
Tunisie |
29,80 |
16,72 |
- |
- |
(5) |
|
Nord de la Tunisie |
31,62 |
- |
0,0213 |
- |
(72) |
|
Bizerte |
16,00 |
- |
0,0093 |
- |
(72) |
|
Kef |
38,00 |
- |
0,0157 |
- |
(72) |
|
Siliana |
41,40 |
- |
0,0178 |
- |
(72) |
|
Beja |
36,00 |
- |
- |
- |
(72) |
|
Centre de la Tunisie |
49,60 |
- |
- |
- |
(72) |
|
Nabeul |
33,64 |
19,39 |
0,0129 |
71,48 |
(39) |
|
Sfax |
35,50 |
17,33 |
0,0139 |
90,00 |
(73) |
|
Grand Tunis |
32,71 |
- |
0,0121 |
- |
(74) |
|
Sud Tunisien |
65,26 |
26,50 |
0,0216 |
98,16 |
(37) |
|
Monastir |
20,10 |
- |
0,0084 |
- |
(75) |
|
Monastir |
24,81 |
17,14 |
- |
- |
(75) |
Impact de la consanguinité sur l'expression des maladies génétiques en Tunisie
Impact sur la prévalence et la santé
Plus de 340 maladies génétiques ont été rapportées affectant la population Tunisienne dont une majeure partie est transmise selon le mode autosomique récessif (6).
Dans une étude récente, la mise à jour du spectre indique 589 maladies génétiques et que 60 % d’entre elles sont autosomiques récessives ( 3)
Dans une large série de patients de notre laboratoire, la consanguinité est souvent retrouvée avec une fréquence supérieure ou égale à 80%.
L’endogamie géographique est aussi présente, avec les mêmes plages de valeurs témoignant l’isolement géographique et social des familles de patients.
Plus encore, les résultats ont révélé la fragmentation de la population Tunisienne en communautés isolées, conduisant à une augmentation du risque de l’expression des maladies génétiques (Non publiés).
Peu d’études se sont focalisées sur l’impact de la consanguinité sur la santé à cause du manque des études épidémiologiques.
Quelques auteurs ont rapporté une corrélation positive entre les unions intrafamiliales et des désordres d’origine génétique comme la polydactylie, la surdité, les maladies neurodégénératives, la bipolarité, des maladies métabolique et le Xeroderma pigmentosum (40, 41, 42 ).
Sur le plan de la santé reproductive, les couples apparentés ont un plus fort indice de fertilité que les couples non apparentés.
Bien que les taux d’avortements spontanées ou fausse-couches et mortinaissances ne sont pas corrélés à la consanguinité, des taux élevés de mortalité néonatale et post-natale et celle des enfants en bas âge (inférieur à 5 ans) ont été observés chez les couples apparentés (38).
Dans ce contexte particulier de fertilité et de mortalité néonatale, des facteurs supplémentaires comme le statut socioéconomique des parents, l’espacement des grossesses, l’utilisation de moyens contraceptifs et la résidence des parents devraient être pris en considération (38).
Une étude plus récente faite dans notre laboratoire, impliquant 1289 individus non apparentés atteints de différentes maladies génétiques et 1067 volontaires non apparentés sains de toutes pathologies, a mis en évidence l’association entre la consanguinité et l’occurrence de chaque groupe pathologique (5).
Il a été ainsi démontré que l’association entre les unions entre apparentés et les maladies autosomiques récessives est hautement significative indiquant un risque multiplié par 8,53 d’exprimer de telles maladies sous un régime matrimonial non panmictique (OR=8,53 ; IC à 95% = [6,7-10,86]).
Ce risque reste élevé est significatif même après ajustement des effets confondants des variables démographiques comme le sexe, l’âge et l’origine géographique.
Les unions intrafamiliales augmentent alors 6 fois le risque d’expression des maladies autosomiques récessives(OR=5,9 ; IC à 95%=[4,22-8,25]).
Par ailleurs, nos résultats ne montrent aucune association entre la consanguinité et l’occurrence des maladies dominantes et multifactorielles.
Plus encore, le type de mariage consanguin est un facteur de risque croissant de développer une maladie autosomique récessive.
En effet, le risque en lien avec les unions entre apparentés augmente d’une manière significative en fonction du degré de consanguinité : OR = 3,9 ; IC à 95% = [4,07-8,55] pour les unions audelà du 2ème degré (F<,0156) ; OR=4,69 ; IC à 95% = [7,34 -16,2] pour les unions entre cousins du deuxième degré (0,0156<F<0,0625) et finalement OR= 7,68 pour les unions entre cousins du premier degré (F>=0,0625) (5).
Impact différentiel de la consanguinité sur l’expression des maladies génétiques
La consanguinité est connue comme étant un facteur de risque majeur quant à l’expression des maladies autosomiques récessives.
En Tunisie, elle est associée à un risque de 10 fois d’expression de la surdité non syndromique due à la mutation 35delG chez les unions entre cousins germains (43).
Dans une étude plus récente, un impact différentiel de la consanguinité sur l’expression de maladies génétiques autosomiques récessives a été mis en évidence (37).
La consanguinité a été observée chez 65% des patients étudiés et la consanguinité variait entre 51,3% et 75,6% pour chaque pathologie.
Il a été alors démontré que la consanguinité augmentait le risque de 6 fois d’exprimer la maladie de Gaucher (OR= 5,53, CI = 1,77–17,26, p = 0,03).
Le risque associé à l’endogamie familiale d’exprimer l’épidermolyse bulleuse était estimé à 16 fois ( OR = 15,17, CI = 3,67–62,91, p< 10-3 ).
Le risque d’exprimer Xeroderma pigmentosum en cas d’union consanguine est augmenté de 25 (OR = 24,41,CI = 9,99–59,61, p < 10-3) (37 ).
Impact sur l’expression des maladies autosomiques dominantes
Dans les populations consanguines, les mariages entre apparentés ne modifient pas la probabilité d’expression d’une maladie autosomique dominante, mais plutôt en affecte le phénotype.
En Tunisie, plus de 150 maladies autosomiques dominantes ont été rapportées (3) dont 15,6 % sont des maladies du système nerveux, 10,2 % sont des maladies métaboliques et 8,16 % sont des pathologies cancéreuses (Fatnassi, Non publié).
Les cas d’homozygotie pour des mutations dominantes sont rares, probablement, parce qu’elles conduiraient à un embryon non viable, donc, seraient létales.
L’homozygotie pour une mutation au gène COL7A1 responsable de l’épidermolyse bulleuse dystrophique a engendré un phénotype très sévère dans une famille consanguine(44).
De même, la prévalence de l’hypercholestérolémie familiale au Centre et au Sud du pays en association avec la consanguinité a causé l’augmentation de la fréquence des patients à l’état homozygote qui montraient un phénotype plus sévère que les patients hétérozygotes (45,46) .
Un autre exemple illustrant comment une mutation dominante à l’état homozygote peut altérer l’expression d’un phénotype est donné par une variation dans le gène CASR.
Les parents, qui étaient hétérozygotes pour la mutation, avaient une hypercalcémie hypocalciurique familiale.
Leur fille consanguine avait la mutation à l’état homozygote et présentait un tableau clinique d’hyperparathyroïdie néonatale sévère (47 ).
Dans une étude récente sur une large famille consanguine atteinte de dystrophie cornéenne granulaire de type I, les individus homozygotes pour la mutation au gène TGFBI présentaient un phénotype sévère et atypique ( 48).
Impact sur la comorbidité
La comorbidité désigne la présence d’une ou de plusieurs maladies en plus de la maladie primaire ou à l’effet de ces maladies supplémentaires (49).
Différents modèles d'associations de comorbidité ont été décrits.
La comorbidité individuelle désigne l'association de maladies comorbides chez un individu, tandis que la comorbidité familiale désigne l'apparition de plusieurs maladies génétiques chez différents membres d'une même famille élargie, de manière combinatoire (50)
En Tunisie, 75 associations comorbides ont été identifiées(50), dont 39 ont été rapportées dans des familles consanguines.
La consanguinité est en effet suggérée être un facteur majeur à l’homozygotie des variants délétères conduisant à des expressions comorbides.
La classification des catégories de comorbidité a montré que la classe des associations maladie génétique – maladie génétique est la plus fréquente (50 %), dont 34% sont des maladies à transmission autosomique récessive.
La fréquence élevée du groupe pathologique formé par les maladies autosomiques récessives suppose que la consanguinité est un facteur majeur à l’homozygotie des variants délétères conduisant à des expressions comorbides.
D’autre part, l’homogamie peut contribuer à la comorbidité et expliquer la co-expression de différentes maladies d’origine génétiques appartenant ou non au même groupe pathologiques (36).
Récemment, 14 associations comorbides ont été rapportées ( 3).
Le nombre élevé (89 au totales) des cas de comorbidité prouvent que celle-ci n’est pas exceptionnelle chez les familles Tunisiennes.
Impact de la consanguinité sur la structure du génome
Découverte et mécanismes de production des ROHs
Les ROHs ont été découvertes pour la première fois au moyen d’analyses de microsatellites à l'échelle du génome, au milieu des années 1990 (51).
Il a été rapporté que les membres de deux familles recrutées afin de construire les premières cartes génétiques humaines étaient porteurs de 4 à 16 ROHs dont les longueurs varient de à peu près 1,5 à 30 Mb et l'individu le plus extrême avait une longueur totale de ROH d’à peu près 195 Mb (51 ).
Bien que les ROHs puissent se produire par différents mécanismes, il est présumé que leur cause principale est la consanguinité.
Les ROHs longues sont très probablement le résultat d'une consanguinité récente, où les événements de recombinaison ne raccourcissent pas les haplotypes identiques hérités de l'ancêtre commun (16)
Les ROHs courtes, en revanche, suggèrent une consanguinité plus ancienne ou un appariement aléatoire d'haplotypes anciens dont la fréquence a été amplifiée par des événements fondateurs(52).
La capacité des ROHs à révéler des informations sur des événements génétiques anciens et récents en fait des outils utiles pour analyser l'histoire des populations, les niveaux de consanguinité et les effets de la consanguinitésur des traits complexes et les troubles congénitaux (16).
Par ailleurs, les régions d’homozygotie se sont avérées ubiquitaires, même dans les populations non consanguines, et quelques études ont montré que les ROHs avec des longueurs supérieures à 1Mb sont plus fréquentes chez les individus étroitement apparentés qu'on ne le pensait auparavant (7 ).
En effet, tous les êtres humains sont consanguins à un certain degré et les ROHs rendent compte de cet aspect de l’histoire démographique individuelle (15).
Les méthodes de génotypages et de séquençage à haut débit ont largement facilité leurs identifications depuis.
Distribution des ROHs dans le génome
Les ROHs de différentes tailles ont des schémas d'apparition différents le long du génome (9).
En effet, elles sont un peu plus fréquentes dans les régions à fort déséquilibre de liaison (DL) et à faible recombinaison (53) et sont particulièrement répandues sur le chromosome X (54) et dans les régions à faible diversité génétique (55).
Des études ont déjà suggéré que les ROHs se regroupent dans des régions du génome où les taux de recombinaison sont faibles (7, 53, 54 , 56, 57 ).
La recombinaison fragmente les segments chromosomiques au fil des générations. Ainsi, les régions à faible recombinaison permettent une plus grande persistance des longs haplotypes ancestraux et augmentent les chances qu'ils se rassemblent pour former des ROHs (21).
La distribution des ROHs est très irrégulière le long du génome avec un certain nombre de régions relativement courtes présentant des excès importants de ROHs, appelées îlots de ROHs ou hotspots de ROHs, sur chaque chromosome (9, 55) ainsi que des coldspots.
L'existence de hotspots et de coldspots de ROHs peut être expliquée en partie comme une conséquence de la stochasticité des événements de recombinaison à travers le génome ou à cause des effets des processus démographiques influençant la diversité génétique (9).
Répartition géographique des ROHs et histoire démographique
Dans le monde
Les ROHs dont la taille est supérieure à 0,5 Mb sont ubiquitaires dans toutes les populations ( 58).
Une étude, utilisant près de 700 000 génotypes de micropuces SNP pour 209 individus HapMap, a révélé pour la première fois à quel point les ROHs à l'échelle d'une mégabase étaient répandues, même parmi les individus non consanguins(53).
Les ROHs de différentes tailles ont des distributions géographiques et des modèles d'occurrence diverses à travers le génome.
Le nombre des ROHs courts et intermédiaires augmente de manière progressive dans les groupes continentaux et présentent des valeurs relativement similaires au sein des continents (9).
Ceci peut être expliqué comme la conséquence d'un modèle migratoire en série vers l'extérieur de l'Afrique, c’est à dire chaque migration diminue la taille effective de la population, générant un déséquilibre de liaison, réduisant la diversité des haplotypes et augmentant la probabilité que des copies identiques du même haplotype s'apparient chez le même individu.
Les longues ROHs, qui résultent en grande partie de la consanguinité, ne suivent pas un tel schéma et sont au contraire plus fréquentes dans les populations où l'isolement et les unions consanguines sont plus courants.
Ce qui fait que les différents schémas continentaux observés pour différentes classes de ROHs reflètent alors des forces distinctes générant des ROHs de tailles différentes (9)
Les longues ROHs fournissent des informations sur une histoire ancestrale plus récente, allant de la taille de la population et de l'endogamie, à la consanguinité récente.
Cependant, contrairement à la diversité génétique, au déséquilibre de liaison et à d'autres statistiques récapitulatives des généalogies, les ROHs sont un phénomène au niveau individuel et fournissent donc un registre distinct de l'histoire démographique des ancêtres d'un individu (58 ).
Certainement les ROHs dépendent de la taille de la population, avec les petites populations ayant des ROHs plus nombreux et plus longs que les populations de taille plus grande.
Le métissage rassemble différents haplotypes et réduit généralement le nombre de ROHs à un très petit nombre de ROHs courtes, tandis que les goulots d'étranglement augmentent le nombre de ROHs, qui restent en général relativement courtes.
La consanguinité, quant à elle, ajoute un petit nombre de ROHs très longs pour ceux qui sont issus d'un mariage entre cousins, ce qui augmente également la variance de la longueur totales des ROHs (21 ).
En Tunisie
Peu d’études se sont intéressées à analyser les ROHs dans la population tunisienne.
Majoritairement ce sont des populations berbères qui ont fait l’objet de telles études en raison de leur caractère isolé dans le paysage démographique complexe de l'Afrique du Nord.
La première étude s’est intéressée à la diversité génétique de l’Afrique du Nord à partir de 730 000 SNPs. Henn et al.,a mis en évidence la présence de longues ROH chez les Berbères Tunisiens dont la taille est deux fois supérieure à celles observées dans les autres populations Nord Africaines.
L’analyse des segments IBD a montré un excès de paires d’individus partageant des segments IBD entre 200-1200 cM.
La distribution bimodale des segments IBD des Berbères Tunisiens était atypique révélant une parenté de type cousins de premier et second degré bien que les participants à l’étude se déclaraient non apparentés.
Ces résultats supposent que les profils des ROHs et des segments IBD suggèrent une endogamie due à l’isolement géographique et probablement à une préférence aux mariages entre apparentés (59).
Plus encore, cette étude met en cause le problème de l’estimation du coefficient de consanguinité à partir des pedigrees qui ignore les relations familiales lointaines qui sont absentes de la plupart des généalogies (58), ou celles à partir de population où les mariages non panmictiques y ont à la fois fréquents et anciens.
Une étude sur la population de Douiret, qui est hautement endogame, a révélé un nombre imoprtant de ROHs> 0,5 Mb, environ 48,24 régions par individus(60).
La longueur moyenne des ROHs est de 1,77 Mb avec des valeurs allant de1,31 à 2,68 Mb.
Le FROH0,5 moyen est de 0,036 signifiant qu’en moyenne, chaque individu a 3,06% de son génome homozygote par descendance.
L’étude des régions d’homozygotie selon leurs tailles montrent des taux élevés de ROHs courts représentant 0,93% du génome de l’individu.
D’autre part, les ROHs de plus grande taille ≥ 5Mb qui sont moins abondants et aussi représentent 0,95% du génome.
Ce résultat indique la présence d’une parente éloignée et récente (60).
Une autre analyse des ROHs d’un échantillon non berbère de la population de Médenine, elle aussi connue par son isolement et la consanguinité en raison des différences religieuses et culturelles, a montré un nombre moyen de ROHs élévé par individu (~30 ROHs).
Les ROHs de courte taille indicatrices d’une consanguinité éloignée représentaient 0,51% de l’ensemble du génome alors que celles de taille intermédiaire et de longue taille,indicatrices de consanguinité plus récente, couvraient respectivement 1,13 et 3,63 % du génome (Sayeb et al.,Non publié)
Une autre étude sur l'impact de la démographie dans la distribution des variations fonctionnelles à partir de données d’exomes dans les populations nord-africaines suggère que les Berbères tunisiens sont porteurs d'un nombre significativement plus élevé de ROHs que les non berbères (61 ).
Cela s'explique par le fait que le groupe berbère présente des signes d'isolement génétique, peut-être après un goulot d'étranglement relativement récent, et suivi d'une forte dérive génétique et de consanguinité (61).
Dans une récente étude, nous avons effectué une analyse des ROHs à partir de données de génotypage à haut débit provenant de 260 individus tunisiens, de deux groupes ethniques : Berbères et non Berbères, collectées à partir de différentes sources (Abassi et al., Non publié).
Des nombres importants de ROHs ont été identifiés par individu (39,72 ROHs en moyenne).
Les deux groupes ethniques ne présentaient pas une différence significative en termes de nombres et longueurs totales des ROHs.
L’enrichissement en ROHs était majoritairement en régions de taille moyenne (1,5-4,99 Mb) et était maximal chez Zraoua et couvrant 1,7 % du génome.
La valeur maximale de FROH dans les deux seuils de longueur de ROHs était comparable au FPED de cousins germains.
Dans le cas de Matmata et Tamezret, les ROHs sont plutôt le résultat d’une consanguinité.
Pour Sened et Tataouine, l’homozygotie semble être produite par dérive génétique.
L’homozygotie dans Douiret semble être provoquée par l'isolement génétique et la dérive génétique.
Notre étude a mis l’accent sur la nécessité d’approfondir l’investigation des îlots ROHs dans la population tunisienne, afin d’identifier de potentiels gènes impliqués dans des cas de comorbidité, des maladies rares ou complexes (Abassi et al., Non publié)
Limite de l’approche généalogique dans l’estimation de l’homozygotie
Comme mentionné ultérieurement, l’estimation du coefficient de consanguinité individuel F repose sur l’examen de la généalogie de l’individu en identifiant les chemins généalogiques connus de l’ancêtre commun jusqu’à l’individu.
Ainsi, pour une bonne estimation de la consanguinité et de l’homozygotie, le calcul de ce coefficient devrait être basé sur des données généalogiques extensives s’étendant sur plusieurs générations comme il a été rapporté dans les études sur les Canadiens Français et les Amish ( 62, 63), autrement, il sera difficile de capturer l’information relative à la consanguinité et à l’homozygotie à l’échelle individuelle et populationnelle.
Cette observation est vraie dans le cas des populations isolées à faible nombre de fondateurs qui limitent le choix matrimonial et donc sont caractérisées par une pratique prolongée de la consanguinité.
La détection des liens de parenté entre les individus y est souvent difficile car les généalogies y sont très complexes avec de multiples boucles de consanguinité inconnues(64).
Ainsi, il est difficile d’obtenir une estimation fiable du coefficient de consanguinité à partir des données généalogiques.
Par ailleurs, la fiabilité de l’estimation de l’homozygotie au niveau populationnel a été remis en question en raison des problèmes de manques de données généalogiques, de fausses paternités ou d’adoptions non déclarées (65).
Cartographie par homozygotie
En 1987, Lander et Botstein ont proposé une approche par descendance encore appelée homozygotie par filiation afin d’établir une localisation primaire des loci responsables de maladies récessives rares ( 66).
Le principe de cette analyse est le suivant : un individu consanguin atteint d’une maladie récessive hérite non seulement deux copies d’une même mutation à partir d’un ancêtre commun mais également une région chromosomique entourant le locus morbide identique par descendance à partir d’un ancêtre commun à ses parents.
Par conséquent, il est possible d’identifier les régions homozygotes grâce aux haplotypes établis avec des marqueurs polymorphes situés dans la région des gènes candidats.
Contrairement à la méthode classique d’analyse de liaison, la méthode de cartographie par homozygotie par descendance ne nécessite pas de disposer de familles avec plusieurs individus atteints, qui sont particulièrement rares quand on s’intéresse à des maladies récessives.
En absence de consanguinité, cette méthode n’est évidemment plus applicable.
En Tunisie, le clonage positionnel par homozygotie par descendance couplée au séquençage d’exome entier a permis d’identifier quatre mutations dans le gène FGD4 confirmant ainsi la maladie de Charcot-Marie-Tooth de type 4 chez quatre familles consanguines d’origine tunisienne (67).
De plus, la cartographie par homozygotie a permis de caractériser chez une famille tunisienne l’ataxie cérébelleuse progressive dont au moins 14 gènes sont impliqués.
Le gène SACS a été ainsi retrouvé dans une région d’homozygotie commune à deux membres atteints de la famille au-dessus du seuil de 20 SNPs successifs homozygotes.
Ce résultat a permis d’affirmer l’ataxie spastique du Charlevoix-Saguenay chez ces deux patients (68).
L’approche d’homozygotie par descendance était également efficace pour l’étude de l’infertilité masculine qui représente un ensemble de pathologies très hétérogènes et multigéniques.
Ainsi, la cartographie par homozygotie d’une cohorte de patients infertiles ayant un phénotype spermatique de type Malformations Multiples des Flagelles, dont 11 sont des tunisiens, a rendu possible la découverte du gène DNAH1 comme étant la cause de cette infertilité chez 28% de ces patients (69).
Conclusion
Bien que l’impact de la consanguinité sur l’émergence des maladies génétiques soit bien établi dans notre population, son impact sur la structure du génome tunisien reste à être élucidé.
Nous avons tenté dans ce document de centraliser l’information sur la consanguinité en Tunisie de sa prévalence à l’impact sur la santé et la dynamique du génome.
Comme le spectre des maladies génétiques montre qu’une grande proportion reste d’étiologie moléculaire inconnue où le gène n’est pas encore identifié (3), une stratégie reposant sur la cartographie par homozygotie pourrait largement y remédier
En pratique médicale, les nouvelles technologies de séquençage à haut débit ont facilité la découverte de la ou des mutations responsables du phénotype étudié.
Cette approche, combinée au génotypage à haut débit chez un échantillon de la population Qatarie, aussi connue pour ses forts taux de consanguinité, a permis de mettre en évidence un enrichissement de variants potentiellement délétères dans les longues ROHs comparé aux régions pauvres en ROHs suggérant ainsi que la consanguinité est un mécanisme conduisant à l’enrichissement du génome par des variants potentiellement pathogènes à l’état homozygote (70).
Il serait judicieux d’étudier la distribution et les propriétés des ROHs dans la population générale Tunisienne afin d’identifier les gènes récessifs impliqués dans les maladies monogéniques et multifactorielles et la comorbidité dans la population Tunisienne.
References
- Romdhane L, Kefi R, Azaiez H, Ben Halim N, Dellagi K, Abdelhak S. Founder mutations in Tunisia: implications for diagnosis in North Africa and Middle East. Orphanet J Rare Dis. 2012;7:52. doi: 10.1186/1750-1172-7-52. [DOI] [PMC free article] [PubMed] [Google Scholar]
- Romdhane L, Mezzi N, Hamdi Y, El-Kamah G, Barakat A, Abdelhak S. Consanguinity and Inbreeding in Health and Disease in North African Populations. Annu Rev Genomics Hum Genet. 2019;20:155–179. doi: 10.1146/annurev-genom-083118-014954. [DOI] [PubMed] [Google Scholar]
- Mezzi N, Messaoud O, Mkaouar R, Zitouna N, Romdhane S, Abdessalem G, et al. Spectrum of Genetic Diseases in Tunisia: Current Situation and Main Milestones Achieved. Genes (Basel) 2021;12(11) doi: 10.3390/genes12111820. [DOI] [PMC free article] [PubMed] [Google Scholar]
- Zakaria D. Faculty of Science, University of Tunis. 1999. Etude de l’endogamie d’origine régionale, de la distribution de la consanguinité apparente et du comportement intergénérationnel dans le choix matrimonial en Tunisie. Intérêt des noms de famille et de l’isonymie maritale. [Google Scholar]
- Ben Halim N, Ben Alaya Bouafif N, Romdhane L, Kefi Ben Atig R, Chouchane I, Bouyacoub Y, et al. Consanguinity, endogamy, and genetic disorders in Tunisia. J Community Genet. 2013;4(2):273–284. doi: 10.1007/s12687-012-0128-7. [DOI] [PMC free article] [PubMed] [Google Scholar]
- Romdhane L, Abdelhak S, et al. Genetic diseases in the Tunisian population. American Journal of Medical Genetics Part A. 2011;155A(1):238–267. doi: 10.1002/ajmg.a.33771. [DOI] [PubMed] [Google Scholar]
- McQuillan R, Leutenegger A-L, Abdel-Rahman R, Franklin CS, Pericic M, Barac-Lauc L, et al. Runs of homozygosity in European populations. The American Journal of Human Genetics. 2008;83(3):359–372. doi: 10.1016/j.ajhg.2008.08.007. [DOI] [PMC free article] [PubMed] [Google Scholar]
- Ceballos FC, Joshi PK, Clark DW, Ramsay M, Wilson JF. Runs of homozygosity: windows into population history and trait architecture. Nat Rev Genet. 2018;19(4):220–234. doi: 10.1038/nrg.2017.109. [DOI] [PubMed] [Google Scholar]
- Pemberton TJ, Absher D, Feldman MW, Myers RM, Rosenberg NA, Li JZ. Genomic patterns of homozygosity in worldwide human populations. Am J Hum Genet. 2012;91(2):275–292. doi: 10.1016/j.ajhg.2012.06.014. [DOI] [PMC free article] [PubMed] [Google Scholar]
- Bittles A. Consanguinity and its relevance to clinical genetics. Clin Genet. 2001;60(2):89–98. doi: 10.1034/j.1399-0004.2001.600201.x. [DOI] [PubMed] [Google Scholar]
- Hamamy H, Antonarakis SE, Cavalli-Sforza LL, Temtamy S, Romeo G, Kate LP, et al. Consanguineous marriages, pearls and perils: Geneva International Consanguinity Workshop Report. Genet Med. 2011;13(9):841–847. doi: 10.1097/GIM.0b013e318217477f. [DOI] [PubMed] [Google Scholar]
- Bou-Assy F, Dumont S, Saillant F. Représentations sociales du mariage endogame et de ses conséquences biologiques sur la santé des descendants chez des fiancés apparentés: Cas de deux villages chiites au Liban. Service social. 2003;50(1):174–198. [Google Scholar]
- Wright S. Coefficients of inbreeding and relationship. The American Naturalist. 1922;56(645):330–338. [Google Scholar]
- Tabah L, Sutter J. La mesure de la consanguinité: Perspectives d'application a la démographie. Population (French Edition) 1950:689–712. [Google Scholar]
- Keller MC, Visscher PM, Goddard ME. Quantification of inbreeding due to distant ancestors and its detection using dense single nucleotide polymorphism data. Genetics. 2011;189(1):237–249. doi: 10.1534/genetics.111.130922. [DOI] [PMC free article] [PubMed] [Google Scholar]
- Ferencakovic M, Hamzic E, Gredler B, Solberg TR, Klemetsdal G, Curik I, et al. Estimates of autozygosity derived from runs of homozygosity: empirical evidence from selected cattle populations. J Anim Breed Genet. 2013;130(4):286–293. doi: 10.1111/jbg.12012. [DOI] [PubMed] [Google Scholar]
- Szmatola T, Gurgul A, Jasielczuk I, Zabek T, Ropka-Molik K, Litwinczuk Z, et al. A Comprehensive Analysis of Runs of Homozygosity of Eleven Cattle Breeds Representing Different Production Types. Animals (Basel) 2019;9(12) doi: 10.3390/ani9121024. [DOI] [PMC free article] [PubMed] [Google Scholar]
- Caballero A, Villanueva B, Druet T. On the estimation of inbreeding depression using different measures of inbreeding from molecular markers. Evol Appl. 2021;14(2):416–428. doi: 10.1111/eva.13126. [DOI] [PMC free article] [PubMed] [Google Scholar]
- Dixit SP, Singh S, Ganguly I, Bhatia AK, Sharma A, Kumar NA, et al. Genome-Wide Runs of Homozygosity Revealed Selection Signatures in Bos indicus. Front Genet. 2020;11:92. doi: 10.3389/fgene.2020.00092. [DOI] [PMC free article] [PubMed] [Google Scholar]
- Zhang Q, Guldbrandtsen B, Bosse M, Lund MS, Sahana G. Runs of homozygosity and distribution of functional variants in the cattle genome. BMC Genomics. 2015;16(1):542. doi: 10.1186/s12864-015-1715-x. [DOI] [PMC free article] [PubMed] [Google Scholar]
- Ceballos FC, Hazelhurst S, Ramsay M. Assessing runs of Homozygosity: a comparison of SNP Array and whole genome sequence low coverage data. BMC Genomics. 2018;19(1):106. doi: 10.1186/s12864-018-4489-0. [DOI] [PMC free article] [PubMed] [Google Scholar]
- Fried K, Davies AM. Some effects on the offspring of uncle-niece marriage in the Moroccan Jewish community in Jerusalem. American Journal of Human Genetics. 1974;26(1):65–72. [PMC free article] [PubMed] [Google Scholar]
- Hussain R. Community perceptions of reasons for preference for consanguineous marriages in Pakistan. Journal of Biosocial Science. 1999;31(4):449–461. doi: 10.1017/s0021932099004496. [DOI] [PubMed] [Google Scholar]
- Al-Gazali L, Hamamy H, Al-Arrayad S. Genetic disorders in the Arab world. Bmj. 2006;333(7573):831–834. doi: 10.1136/bmj.38982.704931.AE. [DOI] [PMC free article] [PubMed] [Google Scholar]
- Chalbi N, Zakaria D. Modèles de famille, endogamie et consanguinité apparente en Tunisie. Essais de mesure. Famille Population. 1998;1:39–59. [Google Scholar]
- M'Rad LB, Chalbi N. Milieu de résidence origine des conjoints et consanguinité en Tunisie. Antropo. 2006;12:63–71. [Google Scholar]
- Lamdouar Bouazzaoui N. [Consanguinity and public health in Morocco] Bull Acad Natl Med. 1994;178(6):1013–1025. discussion 25-7. [PubMed] [Google Scholar]
- Talbi J, Khadmaoui A, Soulaymani A, Chafik A. Etude de la consanguinité dans la population marocaine. Impact sur le profil de la santé. Antropo. 2007;15:1–11. [Google Scholar]
- Benallegue A, Kedji F. Consanguinité et santé publique: Etude algérienne. Archives françaises de pédiatrie. 1984;41(6):435–440. [PubMed] [Google Scholar]
- Guidoum M, Kefi R, Abdelhak S, Bouslama Z. Consanguinity and endogamy of a Northeastern Algerian population (population of El-Kala) Advances in Environmental Biology. 2015:457–466. [Google Scholar]
- Habib Z, Book JA. Consanguinity and incidence of thalassaemia in Egypt. Hereditas. 1983;99(2):215–217. doi: 10.1111/j.1601-5223.1983.tb00894.x. [DOI] [PubMed] [Google Scholar]
- Shawky RM, El-Awady MY, Elsayed SM, Hamadan GE. Consanguineous matings among Egyptian population. Egyptian Journal of Medical Human Genetics. 2011;12(2):157–163. [Google Scholar]
- Badr FM. Genetic studies of Egyptian Nubian populations. I. Frequency and types of consanguineous marriages. Hum Hered. 1972;22(4):387–398. doi: 10.1159/000152515. [DOI] [PubMed] [Google Scholar]
- Abudejaja AH, Khan MA, Singh R, Toweir AA, Narayanappa M, Gupta BS, et al. Experience of a family clinic at Benghazi, Libya, and sociomedical aspects of its catchment population. Fam Pract. 1987;4(1):19–26. doi: 10.1093/fampra/4.1.19. [DOI] [PubMed] [Google Scholar]
- Hammami A, Elgazzeh M, Chalbi N, Mansour BA. Endogamy and consanguinity in Mauritania. La Tunisie Medicale. 2005;83(1):38–42. [PubMed] [Google Scholar]
- Romdhane L, Ben Halim N, Rejeb I, Kefi R, Bouyacoub Y, Ben Rekaya M, et al. Specific aspects of consanguinity: some examples from the Tunisian population. Hum Hered. 2014;77(1-4):167–174. doi: 10.1159/000362167. [DOI] [PubMed] [Google Scholar]
- Ben Halim N, Hsouna S, Lasram K, Rejeb I, Walha A, Talmoudi F, et al. Differential impact of consanguineous marriages on autosomal recessive diseases in Tunisia. Am J Hum Biol. 2016;28(2):171–180. doi: 10.1002/ajhb.22764. [DOI] [PubMed] [Google Scholar]
- Kerkeni E, Monastiri K, Saket B, Guediche MN, Ben Cheikh H. Interplay of socio-economic factors, consanguinity, fertility, and offspring mortality in Monastir, Tunisia. Croat Med J. 2007;48(5):701–708. [PMC free article] [PubMed] [Google Scholar]
- M’Ghirbi J. Diplôme d’études approfondies, Faculté des Sciences de Tunis. 2002. Endogamie, choix matrimonial, consanguinité, facteurs démographiques et socio-économiques dans le Gouvernorat de Nabeul (Tunisie) p. 105. [Google Scholar]
- Ben Arab S, Chalbi N. [Consanguinity and its effects in the Governorate of Bizerte] Tunis Med. 1984;62(7):551–554. [PubMed] [Google Scholar]
- El Gazzah L, Chalbi N, Ben Hamida M. [Effect of consanguinity on certain hereditary-degenerative spinocerebellar diseases in Tunisia] Tunis Med. 1985;63:71–77. [PubMed] [Google Scholar]
- Mechri A, Rouissi K, Mehdi B, Khiari G, Abdelhak S, Gaha L. [Influence of consanguinity on clinical characteristics and affective disorders frequency in relatives of bipolar I patients] Tunis Med. 2007;85(11):951–954. [PubMed] [Google Scholar]
- Ben Arab S, Masmoudi S, Beltaief N, Hachicha S, Ayadi H. Consanguinity and endogamy in Northern Tunisia and its impact on non-syndromic deafness. Genet Epidemiol. 2004;27(1):74–79. doi: 10.1002/gepi.10321. [DOI] [PubMed] [Google Scholar]
- Ouragini H, Cherif F, Kassar S, Floriddia G, Pascucci M, Daoud W, et al. Dystrophic epidermolysis bullosa phenotypes in a large consanguineous Tunisian family. Journal of Dermatological Science. 2009;54(2):114–120. doi: 10.1016/j.jdermsci.2009.01.006. [DOI] [PubMed] [Google Scholar]
- Slimane MN, Pousse H, Maatoug F, Hammami M, Ben Farhat MH. Phenotypic expression of familial hypercholesterolaemia in central and southern Tunisia. Atherosclerosis. 1993;104(1-2):153–158. doi: 10.1016/0021-9150(93)90186-x. [DOI] [PubMed] [Google Scholar]
- Jelassi A, Slimani A, Jguirim I, Najah M, Abid A, Boughamoura L, et al. Moderate phenotypic expression of familial hypercholesterolemia in Tunisia. Clin Chim Acta. 2010;411(9-10):735–738. doi: 10.1016/j.cca.2010.02.008. [DOI] [PubMed] [Google Scholar]
- Sfar S, Bzeouich AA, Kerkeni E, Bouaziz S, Najjar MF, Chouchane L, et al. A novel CASR mutation in a Tunisian FHH/NSHPT family associated with a mental retardation. Mol Biol Rep. 2012;39(3):2395–2400. doi: 10.1007/s11033-011-0990-0. [DOI] [PubMed] [Google Scholar]
- Bouyacoub Y, Falfoul Y, Ouederni M, Sayeb M, Chedli A, Chargui M, et al. Granular type I corneal dystrophy in a large consanguineous Tunisian family with homozygous p.R124S mutation in the TGFBI gene. Ophthalmic Genet. 2019;40(4):329–337. doi: 10.1080/13816810.2019.1639202. [DOI] [PubMed] [Google Scholar]
- Valderas JM, Starfield B, Sibbald B, Salisbury C, Roland M. Defining comorbidity: implications for understanding health and health services. Ann Fam Med. 2009;7(4):357–363. doi: 10.1370/afm.983. [DOI] [PMC free article] [PubMed] [Google Scholar]
- Romdhane L, Messaoud O, Bouyacoub Y, Kerkeni E, Naouali C, Cherif Ben Abdallah L, et al. Comorbidity in the Tunisian population. Clin Genet. 2016;89(3):312–319. doi: 10.1111/cge.12616. [DOI] [PubMed] [Google Scholar]
- Broman KW, Weber JL. Long homozygous chromosomal segments in reference families from the centre d'Etude du polymorphisme humain. Am J Hum Genet. 1999;65(6):1493–1500. doi: 10.1086/302661. [DOI] [PMC free article] [PubMed] [Google Scholar]
- Rosenberg NA, Pemberton TJ, Li JZ, Belmont JW. Runs of homozygosity and parental relatedness. Genetics in Medicine: Official Journal of the American College of Medical Genetics. 2013;15(9):753–754. doi: 10.1038/gim.2013.108. [DOI] [PubMed] [Google Scholar]
- Gibson J, Morton NE, Collins A. Extended tracts of homozygosity in outbred human populations. Hum Mol Genet. 2006;15(5):789–795. doi: 10.1093/hmg/ddi493. [DOI] [PubMed] [Google Scholar]
- Curtis D, Vine AE, Knight J. Study of regions of extended homozygosity provides a powerful method to explore haplotype structure of human populations. Ann Hum Genet. 2008;72(Pt 2):261–278. doi: 10.1111/j.1469-1809.2007.00411.x. [DOI] [PMC free article] [PubMed] [Google Scholar]
- Nothnagel M, Lu TT, Kayser M, Krawczak M. Genomic and geographic distribution of SNP-defined runs of homozygosity in Europeans. Human molecular genetics. 2010;19(15):2927–2935. doi: 10.1093/hmg/ddq198. [DOI] [PubMed] [Google Scholar]
- Lee JA, Carvalho CMB, Lupski JR. A DNA replication mechanism for generating nonrecurrent rearrangements associated with genomic disorders. Cell. 2007;131(7):1235–1247. doi: 10.1016/j.cell.2007.11.037. [DOI] [PubMed] [Google Scholar]
- Simon-Sanchez J, Scholz S, Fung HC, Matarin M, Hernandez D, Gibbs JR, et al. Genome-wide SNP assay reveals structural genomic variation, extended homozygosity and cell-line induced alterations in normal individuals. Hum Mol Genet. 2007;16(1):1–14. doi: 10.1093/hmg/ddl436. [DOI] [PubMed] [Google Scholar]
- Kirin M, McQuillan R, Franklin CS, Campbell H, McKeigue PM, Wilson JF. Genomic runs of homozygosity record population history and consanguinity. PLoS One. 2010;5(11):e13996. doi: 10.1371/journal.pone.0013996. [DOI] [PMC free article] [PubMed] [Google Scholar]
- Henn BM, Botigue LR, Gravel S, Wang W, Brisbin A, Byrnes JK, et al. Genomic ancestry of North Africans supports back-to-Africa migrations. PLoS Genet. 2012;8(1):e1002397. doi: 10.1371/journal.pgen.1002397. [DOI] [PMC free article] [PubMed] [Google Scholar]
- Ben Halim N, Nagara M, Regnault B, Hsouna S, Lasram K, Kefi R, et al. Estimation of Recent and Ancient Inbreeding in a Small Endogamous Tunisian Community Through Genomic Runs of Homozygosity. Ann Hum Genet. 2015;79(6):402–417. doi: 10.1111/ahg.12131. [DOI] [PubMed] [Google Scholar]
- Lucas-Sánchez M, Font-Porterias N, Calafell F, Fadhlaoui-Zid K, Comas D. Whole-exome analysis in Tunisian Imazighen and Arabs shows the impact of demography in functional variation. Scientific Reports. 2021;11(1):21125. doi: 10.1038/s41598-021-00576-0. [DOI] [PMC free article] [PubMed] [Google Scholar]
- Khoury MJ, Cohen BH, Chase GA, Diamond EL. An epidemiologic approach to the evaluation of the effect of inbreeding on prereproductive mortality. American Journal of Epidemiology. 1987;125(2):251–262. doi: 10.1093/oxfordjournals.aje.a114524. [DOI] [PubMed] [Google Scholar]
- Roy-Gagnon M-H, Moreau C, Bherer C, St-Onge P, Sinnett D, Laprise C. Genomic and genealogical investigation of the French Canadian founder population structure. Human Genetics. 2011;129(5):521–531. doi: 10.1007/s00439-010-0945-x. [DOI] [PubMed] [Google Scholar]
- Leutenegger A-L, Prum B, Génin E, Verny C, Lemainque A, Clerget-Darpoux F. Estimation of the inbreeding coefficient through use of genomic data. The American Journal of Human Genetics. 2003;73(3):516–523. doi: 10.1086/378207. [DOI] [PMC free article] [PubMed] [Google Scholar]
- Bellis MA, Hughes K, Hughes S, Ashton JR. Measuring paternal discrepancy and its public health consequences. J Epidemiol Community Health. 2005;59(9):749–754. doi: 10.1136/jech.2005.036517. [DOI] [PMC free article] [PubMed] [Google Scholar]
- Lander ES, Botstein D. Homozygosity mapping: a way to map human recessive traits with the DNA of inbred children. Science. 1987;236(4808):1567–1570. doi: 10.1126/science.2884728. [DOI] [PubMed] [Google Scholar]
- Boubaker C, Hsairi-Guidara I, Castro C, Ayadi I, Boyer A, Kerkeni E. A novel mutation in FGD4/FRABIN causes Charcot Marie Tooth disease type 4H in patients from a consanguineous Tunisian family. Ann Hum Genet. 2013;77(4):336–343. doi: 10.1111/ahg.12017. [DOI] [PubMed] [Google Scholar]
- H'Mida-Ben Brahim D, M'Zahem A, Assoum M, Bouhlal Y, Fattori F, Anheim M. Molecular diagnosis of known recessive ataxias by homozygosity mapping with SNP arrays. J Neurol. 2011;258(1):56–67. doi: 10.1007/s00415-010-5682-5. [DOI] [PubMed] [Google Scholar]
- Ben Khelifa M, Coutton C, Zouari R, Karaouzene T, Rendu J, Bidart M. Mutations in DNAH1, which encodes an inner arm heavy chain dynein, lead to male infertility from multiple morphological abnormalities of the sperm flagella. Am J Hum Genet. 2014;94(1):95–104. doi: 10.1016/j.ajhg.2013.11.017. [DOI] [PMC free article] [PubMed] [Google Scholar]
- Mezzavilla M, Vozzi D, Badii R, Alkowari MK, Abdulhadi K, Girotto G. Increased rate of deleterious variants in long runs of homozygosity of an inbred population from Qatar. Human Heredity. 2015;79(1):14–19. doi: 10.1159/000371387. [DOI] [PubMed] [Google Scholar]
- A BMR. 1985. Situation démographique en Tunisie à la fin de l’année 1985. p. 66. [Google Scholar]
- Riou S. Consanguinite dans lapopulation du nord de la Tunisie. Tun Med. 1989;67:167–172. [PubMed] [Google Scholar]
- Ellouze I. PhD thesis, Université de Tunis El Manar, Tunisia. 2004. Etude de l’endogamie et de la consanguinité dans la région de Sfax; Causes socio-économiques et culturelles et conséquences sur la circulation des genes dans la population. [Google Scholar]
- M’rad LB, Chalbi N. Le choix matrimonial en Tunisie est-il transmissible? Antropo. 2004;7:31–37. [Google Scholar]
- Kerkeni E, Monastiri K, Saket B, Guediche MN, Ben Cheikh H. Interplay of socio-economic factors, consanguinity, fertility, and offspring mortality in Monastir, Tunisia. Croatian Medical Journal. 2007;48(5):701–707. [PMC free article] [PubMed] [Google Scholar]


