RESUMEN
La inmunodeficiencia combinada grave (IDCG) es la forma más grave de inmunodeficiencia primaria, que afecta sobre todo a los linfocitos T, y puede ser detectada al nacer mediante la cuantificación de los círculos de escisión del receptor de linfocitos T (TREC) en una muestra de sangre impregnada en papel (DBS). La detección precoz de esta enfermedad permite establecer de forma temprana un tratamiento adecuado en el paciente, permitiendo así su curación. El cribado neonatal de IDCG comenzó en Cataluña en enero de 2017, siendo la primera región española y europea en incluirla oficial y universalmente en su programa. En el presente trabajo se presentan los resultados obtenidos durante los tres primeros años y medio de experiencia (enero 2017 - junio 2020) empleando el kit EnLite Neonatal TREC (Perkin Elmer), con un cutoff de detección de TREC de 20 copias/µL. De 222.857 recién nacidos analizados, cuarenta y ocho fueron detecciones positivas: tres casos de IDCG (incidencia de 1:74.285); diecisiete casos de linfopenia T no IDCG (incidencia de 1:13.109); veintidós casos falsos positivos (recuento de linfocitos inicialmente normal, con normalización de TREC entre los tres y seis meses de vida); un caso con linfopenia transitoria (con un recuento de linfocitos inicialmente bajo, que se normaliza en los meses siguientes); y cinco pacientes se encuentran todavía en estudio.
Los resultados obtenidos aportan evidencias de los beneficios que supone incluir esta enfermedad en los programas de cribado neonatal. Podría ser necesario un seguimiento todavía más prolongado para acabar de definir la incidencia exacta de IDCG en Cataluña.
Palabras clave: Cribado neonatal, Inmunodeficiencia combinada grave, TREC, IDCG, Deficiencia PNP, Programa de Cribado Neonatal de Cataluña
ABSTRACT
Severe combined immunodeficiency (SCID), the most severe form of T-cell immunodeficiency, can be screened at birth by quantifying T-cell receptor excision circles (TREC) in dried blood spot (DBS) samples. Early detection of this condition speeds up the establishment of appropriate treatment and increases the patient’s life expectancy. Newborn screening for SCID started in January 2017 in Catalonia, the first Spanish and European region to universally include this testing. The results obtained in the first three years and a half of experience (January 2017 - June 2020) are shown here, using EnLite Neonatal TREC kit (Perkin Elmer) with 20 copies/µL as TREC detection cutoff. Of 222,857 newborns screened, 48 tested positive: three patients were diagnosed with SCID (incidence 1:74,285); 17 patients had clinically significant T-cell lymphopenia (non-SCID) with an incidence of 1 in 13,109 newborns; twenty two patients were considered false-positive cases because of an initially normal lymphocyte count with normalization of TREC between 3 and 6 months of life; one case had transient lymphopenia due to an initially low lymphocyte count with recovery in the following months; and five patients are still under study.
The results obtained provide further evidence of the benefits of including this disease in newborn screening programs. Even longer follow-up could be necessary to define the exact incidence of SCID in Catalonia.
Key words: Newborn screening, Severe combined immunodeficiency, TREC, SCID, PNP deficiency, Catalonia Newborn Screening Program
INTRODUCCIÓN
El cribado neonatal de la inmunodeficiencia combinada grave (IDCG) se implementó inicialmente en Wisconsin (Estados Unidos de América) en 2008. Actualmente, ya se realiza en todos los estados de ese país, así como en Taiwán, Israel, Nueva Zelanda, Noruega, y diferentes provincias de Canadá, Suiza, Alemania, Irlanda, Suecia, Italia y Australia 1 . En Cataluña, el cribado neonatal de IDCG se implantó el 1 de enero de 2017, convirtiéndose en la primera comunidad autónoma (CCAA) de España y en la primera región de Europa en incluirla de forma oficial y universal en su programa de cribado. El beneficio coste-efectivo de incluir la IDCG en los programas de cribado se ha demostrado tanto en EEUU 2 como en Europa 3 y, actualmente, se encuentra en evaluación a nivel nacional para decidir su inclusión.
La IDCG es la forma más grave de inmunodeficiencia primaria (afecta a los linfocitos T y B) y se caracteriza en la mayoría de las ocasiones por una linfopenia grave de linfocitos T. Su incidencia en nuestra población es de aproximadamente 1:60.000 recién nacidos, con importantes variaciones geográficas 4 , 5 . Estos pacientes requieren una intervención clínica inmediata para prevenir infecciones graves y daño orgánico, así como para poder instaurar precozmente el tratamiento curativo, mejorando así su supervivencia (92%) 6 . El tratamiento consiste en el trasplante de progenitores hematopoyéticos (TPH) 7 o, en casos seleccionados, el tratamiento enzimático sustitutivo o la terapia génica 1 .
El cribado neonatal de la IDCG se realiza principalmente mediante la cuantificación de los círculos de escisión del gen del receptor de linfocitos T (del inglés, T-cell Receptor Excision Circles, TREC). Los TREC son moléculas de ADN circulares estables formadas por el reordenamiento del gen del receptor del linfocito T durante su diferenciación en el timo, siendo un marcador de linfocitos T de reciente formación (esta población celular está extremadamente disminuida en la mayoría de los recién nacidos con una IDCG). El número de copias de TREC se determina usando métodos cuantitativos basados en la amplificación de ácidos nucleicos (PCR) 8 . La determinación de KREC (del inglés, K-deleting Recombination Excision Circles) en el cribado neonatal podría permitir detectar algunas otras entidades con defectos de linfocitos B (como la agammaglobulinemia ligada al cromosoma X), aunque, en la actualidad, esta determinación no se incluye en la mayoría de programas de cribado universal de la IDCG 1 .
La cuantificación de TREC a través del cribado neonatal también permite identificar otras formas clínicamente relevantes de linfopenia T, como el síndrome de microdeleción 22q11 (DiGeorge) 9 , el síndrome de Down o el síndrome CHARGE, entre otros 10 , 11 .
En Cataluña, el programa de cribado neonatal comenzó con la detección de la fenilcetonuria en 1969 y el hipotiroidismo congénito en 1982. Actualmente, aproximadamente 65.000 recién nacidos al año son cribados para la detección de veinticuatro enfermedades (fenilcetonuria, hipotiroidismo congénito, fibrosis quística, enfermedad de células falciformes, aminoacidopatías, acidurias orgánicas y defectos de la beta oxidación mitocondrial), siendo la IDCG la última en incluirse 12 . En el presente trabajo se presentan los resultados obtenidos en los tres primeros años y medio de experiencia.
MATERIAL Y MÉTODOS
Se incluyeron en el estudio todas las muestras de sangre impregnada en papel (DBS) (n=222.857) recibidas en estos tres años y medio en el laboratorio del Programa de Cribado Neonatal (PCN) de Cataluña. Se excluyeron del estudio las muestras con las siguientes características: toma de la muestra antes de las 44 horas o después de los siete días de vida; transfusiones; pobre amplificación del ADN; y/o mala calidad/escasa cantidad de muestra. Una vez descartadas estas muestras, el estudio se realizó en 220.706 recién nacidos (RN).
Desde el 1 de enero de 2017 al 30 de junio de 2017, se llevó a cabo el estudio piloto prospectivo con 33.040 RN para la validación e implementación de nuestra estrategia, obteniendo resultados comparables a los descritos por el fabricante.
En enero de 2018 se actualizó el algoritmo de decisión, disminuyendo el cutoff de repetición (análisis por duplicado) de 34 a 24 copias/µL de TREC. En enero de 2019 incorporamos un criterio para repetir únicamente por simple los resultados entre 21 y 24 copias/µL (manteniendo la repetición por duplicado de los resultados menores o iguales a 20 copias/µL). Así pues, el algoritmo que utilizamos actualmente se resume en la figura 1.
Figura 1. Algoritmo de decisión para la detección de IDCG a través del cribado neonatal.
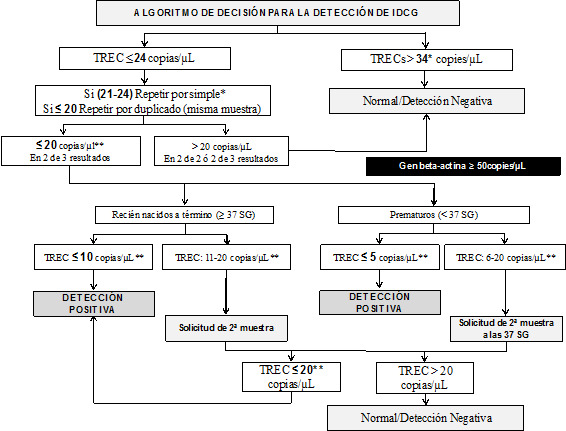
La realización del estudio para la inclusión de la IDCG en el PCN de Cataluña fue aprobado por el Departament de Salut de la Generalitat de Catalunya. El consentimiento informado se solicitó a los padres o tutores legales de los pacientes incluidos en el estudio, a los que se les tuvo que realizar un análisis genético.
La cuantificación de TREC en DBS (discos de 1,5 mm de diámetro) se realizó con el kit EnLite Neonatal TREC (Perkin Elmer, Turku, Finlandia). Esta metodología consiste en una amplificación de ácidos nucleicos mediante PCR con una lectura fotométrica posterior (fluorescencia a tiempo resuelto y transferencias de energía de resonancia -del inglés TR-FRET-). Se realizó la detección de forma simultánea de TREC y del gen beta actina (empleado como control interno de la reacción en cada muestra). Empleando placas de PCR de 96 pocillos, el proceso incluyó una primera parte de elución del ADN de la muestra y una segunda de amplificación de TREC y beta actina, mediante PCR e hibridación con sondas específicas. Posteriormente, la placa fue leída en el fluorómetro Victor EnLite (Perkin Elmer), obteniendo resultados de ambos en concentración (copias/µL).
Las detecciones positivas fueron notificadas a la Unidad Clínica de Referencia Diagnóstica (UCRD) para iniciar la evaluación clínica e inmunológica.
Se calculó la tasa de repetición en la primera muestra, la tasa de solicitud de segundas muestras y la tasa de detección positiva. Para la validación de los resultados se emplearon los programas informáticos Specimen Gate (Perkin Elmer) y Nadons (Limit4, Barcelona, España).
Todos los casos fueron visitados en la consulta antes de los siete días tras la comunicación a la UCRD, donde se siguió el protocolo mostrado en la figura 2 para la evaluación clínica e inmunológica. Se recogieron antecedentes médicos y familiares y se realizó un meticuloso examen físico. Además, se ofreció a las familias soporte psicológico, comenzando desde la primera visita.
Figura 2. Protocolo inmunológico y genético en los casos positivos de IDCG.
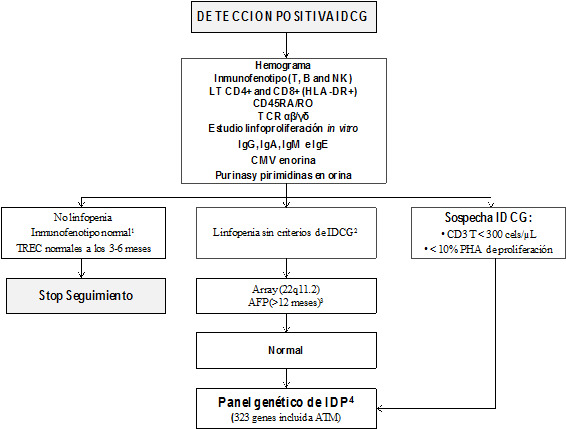
Para el análisis estadístico se empleó el programa informático SPSS 23.0 (SPSS Inc., Chicago, Illinois).
RESULTADOS
Demográficos
De los 220.706 RN cribados, el 51,5% fueron de sexo masculino y el 48,5% femenino. La mediana de tiempo de toma de muestra fue de 50 horas desde el nacimiento (rango intercuartílico (IQR), 49-60). La mediana de edad gestacional fue de 39 semanas (38-40): 0,6% de prematuros extremos (n=1.324); 5,9% de prematuros (n=13.021); y 93,5% RN a término (n=206.361). La mediana del peso al nacer fue de 3.300 g (3.010-3.590) (tabla 1).
Tabla 1. Datos demográficos y valores de TREC en la población estudiada.
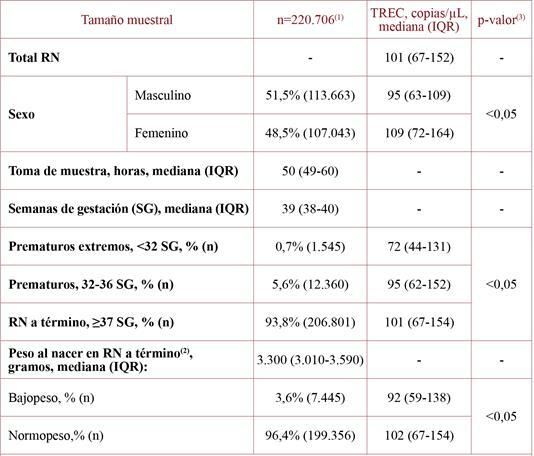
(1) Tamaño muestral después de aplicar los criterios de exclusión; (2) Recién nacidos a término (n=206.801); (3) Los test U de Mann-Whitney o Kruskal-Wallis se usaron para la comparación entre grupos (p-valor calculado con un 5% de nivel de significancia); Abreviaturas: IQR, rango intercuartílico; RN: recién nacidos; SG: semanas de gestación.
La mediana de TREC en la población estudiada fue de 101 copias/µL (IQR: 67-154), siendo 20 copias/µL (cutoff de detección) el percentil 0,6. En la tabla 1 se muestran los diferentes valores de TREC para los subgrupos mencionados.
Valores de cutoff y tasas
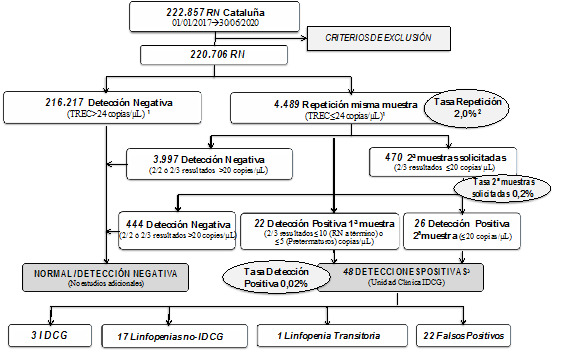
Los resultados globales mostraron una tasa de repetición del 2%, una tasa de solicitud de segundas muestras del 0,2% y una tasa de detección positiva del 0,02% (figura 3). Con la aplicación del cutoff de repetición inicial de 34 copias/µL (percentil 3) en el primer algoritmo empleado (enero 2017-diciembre 2017), se obtuvo una tasa de repetición del 3,34%, con tasas de solicitud de segunda muestra y de detección positiva equivalentes a las globales. Después de reducir el cutoff de detección a 24 copias/µL (percentil 1), se obtuvo una tasa de repetición del 1,4% en 2018, 1,5% en 2019 y 1,6% en el primer semestre de 2020. El cutoff de detección se mantuvo en 20 copias/µL (percentil 0,6), obteniendo tasas de solicitud de segunda muestra y de detección positiva similares todos los años.
Figura 3. Resultados del Programa de Cribado Neonatal de IDCG en Cataluña durante el periodo de estudio (enero 2017 - junio 2020).
Detecciones positivas
De las casi 221.000 muestras, se obtuvieron cuarenta y ocho detecciones positivas (TREC menor o igual a 20 copias/µL), veintidós fueron en primera muestra (45,8%) y veintiséis en segunda muestra (54,2%). En ninguno de los casos hubo historia de tratamiento con inmunosupresión materna.
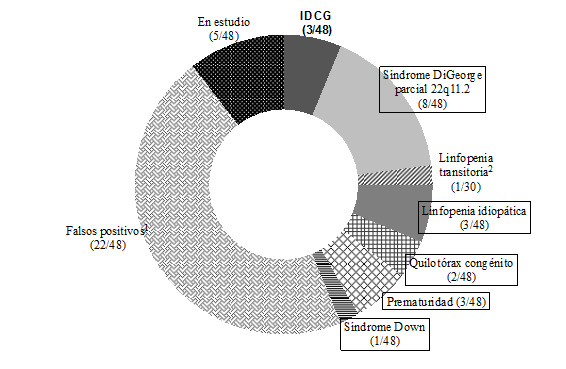
Los RN con detección positiva fueron derivados a la UCRD de IDCG según el protocolo establecido. Los diferentes diagnósticos de las cuarenta y ocho detecciones positivas se muestran en la figura 4 y fueron los siguientes: IDCG (tres), síndrome de DiGeorge parcial 22q11 (ocho), linfopenia idiopática (tres), quilotórax congénito (dos), prematuridad (tres) y síndrome de Down (uno). Veintidós pacientes fueron falsos positivos (TREC inicialmente bajos y recuento normal de linfocitos, con normalización de TREC entre los tres y seis meses de vida), un paciente tuvo linfopenia transitoria (TREC y linfocitos inicialmente bajos, con recuperación en los siguientes meses). Cinco pacientes siguen actualmente en estudio, con diagnóstico de IDCG excluido, pero con necesidad de nuevos análisis de TREC a los tres y seis meses de vida. La incidencia de linfopenia T clínicamente significativa no-IDCG fue de 1:13.109 recién nacidos (35% de las detecciones positivas).
Figura 4. Diagnóstico definitivo de los pacientes con detección positiva de IDCG en Cataluña (enero 2017 - junio 2020).
La incidencia de IDCG en Cataluña fue de 1:74.285 recién nacidos. Los tres recién nacidos detectados y diagnosticados de IDCG presentaron las siguientes características:
El primer paciente detectado de IDCG fue un varón caucásico de treinta y cinco semanas de gestación con cero copias de TREC/µL en el cribado neonatal. El recuento de linfocitos fue de 0,4 x 109/L, con un inmunofenotipo T-B-NK+ y una respuesta proliferativa a mitógenos ausente. Se realizó la secuenciación completa del exoma, pero no se encontraron mutaciones causales. El paciente se sometió a un trasplante de progenitores hematopoyéticos (TPH) a los dos meses de vida (utilizando un acondicionamiento de intensidad reducida), con una buena evolución clínica y una reconstitución inmunológica completa.
El segundo paciente detectado de IDCG fue un varón de treinta y ocho semanas de gestación con cero copias de TREC/µL en el cribado neonatal y padres consanguíneos marroquís. Tenía antecedentes de hermana fallecida en Marruecos, presentando sintomatología descrita compatible con IDCG tipo Omenn. El recuento de linfocitos fue de 0,4 x 109/L con un inmunofenotipo T-B-NK+ y una respuesta proliferativa a mitógenos ausente. Se realizó un panel de secuenciación masiva (NGS) que incluía 323 genes causantes de IDP, y se encontró una mutación patogénica en homocigosis en el gen RAG2 (c.1338C>G / p.Cys446Trp). Ambos padres eran portadores de la mutación en heterozigosis. El paciente se sometió a un TPH a los dos meses de vida (utilizando un acondicionamiento de intensidad reducida), con una buena evolución clínica y una reconstitución inmunológica completa.
El tercer paciente detectado de IDCG se diagnosticó como deficiencia de purina nucleósido fosforilasa (PNP). Fue un varón de cuarenta semanas de gestación con cuatro copias de TREC/µL en el cribado neonatal y padres consanguíneos marroquís. El recuento de leucocitos fue de 4,02 x 109/L con presencia de linfopenia (0,9 x 109/L), un inmunofenotipo T-B+NK+ y una respuesta proliferativa a mitógenos ligeramente disminuida (45% del control). En el análisis genético (mediante el mismo panel NGS de 323 genes anteriormente descrito) se encontró una mutación patogénica en homocigosis en el gen PNP (c.602A>G/p.Glu201Gly), siendo ambos padres heterocigotos para dicha mutación. El paciente se sometió a los dos meses de vida a un TPH haploidentico de su padre (acondicionamiento de intensidad reducida), con una buena evolución clínica y una reconstitución inmunológica completa hasta el momento y sin señales de daño neurológico. Los estudios bioquímicos mostraron un aumento de la concentración de guanosina, inosina, desoxiguanosina y desoxinosina en orina, y una disminución de la concentración de ácido úrico en plasma, así como una actividad enzimática de PNP disminuida en DBS.
DISCUSIÓN
En el presente trabajo exponemos nuestra experiencia en el programa de cribado neonatal de IDCG en Cataluña mediante la cuantificación de TREC, la cual nos ha permitido detectar pacientes asintomáticos afectos de una IDCG, así como condiciones de linfopenia no IDCG. Después de tres años y medio de experiencia y más de 223.000 RN cribados se detectaron tres pacientes con IDCG y diecisiete con otras condiciones. Además, consolidamos nuestra estrategia de detección, así como el protocolo de caracterización, diagnóstico y seguimiento de los casos positivos.
Como iniciativa pionera en Europa, fomentamos la definición de un único enfoque técnico para el cribado neonatal de IDCG, de cara a facilitar futuros estudios comparativos entre diferentes países. En esta línea, decidimos implementar el kit EnLite Neonatal TREC (Perkin Elmer, Turku, Finlandia), disponible comercialmente, que también ha sido validado por otros grupos, mostrando valores de mediana de TREC comparables con los publicados 13 , 14 , 15 , 16 . Este kit permite la elución de ADN y la amplificación de la secuencia de interés en un solo proceso, así como una estandarización y reproducibilidad en cumplimiento con la norma ISO 15189. Con el objetivo de minimizar la variabilidad entre lotes ya descrita anteriormente 14 , decidimos establecer un cutoff de repetición (por duplicado) que fuera superior al cutoff de detección (solicitud de segunda muestra), para disponer de un intervalo o zona gris entre ambos. El cutoff de repetición inicial de 34 copias/μL (percentil 3) se decide cambiar a 24 copias/μL (percentil 1) en 2018, tras análisis y valoración de los datos acumulados; mientras que el cutoff de solicitud de segunda muestra se mantiene en 20 copias/μL. Con este cambio se consigue una reducción en la tasa de repetición del 3,3% (considerada demasiado alta en comparación con lo publicado por otros autores 17 ) al 1,4% (ahora sí, comparable), obteniendo también una reducción en el coste por paciente analizado.
Con nuestro algoritmo de detección se obtiene una tasa de solicitud de segunda muestra del 0,2% y una tasa de detección positiva del 0,02%, similares a las tasas publicadas y resumidas en la revisión de van der Spek et al 17 . Además, los percentiles de nuestra población correspondientes a los cutoffs empleados son comparables a los correspondientes a otras enfermedades incluidas en nuestro programa, tales como hipotiroidismo congénito y fenilcetonuria entre otras.
Más tarde, en 2019 se introduce la repetición por simple en los casos en los que los TREC estuvieran en el intervalo entre 21 y 24 copias/µL. A pesar de esta mejora, tenemos la intención de reevaluar los datos cuando alcancemos una experiencia de cinco años, ya que consideramos que es posible una disminución mayor tanto del cutoff de repetición como del de detección, optimizando aun más nuestro algoritmo y sin riesgo de perder diagnósticos de IDCG.
Desde el punto de vista metodológico, cabe destacar que mediante nuestro método no se pudo cuantificar las copias de beta-actina en algunas de las muestras de control externo de la calidad del programa Proficiency Testing del CDC. Este problema pudo estar relacionado con algún tipo de inhibición de PCR con esta metodología, tal y como nos sugirió el propio CDC. No obstante, esta limitación no afecta los resultados de muestras de pacientes para la detección de IDCG.
Existen varios estudios que han utilizado la cuantificación de TREC y KREC de forma combinada en cribado para detectar simultáneamente los defectos de linfocitos T y B 18 , 19 , incluido un estudio pionero realizado en España por De Felipe et al 20 . Sin embargo, nosotros decidimos centrarnos en los pacientes con IDCG clásica que cumplían completamente con los criterios de cribado neonatal, acorde a la experiencia en Estados Unidos 6 , 8 . Aunque la inclusión de la cuantificación de KREC junto con los TREC permitiría la detección de pacientes con mutaciones hipomórficas de fenotipo leaky IDCG y déficit de adenosina desaminasa de aparición tardía (ADA) 19 , 21 , 22 , también podría resultar en un aumento de detecciones de falsos positivos, los cuales se traducirían en mayores tasas de repetición, solicitud de segundas muestras y detección. Por otro lado, cabe destacar que en el PCN de Cataluña, desde noviembre de 2019, se incluye en el estudio del cribado ampliado por espectrometría de masas en tándem la cuantificación de la concentración de adenosina y desoxiadenosina en DBS (kit NeoBase 2 Non-derivatized MSMS, Perkin Elmer), de manera que los casos de déficit de ADA que no son detectados por el método de TREC serían detectados con esta metodología.
En total, hay cuarenta y ocho detecciones positivas en los 220.706 recién nacidos cribados (222.857 antes de aplicar los criterios de exclusión). Todos son remitidos a la UCRD de IDCG según protocolo: veintidós pacientes resultan falsos positivos, debido a los recuentos de linfocitos totales, inicialmente normales, con normalización de TREC entre los tres y seis meses de vida, mientras que uno muestra linfopenia transitoria al principio, con recuperación en los meses siguientes, y cinco se encuentran actualmente en estudio (aunque ninguno de los cinco pacientes en estudio cumple con los criterios de IDCG definidos según Kwan et al 6 ).
El diagnóstico de los tres casos positivos permitió la derivación oportuna y precoz a un centro de tratamiento especializado para TPH, antes de la aparición de cualquier manifestación clínica que hubiese podido influir en la supervivencia del paciente y en la evolución del TPH. Los pacientes evolucionaron muy favorablemente, logrando una reconstitución de la función inmunológica completa. Cabe mencionar que, en el tercer caso positivo detectado, diagnosticado con deficiencia de PNP, se encuentró una mutación nueva en el gen PNP, aún no descrita en la literatura como causante de la enfermedad (datos aún no publicados).
La incidencia de IDCG encontrada en nuestro estudio (1:74.285) es similar a la descrita en otros países de Europa Occidental y EEUU 6 , 7 , 17 , siendo superior a la publicada previamente por nuestro grupo en 2019 (1:130.903) ya que, como anticipamos, después de alcanzar los 200.000 RN cribados la incidencia se consolida. Además, creemos que la incidencia irá aumentando en los próximos años, basándonos en un estudio retrospectivo realizado en nuestra CCAA previamente al inicio del cribado, donde la incidencia obtenida de casos de IDCG diagnosticados clínicamente fue de 1:57.000 RN (datos no publicados).
Hemos de añadir que la detección temprana de pacientes con síndrome de deleción 22q11.2 permite una intervención rápida para anticipar complicaciones, según lo publicado por nuestro grupo 9 .
Cabe destacar que uno de los puntos fuertes de nuestro enfoque en el cribado de esta enfermedad es el apoyo psicológico ofrecido a las familias, ya que un resultado positivo en cualquier programa de cribado es motivo de preocupación para los padres. El apoyo psicológico se intensifica en los momentos más complicados, como al ingreso en el hospital, y los padres son acompañados en todas las fases relacionadas con el trasplante.
Como conclusiones, decir que la cuantificación de TREC en el cribado neonatal para la detección de IDCG se implementó satisfactoriamente en el programa de Cataluña 12 . Después de una experiencia de tres años y medio, y más de 222.000 recién nacidos cribados, se identificaron tres pacientes con IDCG y otros diecisiete pacientes con otras causas de linfopenia que también se beneficiaron del PCN. Además, consolidamos nuestra estrategia de detección, así como el protocolo de caracterización, diagnóstico y seguimiento de casos positivos. Las tasas de repetición, solicitud de segundas muestras y de detección positivas son similares a los datos publicados. Nuestros resultados proporcionan más evidencia para apoyar la inclusión de la IDCG en los programas de cribado en el resto de las comunidades autónomas, así como en otras regiones y países. Es posible que siga siendo necesario un seguimiento más prolongado para acabar de definir la incidencia exacta de IDCG en Cataluña.
AGRADECIMIENTOS
A la Dra. Marie Audrain (CHU de Nantes, Francia), por su contribución al desarrollo del algoritmo. Al Sr. Antoni Comín (exconsejero de Salud de la Generalitat de Cataluña), por su implicación personal con la implantación del Cribado Neonatal de la IDCG en Cataluña. A la Dra. Rosa Mª Fernández (exdirectora del Departamento de Salud Materno-Infantil), por su apoyo y profesionalidad con el PCN de Cataluña. A la Sra. Anna Fabregas, por dar soporte psicológico a todos los padres de los pacientes con detección positiva de IDCG. A la Sra. Marta Parellada y a la Sra. Yania Quintero, por su enorme labor administrativa y técnica, respectivamente, durante la implantación de la enfermedad en el PCN de Cataluña. A la Asociación Catalana de Inmunodeficiencias Primarias, la Fundación BCN-PID y al grupo PID de las Sociedades Catalanas de Pediatría e Inmunología, por su gran ayuda para la implementación del cribado neonatal de la IDCG en Cataluña. Agradecemos también la implicación y labor de todo el personal técnico del laboratorio de cribado neonatal del Hospital Clínic de Barcelona y del departamento de inmunología del Hospital Vall d’Hebron.
Cita sugerida: Argudo Ramírez A, Martín Nalda A, Marín Soria JL, López Galera RM, José Manuel González de Aledo Castillo JM, Pajares García S, Rivière JG, Martínez Gallo M, Colobran R, Parra Martínez A, Ribes Rubio A, Fernández Bardon RM, Asso Ministral L, Prats Viedma B, García Villoria J, Soler Palacín P. Primer programa europeo de cribado neonatal para la inmunodeficiencia combinada grave: experiencia de tres años en Cataluña. Rev Esp Salud Pública. 2020; 94: 16 de diciembre e202012153.
BIBLIOGRAFÍA
- 1.. van der Burg M, Mahlaoui N, Gaspar HB, Pai SY. Universal Newborn Screening for Severe Combined Immunodeficiency (SCID). Front Pediatr. 2019;7:373. [DOI] [PMC free article] [PubMed]; van der Burg M.Mahlaoui N.Gaspar HB.Pai SY Universal Newborn Screening for Severe Combined Immunodeficiency (SCID) Front Pediatr. 2019;7:373–373. doi: 10.3389/fped.2019.00373. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.. Modell V, Knaus M, Modell F. An analysis and decision tool to measure cost benefit of newborn screening for severe combined immunodeficiency (SCID) and related T-cell lymphopenia. Immunologic Research. 2014 Oct;60(1):145-152. [DOI] [PubMed]; Modell V, Knaus M, Modell F. An analysis and decision tool to measure cost benefit of newborn screening for severe combined immunodeficiency (SCID) and related T-cell lymphopenia. Immunologic Research. 2014 Oct;60(1):145–152. doi: 10.1007/s12026-014-8485-4. [DOI] [PubMed] [Google Scholar]
- 3.. Van der Ploeg C, Blom M, Bredius R, van der Burg M, Schielen P, Verkerk PH et al. Cost-effectiveness of newborn screening for severe combined immunodeficiency. European journal of pediatrics. 2019;178(5):721-729. [DOI] [PubMed]; Van der Ploeg C.Blom M.Bredius R.van der Burg M.Schielen P.Verkerk PH et al Cost-effectiveness of newborn screening for severe combined immunodeficiency. European journal of pediatrics. 2019;178(5):721–729. doi: 10.1007/s00431-019-03346-3. [DOI] [PubMed] [Google Scholar]
- 4.. Puck JM. Newborn screening for severe combined immunodeficiency and T-cell lymphopenia. Immunol Rev. 2019; 287(1):241-252. [DOI] [PMC free article] [PubMed]; Puck JM. Newborn screening for severe combined immunodeficiency and T-cell lymphopenia. Immunol Rev. 2019;287(1):241–252. doi: 10.1111/imr.12729. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.. Audrain M, Léger A, Hémont, Mirallié S, Cheillan D, Rimbert MGM et al. Newborn Screening for Severe Combined Immunodeficiency: Analytic and Clinical Performance of the T Cell Receptor Excision Circle Assay in France (DEPISTREC Study). J Clin Immunol. 2018; 38(7):778-786. [DOI] [PubMed]; Audrain M, Léger A, Hémont -, Mirallié S, Cheillan D. Rimbert MGM et al Newborn Screening for Severe Combined Immunodeficiency Analytic and Clinical Performance of the T Cell Receptor Excision Circle Assay in France (DEPISTREC Study) J Clin Immunol. 2018;38(7):778–786. doi: 10.1007/s10875-018-0550-7. [DOI] [PubMed] [Google Scholar]
- 6.. Kwan A, Abraham RS, Currier R, Brower A, Andruszewski K, Abbott JK et al. Newborn screening for severe combined immunodeficiency in 11 screening programs in the United States. JAMA. 2014; 312(7):729-38. [DOI] [PMC free article] [PubMed]; Kwan A, Abraham RS, Currier R, Brower A, Andruszewski K. Abbott JK et al Newborn screening for severe combined immunodeficiency in 11 screening programs in the United States. JAMA. 2014;312(7):729–738. doi: 10.1001/jama.2014.9132. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.. Buckley R, Schiff SE, Schiff RI, Markert ML, Williams LW, Roberts JL et al. Hematopoietic stem-cell transplantation for the treatment of severe combined immunodeficiency. N. Engl. J. Med. 1999;340:508-516. [DOI] [PubMed]; Buckley R, Schiff SE, Schiff RI, Markert ML, Williams LW. Roberts JL et al Hematopoietic stem-cell transplantation for the treatment of severe combined immunodeficiency N. Engl. J. Med. 1999;340:508–516. doi: 10.1056/NEJM199902183400703. [DOI] [PubMed] [Google Scholar]
- 8.. Chan K, Puck JM. (2005). "Development of a population-based screening for severe combined immunodeficiency". J. Allergy Clin. Immunol. 115: 391-398. [DOI] [PubMed]; Chan K, Puck JM. Development of a population-based screening for severe combined immunodeficiency. J. Allergy Clin. Immunol. 2005;(115):391–398. doi: 10.1016/j.jaci.2004.10.012. [DOI] [PubMed] [Google Scholar]
- 9.. Martin-Nalda A, Cueto-González AM, Argudo-Ramírez A, Marín-Soria JL, Martínez-Gallo M, Colobran R et al. Identification of 22q11.2 deletion syndrome via newborn screening for severe combined immunodeficiency. Two years' experience in Catalonia (Spain). Mol Genet Genomic Med. 2019;7(12):e1016. [DOI] [PMC free article] [PubMed]; Martin-Nalda A, Cueto-González AM, Argudo-Ramírez A, Marín-Soria JL, Martínez-Gallo M. Colobran R et al Identification of 22q11 2 deletion syndrome via newborn screening for severe combined immunodeficiency. Two years' experience in Catalonia (Spain) Mol Genet Genomic Med. 2019;7(12):e1016. doi: 10.1002/mgg3.1016. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.. King J, Hammarström L. Newborn Screening for Primary Immunodeficiency Diseases: History, Current and Future Practice. J Clin Immunol. 2018;38:56-66. [DOI] [PMC free article] [PubMed]; King J, Hammarström L. Newborn Screening for Primary Immunodeficiency Diseases History, Current and Future Practice. J Clin Immunol. 2018;38:56–66. doi: 10.1007/s10875-017-0455-x. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.. Routes J, Verbsky J. Newborn Screening for Severe Combined Immunodeficiency. Curr Allergy Asthma Rep. 2018; 18(6):34. [DOI] [PubMed]; Routes J, Verbsky J. Newborn Screening for Severe Combined Immunodeficiency. Curr Allergy Asthma Rep. 2018;18(6):34–34. doi: 10.1007/s11882-018-0783-9. [DOI] [PubMed] [Google Scholar]
- 12.. Argudo-Ramírez A, Martín-Nalda A, Marín-Soria JL, López-Galera RM, Pajares-García S, González de Aledo Castillo JM et al. "First universal newborn screening program for severe combined immunodeficiency in Europe. Two-years' experience in Catalonia (Spain)". Front Immunol. 2019;10:2406. [DOI] [PMC free article] [PubMed]; Argudo-Ramírez A, Martín-Nalda A, Marín-Soria JL, López-Galera RM, Pajares-García S. González de Aledo Castillo JM et al "First universal newborn screening program for severe combined immunodeficiency in Europe Two-years' experience in Catalonia (Spain)" Front. Immunol. 2019;10:2406–2406. doi: 10.3389/fimmu.2019.02406. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.. Rechavi E, Lev A, Simon AJ, Stauber T, Daas S, Saraf-Levy T et al. First Year of Israeli Newborn Screening for Severe Combined Immunodeficiency - Clinical Achievements and Insights. Front. Immunol. 2017;8:1448. [DOI] [PMC free article] [PubMed]; Rechavi E, Lev A, Simon AJ, Stauber T, Daas S. Saraf-Levy T et al First Year of Israeli Newborn Screening for Severe Combined Immunodeficiency - Clinical Achievements and Insights Front. Immunol. 2017;8:1448–1448. doi: 10.3389/fimmu.2017.01448. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.. Audrain M, Léger A, Hémont, Mirallié SM, Cheillan D, Rimbert MGM et al. Newborn Screening for Severe Combined Immunodeficiency: Analytic and Clinical Performance of the T Cell Receptor Excision Circle Assay in France (DEPISTREC Study). J Clin Immunol. 2018; 38(7):778-786. [DOI] [PubMed]; Audrain M, Léger A, Hémont -, Mirallié SM, Cheillan D. Rimbert MGM et al Newborn Screening for Severe Combined Immunodeficiency Analytic and Clinical Performance of the T Cell Receptor Excision Circle Assay in France (DEPISTREC Study) J Clin Immunol. 2018;38(7):778–786. doi: 10.1007/s10875-018-0550-7. [DOI] [PubMed] [Google Scholar]
- 15.. Blom M, Pico-Knijnenburg I, Sijne-van Veen M, Boelen A, Bredius RGM, van der Buerg M et al. An evaluation of the TREC assay with regard to the integration of SCID screening into the Dutch newborn screening program. Clin Immunol. 2017; 180:106-10. [DOI] [PubMed]; Blom M, Pico-Knijnenburg I, Sijne-van Veen M, Boelen A, Bredius RGM. van der Buerg M et al An evaluation of the TREC assay with regard to the integration of SCID screening into the Dutch newborn screening program. Clin Immunol. 2017;180:106–110. doi: 10.1016/j.clim.2017.05.007. [DOI] [PubMed] [Google Scholar]
- 16.. Adams SP, Rashid S, Premachandra T, Harvey K, Ifederu A,Wilson MC et al. Screening of neonatal UK dried blood spots using a duplex TREC screening assay. J Clin Immunol. 2014; 34:323-30. [DOI] [PubMed]; Adams SP, Rashid S, Premachandra T, Harvey K, Ifederu A. Wilson MC et al Screening of neonatal UK dried blood spots using a duplex TREC screening assay. J Clin Immunol. 2014;34:323–330. doi: 10.1007/s10875-014-0007-6. [DOI] [PubMed] [Google Scholar]
- 17.. Van der Spek J, Groenwold RHH, van der Burg M, van Montfrans JM. TREC based newborn screening for severe combined immunodeficiency disease: a systematic review. J Clin Immunol. 2015; 35:416-30. [DOI] [PMC free article] [PubMed]; Van der Spek J.Groenwold RHH.van der Burg M.van Montfrans JM TREC based newborn screening for severe combined immunodeficiency disease a systematic review. J Clin Immunol. 2015;35:416–430. doi: 10.1007/s10875-015-0152-6. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.. Barbaro M, Ohlsson A, Borte S, Jonsson S, Zetterström RH, King J et al. Newborn Screening for Severe Primary Immunodeficiency Diseases in Sweden - a 2-Years Pilot TREC and KREC Screening Study. J Clin Immunol. 2017; 37:51-60. [DOI] [PMC free article] [PubMed]; Barbaro M, Ohlsson A, Borte S, Jonsson S, Zetterström RH. King J et al Newborn Screening for Severe Primary Immunodeficiency Diseases in Sweden - a 2-Years Pilot TREC and KREC Screening Study. J Clin Immunol. 2017;37:51–60. doi: 10.1007/s10875-016-0347-5. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.. Nourizadeh M, Shakerian L, Borte S, Fazlollahi M, Badalzadeh M, Houshmand M et al. Newborn screening using TREC/KREC assay for severe T and B cell lymphopenia in Iran. Scand J Immunol. 2018; 88:e12699. [DOI] [PubMed]; Nourizadeh M, Shakerian L, Borte S, Fazlollahi M, Badalzadeh M. Houshmand M et al Newborn screening using TREC/KREC assay for severe T and B cell lymphopenia in Iran. Scand J Immunol. 2018;88:e12699. doi: 10.1111/sji.12699. [DOI] [PubMed] [Google Scholar]
- 20.. de Felipe B, Olbrich P, Lucenas JM, Delgado-Pecellin C, Pavon-Delgado A, Marquez J et al. Prospective neonatal screening for severe T and B-lymphocyte deficiencies in Seville. Pediatr Allergy Immunol. 2016;27:70-77. [DOI] [PubMed]; de Felipe B, Olbrich P, Lucenas JM, Delgado-Pecellin C, Pavon-Delgado A. Marquez J et al Prospective neonatal screening for severe T and B-lymphocyte deficiencies in Seville. Pediatr Allergy Immunol. 2016;27:70–77. doi: 10.1111/pai.12501. [DOI] [PubMed] [Google Scholar]
- 21.. Borte S, Wang N, Oskarsdóttir S, von Döbeln U, Hammarström L. Newborn screening for primary immunodeficiencies: beyond SCID and XLA. Ann N Y Acad Sci. 2011; 1246:118-30. [DOI] [PubMed]; Borte S, Wang N, Oskarsdóttir S, von Döbeln U, Hammarström L. Newborn screening for primary immunodeficiencies beyond SCID and XLA. Ann N Y Acad Sci. 2011;1246:118–130. doi: 10.1111/j.1749-6632.2011.06350.x. [DOI] [PubMed] [Google Scholar]
- 22.. Borte S, Puck J, Lorey F, McGhee SA, Hershfield MS, Fasth A et al. KRECs but not TRECs identify patients with delayed-onset adenosine deaminase (ADA) deficiency in neonatal screening. J Clin Immunol. 2013; 33:673.; Borte S, Puck J, Lorey F, McGhee SA, Hershfield MS. Fasth A et al KRECs but not TRECs identify patients with delayed-onset adenosine deaminase (ADA) deficiency in neonatal screening. J Clin Immunol. 2013;33:673–673. [Google Scholar]


