Abstract
目的
探讨同源异形盒基因(HOXs)在脑膜瘤中的甲基化模式,筛选对脑膜瘤复发风险分层有临床指导意义的HOXs基因,建立预测模型并评估其预测效能。
方法
利用GEO数据库下载脑膜瘤相关数据集。通过甲基化差异分析和ROC曲线分析筛选有预后评估价值的HOXs基因,再通过Cox回归分析、分子特征分析对特征基因的临床应用价值进行验证。进一步筛选差异CpG位点并评估其预测效能,通过Lasso-cox回归分析建立预测模型,根据cut off值将患者分成高、低风险组并进行分析。最后通过甲基化特异性PCR(MS-PCR)在细胞和组织水平验证差异CpG位点的甲基化水平,并纳入脑膜瘤组织样本验证该模型的预测效能。
结果
HOXA9甲基化水平在脑膜瘤中显著上调(P<0.001),且具有较高诊断效能(AUC=0.884)。验证分析表明HOXA9甲基化是影响脑膜瘤患者总生存期的独立危险因素(P<0.01),与脑膜瘤恶性程度和不良预后正相关(P<0.05),且基于HOXA9甲基化水平的分组方法在预测患者复发和生存时间时比WHO分级精度更高。筛选出的CpG位点cg03217995和cg21001184对脑膜瘤诊断的AUC均大于0.8,预测脑膜瘤患者复发的AUC均大于0.6。构建的两位点联合预测模型cut off值为1.226,以此分组的患者临床特征均有显著性差异(P<0.001),并且该模型预测评分是脑膜瘤的独立预后因素(P<0.05)。MS-PCR结果显示,位点cg03217995和cg21001184甲基化水平在脑膜瘤细胞中升高(P<0.0001),在不同WHO分级患者间无统计学差异。临床样本分析表明联合模型有较高预测效能(AUC=0.857),预测状态与患者真实临床进展结果高度一致。
结论
HOXA9甲基化是脑膜瘤预后不良的有效预测指标,基于其CpG位点的联合预测模型有望成为恶性进展风险病例早筛的新方法。
Keywords: 脑膜瘤, HOXA9, DNA甲基化, CpG位点, 诊断, 预后
Abstract
Objective
To establish a recurrence risk prediction model for meningioma based on HOXA9 DNA methylation.
Methods
Meningioma-related datasets were downloaded from GEO database for screening homeobox genes (HOXs) with prognostic values using differential methylation and ROC curve analysis and Cox regression analysis. The differentially methylated CpG sites with high predictive efficacy were selected to establish the risk prediction model using Lasso-Cox regression analysis, based on which the patients were divided into high- and low-risk groups by the cutoff value. The methylation levels of CpG sites were verified at the cell and tissue levels using methylation-specific PCR (MS-PCR). Clinical meningioma tissue samples were used to validate the predictive efficacy of the model.
Results
HOXA9 methylation level was significantly up-regulated in meningiomas (P<0.001) and showed a high diagnostic efficiency (AUC=0.884) as an independent risk factor for overall survival (P<0.01) positively correlated with the degree of malignancy and poor prognosis of meningioma (P<0.05). Risk stratification by HOXA9 methylation was more accurate than WHO grading for predicting recurrence and patient survival time. The AUCs of the sites cg03217995 and cg21001184 were both above 0.8 for meningioma diagnosis and above 0.6 for predicting recurrence. The patients' clinical characteristics differed significantly between the high- and low-risk groups (P<0.001), and the prediction score of the model was an independent prognostic factor for meningioma (P<0.05). MS-PCR results showed that the methylation levels of the two sites increased significantly in meningioma cells. In clinical samples, the combined model showed a high prediction efficiency (AUC=0.857), and the predicted risk of progression was highly consistent with the patients' actual condition.
Conclusion
High HOXA9 methylation level is a predictor for poor prognosis of meningiomas, and the combined prediction model based on its CpG sites provides a new approach to early screening of meningioma patients at risk of progression.
Keywords: meningioma, HOXA9, DNA methylation, CpG sites, diagnosis, prognosis
脑膜瘤是成人颅内常见的肿瘤,约占中枢神经系统肿瘤的30%以上[1]。根据世界卫生组织第五版WHO 中枢神经系统肿瘤分类,脑膜瘤被划分为WHO 1~3级,其中80% 为1级,在组织学上呈良性;其余20%为2级(非典型)和3级(间变性),具有恶性倾向[2]。目前,主要依据WHO分级判断肿瘤的进展风险,然而部分患者的临床病程与WHO分级并不一致,少数1级脑膜瘤全切除后频繁复发,甚至出现恶性进展,而一些2~3级脑膜瘤几乎不复发[3-5]。提示依据WHO分级系统预测脑膜瘤的进展风险并不准确,探索新的预测方法对于脑膜瘤风险分层以及改善预后具有重要意义。
基因突变、染色体拷贝数变异(CNV)和表观遗传修饰异常均与脑膜瘤的发生发展密切相关,这些特征在其早期诊断及预后评估中具有重要的应用价值[6-9]。同源异形盒基因(HOXs)家族是一组高度保守的转录因子,分为A、B、C、D 4簇,可调控哺乳动物胚胎发育、器官形成、细胞增殖及分化[10]。DNA甲基化是调控HOXs基因表达的关键表观遗传方式,其失调参与HOXs介导的肿瘤发生发展[11]。研究发现,HOXs基因甲基化异常在多种中枢神经系统肿瘤中普遍存在[12-14]。研究报道称HOXA10启动子区超甲基化参与调控胶质母细胞瘤的恶性进展[15]。一项基于7个HOXs基因甲基化的诊断模型在IDH突变型胶质瘤的诊断中优势突出[13]。然而,HOXs在脑膜瘤中的甲基化模式及其临床价值至今研究较少,缺乏关键发现。因此,本研究整合DNA甲基化以及基因表达数据集分析筛选与脑膜瘤诊断和进展相关的HOXs基因,评估该基因与脑膜瘤临床病理以及分子特征的相关性。筛选出差异CpG位点后,构建疾病进展预测模型。最后,通过甲基化特异性PCR(MS-PCR)在细胞和组织水平对差异CpG位点进行验证,并纳入脑膜瘤患者组织样本对该模型的预测效能进行验证,旨在为改善脑膜瘤患者个体预后提供更多可能性。
1. 资料和方法
1.1. 数据来源和预处理
通过GEO数据库 (https://www.ncbi.nlm.nih.gov)下载脑膜瘤基因表达相关数据集GSE85135、甲基化相关数据集GSE178143、GSE241956和GSE183656。其中数据集GSE85135基于GPL10558平台,包含96例脑膜瘤组织和25例脑膜组织,数据集GSE178143和GSE241956基于GPL13534平台,共包含34例脑膜瘤组织和12例硬脑膜组织及5例软脑膜组织,而数据集GSE183656基于GPL21145平台,包含565例脑膜瘤组织。通过Strawberry Perl 5.30.0.1提取甲基化谱和转录组矩阵,并通过KNN算法填补矩阵中的缺失值。
1.2. 基因表达和甲基化差异分析
使用R 4.3.0软件中的“limma”R包进行基因表达和甲基化差异分析,利用Wilcoxon test检验各分组之间的差异,基因表达差异筛选阈值为差异倍数 |log2FC| >0.585,adjust P<0.05,甲基化水平通过合并甲基化数据集GSE178143和GSE241956筛选,以adjust P<0.05为差异有统计学意义。利用“ggpubr”R包可视化差异表达情况,“ggplot2”R包绘制箱线图、火山图、韦恩图和热图。
1.3. 诊断和预后分析
使用“pROC”R 包绘制受试者工作特征(ROC)曲线并计算曲线下面积(AUC),预测正常脑膜组织和脑膜瘤以及原发和复发脑膜瘤结局,AUC<0.5表示无诊断价值。根据脑膜瘤患者的无复发生存期(RFS)和总生存期(OS),利用 “survival” R包进行单因素和多因素Cox回归分析,通过“forestploter”R包绘制森林图,利用Kaplan-Meier算法进行生存分析并绘制生存曲线。
1.4. HOXA9甲基化与脑膜瘤临床特征相关性分析
根据HOXA9甲基化值中位数将脑膜瘤患者分为高、低甲基化组,分析在脑膜瘤分子特征、分型、WHO分级等临床特征中的占比和差异并绘制冲积图和箱线图。统计NF2缺失、HLA多态性、HLA_DQA1、HLA_DQB1、HLA_DRB1、HLA_DRB5、CDKN2A/B缺失和USF1扩增在每个脑膜瘤样本中是否发生,发生赋1分,以每个样本的总分分析高、低甲基化分组中的基因组稳定性差异,并将总分4分及以上的样本判断为基因组不稳定。通过“reshape2”、“GSEABase”、“GSVA”等R包进行细胞增殖相关基因富集分析并绘制箱线图,利用“pec”R包对WHO分级和HOXA9甲基化水平分组进行Brier 预测分析并绘制曲线。
1.5. CpG位点筛选及预测模型构建
根据CpG位点信息,对脑膜瘤和脑膜组织中的CpG位点进行差异分析,以|Δβ| ≥0.08,adjust P<0.05为条件筛选出2个差异位点并绘制火山图,通过甲基化水平分析绘制热图和箱线图,并根据ROC曲线分析2位点诊断效能。最后通过Lasso-cox回归分析建立两位点联合预测模型,根据cut off预测评分将脑膜瘤患者分为高、低风险组,分析与脑膜瘤临床特征的相关性并进行生存曲线分析。
1.6. 细胞和组织样本
人脑膜原代细胞 HMC(武汉赛奥斯生物科技有限公司)使用配套原代成纤维细胞培养基,人脑膜瘤细胞系IOMM-Lee(上海酶研生物科技有限公司)和Ben-Men-1(上海青旗生物技术发展有限公司)使用含 10% 胎牛血清和 1% 青霉素-链霉素溶液的DMEM,在37 ℃,5% CO2条件下培养。9例脑膜瘤组织样本均来自吉林大学中日联谊医院(伦理批号:20221213003)。
1.7. 基因组DNA提取和甲基化特异性PCR(MS-PCR)
取对数生长期细胞,使用DNAiso Reagent试剂盒 (Takara) 提取基因组DNA并紫外分光光度计法测量DNA浓度和纯度以确保质量良好。取25 mg石蜡包埋组织样品,加入1.2 mL二甲苯充分脱蜡后,使用基因组DNA小量抽提试剂盒(碧云天)提取组织DNA并测量浓度和纯度。根据DNA甲基化试剂盒 (康为世纪) 的说明取20 μL DNA经亚硫酸氢钠处理并纯化后,取1 μL DNA 使用Gloria U 2×HS Master Mix试剂盒(爱博泰克)进行MS-PCR。PCR产物用3%琼脂糖凝胶电泳分离,在紫外光下观察,通过影像学分析测定每个条带的光密度值。2个CpG位点的引物由上海生工生物合成(表1)。
表1.
MS-PCR引物序列
Tab.1 Primer sequences for MS-PCR
| Site | Primer sequence (5'-3') | Product length (bp) | |
|---|---|---|---|
| cg03217995 | M | F:TGGTGTTTTGTATAGGGGTATCG | 95 |
| R:AACCCAATATTTCTCTTCCCCG | |||
| U | F:AGGGTTTGGTGTTTTGTATAGGGGTATTG | 107 | |
| R:CTCCTAAACCCAAGATTTCTCTTCCCCA | |||
| cg21001184 | M | F:GGTCGTGCGCGTTACGTGTTCGT | 92 |
| R:AAATTATAACTACAAAACATCGA | |||
| U | F:TTTATTGGTTGTGTGTGTTATGTGTTTGT | 104 | |
| R:CTATAAAAATTATAACTACAAAACATCAA | |||
M: Methylated; U: Unmethylated; F: Forward; R: Reverse.
1.8. 统计学方法
使用GraphPad Prism 8.0软件进行统计学分析和可视化处理,计量数据以均数±标准差进行统计描述。每个实验均独立重复3次。利用 Shapiro-Wilk test和Brown-Forsythe test检验数据是否符合正态分布和满足方差齐性。本研究中MS-PCR数据符合正态分布,采用单因素方差分析比较各组细胞以及同一WHO分级中各脑膜瘤组织之间的甲基化水平差异。两组间数据比较采用Wilcoxon检验。生存曲线使用log-rank检验进行比较。以P<0.05时认为差异有统计学意义。
2. 结果
2.1. HOXs家族特征基因的筛选
基于甲基化数据集分析HOXs基因在脑膜瘤和正常脑膜组织中的甲基化水平,结果显示,HOXs家族中共有25个基因甲基化水平差异有统计学意义(P<0.05),除HOXD3和HOXD4外,其余23个基因在脑膜瘤组织中的甲基化水平高于脑膜组织(P<0.05,图1A)。以AUC≥0.8作为筛选条件,得到8个有较高诊断评分的候选特征基因:HOXD11(AUC=0.891)、HOXA9(AUC=0.884)、HOXD12(AUC=0.884)、HOXD13(AUC=0.839)、HOXA11(AUC=0.837)、HOXA13(AUC=0.824)、HOXA1(AUC=0.822)和HOXC10(AUC=0.806)(图1B)。HOXs甲基化水平与患者WHO分级相关性分析结果显示,HOXA1、HOXA9、HOXA13、HOXC9和HOXD1的甲基化水平与脑膜瘤WHO级别显著正相关(P<0.05,图1C)。接下来通过基因表达数据集GSE85135筛选得到差异表达的HOXs(图1D)。基于以上得到的基因进行韦恩图分析,获得了1个基因:HOXA9(图1E)。
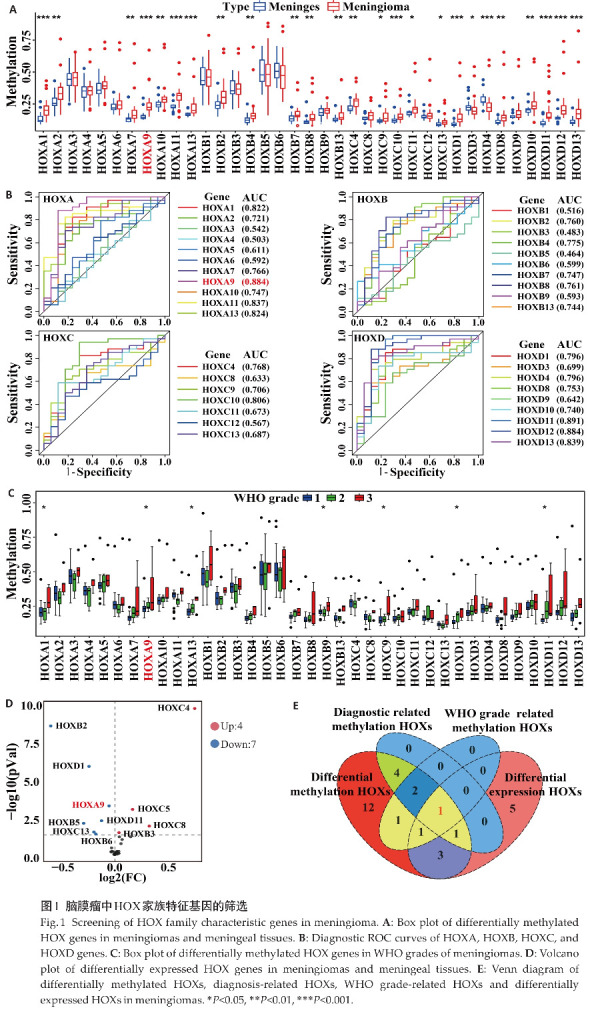
2.2. HOXA9甲基化与脑膜瘤临床特征相关性分析
通过数据集GSE183656分析HOXA9甲基化水平与脑膜瘤患者临床特征相关性,单因素Cox回归分析显示HOXA9甲基化水平、年龄、WHO分级、放疗史以及复发均是患者生存的影响因素(P<0.001)。多因素Cox回归分析显示HOXA9甲基化水平与患者OS显著负相关,是影响预后的独立危险因素(P=0.004,图2A)。脑膜瘤分子特征分析结果显示,携带CDKN2A/B缺失的脑膜瘤患者HOXA9甲基化水平高于非携带者(P<0.0001),而携带TRAF7/AKT1突变的患者HOXA9甲基化水平低于非携带者(P<0.05,图2B)。根据HOXA9甲基化水平中位数将患者分为高甲基化和低甲基化组后,发现高甲基化患者NF2失活、染色体22q缺失、1q拷贝数扩增的阳性比例较高,基因组稳定性更低(P<0.0001,图2C)。Kaplan-Meier分析显示HOXA9低甲基化组患者的RFS以及OS均大于高甲基化组患者(P<0.05,图2D)。与高甲基化组相比,低甲基化组中Merlin-intact型占比更高(17% vs 51%),Hypermitotic型占比更低(47% vs 9%),且WHO 2、3级的患者占比更低(图2E)。基因富集分析结果显示,细胞增殖相关基因在HOXA9高甲基化组中的富集分数高于低甲基化组(P<0.01,图2F)。另外,Brier 预测分析结果显示,在预测患者复发和生存时间时,基于HOXA9甲基化水平的分组方法比WHO分级精度更高(图2G)。
图2.
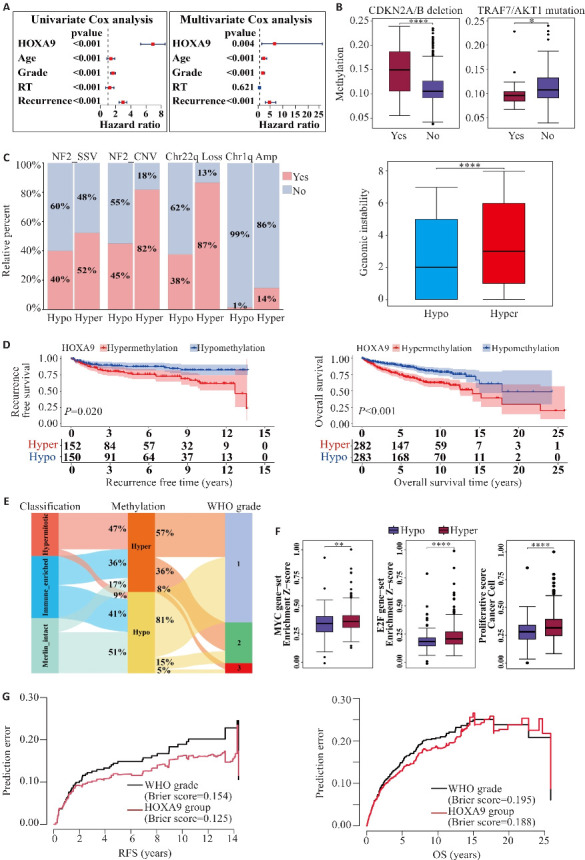
HOXA9甲基化与脑膜瘤临床特征相关性分析
Fig.2 Correlation analysis of HOXA9 methylation and clinical characteristics of meningioma. A: Univariate Cox analysis and multivariate Cox analysis of HOXA9 methylation and clinical characteristics with overall survival (OS) of meningioma patients. B: HOXA9 methylation difference in meningioma patients with CDKN2A/B deletion and TRAF7/AKT1 mutation. C: Proportion of meningioma patients with NF2-SSV, NF2-CNV, Chr22q Loss, and Chr1q Amp in hypomethylation (Hypo) and hypermethylation (Hyper) groups and box plot of the differences in gene instability. D: Kaplan-Meier survival curves for recurrence-free survival (RFS) and OS in Hypo and Hyper groups. E: Alluvial chart of Choudhury classification and WHO grade in Hypo and Hyper groups. F: Box plots comparing activation of molecular signatures of proliferation between HOXA9 groups. G: Brier prediction analysis of RFS and OS in HOXA9 groups and WHO grade in meningiomas. *P<0.05, **P<0.01, ****P<0.0001.
2.3. HOXA9差异甲基化 CpG位点的筛选
基于GSE178143和GSE241956数据集,以 |Δβ| ≥0.08,adjust P<0.05为条件筛选得到2个HOXA9甲基化差异CpG位点:cg03217995、cg21001184(图3A)。cg03217995和cg21001184分别位于HOXA9的Body区和TSS200区(图3B)。进一步分析两位点在脑膜瘤及脑膜组织中的甲基化水平,并绘制热图(图3C)。与对照相比,两位点在脑膜瘤组织中的甲基化水平均升高,差异有统计学意义(P<0.0001,图3D)。ROC曲线分析显示,cg03217995[AUC=0.884,敏感性=88.24%,特异性=94.12%] 和cg21001184[AUC=0.841,敏感性=67.65%,特异性=100%]对正常和肿瘤组织均有良好的诊断效能,两位点区分原发和复发的 AUC 分别为0.640[敏感性=55.96%,特异性=65.57%]和0.624[敏感性=69.72%,特异性=52.19%](图3E)。
图3.
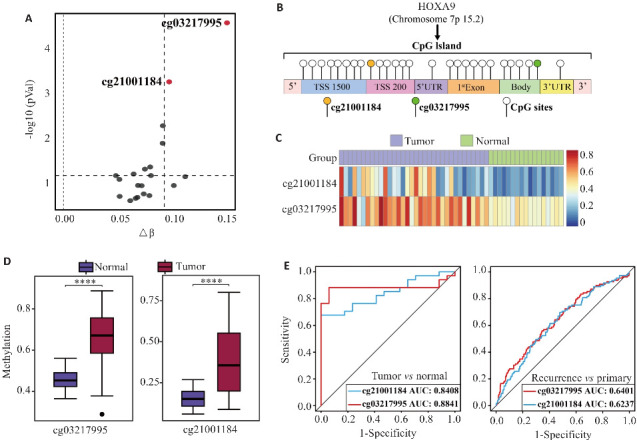
CpG位点的筛选
Fig.3 Screening of CpG sites. A: Volcano map of differential methylation of CpG sites. B: The location of CpG sited. C: Heat map of CpG sites. D: Differential methylation between cg03217995 and cg21001184 in meningiomas and meningeal tissues. E: ROC analysis of cg03217995 and cg21001184. ****P<0.0001.
2.4. cg03217995和cg21001184两位点联合预测模型的构建
采用Lasso-cox回归分析建立两位点联合预测模型:Risk score=cg03217995×1.087+cg21001184×1.222。根据模型将脑膜瘤患者分为高、低风险两组,并绘制热图展示患者预测评分分布与临床病理特征的相关性,结果显示,两组患者的生存率、复发率、WHO分级以及多种分子特征差异有统计学意义(P<0.001,图4A)。Kaplan-Meier分析显示相较于高风险组,低风险组患者的RFS以及OS更长,预后更好(P<0.05,图4B)。Cox回归分析显示,脑膜瘤患者的预测评分和WHO分级与RFS独立相关,位点cg03217995和cg21001184预测特征能够独立于其他临床病理特征作为脑膜瘤的独立预后因素(P<0.05,表2)。
图4.
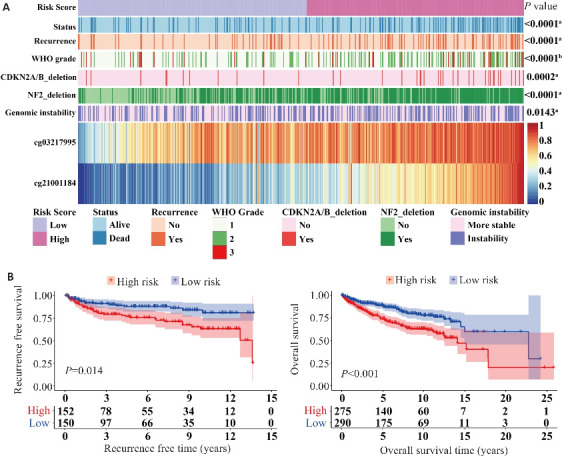
两位点联合预测模型的构建
Fig.4 Construction of the two-site combined prediction model. A: Heat map of CpG sites arranged by risk score. The relationship between the risk scores and patients' clinic pathological characteristics was evaluated (a, Wilcoxon test; b, One-way ANOVA test). B: RFS and OS survival curves grouped by risk score.
表2.
两 CpG位点RFS建模风险评分的单因素和多因素分析
Tab.2 Univariate and multivariate analysis of two CpG sites RFS modeling risk scores
| Characteristics | Total (n) | Multi score | Univariate analysis | Multivariate analysis | ||
|---|---|---|---|---|---|---|
| HR (95% CI) | P | HR (95% CI) | P | |||
| Risk score | 243 | 1.341±0.422 | 3.654 (1.586-8.420) | 0.002 | 2.725 (1.184-2.269) | 0.018 |
| WHO grade | ||||||
| 1 | 212 | 1.317±0.416 | 4.391 (2.504-7.700) | <0.0001 | 3.203 (1.257-8.161) | 0.015 |
| 2 | 29 | 1.505±0.450 | ||||
| 3 | 2 | 1.548±0.222 | ||||
| RT | ||||||
| Yes | 29 | 1.499±0.390 | 4.382 (2.071-9.072) | <0.0001 | 1.282 (0.380-2.322) | 0.689 |
| No | 214 | 1.320±0.423 | ||||
| Age (year) | ||||||
| ≤60 | 139 | 1.304±0.393 | 0.934 (0.484-1.802) | 0.838 | ||
| >60 | 104 | 1.390±0.456 | ||||
RT: Radiotherapy.
2.5. cg03217995和cg21001184两位点联合预测模型效能验证
MS-PCR结果显示,位点cg03217995和cg21001184在人脑膜细胞中的甲基化水平均低于脑膜瘤细胞(P<0.0001),与恶性程度较高的IOMM-Lee细胞相比,Ben-Men-1细胞中位点cg03217995的甲基化水平较低(P<0.0001,图5A);而位点cg21001184甲基化水平在两株脑膜瘤细胞中无统计学差异。选取WHO分级1-3级的患者(表3),进一步评估两位点联合模型对脑膜瘤进展的预测效能,结果显示,两位点甲基化水平在不同WHO分级患者间无统计学差异,其中患者3 cg03217995位点的甲基化水平高于WHO 1级其他患者(P<0.0001),患者6 两位点的甲基化水平均显著高于WHO 2级其他患者(P<0.0001,图5A)。将cg03217995和cg21001184位点 β 值带入两位点联合预测模型公式得到各研究对象的预测评分,ROC曲线分析显示,两位点联合模型的AUC达到0.857,敏感度和特异度分别为100% 和85.71%(图5B)。以1.226作为cut off值将研究对象分为预测进展组(预测评分≥1.226)和预测无进展组(预测评分<1.226),结果显示患者3(预测评分1.228)及患者6(预测评分1.420)均属于高风险组,与真实临床进展结果高度一致(图5C)。
图5.
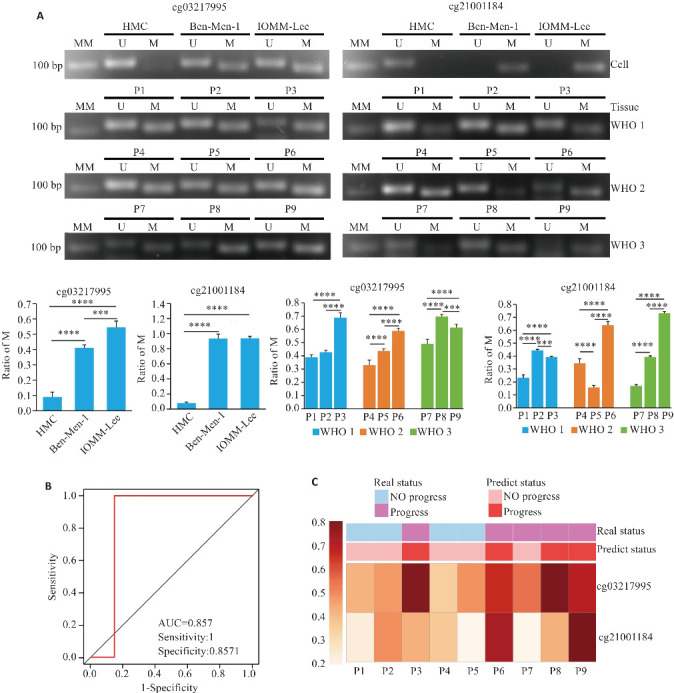
两位点联合预测模型效能验证
Fig.5 Validation of the combined prediction model of the two sites. A: MS-PCR results of CpG sites cg03217995 and cg21001184 in meningioma cells and meningioma tissues. The density of each strip was quantified by imaging analysis, and the relative band density value was calculated as the ratio of methylation to methylation plus unmethylation (M/U+M). U: Unmethylated; M: Methylated; MM: molecular marker. B: ROC of CpG sites cg03217995 and cg21001184 for diagnosis of meningioma patients. C: Prediction of meningioma patients at CpG sites cg03217995 and cg21001184. P: Patient. ***P<0.001, ****P<0.0001.
表3.
脑膜瘤组织样本临床信息和实验结果
Tab.3 Clinical information and experimental results of meningioma tissue samples
| Case No. | WHO grade | Progression | β value | Risk score | |
|---|---|---|---|---|---|
| cg03217995 | cg21001184 | ||||
| 1 | 1 | no | 0.389±0.018 | 0.233±0.022 | 0.708 |
| 2 | 1 | no | 0.428±0.016 | 0.445±0.008 | 1.008 |
| 3 | 1 | yes (Invasion) | 0.668±0.037 | 0.393±0.006 | 1.228 |
| 4 | 2 | no | 0.330±0.039 | 0.344±0.036 | 0.779 |
| 5 | 2 | no | 0.437±0.015 | 0.157±0.016 | 0.668 |
| 6 | 2 | yes (Multiple recurrence) | 0.588±0.019 | 0.639±0.029 | 1.420 |
| 7 | 3 | no | 0.492±0.034 | 0.169±0.011 | 0.741 |
| 8 | 3 | no | 0.696±0.016 | 0.393±0.008 | 1.238 |
| 9 | 3 | no | 0.614±0.024 | 0.731±0.013 | 1.561 |
3. 讨论
DNA甲基化是最常见的表观遗传学改变方式,在不影响基因序列的前提下通过碱基的特定化学修饰改变染色质结构、DNA构象、DNA稳定性以及DNA-蛋白质互作而调控基因的转录和表达[16]。DNA的异常甲基化贯穿于肿瘤发生和发展的全过程,与CNV、基因突变相比具有更高的敏感性、特异性和稳定性,可通过甲基化特异性PCR进行快速检测[17]。因此,DNA甲基化标志物可辅助肿瘤早期诊断、评估进展风险并作为化疗敏感性的标志[18, 19]。近年来,研究发现HOXs基因在多种恶性肿瘤中异常表达,其甲基化水平有肿瘤特异性诊断与治疗的价值。房明浩等[20]报道称HOXA簇基因TSS区CpG岛甲基化状态在各种白血病细胞中差异明显,体现出不同种类白血病的特异性,可作为区分AML、ALL和CML的分子标志。HOXA4低甲基化是乳腺癌早期诊断潜在标志物[21], HOXA10基因启动子区低甲基化与卵巢上皮癌的不良预后有关[22]。此外,HOXB5、HOXB7启动子低甲基化是小细胞肺癌转移的预测指标[23]。HOXD10启动子高甲基化与胃癌患者的低生存率显著相关,可作为胃癌预后的生物标志物[24]。另外,HOXD10甲基化与原发性肝癌患者血管癌性栓塞、肿瘤细胞分化、3年总生存率显著相关,可作为HCC的预后标志物[25]。本研究通过公共数据集对HOXs基因在脑膜瘤组织中的甲基化模式以及诊断价值进行整体评估,发现与脑膜组织相比该家族大部分基因在脑膜瘤组织中高甲基化,HOXD亚簇基因整体具有较好的诊断效能。在此基础之上,我们整合患者WHO分级信息和基因差异表达数据确定HOXA9为候选基因。
HOXA9基因是位于7号染色体上的HOXA亚簇的最末端成员,其表达受到miRNA、DNA结合辅助因子以及表观遗传调控因子等多种方式的调节,其中DNA甲基化是最重要的表观遗传调控方式。Han等[26]研究发现,HOXA9甲基化水平与胃癌患者的UICC分期、组织分化程度、T分期、M 分期及复发正相关;Wu等[27]发现HOXA9甲基化水平与膀胱癌TNM 分期、淋巴结转移及病理分期正相关。此外,Kim等[28]报道HOXA9高甲基化促进NMIBC患者复发和进展。本研究发现HOXA9基因在脑膜瘤组织中的甲基化水平显著高于脑膜组织而基因表达水平显著低于脑膜组织。Cox回归分析发现其甲基化水平与患者WHO分级正相关,与患者OS负相关,提示HOXA9基因高甲基化是脑膜瘤患者预后不良的独立因素。
近年来,越来越多的研究证实脑膜瘤的分子病理学特征与其发生和进展密切相关。有研究[29]从528例脑膜瘤人群中鉴定出4.9%的患者携带CDKN2A/B基因缺失,这类患者有明显更差的预后和更快的疾病进展速度。相反,携带TRAF7和AKT1/KLF4共突变、SMO突变或POLR2A突变的患者通常有较好的预后和较低的进展风险[30]。本研究发现携带CDKN2A/B缺失的脑膜瘤患者HOXA9甲基化水平显著高于非携带者,而携带TRAF7/AKT1突变的患者HOXA9甲基化水平显著低于非携带者。并且HOXA9高甲基化组患者的RFS时间显著低于低甲基化组,进一步验证HOXA9基因高甲基化患者更容易出现复发、恶性进展等不良预后情况。本研究分析两组患者CNV情况后还发现高甲基化组患者染色体22q缺失、NF2 失活、1q拷贝数扩增的阳性比例较高,基因组稳定性更低,与彭等报道的高级别脑膜瘤有更不稳定的基因组相一致[31],再一次验证了HOXA9甲基化水平与脑膜瘤恶性程度相关。
2022年Choudhury等[9]根据DNA甲基化谱将565例脑膜瘤样本分为:Merlin-intact、Immune-enriched和Hypermitotic 3个亚型。Merlin-intact型患者对细胞毒性治疗最敏感,临床预后最佳;Immune-enriched型患者免疫细胞浸润水平较高,临床预后次之;Hypermitotic型患者细胞增殖活性最高,临床预后最差。我们将患者按HOXA9甲基化分组与Choudhury分型交叉分析后发现,HOXA9高甲基化组中Hypermitotic型患者占比最高而低甲基化组Merlin-intact患者占比最高。Alluvial plot分析显示HOXA9甲基化分组方法与WHO分级有相关性但不完全重叠。并且,HOXA9甲基化分组方法在预测患者复发和生存时间时比WHO分级精度更高。以上结果说明基于HOXA9甲基化的分组能够反映脑膜瘤组织学、细胞遗传学和基因组学异质性,有潜力成为一种新的分类方法。
尽管改善WHO分级系统预后的新方法不断出现,但方法复杂、缺少执行标准、检测成本昂贵等问题限制了其在临床实践中的应用[6, 9]。为简化评估流程、降低患者检测成本,本研究进一步筛选了HOXA9基因差异甲基化CpG位点cg03217995和cg21001184。发现两位点在脑膜瘤组织中甲基化水平显著升高。两位点对脑膜瘤诊断的AUC均大于0.8,预测脑膜瘤患者复发的AUC均大于0.6,说明两位点均有潜力成为诊断及疾病进展的分子标志物。基于此,本研究构建两位点联合预测模型,结果表明联合模型预测评分与脑膜瘤患者的恶性临床特征呈正相关,与患者OS、RFS呈负相关,是独立的临床风险评估指标。为进一步探讨cg03217995和cg21001184两位点联合模型在脑膜瘤进展预测中的应用,本研究纳入了9例脑膜瘤组织样本,通过MS-PCR对联合模型的预测效能进行验证。结果显示,该模型AUC值大于0.8,且具有较高的灵敏度和特异性。值得注意的是,研究对象中,后期发生恶性进展的病例WHO 1级患者3和WHO 2级患者6实属HOXA9两CpG位点联合模型的预测进展组,提示依据该模型分组可达到恶性进展风险病例早筛的目的。
综上所述,本研究首先通过公共数据集分析了HOXs基因在脑膜瘤组织中的甲基化模式以及诊断价值,筛选出候选基因HOXA9。进一步分析HOXA9甲基化水平与脑膜瘤临床病理以及分子特征的相关性后,明确了其在脑膜瘤预后评估中对比WHO分级的优势。接着通过差异DNA甲基化图谱筛选出差异显著的甲基化位点cg03217995和cg21001184,Lasso-cox回归建立基于两位点甲基化的联合预测模型。最后,再采用MS-PCR在细胞和组织水平对两位点的甲基化进行验证,并结合脑膜瘤患者临床信息对两位点甲基化联合模型的预测效能进行验证。基于此,本研究认为此联合预测模型可以用于评估超出中枢神经系统WHO分级患者的复发及恶性进展,对于患者风险分层以及改善预后具有重要意义。但本研究存在以下不足:临床样本量有限,临床信息不完整,需加大样本进一步验证两位点甲基化的联合预测模型的效能;另一方面,HOXA9甲基化对基因表达的影响机制以及对脑膜瘤恶性进展的调控机制是后续研究的关注重点。
基金资助
2024年浙江省基础公益研究计划项目(Y24H160094); 浙江中医药大学2023年校级研究生科学研究基金项目(2023YKJ06);2024年吉林省自然科学基金(YDZJ202401231ZYTS)
参考文献
- 1. Ostrom QT, Gittleman H, Truitt G, et al. CBTRUS statistical report: primary brain and other central nervous system tumors diagnosed in the United States in 2011-2015[J]. Neuro Oncol, 2018, 20(suppl_4): iv1-86. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2. Louis DN, Perry A, Wesseling P, et al. The 2021 WHO Classification of Tumors of the Central Nervous System: a summary[J]. Neuro Oncol, 2021, 23(8): 1231-51. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3. Roehrkasse AM, Peterson JEG, Fung KM, et al. The discrepancy between standard histologic WHO grading of meningioma and molecular profile: a single institution series[J]. Front Oncol, 2022, 12: 846232. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4. Robert SM, Vetsa S, Nadar A, et al. The integrated multiomic diagnosis of sporadic meningiomas: a review of its clinical implications[J]. J Neurooncol, 2022, 156(2): 205-14. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5. Pellerino A, Bruno F, Palmiero R, et al. Clinical significance of molecular alterations and systemic therapy for meningiomas: where do we stand?[J]. Cancers, 2022, 14(9): 2256. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6. Maas SLN, Stichel D, Hielscher T, et al. Integrated molecular-morphologic meningioma classification: a multicenter retrospective analysis, retrospectively and prospectively validated[J]. J Clin Oncol, 2021, 39(34): 3839-52. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7. Nassiri F, Liu J, Patil V, et al. A clinically applicable integrative molecular classification of meningiomas[J]. Nature, 2021, 597(7874): 119-25. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8. Driver J, Hoffman SE, Tavakol S, et al. A molecularly integrated grade for meningioma[J]. Neuro Oncol, 2022, 24(5): 796-808. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9. Choudhury A, Magill ST, Eaton CD, et al. Meningioma DNA methylation groups identify biological drivers and therapeutic vulnerabilities[J]. Nat Genet, 2022, 54(5): 649-59. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10. Gehring WJ, Hiromi Y. Homeotic genes and the homeobox[J]. Annu Rev Genet, 1986, 20: 147-73. [DOI] [PubMed] [Google Scholar]
- 11. Hu X, Wang Y, Zhang XY, et al. DNA methylation of HOX genes and its clinical implications in cancer[J]. Exp Mol Pathol, 2023, 134: 104871. [DOI] [PubMed] [Google Scholar]
- 12. Di Vinci A, Brigati C, Casciano I, et al. HOXA7, 9, and 10 are methylation targets associated with aggressive behavior in meningiomas[J]. Transl Res, 2012, 160(5): 355-62. [DOI] [PubMed] [Google Scholar]
- 13. Mamatjan Y, Voisin MR, Nassiri F, et al. Integrated molecular analysis reveals hypermethylation and overexpression of HOX genes to be poor prognosticators in isocitrate dehydrogenase mutant glioma[J]. Neuro Oncol, 2023, 25(11): 2028-41. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14. Ghisai SA, van Hijfte L, Vallentgoed WR, et al. Epigenetic landscape reorganization and reactivation of embryonic development genes are associated with malignancy in IDH-mutant astrocytoma[J]. bioRxiv, 2024: 2024. 03. 19,. 585212. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15. Kurscheid S, Bady P, Sciuscio D, et al. Chromosome 7 gain and DNA hypermethylation at the HOXA10 locus are associated with expression of a stem cell related HOX-signature in glioblastoma[J]. Genome Biol, 2015, 16(1): 16. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16. Schmitz RJ, Lewis ZA, Goll MG. DNA methylation: shared and divergent features across eukaryotes[J]. Trends Genet, 2019, 35(11): 818-27. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17. Constâncio V, Nunes SP, Henrique R, et al. DNA methylation-based testing in liquid biopsies as detection and prognostic biomarkers for the four major cancer types[J]. Cells, 2020, 9(3): 624. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18. Palanca-Ballester C, Rodriguez-Casanova A, Torres S, et al. Cancer epigenetic biomarkers in liquid biopsy for high incidence malignancies[J]. Cancers, 2021, 13(12): 3016. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19. Ibrahim J, Peeters M, van Camp G, et al. Methylation biomarkers for early cancer detection and diagnosis: current and future perspectives[J]. Eur J Cancer, 2023, 178: 91-113. [DOI] [PubMed] [Google Scholar]
- 20. 房明浩, 刘文励, 孟凡凯, 等. 白血病细胞HOX A基因亚群启动子区域异常甲基化的研究[J]. 中华血液学杂志, 2009, 30(7): 468-72. [PubMed] [Google Scholar]
- 21. Li SY, Wu HC, Mai HF, et al. Microarray-based analysis of whole-genome DNA methylation profiling in early detection of breast cancer[J]. J Cell Biochem, 2019, 120(1): 658-70. [DOI] [PubMed] [Google Scholar]
- 22. Cheng WJ, Jiang Y, Liu CX, et al. Identification of aberrant promoter hypomethylation of HOXA10 in ovarian cancer[J]. J Cancer Res Clin Oncol, 2010, 136(8): 1221-7. [DOI] [PubMed] [Google Scholar]
- 23. Na FF, Pan XY, Chen JY, et al. KMT2C deficiency promotes small cell lung cancer metastasis through DNMT3A-mediated epigenetic reprogramming[J]. Nat Cancer, 2022, 3(6): 753-67. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24. Lin ZH, Luo MZ, Chen XQ, et al. Combined detection of plasma ZIC1, HOXD10 and RUNX3 methylation is a promising strategy for early detection of gastric cancer and precancerous lesions[J]. J Cancer, 2017, 8(6): 1038-44. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25. Guo YL, Peng YJ, Gao D, et al. Silencing HOXD10 by promoter region hypermethylation activates ERK signaling in hepatocellular carcinoma[J]. Clin Epigenetics, 2017, 9: 116. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26. Han Y, Tu WW, Wen YG, et al. Identification and validation that up-expression of HOXA13 is a novel independent prognostic marker of a worse outcome in gastric cancer based on immunohistochemistry[J]. Med Oncol, 2013, 30(2): 564. [DOI] [PubMed] [Google Scholar]
- 27. Wu YS, Jiang GL, Zhang N, et al. HOXA9, PCDH17, POU4F2, and ONECUT2 as a urinary biomarker combination for the detection of bladder cancer in Chinese patients with hematuria[J]. Eur Urol Focus, 2020, 6(2): 284-91. [DOI] [PubMed] [Google Scholar]
- 28. Kim YJ, Yoon HY, Kim JS, et al. HOXA9, ISL1 and ALDH1A3 methylation patterns as prognostic markers for nonmuscle invasive bladder cancer: array-based DNA methylation and expression profiling[J]. Int J Cancer, 2013, 133(5): 1135-42. [DOI] [PubMed] [Google Scholar]
- 29. Clark VE, Erson-Omay EZ, Serin A, et al. Genomic analysis of non-NF2 meningiomas reveals mutations in TRAF7, KLF4, AKT1, and SMO[J]. Science, 2013, 339(6123): 1077-80. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 30. Sahin B, Katar S, Şahin SA, et al. Influence of human telomerase reverse transcriptase mutation on the aggressiveness and recurrence in meningiomas[J]. Cureus, 2021, 13(5): e15342. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 31. 彭怡琛, 郑 义, 李生兰, 等. 高级别脑膜瘤中异常甲基化调控相关基因识别研究[J]. 首都医科大学学报, 2023, 44(5): 747-52. [Google Scholar]


