Abstract
慢性脑低灌注(CCH)在血管性痴呆(VD)的发生和发展中起着重要作用。近年来的研究表明,脑缺血过程中涉及多阶段的免疫炎症反应,因而脑缺血的免疫治疗逐渐受到关注。本研究旨在通过生物信息学方法,从免疫学的视角确定CCH的潜在免疫治疗靶点。我们共鉴定出823个与CCH相关的差异基因,并筛选出9个与免疫功能相关的核心基因,分别是RASGRP1、FGF12、SEMA7A、PAK6、EDN3、BPHL、FCGRT、HSPA1B和MLNR。基因富集分析显示,这些核心基因主要参与细胞生长、神经投射延伸及间充质干细胞迁移等生物学功能。生物信号通路分析表明,核心基因主要参与T细胞受体、Ras及MAPK等信号通路的调控。通过LASSO回归,我们进一步筛选出RASGRP1和BPHL作为关键的免疫相关核心基因。同时,结合差异miRNA及miRwalk数据库,我们确定miR-216b-5p为调控RASGRP1的关键免疫相关miRNA。综上所述,本研究预测的miR-216b-5p/RASGRP1信号通路在CCH的免疫调节中具有重要意义,有望为CCH的免疫治疗提供新的靶点。
Keywords: 慢性脑低灌注, 免疫治疗, 生物信息学
Abstract
Chronic cerebral hypoperfusion (CCH) plays an important role in the occurrence and development of vascular dementia (VD). Recent studies have indicated that multiple stages of immune-inflammatory response are involved in the process of cerebral ischemia, drawing increasing attention to immune therapies for cerebral ischemia. This study aims to identify potential immune therapeutic targets for CCH using bioinformatics methods from an immunological perspective. We identified a total of 823 differentially expressed genes associated with CCH, and further screened for 9 core immune-related genes, namely RASGRP1, FGF12, SEMA7A, PAK6, EDN3, BPHL, FCGRT, HSPA1B and MLNR. Gene enrichment analysis showed that core genes were mainly involved in biological functions such as cell growth, neural projection extension, and mesenchymal stem cell migration. Biological signaling pathway analysis indicated that core genes were mainly involved in the regulation of T cell receptor, Ras and MAPK signaling pathways. Through LASSO regression, we identified RASGRP1 and BPHL as key immune-related core genes. Additionally, by integrating differential miRNAs and the miRwalk database, we identified miR-216b-5p as a key immune-related miRNA that regulates RASGRP1. In summary, the predicted miR-216b-5p/RASGRP1 signaling pathway plays a significant role in immune regulation during CCH, which may provide new targets for immune therapy in CCH.
Keywords: Chronic cerebral hypoperfusion, Immunotherapy, Bioinformatics
0. 引言
血管性痴呆(vascular dementia,VD)在全球认知障碍类疾病中位列第二,其发病率、致残率和致死率较高,严重影响着人类的生存和生活质量。慢性脑低灌注(chronic cerebral hypoperfusion,CCH)被公认为导致VD的重要危险因素之一。CCH可通过多种机制导致神经元损伤和白质病变,如神经递质紊乱、氧化应激及神经炎性反应等,进而导致神经退行性改变及认知功能障碍。然而,目前学者对CCH诱发VD的潜在病理生理学机制的认识仍然有限[1]。
近年来随着对CCH病理机制理解的加深,研究者们逐渐认识到免疫系统在这一过程中扮演着关键角色,特别是小胶质细胞及炎症反应对脑组织损伤的影响[2]。例如,CCH诱导的M1型小胶质细胞能够释放多种促炎细胞因子,如肿瘤坏死因子-α(tumor necrosis factor-α,TNF-α)和白细胞介素1β(interleukin-1β,IL-1β),这些因子不仅加剧了炎症反应,还进一步加重了神经元的损伤[3];此外,细胞因子介导的炎症信号通路,包括核转录因子-κB(nuclear factor-kappa B,NF-κB)和酪氨酸蛋白激酶/信号转导子和转录激活子通路(Janus kinase and signal transducer and activator of transcription,JAK/STAT)在CCH的病理过程中也发挥着重要作用[4]。研究表明,针对免疫反应过程中的靶点进行探究,有助于识别出用于认知障碍诊断及治疗的新型生物标志物,从而促进临床转化应用,制定个体化的免疫治疗方案[5]。因此,在CCH的研究领域,免疫治疗靶点的筛选逐渐成为一个重要的研究方向。近年来,转录组学、代谢组学及蛋白质组学的生物信息学研究在CCH疾病研究中发挥了重要的作用[6-7]。为了进一步探寻CCH的免疫治疗靶点,本研究采用生物信息学方法,从免疫学的视角识别相关靶点。
本研究通过GSE199508数据集获取差异基因,使用加权基因相关性网络分析(weighted correlation network analysis,WGCNA)构建基因模块,筛选免疫相关核心基因,并利用最小绝对值收缩和选择算子(least absolute shrinkage and selection operator,LASSO)回归算法确定关键免疫相关核心基因;同时基于差异miRNA和miRWalk数据库进一步筛选出关键免疫相关miRNA,为CCH的免疫治疗提供新的靶点。
1. 资料与方法
1.1. 数据下载及预处理
本研究通过R下载函数获得GSE199508基因表达矩阵及平台信息。通过标准化及探针转化,得到包含28897个基因及6例大鼠脑皮层组织样本的基因表达矩阵,通过探针转化得到基因名称。6例大鼠脑皮层组织包括3例对照(Sham)大鼠皮层组织和3例CCH大鼠皮层组织。
1.2. 筛选Sham大鼠皮层组织与CCH大鼠皮层组织差异基因
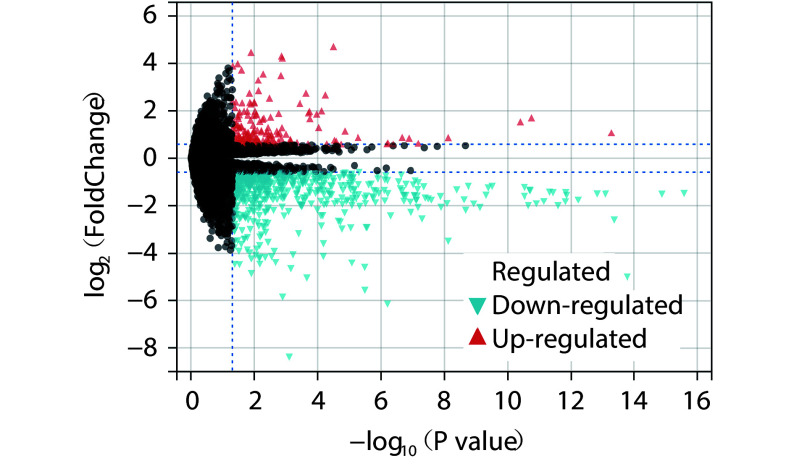
以|logFC| ≥ 0.6和P值<0.05作为筛选标准,利用“Limma”R包识别GSE199508中的差异表达基因(differentially expressed genes,DEGs),其中logFC ≥ 0.6、P < 0.05为上升,log FC ≤ –0.6、 P < 0.05为下降。使用“ggplot2”R包绘制差异基因的火山图,其中,红色代表上调基因,绿色代表下调基因。
1.3. WGCNA及基因模块分析
按照WGCNA方法,采用分步骤的方式来构建网络及模块识别,并进行分层聚类。利用动态剪切树法切割基因模块,用hclust合并高度相似的模块。通过WGCNA分析得到基因模块之后,采用Pearson相关性分析方法,将基因模块与临床性状进行相关性分析。
1.4. 筛选免疫相关核心基因并进行富集分析
将Sham大鼠皮层组织与CCH大鼠皮层组织差异基因、模块基因及免疫相关基因取交集,筛选出免疫相关核心基因。接下来对筛选出的免疫相关核心基因进一步进行基因本体论分析(gene ontology,GO)和京都基因与基因组百科全书分析(Kyoto Encyclopedia of Genes and Genomes,KEGG)富集分析。其中GO分析包括生物学过程(biological process,BP)、分子功能(molecular function,MF)和细胞组分(cellular component,CC)三类。
1.5. 蛋白质-蛋白质相互作用网络分析及LASSO回归筛选结果
GeneMANIA是一个公共资源库,用于构建蛋白质-蛋白质相互作用(protein-protein interaction,PPI)网络、提出有关基因功能的假设、显示基因列表以及为功能分析确定基因的优先级[8]。我们使用GeneMANIA探索蛋白质的共表达,并构建了研究中筛选出的免疫相关核心基因PPI网络。为了得到最精简的预测模型,本研究进一步通过交叉验证选择最佳λ值,在保证拟合度的同时纳入最少变量。通过交叉验证,使用LASSO回归算法从差异有统计学意义的免疫相关核心基因中筛选出具有非零系数的关键免疫相关基因。
1.6. 关键免疫相关基因分析验证及miRNA预测
本研究进一步基于GEO在线数据库分析免疫相关基因在Sham及CCH大鼠脑皮层组织中的基因表达水平。并利用GSE199508数据集,基于Limma包分析miRNA差异表达矩阵。以“P < 0.05及|logFC| ≥ 0.6”为筛选条件,得到差异表达miRNA,进一步基于miRWalk数据库获得与关键免疫相关基因有关的miRNA,同时将下调的差异miRNA与CCH高表达基因相关miRNA取交集,将上调的差异miRNA与CCH低表达基因相关miRNA取交集,筛选出关键免疫相关miRNA。
2. 结果
2.1. 基因获取及差异基因分析
结果显示,GSE199508数据集中Sham组及CCH组的差异基因DEGs共823个,其中186个上调,637个下调。如图1所示,红色代表186个上调基因,绿色代表637个下调基因。
图 1.
mRNA difference volcano map
mRNA 差异火山图
2.2. WGCNA分析
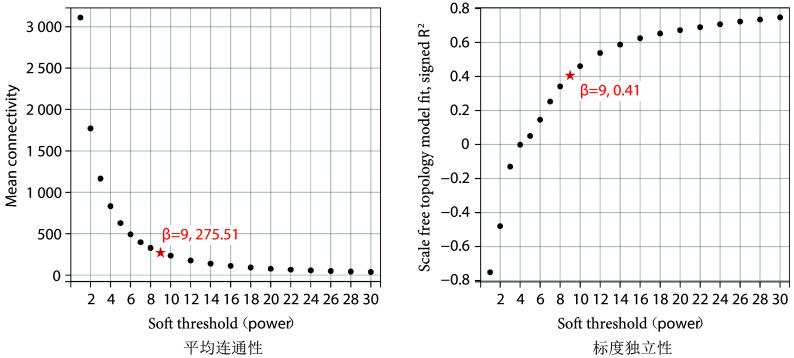
按照WGCNA分析的相应步骤构建共表达网络,并进行分层聚类。利用动态剪切树法切割基因模块,用hclust函数合并高度相似的模块,经平均连通性和标度独立性显示,WGCNA分析的最适软阈值β = 9(图2),在该软阈值的条件下,共将基因分为15个共表达模块。在表1中,一种颜色代表着一个基因模块。通过WGCNA分析得到基因模块之后,采用Pearson相关性分析方法,将基因模块与临床性状进行相关性分析。如表1所示,在15个基因模块中,cyan模块、turquoise模块与CCH有相关性(P < 0.05),它们的相关系数分别为0.95和–0.89,表明cyan模块的基因与CCH呈正相关,而turquoise模块的基因与CCH是负相关,其中cyan模块与CCH的相关性最高。
图 2.
Optimal soft thresholding power for WGCNA
WGCNA分析的最适软阈值
表 1. Correlation analysis between WGCNA gene modules and CCH.
WGCNA基因模块与CCH的相关分析
| 基因模块 | 相关性 | P值 | |
| Group CCH | Group Sham | ||
| 注:加粗字体为与CCH有相关性的模块 | |||
| purple | 0.26 | –0.26 | 0.61 |
| pink | 0.29 | –0.29 | 0.58 |
| royalblue | 0.37 | –0.37 | 0.47 |
| black | 0.63 | –0.63 | 0.18 |
| cyan | 0.95 | –0.95 | 0.003 |
| white | –0.07 | 0.07 | 0.89 |
| darkturquoise | 0.14 | –0.14 | 0.79 |
| sienna3 | 0.78 | –0.78 | 0.07 |
| darkolivegreen | 0.48 | –0.48 | 0.33 |
| grey60 | 0.19 | –0.19 | 0.71 |
| darkgrey | –0.13 | 0.13 | 0.81 |
| darkred | –0.28 | 0.28 | 0.59 |
| turquoise | –0.89 | 0.89 | 0.02 |
| brown | –0.44 | 0.44 | 0.38 |
| darkmagenta | –0.39 | 0.39 | 0.44 |
2.3. 免疫相关基因鉴定
进一步将823个差异基因、847个cyan模块基因以及1 793个免疫相关基因取交集,从而筛选出9个免疫相关核心基因(图3),分别是RASGRP1、FGF12、SEMA7A、PAK6、EDN3、BPHL、FCGRT、HSPA1B与MLNR。
图 3.
Venn map of differentially expressed genes, WGCNA and immune-related genes
差异表达基因、WGCNA和免疫相关基因的Venn图谱
2.4. GO和KEGG富集分析
接下来对筛选出的免疫相关核心基因进一步进行GO和KEGG富集分析。图4中GO分析展示免疫相关核心基因在生物学过程、分子功能和细胞组分的前7个最显著的分析结果,结果显示了核心基因主要参与细胞生长、神经投射延伸、间充质干细胞迁移等生物学功能;图4中KEGG分析展示免疫相关核心基因富集到的前9个最显著生物信号通路,涉及到的通路包括T细胞受体信号通路、Ras及丝裂原活化蛋白激酶(mitogen-activated protein kinases,MAPK)等信号通路。
图 4.

GO enrichment analysis (left) and KEGG enrichment analysis (right)
GO富集分析(左)和 KEGG富集分析(右)
2.5. PPI网络构建
利用GeneMANIA数据库将筛选出的9个免疫相关核心基因进一步构建了基因编码蛋白质的PPI网络,并进行可视化分析(见图5)。
图 5.
PPI network of immune-related core genes
免疫相关核心基因的PPI网络
2.6. LASSO回归筛选结果
使用LASSO回归算法从以上9个差异有统计学意义的免疫相关核心基因中筛选出具有非零系数的关键免疫相关基因RASGRP1和BPHL,对应系数分别为9.764和–5.561(见图6)。
图 6.
Relationship between log10 λ value and LASSO regression coefficient (left) and model error (right)
log10λ值与LASSO回归系数(左)和模型误差(右)的关系
2.7. 关键免疫相关基因分析验证
本研究进一步基于GEO在线数据库分析RASGRP1和BPHL在Sham及CCH大鼠脑皮层组织中的基因表达水平。如图7所示,与Sham相比,RASGRP1在CCH大鼠脑皮层组织中高表达,而BPHL在CCH大鼠脑皮层组织中低表达。
图 7.
RASGRP1 and BPHL expression in GSE199508 gene expression matrix
RASGRP1和BPHL在GSE199508基因表达矩阵中的表达
2.8. 关键免疫相关miRNA预测
利用GSE199508数据集,基于Limma包进行miRNA差异表达分析。筛选后共得到差异表达miRNA 33个,其中显著上调miRNA 3个,显著下调miRNA 30个,如图8a所示。进一步基于miRWalk数据库获得与RASGRP1和BPHL有关的miRNA,同时将下调的差异miRNA与RASGRP1相关miRNA取交集(见图8b),将上调的差异miRNA与BPHL相关miRNA取交集(见图8c),最终筛选出miR-216b-5p(19~39 bp大小的内源性非编码RNA)为关键免疫相关miRNA。
图 8.
Prediction of key immune-related miRNAs
预测关键的免疫相关miRNA
a. 差异表达miRNA火山图;b. 预测靶向RASGRP1的miRNA;c. 预测靶向BPHL的miRNA
a. volcano plot of differentially expressed miRNAs; b. prediction of miRNA targeting RASGRP1; c. prediction of miRNA targeting BPHL
3. 讨论
过去十年的研究已经表明,CCH可能通过影响神经能量衰竭、产生活性氧及激活小胶质细胞产生促炎细胞因子,进而破坏神经元和诱导白质病变,促进神经退化、VD以及阿尔茨海默病(Alzheimer’s disease,AD)的进展[9-17]。随着高通量组学技术的发展和数据分析能力的提高,研究者开始利用这些工具深入挖掘CCH的分子机制。通过基因表达分析、功能富集、网络生物学和机器学习方法的应用,研究者已识别出多个可能的CCH靶点,并为未来的临床研究和新疗法开发提供了重要基础[18-19]。近些年,随着对CCH及其病理机制的理解不断加深,研究者们也逐步发现免疫系统在这一过程中扮演着关键角色,已有大量研究报道了免疫相关基因在VD中的调控作用[20-21],进一步利用高通量数据从免疫角度寻找CCH治疗靶点必将拓展CCH免疫治疗的方向[22]。
本项研究基于GSE199508基因表达矩阵及平台信息,比较CCH大鼠皮层组织和Sham大鼠皮层组织的基因表达谱,得到了差异表达基因DEGs共823个,其中186个上调,637个下调。经系列分析本研究得到了免疫相关核心基因,分别是RASGRP1、FGF12、SEMA7A、PAK6、EDN3、BPHL、FCGRT、HSPA1B与MLNR。通过将免疫相关核心基因进行KEGG和GO功能富集分析,结果显示核心基因主要参与的生物功能包括细胞生长、神经投射延伸及间充质干细胞迁移等;主要涉及到的信号通路包括T细胞受体、Ras及MAPK等信号通路。进一步对筛选出的免疫相关核心基因进行文献检索发现,RASGRP1主要作为Ras家族小三磷酸鸟苷酸酶(guanosine triphosphate enzyme,GTP酶)的激活因子,参与调控细胞增殖、分化和存活等基本生物学过程。有研究报道,RASGRP1可通过与三磷酸鸟苷酸(guanosine triphosphate,GTP)结合促进Ras的活化,从而启动下游信号通路,包括丝裂原活化蛋白激酶/胞外信号调节激酶通路(mitogen-activated protein kinases/extracellular signal-regulated kinases,MAPK/ERK)和磷脂酰肌醇3-激酶/蛋白激酶B(phosphatidylinositol 3-kinases/v-Akt murine thymoma viral oncogene homolog,PI3K/Akt),这些通路在免疫细胞功能、发育和炎症反应中至关重要[23]。FGF12属于成纤维生长因子家族,主要以神经生长因子的身份参与神经发育和神经保护,在免疫方面,FGF12被发现可以影响巨噬细胞的极化状态,进而调节炎症反应[24]。SEMA7A是一种重要的细胞黏附蛋白,属于半胱天冬酶家族。有研究报道,它能够通过激活巨噬细胞和小胶质细胞,促进炎症细胞的招募和激活,在脑免疫调节中起重要作用[25]。可见,上述筛选出的免疫相关核心基因在调节免疫系统反应中展现出重要性,它们潜在的生物学功能和调控机制,将有可能推动CCH认知功能障碍研究的新方向。
本研究进一步利用LASSO回归从以上9个差异有统计学意义的免疫相关核心基因中筛选出具有非零系数的关键免疫相关基因,分别为RASGRP1和BPHL。进一步分析发现,在CCH大鼠的脑皮层组织中,与Sham组织相比,RASGRP1的表达水平较高,而BPHL的表达水平较低。我们又基于GSE199508数据集将下调的差异miRNA与RASGRP1相关miRNA取交集,将上调的差异miRNA与BPHL相关miRNA取交集,最终筛选出调控RASGRP1的miR-216b-5p为关键免疫相关miRNA。因此,我们推测miR-216b-5p/RASGRP1信号通路可能在CCH损伤中发挥了重要作用。
已经有很多研究表明,RASGRP1作为免疫基因,在调节免疫反应中起着重要作用[26-27]。然而,它在神经系统中的功能和作用仍在被不断探索中。本研究表明RASGRP1在CCH大鼠的脑皮层组织中表达水平较高。这一发现提示RASGRP1可能在脑血管系统中具有一定的功能,尤其是脑血流受损的条件下。结合先前的研究,关于RASGRP1在海马和杏仁核中通过Ras信号通路参与控制突触功能的发现[28],我们可以推测,在脑皮层组织中高表达的RASGRP1可能也与突触功能相关,并可能涉及到Ras信号通路在神经系统中的调节作用,从而影响脑细胞的信号传导和突触可塑性。经进一步检索,调控RASGRP1的miR-216b-5p在癌症治疗中的作用已有报道[29-30],但在神经系统中的功能仍处于探索阶段,有待进一步的深入探究。
综上所述,本研究仅利用生物信息学方法研究CCH靶点,并未进行后续的实验验证,在后续研究中,可以进行进一步的生物学论证为CCH的免疫治疗提供实验依据。
重要声明
利益冲突声明:本文全体作者均声明不存在利益冲突。
作者贡献声明:赵梅和陈鑫负责文章的整体构思和设计,负责文章审阅和修改;赵梅,薛艳鹏,田青青和杨赫负责文章涉及工作的具体实施,包括数据获取与分析、结果整理与作图、文章撰写、投稿及修稿;蒋晴和于孟凡参与了文献的检索和整理,协助进行生信数据分析和作图。
Funding Statement
海南省自然科学基金(高层次人项目)(822RC872)
Contributor Information
梅 赵 (Mei ZHAO), Email: hrb_xiaozhaomei@163.com.
鑫 陈 (Xin CHEN), Email: chenxin_tracy@hrbmu.edu.cn.
References
- 1.Jonker M, Nooij F J The internal image-like anti-idiotypic response to a CD3-specific monoclonal antibody in primates is dependent on the T cell-binding properties of the injected antibody. Eur J Immunol. 1987;17(10):1519–1522. doi: 10.1002/eji.1830171022. [DOI] [PubMed] [Google Scholar]
- 2.Salminen A Hypoperfusion is a potential inducer of immunosuppressive network in Alzheimer's disease. Neurochem Int. 2021;142:104919. doi: 10.1016/j.neuint.2020.104919. [DOI] [PubMed] [Google Scholar]
- 3.Mao M, Xu Y, Zhang X Y, et al MicroRNA-195 prevents hippocampal microglial/macrophage polarization towards the M1 phenotype induced by chronic brain hypoperfusion through regulating CX3CL1/CX3CR1 signaling. J Neuroinflammation. 2020;17(1):244. doi: 10.1186/s12974-020-01919-w. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Li M, Meng N, Guo X, et al Dl-3-n-butylphthalide promotes remyelination and suppresses inflammation by regulating AMPK/SIRT1 and STAT3/NF-κB signaling in chronic cerebral hypoperfusion. Front Aging Neurosci. 2020;12:137. doi: 10.3389/fnagi.2020.00137. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.Chen X, Holtzman D M Emerging roles of innate and adaptive immunity in Alzheimer’s disease. Immunity. 2022;55(12):2236–2254. doi: 10.1016/j.immuni.2022.10.016. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Tukacs V, Mittli D, Hunyadi-Gulyás É, et al Chronic cerebral hypoperfusion-induced disturbed proteostasis of mitochondria and MAM is reflected in the CSF of rats by proteomic analysis. Mol Neurobiol. 2023;60(6):3158–3174. doi: 10.1007/s12035-023-03215-z. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Kim M, Kim B, Kim J I, et al Mumefural improves recognition memory and alters ERK-CREB-BDNF signaling in a mouse model of chronic cerebral hypoperfusion. Nutrients. 2023;15(14):3271. doi: 10.3390/nu15143271. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Warde-Farley D, Donaldson S L, Comes O, et al The GeneMANIA prediction server: biological network integration for gene prioritization and predicting gene function. Nucleic Acids Res. 2010;38:W214–W220. doi: 10.1093/nar/gkq537. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Hainsworth A H, Allan S M, Boltze J, et al Translational models for vascular cognitive impairment: a review including larger species. BMC Med. 2017;15(1):16. doi: 10.1186/s12916-017-0793-9. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Chen Y, Liu Q, Liu J, et al Revealing the modular similarities and differences among Alzheimer’s disease, vascular dementia, and Parkinson’s disease in genomic networks. Neuromolecular Med. 2022;24(2):125–138. doi: 10.1007/s12017-021-08670-2. [DOI] [PubMed] [Google Scholar]
- 11.Sun Z, Gao C, Gao D, et al Reduction in pericyte coverage leads to blood–brain barrier dysfunction via endothelial transcytosis following chronic cerebral hypoperfusion. Fluids Barriers CNS. 2021;18(1):21. doi: 10.1186/s12987-021-00255-2. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Farkas E, Luiten P G M, Bari F Permanent, bilateral common carotid artery occlusion in the rat: a model for chronic cerebral hypoperfusion-related neurodegenerative diseases. Brain Res Rev. 2007;54(1):162–180. doi: 10.1016/j.brainresrev.2007.01.003. [DOI] [PubMed] [Google Scholar]
- 13.Lee J, Lee J, Song M, et al NXP031 improves cognitive impairment in a chronic cerebral hypoperfusion-induced vascular dementia rat model through Nrf2 Signaling. Int J Mol Sci. 2021;22(12):6285. doi: 10.3390/ijms22126285. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Qu C, Song H, Shen J, et al Mfsd2a reverses spatial learning and memory impairment caused by chronic cerebral hypoperfusion via protection of the blood–brain barrier. Front Neurosci. 2020;14:461. doi: 10.3389/fnins.2020.00461. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Procter T V, Williams A, Montagne A Interplay between brain pericytes and endothelial cells in dementia. Am J Pathol. 2021;191(11):1917–1931. doi: 10.1016/j.ajpath.2021.07.003. [DOI] [PubMed] [Google Scholar]
- 16.Zhang D, Cao Y, Mu J, et al Inflammatory biomarkers and cerebral small vessel disease: a community-based cohort study. Stroke Vasc Neurol. 2022;7(4):302–309. doi: 10.1136/svn-2021-001102. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Xu J J, Guo S, Xue R, et al Adalimumab ameliorates memory impairments and neuroinflammation in chronic cerebral hypoperfusion rats. Aging (Albany NY) 2021;13(10):14001–14014. doi: 10.18632/aging.203009. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Baik S H, Selvaraji S, Fann D Y, et al Hippocampal transcriptome profiling reveals common disease pathways in chronic hypoperfusion and aging. Aging (Albany NY) 2021;13(11):14651–14674. doi: 10.18632/aging.203123. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Wang Y H, Cheng C, Zuo X Z, et al Inhibition of A2AR gene methylation alleviates white matter lesions in chronic cerebral hypoperfusion rats. Eur Rev Med Pharmacol Sci. 2022;26(8):2702–2711. doi: 10.26355/eurrev_202204_28600. [DOI] [PubMed] [Google Scholar]
- 20.Poh L, Fann D Y, Wong P, et al AIM2 inflammasome mediates hallmark neuropathological alterations and cognitive impairment in a mouse model of vascular dementia. Mol Psychiatry. 2021;26(8):4544–4560. doi: 10.1038/s41380-020-00971-5. [DOI] [PubMed] [Google Scholar]
- 21.Zhang Z, Guo Z, Jin P, et al Transcriptome profiling of hippocampus after cerebral hypoperfusion in mice. J Mol Neurosci. 2023;73(6):423–436. doi: 10.1007/s12031-023-02123-0. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22.Li K, Du Y, Li L, et al Bioinformatics approaches for anti-cancer drug discovery. Curr Drug Targets. 2020;21(1):3–17. doi: 10.2174/1389450120666190923162203. [DOI] [PubMed] [Google Scholar]
- 23.Kortum R L, Rouquette-Jazdanian A K, Samelson L E Ras and extracellular signal-regulated kinase signaling in thymocytes and T cells. Trends Immunol. 2013;34(6):259–268. doi: 10.1016/j.it.2013.02.004. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.Li S, Zhou B, Xue M, et al Macrophage-specific FGF12 promotes liver fibrosis progression in mice. Hepatology. 2023;77(3):816–833. doi: 10.1002/hep.32640. [DOI] [PubMed] [Google Scholar]
- 25.Deng J, Xu T, Yang J, et al Sema7A, a brain immune regulator, regulates seizure activity in PTZ-kindled epileptic rats. CNS Neurosci Ther. 2020;26(1):101–116. doi: 10.1111/cns.13181. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.Somekh I, Marquardt B, Liu Y, et al Correction to: novel mutations in RASGRP1 are associated with immunodeficiency, immune dysregulation, and EBV-induced lymphoma. Clin Immunol. 2018;38(6):711. doi: 10.1007/s10875-018-0541-8. [DOI] [PubMed] [Google Scholar]
- 27.Baars M J D, Douma T, Simeonov D R, et al Dysregulated RASGRP1 expression through RUNX1 mediated transcription promotes autoimmunity. Eur J Immunol. 2021;51(2):471–482. doi: 10.1002/eji.201948451. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 28.Pierret P, Vallée A, Mechawar N, et al Cellular and subcellular localization of Ras guanyl nucleotide-releasing protein in the rat hippocampus. Neuroscience. 2001;108(3):381–390. doi: 10.1016/S0306-4522(01)00429-8. [DOI] [PubMed] [Google Scholar]
- 29.Felix T F, Lopez Lapa R M, de Carvalho M, et al MicroRNA modulated networks of adaptive and innate immune response in pancreatic ductal adenocarcinoma. PLoS One. 2019;14(5):e0217421. doi: 10.1371/journal.pone.0217421. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 30.Qi Y, Lai Y L, Shen P C, et al Identification and validation of a miRNA-based prognostic signature for cervical cancer through an integrated bioinformatics approach. Sci Rep. 2020;10(1):22270. doi: 10.1038/s41598-020-79337-4. [DOI] [PMC free article] [PubMed] [Google Scholar]