Abstract
目的
探讨谷胱甘肽转移酶(GST)对变异链球菌环境适应能力的影响。
方法
构建变异链球菌GST基因敲除株ΔgsT。通过转录组测序分析变异链球菌UA159野生型与其GST基因敲除株ΔgsT的基因表达差异,酸胁迫实验、过氧化氢胁迫实验分析耐酸能力、抗氧化应激能力,营养限制条件下进行生长动力学分析,荧光原位杂交实验分析在菌斑生物膜的种间竞争能力。
结果
与变异链球菌野生型UA159相比,敲除株ΔgsT中198个基因显著差异表达,富集于代谢、应激响应及能量稳态相关通路。耐酸实验中,敲除株ΔgsT存活率显著降低(P<0.01)。过氧化氢处理15 min后,敲除株ΔgsT存活率为38.12%(野生型为71.75%)。营养限制下,敲除株ΔgsT最大比生长速率及稳定期生物量显著降低(P<0.05)。生物膜竞争实验中,变异链球菌敲除株ΔgsT在混合生物膜中所占的比例(8.50%)较野生型(16.89%)显著下降(P<0.05)。
结论
GST可通过调控代谢和应激响应相关基因,显著增强变异链球菌的耐酸、抗氧化及营养胁迫适应能力。
Keywords: 谷胱甘肽转移酶, 变异链球菌, 耐酸性, 氧化应激, 种间竞争, 基因表达调控, 代谢重编程
Abstract
Objective
This study aims to investigate the impact of glutathione S-transferase (GST) on the environmental adaptability of Streptococcus mutans (S. mutans).
Methods
A GST knockout strain ΔgsT was constructed. Transcriptomic sequencing was performed to analyze the gene expression differences between the wild-type S. mutans UA159 and its GST knockout strain ΔgsT. Comprehensive functional assessments, including acid tolerance assays, hydrogen peroxide challenge assays, nutrient limitation growth assays, and fluorescence in situ hybridization, were conducted to evaluate the acid tolerance, antioxidant stress resistance, growth kinetics, and interspecies competitive ability of ΔgsT within plaque biofilms.
Results
Compared with the wild-type S. mutans, 198 genes in ΔgsT were significantly differentially expressed and enriched in pathways related to metabolism, stress response, and energy homeostasis. The survival rate of ΔgsT in acid tolerance assays was markedly reduced (P<0.01). After 15 min of hydrogen peroxide challenge, the survival rate of ΔgsT decreased to 38.12% (wild type, 71.75%). Under nutrient-limiting conditions, ΔgsT exhibited a significantly lower final OD600 value than the wild-type strain (P<0.05). In the biofilm competition assays, the proportion of S. mutans ΔgsT in the mixed biofilm (8.50%) was significantly lower than that of the wild type (16.89%) (P<0.05).
Conclusion
GST enhances the acid resistance, oxidative stress tolerance, and nutrient adaptation of S. mutans by regulating metabolism-related and stress response-related genes.
Keywords: glutathione S-transferase, Streptococcus mutans, acid tolerance, oxidative stress, interspecies competition, gene expression regulation, metabolic reprogramming
变异链球菌(Streptococcus mutans)是目前公认的主要致龋菌,牙齿表面菌斑中的变异链球菌通过糖酵解代谢产酸导致牙釉质脱矿,并依赖低pH环境抑制共生菌(如血链球菌和戈登链球菌)的定植[1-3]。后者产生的过氧化氢对变异链球菌构成氧化胁迫,促使其进化出多重防御机制,包括合成超氧化物歧化酶、硫醇过氧化物酶及烟酰胺腺嘌呤二核苷酸氧化酶等,并通过外源性摄取谷胱甘肽(glutathione,GSH)增强抗氧化能力[4-5]。近年研究[6-10]发现,S-谷胱甘肽化作为可逆的翻译后修饰,通过调控蛋白功能参与应激适应,其缺失可显著降低变异链球菌的致龋性。谷胱甘肽转移酶(glutathione S-transferase,GST)作为催化GSH结合反应的关键酶,在细菌解毒和应激耐受中发挥重要作用[11-14],但在变异链球菌中的功能尚未明确。
本研究对GST基因敲除株ΔgsT进行转录组测序,并采用酸胁迫、过氧化氢胁迫、营养限制条件下生长动力学分析和荧光原位杂交(fluorescen-ce in situ hybridization,FISH)实验,探讨GST在代谢重编程和生态适应中的分子功能,为生态防龋策略提供新思路和新靶点。
1. 材料和方法
1.1. 实验菌株和生长条件
实验菌株:变异链球菌UA159(ATCC 700-610)、血链球菌(ATCC 10556)和戈登链球菌(DL1)。上述菌株均获自中国口腔微生物组数据库(Oral Microbiome Bank of China,OMBC)[15]。等位基因交换法[10]构建变异链球菌GST基因敲除株ΔgsT(基因位点:SMU_1296),通过一代测序扩增16S区域,与NCBI数据库比对,序列相似性为99.58%。
所有菌株均于37 ℃、5%CO₂的孵箱中,使用脑心浸液培养基(brain heart infusion,BHI)进行兼性厌氧培养。培养基按照说明书进行配制,高压灭菌后分装至无菌试管或培养皿备用。
1.2. 实验方法
1.2.1. 基因表达差异分析
将变异链球菌UA159野生型及其敲除株ΔgsT分别接种于BHI液体培养基中,于37 ℃、5%CO2条件下过夜活化。取30 mL对数生长期菌液(OD600=0.5)离心(10 000×g,4 ℃,10 min)收集菌体,pH 7.4无菌磷酸盐缓冲液(phosphate-buffered saline,PBS)洗涤2次以去除培养基残留。菌体沉淀经液氮速冻后,立即转移至-80 ℃超低温冰箱保存。
使用十六烷基三甲基溴化铵法(cetyltrimethylammonium bromide,CTAB)法[16]进行RNA提取,NanoDrop 2000分光光度计进行RNA纯度检测,琼脂糖凝胶电泳及Agilent 5300片段分析仪系统进行RNA完整性检测。委托上海美吉生物医药科技有限公司完成转录组测序,使用DESeq2 1.42.0软件进行表达量差异分析,与同源蛋白簇(clusters of orthologous groups,COG)数据库对比进行COG功能注释分析,使用Goatools 1.4.4软件GO富集分析,使用R脚本进行京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Geno-mes,KEGG)通路功能富集分析,参数均设置为Padjust<0.05,BH多重检验校正。采用Illumina NovaSeq 6000平台进行双端150 bp测序(PE150),每个菌株设置3个生物学重复。
1.2.2. 表型验证
1.2.2.1. 酸胁迫实验
参照Belli和Marquis[17]的方法,进行菌体预处理、酸性胁迫处理和存活率测定。
将变异链球菌UA159野生型及其敲除株ΔgsT分别接种于BHI液体培养基中,于37 ℃、5%CO₂条件下培养至对数生长期(OD600=0.5)。取5 mL菌液经离心(4 000×g,4 ℃,10 min)收集菌体沉淀,用预冷的0.1 mol/L、pH 7.0甘氨酸缓冲液洗涤2次以去除培养基残留。
将菌体重悬于常温5 mL的0.1 mol/L、pH 2.8甘氨酸缓冲液,分别孵育15、30、45 min。为精确控制酸暴露时间,孵育后立即加入预冷的磷酸盐缓冲液(phosphate buffered saline,PBS)终止反应。将处理后的菌液进行10倍梯度稀释(10-1至10-5),取10 μL接种于BHI琼脂平板,每个稀释度设置3个生物学重复。平板于37 ℃、5%CO₂条件下厌氧培养48 h后计数菌落形成单位(colony forming unit,CFU),以未处理组为对照组,计算存活率(%)。
1.2.2.2. 过氧化氢胁迫实验
细菌抗氧化应激能力评估参照Wen等[18]的方法,菌体预处理及存活率测定与上述耐酸实验相同。
将菌体重悬于5 mL含0.2%过氧化氢的0.1 mol/L、pH 7.0甘氨酸缓冲液,分别孵育15、30、45 min。加入过氧化氢酶(5 mg/mL)并混匀,静置5 min以彻底分解残留过氧化氢,终止反应。
1.2.2.3. 营养限制条件下生长动力学分析
将变异链球菌UA159野生型及其敲除株ΔgsT分别接种于BHI液体培养基中,于37 ℃、5%CO2条件下过夜活化并传代至对数生长期(OD600=0.5)。
将BHI培养基与无菌去离子水按1∶1体积比混合,配制为1/2浓度的BHI培养基;正常的BHI培养基作为对照。取对数期菌液,分别用正常浓度的BHI、1/2浓度的BHI培养基调整菌体密度至初始接种量(OD600=0.10)。将200 μL菌液分装至96孔板,每孔覆盖50 μL无菌矿物油以防止蒸发并维持微需氧环境。
使用BioTek LogPhase 600全自动微生物生长曲线分析仪,于37 ℃条件下连续监测OD600值,振荡频率为180 r/min,测量间隔为1 h,持续24 h。每个处理组设置3个生物学重复,每个生物学重复包含3个技术重复。采用GraphPad Prism 10.0软件计算各时间点OD600的均值±标准差,并通过四参数Logistic模型拟合生长曲线,计算最大比生长速率(μmax)及稳定期生物量(OD600_max)。
1.2.2.4. FISH实验
将血链球菌ATCC10556、戈登链球菌DL1、变异链球菌UA159野生型及其敲除株ΔgsT分别接种于BHI液体培养基,于37 ℃、5%CO2条件下过夜活化并传代至对数生长期(OD600=0.5)。
将变异链球菌UA159、敲除株ΔgsT分别与血链球菌和戈登链球菌按等比例(1∶1∶1)混合后,用含1%蔗糖的BHI培养基按1∶100稀释(终菌液浓度约1×105 CFU/mL),接种至无菌玻底培养皿(直径15 mm)。37 ℃兼性厌氧环境中静态培养24 h,以模拟口腔生物膜形成的自然条件,构建生物膜共培养体系。移除培养基后,用pH 7.4无菌PBS缓冲液轻柔洗涤生物膜3次,去除浮游菌体,加入预冷的4%多聚甲醛4 ℃下固定6 h。参照Klug等[19]的方法,采用物种特异性16S rRNA荧光探针进行杂交。
使用尼康N-SIM超分辨共聚焦显微镜,每组随机捕获至少3个视野采集Z-stack图像。通过NIS-Elements AR 5.21软件进行三维重构,并采用ImageJ 1.8.0定量各物种在生物膜中的空间占比。
1.3. 数据分析
采用SPSS 29.0和Prism 10.0软件对数据进行统计分析,组间差异比较采用Welch校正的非配对t检验,趋势分析采用泊松回归检验。P<0.05被认为差异具有统计学意义。
2. 结果
2.1. 基因表达差异分析
2.1.1. RNA提取检测结果
为确保转录组测序数据的可靠性,对变异链球菌野生型和敲除株ΔgsT的RNA样品进行了严格的质量控制。RNA质量和数量检测结果见表1。样品的总量为52.33~120.54 μg,符合Illumina高通量测序的起始量要求(≥1 μg)。纯度分析显示,OD260/280为2.10~2.12,OD260/230为2.57~2.61,表明样本无显著蛋白质或有机溶剂污染。完整性检测显示,RNA完整性数(RNA quality number,RQN)均≥9.5。结合琼脂糖凝胶电泳结果可见清晰的28S与18S核糖体RNA条带(图1),表明无基因组DNA残留或RNA降解迹象。
表 1.
RNA质量和数量检测结果
Tab 1 Assessment of RNA quality and quantity
| 样品名称 | 浓度/(ng/μL) | 总量/μg | OD260/280 | OD260/230 | RQN |
|---|---|---|---|---|---|
| 野生型1 | 1 586.50 | 79.33 | 2.10 | 2.61 | 10.00 |
| 野生型2 | 1 691.20 | 59.19 | 2.11 | 2.59 | 9.90 |
| 野生型3 | 1 774.90 | 88.75 | 2.12 | 2.59 | 10.00 |
| 敲除株ΔgsT1 | 1 492.00 | 52.22 | 2.11 | 2.60 | 10.00 |
| 敲除株ΔgsT2 | 1 855.90 | 64.96 | 2.11 | 2.57 | 10.00 |
| 敲除株ΔgsT3 | 1 418.10 | 120.54 | 2.11 | 2.59 | 9.50 |
图 1.

琼脂糖凝胶电泳结果
Fig 1 Agarose gel electrophoresis of total RNA
1~3:野生型;4~6:敲除株ΔgsT;M:Marker。
2.1.2. gsT缺失对基因表达的影响
基因表达量的差异火山图(图2)显示,与变异链球菌野生型UA159相比,敲除株ΔgsT中198个基因显著差异表达,其中,99个基因显著上调,99个基因显著下调,敲除后的基因表达变化呈现均衡的上下调趋势。此外,有1 709个基因未表现出显著差异,表明gsT缺失对整体基因表达的影响有限。
图 2.
基因表达量差异火山图
Fig 2 Volcano plot of differential expression of gene
从差异表达基因的数目(表2)可以看出,当差异倍数(fold change,FC)大于1.5时,上调与下调基因分别减少至22和34个;FC大于2时,上调基因仅保留8个(ptcA、SMU_921、SMU_922、SMU_923、tpx、add、SMU_1297、pheS),下调基因仅保留7个(SMU_151、SMU_152、SMU_498、SMU_499、SMU_RS04545、dprA、SMU_1905c)。该结果提示,随着筛选标准升高,差异基因数量显著减少(P<0.001)。
表 2.
差异表达的基因数目
Tab 2 Number of differentially expressed genes
| 调节类型 | FC>1.2 | FC>1.5 | FC>2 |
|---|---|---|---|
| 上调 | 99 | 22 | 8 |
| 下调 | 99 | 34 | 7 |
2.1.3. gsT缺失主要影响防御和代谢功能
COG功能注释分析显示,差异表达基因广泛分布于多个功能类别中,并主要集中在代谢相关功能类别中(图3)。下调基因主要集中在脂质代谢(COG类别I)、转录(COG类别K)、复制/重组/修复(COG类别L)、无机离子转运和代谢(COG类别P)、防御机制(COG类别V)和移动基因组(COG类别X)。上调基因主要集中在能量生产与转换(COG类别C)、氨基酸转运和代谢(COG类别E)、碳水化合物转运和代谢(COG类别G)、翻译/核糖体结构/生物合成(COG类别J)、翻译后修饰/蛋白质周转/分子伴侣(COG类别O)和信号转导机制(COG类别T)。
图 3.
基因集COG分类统计柱状图
Fig 3 Bar chart of COG classification statistics for gene sets
GO富集分析气泡图(图4)显示,差异基因主要富集于对细菌的防御反应及响应、对外部生物刺激的响应、防御反应等条目,且绝大多数为下调基因,提示gsT缺失可抑制宿主防御机制,削弱菌株对生物胁迫的应答能力。这一结果与COG注释分析中的信号转导机制(COG类别T)和防御机制(COG类别V)相符。
图 4.
GO富集分析气泡图
Fig 4 Bubble plot of GO enrichment analysis
KEGG富集分析气泡图(图5)显示,差异基因富集于硫辛酸代谢、三羧酸循环和丙酮酸代谢,且相关基因均上调。这一结果与COG注释分析中的能量生产与转换(COG类别C)、碳水化合物转运和代谢(COG类别G)结果高度一致,表明gsT缺失后变异链球菌可能通过激活糖酵解-三羧酸循环-氧化磷酸化轴提升能量代谢效率。
图 5.
KEGG富集分析气泡图
Fig 5 Bubble plot of KEGG enrichment analysis
2.2. 表型验证
2.2.1. gsT缺失变异链球菌耐酸能力降低
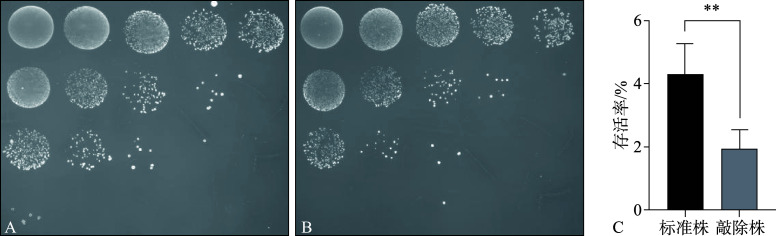
gsT缺失对耐酸能力的影响见图6。与标准株UA159相比,敲除株ΔgsT在pH2.8的甘氨酸缓冲液中的存活能力明显降低。15 min处理组标准株的平均存活率为4.31%,而敲除株的平均存活率仅为1.93%,存活率显著降低(P<0.01)。30 min处理组虽然无法计算存活率,但稀释100倍后标准株仍形成了数量可观的菌落,而敲除株仅形成了十余个菌落。45 min处理组同样无法计算存活率,但稀释10倍后标准株仍有少量菌落形成,而敲除株则无菌落形成。
图 6.
gsT缺失对耐酸能力的影响
Fig 6 Effect of gsT deficiency on acid tolerance
A、B分别为稀释后接种于培养基平板的标准株和敲除株,从上到下依次为对照组、15 min处理组、30 min处理组和45 min处理组,从左到右稀释倍数从10开始依次递增;C为15 min处理组的存活率,**P<0.01。
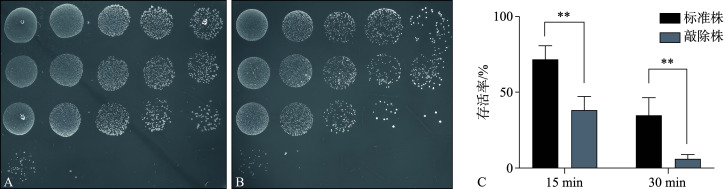
2.2.2. gsT缺失抗氧化应激能力降低
gsT缺失对抗氧化应激能力的影响见图7。与标准株UA159相比,敲除株ΔgsT面对氧化应激时存活能力明显降低。15 min处理组标准株的平均存活率为71.75%,敲除株的平均存活率为38.12%;30 min处理组标准株的平均存活率为34.73%,敲除株的平均存活率为5.88%。45 min处理组虽然无法计算存活率,但可以看到在稀释10倍后,标准株比敲除株有更多、形态更好的菌落形成。
图 7.
gsT缺失对抗氧化应激能力的影响
Fig 7 Effect of gsT deficiency on oxidative stress resistance
A、B分别为稀释后接种于培养基平板的标准株和敲除株,从上到下依次为对照组、15 min处理组、30 min处理组和45min处理组,从左到右稀释倍数从10开始依次递增;C为15 min处理组及30 min处理组的存活率,**P<0.01。
2.2.3. gsT缺失生长速率降低且对营养限制更敏感
gsT缺失对生长的影响见图8。
图 8.
gsT缺失对生长的影响
Fig 8 Effect of gsT deficiency on growth
当使用正常浓度BHI培养基时,标准株呈现典型的生长周期,对数生长期(3~8 h)的OD600迅速上升,μmax为0.70 h-1,OD600_max为0.93。敲除株在经历约3 h的延迟期后进入对数生长期,OD600_max同样达到0.93,但μmax仅为0.35 h-1,明显低于标准株,相应的对数生长期也明显延长。
在营养限制环境(1/2浓度BHI培养基)下,标准株的生长周期无明显改变,生长速率降低(μmax为0.32 h-1),OD600_max为0.32。敲除株的生长受到进一步抑制,与正常营养条件下一样对数生长期延长,μmax仅为0.16 h-1,OD600_max为0.28,与标准株相比差异有统计学意义(P<0.05),表明gsT的缺失导致变异链球菌对营养缺乏更加敏感。
2.2.4. gsT缺失种间竞争能力降低
gsT缺失种间竞争结果见图9,可见敲除株形成的混合生物膜结构较为稀疏,且敲除株在其中占比较少。相对丰度定量分析,敲除株在混合生物膜中占比(8.50%)较标准株(16.89%)下降(P<0.05),而血链球菌占比(37.16)较标准株(30.92%)上升,戈登链球菌占比(54.34%)也较标准株(52.19%)上升。
图 9.
gsT缺失对种间竞争能力的影响
Fig 9 Effect on interspecific competitive ability
A、B分别为变异链球菌标准株、敲除株与血链球菌及戈登链球菌共培养的混合生物膜荧光染色,C为混合生物膜中各菌种相对丰度。
3. 讨论
本研究通过整合转录组测序与多维度表型验证,较为系统揭示了GST在变异链球菌代谢网络调控中的核心作用。研究结果表明,gsT基因缺失不仅显著影响菌株的氧化应激响应能力,更通过重塑中心代谢通路引发全局性生理重构,这一发现为深入解析口腔致病菌的环境适应性机制提供了新的理论依据。
基于转录组学分析,gsT缺失株呈现显著的代谢重编程特征。值得注意的是,脂质代谢相关基因的普遍下调提示细胞膜生物合成可能受阻,导致膜流动性降低及质子通透性改变,进而影响胞内pH稳态维持能力。同时,无机离子转运系统的表达抑制可能加剧酸性环境下的质子失衡现象,这一推论在后续耐酸实验中得到表型验证。与此同时,能量代谢相关通路呈现显著激活特征:1)碳水化合物转运与代谢基因的同步上调,与胞外多糖合成基因表达增强形成协同效应;2)三羧酸循环相关基因的转录水平升高,提示丙酮酸脱氢酶复合体活性增强可能驱动乙酰辅酶A通量增加;3)氧化磷酸化系统的激活表明能量转换效率提升。这种“代谢增强-结构妥协”的适应性策略与真核生物Warburg效应具有表型相似性[20],具体调控机制可能涉及原核生物特有的双组分信号系统,在后续的机制研究中可通过实时荧光定量多聚核苷酸链式反应、靶向代谢组学和蛋白质组学进行验证。
在基因组稳定性维持方面,gsT缺失导致同源重组、错配修复等关键DNA修复基因表达下调,同时转座子及原噬菌体相关基因转录抑制,提示基因组可塑性可能降低。转座子对真核基因组稳定性有着重要作用,在细菌中与接触依赖性信号转导密切相关[21-22]。研究[23-25]表明,原噬菌体是细菌水平基因转移的重要载体,可增强细菌适应性和毒力,在细菌进化中发挥重要作用。有趣的是,核糖体生物合成基因的显著上调与氨基酸转运系统激活形成代谢补偿网络,推测这种翻译效率提升可能是通过增加应激蛋白合成来缓解氧化损伤,在未来研究中可结合分子荧光追踪技术动态解析氧化损伤条件下DNA修复复合体的组装过程,进一步阐明其在基因组稳定性中的作用机制。
本研究发现的GSH代谢网络调控机制与多物种研究形成理论呼应。在真核系统中,Han等[26]证实GSH合成与谷氨酰胺代谢偶联可增强肿瘤细胞氧化应激耐受;Wang等[27]在弗朗西斯氏菌中发现的保守性GSH利用途径则提示该分子在微生物环境适应中的普适性作用。值得注意的是,本研究发现GSH系统对耐酸能力的特异性调控,这一新机制在革兰氏阳性菌中尚未见报道。结合Wu等[28]对Gstm2调控线粒体功能的发现,提示GSH相关酶系可能通过保守的氧化还原调控模块影响不同生物体系的能量代谢。
综上,本研究阐释了gsT在变异链球菌代谢网络中的枢纽作用,揭示其通过协调氧化防御与能量重分配维持环境适应性的新机制。这些发现不仅为口腔微生物生态学研究提供新的理论框架,更为开发靶向GSH代谢通路的防龋策略奠定基础。
基金资助
国家自然科学基金(82170947)
National Natural Science Foundation of China (82170947)
利益冲突声明
作者声明本文无利益冲突。
参考文献
- 1. Banas JA. Virulence properties of Streptococcus mutans [J]. Front Biosci, 2004, 9: 1267-1277. [DOI] [PubMed] [Google Scholar]
- 2. Featherstone JD. Dental caries: a dynamic disease process[J]. Aust Dent J, 2008, 53(3): 286-291. [DOI] [PubMed] [Google Scholar]
- 3. Xu YF, Itzek A, Kreth J. Comparison of genes required for H2O2 resistance in Streptococcus gordonii and Streptococcus sanguinis [J]. Microbiology, 2014, 160(12): 2627-2638. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4. Sherrill C, Fahey RC. Import and metabolism of glutathione by Streptococcus mutans [J]. J Bacteriol, 1998, 180(6): 1454-1459. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5. Vergauwen B, Verstraete K, Senadheera DB, et al. Mole-cular and structural basis of glutathione import in Gram-positive bacteria via GshT and the cystine ABC impor-ter TcyBC of Streptococcus mutans [J]. Mol Microbiol, 2013, 89(2): 288-303. [DOI] [PubMed] [Google Scholar]
- 6. Zhang J, Ye ZW, Singh S, et al. An evolving understan-ding of the S-glutathionylation cycle in pathways of redox regulation[J]. Free Radic Biol Med, 2018, 120: 204-216. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7. Mailloux RJ. Protein S-glutathionylation reactions as a global inhibitor of cell metabolism for the desensitization of hydrogen peroxide signals[J]. Redox Biol, 2020, 32: 101472. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8. Bischoff ME, Shamsaei B, Yang JC, et al. Copper drives remodeling of metabolic state and progression of clear cell renal cell carcinoma[J]. Cancer Discov, 2025, 15(2): 401-426. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9. Romig M, Eberwein M, Deobald D, et al. Reactivation and long-term stabilization of the [NiFe] Hox hydrogenase of Synechocystis sp. PCC6803 by glutathione after oxygen exposure[J]. J Biol Chem, 2025, 301(1): 108086. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10. Li ZY, Zhang CZ, Li C, et al. S-glutathionylation proteome profiling reveals a crucial role of a thioredoxin-like protein in interspecies competition and cariogene-city of Streptococcus mutans [J]. PLoS Pathog, 2020, 16(7): e1008774. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11. Hayes JD, Flanagan JU, Jowsey IR. Glutathione transfe-rases[J]. Annu Rev Pharmacol Toxicol, 2005, 45: 51-88. [DOI] [PubMed] [Google Scholar]
- 12. Armstrong RN. Structure, catalytic mechanism, and evolution of the glutathione transferases[J]. Chem Res Toxicol, 1997, 10(1): 2-18. [DOI] [PubMed] [Google Scholar]
- 13. Zhang SS, Zhang C, Sun FJ, et al. Glutathione-S-transferase (GST) catalyzes the degradation of chlorimuron-ethyl by Klebsiella jilinsis 2N3[J]. Sci Total Environ, 2020, 729: 139075. [DOI] [PubMed] [Google Scholar]
- 14. Kammerscheit X, Chauvat F, Cassier-Chauvat C. From cyanobacteria to human, MAPEG-type glutathione-S-transferases operate in cell tolerance to heat, cold, and lipid peroxidation[J]. Front Microbiol, 2019, 10: 2248. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15. Peng X, Zhou XD, Xu X. The oral microbiome bank of China[J]. Int J Oral Sci, 2018, 10(2): 16. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16. Jones AS. The isolation of bacterial nucleic acids using cetyltrimethylammonium bromide (cetavlon)[J]. Biochim Biophys Acta, 1953, 10(4): 607-612. [DOI] [PubMed] [Google Scholar]
- 17. Belli WA, Marquis RE. Adaptation of Streptococcus mutans and Enterococcus hirae to acid stress in continuous culture[J]. Appl Environ Microbiol, 1991, 57(4): 1134-1138. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18. Wen ZT, Suntharaligham P, Cvitkovitch DG, et al. Trigger factor in Streptococcus mutans is involved in stress tolerance, competence development, and biofilm formation[J]. Infect Immun, 2005, 73(1): 219-225. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19. Klug B, Rodler C, Koller M, et al. Oral biofilm analysis of palatal expanders by fluorescence in situ hybridization and confocal laser scanning microscopy[J]. J Vis Exp, 2011(56): 2967. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20. Hsu PP, Sabatini DM. Cancer cell metabolism: warburg and beyond[J]. Cell, 2008, 134(5): 703-707. [DOI] [PubMed] [Google Scholar]
- 21. Sienski G, Dönertas D, Brennecke J. Transcriptional silencing of transposons by piwi and maelstrom and its impact on chromatin state and gene expression[J]. Cell, 2012, 151(5): 964-980. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22. Ocasio AB, Cotter PA. CDI/CDS system-encoding ge-nes of Burkholderia thailandensis are located in a mobile genetic element that defines a new class of transposon[J]. PLoS Genet, 2019, 15(1): e1007883. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23. Juhas M. Horizontal gene transfer in human pathogens[J]. Crit Rev Microbiol, 2015, 41(1): 101-108. [DOI] [PubMed] [Google Scholar]
- 24. Davies EV, Winstanley C, Fothergill JL, et al. The role of temperate bacteriophages in bacterial infection[J]. FEMS Microbiol Lett, 2016, 363(5): fnw015. [DOI] [PubMed] [Google Scholar]
- 25. Renard A, Diene SM, Courtier-Martinez L, et al. 12/111phiA prophage domestication is associated with autoaggregation and increased ability to produce biofilm in Streptococcus agalactiae [J]. Microorganisms, 2021, 9(6): 1112. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26. Han XL, Wang DY, Yang L, et al. Activation of polyamine catabolism promotes glutamine metabolism and creates a targetable vulnerability in lung cancer[J]. Proc Natl Acad Sci USA, 2024, 121(13): e2319429121. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27. Wang YX, Ledvina HE, Tower CA, et al. Discovery of a glutathione utilization pathway in Francisella that shows functional divergence between environmental and pathogenic species[J]. Cell Host Microbe, 2023, 31(8): 1359-1370.e7. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 28. Wu HY, Yao Z, Li HK, et al. Improving dermal fibroblast-to-epidermis communications and aging wound repair through extracellular vesicle-mediated delivery of Gstm2 mRNA[J]. J Nanobiotechnol, 2024, 22: 307. [DOI] [PMC free article] [PubMed] [Google Scholar]