Abstract
La structure cristallographique de l’εnzyme ARN polymérase II, libre ou en plein acte de transcription, révèle comment cette machine moléculaire utilise l’information contenue dans l’ADN génique pour fabriquer les molécules d’ARNm qui dirigeront ensuite la synthèse des protéines. La résolution de la structure atomique de cette enzyme centrale au processus d’expression génique aura des retombées importantes : elle permettra l’interprétation plus adéquate de certaines données biochimiques et génétiques et elle ouvrira la voie à la modélisation moléculaire de drogues ciblant la transcription.
La découverte de l’ARN polymérase II (ARN pol II), l’enzyme eucaryote qui transcrit les gènes codant pour les protéines, a représenté une étape majeure dans la compréhension du processus de transcription. Trente ans plus tard, la résolution de la structure tridimensionnelle de cette enzyme est très certainement une avancée considérable ouvrant la voie à de nouvelles perspectives tant sur le plan scientifique que thérapeutique.
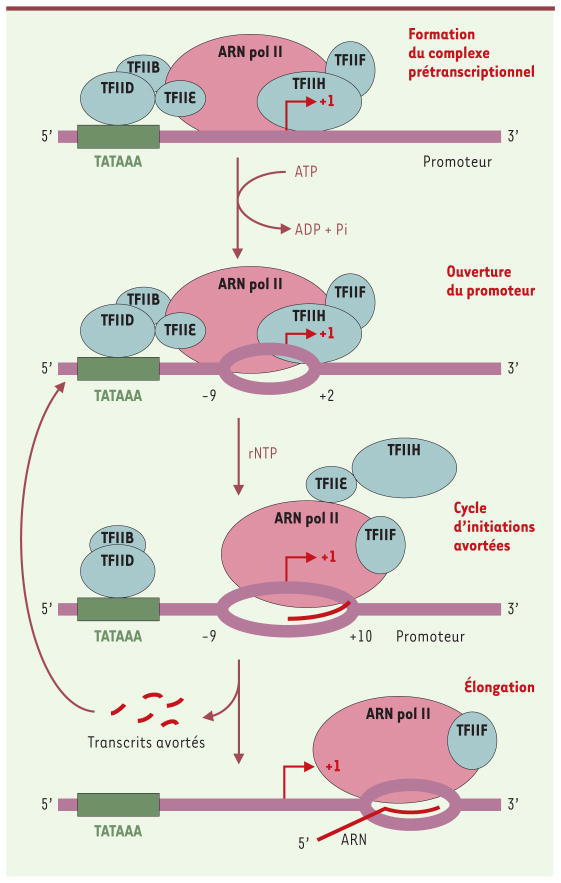
ds La transcription comporte différentes étapes conduisant à la synthèse de l’ARNm (Figure 1). L’ARN pol II doit d’abord se fixer au promoteur génique en compagnie des facteurs généraux de la transcription TFIIB, TFIID, TFIIE, TFIIF et TFIIH [1]. TFIIH catalyse ensuite l’ouverture de la double hélice d’ADN entre les positions −9 et +2 de part et d’autre du site d’initiation de la transcription (site +1) pour former la bulle transcriptionnelle [2]. Ceci rendra possible l’appariement des ribonucléotides (rNTP) libres au brin matrice et donc l’initiation de la transcription. La catalyse nécessite la présence de deux ions Mg2+ qui se retrouvent au site actif de la polymérase. Au fur et à mesure que la transcription progresse, la bulle s’étend en aval d’une paire de base de plus que la position du dernier rNTP ajouté [3]. Après l’ajout du quatrième rNTP à l’ARNm en formation, le complexe prétranscriptionnel subit un changement de conformation qui conduit à la stabilisation du complexe ouvert. TFIIH n’est alors plus nécessaire au maintien de la bulle de transcription. Jusqu’à l’atteinte de la position +10, l’ARN pol II entre dans un cycle d’initiations avortées (abortive initiation) puisque la polymérase peut s’arrêter à tout moment, relâcher l’ARNm en formation, reculer sur la matrice d’ADN pour repositionner son centre catalytique au site d’initiation et entreprendre un nouveau cycle. À la position +11, une autre transition importante referme abruptement le promoteur entre −9 et +2, et le complexe entre alors dans le mode de l’élongation productive. Lors de la transition entre l’initiation et l’élongation, les facteurs généraux de transcription, à l’exception de TFIIF, sont progressivement relâchés et la polymérase peut alors s’associer aux facteurs d’élongation. Ces derniers préviennent les pauses et les arrêts de la polymérase durant la synthèse de l’ARNm [4]. Au niveau du complexe d’élongation, un hybride ADN-ARN de 8 à 9 nucléotides se forme au sein de la bulle qui reste ouverte entre −9 et +2 par rapport au site +1. Il faut souligner que, pendant l’élongation, ce que l’on appelle le site +1 correspond au registre, c’est-à-dire la position du dernier rNTP ajouté dans le complexe en élongation. Les mécanismes de terminaison demeurent peu connus chez les eucaryotes, mais impliqueraient la présence de sites de pause en aval du signal de polyadénylation.
Figure 1. Étapes de la transcription par l’ARN polymérase II.
L’ARN polymérase II (ARN pol II) et les facteurs généraux de transcription s’assemblent d’abord sur le promoteur, pour former le complexe prétranscriptionnel. Puis, TFIIH catalyse l’ouverture du promoteur entre −9 et +2 par rapport au site d’initiation pour former la bulle transcriptionnelle, qui s’étend en aval au fur et à mesure que la transcription progresse. Jusqu’à la position +10, l’ARN pol II effectue un cycle d’initiations avortées. A la position +11, le promoteur se referme entre −9 et +2 et le complexe entre alors dans un mode d’élongation productive. La bulle transcriptionnelle reste alors ouverte entre −9 et +2 par rapport à la position du dernier rNTP ajouté.
Les sous-unités de l’ARN polymérase II
Trois caractéristiques de l’ARN pol II sont conservées entre les espèces eucaryotes [5]. L’enzyme est toujours constituée de 10 à 12 sous-unités nommées Rbp1 à 12. Les deux plus grandes sous-unités, qui ont des poids moléculaires approximatifs de 220 et 140 kDa, sont respectivement homologues, en terme de séquence et de fonction, aux sous-unités β’ et β de l’ARN polymérase bactérienne. Cinq sous-unités de l’ARN pol II, entre 7 et 28 kDa, sont aussi des sous-unités des ARN pol I (synthèse des ARNr) et III (synthèse des ARNt et ARNr 5S). Les autres sous-unités non-communes aux trois ARN polymérases eucaryotes sont tout de même très conservées.
Différentes études génétiques et biochimiques ont permis d’attribuer des rôles aux sous-unités de l’ARN pol II. Par exemple, les sous-unités Rpb1 et Rpb2 forment le centre catalytique de l’enzyme [5]. Rpb9 est impliquée dans l’élongation à travers les sites d’arrêts [6]. Rpb3 et Rpb11 présentent des homologies avec le domaine N-terminal de la sous-unité α de l’ARN polymérase de Escherichia coli, impliquée dans l’assemblage de l’enzyme. Les sous-unités Rpb4 et Rpb7 forment un sous-complexe qui peut se dissocier du reste de l’enzyme. Contrairement à Rpb7, Rpb4 n’est pas essentielle à la viabilité cellulaire. Cependant, à température élevée, Rpb4 devient nécessaire afin de maintenir Rpb7 associée à l’ARN pol II [7].
Bien que des rôles généraux aient été attribués à certaines sous-unités, leur implication précise dans les différentes étapes de la transcription restait floue jusqu’à la détermination récente par Roger Kornberg et ses collaborateurs de la structure de l’ARN pol II à une résolution de 2,8 Å [8] et de la structure de l’enzyme en élongation à une résolution de 3,3 Å [9]. Plusieurs structures ont été déterminées à plus faibles résolutions dans le passé, par cristallographie et par microscopie électronique [10–12]. Cependant, les structures récentes marquent un pas important dans la compréhension du mécanisme transcriptionnel en permettant de voir, à l’échelle atomique, les chaînes latérales des acides aminés de l’enzyme libre et, à une résolution légèrement moindre, les domaines de la polymérase interagissant avec les acides nucléiques lors de l’élongation.
La structure de l’ARN polymérase II montre un sillon et une pince mobile capables de retenir les acides nucléiques
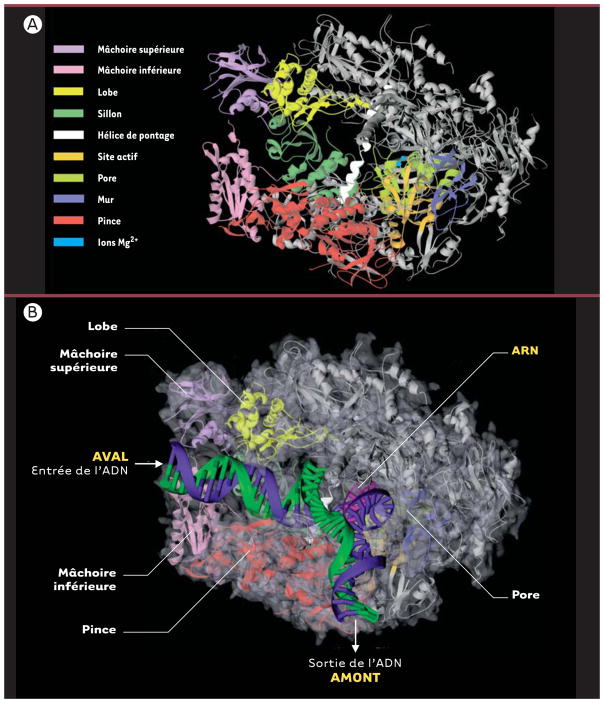
La structure à 2,8 Å a permis l’identification de domaines ou modules, qui ont été nommés selon leur localisation ou leur fonction présumée (Figure 2A et 2B). Ces différents domaines sont souvent composés de parties de différentes sous-unités. Une paire de mâchoires supérieure (upper jaw-partie de Rbp1 et Rbp9) et inférieure (lower jaw, Rpb5) bordent l’entrée du sillon (cleft, Rpb1) au fond duquel se retrouvent deux ions Mg2+ qui indiquent l’emplacement du site actif. Les acides nucléiques prendraient place dans ce sillon où sont localisés la majorité des charges positives de l’enzyme. Près du site actif, une hélice de Rpb1, l’hélice de pontage (bridge helix), traverse le sillon et rejoint Rpb2. Cette hélice serait impliquée dans la translocation de l’enzyme le long de l’ADN lors de l’élongation (voir plus loin). Près du site catalytique, se trouve un pore qui s’étend du fond du sillon jusqu’à l’arrière de l’enzyme. Celui-ci servirait à l’entrée des rNTP vers le site actif.
Figure 2. Structure cristallographique de l’ARN polymérase II.
A. Enzyme libre à une résolution de 2,8 Å. L’enzyme contient un sillon (Rpb1) bordé à son entrée par une paire de mâchoires, supérieure (partie de Rpb1 et Rpb9) et inférieure (Rbp5), et au fond duquel se trouvent deux ions Mg2+ qui indiquent l’emplacement du site actif. Près du site actif, l’hélice de pontage (Rpb1) traverse le sillon pour rejoindre Rpb2. Le pore localisé à l’arrière de l’enzyme permettrait l’entrée des rNTP et la sortie de l’extrémité 3’ libre de l’enzyme(Protein Data Bank Accession Code : 1i50).
B. Enzyme en élongation à une résolution de 3,3 Å. La pince formée de parties de Rbp1 et Rbp2 effectue une rotation de 30° qui maintient la matrice d’ADN et le transcrit au sein du complexe transcriptionnel. L’extrapolation du trajet probable des acides nucléiques au sein de l’enzyme montre que l’ADN double brin situé en aval fait contact avec la mâchoire inférieure puis passe entre le lobe de Rbp2 et une partie de la pince formée par Rbp1. Des flèches indiquent les sites d’entrée et de sortie de l’ADN (Protein Data Bank Accession Code : 1i6H).
Violet : mâchoire supérieure; rose : mâchoire inférieure; jaune : lobe; vert foncé : sillon; blanc : hélice de pontage; orange : site actif; vert pâle : pore; bleu : mur; rouge : pince; bleu pâle : ions Mg2+ (Protein Data Bank Accession Code : 1i50). Bleu : brin matrice; vert : brin codant; rose : ARN. Le code de couleur des domaines de l’enzyme est le même qu’en (A).
Les structures de la polymérase libre (Figure 2A) et en association avec le duplexe ADN-ARN (Figure 2B) diffèrent principalement au niveau de la position d’une structure mobile appelée pince ou clamp, formée de parties de Rpb1 et Rpb2. La pince peut effectuer une rotation de 30°, qui ferme le sillon et retient la matrice d’ADN et le transcrit au sein du complexe transcriptionnel afin de permettre une élongation efficace. À la base de la pince, une série de cinq boucles (switches) de Rpb1 coupleraient la rotation de la pince à la présence d’un hybride ADN-ARN dans le sillon. En effet, si les boucles 1 et 2 sont peu ordonnées et la boucle 3 désordonnée dans la structure de la polymérase libre, toutes trois deviennent ordonnées dans la structure en élongation.
Structure du complexe ARN polymérase II-ADN-ARN : une fenêtre ouverte sur le mécanisme d’élongation
La cristallographie de l’ARN pol II en élongation à 3,3 Å [9] a été réalisée en plaçant l’enzyme en présence d’une matrice d’ADN présentant une extrémité 3’ simple brin. Cette extrémité est reconnue par la polymérase qui peut alors déclencher la transcription sans l’aide des facteurs généraux de transcription. L’omission d’ATP dans la réaction entraîne l’arrêt de l’enzyme au premier T rencontré le long de la matrice d’ADN. Afin d’éliminer les enzymes inactives, la réaction a été purifiée à l’aide d’une colonne d’héparine, un polymère chargé négativement qui mime la structure des acides nucléiques. Les molécules de polymérase n’ayant pas le duplex ADN-ARN au niveau du sillon sont retenues par la colonne d’héparine, contrairement à celles en élongation.
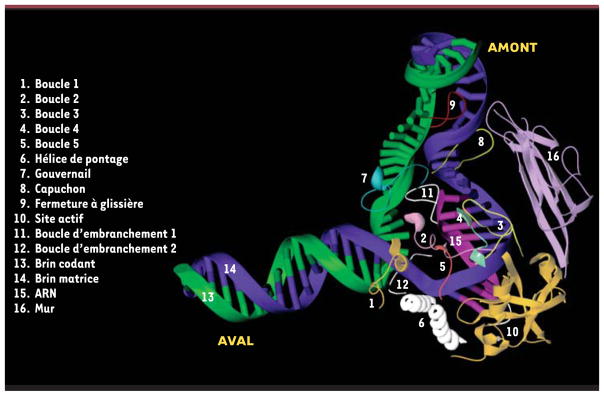
La structure à 3,3 Å élucidée par l’équipe de Kornberg montre le trajet d’un fragment d’ADN de 13 dNTP (de −9 à +4) appariés à 9 rNTP (Figures 2B et 3). Le reste de l’ADN au niveau de la figure 2B a été extrapolé afin de mieux visualiser le trajet probable des acides nucléiques au sein de l’enzyme. Ainsi, on peut voir que l’ADN double-brin situé en aval (+4 à +20) se retrouve dans le sillon entre la pince et Rpb2. Plus précisément, il fait d’abord contact avec la mâchoire inférieure et passe ensuite entre le lobe de Rpb2 et une partie de la pince formée par Rpb1.
Figure 3. Contacts ARN polymérase II-ADN-ARN dans la région de la bulle de transcription.
Plusieurs domaines des sous-unités Rpb1 et Rpb2 font contact avec l’ADN et l’ARN au niveau de la bulle transcriptionnelle en formant un réseau complexe d’interactions qui crée une zone hautement complémentaire à la liaison des acides nucléiques. Cette zone contribue à la stabilité du complexe transcriptionnel et donc à l’efficacité de l’élongation par la polymérase. L’hélice de pontage de Rpb1, qui peut prendre deux conformations alternatives, jouerait un rôle particulièrement important lors de la translocation de l’enzyme le long de la matrice d’ADN.
Au niveau de la bulle de transcription (Figure 3), l’extrémité aval du brin matrice, qui serait située aux environs de la position +4, passe au bas de la pince et contourne l’hélice de pontage. Celle-ci fait alors contact via des résidus conservés avec les nucléotides +1 et +2 et les positionne en une conformation particulière qui fait en sorte que le nucléotide codant est dirigé vers le site actif. Une boucle de Rpb2, la boucle d’embranchement 2 (fork loop 2) serait impliquée dans le maintien de l’extrémité aval de la bulle. En effet, le positionnement probable de la boucle d’embranchement indique qu’elle entrerait en collision avec le nucléotide à la position +3 si celui-ci était encore apparié au brin d’ADN complémentaire. En ce qui concerne l’extrémité amont de la bulle, la faible densité électronique détectée ne permet pas de déduire les contacts de l’ARN pol II avec l’ADN dans cette région. Cependant, deux boucles peu ordonnées, le capuchon (lid) et la fermeture à glissière (zipper) pourraient participer au maintien de l’extrémité amont de la bulle. La position de ces deux boucles, qui s’étendent à partir de la pince vers le site actif, leur permettrait en effet d’interagir avec cette portion de la bulle.
La longueur de l’hybride ADN-ARN est de 9 nucléotides, ce qui correspond à la longueur déterminée par les études biochimiques [13]. L’hybride prend place dans le sillon entre l’hélice de pontage et une structure de Rpb2 appelée le mur (wall). En tout, une quinzaine de régions protéiques appartenant à Rpb1 et Rpb2 sont impliquées dans des interactions avec l’hybride, dont les principales sont représentées sur la Figure 3. Ces contacts de la polymérase avec l’ADN et l’ARN dans la région de l’hybride constituent un réseau d’interactions complexe qui crée une zone hautement complémentaire à la liaison des acides nucléiques. Cette zone contribue à la stabilité du complexe transcriptionnel et donc à l’efficacité de la polymérase durant l’élongation. Cependant, bien que les interactions doivent permettre à l’enzyme de rester accrochée, celle-ci doit tout de même pouvoir se transloquer le long de la matrice d’ADN. Différentes observations permettent de résoudre ce paradoxe. Tout d’abord, les contacts protéiques sont faits avec le squelette sucre-phosphate et non avec les bases. Ensuite, la plupart des chaînes latérales interagissent avec deux groupements phosphates simultanément, ce qui abaisse la barrière énergétique de la translocation. Finalement, des résidus chargés positivement forment une coquille autour de l’hybride à une distance de 4 à 8 Å, qui le maintient dans le sillon sans bloquer le mouvement de l’enzyme.
En comparant la structure de l’ARN pol II avec celle de l’ARN polymérase bactérienne à 3,3 Å [14], Gnatt et al. ont déduit un rôle important pour l’hélice de pontage de Rpb1 dans le mécanisme de translocation. L’hélice de pontage pourrait avoir en effet deux conformations : soit droite, soit courbée. L’hélice courbée entrerait en collision avec le nucléotide en position +1, ce qui permettrait le déplacement des acides nucléiques lors de l’élongation de l’ARN.
La résolution de la structure de l’ARN pol II en élongation a aussi permis de mieux comprendre comment l’ajout du 11e rNTP dans l’ARNm provoque la transition de cycles d’initiations avortées vers une élongation productive [3]. La cristallographie indique que lors de cette étape, l’ARN se séparerait de l’hybride et entrerait dans un canal de sortie localisé à la surface de l’enzyme. Deux boucles s’étendant à partir de la pince, le gouvernail et le capuchon, joueraient un rôle dans la séparation de l’ARN de la matrice d’ADN (Figure 3). Le canal de sortie de l’ARNm est localisé tout près du dernier résidu ordonné de Rpb1, c’est-à-dire juste avant son domaine carboxy-terminal. Ce dernier est un domaine non structuré qui interagit avec des protéines impliquées dans la maturation des ARNm, comme des enzymes de coiffage, d’épissage et de polyadénylation [15, 16]. Ceci laisse croire que ces enzymes se retrouvent à proximité de l’ARNm à sa sortie du canal.
Conclusions
La cristallographie de l’ARN pol II libre à 2,8 Å et en élongation à 3,3 Å représente une grande avancée dans la compréhension du mécanisme de transcription. Plusieurs domaines de l’enzyme sont maintenant identifiés comme interagissant avec la bulle de transcription, l’hybride ADN-ARN, l’ARNm en formation, ce qui permet d’entrevoir leur rôle dans les différentes étapes du processus transcriptionnel. Sur le plan technique, la cristallographie d’une enzyme de 500 kDa avec un fragment d’ADN et d’ARN marque un pas très important. Dans cette lancée, les prochaines étapes seront sûrement la cristallographie de l’ARN pol II en complexe avec les facteurs généraux de transcription et/ou l’ADN du promoteur. De plus, la connaissance détaillée de la structure de l’enzyme facilitera l’interprétation des études biochimiques et génétiques. Finalement, il y a fort à parier que des retombées significatives pour la médecine verront le jour grâce à la modélisation, par homologie structurale, de petites molécules capables soit d’inhiber spécifiquement l’ARN pol II de certains micro-organismes nuisibles, soit de contrôler certains dérèglements transcriptionnels responsables de pathologies, comme certains cancers et certains dysfonctionnements génétiques.
Acknowledgments
Les auteurs tiennent à remercier Diane Bourque pour son excellente assistance dans la réalisation des figures. Le laboratoire de Benoit Coulombe est financé par les Instituts de recherche en santé du Canada, la Société de recherche sur le cancer inc. et Génome Québec. Benoit Coulombe est chercheur-boursier du Fonds de la recherche en santé du Québec et Marie-France Langelier détient une bourse du Conseil de recherche en sciences naturelles et génie du Canada et du Fonds pour la formation de chercheurs et l’aide à la recherche.
References
- 1.Orphanides G, Lagrange T, Reinberg D. The general transcription factors of RNA polymerase II. Genes Dev. 1996;10:2657–83. doi: 10.1101/gad.10.21.2657. [DOI] [PubMed] [Google Scholar]
- 2.Douziech M, Coin F, Chipoulet JM, et al. Mechanism of promoter melting by the xeroderma pigmentosum complementation group B helicase of transcription factor IIH revealed by protein-DNA photo-cross-linking. Mol Cel Biol. 2000;20:8168–77. doi: 10.1128/mcb.20.21.8168-8177.2000. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.Holstege FC, Fiedler U, Timmers HT. Three transitions in the RNA polymerase II transcription complex during initiation. EMBO J. 1997;16:7468–80. doi: 10.1093/emboj/16.24.7468. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Conaway JW, Shilatifard A, Dvir A, Conaway RC. Control of elongation by RNA polymerase II. Trends Biochem Sci. 2000;25:375–80. doi: 10.1016/s0968-0004(00)01615-7. [DOI] [PubMed] [Google Scholar]
- 5.Coulombe B, Burton ZF. DNA bending and wrapping around RNA polymerase: a «revolutionary» model describing transcriptional mechanisms. Microbiol Mol Biol Rev. 1999;63:457–78. doi: 10.1128/mmbr.63.2.457-478.1999. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Awrey DE, Weilbaecher RG, Hemming SA, Orlicky SM, Kane CM, Edwards AM. Transcription elongation through DNA arrest sites. J Biol Chem. 1997;272:14747–54. doi: 10.1074/jbc.272.23.14747. [DOI] [PubMed] [Google Scholar]
- 7.Maillet I, Buhler JM, Sentenac A, Labarre J. Rpb4p is necessary for RNA polymerase II activity at high temperature. J Biol Chem. 1999;274:22586–90. doi: 10.1074/jbc.274.32.22586. [DOI] [PubMed] [Google Scholar]
- 8.Cramer P, Bushnell DA, Kornberg RD. Structural basis of transcription: RNA polymerase II at 2. 8 A resolution. Science. 2001;292:1863–76. doi: 10.1126/science.1059493. [DOI] [PubMed] [Google Scholar]
- 9.Gnatt AL, Cramer P, Fu J, Bushnell DA, Kornberg RD. Structural basis of transcription: an RNA polymerase II elongation complex at 3. 3 A resolution. Science. 2001;292:1876–82. doi: 10.1126/science.1059495. [DOI] [PubMed] [Google Scholar]
- 10.Poglitsch CL, Meredith GD, Gnatt A, et al. Electron crystal structure of an RNA polymerase II transcription elongation complex. Cell. 1999;98:791–8. doi: 10.1016/s0092-8674(00)81513-5. [DOI] [PubMed] [Google Scholar]
- 11.Fu J, Gnatt AL, Bushnell DA, et al. Yeast RNA polymerase II at 5 A resolution. Cell. 1999;98:799–810. doi: 10.1016/s0092-8674(00)81514-7. [DOI] [PubMed] [Google Scholar]
- 12.Cramer P, Bushnell DA, Fu J, et al. Architecture of RNA polymerase II and implications for the transcription mechanism. Science. 2000;288:640–9. doi: 10.1126/science.288.5466.640. [DOI] [PubMed] [Google Scholar]
- 13.Nudler E, Mustaev A, Lukhtanov E, Goldfarb A. The RNA-DNA hybrid maintains the register of transcription by preventing backtracking of RNA polymerase. Cell. 1997;89:33–41. doi: 10.1016/s0092-8674(00)80180-4. [DOI] [PubMed] [Google Scholar]
- 14.Zhang G, Campbell EA, Minakhin L, Richter C, Severinov K, Darst SA. Crystal structure of Thermus aquaticus core RNA polymerase at 3. 3 A resolution. Cell. 1999;98:811–24. doi: 10.1016/s0092-8674(00)81515-9. [DOI] [PubMed] [Google Scholar]
- 15.Cho EJ, Takagi T, Moore CR, Buratowski S. mRNA capping enzyme is recruited to the transcription complex by phosphorylation of the RNA polymerase II carboxy-terminal domain. Genes Dev. 1997;11:3319–26. doi: 10.1101/gad.11.24.3319. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.McCracken S, Fong N, Rosonina E, et al. 5′-Capping enzymes are targeted to pre-mRNA by binding to the phosphorylated carboxy-terminal domain of RNA polymerase II. Genes Dev. 1997;11:3306–18. doi: 10.1101/gad.11.24.3306. [DOI] [PMC free article] [PubMed] [Google Scholar]