Abstract
Ein einfacher Ansatz nutzt einen erweiterten genetischen Code von Escherichia coli zur Biosynthese von Selenoproteinen mit zahlreichen Sec-Resten. Kürzlich wurden so genannte allo-tRNAs entdeckt. Diese verfügen über eine ungewöhnliche Struktur, sind genauso effiziente Serinakzeptoren wie die normale tRNASer aus E. coli und werden von der Aeromonas-salmonicida-Selenocysteinsynthase (SelA) von Ser-allo-tRNA zu Sec-allo-tRNA umgesetzt. Anschließend ermöglicht es Sec-allo-tRNA, fünf UAG-Stop-Codons auf der fdhF-mRNA für E.-coli-Formatdehydrogenase H als Sec zu translatieren und katalytisch aktive E.-coli-Formatdehydrogenase mit fünf Sec-Resten in E. coli zu produzieren. Weiterhin konnte gezeigt werden, dass sich in E. coli durch Kombination genetischer Varianten von allo-tRNA und SelA mit einem modifizierten Selenstoffwechsel das humane Selenoenzym GPx1 mit über 80% Sec-Einbaurate rekombinant produzieren lässt. Beide Beispiele belegen den Wert von allo-tRNAUTu als molekulare Plattform zur Entwicklung neuartiger Selenoproteine.
Zuschriften

’Allo ’Allo! Der Wert von allo-tRNAUTu als Plattform zur Entwicklung neuartiger Selenoproteine wird demonstriert. Weil Selenocystein (Sec, U) Proteinen neue chemische Eigenschaften verleiht, stoßen verbesserte Methoden zum gerichteten Einbau von Sec in Enzymen auf breites Interesse. Hier wird ein einfacher Ansatz beschrieben, der einen erweiterten genetischen Code von Escherichia coli zur Biosynthese von Selenoproteinen mit zahlreichen Sec-Resten nutzt.
Selenocystein ist ein faszinierender Baustein rekombinanter Proteine:[1] Im Vergleich zu Cystein (Cys) ist Sec reaktiver, widerstandsfähiger gegen irreversible Überoxidation,[2] chemisch modifizierbar[3] und ermöglicht in Proteinen die Bildung von Diselenidbrücken, die stabiler als Disulfidbrücken sind.[4] Über Dehydroalaninintermediate können Sec-Reste darüber hinaus in Proteinen chemisch zu verschiedenen Aminosäureseitenketten umgewandelt werden.[3a, 5] Die neuesten Methoden zur Erweiterung des genetischen Codes umfassen unter anderem In-vivo-Techniken zum ortsspezifischen, gerichteten, UAG-abhängigen Sec-Einbau in rekombinante Proteine unter Zuhilfenahme von Elongationsfaktor EF-Tu und sind unabhängig vom Sec-spezifischem Elongationsfaktor SelB sowie dem Sec-Insertions-Sequenz(SECIS)-Motiv.[6] Der Wildtyp von tRNASec schließt die Erkennung durch EF-Tu aus,[7] jedoch konnte tRNASec aus E. coli durch Mutationen in Akkzeptorarm und T-Stamm kompatibel zu EF-Tu gemacht werden.[6c, 7] Dies ermöglichte anschließend die Produktion bakterieller und humaner Selenoproteine in E. coli.[6a–d, 8]
Sec-tRNASec wird in Bakterien über zwei Stufen synthetisiert: Die Seryl-tRNA-Synthetase (SerRS) belädt tRNASec mit Serin (Ser), und SelA katalysiert anschließend die Umwandlung des Ser-Restes zu Sec[9] unter Verwendung von Selenophosphat, das von der Selenophosphatsynthase (SelD) bereitgestellt wird.[10] Eines der zentralen Erkennungsmerkmale von tRNASec für SelA aus E. coli ist das nichtkanonische, aus 13 Basenpaaren (bp) gebildete Motiv aus Akzeptorarm und T-Stamm,[11] das folglich auch in tRNASec aus E. coli und dessen EF-Tu-kompatiblen synthetischen Derivaten (UTu, UTuX, UTu6 und SecUx) enthalten ist.[6a,c,d, 8] Während dieses 13-bp-Motiv allerdings für die SelB-vermittelte Umkodierungsreaktion optimiert ist,[12] sind kanonische tRNAs, die ein lediglich 12 bp langes Strukturmotiv aus Akzeptorarm und T-Stamm bilden, aller Wahrscheinlichkeit nach besser für die EF-Tu-vermittelte kanonische Translation geeignet.[13]
Um diese Hürde zu überwinden, haben wir SelA-Spezies verwendet, die tRNASec-Varianten erkennen können, die das reguläre 12-bp-Motiv von kanonischer tRNA aufweisen. Die eng mit E. coli verwandte Bakterienspezies Aeromonas salmonicida (As) subsp. pectinolytica 34mel enthält natürlicherweise ein solches SelA- und tRNASec-Paar.[14] Die ersten Versuche, EF-Tu-kompatible As-tRNASec-Varianten zu generieren, waren erfolglos (Daten nicht gezeigt), was darauf schließen lässt, dass selbst das 12-bp-Motiv der As-tRNASec-Struktur nicht ausreicht, um die Serylierung durch SerRS und anschließende EF-Tu-vermittelte Translation zu ermöglichen.
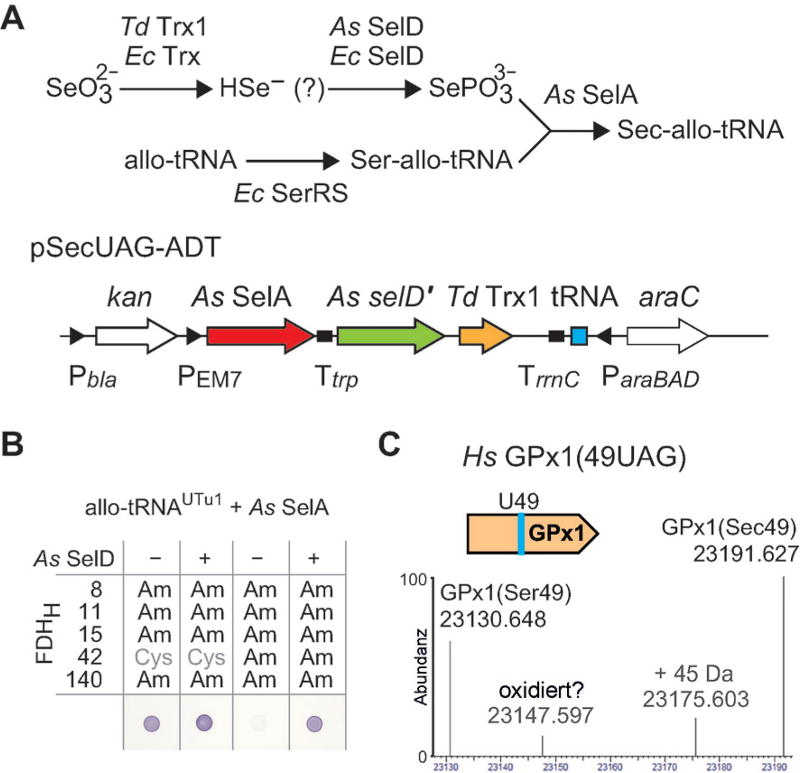
Kürzlich entdeckten wir während der Metagenomanalyse zur Suche nach natürlich vorkommenden tRNASec-Varianten eine Gruppe von neuartigen allo-tRNAs, die eine ungewöhnliche Kleeblattstruktur einnehmen.[15] Einige dieser allo-tRNAs fungieren in E. coli als tRNASer,[15] und wir vermuteten, dass sie möglicherweise auch Erkennungssequenzen für SelA enthalten (Abbildung 1 A). Dabei zeigten einige der per Metagenomanalyse identifizierten allo-tRNA-Spezies (allo-tRNAUTu1) eine ähnliche Ambersuppressionseffizienz wie E.-coli-supD-tRNASer (Abbildung 1 A und Hintergrundinformationen, Abbildung S1), die eine der aktivsten Suppressor-tRNAs in E. coli ist.[16] Folglich haben wir diese allo-tRNA-Variante zusammen mit As-SelA verwendet.
Abbildung 1.
Translation des UAG-Stopcodons als Serin und Selenocystein. A) Kleeblattstrukturen von E.-coli.-supD-tRNASer und allo-tRNAUTu1. B) FDHH-Expression in ΔselABCΔfdhF (ME6) aus E. coli bei 25°C mittels allo-tRNAUTu1, mit und ohne As-SelA, sowie in Gegenwart von Genvarianten des Reporters fdhF. Am: Amber-UAG-Codon.
Als Testsystem für die Analyse des EF-Tu-basierten Sec-Einbaus verwendeten wir das fdhF-Gen von E. coli, das für die Formatdehydrogenase H (FDHH; Abbildung 1 B) kodiert.[6a,c,d, 17] Dazu wurde das UGA-Codon 140, welches für das katalytisch aktive Sec im aktiven Zentrum kodiert, zu einem UAG-Codon mutiert (Abbildung 1 B). Wie zu erwarten, führte anschließend die Koexpression von allo-tRNAUTu1 und As-SelA zum Sec-Einbau und ermöglichte die Produktion von Wildtyp-FDHH, welches das artifizielle Substrat Benzylviologen zu einem violetten Farbstoff reduziert (Abbildung 1 B). Um das volle Potenzial von allo-tRNA zu analysieren und zu testen, ob mit dem allo-tRNA-System der Einbau von multiplen Sec-Resten in ein Protein möglich ist, wurden die vier Cys-Codons (Position 8, 11, 15 und 42) des fdhF-Gens zu UGA-Codons mutiert. Bereits zuvor hatten wir gezeigt, dass jeder dieser vier Cys-Reste separat durch Sec, aber nicht durch Ser ersetzt werden kann, ohne die FDHH-Aktivität zu beeinträchtigen (Abbildung 1 B).[23] Daher erzeugten wir fdhF-Genvarianten, die bis zu fünf UAG enthalten. Alle UAG-Codons müssen als Sec translatiert werden, und sowohl frühzeitige translationale Termination durch Releasefaktor 1 (RF-1) als auch Ser-Einbau durch Ser-allo-tRNAUTu1 würden in einer inaktiven FDHH resultieren. Überraschenderweise war es mithilfe der allo-tRNAUTu1 und As-SelA möglich, bis zu fünf UAG-Codons auf einer mRNA zu translatieren (Abbildung 1 B). Folglich könnten die verschiedenen FDHH-Varianten bis zu fünf Sec enthalten. Allerdings musste davon ausgegangen werden, dass es auch zu vorzeitiger translationaler Termination an UAG-Codons oder fälschlichem Einbau von Ser gekommen ist, da mit steigender Anzahl an UAG-Codons auf der fdhF-mRNA die FDHH-Aktivität der E.-coli-Zellkolonien abgenommen hat (Abbildung 1 B). Auch wenn allo-tRNAUTu1 den früheren tRNASec-Varianten offensichtlich in Bezug auf Selenoprotein-Überproduktion überlegen ist (Abbildung S2), lässt sie möglicherweise nicht denselben Grad an Reinheit und Sec-Einbauraten zu.[6c] Daher haben wir zur weiteren Verfeinerung einen Ansatz gewählt, bei dem der Selenstoffwechsel modifiziert wird, allo-tRNA und SelA optimiert werden und die Expression des gesamten Sec-Einbausystems fein aufeinander abgestimmt wird, um sowohl die Ser-Sec-Umwandlung auf der allo-tRNA als auch den anschließenden Einbau von Sec zu fördern.
Um größere Mengen des Selendonors Selenophosphat für As-SelA bereitzustellen, haben wir As-SelD und Treponema denticola (Td) Trx1 (Selenovariante von Thioredoxin) rekombinant koexprimiert (Abbildung 2 A).[18] Tatsächlich resultierte die Expression von As-SelD in einer deutlich gesteigerten Ausbeute an FDHH mit fünf Sec-Aminosäureresten (Abbildung 2 B). Anschließend generierten wir eine Mutante von Td-Trx1, bei der Sec über ein UAG kodiert ist (Abbildung 2 A). Um anschließend die Effizienz des Sec-Einbaus zu bestimmen, verwendeten wir die humane Glutathionperoxidase 1 (GPx1), die im aktiven Zentrum Sec an Aminosäureposition 49 enthält, als Reporterenzym. GPx1-mRNA mit UAG-Codon 49[6a] wurde mithilfe eines pET-Vectors mit ähnlichen Expressionraten wie GPx1(Cys49) überexprimiert und der Sec-Einbau per Massenspektrometrie analysiert. Während ohne Td-Trx1 mehr als 50% des GPx1-Reporters an Position 49 Ser enthielten (Abbildung S3), zeigten die Massenspektrometriesignale von GPx1-Proben, die in Gegenwart von Td-Trx1 produziert wurden, Sec-Einbauraten von deutlich über 50% (Abbildungen 2 C und S4). Daraus lässt sich folgern, dass As-SelD und Td-Trx1 in der Lage sind, bei hohem Sec-Bedarf schnell ausreichende Mengen des Selendonors zur Verfügung zu stellen. Da das pSecUAG-ADT-System (Abbildung 2 A) im Nährmedium enthaltenes Selenit (10 µm) zu rotgefärbtem elementarem Selen umsetzt, steht den Zellen aller Wahrscheinlichkeit nach ausreichend Selenit zur Verfügung. Tatsächlich ließ sich mit weiteren Proteinen und Enzymen, die am Selenstoffwechsel beteiligt sind (z.B. Thioredoxinreduktase, Selenocysteinlyase als spekulative S-Transporter, die möglicherweise auch am Selentransfer beteiligt sein könnten; Abbildung S5A) keine weitere Verbesserung der Sec-Einbauraten erzielen (Abbildungen S4A und S5B).
Abbildung 2.
Modifizierter Selenstoffwechsel. A) Mutmaßlicher Weg des Selentransfers zu As-SelA in genetisch modifierten E.-coli-Zellen, die pSecUAG-ADT enthalten. B) FDHH-Expression in ME6-Zellen bei 25°C. C) Massenspektrometrie (intakte Masse) einer Mischung von humanem GPx1 (Ser49 und Sec49), die aus E.-coli-ME6-Zellen mit pSecUAG-ADT gereinigt wurde. Die berechneten Massen liegen bei 23133 Da für GPx1(Ser49) und 23193 Da für GPx1(Sec49). In zahlreichen Fällen wurden Signale beobachtet, die möglicherweise monooxidiertes (+16 Da), dioxidiertes (+32 Da) oder ein etwas größeres (+42–47 Da) GPx1 repräsentieren. Die Sec-Einbaurate wurde auf 55% gesch900E4;tzt.
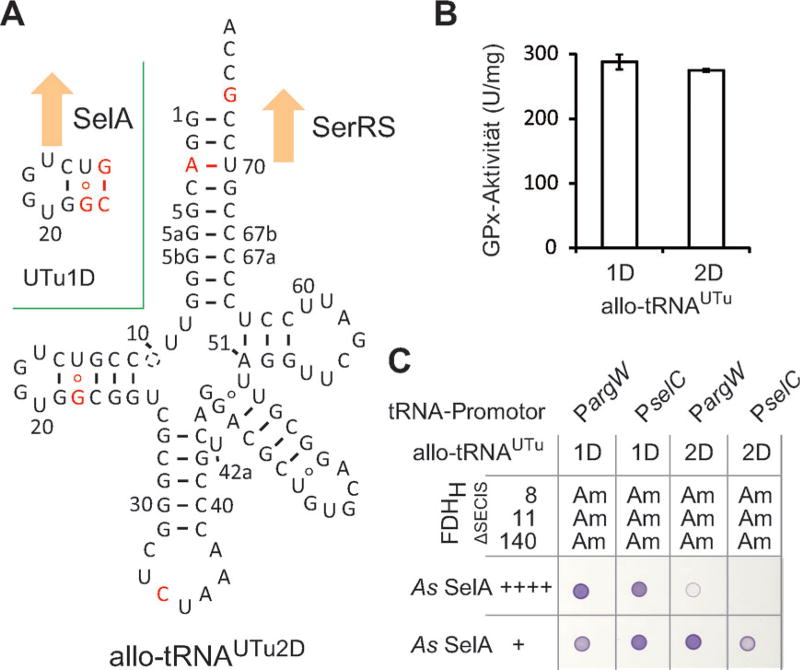
Wir haben Varianten von allo-tRNA hergestellt, die entweder im Detail oder in der Gesamtstruktur modifiziert waren (Abbildung 3 A). Eine allo-tRNAUTu1-Variante (allo-tRNAUTu1D) enthielt dabei einen Teil des A.-salmonicida-tRNASec-D-Stamms (Abbildung 3 A) und ermöglichte effizienteren Sec-Einbau als die parentale allo-tRNAUTu1 (Abbildungen S6 und S7). Das U14-G21-Wobble-Basenpaar in allo-tRNAUTu1D könnte dabei zur verbesserten SelA-Bindung beitragen. Außerdem entwickelten wir eine weitere, als allo-tRNAUTu2D bezeichnete allo-tRNA-Variante (Abbildungen 3 A und S6). Diese beruhte auf allo-tRNAAla und weist eine von allo-tRNAUTu1 abweichende Kleeblattstruktur auf.[15] Überraschenderweise zeigten GPx1-Proteine, die mit allot-RNAUTu1D oder allo-tRNAUTu2D produziert wurden, ähnliche Glutathionperoxidase-Aktivität (Abbildung 3 B); ähnliche Raten für den Sec-Einbau in die GPx1-Proteine wurden außerdem per Massenspektrometrie bestätigt (Abbildung S7). Weil allo-tRNAUTu2D mithilfe eines schwachen tRNA-Promotors exprimiert wurde, konnte sie höchstwahrscheinlich einfacher von überschüssigem SelA gebunden werden als allo-tRNAUTu1D (Abbildung 3 C). Allo-tRNAUtu2D benötigte geringere Mengen an As-SelA, um Sec-tRNA zu synthetisieren. Wir wählten allo-tRNAUTu2D, da SelA-Überexpression eine mögliche Belastung für Zellen ist.
Abbildung 3.
Modifikationen der allo-tRNA-Struktur. A) Kleeblattstrukturen zweier allo-tRNA-Varianten. Zusätzlich eingefügte Mutationen sind in Rot gekennzeichnet. B) Glutathionperoxidase(GPx)-Aktivität von GPx1, die mit allo-tRNAUTu1D und allo-tRNAUTu2D in ME6-Zellen bei 25°C produziert wurde. Jeder Balken repräsentiert den Durchschnitt von drei unabhängigen Experimenten auf Basis unterschiedlicher E.-coli-Kolonien. C) FDHH-Expression in ME68z-Zellen bei 25°C. As-SelA wurde mit einem starken (+ + + +) oder schwachen (+) Promotor produziert. Allo-tRNAsUTu wurden mit den Promotoren PargW oder PselC produziert.
Anschließend modifizierten wir die Struktur von As-SelA, um eine bessere Positionierung des allo-tRNA-Substrates zu erreichen (Abbildung 4 A). Die N-terminale Domäne von SelA bindet an die Ellbogenregion von Ser-tRNASec, um den Ser-Rest im aktiven Zentrum in der Kerndomäne von SelA zu positionieren. Der N-Terminus wechselwirkt dabei durch hydrophobe Interaktion einiger weniger Aminosäurereste mit der Kerndomäne (Abbildung 4 A).[9] Dabei gingen wir davon aus, dass Modifikationen, welche die Protein-Protein-Interaktion der beiden Domänen beeinflussen, die Orientierung der N-terminalen Domäne verändern würden und folglich die Umwandlung von Ser zu Sec auf der allo-tRNA verbessern könnten. Auf Grundlage der Proteinstruktur eines Komplexes aus SelA und tRNASec[9] modifizierten wir die Aminosäurereste Pro68, Leu69, Gln72 und Cys173 von As-SelA, die Ile25, Tyr26, Lys29 und Glu129 von SelA aus Aquifex aeolicus entsprechen (Abbildungen 4 A und S8). Zum Screening der As-SelA-Mutantenbibliothek verwendeten wir eine NMC-A-β-Lac-tamasevariante,[6c] deren essenzielle Disulfidbrücke in E.-coli-C321.ΔA.opt ΔselAB durch eine Diselenitbrücke ersetzt werden muss (Abbildung 4 B). Wir fanden verschiedene vielversprechende SelA-Varianten (Tabelle S1, Abbildungen 4 B und S9), von denen unter anderem die Varianten #1.9 und #2.1 zahlreiche Mutationen aufweisen, A68-I69-E72-R173 und P68-F69-S72-V173. Darüber hinaus enthält die SelA-Variante #1.9 noch eine zusätzliche spontane Pro2-Thr2-Mutation. Beide As-SelA-Varianten erhöhten die Expression von FDHH mit fünf Sec-Aminosäureresten bei 30°C (Abbildungen 4 C und S10) und 37°C (Abbildung S11) drastisch und waren weiterhin orthogonal zu E.-coli-tRNASer (Abbildung S12). Schließlich wurden #2.1 und Thr2 kombiniert, um SelAEvol zu erhalten.
Abbildung 4.
Modifikation von SelA aus A. salmonicida. A) Aminosäurereste, die gemäß der Proteinkristallstruktur von Aquifex-aeolicus-SelA und Thermoanaerobacter-tengcongensis-tRNASec (PDB: 3w1k) an der Fixierung der N-terminalen SelA-Domäne beteiligt sind. B) Selektion hochaktiver As-SelA-Varianten mittels NMC-A-β-Lactamasereporterassay in E.-coli-C321.ΔA.opt ΔselAB. Verdünnungsreihe von Zellen, die entweder den SelA-Wildtyp (WT) oder SelA-Mutanten exprimieren, auf Ampicillin-Agarplatten (Inkubation bei 30°C. C) FDHH-Expression bei 30°C in ME6-fdhF-Zellen (5 UAG-Codons) mit pSecUAG-d-allo-tRNAUTu1D und pMWcat-AsSelA(GUG) sowie SelA-Wildtyp oder SelA-Mutanten. Natriumselenit wurde in einer Endkonzentration von 5 µm zugegeben. D) FDHH-Expressionsniveaus in ME68z-fdhF-Zellen (5 UAG-Codons & ΔSECIS) bei 37°C waren am höchsten, wenn allo-tRNAUTu2D und SelAEvol unter milden Bedingungen exprimiert wurden (Arabinose (ara) 0.01% und „+ +“).
SelAEvol wurde zusammen mit allo-tRNAUTu2D subkloniert, um eine Serie von pSecUAG-Evol-Vektorvarianten (Nr. 1–4) zu erhalten, die gewährleisten, dass die relativen Expressionsniveaus von tRNA und SelA miteinander korrelieren. Die Expression von SelAEvol wird dabei von vier verschiedenen Transkriptions/Translations-Initiationssignalen kontrolliert (Nr. 1 > 2 > 3 > 4), und die Expressionsniveaus von allo-tRNAs lassen sich über die Arabinosekonzentration einstellen. Da die unerwünschte Suppression von Ambercodons am Ende essenzieller Gene zu Problemen im Zellstoffwechsel führt, wurde E.-coli-ME6 mit einem Plasmid versetzt, das für essenzielle Gene kodiert, die einen UAG-UAA-Austausch aufweisen;[19] der resultierende E.-coli-Stamm wurde als ME68z bezeichnet (siehe Materialien und Methoden in den Hintergrundinformationen). Die besten FDHH-Ausbeuten wurden bei 37°C, mit pSecUAG-Evol3 und mit 0.01% Arabinose erzielt (Abbildung 4 D). Das Vektorkonstrukt pSecUAG-Evol2 ermöglichte in Gegenwart von 0.001% Arabinose bei 20°C die Produktion und Reinigung von Selenoglutaredoxin,[6a,d, 20] einer Variante von E.-coli-Glutaredoxin-Grx1(Cys11Sec/Cys14Ser), in einer Ausbeute von 0.9 mg pro 2 L Bakterienkultur mittels Thiolsepharose-Affinitätschromatographie (Abbildungen S13 und S14).[6a]
GPx1 wurde mithilfe von pSecUAG-Evol2 in Gegenwart von 0.001% Arabinose bei 25°C produziert (Abbildung 5 A). Proteinausbeute und Effizienz der UAG-Suppression lagen bei 2–3 mgL−1 bzw. 70%. Durch Massenspektrometrie (intakte Masse) wurde anschließend bestätigt, dass hauptsächlich (> 80%) Sec eingebaut wurde (Abbildung 5 B). Die Masse von GPx1(49Sec) stellte sich als um 44–46 Da erhöht heraus (Abbildung 5B), was einem Cys-zu-Sec-Austausch an einer beliebigen der fünf Cys-Positionen entsprechen kann.[21] Hierbei muss berücksichtigt werden, dass hohe Expressionsraten an SelAEvol möglicherweise in Sec-allo-tRNA-Konzentrationen resultieren, die den Bedarf für die UAG-Translation weit überschreiten. Die Hydrolyse überschüssiger Sec-allo-tRNA könnte möglicherweise zu freiem Sec führen, das nachfolgend zu von der Cysteinyl-tRNA-Synthetase katalysiertem, fehlerhaftem Sec-Einbau an Cys-Codons führen könnte. Daher wurde versucht, die Expressionsraten von SelAEvol und allo-tRNAUTu2D moderat zu halten. Erstaunlicherweise verschwand bei maximaler SelAEvol-Ü;berproduktion (pSecUAG-Evol1) die GPx1-Proteinbande in der SDS-PAGE-Analyse, während eine zuvor nicht beobachtete Bande bei etwa 55 kDa auftauchte (Abbildung 5 A). Überschüssiges SelAEvol hat hier möglicherweise allo-tRNAUTu2D sequestriert. Zusätzlich hat sich gezeigt, dass SelAEvol eine Affinität zu Nickelaffinitätsmaterial aufweist, die möglicherweise durch Histidinreste in der N-terminalen Domäne vermittelt wird. Folglich änderten wir die A-H-S-H-S-Sequenz von SelAEvol zu P-Y-R (von einer anderen Aeromonas-SelA-Variante) oder zu A-S-S-A-S (Abbildung S15). GPx1-Protein, das mit SelAEvol und der ASSAS-Variante produziert wurde, zeigte ähnliche GPx-Aktivität, wohingegen die PYR-Variante etwas weniger aktiv war (Abbildung 5 C).
Abbildung 5.
Optimierung des Sec-Einbausystems in ME68z-Zellen. A) SDS-PAGE von gereinigtem GPx1-Protein, das mit pSecUAG-Evol1 und 2 produziert wurde. Arabinose wurde in Konzentrationen von 0.01% (+ +) und 0.001% (+) verwendet. Zellüberstand und Durchfluss wurden ebenfalls auf dem Gel analysiert. Unerwarteterweise wurde SelAEvol durch Nickelaffinitätschromatographie cogereinigt. B) Massenspektrometrie (intakte Masse) der Probe aus Panel A, rechte Spur, die mittels pSecUAG-Evol2 produziert wurde. Die Sec-Einbaurate lag bei 84%. Das Signal bei 23 237.601 stammt möglicherweise von GPx1(Sec49), das einen Cys-Sec-Austausch an einer der fünf Cys-Positionen aufweist. C) Glutathionperoxidase-Aktivität von GPx1, die mit SelAEvol-Varianten produziert wurde. Jeder Balken repräsentiert den Durchschnitt von drei unabhängigen Experimenten auf Basis unterschiedlicher E.-coli-Kolonien. D) Proteolysemuster von MXB-Varianten, die mit SelA-Varianten und SufS(C364A) produziert wurden. Die Proteolyse mittels Intein erfolgte in vitro in Gegenwart von 100 mm Dithiothreitol und bei Raumtemperatur über 16 h.
Abschließend wurde pSecUAG-Evol2 noch modifiziert, um für die ASSAS-Variante und die Selenocysteinlyase SufS(C364A)[22] zu kodieren; dies führte zu pSecUAG2. Der vorherrschende Sec-Einbau wurde mithilfe eines In-vitro-Inteinproteolyseassays bestätigt, der eine Mxe-GyrA-Inteinvariante (MXB) verwendet, deren katalytisches Cys384 durch Sec ersetzt wurde.[6b] Die Ausbeute an Protein (Volllängenprotein) bei 25°C Expressionstemperatur lag bei etwa 10 mgL−1. Da nur < 10% des Volllängenproteins nach der Proteolysereaction intakt blieben (Abbildungen 5 D und S16) und weil die Ser384-Variante von MXB katalytisch inaktiv ist,[6b] wurde die Sec-Einbaurate unter der Annahme einer vollständigen Inteinreaktion auf > 90% geschätzt/bestimmt.
Im Unterschied zu früheren tRNAUTu-Varianten, die einen 13-bp-Arm enthalten, ist das allo-tRNA-System bereits intrinsisch von der SelB/SECIS-Maschinerie abgekoppelt und daher voll kompatibel mit dem kanonischen Proteintranslationsapparat. Zukünftige Optimierungen sollten sich daher auf die Ausbeute und Spezifität der gewünschten rekombinanten Selenoproteine konzentrieren. Einfachheit und Kompatibilität des Systems dürften außerdem mit überschaubarem Aufwand eine Anwendung in anderen Organismen ermöglichen.
Supplementary Material
Acknowledgments
Wir danken Anna Merkuryev für Ihre Hilfe beim SelA-Engineering und Navin Rauniyar, Ana Crnković sowie Omer Ad für ihr Engagement während der MS-Analysen. T.M. ist Stipendiat der Japan Society for the Promotion of Science. Wir bedanken uns für die Unterstützung dieses Projektes durch die US National Institutes of Health (Grants R01GM22854 und R35GM122560 an D.S.); genetische Experimente wurden durch Mittel des Department of Energy (Grant DE-FG02-98ER20311 an D.S.) unterstützt. Dieser Artikel wurde von Dr. Markus J. Bröcker aus dem Englischen übersetzt.
Footnotes
Hintergrundinformationen und die Identifikationsnummer (ORCID) eines Autors sind unter: https://doi.org/10.1002/ange.201713215 zu finden.
Interessenkonflikt
Die Autoren erklären, dass keine Interessenkonflikte vorliegen.
Contributor Information
Dr. Takahito Mukai, Department of Molecular Biophysics and Biochemistry, Yale University, New Haven, CT 06520 (USA)
Dr. Anastasia Sevostyanova, Department of Molecular Biophysics and Biochemistry, Yale University, New Haven, CT 06520 (USA)
Dr. Tateki Suzuki, Department of Molecular Biophysics and Biochemistry, Yale University, New Haven, CT 06520 (USA)
Dr. Xian Fu, Department of Molecular Biophysics and Biochemistry, Yale University, New Haven, CT 06520 (USA)
Prof. Dieter Söll, Department of Molecular Biophysics and Biochemistry, Yale University, New Haven, CT 06520 (USA) Department of Chemistry, Yale University, New Haven, CT 06520 (USA).
References
- 1.Mousa R, Notis Dardashti R, Metanis N. Angew. Chem. Int. Ed. 2017;56:15818. doi: 10.1002/anie.201706876. Angew. Chem. 2017, 129, 16027. [DOI] [PubMed] [Google Scholar]
- 2.a) Metanis N, Keinan E, Dawson PE. Angew. Chem. Int. Ed. 2010;49:7049. doi: 10.1002/anie.201001900. Angew. Chem. 2010, 122, 7203. [DOI] [PMC free article] [PubMed] [Google Scholar]; b) Metanis N, Hilvert D. Chem. Sci. 2015;6:322. doi: 10.1039/c4sc02379j. [DOI] [PMC free article] [PubMed] [Google Scholar]; c) Cohen DT, Zhang C, Pentelute BL, Buchwald SL. J. Am. Chem. Soc. 2015;137:9784. doi: 10.1021/jacs.5b05447. [DOI] [PMC free article] [PubMed] [Google Scholar]; d) Reich HJ, Hondal RJ. ACS Chem. Biol. 2016;11:821. doi: 10.1021/acschembio.6b00031. [DOI] [PubMed] [Google Scholar]; e) Marques MC, Tapia C, Gutiérrez-Sanz O, Ramos AR, Keller KL, Wall JD, De Lacey AL, Matias PM, Pereira IAC. Nat. Chem. Biol. 2017;13:544. doi: 10.1038/nchembio.2335. [DOI] [PubMed] [Google Scholar]; f) Ingold I, Berndt C, Schmitt S, Doll S, Poschmann G, Buday K, Roveri A, Peng X, Porto Freitas F, Seibt T, Mehr L, Aichler M, Walch A, Lamp D, Jastroch M, Miyamoto S, Wurst W, Ursini F, Arner ESJ, Fradejas-Villar N, Schweizer U, Zischka H, Friedmann Angeli JP, Conrad M. Cell. 2018;172:409. doi: 10.1016/j.cell.2017.11.048. [DOI] [PubMed] [Google Scholar]
- 3.a) Liu J, Chen Q, Rozovsky S. J. Am. Chem. Soc. 2017;139:3430. doi: 10.1021/jacs.6b10991. [DOI] [PMC free article] [PubMed] [Google Scholar]; b) Rakauskaitė R, Urbanavičiūtė G, Rukšėnaitė A, Liutkevičiūtė Z, Juškėnas R, Masevičius V, Klimašauskas S. Chem. Commun. 2015;51:8245. doi: 10.1039/c4cc07910h. [DOI] [PubMed] [Google Scholar]
- 4.a) Shchedrina VA, Novoselov SV, Malinouski MY, Gladyshev VN. Proc. Natl. Acad. Sci. USA. 2007;104:13919. doi: 10.1073/pnas.0703448104. [DOI] [PMC free article] [PubMed] [Google Scholar]; b) Metanis N, Hilvert D. Curr. Opin. Chem. Biol. 2014;22:27. doi: 10.1016/j.cbpa.2014.09.010. [DOI] [PubMed] [Google Scholar]; c) Arai K, Takei T, Okumura M, Watanabe S, Amagai Y, Asahina Y, Moroder L, Hojo H, Inaba K, Iwaoka M. Angew. Chem. Int. Ed. 2017;56:5522. doi: 10.1002/anie.201701654. Angew. Chem. 2017, 129, 5614. [DOI] [PubMed] [Google Scholar]
- 5.Wright TH, Bower BJ, Chalker JM, Bernardes GJ, Wiewiora R, Ng WL, Raj R, Faulkner S, Vallee MR, Phanumartwiwath A, Coleman OD, Thezenas ML, Khan M, Galan SR, Lercher L, Schombs MW, Gerstberger S, Palm-Espling ME, Baldwin AJ, Kessler BM, Claridge TD, Mohammed S, Davis BG. Science. 2016;354:597. doi: 10.1126/science.aag1465. [DOI] [PubMed] [Google Scholar]
- 6.a) Aldag C, Bröcker MJ, Hohn MJ, Prat L, Hammond G, Plummer A, Söll D. Angew. Chem. Int. Ed. 2013;52:1441. doi: 10.1002/anie.201207567. Angew. Chem. 2013, 125, 1481. [DOI] [PMC free article] [PubMed] [Google Scholar]; b) Haruna K, Alkazemi MH, Liu Y, Söll D, Englert M. Nucleic Acids Res. 2014;42:9976. doi: 10.1093/nar/gku691. [DOI] [PMC free article] [PubMed] [Google Scholar]; c) Thyer R, Robotham SA, Brodbelt JS, Ellington AD. J. Am. Chem. Soc. 2015;137:46. doi: 10.1021/ja510695g. [DOI] [PMC free article] [PubMed] [Google Scholar]; d) Miller C, Bröcker MJ, Prat L, Ip K, Chirathivat N, Feiock A, Veszprémi M, Söll D. FEBS Lett. 2015;589:2194. doi: 10.1016/j.febslet.2015.06.039. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Rudinger J, Hillenbrandt R, Sprinzl M, Giegé R. Embo J. 1996;15:650. [PMC free article] [PubMed] [Google Scholar]
- 8.Fan Z, Song J, Guan T, Lv X, Wei J. ACS Synth. Biol. 2018;7:249. doi: 10.1021/acssynbio.7b00290. [DOI] [PubMed] [Google Scholar]
- 9.Itoh Y, Bröcker MJ, Sekine S, Hammond G, Suetsugu S, Söll D, Yokoyama S. Science. 2013;340:75. doi: 10.1126/science.1229521. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Silva IR, Serrão VH, Manzine LR, Faim LM, da Silva MT, Makki R, Saidemberg DM, Cornélio ML, Palma MS, Thiemann OH. J. Biol. Chem. 2015;290:29178. doi: 10.1074/jbc.M114.613406. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Baron C, Böck A. J. Biol. Chem. 1991;266:20375. [PubMed] [Google Scholar]
- 12.Fischer N, Neumann P, Bock LV, Maracci C, Wang Z, Paleskava A, Konevega AL, Schröder GF, Grubmüller H, Ficner R, Rodnina MV, Stark H. Nature. 2016;540:80. doi: 10.1038/nature20560. [DOI] [PubMed] [Google Scholar]
- 13.Ohtsuki T, Manabe T, Sisido M. FEBS Lett. 2005;579:6769. doi: 10.1016/j.febslet.2005.11.010. [DOI] [PubMed] [Google Scholar]
- 14.Mukai T, Englert M, Tripp HJ, Miller C, Ivanova NN, Rubin EM, Kyrpides NC, Söll D. Angew. Chem. Int. Ed. 2016;55:5337. doi: 10.1002/anie.201511657. Angew. Chem. 2016, 128, 5423. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Mukai T, Vargas-Rodriguez O, Englert M, Tripp HJ, Ivanova NN, Rubin EM, Kyrpides NC, Söll D. Nucleic Acids Res. 2017;45:2776. doi: 10.1093/nar/gkw898. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Eggertsson G, Söll D. Microbiol. Rev. 1988;52:354. doi: 10.1128/mr.52.3.354-374.1988. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Boyington JC, Gladyshev VN, Khangulov SV, Stadtman TC, Sun PD. Science. 1997;275:1305. doi: 10.1126/science.275.5304.1305. [DOI] [PubMed] [Google Scholar]
- 18.a) Kim MJ, Lee BC, Hwang KY, Gladyshev VN, Kim HY. Biochem. Biophys. Res. Commun. 2015;461:648. doi: 10.1016/j.bbrc.2015.04.082. [DOI] [PMC free article] [PubMed] [Google Scholar]; b) Tamura T, Sato K, Komori K, Imai T, Kuwahara M, Okugochi T, Mihara H, Esaki N, Inagaki K. Biosci. Biotechnol. Biochem. 2011;75:1184. doi: 10.1271/bbb.100847. [DOI] [PubMed] [Google Scholar]; c) Kumar S, Björnstedt M, Holmgren A. Eur. J. Biochem. 1992;207:435. doi: 10.1111/j.1432-1033.1992.tb17068.x. [DOI] [PubMed] [Google Scholar]
- 19.Mukai T, Yanagisawa T, Ohtake M, Wakamori K, Adachi J, Hino N, Sato A, Kobayashi T, Hayashi A, Shirouzu M, Umehara T, Yokoyama S, Sakamoto K. Biochem. Biophys. Res. Commun. 2011;411:757. doi: 10.1016/j.bbrc.2011.07.020. [DOI] [PubMed] [Google Scholar]
- 20.Casi G, Roelfes G, Hilvert D. ChemBioChem. 2008;9:1623. doi: 10.1002/cbic.200700745. [DOI] [PubMed] [Google Scholar]
- 21.Guo X, Yu Y, Liu X, Zhang Y, Guan T, Xie G, Wei J. IUBMB Life. 2014 doi: 10.1002/iub.1255. [DOI] [PubMed]
- 22.Mihara H, Kurihara T, Yoshimura T, Esaki N. J. Biochem. 2000;127:559. doi: 10.1093/oxfordjournals.jbchem.a022641. [DOI] [PubMed] [Google Scholar]
- 23.Miller, Reynolds, et al. unveröffentlichte Ergebnisse [Google Scholar]
Associated Data
This section collects any data citations, data availability statements, or supplementary materials included in this article.