Abstract
目的
探讨结直肠癌发展早期肠道菌群的变化规律。
方法
通过结肠镜活检收集31例结肠腺瘤患者的腺瘤组织以及20例健康志愿者的肠道黏膜,提取基因组DNA,对细菌16SrRNA基因序列V3~V4高变区进行PCR扩增,lluminaMiSeq平台上高通童测序分析。
结果
结肠腺瘤患者的肠道黏膜菌群多样性比健康对照人群显著增高(P < 0.01),腺瘤组黏膜菌群结构和健康对照者相比差异显著。门水平上:结肠腺瘤组变形菌门明显增多(P < 0.01),厚壁菌门/拟杆菌门比值下降。属水平上:结肠腺瘤组乳球菌属、链球菌属和假单胞菌属增多(P < 0.01),而肠球菌属,杆菌属丰度明显下降(P < 0.01)。不同病理类型腺瘤比较发现:结肠高级別和低级別上皮内瘤变组肠道黏膜菌群整体结构相似,埃希菌-志贺菌属在高级別上皮内瘤变组呈现上升趋势,但差异尤统计学意义(P > 0.05)。
结论
结肠腺瘤患者肠道菌群多样性和构成与正常健康人存在明显差异,腺瘤患者黏膜菌群结构发生明显失衡,形成了易于肿瘤生长的肠道微生态环境。
Keywords: 结肠腺瘤, 肠道菌群, 结构失调, 16SrRNA, 高通童测序
Abstract
Objective
To investigate the taxonomic richness and diversity of gut microbiota in patients with colorectal adenoma and elucidate the role of gut microorganisms in precancerous lesions in the colon and rectum.
Method
Adenomatous tissues from 31 patients with colorectal adenoma and normal intestinal mucosal tissues from 20 healthy control subjects were collected through colonoscopy. The total bacterial genomic DNA was extracted, and the V3-V4 hypervariable region in bacterial 16S rRNA gene was amplified using polymerase chain reaction and sequenced on an Illumina MiSeq platform.
Results
Patients with colorectal adenomas had a higher alpha diversity and richness indices compared to the healthy controls (P < 0.01). The mucosal microbiota in colorectal adenoma tissue showed a distinctive structural difference from that in normal intestinal mucosal tissues. At the phylum level, a large decrease in Firmicutes with concomitant relative expansion of Proteobacteria was observed in patients with colorectal adenomas, resulting in a significant decrease in the Firmicutes/Bacteroidetes ratio (P < 0.01). At the genus level, Lactococcus and Pseudomonas were enriched whereas Enterococcus, Bacillus, and Solibacillus were reduced obviously in the preneoplastic tissues (P < 0.01). We also found a similar gut microbiome composition between low-grade and high-grade intraepithelial neoplasia; the ratio of Escherichia-Shigella tended to increase in high-grade intraepithelial neoplasia, but this change was not statistically significant (P=0.28).
Conclusion
Significant changes in the structure of the intestinal flora occur in patients with colorectal adenomas, indicating that the association of dysbiosis of the gut microbiota with the occurrence of a pro-oncogenic microenvironment.
Keywords: colorectal adenoma, gut microbiota, structural imbalance, 16S rRNA, high-throughput
结直肠癌(CRC)是一种常见的消化道恶性肿瘤。在我国,随着生活方式和饮食习惯的改变,CRC的发病率和死亡率均呈上升趋势[1],全球CRC发病率位于男性恶性肿瘤的第4位,女性位于第3位[2]。通常认为,结肠肿瘤主要包括结肠癌和结肠腺瘤,且结肠腺瘤是大肠癌最主要的癌前疾病[3]。美国结直肠癌研究小组报告从腺瘤发展至腺癌平均需要10年[4]。
结肠中肠道菌群密度(约1012/mL)远远高于小肠(102/mL),且结肠肿瘤的发生是小肠的12倍,据此有学者提出结肠肿瘤是菌群相关性疾病[5],多项研究揭示了结直肠癌患者的肠道菌群与健康人群之间存在显著不同[6-7]。目前对于肠癌癌前期病变的菌群变化尚未有模型详加讨论,而阐释结直肠癌发生发展各阶段肠道菌群的变化过程在临床上具有重要意义。因此,本研究通过运用微生物16SrRNA的高通量测序及生物信息学分析,观察结肠腺瘤患者和健康志愿者、以及腺瘤不同进展阶段的黏膜菌群构成差异,分析肠道菌群结构和生物分类丰富性、多样性改变,初步探索结肠癌前病变期扮演重要角色的关键菌属,为发展粪便或血清学细菌标志物及治疗提供新的思路及理论依据。
1. 资料和方法
1.1. 研究对象
本项研究纳人了2014年1~6月在上海交通大学附属第一人民医院进行肠镜检查的31例结肠腺瘤患者,其中男12例,女19例,以及20例无结肠腺瘤的健康人作为对照,其中男性9人,女性11人,年龄27~67岁,差异没有统计学意义。纳人标准为:(1) 年龄20~70周岁;(2) 体质量指数在正常范围之内,无超重或肥胖; (3) 既往无结直肠手术史; (4) 无家族性腺瘤性息肉病或遗传性非息肉性大肠癌的患者; (5) 无炎症性肠病(IBD)、代谢性疾病(如糖尿病、肥胖、高脂血症)或感染性疾病的患者、无严重肝肾疾病、无免疫缺陷;(6) 在收集标本之前3个月内未应用过抗生素或皮质类固醇激素或益生菌; (7) 无特殊饮食习惯; (8) 签署知情同意书。结肠腺瘤分布部位为:直肠5例,乙状结肠13例,降结肠4例,横结肠4例,升结肠5例。腺瘤直径等于或超过1 cm为17例,小于1 cm的有14例,均为单发的腺瘤。腺瘤的病理类型:腺瘤性息肉伴低级别上皮内瘤变26例,腺瘤性息肉伴高级别上皮内瘤变5例。
1.2. 样本采集
所有志愿者按常规进行肠道清洁准备,检查当日凌晨4点服用复方聚乙二醇电解质溶液2盒,待肠道准备完成后行无痛结肠镜。符合人选标准的志愿者在发现结肠息肉后进行活检2块,一块常规保存于福尔马林送病理科,另一块活检标本以记号笔标记后立即置于液氮,随后转移至-80 ℃冰箱保存待分析。另外20例对照标本取自健康人群相对应部位肠道的正常黏膜。同时记录结肠息肉的部位、大小、形状(有蒂或无蒂)及志愿者的身高、体质量、饮食习惯。每位参与者签署了知情同意书,本项研究通过上海交通大学附属第一人民医院伦理委员会审核。
1.3. 样本DNA提取和16SrRNA基因扩增
结肠黏膜样品的基因组DNA通过E.Z.N.A.®DNA Kit(Omega Bio-tek, Norcross, GA, U.S.)试剂盒按照说明书进行提取。简言之,将样品加人与酶裂解缓冲液、溶菌酶和消色肽酶,并在37 ℃反应1 h。然后加人蛋白酶K和裂解缓冲液AL,并将悬浮液于56 ℃下30 min,然后将基因组DNA进行离心柱纯化并洗脱。利用1%琼脂糖凝胶电泳检测抽提的基因组DNA,DNA储存在-20 ℃直至进一步分析。按指定测序区域,合成带有barcode的特异引物,进行细菌16SrRNA基因V3~V4区的扩增,所采用的细菌引物是515F 5’-barcode-GTGCCAGCMGCCGCGG-3'和907R 5'-CCGTCAATTCMTTTRAGTTT-3'。循环参数如下:95 ℃初始变性2 min, 5 ℃变性30 s 25个循环,55 ℃退火30 s,72 ℃延伸30 s,并在72 ℃下最后延伸5 min。PCR扩增反应体系(20 μL)包括:4 μL 5×FastPfu缓冲液, 2 μL 2.5 mmol单脱氧核苷酸,0.8 μL引物(5 pmol),0.4 μL FastPfu聚合酶,10 ng总DNA作为模板。每个样本3个重复,将同一样本的PCR产物混合后用2%琼脂糖凝胶电泳检测,使用AxyPrepDNA凝胶回收试剂盒(AXYGEN公司)切胶回收PCR产物,Tris-HCr冼脱,2%琼脂糖电泳检测。参照电泳初步定量结果,将PCR产物用QuantiFluorTM-ST蓝色荧光定量系统(Promega公司)进行检测定量,按照每个样本的测序量要求相应比例的混合,送美吉生物医药科技公司在lluminaMiSeq平台上进行高通量测序。
1.4. 生物信息学和统计分析
数据处理:首先去掉含有模糊碱基 “N”)的序列,过滤read尾部质量值20以下的碱基,设置50 bp的窗口,以1 bp步长滑行,如果窗口内的平均质量值低于20,从窗口开始截去后端碱基,过滤质控后50 bp以下的read(Trimmomatic软件);根据reads间的overlap关系,将成对reads拼接成一条序列,overlap长度最短为10 bp,最大错配比率为0.2,筛除不符合序列(FLASH软件)根据序列首尾两端的barcode和引物区分样品,并调整序列方向,barcode允许的错配数为0,最大引物错配数为2。对质控得到的优化序列提取非重复序列,去除没有重复的单序列,按照97%相似性对非重复序列进行分类操作单元(OTU)聚类,在聚类过程中去除嵌合体,得到OTU的代表序列(Usearch 7.1)。将OTU代表序列与Silva(<a href="http://www.arb-silva.de" target="_blank">http://www.arb-silva.de</a>)数据库比对,通过Qiime平台,采用RDP classifier贝叶斯算法对OTU代表序列进行分类学分析,将所有优化序列map至OTU代表序列,选出与OTU代表序列相似性在97%以上的序列,生成OTU-taxa表格。根据OTU-taxa表格计算ACE、Chao、Shannon和Simpson指数。ACE、Chao这两项是估计样本中所含OTU数目的指数,主要用来评估样品物种丰度。Simpson和Shannon指数常用于反映alpha多样性,Simpson指数值越大,说明群落多样性越低, Shannon值越大,说明群落多样性越高。计算稀释曲线来评估样本的测序数据量是否合理。通过主成分分析(PCA)及多样本相似度树状图反应样本间的分散和聚集分布情况。利用R语言绘制热图,热图用颜色变化直观反映样品物种及丰度情况。利用matastats (<a href="http://metastats.cbcb.umd.edu/" target="_blank">http://metastats.cbcb.umd.edu/</a>)在门、属水平上分析两组样本,进行组间显著性差异分析,得到两组的差异物种,数值以均值±标准误表示,数据采用SPSS20软件统计处理,并用两独立样本的<italic>t</italic>检验统计差异,<italic>P</italic> < 0.05认为差异有统计学意义。
1.5. 核酸登录号
本文中涉及的序列数据已提交至GenBank的Sequence Read Archive数据库,核酸登录号为SRP064975。
2. 结果
2.1. 测序数据和多样性指数
两组51个样本共获得706 980条可用的高质量序列,序列长度为440 bp左右。从97%相似水平上划分了OTU。根据16SrRNA基因序列相似度与细菌分类地位之间的对应关系, 97%的相似度通常被认为是对应于种的划分。在本项研究中结肠腺瘤组标本共获得805个OTU, 正常对照组共获得463个OTU, 所有样品的文库覆盖率达99%以上, 显示出在此测序量下,每个样品的测序量已经达到饱和。从结肠腺瘤组和正常对照组的OTU统计序列及相关多样性指数比较可以看出:腺瘤组较对照组的Shannon指数(P < 0.05)、Simpson指数(P < 0.05)、Chao I(P < 0.01)、ACE(P < 0.01) 指数和OTU (P < 0.01) 差异增高。
2.2. 门水平上分析
两组样本共包含19个门,从数量上说,厚壁菌门(Firmicutes)在全部细菌类群中占有最主要的地位。所有样本的优势菌门是厚壁菌门、拟杆菌门(Bacteroidetes)和变形菌门(Proteobacteria)。结肠腺瘤组中丰度最高的3个门依次为:厚壁菌门占53.7%、变形菌门占30.1%、拟杆菌门占10.8%。而正常对照中,厚壁菌门占88.6%、拟杆菌门占5.8%、变形菌门占3.3% (图 1)。通过Metastat分析显示总共有8个门包括厚壁菌门、变形菌门、拟杆菌门、放线菌门(Actinobacteria)、绿弯菌门(Chloroflexi)、蓝菌门(Cyanobacteria)、Candidate-division TM7和柔壁菌门(Tenericutes)在两组间有显著差异(图 2)。
1.
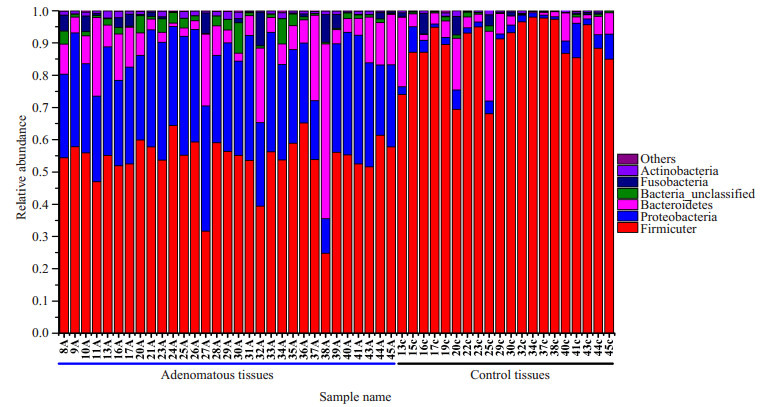
腺瘤组和正常对照组样本在门水平上的相对丰度
Relative abundance of bacterial phyla in microbiota in healthy volunteers and patients with adenoma.
2.
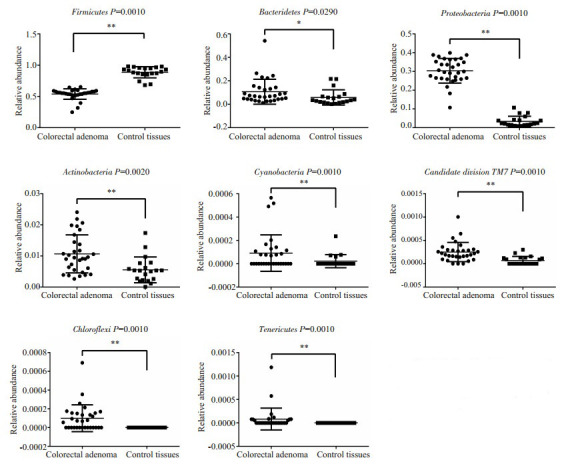
结肠腺瘤和正常对照组在门水平上丰度明显差异的菌
Relative abundance of significant differential phyla between adenomatous and control tissues. *P < 0.05, **P < 0.01.
2.3. 属水平上分析
通过与SILVA数据库比对,我们在51个标本中发现200多个菌属。正常对照组中大多数优势菌属属于厚壁菌门,丰度最高的依次是:肠球菌(Enterococcus)占40.7%、杆菌(Bacillus)占10.8%、Solibacillus占6.6%、Lachnospiraceae_incertae_sedis占6.0%、乳球菌(Lactococcus)占4.0%、拟杆菌属(Bacteroides)占3.6%、Lysinibacillus占2.6%、Sporosarcina占2.4%。而在结肠腺瘤组中优势菌属属于变形菌门,丰度最高的依次是:乳球菌占39.7 %、假单胞菌(Pseudomonas)占17.4%、拟杆菌属占4.9%、Psychrobacter占3.2%、Prevotella占3.2%、埃希菌-志贺菌(Escherichia-Shigella)占3.1%(图 3)。热图可以用颜色变化来反映二维矩阵或表格中的数据信息,可以直观地将数据值的大小以定义的颜色深浅表示出来,我们将腺瘤和对照两组中占优势的42个菌属绘制成热图(图 4)。
3.
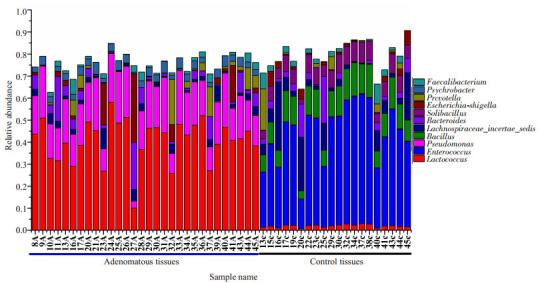
腺瘤组和正常对照组样本在属水平上的相对丰度
Relative abundance of bacterial genera in microbiota in healthy volunteers and patients with adenoma.
4.
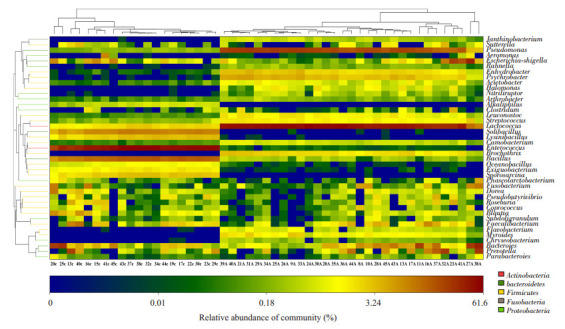
腺瘤和正常对照组属水平的heatmap聚类图
Heat map of the genera level bacterial abundances in healthy volunteers and patients with adenoma.
2.4. Beta多样性分析
2.4.1. 主成分分析(PCA)和非度量多维尺度分析(NMDS)分析
PCA是一种对数据进行简化分析的技术,通过降维寻找变量间的共线性关系。通过分析不同样本OTU组成可以反映样本间的差异和距离,如样本组成越相似,反映在PCA图中的距离越近(图 5),PC1、PC2分别代表对于两组样本微生物组成发生偏移的疑似影响因素,需要结合样本特征信息归纳总结,例如A组和B组样本在PC1轴的方向上分离开来,则可分析为PC1是导致A组和B组分开的主要因素,同时验证了这个因素有较高的可能性影响了样本的组成。基于本项研究,我们认为:腺瘤和健康组菌群黏膜结构明显不用,而疾病/健康的分组是造成PCA图中各样本分布差异的主要原因,且PC1因素达到90.45%,即患者长了腺瘤导致了菌群黏膜明显改变。NMDS也用于比对样本组之间的差异,可以基于进化关系或数量距离矩阵,横轴和纵轴分别表示基于进化或者数量距离矩阵的数值在二维表中成图(图 6)。
5.
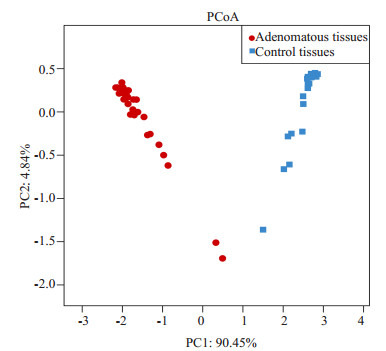
PCA分析的基于PC1,2的赋值样本分布图
PCoA plots based on Bray-Curtis metrics between adenomatous (red) and control (blue) tissues.
6.
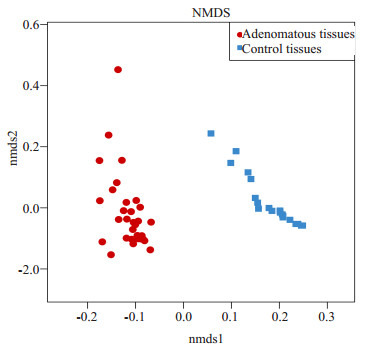
腺瘤和正常对照组NMDS分析
NMDS analysis of adenomatous (red) and control (blue) tissues.
2.4.2. 多样本相似度树状图
两组组成的聚类分析:利用树枝结构描述和比较多个样本间的相似性和差异关系。首先根据beta多样性距离矩阵进行层次聚类分析,使用非加权组平均法UPGMA算法构建树状结构,得到树状关系形式用于可视化分析。使用Qiime软件计算beta多样性距离矩阵,算法为bray curtis。最后用R语言作图画树(图 7),提示两组黏膜菌群结构明显不同。
7.
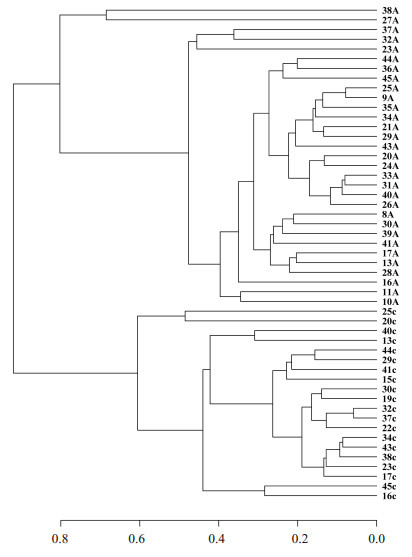
结肠腺瘤和正常对照组黏膜菌群的分成聚类树状图
Cluster tree based on Bray-Curtis distances between adenomatous (A) and control (C) tissues.
2.5. 不同病理类型腺瘤黏膜菌群比较
31个结肠腺瘤标本病理类型为:腺瘤性息肉伴低级别上皮内瘤变(LIN)26例,腺瘤性息肉伴高级别上皮内瘤变(HIN)5例,结肠癌发生发展过程中存在正常-LIN-HIN-原位癌-进展癌的演变过程,为了解在这动态发展过程中肠道菌群的变化,我们通过matastats比较了5例高级别上皮内瘤变和26例低级别上皮内瘤变患者的菌群结构。结果发现LIN组菌群丰度组成分别为:Lactococcus 40.7%、Pseudo-monas 17.8%、Bacteroides 4.9%、Psychrobacter 3.3%、Prevotella 2.4%、Enhydrobacter 2.4%、Brochothrix 2.0%、Escherichia-Shigella 1.8%。HIN组菌群丰度组成分别为:Lactococcus 35.4%、Pseudomonas 14.9%、Escherichia-Shigella 10.1%、Bacteroides 4.9%、cteria_unclassified 4.6%、Psychrobacter 2.7%、Lachnospiraceae_incertas_sedis 2.5%、Enhydrobacter 1.8%。低级别和高级别上皮内瘤变两组的菌群构成基本类似(图 8),虽然我们发现埃希菌-志贺菌属在结肠高级别上皮内瘤变组有升高趋势,但未呈现显著性差异(P=0.28)。
8.
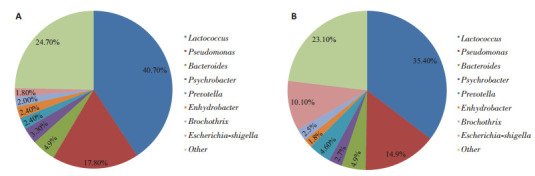
结肠腺瘤高级别上皮内瘤变和低级别上皮内瘤变组肠道菌群属水平的构成
Gut microbial community structure of patients with high-(A) and low-grade (B) intraepithelial neoplasia at the level of genus.
3. 讨论
肠道菌群定居于肠道黏膜表面,构成肠道微生态平衡,对维护机体健康起重要的作用[8]。肠道菌群和结肠癌关系密切,既往有多项研究证实,结肠肿瘤(结肠癌和结肠腺瘤)患者的肠道菌群构成与健康人群存在差异。Marchesi等[9]运用测序技术发现结直肠癌患者肿瘤组织黏膜中具有益生菌特性的肠道共生菌(如Coriobateria)含量很高,而类似大肠杆菌的潜在致病菌低表达。Rubinstein等[10]研究发现具核梭杆菌能仓隨一个促炎的微环境并且通过E-钙粘蛋白和β-catenin信号促进结直肠癌形成。同时研究发现具核梭杆菌在结肠癌组织中高表达,并和微卫星不稳定性有关[11],且癌组织中具核梭杆菌DNA的量可能是一个疾病预后预测因子[12]。Baxter等[13]将正常人和结肠肿瘤患者的粪菌移植给无菌小鼠并诱导肠癌模型,结果发现肿瘤的数目与基线菌群构成密切相关,也就是说菌群的结构决定了肿瘤形成的易感性。目前研究晚期结肠癌和肠道菌群关系的文章相对较多,而菌群与腺瘤关系的研究远没有结肠癌多。Shen等[14]发现腺瘤性息肉患者肠道微生物拟杆菌比例低,变形菌比例高。Goedert等[15]也发现结肠腺瘤患者的粪便菌群与正常人显著不同。
肠道菌群的组成包括粪便菌群和黏膜菌群,黏膜菌群比粪便菌群种类更丰富,相关研究认为粪便细菌主要通过参与宿主代谢引起CRC发生,而黏膜内细菌通过与宿主直接作用促使CRC发生[16]。我们本项研究以黏膜菌群为主,发现健康人中以Firmicutes、Bacteroidetes和Proteobacteria为优势菌门,这与既往的研究相符[17]。反映a多样性的指标,如Shannon指数值腺瘤患者较正常组明显升高,Simpson指数值下降,提示腺瘤患者肠道菌群多样性显著增高,与健康人的菌群相比发生了整体结构失衡的现象。
在门的水平,本研究发现结肠腺瘤患者厚壁菌门明显下降,厚壁菌/拟杆菌门比值下降,而变形菌门比例显著升高。变形菌门属于细菌分类中较大的门,其中的Y-变形菌纲包含了很多重要的已知病原菌或条件致病菌。另外,两组之间有显著差异的门还包括Chloroflexi、Tenericutes和Candidate-division TM7均是口腔常驻菌[18],提示口腔细菌也可能与大肠癌的发生有间接或直接的关系。而最近的一项大规模流行病学调查也发现牙周疾病可能是结直肠癌的一个危险因素[19],需进一步的研究来证实及探讨其机制。
在属的水平,我们发现包括链球菌属,假单胞菌属等条件致病菌在结肠腺瘤患者中明显增加。链球菌属是一个在结肠癌发生过程中起致病作用的菌。体外研究发现牛链球菌细胞壁所分泌的抗原能够通过过表达COX-2来促进Caco-2细胞系的癌变[20]。另外有些研究提示,不仅仅是牛链球菌可能促进肿瘤的生长,肿瘤本身的代谢也为牛链球菌提供了一个高氨基酸,高糖适宜生长的环境[21],两者互相影响,互为因果,共同促进疾病的发展。在腺瘤组中明显升高的假单胞菌属,其中的某些种对人或动物有致病性,有研究发现结肠癌患者术后肠道假单胞菌含量明显增加[22],提示假单胞菌属可能与结肠癌有关。在本项研究中,我们发现在腺瘤组织中乳球菌存在明显的升高,乳球菌是革兰氏阳性菌,以一些碳水化合物为原料发酵产酸。阐述结肠肿瘤发生发展的“driver-passenger”[23]模型学说认为:癌变过程介导了肠道局部微环境的改变,这种改变有利于另外某些细菌的增殖存活,这些细菌被定义为“passengers”,如解没食子酸链球菌亚种、氏菌属等,“passengers”不排除其具有保护肠道内环境的益生菌效应,但也有可能对肿瘤起到促进作用,我们认为乳球菌可能是存在于结肠腺瘤微环境中的“passengers”。而某些特定的肠道固有菌能诱发上皮细胞DNA损伤,这些肠道固有菌被定义为“drivers”,drivers通过不同的机制促进了大肠的癌变过程,如粪肠球菌产生的胞外超氧化物转化为过氧化氢,可引起结肠上皮细胞的DNA损伤[24]。
在研究中观察到Solibacillus属在腺瘤中明显减少。Salibacillus是芽孢杆菌中的一个成员,能产生芽孢,但目前对其作用所知甚少,特别在结肠癌逐渐发生进展过程中该菌的作用研究尚未见文献报道,Solibacillus在结肠黏膜癌变的早期阶段过程中存在明显下降,我们推测其可能扮演有益菌的角色。
通过β多样性分析,PCA研究发现在坐标轴上,代表腺瘤标本的红点和代表对照标本的蓝点完全分开,没有重叠,且导致两组分开的PC1因素达到90.45%。多样本相似度树状图和NMDS分析也获得了类似的结果,该现象提示疾病/健康因素即结肠腺瘤发生是造成肠道黏膜菌群构成改变的主要因素,且该因素占高达90.45%比例,提示结肠腺瘤是导致肠道菌群稳态失衡的主要因素。
对于不同病理类型结肠腺瘤菌群构成的研究非常少,我们研究了结肠低级别和高级别上皮内瘤变的细菌构成,发现虽然两组菌群构成类似,无显著差异,但是埃希菌-志贺菌属在结肠高级别上皮内瘤变组有升高趋势。Collins等[25]发现埃希菌可以产生细胞毒性坏死因子,能诱导宿主基因组的复制,激活RhoGTP酶,从而增加细胞转移活性,提示大肠埃希菌对于肿瘤发生有促进作用。因此局部病变组织在从低级别到高级别瘤变演进过程中,存在某些致病菌的逐渐增多,从而引起肠上皮细胞及肠粘膜的损伤可能是结肠肿瘤动态演变过程中的一个特征。但是本项研究的缺陷是高级别上皮内瘤变组样本量较小,如果进一步增加样本量,同时增加结肠原位癌阶段的菌群分析,更能动态监测肿瘤发生发展过程中不同时期的菌群构成改变,有助于阐明结肠癌发生机制。
综上所述,我们发现结肠腺瘤患者肠道内定植的菌群具有整体结构失衡的特征,这一结构失衡可以看作是肠道微生态平衡紊乱,并且有可能是结直肠癌发生之前的早期事件。虽然结直肠癌发病是多因素的,并不单纯由菌群的活动所引起,但菌群稳态失调可能在结直肠癌发生发展演变过程中发挥重要的作用,并为早期诊断结直肠肿瘤提供重要的线索和参考。
Biography
陆颖影,医学硕士,主治医师,E-mail:le_voyageur@qq.com
Funding Statement
国家自然科学基金(81600500);上海交通大学医工交叉基金(YG2014ZD10)
Supported by National Natural Science Foundation of China (81600500)
Contributor Information
陆 颖影 (Yingying LU), Email: le_voyageur@qq.com.
王 兴鹏 (Xingpeng WANG), Email: richardwangxp@163.com.
References
- 1.Chen W, Zheng R, Baade PD, et al. Cancer statistics in China, 2015. CA Cancer J Clin. 2016;66(2):115–32. doi: 10.3322/caac.21338. [Chen W, Zheng R, Baade PD, et al. Cancer statistics in China, 2015 [J]. CA Cancer J Clin, 2016, 66(2): 115-32.] [DOI] [PubMed] [Google Scholar]
- 2.Parkin DM. International variation. Oncogene. 2004;23(38):6329–40. doi: 10.1038/sj.onc.1207726. [Parkin DM. International variation [J]. Oncogene, 2004, 23(38): 6329-40.] [DOI] [PubMed] [Google Scholar]
- 3.Lieberman DA, Weiss DG, Bond JH, et al. Use of colonoscopy to screen asymptomatic adults for colorectal cancer. N Engl J Med. 2000;343(3):162–8. doi: 10.1056/NEJM200007203430301. [Lieberman DA, Weiss DG, Bond JH, et al. Use of colonoscopy to screen asymptomatic adults for colorectal cancer[J]. N Engl J Med, 2000, 343(3): 162-8.] [DOI] [PubMed] [Google Scholar]
- 4.Winawer S, Fletcher R, Rex D, et al. Colorectal cancer screening and surveillance: clinical guidelines and rationale-Update based on new evidence. Gastroenterology. 2003;124(2):544–60. doi: 10.1053/gast.2003.50044. [Winawer S, Fletcher R, Rex D, et al. Colorectal cancer screening and surveillance: clinical guidelines and rationale-Update based on new evidence[J]. Gastroenterology, 2003, 124(2): 544-60.] [DOI] [PubMed] [Google Scholar]
- 5.Sobhani I, Amiot A, Le Baleur Y, et al. Microbial dysbiosis and colon carcinogenesis: could colon cancer be considered a bacteria-related disease. Therap Adv Gastroenterol. 2013;6(3):215–29. doi: 10.1177/1756283X12473674. [Sobhani I, Amiot A, Le Baleur Y, et al. Microbial dysbiosis and colon carcinogenesis: could colon cancer be considered a bacteria-related disease [J]? Therap Adv Gastroenterol, 2013, 6(3): 215-29.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Sobhani I, Tap J, Roudot-Thoraval F, et al. Microbial dysbiosis in colorectal cancer (CRC) patients. PLoS One. 2011;6(1):e16393. doi: 10.1371/journal.pone.0016393. [Sobhani I, Tap J, Roudot-Thoraval F, et al. Microbial dysbiosis in colorectal cancer (CRC) patients[J]. PLoS One, 2011, 6(1): e16393.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Ahn J, Sinha R, Pei Z, et al. Human gut microbiome and risk for colorectal cancer. JNatl CancerInst. 2013;105(24):1907–11. doi: 10.1093/jnci/djt300. [Ahn J, Sinha R, Pei Z, et al. Human gut microbiome and risk for colorectal cancer[J]. JNatl CancerInst, 2013, 105(24): 1907-11.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Nicholson JK, Holmes E, Wilson ID. Gut microorganisms, mammalian metabolism and personalized health care. Nat Rev Microbiol. 2005;3(5):431–8. doi: 10.1038/nrmicro1152. [Nicholson JK, Holmes E, Wilson ID. Gut microorganisms, mammalian metabolism and personalized health care [J]. Nat Rev Microbiol, 2005, 3(5): 431-8.] [DOI] [PubMed] [Google Scholar]
- 9.Marchesi JR, Dutilh BE, Hall N, et al. Towards the human colorectal cancer microbiome. PLoS One. 2011;6(5):e20447. doi: 10.1371/journal.pone.0020447. [Marchesi JR, Dutilh BE, Hall N, et al. Towards the human colorectal cancer microbiome[J]. PLoS One, 2011, 6(5): e20447.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Rubinstein MR, Wang X, Liu W, et al. Fusobacterium nucleatum promotes colorectal carcinogenesis by modulating E-cadherin/ beta-catenin signaling via its FadA adhesin. Cell Host Microbe. 2013;14(2):195–206. doi: 10.1016/j.chom.2013.07.012. [Rubinstein MR, Wang X, Liu W, et al. Fusobacterium nucleatum promotes colorectal carcinogenesis by modulating E-cadherin/ beta-catenin signaling via its FadA adhesin[J]. Cell Host Microbe, 2013, 14(2): 195-206.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Viljoen KS, Dakshinamurthy A, Goldberg P, et al. Quantitative profiling of colorectal cancer-associated bacteria reveals associations between fusobacterium spp., enterotoxigenic Bacteroides fragilis (ETBF) and clinicopathological features of colorectal cancer. PLoSOne. 2015;10(3):e0119462. doi: 10.1371/journal.pone.0119462. [Viljoen KS, Dakshinamurthy A, Goldberg P, et al. Quantitative profiling of colorectal cancer-associated bacteria reveals associations between fusobacterium spp., enterotoxigenic Bacteroides fragilis (ETBF) and clinicopathological features of colorectal cancer [J]. PLoSOne, 2015, 10(3): e0119462.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Mima K, Nishihara R, Qian ZR, et al. Fusobacterium nucleatum in colorectal carcinoma tissue and patient prognosis. Gut. 2016;65(12):1973–80. doi: 10.1136/gutjnl-2015-310101. [Mima K, Nishihara R, Qian ZR, et al. Fusobacterium nucleatum in colorectal carcinoma tissue and patient prognosis [J]. Gut, 2016, 65 (12): 1973-80.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Baxter NT, Zackular JP, Chen GY, et al. Structure ofthe gut microbiome following colonization with human feces determines colonic tumor burden[Z]. 2014: 20.
- 14.Shen XJ, Rawls JF, Randall T, et al. Molecular characterization of mucosal adherent bacteria and associations with colorectal adenomas. http://pubmedcentralcanada.ca/pmcc/articles/PMC2927011/ Gut Microbes. 2011;1(3):138–47. doi: 10.4161/gmic.1.3.12360. [Shen XJ, Rawls JF, Randall T, et al. Molecular characterization of mucosal adherent bacteria and associations with colorectal adenomas[J]. Gut Microbes, 2011, 1(3): 138-47.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Goedert JJ, Gong Y, Hua X, et al. Fecal microbiota characteristics of patients with colorectal adenoma detected by screening: a population-based study. EBioMedicine. 2015;2(6):597–603. doi: 10.1016/j.ebiom.2015.04.010. [Goedert JJ, Gong Y, Hua X, et al. Fecal microbiota characteristics of patients with colorectal adenoma detected by screening: a population-based study[J]. EBioMedicine, 2015, 2(6): 597-603.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Chen W, Liu F, Ling Z, et al. Human intestinal lumen and mucosa-associated microbiota in patients with colorectal cancer. PLoS One. 2012;7(6):e39743. doi: 10.1371/journal.pone.0039743. [Chen W, Liu F, Ling Z, et al. Human intestinal lumen and mucosa-associated microbiota in patients with colorectal cancer [J]. PLoS One, 2012, 7(6): e39743.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Eckburg PB, Bik EM, Bernstein CN, et al. Diversity of the human intestinal microbial flor. Science. 2005;308(5728):1635–8. doi: 10.1126/science.1110591. [Eckburg PB, Bik EM, Bernstein CN, et al. Diversity of the human intestinal microbial flor[J]. Science, 2005, 308(5728): 1635-8.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Dewhirst FE, Chen T, Izard J, et al. The human oral microbiome. JBacteriol. 2010;192(19):5002–17. doi: 10.1128/JB.00542-10. [Dewhirst FE, Chen T, Izard J, et al. The human oral microbiome [J]. JBacteriol, 2010, 192(19): 5002-17.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Momen-Heravi F, Babic A, Tworoger SS, et al. Periodontal disease, tooth loss and colorectal cancer risk: Results from the Nurses' Health Study. Int J Cancer. 2017;140(3):646–52. doi: 10.1002/ijc.v140.3. [Momen-Heravi F, Babic A, Tworoger SS, et al. Periodontal disease, tooth loss and colorectal cancer risk: Results from the Nurses' Health Study[J]. Int J Cancer, 2017, 140(3): 646-52.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Biarc J, Nguyen IS, Pini A, et al. Carcinogenic properties of proteins with pro-inflammatory activity from Streptococcus infantarius (formerly S.bovis) Carcinogenesis. 2004;25(8):1477–84. doi: 10.1093/carcin/bgh091. [Biarc J, Nguyen IS, Pini A, et al. Carcinogenic properties of proteins with pro-inflammatory activity from Streptococcus infantarius (formerly S.bovis) [J]. Carcinogenesis, 2004, 25(8): 1477-84.] [DOI] [PubMed] [Google Scholar]
- 21.Hirayama A, Kami K, Sugimoto M, et al. Quantitative metabolome profiling of colon and stomach cancer microenvironment by capillary electrophoresis time-of-flight mass spectrometry. Cancer Res. 2009;69(11):4918–25. doi: 10.1158/0008-5472.CAN-08-4806. [Hirayama A, Kami K, Sugimoto M, et al. Quantitative metabolome profiling of colon and stomach cancer microenvironment by capillary electrophoresis time-of-flight mass spectrometry[J]. Cancer Res, 2009, 69(11): 4918-25.] [DOI] [PubMed] [Google Scholar]
- 22.Ohigashi S, Sudo K, Kobayashi D, et al. Significant changes in the intestinal environment after surgery in patients with colorectal cancer. J Gastrointest Surg. 2013;17(9):1657–64. doi: 10.1007/s11605-013-2270-x. [Ohigashi S, Sudo K, Kobayashi D, et al. Significant changes in the intestinal environment after surgery in patients with colorectal cancer[J]. J Gastrointest Surg, 2013, 17(9): 1657-64.] [DOI] [PubMed] [Google Scholar]
- 23.Tjalsma H, Boleij A, Marchesi JR, et al. A bacterial driver-passenger model for colorectal cancer: beyond the usual suspects. Nat Rev Microbiol. 2012;10(8):575–82. doi: 10.1038/nrmicro2819. [Tjalsma H, Boleij A, Marchesi JR, et al. A bacterial driver-passenger model for colorectal cancer: beyond the usual suspects [J]. Nat Rev Microbiol, 2012, 10(8): 575-82.] [DOI] [PubMed] [Google Scholar]
- 24.Wang X, Huycke MM. Extracellular superoxide production by Enterococcus faecalis promotes chromosomal instability in mammalian cells. Gastroenterology. 2007;132(2):551–61. doi: 10.1053/j.gastro.2006.11.040. [Wang X, Huycke MM. Extracellular superoxide production by Enterococcus faecalis promotes chromosomal instability in mammalian cells[J]. Gastroenterology, 2007, 132(2): 551-61.] [DOI] [PubMed] [Google Scholar]
- 25.Collins D, Hogan AM, Winter DC. Microbial and viral pathogens in colorectal cancer. Lancet Oncol. 2011;12(5):504–12. doi: 10.1016/S1470-2045(10)70186-8. [Collins D, Hogan AM, Winter DC. Microbial and viral pathogens in colorectal cancer[J]. Lancet Oncol, 2011, 12(5): 504-12.] [DOI] [PubMed] [Google Scholar]


