Abstract
目的
探索长链非编码RNA MALAT1在皮肤鳞癌发生发展中的作用,为皮肤鳞癌的临床治疗提供基础理论依据。
方法
收集55例皮肤鳞癌样本和10例正常皮肤组织样本,采用qRT-PCR与原位杂交方法检测MALAT1在皮肤鳞癌组织和正常组织中的表达情况。在皮肤鳞癌细胞株A431中,采用siRNAs(siNC, siMALAT1-1, siMALAT1-2)和Lipofectamine2000进行细胞转染来敲减基因,采用Transwell实验,细胞划痕实验,CCK增殖实验检测皮肤鳞癌细胞的迁移侵袭能力,运动能力和增殖能力。采用qRT-PCR方法检测A431中MALAT1与上皮间质转化(EMT)样转化相关因子(E-cadherin, Vimentin, β-catenin)的变化;明确其关系。
结果
相比正常组织,皮肤鳞癌组织的不同分化水平上(高分化,中分化,低分化),MALAT1的表达率均升高(P < 0.001)。在皮肤鳞癌细胞A431中,转染MALAT1 siRNAs下调其表达后,与对照相比,其增殖能力(P < 0.001),迁移能力(P < 0.01),侵袭能力(P < 0.01),细胞划痕运动能力均下降(P < 0.01)。且EMT相关因子中上皮表型标志物E-cadherin,β-catenin的表达升高(P < 0.01),间质表型分子vimentin表达降低(P < 0.01)。
结论
长链非编码RNA MALAT1在皮肤鳞癌的发生发展中起促进作用,可以为皮肤鳞癌的临床治疗提供基础理论依据。
Keywords: LncRNA, MALAT1, 皮肤鳞癌, 上皮间质转化
Abstract
Objective
To investigate the role of long noncoding RNA MALAT1 in the occurrence and progression of cutaneous squamous cell carcinoma (CSCC).
Methods
Fifty-five tissue samples of CSCC and 10 normal epidermal tissues were collected for examination of the expression of MALAT1 using q-PCR and in situ hybridization. Human CSCC A431 cells were transfected with small interfering RNAs (siNC, siMALAT1-1, and siMALAT1-2) using Lipofectamine2000 to knock down MALAT1 gene, and the changes in the cell migration, invasion, mobility and proliferation were analyzed using Transwell assay, wound healing assay, and CCK-8 assay; the changes in the expressions of the related factors of epithelial-mesenchymal transition (EMT), including E-cadherin, vimentin, and β-catenin, were detected using qRT-PCR.
Results
Compared with normal tissues, CSCC tissues of different grades of differentiation all showed significantly increased expression of MALAT1 (P < 0.001). In A431 cells, MALAT1 knockdown with siRNAs resulted in significantly lowered cell proliferation (P < 0.001), migration (P < 0.01), invasion (P < 0.01), and mobility (P < 0.01). Knocking down MALAT1 gene also caused significantly increased expressions of E-cadherin and β-catenin (P < 0.01) and lowered the expression of vimentin (P < 0.01) in A431 cells.
Conclusion
The long noncoding RNA MALAT1 promotes the occurrence and progression of CSCC and can potentially serve as a therapeutic target in treatment of CSCC.
Keywords: long noncoding RNA, MALAT1, cutaneous squamous cell carcinoma, epithelial-mesenchymal transition
皮肤鳞癌(SCC)是起源于角质形成细胞的一种恶性肿瘤,是仅次于基底细胞癌的第2常见的皮肤恶性肿瘤[1]。引起发病的原因有:紫外线照射、电离辐射、化学致癌物以及慢性刺激等,其中主要原因是日光紫外线的照射[2-3]。据报道,在世界范围内,皮肤鳞癌的发病率在逐年递增[4]。SCC的发病率在我国并不高,但是患病后,极大的影响患者的生存质量[5]。长链非编码RNA(LncRNA)是一类转录本长度大于200个核苷酸、不能编码产生蛋白质、没有明显开放阅读框的、具有基因表达调节功能的一类RNA分子。许多研究表明,在肿瘤细胞中,LncRNA通过调节其增殖、迁移等生物学功能从而影响肿瘤的发生发展[6]。
人类肺腺癌转移相关转录本1(MALAT1),又称为核富集常染色体转录产物2(NEAT2),长度约为8000 nt,定位于染色体11q13.1,是2003年首次报道的一种LncRNA[7]。据文献报道,MALAT1基因与多种肿瘤的发生、发展及转移密切相关[8-9]。但是在皮肤鳞癌中,关于MALAT1的功能和分子机制方面的报道很少。
本文通过检测皮肤鳞癌组织中MALAT1的表达情况,以及在MALAT1沉默后A431的增殖、迁移、侵袭等能力的变化,明确MALAT1与EMT样相关转化因子的关系,从而研究长链非编码RNA MALAT1在皮肤鳞癌中的表达情况及其在皮肤鳞癌的发展过程中所起的作用。
1. 资料和方法
1.1. 材料
55例皮肤鳞状细胞癌组织取自已经确诊且病例资料完整的的病理组织(2009年1月~2016年12月于南方医科大学南方医院、珠江医院、广州市增城区人民医院、广州市武警医院及佛山市南海区人民医院),10例正常皮肤组织样本取自南方医院整形外科手术室。
A431(人皮肤鳞状细胞癌细胞株)购自上海中国科学院典型培养物保藏委员会细胞库。
本研究由南方医科大学南方医院医学伦理委员会批准,所有患者均签署知情同意书。
1.2. 方法
1.2.1. 细胞培养
A431细胞用完全DMEM培养基(加入血清与青霉素+链霉素)培养,放在37 ℃,5% CO2培养箱中,当其处于指数生长期进行实验。
1.2.2. 细胞转染
使用小干扰RNAs(siNC,siMALAT1-1,siMALAT1-2)和Lipofectamine 2000沉默MALAT1。即前1 d种皿,等到次日细胞汇合度达到70%~80%用siNC、siMALAT1-1与siMALAT1-2和Lipofectamine 2000一起进行转染。
1.2.3. CCK-8增殖实验
A431细胞前1 d种皿于96孔板中,次日汇合度达到70%~80%进行转染,3~5 h后换为完全DMEM培养基,记为0 h。然后分别检测0,12,24,48,72 h时细胞的增值能力。在每个时间点每孔细胞中加入CCK-8Solution,避光放在培养箱2 h后,用酶标仪测定450 nm处的吸光度值,记录。
1.2.4. 原位杂交
制备探针,将组织样品进行石蜡包埋,将包埋过的切片用HCL室温孵育5 min,PBS洗3遍,5 min/次;蛋白酶K孵育20 min,PBS洗3遍,5 min/次;多聚甲醛孵育10 min,PBS洗2次,5 min/次;醋酸酐和三乙醇胺溶液室温孵育10 min,PBS洗2次,5 min/次。用无探针预杂交液37 ℃杂交2 h;杂交液稀释过的探针85 ℃变性5 min,加入组织,37 ℃密封过夜;SSC缓冲液中洗2次,20 min/次;随后用甲酰胺/SSC缓冲液37 ℃漂洗3次,20 min/次;最后用TBST漂洗5次,10 min/次;在3% BSA中,室温封闭1h;加入抗地高辛一抗(1:1000)4 ℃孵育过夜;TBST洗4次,10 min/次;BCIP/NBT染色液缓冲液洗涤2次,10 min/次;BCIP/NBT暗处染色4~ 48 h;TE缓冲液(pH8.0)洗涤2次后终止反应;水洗残液,脱水、封片;倒置显微镜下观察,拍照。
1.2.5. Transwell迁移与侵袭
前1 d种皿,次日汇合度达到70%~80%,用siRNAs和Lipofectamine 2000进行转染,24 h后收集细胞,计数,用无血清DMEM培养基重悬,按2×105/400 mL/孔细胞接种于Transwell小室,下面孔室放完全DMEM培养基,放再37 ℃,5% CO2培养箱中12~16 h,一出培养基,预冷PBS洗涤2次,甲醛固定2 min,移除,PBS洗涤2次,100%甲醇进行细胞透化固定,室温孵育20 min,移除,PBS洗涤两次0.1%结晶紫染色孵育15 min,PBS洗涤2次,用棉棒清除小室内未迁移的细胞,晾干细胞,×200镜下计数迁移到小室底面的细胞。观察5个高倍视野,计数迁移细胞平均数,每组实验设置3个复孔。
1.2.6. 细胞划痕实验
计数细胞,按总体积1 mL/孔,5× 105~1×106/孔种皿于6孔板中,次日汇合度达到70%~ 80%时用siRNAs+Lipofectamine 2000进行转染,3~5 h后换为完全DMEM培养基,记为起始时间。在6孔板后面划3条平行直线和一条垂直直线,转染12 h后进行划痕,PBS洗涤2次,去除划下的细胞,加入DMEM培养基,记为拍照时间0 h,置于培养箱中培养,分别在0,12,24,30 h在倒置显微镜下拍照。×100镜下计算细胞运动的面积。观察5个高倍视野,计算细胞运动面积,每组实验设置3个复孔。使用Image J软件进行数据的分析。
1.2.7. qRT-PCR
前1 d种皿,次日汇合度达到70%~ 80%,进行细胞转染。48 h后提取RNA,然后进行qRTPCR反应,使RNA逆转录为cDNA,然后进行实时荧光定量PCR反应。
1.2.8. 统计分析
所有结果均用SPSS 21.0统计软件进行分析。两组样本组间比较采用双侧独立样本t检验;三组样本组间比较采用单因素方差分析检验。每组样本均进行方差齐性检验。组间多重比较,若方差齐,采用方差分析中LSD方法,方差不齐,则采取Dunnett's T3方法。所有数据以均数±标准差表示。
2. 结果
2.1. 正常组织和皮肤鳞癌组织中MALAT1的表达情况
qRT-PCR用来检测正常皮肤组织和皮肤鳞癌组织中MALAT1的表达情况。结果显示:与正常组织相比,皮肤鳞癌组织中MALAT1的表达显著升高(图 1)提示MALAT1在皮肤鳞癌中高表达。
1.
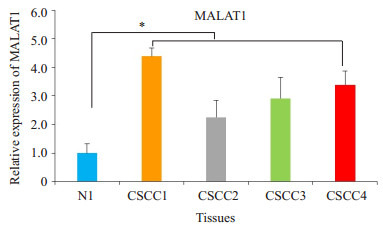
正常组织和皮肤鳞癌组织中MALAT1的RNA表达情况
Up-regulation of MALAT1 expression in cutaneous squamous cell carcinoma. N1: Nnormal epidermal tissues; CSCC: CSCC tissues. *P < 0.05
2.2. 原位杂交结果显示皮肤鳞癌组织中MALAT1的阳性表达率高于正常组织
本实验共收集55例皮肤鳞癌样本和10例正常组织样本,其中皮肤鳞癌组织中有高分化18例,中分化26例,低分化11例。使用原位杂交方法分别检测正常皮肤组织和不同分化程度的皮肤鳞癌组织中MALAT1的达情况。结果显示:正常组织中,MALAT1仅表达于表皮细胞中,而不同分化的皮肤鳞癌组织中则满视野,如图 2。且与正常组织(13.6%)相比,皮肤鳞癌组织中高分化(52.78%),中分化(40.42%),低分化(62.18%)中MALAT1阳性表达率均高于正常组织,差异具有统计学意义。
2.

正常组织和不同分化程度的皮肤鳞癌组织中MALAT1的表达情况
Expression of MALAT1 in normal tissues and CSCC tissues (Original magnification: ×200). **P < 0.01, ***P < 0.001
2.3. 建立的沉默MALAT1模型
为了研究MALAT1在皮肤鳞癌的生物学作用,我们在A431细胞中,采用siRNAs(siMALAT1-1与siMALAT1-2)和Lipofectamine2000进行基因的敲减,然后用qRT-PCR的方法检测沉默模型是否成功(图 3)。
3.

采用qRT-PCR检测MALAT1的敲减情况
Expression of MALAT1 in A431 cells with MALAT1 knockdown assessed by qRT-PCR. **P < 0.01, ***P < 0.001
2.4. 下调MALAT1的表达后抑制A431细胞的运动能力
细胞划痕实验用来检测MALAT1沉默后A431细胞的运动能力。结果显示:与阴性对照转染siNC相比,转染siMALAT1-1与siMALAT1-2的运动能力明显降低。在24 h时,siNC愈合率达到56%,而siMALAT1-1与siMALAT1-2愈合率约为27%;在30 h,siNC的愈合率达到68%,而siMALAT1-1与siMALAT1-2的愈合率仅达到35%~47%。采用单因素方差分析方法进行统计分析,与siNC组相比,siMALAT1-1沉默组组间具有统计学差异(P < 0.001),siMALAT1-2沉默组组间具有统计学差异(P < 0.01,图 4)。
4.

伤口愈合实验检测转染MALAT1 siRNAs后A431细胞运动能力情况
Mobility of A431 cells transfected with siMALAT1s assessed using wound healing assay (×100). **P < 0.01, ***P < 0.001
2.5. 下调MALAT1表达后能抑制A431细胞的迁移与侵袭能力
Transwell小室迁移实验和侵袭实验是为了检测用siRNAs下调MALAT1的表达后对皮肤鳞癌细胞体外迁移能力与侵袭能力的影响。迁移实验和侵袭实验结果显示:与阴性对照siNC相比,转染siMALAT1-1与siMALAT1-2的A431细胞结晶紫染色的数量显著降低。采用单因素方差分析方法进行统计分析,与siNC相比,实验组均明显降低且结果具有统计学意义(图 5、6)。提示MALAT1在皮肤鳞癌细胞中与癌细胞的迁移与侵袭息息相关。
5.

Transwell基质胶小室迁移实验分析下调MALAT1的表达后A431细胞迁移能力的变化
Migration ability of A431 cells transfected with siMALAT1s assessed using Transwell assay (×200). **P < 0.01
6.

Transwell基质胶小室侵袭实验分析下调MALAT1的表达后A431细胞侵袭能力的变化
Invasiveness of A431 cells transfected with siMALAT1s assessed using Transwell assay (×200). **P < 0.01
2.6. 下调MALAT1的表达影响A431细胞的增殖能力
为了研究MALAT1基因对皮肤鳞癌增殖的影响。我们用siRNAs和Lipofectamine 2000进行Malat1基因的敲减。采用Cell Counting Kit-8方法进行其增殖能力的分析结果显示:与对照组siNC相比,转染siMALAT1-1和siMALAT1-2的实验组增值能力降低(***P < 0.001,图 7)。
7.

CCK-8检测siRNAs处理A431细胞后增殖能力情况
Proliferation of A431 cells transfected with siMALAT1s assessed using CCK-8 kit. ***P < 0.001
2.7. qRT-PCR结果显示MALAT1与EMT之间可能密切相关
在A431细胞中下调MALAT1表达后,检测了EMT中的代表性基因E-cadherin,β-catenin,vimentin的表达情况。结果显示:与阴性对照siNC相比,下调A431细胞中的MALAT1的表达后,EMT的上皮样表型标志物E-cadherin,β-catenin的表达明显升高;而间质表型分子vimentin的表达明显降低。所有数据采用单因素方差分析的方法进行统计分析,E-cadherin(P < 0.01);β-catenin(P < 0.01);vimentin(P < 0.01)差异均具有统计学意义(图 8)。
8.
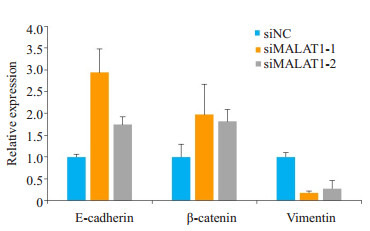
采用qRT-PCR检测A431中MALAT1敲减后E-cadherin、β-catenin与vimentin的表达情况
Expression of E-cadherin、β-catenin and vimentin after transfection of siNC、siMALAT1-1and siMALAT1-2 in A431 cells
3. 讨论
MALAT1最早是在非小细胞肺癌的研究中发现。Bernard等进行荧光杂交发现其具有特殊的亚细胞定位,即定位在核小斑结构中[10]。MALAT1特殊的亚细胞定位、特异性招募SR蛋白家族成员以及其在多种肿瘤细胞中异常表达的特点,可以猜测在肿瘤学中其可能发挥了重要的作用[11]。MALAT1在多种肿瘤组织中表达量升高[12]。同时,MALAT1对肿瘤细胞生物学行为的调控已得到较多研究者证实。在胃癌中,其可能通过miR-122-IGF-1R信号通路促进胃癌细胞增殖、迁移、侵袭以及抑制凋亡等[13]。在宫颈癌中,MALAT1表达升高,促进肿瘤细胞的增殖、迁移与侵袭[14]。在非小细胞肺癌[15]、前列腺癌[16]、多发性骨髓瘤[17]、宫颈癌[18]、胶质瘤[19]中,MALAT1与癌症临床分期及预后相关。可见,MALAT1在多种肿瘤细胞中表达上调,起着一个癌基因的作用,与肿瘤的发生发展密切相关。
我们采用qRT-PCR和原位杂交的方法检测了皮肤鳞癌组织与正常皮肤组织中MALAT1的表达情况以及在MALAT1沉默后A431的增殖、迁移、侵袭等能力的变化。结果表明:在皮肤鳞癌组织中MALAT1高表达;对MALAT1进行敲减后,A431细胞的运动能力、增殖能力、迁移能力与侵袭能力降低。由此可见,MALAT1在皮肤鳞癌中高表达并起到一个促进细胞增殖、迁移、侵袭与运动能力的作用。MALAT1在皮肤鳞癌中的促癌作用的这个结果与其在其他的肿瘤中的研究结果一致。
EMT是在特定的生理或病理的情况下,上皮细胞特性与间质细胞特性发生转变的一系列生物学现象。主要特征有:E-cadherin、β-catenin等的表达降低、vimentin等的表达升高以及细胞骨架重构,从而使细胞-细胞间的连接丧失,黏附力减弱, 促进细胞迁移[20-21]。在肺腺癌中,MALAT1是高表达的,其可能通过miR-204介导从而促进肺腺癌细胞EMT样转化和转移[22]。在乳腺癌中,MALAT1可通过PI3K/Akt途径调节EMT,促进肿瘤细胞的迁移与侵袭[23]。在膀胱癌[24]中,MALAT1能促进上皮间质转化。EMT与肿瘤细胞的迁移侵袭能力息息相关, 是肿瘤迁移与侵袭的关键因素之一[25-26]。我们在下调MALAT1的表达后, 采用qRT-PCR的方法检测了E-钙黏蛋白、β-连环素以及波形蛋白的表达情况。结果发现:上皮表型标志物E-cadherin、β-catenin表达升高,间质表型标志物vimentin表达降低。提示:MALAT1可能通过EMT从而促进肿瘤细胞的迁移与侵袭。
综上所述,我们探讨了MALAT1在皮肤鳞癌中的生物学作用及影响因素。可以明确,MALAT1在皮肤鳞癌的发生发展中起着一个正向调控的作用且与EMT息息相关。但是这个调控作用具体是如何发挥呢,目前关于MALAT1在皮肤鳞癌中的分子机制研究还很少,需要进一步研究来探讨。在临床应用上,基于本文的研究结果,我们希望研制出能快速、灵敏地检测MALAT1的试剂盒,从而使MALAT1可以作为皮肤鳞癌癌前诊断的标志物以及临床治疗和预后评估的靶点。
Biography
李姗姗,硕士研究生,E-mail: 765024107@qq.com
Funding Statement
国家自然科学基金(81573076,81472922,81673105)
Supported by National Natural Science Foundation of China (81573076, 81472922, 81673105)
Contributor Information
李 姗姗 (Shanshan LI), Email: 765024107@qq.com.
丁 振华 (Zhenhua DING), Email: dingzh@smu.edu.cn.
References
- 1.Fransen M, Karahalios E, Sharma N, et al. Non melanoma skin cancer in Australia. https://www.ncbi.nlm.nih.gov/pubmed/23163687. Austra J Dermatol. 2012;53(1, SI):7. doi: 10.5694/mja12.10654. [Fransen M, Karahalios E, Sharma N, et al. Non melanoma skin cancer in Australia[J]. Austra J Dermatol, 2012, 53(1, SI): 7.] [DOI] [PubMed] [Google Scholar]
- 2.Chen AC, Halliday GM, Damian DL. Non-melanoma skin cancer:carcinogenesis and chemoprevention. Pathology. 2013;45(3):331–41. doi: 10.1097/PAT.0b013e32835f515c. [Chen AC, Halliday GM, Damian DL. Non-melanoma skin cancer:carcinogenesis and chemoprevention[J]. Pathology, 2013, 45(3):331-41.] [DOI] [PubMed] [Google Scholar]
- 3.Andersen LK, Davis MD. Sex differences in the incidence of skin and skin-related diseases in Olmsted County, Minnesota, United States, and a comparison with other rates published worldwide. Int J Dermatol. 2016;55(9):939–55. doi: 10.1111/ijd.2016.55.issue-9. [Andersen LK, Davis MD. Sex differences in the incidence of skin and skin-related diseases in Olmsted County, Minnesota, United States, and a comparison with other rates published worldwide[J].Int J Dermatol, 2016, 55(9): 939-55.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Perera E, Gnaneswaran N, Staines C, et al. Incidence and prevalence of non-melanoma skin cancer in Australia: A systematic review. Australas J Dermatol. 2015;56(4):258–67. doi: 10.1111/ajd.12282. [Perera E, Gnaneswaran N, Staines C, et al. Incidence and prevalence of non-melanoma skin cancer in Australia: A systematic review[J]. Australas J Dermatol, 2015, 56(4): 258-67.] [DOI] [PubMed] [Google Scholar]
- 5.林 春玲, 赵 延明, 牛 云彤. 68例皮肤恶性肿瘤与癌前病变的临床分析. http://d.old.wanfangdata.com.cn/Periodical/syazzz201706048. 实用癌症杂志. 2017;32(6):1031–3. [林春玲, 赵延明, 牛云彤. 68例皮肤恶性肿瘤与癌前病变的临床分析[J].实用癌症杂志, 2017, 32(6): 1031-3.] [Google Scholar]
- 6.顾 姣姣, 严 威, 王 源源, et al. 长链非编码RNA BANCR在肿瘤中的研究进展. 现代肿瘤医学. 2017;25(12):1997–2000. doi: 10.3969/j.issn.1672-4992.2017.12.037. [顾姣姣, 严威, 王源源, 等.长链非编码RNA BANCR在肿瘤中的研究进展[J].现代肿瘤医学, 2017, 25(12): 1997-2000.] [DOI] [Google Scholar]
- 7.Wu Y, Huang C, Meng X, et al. Long noncoding RNA MALAT1:insights into its biogenesis and implications in human disease. Curr Pharm Des. 2015;21(34):5017–28. doi: 10.2174/1381612821666150724115625. [Wu Y, Huang C, Meng X, et al. Long noncoding RNA MALAT1:insights into its biogenesis and implications in human disease[J]. Curr Pharm Des, 2015, 21(34): 5017-28.] [DOI] [PubMed] [Google Scholar]
- 8.Wang S, Ma G, Zhu H, et al. miR-107 regulates tumor progression by targeting NF1 in gastric cancer. Sci Rep. 2016;6:36531. doi: 10.1038/srep36531. [Wang S, Ma G, Zhu H, et al. miR-107 regulates tumor progression by targeting NF1 in gastric cancer [J]. Sci Rep, 2016, 6: 36531.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Yamakuchi M, Lotterman CD, Bao C, et al. P53-induced microRNA-107 inhibits HIF-1 and tumor angiogenesis. Proc Natl Acad Sci USA. 2010;107(14):6334–9. doi: 10.1073/pnas.0911082107. [Yamakuchi M, Lotterman CD, Bao C, et al. P53-induced microRNA-107 inhibits HIF-1 and tumor angiogenesis[J]. Proc Natl Acad Sci USA, 2010, 107(14): 6334-9.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Bernard D, Prasanth KV, Tripathi V, et al. A long nuclear-retained non-coding RNA regulates synaptogenesis by modulating gene expression. EMBO J. 2010;29(18):3082–93. doi: 10.1038/emboj.2010.199. [Bernard D, Prasanth KV, Tripathi V, et al. A long nuclear-retained non-coding RNA regulates synaptogenesis by modulating gene expression[J]. EMBO J, 2010, 29(18): 3082-93.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.王 俊青, 任 建敏, 张 彦洁, et al. MALAT1生物学功能及其与炎症-肿瘤的关系. http://www.docin.com/p-621656142.html. 生命科学. 2012;21(10):1163–8. [王俊青, 任建敏, 张彦洁, 等. MALAT1生物学功能及其与炎症-肿瘤的关系[J].生命科学, 2012, 21(10): 1163-8.] [Google Scholar]
- 12.白 茹梦, 王 朝霞. 长非编码RNA MALAT1在肿瘤研究中的新进展. http://www.doc88.com/p-1334936490115.html. 临床肿瘤学杂志. 2016;21(12):1139–45. [白茹梦, 王朝霞.长非编码RNA MALAT1在肿瘤研究中的新进展[J].临床肿瘤学杂志, 2016, 21(12): 1139-45.] [Google Scholar]
- 13.Xia H, Chen Q, Chen Y, et al. The lncRNA MALAT1 is a novel biomarker for gastric cancer metastasis. http://pubs.sciepub.com/jcrt/1/2/2/ Oncotarget. 2016;7(35):56209–18. doi: 10.18632/oncotarget.10941. [Xia H, Chen Q, Chen Y, et al. The lncRNA MALAT1 is a novel biomarker for gastric cancer metastasis[J]. Oncotarget, 2016, 7(35): 56209-18.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Zhang Y, Wang T, Huang HQ, et al. Human MALAT-1 long noncoding RNA is overexpressed in cervical cancer metastasis and promotes cell proliferation, invasion and migration. http://www.ncbi.nlm.nih.gov/pubmed/27313790. J BUON. 2015;20(6):1497–503. [Zhang Y, Wang T, Huang HQ, et al. Human MALAT-1 long noncoding RNA is overexpressed in cervical cancer metastasis and promotes cell proliferation, invasion and migration[J]. J BUON, 2015, 20(6): 1497-503.] [PubMed] [Google Scholar]
- 15.Liu MJ, Sun WL, Liu YQ, et al. The role of lncRNA MALAT1 in bone metastasis in patients with non-small cell lung cancer. Oncol Rep. 2016;36(3):1679–85. doi: 10.3892/or.2016.4909. [Liu MJ, Sun WL, Liu YQ, et al. The role of lncRNA MALAT1 in bone metastasis in patients with non-small cell lung cancer[J]. Oncol Rep, 2016, 36(3): 1679-85.] [DOI] [PubMed] [Google Scholar]
- 16.Aiello A, Bacci L, Re A, et al. MALAT1 and HOTAIR long noncoding RNAs play opposite role in estrogen-mediated transcriptional regulation in prostate cancer cells. Sci Rep. 2016;6:38414. doi: 10.1038/srep38414. [Aiello A, Bacci L, Re A, et al. MALAT1 and HOTAIR long noncoding RNAs play opposite role in estrogen-mediated transcriptional regulation in prostate cancer cells[J]. Sci Rep, 2016, 6: 38414.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Cho SF, Chang YC, Chang CS, et al. MALAT1 long non-coding RNA is overexpressed in multiple myeloma and May serve as a marker to predict disease progression. BMC Cancer. 2014;14(1):809. doi: 10.1186/1471-2407-14-809. [Cho SF, Chang YC, Chang CS, et al. MALAT1 long non-coding RNA is overexpressed in multiple myeloma and May serve as a marker to predict disease progression[J]. BMC Cancer, 2014, 14(1): 809.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Lu H, He Y, Lin L, et al. Long non-coding RNA MALAT1 modulates radiosensitivity of HR-HPV + cervical cancer via sponging miR-145. Tumour Biol. 2016;37(2):1683–91. doi: 10.1007/s13277-015-3946-5. [Lu H, He Y, Lin L, et al. Long non-coding RNA MALAT1 modulates radiosensitivity of HR-HPV + cervical cancer via sponging miR-145[J]. Tumour Biol, 2016, 37(2): 1683-91.] [DOI] [PubMed] [Google Scholar]
- 19.Feng TB, Shao F, Wu QY, et al. miR-124 downregulation leads to breast cancer progression via LncRNA-MALAT1 regulation and CDK4/E2F1 signal activation. https://link.springer.com/article/10.1007/s13277-016-5386-2. Oncotarget. 2016;7(13):16205–16. doi: 10.18632/oncotarget.7578. [Feng TB, Shao F, Wu QY, et al. miR-124 downregulation leads to breast cancer progression via LncRNA-MALAT1 regulation and CDK4/E2F1 signal activation[J]. Oncotarget, 2016, 7(13): 16205-16.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Tiwari N, Gheldof A, Tatari M, et al. EMT as the ultimate survival mechanism of cancer cells. Semin Cancer Biol. 2012;22(3):194–207. doi: 10.1016/j.semcancer.2012.02.013. [Tiwari N, Gheldof A, Tatari M, et al. EMT as the ultimate survival mechanism of cancer cells[J]. Semin Cancer Biol, 2012, 22(3):194-207.] [DOI] [PubMed] [Google Scholar]
- 21.Thiery JP, Acloque H, Huang RY, et al. Epithelial-Mesenchymal transitions in development and disease. Cell. 2009;139(5):871–90. doi: 10.1016/j.cell.2009.11.007. [Thiery JP, Acloque H, Huang RY, et al. Epithelial-Mesenchymal transitions in development and disease[J]. Cell, 2009, 139(5): 871-90.] [DOI] [PubMed] [Google Scholar]
- 22.Li J, Wang J, Chen Y, et al. LncRNA MALAT1 exerts oncogenic functions in lung adenocarcinoma by targeting miR-204. http://www.ncbi.nlm.nih.gov/pmc/articles/PMC4889723/figure/fig04/ Am J Cancer Res. 2016;6(5):1099–107. [Li J, Wang J, Chen Y, et al. LncRNA MALAT1 exerts oncogenic functions in lung adenocarcinoma by targeting miR-204[J]. Am J Cancer Res, 2016, 6(5): 1099-107.] [PMC free article] [PubMed] [Google Scholar]
- 23.Xu S, Sui S, Zhang J, et al. Downregulation of long noncoding RNA MALAT1 induces epithelial-to-mesenchymal transition via the PI3K-AKT pathway in breast cancer. https://www.ncbi.nlm.nih.gov/pubmed/26191181. Int J Clin Exp Pathol. 2015;8(5):4881–91. [Xu S, Sui S, Zhang J, et al. Downregulation of long noncoding RNA MALAT1 induces epithelial-to-mesenchymal transition via the PI3K-AKT pathway in breast cancer[J]. Int J Clin Exp Pathol, 2015, 8(5): 4881-91.] [PMC free article] [PubMed] [Google Scholar]
- 24.Han Y, Liu Y, Zhang H, et al. Hsa-miR-125b suppresses bladder cancer development by down-regulating oncogene SIRT7 and oncogenic long non-coding RNA MALAT1. FEBS Lett. 2013;587(23):3875–82. doi: 10.1016/j.febslet.2013.10.023. [Han Y, Liu Y, Zhang H, et al. Hsa-miR-125b suppresses bladder cancer development by down-regulating oncogene SIRT7 and oncogenic long non-coding RNA MALAT1[J]. FEBS Lett, 2013, 587(23): 3875-82.] [DOI] [PubMed] [Google Scholar]
- 25.苏 云涛, 许 妍洁, 仇 金荣, et al. 上皮间质转化分子标记物在乳腺浸润性导管癌组织中的表达及临床意义. http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=lczlxzz201206007. 临床肿瘤学杂志. 2012;17(6):505–10. [苏云涛, 许妍洁, 仇金荣, 等.上皮间质转化分子标记物在乳腺浸润性导管癌组织中的表达及临床意义[J].临床肿瘤学杂志, 2012, 17(6): 505-10.] [Google Scholar]
- 26.Amemiya H, Menolascino F, Peña A. Role of the expression of cMet receptor in the progression of gastric cancer. http://www.mdpi.com/1422-0067/14/9/18056/notes. Invest Clin. 2010;51(3):369–80. [Amemiya H, Menolascino F, Peña A. Role of the expression of cMet receptor in the progression of gastric cancer[J]. Invest Clin, 2010, 51(3): 369-80.] [PubMed] [Google Scholar]


