Abstract
目的
分析甲基转移酶SETD2表达改变对人鼻咽癌(NPC)细胞中蛋白表达谱的影响并富集差异信号通路。
方法
提取SETD2基因敲除细胞株CNE1SETD2-KO和野生型细胞CNE1WT的总蛋白,利用Tandem Mass Tag(TMT)标记蛋白质定量技术和串联质谱分析技术筛选出差异表达蛋白质,应用GO数据库对差异表达蛋白质进行注释和富集,应用KEGG数据库进行差异蛋白质涉及信号通路富集。
结果
以表达倍数(FC)≥1.2倍且P < 0.05为筛选标准,SETD2基因敲除的CNE1细胞鉴别出2049个差异表达蛋白质,其中904个表达上调,1145个表达下调。GO功能注释结果表明,SETD2敲除后在生物过程(细胞过程及调节,细胞运动、代谢过程及细胞组分的生物合成)、分子功能(催化活性及分子结合、转录因子活性)和细胞组分(胞膜、细胞器、大分子复合物)等具有特征性。KEGG信号富集结果表明SETD2敲除主要影响与肿瘤关系密切的信号有MAPK、PI3K-Akt、Ras、Rap1、mTOR、Hippo、HIF-1、Wnt、AMPK、FoxO、ErbB、p53、JAK-STAT等。
结论
SETD2敲除后显著改变了NPC细胞中蛋白质表达特征并影响多条与肿瘤关系密切的信号通路,研究结果为NPC的发病机制和治疗标靶筛选提供了参考。
Keywords: 鼻咽癌, SETD2, TMT标记定量蛋白组学, 生物信息学
Abstract
Objective
To analyze the effects of alterations in the expressions of methyltransferase SETD2 on protein expression profiles in human nasopharyngeal carcinoma (NPC) cells and enrich the differential signaling pathways.
Methods
The total protein was extracted from SETD2-knockout cell line CNE1SETD2-KO and the wild-type cell line CNE1WT, and the differentially expressed proteins were screened by tandem mass tag (TMT) labeled protein quantification technique and tandem mass spectrometry. GO analysis was used to annotate and enrich the differentially expressed proteins, and the KEGG database was used to enrich and analyze the pathways of the differential proteins.
Results
With a fold change (FC)≥1.2 and P < 0.05 as the screening standard, 2049 differentially expressed proteins were identified in CNE1SETD2-KO cells, among which 904 were up-regulated and 1145 were down-regulated. GO functional annotation results indicated that SETD2 knockout caused characteristic changes in multiple biological processes (cell processes and regulation, cell movement, metabolic processes, and biosynthesis of cellular components), molecular functions (catalytic activity and molecular binding, transcription factor activity), and cellular components (cell membrane, organelle, macromolecular complex). KEGG analysis showed that the differentially expressed proteins were involved in an array of signaling pathways closely related to tumors, including MAPK, PI3K-Akt, Ras, Rap1, mTOR, Hippo, HIF-1, Wnt, AMPK, FoxO, ErbB, P53 and JAK-STAT.
Conclusion
SETD2 knockout significantly changes the protein expression characteristics of NPC cells and affects a number of signal pathways closely related to tumors. The results provide evidence for investigation of the pathogenesis and therapeutic target screening of NPC.
Keywords: nasopharyngeal carcinoma, SETD2, TMT-tagged quantitative proteomics, bioinformatics
鼻咽癌(NPC)是起源于鼻咽粘膜上皮细胞的恶性肿瘤,具有独特的流行病学特征。我国华南五省尤以广东省是世界范围内的高发地区,俗称“广东癌”。由于NPC发病部位的特殊性,早期症状不明显,大部分患者在确诊时已有颈部淋巴结转移,已处于临床中晚期[1]。得益于诊疗技术的革新,现今早期NPC患者的5年生存率大于90%,但中晚期患者5年生存率仍不高[2-4]。因此,寻找合适的生物标志物以提高NPC的早期诊断、治疗效果评价与预后分析具有极其重要的意义。近来,基于宏观角度的组学如蛋白组学、基因组学等研究可能为此提供潜在的方案。
SETD2蛋白又名Huntington Interacting Protein B、P23、1HBP、HIF-1、KMT3A或HYPB,它的编码基因位于第三号染色体的3p21.31区域。SETD2蛋白是一个280 kDa、含有SET结构域的组蛋白H3K36特异性三甲基转移酶[5]。在人体中,SETD2是H3K36唯一的三甲基转移酶,参与调节H3K36三甲基化(H3K36me3)过程。新近研究表明,在人类多种肿瘤等中存在SETD2基因突变[6-10],这种突变是SETD2表达下调的原因之一。有关SETD2与NPC的报道罕见。赵利刚等[11]发现,相对于鼻咽粘膜慢性炎组织,临床NPC组织中SETD2 mRNA的含量显著下降,且随着淋巴结转移分级升高而降低,表明SETD2表达下调可能与NPC的发展有关。该报道未探讨SETD2在NPC中的确切生物学功能及机制。我们前期基于CRISPR/Cas9技术构建了SETD2基因稳定敲除的NPC细胞株,发现SETD2表达缺失促进NPC细胞的增殖,改变细胞周期进展,凸显了NPC中SETD2具有重要的生物学功能[12]。迄今,SETD2表达缺失对NPC生物学功能及其作用机制等相关的信号通路的调控尚不清楚。
蛋白质组学作为一种高通量筛选方法,近来在人类疾病相关研究中越来越受到关注[13-14]。有关蛋白质组学在NPC相关标志物的鉴定和发病机制的阐明上已得到重视[15-16]。本研究拟采用串联质谱标签(Tandem Mass Tag,TMT)标记的蛋白质定量技术和质谱分析技术,检测SETD2表达改变前后NPC细胞中蛋白质表达谱,表征NPC中SETD2诱导的差异蛋白,结合生物信息学GO和KEGG分析来探究在差异蛋白涉及的功能、分布及参与的肿瘤相关信号通路,以期从宏观角度上发现SETD2表达改变涉及的差异蛋白标靶,为阐明NPC的发病机制和评估NPC治疗及预后靶点提供新的思路和方法。
1. 材料和方法
1.1. 细胞系、主要试剂和仪器
野生型NPC细胞系CNE1WT及SETD2稳定敲除细胞株CNE1SETD2-KO由广东医科大学基础医学院病理学系实验室保存;RPMI 1640培养基、胎牛血清(Hyclone,美国);HCOONH4、三氟乙酸、二硫苏糖醇、碘乙酰胺、NH3· H2O、C18 EmporeTM固相萃取圆盘、NH4HCO3(Sigma,美国);乙腈(Merck,德国);C18 Cartridge(Waters,美国);Lysing Matrix A(MP,美国);Trypsin(Promega,美国);30 000超滤离心管(Sartorius,德国);0.22 μm超滤离心管(Corning,美国);Multiple Affinity Removal LC Column (Agilent,美国);PMSF和SDT裂解液(碧云天,南京);BSA、SDS、Tris(生工,上海);甲酸、1 mol/L TEAB、TMT试剂盒、冷冻离心机、Nano Drop、高分辨质谱仪Q Exactive Plus、超声仪(ThermoFisher Scientific,美国);CO2培养箱(BNA-311型,日本);Urea、电泳仪、电泳槽、转膜槽(Bio-Rad,美国)。
1.2. 主要软件包
Proteome Discoverer 2.1(Thermo Fisher Scientific,美国);MASCOT 2.6(Matrix Science,美国);Perseus 1.3(Max Planck Institute of Biochemistry,德国);R version 3.0.3(<a href="https://cran.r-project.org/" target="_blank">https://cran.r-project.org/</a>)。
1.3. 细胞培养
CNE1SETD2-KO细胞株由广东医科大学基础医学院病理学系构建[12]。CNE1WT和CNE1SETD2-KO细胞参考既往方法[17-19]进行常规培养。简言之,CNE1WT和CNE1SETD2-KO细胞用含10%胎牛血清的RPMI 1640培养液重悬,接种直径6 cm培养皿,置于37 ℃、5% CO2饱和湿度条件下培养,每2 d换液一次。取对数生长期细胞用于相关实验。
1.4. 蛋白质样本处理和收集
将CNE1WT和CNE1SETD2-KO细胞接种于直径为10 cm的细胞培养皿中,以37 ℃、5% CO2条件下在培养箱中培养,每48 h更换培养基,待细胞90%汇合后加入SDS裂解液裂解细胞并收集至EP管中,95 ℃条件下煮10 min,期间每隔4 min打开EP管盖以排出气体。将样本置于冰上,并于30%功率、间隔5 s条件下超声40 s。12 000 r/min、4 ℃离心15 min后收集上清液。用BCA试剂盒测定蛋白浓度,并取等量蛋白质进行SDS-PAGE电泳以检验蛋白质丰度和样本间的平行性。
1.5. SDS-PAGE电泳
各样品取蛋白质20 µg分别加入6×上样缓冲液,沸水浴5 min,进行12% SDS-PAGE电泳(恒压250 V,40 min),考马斯亮蓝染色。
1.6. 滤膜辅助蛋白酶解法酶解蛋白
各样品取30 μL蛋白质溶液,分别加入DTT至终浓度为100 mmol/L,沸水浴5 min,冷却至室温。加入200 μL UA buffer混匀,转入超滤离心管中,离心12 500×g 25 min,弃滤液(重复该步骤两次)。加入100 μL IAA buffer(100 mmol/L IAA in UA),600 r/min振荡1 min,室温避光反应30 min,离心12 500×g 25 min。加入100 μL UA buffer离心12 500×g 15 min,重复该步骤两次。加入100 μL 0.05 mol/L TEAB溶液,离心12 500×g 15 min,重复该步骤两次。加入40 μL Trypsin buffer(4 μg Trypsin in 40 μL 0.1 mol/L TEAB溶液),600 r/min振荡1 min,37 ℃放置16 h。换新收集管,离心12 500×g 15 min;再加入20μL 0.1 mol/LTEAB溶液,离心12 500×g 15 min,收集滤液,采用Nano Drop 2000进行肽段定量。上清液放入真空浓缩蒸发仪干燥后置于-80℃保存。
1.7. TMT标记
用100 mmol/L TEAB复溶干燥后的样本,取6标的TMT试剂盒,利用TMT试剂对所获得的全部肽段进行室温标记1 h。加入适量的5%羟胺终止反应,干燥除去乙腈后,调节PH至2以下并用Sep-Pak C18柱除盐,-80 ℃保存样本。
1.8. 液相色谱-串联质谱
样品经LC系统进样,色谱柱分离,从LC流出的被分离组分通过接口进入离子源,离子化后在第一级质量分析器根据质荷比分离,之后串联进入第二级质量分析器进行二级质谱分析,获得质谱原始数据。
1.9. 生物信息学分析
对目标蛋白质集合进行GO注释或KEGG通路注释,通过Fisher精确检验,比较各个GO分类或KEGG通路在目标蛋白质集合和总体蛋白质集合中的分布情况,来评价某个GO条目或KEGG通路蛋白质富集度的显著性水平。
1.10. 数据处理及分析
质谱分析原始数据为raw文件,用软件Mascot 2.6和Proteome Discoverer 2.1进行查库鉴定及定量分析。MASCOT 2.6数据库搜索相关参数见表 1。在定量分析过程中,采用Proteome Discoverer 2.1软件对肽段报告离子峰强度值进行抽提,并对定量值进行归一化处理,相关参数见表 2。将表达倍数(FC)≥1.2,P < 0.05定义为差异蛋白。对目标蛋白质集合的GO注释可以从参与的生物学过程、具有的分子功能和所处的细胞组分三个方面对这些蛋白质进行分类,通过Fisher精确检验来评价某个GO条目蛋白质富集度的显著性水平。KEGG通路富集分析方法即以KEGG通路为单位,以所有定性蛋白质为背景,通过Fisher精确检验来分析计算各个通路蛋白质富集度的显著性水平,从而确定受到显著影响的代谢和信号转导途径。
1.
MASCOT 2.6数据库搜索相关参数
Related parameters for MASCOT 2.6 database searching
| Parameter | Value |
| Protein database | Uniprot_HomoSapiens_20386_20180905 |
| Enzyme | Trypsin |
| Max missed cleavages | 2 |
| Instrument | ESI-TRAP |
| Precursor mass toleranc | ±10 ppm |
| Fragment mass tolerance | 0.05 Da |
| Use average precursor mass | False |
| Modification groups from quan method | TMT 6plex |
| Dynamic modifications | Oxidation(M)Acetyl(Protein N-term) |
| Static modifications | Carbamidomethyl(C) |
| Database pattern | Decoy |
| Peptide FDR | ≤0.01 |
2.
Proteome Discoverer 2.1定量分析参数
Proteome Discoverer 2.1 quantitative analysis parameters
| Parameter | Value |
| Peak integration | Integration window tolerance:20 ppm integration method:most confident centroid |
| Scan event filters | Mass analyzer:FTMSMS Order:MS2 Activation type:HCD |
| Quantification-general | Peptides to use:Unique |
| Normalization scaling | Normalization mode:total peptide amountscaling mode:on channels average(per file) |
2. 结果
2.1. 成功获得高质量蛋白质样本
两组细胞经SDS裂解后获得蛋白质样本,BCA法测定各组蛋白质浓度,各样本蛋白质浓度均大于5 μg/μL,蛋白质总量均大于200 μg,样本评价为A级。取一定量蛋白质样本进行SDS-PAGE,凝胶经考马斯亮蓝染色,结果显示各样本电泳条带均清晰,样本间平行性较好。蛋白预质谱显示酶解正常,色谱质谱行为正常,质谱结果评价为A级(图 1)。
1.
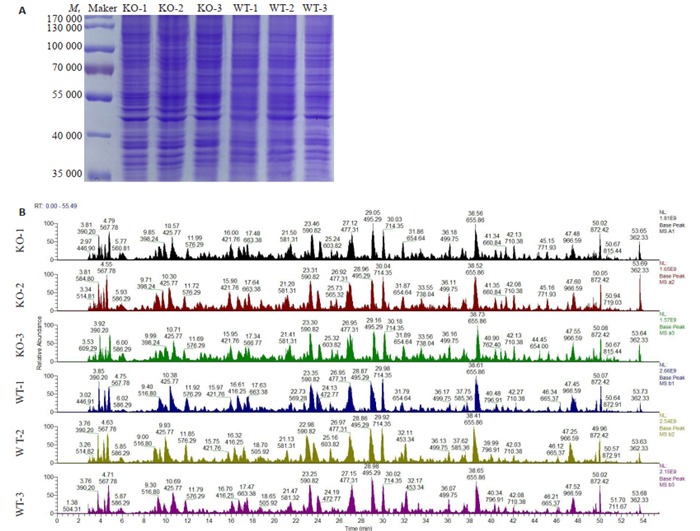
定量蛋白质学待测样本质量评估
Quality assessment of the protein samples for quantitative proteomics analysis. A: SDS-PAGE results of protein samples; B: Mass spectrometry peak map of protein samples.
2.2. 蛋白鉴定和筛选
通过TMT蛋白质定量技术串联质谱分析,共得到二级质谱谱图总数298 224个,肽段匹配到的谱图数为126 517个,肽段总数44 175,唯一肽段总数42 221,蛋白质总数5566。总体鉴定到的肽段离子得分、肽段数量分布、肽段序列长度分布、蛋白质相对分子量、蛋白质等电点分布、蛋白质序列覆盖度、蛋白质丰度分布和蛋白质分布火山图(图 2)。
2.
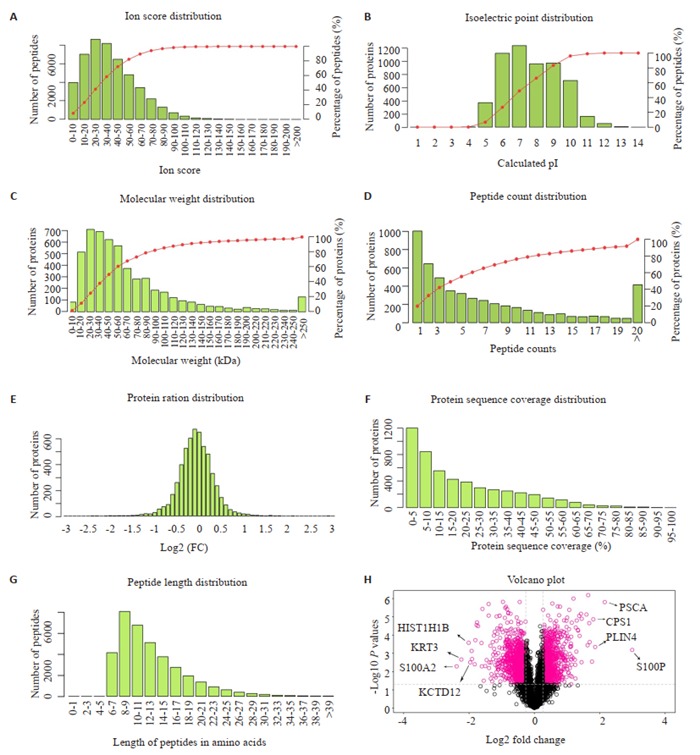
蛋白质质谱鉴定的定量分析
Quantitative analysis of protein mass spectrometry. A: Ion score distribution; B: Isoelectric point distribution; C: Peptide weight distribution; D: Peptide count distribution; E: Protein ratio distribution; F: Protein sequence coverage distribution; G: Peptide length distribution; H: Volcano plot, where top 4 up-regulated (S100P, PSCA, CPS1, PLIN4) and down-regulated proteins (S100A2, KRT3, HIST1HI1B, KCTD12) are indicated.
2.3. 差异蛋白质富集
以表达FC≥1.2倍(上调或下调)且P < 0.05作为标准,与CNE1WT相比较,CNE1SETD2-KO细胞中显著上调蛋白数量为904个(前5名的蛋白分别为S100P、PSCA、PLIN4、CPS1和CSPR1),显著下调蛋白数量1145个(前5名的蛋白为S100A2、KRT3、HIST1H1B、KCTD12和KRT5),总共差异表达蛋白共2049个。上调蛋白质中影响细胞增殖、周期蛋白、细胞粘附、迁移、上皮间叶转化等相关蛋白共62个。将相关蛋白质聚类后制作热图(图 3)。
3.
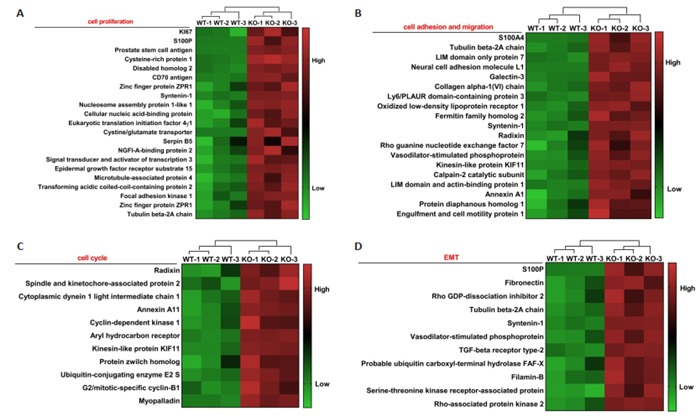
部分差异蛋白质聚类热图
Clustering heat map of partial differential proteins. A: Cell proliferation; B: Cell adhesion and migration; C: Cell cycle; D: EMT.
2.4. GO注释及富集分析
GO功能注释结果表明,SETD2敲除后在生物过程(细胞过程及调节,细胞运动、代谢过程及细胞组分的生物合成)、分子功能(催化活性及分子结合、转录因子活性)和细胞组分(胞膜、细胞器、大分子复合物)等具有特征性。通过Fisher精确检验来对GO注释进行显著性富集分析。结果显示胞质组分、单一生物过程、细胞蛋白复合物解聚、辅助因子代谢过程等GO条目最容易受到SETD2敲除的影响。其中富集最显著的前30个GO条目(图 4)。
4.

富集的前30条GO条目统计
Enriched top 30 GO terms.
2.5. KEGG通路注释及富集分析
为了更系统全面地了解SETD2所参与的生物学过程,将上述差异表达蛋白与KEGG数据库比对后进行差异信号分析通过Fisher精确检验来分析计算各个通路蛋白质富集度的显著性水平,结果显示差异表达蛋白主要参与了近300条KEGG通路,与肿瘤密切相关的经典通路主要有MAPK、PI3K-Akt、Ras、Rap1、mTOR、Hippo、HIF-1、Wnt、AMPK、FoxO、ErbB、p53、JAK-STAT、PPAR、Apelin、NF-kB、VEGF、Hedgehog及Notch通路等(表 3)。
3.
差异表达蛋白与肿瘤相关KEGG通路富集与注释
Enrichment and annotation of differentially expressed proteins associated with tumor-related KEGG pathways
| Protein Name | Number | Map ID | Map name |
| NFKB2, FlNA, MKK3, ASK2, HSPA1s, MST2, PKA, PPP5C, JNK, MAPKAPK2, MLTK, DUSP9, FLNA, PAK2, MEKK2, HSPA1s, MAPK1_3, GNG12, RELA, PPP3R, GRB2, KRAS2, TGFB1.ECSIT, RAP1A, HRAS, FGF2, MAP2K1IP1, RRAS, ERBB1, JUND, FLNA, RSP5, MAPK1_3, RHOA | 35 | ko04010 | MAPK |
| COL6A. GYS, FN1.ITGBS, CASP9, PPP2R2, IIGAV, PTK2, GSK3B, PRKAA, EIF4E, ERK. PPP2R1, GNG12, RELA, CREB1, GRB2, KRAS, GNG4, LAMC1, E4.1.132, HRAS, HSP90B, FGF2, BAD, EGFR, GNGS, ITGA3, LAMB3, CD104, CD49f, GNG10, LAMC2, LAMA3_ 5 | 34 | ko04151 | PIK-Akt |
| TBK1, PKA, AF6, JNK, PAK2, ERK, GNG12, RELA, GRB2, KRAS, SHOC2, RHOA, RALB, GNG4, ARF6, RAP1A, GNB2, HRAS, FGF2, BAD, RRAS, EGFR, GNG5, RALA | 24 | k04014 | Ras |
| VASP, MAP2K3, PARD6, AF6, ERK, PKD, BCAR1, CTNNB1, KRAS, GNAQ, GNAS, SRC, SIPA1L3, RHOA. RALB, RAP1A, HRAS, FGF2, GNAI, RRAS, EGFR, RALA, | 22 | ko04015 | Rap1 |
| CLIP1, ATPeV1C, ATPeV1A, GSK3B, PRKAA, EIF4E, MAPK1_3, ATPeV1E, SEH1, LAMTOR1, WNT7, LAMTOR5, GRB2, KRAS2, ATPeV1F, FZD6, RHOA, LAMTOR2, HRAS, LAMIOR3 | 20 | ko04150 | mTOR |
| AMOT, TGFBR2, PARD6, PPP2R2, STK3, GSK3B, SMAD4, PPP2R1, PALS1, CTNNB1, WNT7, DLG1, TGFB1, FZD6, PATJ, PPP1C, SERPINE1, MST2 | 18 | ko04390 | Hippo |
| HM0X1, IFNGR1, CAMK2, GAPDH, STAT3, EIF4E, MAPK1_3, CUL2, RELA, PDHA, PDHB, TE, HK, ERBB1, SERPINE1 | 15 | ko04066 | HIF-1 |
| CAMK2, ROCK2, PKA, JNK, GSK3B, SMAD4, CUL1, PPP3R, CSNK2B, CTNNB1, WNT7, CSNK2A, FZD6. TBL1, RHOA | 15 | k04310 | Wnt |
| PFK, EEF2, GYS, PFK PPP2R2, AMPK, PPP2R1. pfkA, CREB1, RAB14, ELAVL1, MLYCD, E4.1132, SCD | 14 | ko04152 | AMPK |
| TGFBR2, STAT3, JNK, SMAD4, PRKAA, MAPK1_3, GABARAP, GRB2, KRAS2, TGFB1, HOMER, E4.11.32, HRAS, ERBB1 | 14 | ko04068 | FoxO |
| CAMK2, STAT5B, JNK, PTK2, PAK2. ERK, GRB2. KRAS2, SRC, HRAS, BAD, ERBB1 | 12 | k04012 | ErbB |
| RRM2, CASP9, CDK1, BID, RRM2, CCNB1, GTSE1, TNFRSF6, STEAP3, CYC, PAI1 | 11 | ko04115 | p53 |
| IFNGR1, STAT3, STAT5B, STAM, PTPN6, GRB2, PTPN2, HRAS, ERBB1 | 9 | ko04630 | JAK STAT |
| PLIN4, ACSL, SLC27A2, ACADM, ACSL, E1.33.6, SCP2, E4.1.1.32, SCD, | 9 | k03320 | PPAR |
| GNAI, GNG4, GNB2, HRAS, JAG1, GNGS, SERPINE1, GNG10 | 8 | ko04371 | Apelin |
| RIPK1, NFKB2, MAIT1, RELA, CSNK2B, CSNK2A, PIAS4, CD54 | 8 | ko04064 | NF-kB |
| TGFBR2, MADHIP, SMAD4, PPP2R1, ERK, CUL1, TGFB1, RHOA | 8 | ko04350 | TGF-b |
| CASP9, MAPKAPK2, PTK2, ERK, PPP3R, SRC, HRAS, BAD | 8 | ko04370 | VEGF |
| PKA, GSK3B, CUL3, CUL1, GSK3B | 5 | ko04340 | Hedgehog |
| JAG2, HDAC1 2, JAG1 | 3 | ko04330 | Notch |
3. 讨论
为获得SETD2表达改变前后NPC细胞中蛋白质表达谱特性,实验首先将前期基于CRISPR/Cas9技术成功构建的SETD2基因敲除细胞CNE1SETD2-KO与未进行基因敲除的野生型细胞CNE1WT常规培养,在获得合格的细胞及蛋白样本的基础上,经TMT标记的串联质谱分析技术,鉴定到两株细胞中总蛋白数量为5566个,其中显著表达差异蛋白质共2049个,CNE1SETD2-KO细胞中显著上调蛋白904个,显著下调蛋白1145个。这些庞大的差异蛋白数量提示SETD2基因在NPC生物学功能中发挥了重要作用。随后,对上调蛋白进行初步分类,发现部分蛋白质参与了细胞增殖、周期蛋白、细胞粘附、迁移、上皮间叶转化等过程,这些蛋白质的上调表达对促进NPC细胞演进无疑起到重要的作用。由于下调蛋白数量巨大,我们暂未对下调蛋白进行详尽的分类。
如前所述,蛋白组学可基于宏观角度提供疾病相关的大数据,再经过生物信息学分析挖掘,有很能得到相关疾病特异性的分子治疗和预后判断靶点。经过TMT标记蛋白质串联质谱技术,我们表征了鉴定到的蛋白质(或多肽)的基本参数,包括肽段离子得分、肽段数量分布、肽段序列长度分布、蛋白质相对分子量、蛋白质等电点分布、蛋白质序列覆盖度、蛋白质丰度分布等情况。经过绘制蛋白质分布火山图,进一步显示了SETD2基因表达改变后上调和下调显著的蛋白质。本研究中,我们发现SETD2缺失后上调最显著的5个蛋白质是S100P、PSCA、PLIN4、CPS1和CSPR1,下调最显著的5个蛋白质为S100A2、KRT3、HIST1H1B、KCTD12和KRT5。通过检索PubMed数据库,未发现有关SETD2与上述10个显著差异蛋白的相关报道,但S100P、S100A2和KRT5与NPC的关系已有报道[20-22]。鉴于SETD2既可以甲基化组蛋白,又可以甲基化非组蛋白[23-26],这为后期的进一步挖掘NPC发病的分子机制提供了可能的方向。
SETD2是人体中迄今已知的唯一调节H3K36me3活性的基因[24]。通常认为,低甲基化有利于基因的转录活化,而高甲基化则抑制基因的转录活化。我们前期发现,SETD2基因敲除后显著抑制了NPC细胞中H3K36me3的表达,而对H3K36me1和H3K36me2表达无明显影响(未发表资料)。在人类肿瘤中SETD2基因突变较为常见,而SETD2基因突变后降低了H3K36me3的表达和活性,是患者预后不良的分子事件[27-29]。新近发现,在白血病肿瘤细胞中H3K36me3功能下降后通过上调H3K79me2而扰动基因表达,最终促进了白血病的进展[30]。上述发现为阐明H3K36me3表达与活性异常参与肿瘤进展的机制提供了依据。本研究发现,SETD2敲除后导致差异上调和下调表达基因众多;经检索PubMed数据库,目前尚未见H3K36me3直接调节前述10个改变最显著基因的报道。当前我们正在以H3K36me3为靶点进行ChIP-seq实验,以期发现NPC中H3K36me3直接调控的下游基因,为阐明NPC的发病机制提供更直观的实验依据。
GO富集注释和KEGG信号富集注释是生物信息学常用的手段[31]。通过GO富集发现,SETD2表达缺失诱导的差异蛋白表达主要功能涉及多个方面,在生物过程、分子功能和细胞组分等具有特征性。通过Fisher精确检验来对GO注释进行显著性富集分析,结果显示胞质组分、单一生物过程、细胞蛋白复合物解聚、辅助因子代谢过程等30个GO条目最容易受到SETD2敲除的影响。GO富集注释结果为SETD2下游分子的分布及功能提供一个总体趋势判断。KEGG信号富集中发现SETD2表达缺失可影响近300个KEGG通路,其中与肿瘤关系密切的经典通路主要有MAPK、PI3K-Akt、Ras、Rap1、mTOR、Hippo、HIF-1、Wnt、AMPK、FoxO、ErbB、p53、JAK-STAT、PPAR、Apelin、NF-kB、VEGF、Hedgehog及Notch通路等。临床NPC组织或体外培养的细胞系中上述富集到的信号通路的异常活化已被大量独立报道所证实。以PI3K-Akt信号为例,本研究发现SETD2表达缺失后富集到34个与PI3K-Akt信号通路有关的差异表达蛋白质。我们前期报道靶向抑制PI3K-Akt信号可显著抑制NPC细胞的上皮间质转化和肿瘤细胞肺转移[32]。因而,PI3K-Akt信号异常活化在NPC中具有普遍性,具有分子靶向干预价值。本研究结果提示,NPC中SETD2表达缺失可改变20条与肿瘤相关的经典的信号通路,提示SETD2基因的多能性和重要性,也提示基于SETD2为靶点的分子治疗具有潜在的可行性。目前我们正在对感兴趣的通路进行干预和验证。
SETD2蛋白又名Huntington相互作用蛋白。我们发现NPC中SETD2缺失后Huntington疾病相关信号同样被富集到,相关的差异蛋白质共72个(未发表资料)。另外有趣的是,鉴于NPC是EB病毒相关性肿瘤,本次实验我们也富集到37个与EB病毒感染有关的差异蛋白(未发表资料)。这些有趣的信息均有待后续深入分析。
既往NPC中蛋白组学相关研究对象多为体外模型细胞或患者血清样本,考察指标多为筛查放射抵抗、转移或复发相关标志物[33-36]。本研究应用最新的TMT标记定量蛋白组学方法,方法上较早期的组学研究更为高效和可靠。同时,研究对象为基于CRISPR/Cas9基因组编辑技术构建的SETD2稳定敲除细胞,在模型上具有原始创新性。最后,本研究首次揭示了SETD2表达缺失后NPC细胞的蛋白组学变化特征,初步提示了差异蛋白涉及的生物学功能,富集了差异蛋白主要涉及的信号通路,从宏观角度为临床上NPC的诊断和治疗方案制定提供了一定的实验参考。
Biography
曾玉梅,硕士,副主任医师,E-mail: 41274931@qq.com
Funding Statement
广东省“扬帆计划”高层次人才项目(4YF16007G)
Contributor Information
曾 玉梅 (Yumei ZENG), Email: 41274931@qq.com.
揭 伟 (Wei JIE), Email: wei.jie@gdmu.edu.cn.
References
- 1.Lo KW, To KF, Huang DP. Focus on nasopharyngeal carcinoma. Cancer Cell. 2004;5(5):423–8. doi: 10.1016/S1535-6108(04)00119-9. [Lo KW, To KF, Huang DP. Focus on nasopharyngeal carcinoma[J]. Cancer Cell, 2004, 5(5): 423-8.] [DOI] [PubMed] [Google Scholar]
- 2.Al-Sarraf M, Reddy MS. Nasopharyngeal carcinoma. Curr Treat Options Oncol. 2002;3(1):21–32. doi: 10.1007/s11864-002-0038-8. [Al-Sarraf M, Reddy MS. Nasopharyngeal carcinoma[J]. Curr Treat Options Oncol, 2002, 3(1): 21-32.] [DOI] [PubMed] [Google Scholar]
- 3.Sun XS, Liu DH, Liu SL, et al. Patterns of Failure and Survival Trends in 3, 808 Patients with Stage Ⅱ Nasopharyngeal Carcinoma Diagnosed from 1990 to 2012: A Large-Scale Retrospective Cohort Study. Cancer Res Treat. 2019 doi: 10.4143/crt.2018.688. [Sun XS, Liu DH, Liu SL, et al. Patterns of Failure and Survival Trends in 3, 808 Patients with Stage Ⅱ Nasopharyngeal Carcinoma Diagnosed from 1990 to 2012: A Large-Scale Retrospective Cohort Study[J]. Cancer Res Treat, 2019, doi: 10.4143/crt.2018.688.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Sun XS, Liang YJ, Liu SL, et al. Establishment and validation of a nomogram for predicting survival in patients with de novo metastatic nasopharyngeal carcinoma. Oral Oncol. 2019;94:73–9. doi: 10.1016/j.oraloncology.2019.05.015. [Sun XS, Liang YJ, Liu SL, et al. Establishment and validation of a nomogram for predicting survival in patients with de novo metastatic nasopharyngeal carcinoma[J]. Oral Oncol, 2019, 94: 73-9.] [DOI] [PubMed] [Google Scholar]
- 5.Faber PW, Barnes GT, Srinidhi J, et al. Huntingtin interacts with a family of WW domain proteins. Hum Mol Genet. 1998;7(9):1463–74. doi: 10.1093/hmg/7.9.1463. [Faber PW, Barnes GT, Srinidhi J, et al. Huntingtin interacts with a family of WW domain proteins[J]. Hum Mol Genet, 1998, 7(9): 1463-74.] [DOI] [PubMed] [Google Scholar]
- 6.Hakimi AA, Ostrovnaya I, Reva B, et al. Adverse outcomes in clear cell renal cell carcinoma with mutations of 3p21 epigenetic regulators BAP1 and SETD2: a report by MSKCC and the KIRC TCGA research network. Clin Cancer Res. 2013;19(12):3259–67. doi: 10.1158/1078-0432.CCR-12-3886. [Hakimi AA, Ostrovnaya I, Reva B, et al. Adverse outcomes in clear cell renal cell carcinoma with mutations of 3p21 epigenetic regulators BAP1 and SETD2: a report by MSKCC and the KIRC TCGA research network[J]. Clin Cancer Res, 2013, 19(12): 3259-67.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Al SW, Sasi W, Jiang WG, et al. The mRNA expression of SETD2 in human breast cancer: correlation with clinico-pathological parameters. BMC Cancer. 2009;9:290. doi: 10.1186/1471-2407-9-290. [Al SW, Sasi W, Jiang WG, et al. The mRNA expression of SETD2 in human breast cancer: correlation with clinico-pathological parameters[J]. BMC Cancer, 2009, 9: 290.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Zhu X, He F, Zeng H, et al. Identification of functional cooperative mutations of SETD2 in human acute leukemia. Nat Genet. 2014;46(3):287–93. doi: 10.1038/ng.2894. [Zhu X, He F, Zeng H, et al. Identification of functional cooperative mutations of SETD2 in human acute leukemia[J]. Nat Genet, 2014, 46(3): 287-93.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Joseph CG, Hwang H, Jiao Y, et al. Exomic analysis of myxoid liposarcomas, synovial sarcomas, and osteosarcomas. Genes Chromosomes Cancer. 2014;53(1):15–24. doi: 10.1002/gcc.22114. [Joseph CG, Hwang H, Jiao Y, et al. Exomic analysis of myxoid liposarcomas, synovial sarcomas, and osteosarcomas[J]. Genes Chromosomes Cancer, 2014, 53(1): 15-24.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Rogers K. Comprehensive molecular profiling of lung adenocarcinoma. Nature. 2014;511(7511):543–50. doi: 10.1038/nature13385. [Rogers K. Comprehensive molecular profiling of lung adenocarcinoma[J]. Nature, 2014, 511(7511): 543-50.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.赵 利刚, 林 中原, 覃 灵燕, et al. SETD2 mRNA在鼻咽癌组织中的表达及其与LMP1的关系. 山东医药. 2016;56(2):20–2. doi: 10.3969/j.issn.1002-266X.2016.02.007. [赵利刚, 林中原, 覃灵燕, 等. SETD2 mRNA在鼻咽癌组织中的表达及其与LMP1的关系[J].山东医药, 2016, 56(2): 20-2.] [DOI] [Google Scholar]
- 12.王 思思, 廖 晓敏, 邵 钟铭, et al. 基于CRISPR/Cas9技术构建SETD2基因敲除鼻咽癌细胞株并分析其增殖特性. 中国病理生理杂志. 2018;34(12):2172–9. doi: 10.3969/j.issn.1000-4718.2018.12.010. [王思思, 廖晓敏, 邵钟铭, 等.基于CRISPR/Cas9技术构建SETD2基因敲除鼻咽癌细胞株并分析其增殖特性[J].中国病理生理杂志, 2018, 34(12): 2172-9.] [DOI] [Google Scholar]
- 13.Gregorich ZR, Ge Y. Top-down proteomics in health and disease: challenges and opportunities. Proteomics. 2014;14(10):1195–210. doi: 10.1002/pmic.201300432. [Gregorich ZR, Ge Y. Top-down proteomics in health and disease: challenges and opportunities[J]. Proteomics, 2014, 14(10): 1195-210.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Li X, Wang W, Chen J. Recent progress in mass spectrometry proteomics for biomedical research. Sci China Life Sci. 2017;60(10):1093–113. doi: 10.1007/s11427-017-9175-2. [Li X, Wang W, Chen J. Recent progress in mass spectrometry proteomics for biomedical research[J]. Sci China Life Sci, 2017, 60(10): 1093-113.] [DOI] [PubMed] [Google Scholar]
- 15.Xiao L, Xiao T, Wang ZM, et al. Biomarker discovery of nasopharyngeal carcinoma by proteomics. Expert Rev Proteomics. 2014;11(2):215–25. doi: 10.1586/14789450.2014.897613. [Xiao L, Xiao T, Wang ZM, et al. Biomarker discovery of nasopharyngeal carcinoma by proteomics[J]. Expert Rev Proteomics, 2014, 11(2): 215-25.] [DOI] [PubMed] [Google Scholar]
- 16.Xiao Z, Chen Z. Deciphering nasopharyngeal carcinoma pathogenesis via proteomics. Expert Rev Proteomics. 2019;16(6):475–85. doi: 10.1080/14789450.2019.1615891. [Xiao Z, Chen Z. Deciphering nasopharyngeal carcinoma pathogenesis via proteomics[J]. Expert Rev Proteomics, 2019, 16(6): 475-85.] [DOI] [PubMed] [Google Scholar]
- 17.贾 亚楠, 王 可可, 王 思思, et al. SATB1高表达通过促进上皮-间充质转化而介导鼻咽癌细胞侵袭和转移. 中国病理生理杂志. 2018;34(9):1578–85. doi: 10.3969/j.issn.1000-4718.2018.09.007. [贾亚楠, 王可可, 王思思, 等. SATB1高表达通过促进上皮-间充质转化而介导鼻咽癌细胞侵袭和转移[J].中国病理生理杂志, 2018, 34(9): 1578-85.] [DOI] [Google Scholar]
- 18.Shen Z, Zeng Y, Guo J, et al. Over-expression of the special AT rich sequence binding protein 1(SATB1) promotes the progression of nasopharyngeal carcinoma: association with EBV LMP-1 expression. J Transl Med. 2013;11:217. doi: 10.1186/1479-5876-11-217. [Shen Z, Zeng Y, Guo J, et al. Over-expression of the special AT rich sequence binding protein 1(SATB1) promotes the progression of nasopharyngeal carcinoma: association with EBV LMP-1 expression[J]. J Transl Med, 2013, 11: 217.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Wu Y, Shen Z, Wang K, et al. High FMNL3 expression promotes nasopharyngeal carcinoma cell metastasis: role in TGF-β1-induced epithelia-to-mesenchymal transition. Sci Rep. 2017;7:42507. doi: 10.1038/srep42507. [Wu Y, Shen Z, Wang K, et al. High FMNL3 expression promotes nasopharyngeal carcinoma cell metastasis: role in TGF-β1-induced epithelia-to-mesenchymal transition[J]. Sci Rep, 2017, 7: 42507.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Liu Y, Wang C, Shan X, et al. S100P is associated with proliferation and migration in nasopharyngeal carcinoma. Oncol Lett. 2017;14(1):525–32. doi: 10.3892/ol.2017.6198. [Liu Y, Wang C, Shan X, et al. S100P is associated with proliferation and migration in nasopharyngeal carcinoma[J]. Oncol Lett, 2017, 14(1): 525-32.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.Lin SJ, Chang KP, Hsu CW, et al. Low-molecular-mass secretome profiling identifies C-C motif chemokine 5 as a potential plasma biomarker and therapeutic target for nasopharyngeal carcinoma. J Proteomics. 2013;94:186–201. doi: 10.1016/j.jprot.2013.09.013. [Lin SJ, Chang KP, Hsu CW, et al. Low-molecular-mass secretome profiling identifies C-C motif chemokine 5 as a potential plasma biomarker and therapeutic target for nasopharyngeal carcinoma[J]. J Proteomics, 2013, 94: 186-201.] [DOI] [PubMed] [Google Scholar]
- 22.Xiao Z, Li M, Li G, et al. Proteomic characterization reveals a molecular portrait of nasopharyngeal carcinoma differentiation. J Cancer. 2017;8(4):570–7. doi: 10.7150/jca.17414. [Xiao Z, Li M, Li G, et al. Proteomic characterization reveals a molecular portrait of nasopharyngeal carcinoma differentiation[J]. J Cancer, 2017, 8(4): 570-7.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.Chopra M, Bohlander SK. Disturbing the histone code in leukemia: translocations and mutations affecting histone methyl transferases. Cancer Genet. 2015;208(5):192–205. doi: 10.1016/j.cancergen.2014.10.005. [Chopra M, Bohlander SK. Disturbing the histone code in leukemia: translocations and mutations affecting histone methyl transferases[J]. Cancer Genet, 2015, 208(5): 192-205.] [DOI] [PubMed] [Google Scholar]
- 24.Edmunds JW, Mahadevan LC, Clayton AL. Dynamic histone H3 methylation during gene induction: HYPB/SETD2 mediates all H3K36 trimethylation. http://d.old.wanfangdata.com.cn/NSTLQK/NSTL_QKJJ021679632/ EMBO J. 2008;27(2):406–20. doi: 10.1038/sj.emboj.7601967. [Edmunds JW, Mahadevan LC, Clayton AL. Dynamic histone H3 methylation during gene induction: HYPB/SETD2 mediates all H3K36 trimethylation[J]. EMBO J, 2008, 27(2): 406-20.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.McDaniel SL, Strahl BD. Shaping the cellular landscape with Set2/SETD2 methylation. Cell Mol Life Sci. 2017;74(18):3317–34. doi: 10.1007/s00018-017-2517-x. [McDaniel SL, Strahl BD. Shaping the cellular landscape with Set2/SETD2 methylation[J]. Cell Mol Life Sci, 2017, 74(18): 3317-34.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.Chen K, Liu J, Liu S, et al. Methyltransferase SETD2-mediated methylation of STAT1 is critical for interferon antiviral activity. Cell. 2017;170(3):492–506. doi: 10.1016/j.cell.2017.06.042. [Chen K, Liu J, Liu S, et al. Methyltransferase SETD2-mediated methylation of STAT1 is critical for interferon antiviral activity[J]. Cell, 2017, 170(3): 492-506. e14.] [DOI] [PubMed] [Google Scholar]
- 27.Li J, Duns G, Westers H, et al. SETD2: an epigenetic modifier with tumor suppressor functionality. http://d.old.wanfangdata.com.cn/Periodical/jsyxkxyyk201101008. Oncotarget. 2016;7(31):50719–34. doi: 10.18632/oncotarget.9368. [Li J, Duns G, Westers H, et al. SETD2: an epigenetic modifier with tumor suppressor functionality[J]. Oncotarget, 2016, 7(31): 50719-34.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 28.Fontebasso AM, Schwartzentruber J, Khuong-Quang DA, et al. Mutations in SETD2 and genes affecting histone H3K36 methylation target hemispheric high-grade gliomas. Acta Neuropathol. 2013;125(5):659–69. doi: 10.1007/s00401-013-1095-8. [Fontebasso AM, Schwartzentruber J, Khuong-Quang DA, et al. Mutations in SETD2 and genes affecting histone H3K36 methylation target hemispheric high-grade gliomas[J]. Acta Neuropathol, 2013, 125(5): 659-69.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.Huang KK, McPherson JR, Tay ST, et al. SETD2 histone modifier loss in aggressive GI stromal tumours. Gut. 2016;65(12):1960–72. doi: 10.1136/gutjnl-2015-309482. [Huang KK, McPherson JR, Tay ST, et al. SETD2 histone modifier loss in aggressive GI stromal tumours[J]. Gut, 2016, 65(12): 1960-72.] [DOI] [PubMed] [Google Scholar]
- 30.Bu J, Chen A, Yan X, et al. SETD2-mediated crosstalk between H3K36me3 and H3K79me2 in MLL-rearranged leukemia. Leukemia. 2018;32(4):890–9. doi: 10.1038/leu.2017.339. [Bu J, Chen A, Yan X, et al. SETD2-mediated crosstalk between H3K36me3 and H3K79me2 in MLL-rearranged leukemia[J]. Leukemia, 2018, 32(4): 890-9.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 31.Chen L, Zhang YH, Lu G, et al. Analysis of cancer-related lncRNAs using gene ontology and KEGG pathways. Artif Int Med. 2017;76:27–36. doi: 10.1016/j.artmed.2017.02.001. [Chen L, Zhang YH, Lu G, et al. Analysis of cancer-related lncRNAs using gene ontology and KEGG pathways[J]. Artif Int Med, 2017, 76: 27-36.] [DOI] [PubMed] [Google Scholar]
- 32.Jiang H, Gao M, Shen Z, et al. Blocking PI3K/Akt signaling attenuates metastasis of nasopharyngeal carcinoma cells through induction of mesenchymal-epithelial reverting transition. Oncol Rep. 2014;32(2):559–66. doi: 10.3892/or.2014.3220. [Jiang H, Gao M, Shen Z, et al. Blocking PI3K/Akt signaling attenuates metastasis of nasopharyngeal carcinoma cells through induction of mesenchymal-epithelial reverting transition[J]. Oncol Rep, 2014, 32(2): 559-66.] [DOI] [PubMed] [Google Scholar]
- 33.蒙 慧玲, 朱 小东, 李 龄, et al. TMT定量蛋白质组学方法筛选鼻咽癌复发相关表达蛋白. 中国癌症防治杂志. 2017;9(3):167–71. doi: 10.3969/j.issn.1674-5671.2017.03.01. [蒙慧玲, 朱小东, 李龄, 等. TMT定量蛋白质组学方法筛选鼻咽癌复发相关表达蛋白[J].中国癌症防治杂志, 2017, 9(3): 167-71.] [DOI] [Google Scholar]
- 34.王 中卫, 王 亚利, 金 迎迎, et al. 蛋白组学分析放射抗拒性鼻咽癌细胞差异表达蛋白. http://d.old.wanfangdata.com.cn/Periodical/xaykdxxb201406016. 西安交通大学学报:医学版. 2014;35(6):810–5. [王中卫, 王亚利, 金迎迎, 等.蛋白组学分析放射抗拒性鼻咽癌细胞差异表达蛋白[J].西安交通大学学报:医学版, 2014, 35(6): 810-5.] [Google Scholar]
- 35.Peng X, Gong FM, Ren M, et al. Proteomic analysis of docetaxel resistance in human nasopharyngeal carcinoma cells using the twodimensional gel electrophoresis method. Anticancer Drugs. 2016;27(8):748–55. doi: 10.1097/CAD.0000000000000388. [Peng X, Gong FM, Ren M, et al. Proteomic analysis of docetaxel resistance in human nasopharyngeal carcinoma cells using the twodimensional gel electrophoresis method[J]. Anticancer Drugs, 2016, 27(8): 748-55.] [DOI] [PubMed] [Google Scholar]
- 36.Meng H, Zhu X, Li L, et al. Identification of CALM as the potential serum biomarker for predicting the recurrence of nasopharyngeal carcinoma using a mass spectrometry-based comparative proteomic approach. Int J Mol Med. 2017;40(4):1152–64. doi: 10.3892/ijmm.2017.3094. [Meng H, Zhu X, Li L, et al. Identification of CALM as the potential serum biomarker for predicting the recurrence of nasopharyngeal carcinoma using a mass spectrometry-based comparative proteomic approach[J]. Int J Mol Med, 2017, 40(4): 1152-64.] [DOI] [PMC free article] [PubMed] [Google Scholar]


