Abstract
目的
通过研究一例掌跖角化-牙周破坏综合征(PLS)患者及其家系组织蛋白酶C(CTSC)基因突变的特点,提供PLS分子致病机制的理论依据,丰富PLS基因诊断和治疗的科学资料。
方法
取得临床发现的一例PLS先证者及其家属知情同意,分别提取先证者及其父母、姐姐的基因组DNA,应用聚合酶链反应和DNA直接测序技术,分析先证者及其直系血亲CTSC基因的突变情况。
结果
PLS先证者CTSC基因存在复合型杂合突变,突变位点是c.754C>T和c.1040A>G,其父母均为携带者,姐姐未发现突变。
结论
PLS先证者的临床表征是由CTSC基因突变引起。
Keywords: 组织蛋白酶C, 掌跖角化-牙周破坏综合征, 基因突变
Abstract
Objective
This study aims to investigate the gene mutational characteristics of cathepsin C (CTSC) gene in a Chinese patient with Papillon-Lefèvre syndrome (PLS), then further confirm the genetic basis for the phenotype of PLS, and obtain genetic information that can be used as guide in the diagnosis and treatment of PLS.
Methods
With their consent, peripheral blood samples were obtained from the proband and his family members (his parents and older sister) for genomic DNA extraction. The coding region and exon/intron boundaries of the CTSC gene were amplified and sequenced using polymerase chain reaction and direct sequencing of DNA.
Results
Compound heterozygous mutations of CTSC gene were identified in the patient. The proband carries one heterozygous nonsense mutation c.754C>T in exon 5 and one heterozygous missense mutation c.1040A>G in exon 7. Both parents were heterozygous carriers without the clinical symptoms of PLS. None of the mutations were detected in the proband's sister.
Conclusion
The study proves that mutations of CTSC gene are responsible for the phenotype of Papillon-Lefèvre syndrome.
Keywords: cathepsin C, Papillon-Lefèvre syndrome, gene mutation
掌跖角化-牙周破坏综合征(Papillon-Lefèvre syndrome,PLS)是1924年由法国人Papillon和Lefèvre首次报告的常染色体隐性遗传性疾病,其发病率约为百万分之一至四[1]。男女发病率相似,未见种族特异性,多发于有近亲结婚史的家系。PLS以早发的快速进展的重度牙周组织破坏和掌跖过度角化为特征,牙周组织严重破坏且发现较晚时常导致乳恒牙的早期脱落。迄今为止,PLS的病因和致病机制还不完全清楚,但已有的研究结果表明位于染色体11q14.2的组织蛋白酶C(cathepsin C,CTSC)基因的突变是PLS的致病基础[2]。本研究对临床确诊的一个PLS患者家系进行CTSC基因分析。
1. 材料和方法
1.1. 研究对象
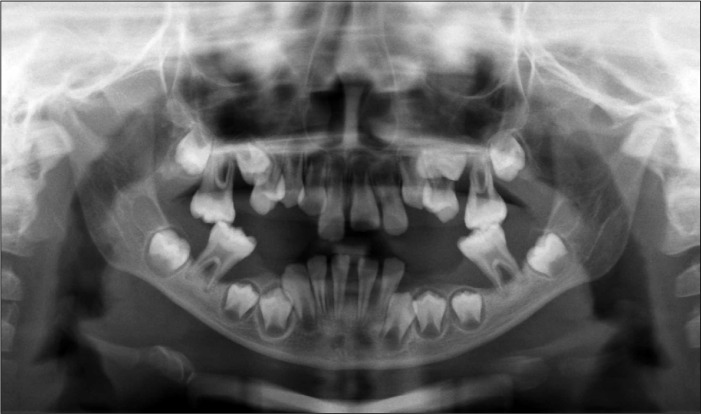
先证者为一名临床确诊为PLS的患儿,该患儿于2010年9月(8岁)因新萌牙齿松动、伴牙齿咬合疼痛、牙龈出血就诊。就诊时口腔检查:所有乳牙脱落,3、8、9、10、14、19、23、24、25、26、30萌出,多数牙松动并探及深牙周袋、牙周溢脓;未萌恒牙区的下颌牙槽嵴低平狭窄,牙龈未见明显炎症表现(图1)。X线片示全口牙槽骨广泛性吸收,4颗第一磨牙仅根尖处见少量牙槽骨(图2)。双手掌、足底皮肤过度角化,脱屑,病损厚度不一,呈鱼鳞状。足底皮肤损害累及足背和跟腱,双侧膝关节区见轻度皮肤损害。手背、指甲及肘关节无明显异常。全身皮肤无脓肿,无多汗及臭汗。现先证者13岁,发育良好,智力正常,恒牙均萌出,牙列拥挤,仅19、24、25轻微松动,牙龈呈轻度炎症。家系追溯4代,现存17人,无近亲结婚史,了解相关病史,对各家系成员进行全身及牙周专科检查,仅发现先证者一名受累。先证者共有直系血亲3人,包括父亲、母亲及一名姐姐,3人的牙周及全身检查结果显示均无侵袭性牙周炎及掌跖角化症状。
图 1. 初诊时先证者口内照.
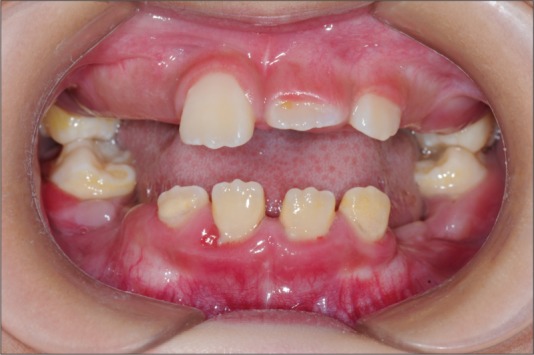
Fig 1 Intraoral view of propositus at the baseline
图 2. 初诊时先证者口腔曲面断层片.
Fig 2 Panoramic radiograph of propositus at the baseline
1.2. 方法
1.2.1. 基因组DNA的提取
因该PLS家系仅患儿一名先证者,故选择先证者及其直系血亲作为实验对象。取得受试者知情同意后,抽取外周静脉血各5 mL,枸橼酸钠抗凝,使用血液DNA提取试剂盒(天根生化科技北京有限公司)提取基因组DNA。
1.2.2. 引物设计和合成
参照Toomes等[3]和Hart等[4]关于CTSC基因1~7外显子引物设计方案设计引物,由上海生工生物工程有限公司合成。
1.2.3. 聚合酶链反应(polymerase chain reaction,PCR)
PCR反应体系为50 µL体系,包括10×PCR buffer 5 µL、25 mmol·L−1 dNTP 4 µL、10 µmol·L−1上游引物1 µL、10 µmol·L−1下游引物1 µL、5 U·µL−1 Taq酶0.5 µL、ddH2O 33.5 µL,模板DNA 5 µL。PCR反应条件:94 °C预变性3 min,94 °C变形30 s,按各外显子的退火温度退火30 s,72 °C延伸30 s,32个循环后于72 °C再延伸10 min。反应在GeneAmp PCR System 9600(PerkinElmer公司,美国)上进行。
1.2.4. DNA直接测序
使用琼脂糖凝胶分别纯化所得CTSC基因外显子1~7的PCR产物,并将纯化后的PCR产物送上海生工生物工程有限公司测序,其中外显子5使用正向巢氏测序引物,其余外显子使用PCR扩增引物,测序结果与人类基因组中的CTSC基因序列进行对比分析。
2. 结果
2.1. CTSC基因外显子5突变情况
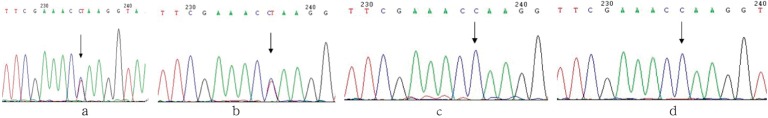
先证者位于外显子5的第754位碱基发生了一个杂合突变,该碱基对中的碱基C被T取代(c.754C>T)(图3a),与之相对应的第252位氨基酸密码子由CAA改变为TAA,其编码的氨基酸由谷氨酰胺改变为终止密码子(p.Q252X),该无义突变使该条染色体上的CTSC基因编码的氨基酸缩短为251个氨基酸。该突变位点以前的研究均未报道过,属于新突变位点。先证者的父亲于外显子5的第754位碱基也发现了一个与先证者相同的杂合突变(图3b),先证者的母亲及姐姐均未在外显子5上发现突变位点(图3c、d)。
图 3. CTSC基因外显子5测序.
Fig 3 Sequencing of CTSC gene mutation of exon 5
a、b箭头分别指示先证者及其父亲c.754C>T杂合突变位点,c、d箭头分别指示其母亲和姐姐c.754C正常序列。
2.2. CTSC基因外显子7突变情况
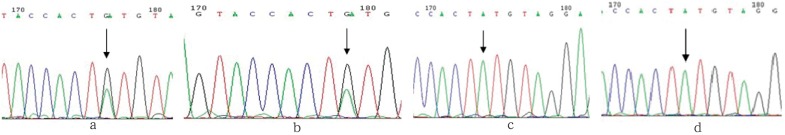
先证者位于外显子7的第1 040位碱基发生了一个杂合突变(图4a),该碱基对中的碱基A被G取代(c.1040A>G),与之相对应的第347位氨基酸密码子由TAT改变为TGT,其编码的氨基酸由酪氨酸改变为半胱氨酸(p.Y347C)。先证者的母亲于外显子7的第1 040位碱基也发现了一个与先证者相同的杂合突变(图4b),先证者的父亲及姐姐均未在外显子7上发现突变位点(图4c、d)。
图 4. CTSC基因外显子7测序.
Fig 4 Sequencing of CTSC gene mutation of exon 7
a、b箭头分别指示先证者及其母亲c.1040A>G杂合突变位点,c、d箭头分别指示其父亲和姐姐c.1040A正常序列。
2.3. CTSC基因内含子1的改变
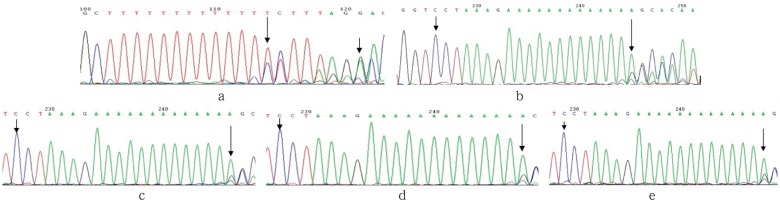
先证者外显子2的PCR产物正向测序发现:CTSC基因内含子1距外显子2序列始端有一个13个碱基T的连续序列,先证者这个连续序列含有一个碱基T的杂合缺失,造成外显子2的序列无法检测(图5a)。接着对先证者及其双亲、姐姐的外显子2的PCR产物行反向测序,结果均未在外显子2上发现突变,在内含子1末尾的T碱基连续序列均发现一个碱基T杂合缺失,但该缺失不位于内含子1和外显子2的接合处,不影响CTSC基因的转录和翻译(图5b~e)。
图 5. CTSC基因内含子1测序.
Fig 5 Sequencing of CTSC gene mutation of intron 1
a:先证者CTSC基因内含子1末尾的正向测序图,发现T碱基连续序列的一个碱基T杂合缺失,长箭头指示因缺失引起的测序结果异常,短箭头指示CTSC基因外显子2的第一个碱基(c.173G);b~e:反向测序图,分别示先证者及其父亲、母亲、姐姐CTSC基因内含子1互补链末尾的A碱基连续序列中的一个碱基杂合缺失,长箭头指示因缺失引起的测序结果异常,短箭头指示CTSC基因外显子2的第一个碱基(c.173G)。
由此可见,PLS先证者CTSC基因存在复合型杂合突变,突变位点是外显子5上的c.754C>T 和外显子7上的c.1040A>G,其父母均为携带者,姐姐未发现突变。另外,先证者及其父母、姐姐均在CTSC基因内含子1末尾的T碱基连续序列中发现一个碱基T的杂合缺失。
3. 讨论
PLS属于常染色体隐性遗传疾病,位于常染色体11q14.2的CTSC基因突变是PLS的致病基础[2]。迄今为此,已经发现70多种CTSC基因突变型与PLS有关,其中无义突变和错义突变较多[5]。本实验发现的c.754C>T以前从未报道过,是CTSC基因的新突变位点,c.1040A>G早在Toomes等[3]证实CTSC基因突变是PLS的遗传学基础的研究中已经被发现,并被认为可能是PLS患者CTSC的突变热点[2]。
CTSC基因突变类型中,以纯合突变或纯合子突变合并杂合突变为主,本实验中先证者由两个杂合突变组合的复合型杂合突变较少。目前研究[6]不能确定CTSC基因是否有特异性突变。另外,先证者及其父母、姐姐均在CTSC基因内含子1末尾的T碱基连续序列中发现一个碱基T的杂合缺失,该缺失距离内含子1和外显子2的接合处较远,不会影响CTSC基因内含子的剪接、成熟mRNA的加工及后期mRNA的转录,考虑为CTSC基因多态性位点[7]。
c.754C和c.1040A都位于CTSC基因的高度保守序列[7],c.754C>T致使编码谷氨酰胺的第252位密码子CAA被终止密码子TAA替代,致使后来合成的CTSC肽链缩短;c.1040A>G导致第347位氨基酸密码子TAT被TGT替换,转录过程中半胱氨酸替换正常序列中的酪氨酸(p.Y347C)。半胱氨酸替换酪氨酸,不但改变肽链的结构,还影响肽链的折叠和组装,妨碍CTSC功能蛋白质的形成。c.754C>T和c.1040A>G的复合型杂合突变,可能严重影响CTSC基因合成有功能的蛋白质,致使CTSC的活性显著降低甚至缺乏。
CTSC在中性粒细胞、具有细胞毒性的淋巴细胞、自然杀伤细胞、牙槽骨内巨噬细胞以及肥大细胞等免疫细胞中表达较高,可以活化中性粒细胞衍生的丝氨酸蛋白酶和粒酶,影响免疫反应[8]–[9]。由于CTSC基因突变引起的免疫紊乱增加了PLS患者的炎症易感性,致使PLS患者更容易发生严重的少发的炎症性疾病[6]。所以,可推测本实验中的先证者因CTSC基因的复合型杂合突变致使牙周组织中CTSC活性几乎完全丧失,引起牙周组织的免疫反应紊乱和严重的牙周炎症。先证者乳牙期虽未追溯到明显的牙龈炎症表现,但存在乳牙早脱、萌出恒牙牙周炎重等早发性重度牙周炎表现,与上述推测符合。
大部分PLS患者都有过度的掌跖角化症状和早发的快速进展性牙周炎这两个典型的临床表现,但是少数病例中患者表现为不典型的临床症状或一些少发的临床表现,因此,有学者[5],[10]就CTSC基因的突变位点及类型与PLS患者的临床表型之间的关系进行了研究,结果显示:PLS患者致病基因的遗传异质性低、等位基因异质性高,即PLS的致病基因仅是CTSC基因,CTSC基因发生不同类型的突变产生多种异常表型的概率高;PLS患者显著的表型异质性与特别的基因突变类型间无确切联系,但可能与一些附加基因的修饰和后天环境因素的影响有关。CTSC基因突变是PLS遗传学基础已经被证实,其可作为PLS诊断的新措施,特别适用于一些临床症状不典型但疑为PLS病例的诊断。
目前,PLS的治疗措施有限,掌跖角化病损和牙周破坏都以对症治疗为主。早期给予牙周干预治疗,可以尽量保存PLS患者牙周情况较好的乳恒牙甚至全口健康的恒牙。CTSC基因检测可作为一种筛查手段,增大早期发现PLS的机率。近期研究[11]显示,PLS患者的牙周炎症反应的改变与血清IgE含量变化一致,IgE有可能作为监测PLS牙周炎症反应程度的指示因子。全面了解PLS的病因和致病机制,以及病因与临床表型的关系,从病因出发,采用基因治疗的方法纠正或补偿CTSC基因的异常和缺陷,有望打破PLS治疗方面的困境。
References
- 1.Papillon MM, Lefevre P. Two cases of symmetrically familial palmar and plantar hyperkeratosis (Meleda disease) within brother and sister combined with severe dental alterations in both cases[J] Bull Soc Fr Dermatol Syphiligr. 1924;31(2):82–87. [Google Scholar]
- 2.Dhanawade SS, Shah SD, Kakade GM. Papillon-lefevre syndrome with liver abscess[J] Indian Pediatr. 2009;46(8):723–725. [PubMed] [Google Scholar]
- 3.Toomes C, James J, Wood AJ, et al. Loss-of-function mutations in the cathepsin C gene result in periodontal disease and palmoplantar keratosis[J] Nat Genet. 1999;23(4):421–424. doi: 10.1038/70525. [DOI] [PubMed] [Google Scholar]
- 4.Hart PS, Zhang Y, Firatli E, et al. Identification of cathepsin C mutations in ethnically diverse papillon-Lefèvre syndrome patients[J] J Med Genet. 2000;37(12):927–932. doi: 10.1136/jmg.37.12.927. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.Nagy N, Vályi P, Csoma Z, et al. CTSC and Papillon-Lefèvre syndrome: detection of recurrent mutations in Hungarian patients, a review of published variants and database update[J] Mol Genet Genomic Med. 2014;2(3):217–228. doi: 10.1002/mgg3.61. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Hart TC, Bowden DW, Ghaffar KA, et al. Sublocalization of the Papillon-Lefevre syndrome locus on 11q14-q21[J] Am J Med Genet. 1998;79(2):134–139. doi: 10.1002/(sici)1096-8628(19980901)79:2<134::aid-ajmg9>3.0.co;2-q. [DOI] [PubMed] [Google Scholar]
- 7.Nakano A, Nomura K, Nakano H, et al. Papillon-Lefèvre syndrome: mutations and polymorphisms in the cathepsin C gene[J] J Invest Dermatol. 2001;116(2):339–343. doi: 10.1046/j.1523-1747.2001.01244.x. [DOI] [PubMed] [Google Scholar]
- 8.李 颂, 涂 平, 朱 学骏. Papillon-Lefèvre综合征伴沟纹舌1例[J] 临床皮肤科杂志. 2006;35(2):94–95. [Google Scholar]; Li S, Tu P, Zhu XJ. A case of Papillon-Lefèvre syndrome with fissured tongue[J] J Clin Dermatol. 2006;35(2):94–95. [Google Scholar]
- 9.Sørensen OE, Clemmensen SN, Dahl SL, et al. Papillon-Lefèvre syndrome patient reveals species-dependent requirements for neutrophil defenses[J] J Clin Invest. 2014;124(10):4539–4548. doi: 10.1172/JCI76009. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Sulák A, Tóth L, Farkas K, et al. One mutation, two phenotypes: a single nonsense mutation of the CTSC gene causes two clinically distinct phenotypes[J] Clin Exp Dermatol. 2016;41(2):190–195. doi: 10.1111/ced.12710. [DOI] [PubMed] [Google Scholar]
- 11.Wang X, Liu Y, Liu Y, et al. Long-term change of disease behavior in Papillon-Lefèvre syndrome: seven years follow-up[J] Eur J Med Genet. 2015;58(3):184–187. doi: 10.1016/j.ejmg.2014.12.003. [DOI] [PubMed] [Google Scholar]