Abstract
龋病是发病率最高的口腔慢性感染性疾病,以变异链球菌为代表的一系列细菌曾被认为是龋病的单一致病菌。然而,近几十年来,基于传统致病菌的防治手段未能有效降低龋病的发病率,人们逐渐认识到单一致病菌理论并不能全面反映疾病与微生物的关系。龋病病因学的微生物研究逐步从传统致病菌理论过渡至微生态失衡理论。目前,随着检测手段的不断发展,大量龋病微生物群落研究陆续开展,龋病“核心微生物组”的概念得以提出,即在龋病发生发展中起到关键作用的一组微生物,这将是未来龋病微生物因素研究的方向。
Keywords: 龋病, 口腔微生物, 病因, 变异链球菌, 核心微生物组
Abstract
Dental caries is the most common chronic infectious disease of the oral cavity. The bacterium Streptococcus mutans is the sole pathogen that causes this disease. However, substantial evidence suggests that prevention and treatment strategies developed from traditional “cariogenic pathogen theory” are inefficient in reducing the prevalence of dental caries. An increasing number of individuals adopt the ecological view of the microbiota in the pathogenesis of dental caries. Recent technological improvements have enabled the detection and analysis of oral microorganisms, and many studies have focused on this area. The core microbiota is defined as a cluster of microbes playing critical roles in the initial and development phases of dental caries and may provide future direction for microorganism-related etiological studies.
Keywords: dental caries, oral microbiota, etiology, Streptococcus mutans, core microbiota
龋病是一种以牙体硬组织进行性破坏为特征的慢性感染性疾病。在全球范围内,约有24.3亿人群受到了龋病不同程度的影响[1]。在我国,第三次全国口腔流行病学调查[2]显示,龋病的发病率在5岁儿童中达66.0%,成年人群(35~44岁)达88.1%,老年人群更高达98.4%。目前,基于传统致病菌的防治手段未能有效降低龋病的发病率,越来越多的学者[3]–[5]认识到龋病的发生发展与口腔微生态的失衡存在关系,有关龋病病因学的微生物群落研究得到逐步开展,人们对于龋病相关微生物的认识也经历着不断的变革与发展,本文将其研究进展作一综述。
1. 龋病致病菌理论
龋病作为一种感染性疾病,其致病微生物的相关研究由来已久。早在1924年,英国皇家牙学院的Killian Clarke就在龋损牙体组织中发现了成灵活链状排列的球菌,并命名为变异链球菌(Streptococcus mutans,S. mutans)[6]。19世纪50年代,Keyes和Fitzgerald通过同笼饲养无龋和龋活跃田鼠以及饲喂无龋和龋活跃田鼠粪便第一次验证了龋病是感染性疾病而不是遗传性疾病。接着,Keyes等又进一步通过无菌鼠模型验证了一种从田鼠龋损部位分离的变异链球菌符合Koch法则,是龋病的致病菌。在这一重大发现后,大量的研究小组在人类、猿猴等动物龋损部位也分离培养出了变异链球菌,验证了变异链球菌与人类龋病的相关性。然而进一步的人群研究却发现有些个体,在他们的唾液中,变异链球菌检出率很高,但他们却并没有表现出临床龋损,反之亦然[7]。目前,大量基于龋病单一致病微生物的防治手段也未能有效地在人群中控制疾病的发生发展。流行病学调查显示,2015年全球未经治疗的恒牙龋检出率为34.1%,相较于1990年34.3%的检出率,25年间未出现明显降低[8]。同时,循证医学证据支持,目前包含了变异链球菌计数的龋病风险评估并不能很好地预测龋病的发生发展,目前尚无有效的龋病风险评估方法[9]。因此,这种变异链球菌为主要代表的龋病致病微生物的理论并不能全面地反映疾病与微生物的关系。
2. 龋病微生态理论
20世纪90年代早期,英国口腔微生物学家Marsh[5]首次提出了生态菌斑学说,强调牙菌斑生物膜微环境可以受到外界因素的影响,改变菌群的动态演变方向,最终决定疾病的发生与否。人们进一步认识到龋病的发生发展与微生态的酸碱平衡被打破,生物膜成酸性状态有关。Kleinberg[10]提出,产酸及产碱细菌数量及在微生态中的组成是龋病是否活跃的核心。Takahashi等[11]提出,致龋性微生态是一个从动态稳定阶段过渡至产酸阶段最后发展至耐酸阶段的微生态。基于以上理论,有一部分研究[12]认为微生物的酸性适应及低pH非变异链球菌的选择性增殖是微生态向致龋方向过渡的关键。
与此同时,随着变性梯度凝胶电泳(denatured gradient gel electrophoresis,DGGE)、DNA杂交芯片技术、16s rRNA基因测序技术以及下一代测序技术等不依赖传统培养方法的实验技术的不断发展与成熟,人们了解到口腔内仍存在着大量不可培养微生物,口腔微生态远远比人们先前认识的要更加复杂多样,生态平衡的维系可能涉及到了更多的微生物。2012年,人类微生物计划[13]–[14]公布了其5年来最新研究成果及人类微生物组数据,确定了口腔生态位最多可含有1 300个细菌种和 20 000个基因。这其中不可培养微生物约占总群落的1/3,为进一步了解微生物群落整体在龋病发生发展中的地位及作用,大量研究投入到了这个领域。
低龄儿童龋(early childhood caries,ECC)是儿童最为常见的口腔疾病,其患病率高,进展迅速,累及广泛,治疗难度大,是一个全球性的公共卫生问题[15]。为了解无龋儿童与龋损儿童的口腔微生物组成差异,研究者借助分子生物学及组学手段检测了不同儿童的口腔菌群组成。Jiang等[16]利用DGGE技术比较了无龋、中龋(4≤dmfs≤6)及严重龋易感(dmfs≥10)儿童(共45例)菌斑中34种不同微生物组成,其结果显示缓症链球菌和弯曲菌出现在无龋组但在严重龋易感组消失,二氧化碳噬纤维菌属、伯克氏菌属、普雷沃菌属在严重龋易感组检出率明显降低。Ma等[17]采用人类口腔微生物芯片(human oral microbe identification microarray,HOMIM)比较无龋和严重ECC儿童(各20例)唾液及菌斑微生物认为,变异链球菌、卟啉单胞菌属及放线菌属与严重ECC有极强的相关性;然而另一同样采用HOMIM技术分析了无龋及龋活跃儿童混合牙列唾液样本的实验结果则显示,拟杆菌门、二氧化碳噬纤维菌属、弯曲菌属、月形单胞菌属、类杆菌属、Parvimonas在龋活跃组有更高的检出[18]。Jiang等[19]基于454焦磷酸测序技术对于20对不同龋状况儿童菌斑微生物的分析则发现了这些儿童菌斑中共包含14个菌门,63个菌属,其中10个菌属构成了口腔81%的微生物,16个属的微生物出现在所有个体中,链球菌属、颗粒链菌属、放线菌属的丰度在严重ECC患儿菌斑中相对增加,河口微菌属的丰度则在无龋组相对增加。Tanner等[20]对无龋儿童以及严重ECC儿童接受治疗前后的口腔微生物分析则新发现了Slackia exigua和严重ECC的关系。同时,患龋组儿童可以分为现有龋损牙面较多,但治疗后复发较低组和现有龋损牙面较少但治疗后复发率较高两组。后者往往可检出较高的变异链球菌、普雷沃菌属微生物。Chalmers等[21]则探究了严重ECC患儿感染乳牙牙髓中的微生物,其微生物组成与龋损部位微生物相似,其中变异链球菌仍占主要地位,类斯氏菌属、乳酸菌属、韦荣菌属则可能和牙髓感染的病因有关。
Aas等[22]进行的一项包含年轻恒牙健康及龋病状态下微生物群落组成的研究,通过16s rRNA基因文库比较分析了健康人群及患龋人群的健康釉质表面、白色病损表面菌斑、成洞性牙本质以及深部牙本质菌斑。他们在患龋人群菌斑中检测到197种表型的微生物,其中有50%的微生物是不可培养的。同时,Aas等[22]的研究未能在患龋者健康釉质及白色病损表面菌斑检出变异链球菌,但在成洞性病损牙本质及深部牙本质菌斑中大多能检出变异链球菌,另有10%的患龋者不同部位的菌斑中均未能检出变异链球菌。进一步研究比较了患龋者菌斑的微生物组成,虽然不同个体间差异较大,但韦荣菌属、乳杆菌属、双歧杆菌属、丙酸杆菌属、低pH非变异链球菌、放线菌属、奇异菌属的一些菌种也极有可能在龋病的发生发展中发挥着重要作用。Xiao等[23]通过454焦磷酸测序技术检测了共160例健康及不同程度龋损患者牙面菌斑的微生物情况。该研究发现,被认为是主要致龋菌的变异链球菌在总的菌落中呈现出一个较低的丰度、并且在健康及不同疾病状态分组中不存在差异。而另一种被广泛认可的致龋菌表兄链球菌在龋损组丰度增加具有统计学意义,可能是一种潜在的龋特异性微生物。Johansson等[24]则选择比较了不同龋流行率人群的微生物组成。他们以罗马尼亚人作为高龋流行率人群代表,瑞典人为低龋流行率人群代表,选取了14位罗马尼亚龋活跃青少年,12名瑞典龋活跃青少年和12名瑞典无龋青少年作为研究对象,通过FLX+高通量测序分析了他们全部牙菌斑的微生物构成情况,同时通过聚合酶链式反应(polymerase chain reaction,PCR)技术比较了变异链球菌及表兄链球菌的检出情况。其结果显示,在获得较少口腔卫生防治的罗马尼亚高龋青少年中,变异链球菌与表兄链球菌均有很高的检出率,而在接受了良好口腔卫生防治的瑞典青少年中,这两种常见的致龋菌则被较少检出,瑞典龋活跃青少年的菌群组成主要为放线菌属、月形单胞菌属、普雷沃菌属及二氧化碳噬纤维菌属。
现如今随着人口老龄化的加剧,老年人的口腔健康受到了人们日益广泛的关注。根面龋是影响中老年人口腔健康的最主要疾病之一。根面龋不同于发生在冠方的龋损,具有主要破坏结构为牙骨质及牙本质,进展迅速且隐蔽,治疗过程隔湿等操作难度大,预后相对不佳的特点[25]。由于根面龋发生人群主要集中在中老年,龋病发生部位牙体硬组织矿化程度较低,且根面龋菌斑常常与龈下菌斑相交联,其微生态具有特殊性[26],然而一直以来对于根面龋相关的微生物研究较少被报道。Preza等[27]通过16s rRNA基因测序及文库比对技术分析了老年人根面龋患者(11例)菌斑与健康人群(10例)根面菌斑微生物组成差异。总体样本中54%的微生物是不可培养的,月形单胞菌属、韦荣菌属在所有菌斑中均很常见。虽然个体间的差异较大,但是研究认为根面龋菌斑微生物远比人们先前认为的要复杂,根面龋的致病微生物可能不仅仅是先前根据培养技术推测的变异链球菌、乳杆菌属及部分放线菌。Preza等[28]在一次更大规模的研究中采取了基于16s rRNA基因的微阵列芯片对根面龋及健康老年人群牙本质及菌斑样本进行了分析,进一步认为,健康人群的菌斑样本中所检测得到的微生物种类更加丰富,乳杆菌属及太阳杆菌属部分物种与根面龋存在一定的相关性。2011年,Bizhang等[29]首次通过定量PCR的非培养技术检测了根面龋表面菌斑与健康根面菌斑中9种微生物的数量差异,其结果显示放线菌属、变异链球菌、表兄链球菌、乳酸杆菌、龋齿罗氏菌、微小微单胞菌、痤疮丙酸杆菌、黏膜奈瑟菌在龋损根面牙菌斑中检出数量显著高于健康牙根表面。Chen等[30]开展的一项基于454焦磷酸测序技术分析老年人群根面龋患者(21例)有龋部位和无龋部位菌斑以及无龋患者(21例)菌斑的研究则认为,根面龋部位菌斑与健康组菌斑的生物量、物种丰度及物种结构不存在显著差异。但根面龋菌斑的生物群落变异度更大,且可很好地与健康人群菌斑微生物进行区分,其中产酸丙酸杆菌、变异链球菌、Olsenella profuse、嗜糖普雷沃菌、卷曲乳杆菌与根面龋最具相关性。
这些龋病微生物群落相关横断面研究从人群层面证实了龋病微生态的失衡是由多种微生物参与的,突破了基于传统培养方式得到的对于致病微生物为变异链球菌、乳酸杆菌[31]的认识局限。但不同研究所涉及人群、疾病所处阶段、取样部位、检测手段、统计方法等均有所不同差异,结合口腔微生态本身的复杂多样性,研究所得结果存在较高的异质性,研究所涉及的微生物种类也过于繁多,研究者未能进一步根据这些实验结果对龋病的生态防治提供更多地指导及帮助。
3. 龋病“核心微生物组”
为了更好地了解微生物群落在龋病发生发展中的作用,研究人员拟通过寻找龋病相关的“核心微生物组”,以期获得复杂微生态中发挥关键作用的物种组合或其功能组合,从而为龋病的有效防治提供新的理论基础。
目前,Xiao等[23]研究显示,在所有不同健康及疾病状态下的160位志愿者龈上菌斑共享了7 522个操作分类单元,龈上菌斑存在“核心微生物组”,在属水平的“核心微生物组”则由99个属构成,其中在龋病状态下丰度显著增加的奇异菌属、隐小杆菌属、乳杆菌属等13个菌属则可能是龋病相关的“核心微生物组”。Teng等[32]通过对50名4岁儿童进行了为期2年的追踪研究,其中17名一直处于无龋状态(H2H),21名从无龋状态进入龋活跃状态(H2C),12名一直处于龋活跃状态(C2C)。通过454焦磷酸测序技术分析菌斑及唾液样本的16s rRNA基因序列后,发现4~6岁健康儿童的口腔微生物多样性随年龄增加而增加,微生态不断成熟,H2C组儿童口腔微生物的多样性也会增长,但不如H2H组成熟,C2C组儿童的菌斑以及唾液变化则几乎不受时间影响。此外,研究者发现,随龋病发生而带来的改变不受年龄因素的影响,儿童从健康状态进入到患龋状态时唾液、菌斑中存在部分微生物种群在不同疾病分组的富集程度出现改变,这些从“健康”到“龋病”状态出现变化的微生物可能是龋病发生发展的“核心微生物组”。此外,Yang等[33]对健康及龋病状态志愿者的唾液样本进行全基因组测序则发现,虽然从物种水平难以归纳出“核心微生物组”,然而在功能组合水平可以寻找到来自不同微生物的具有龋病相关特征的“核心微生物功能组”。
4. 结语
人们对于龋病病因的认识已逐渐从单一致病菌理论向微生态失衡理论转变。现阶段的大量群落研究为深入探索龋病微生态提供了有力的参考与提示,龋病“核心微生物组”将可能是调节龋病失衡微生态的关键作用靶点(图1)。
图 1. 龋病病因的微生物因素研究理论发展模式图.
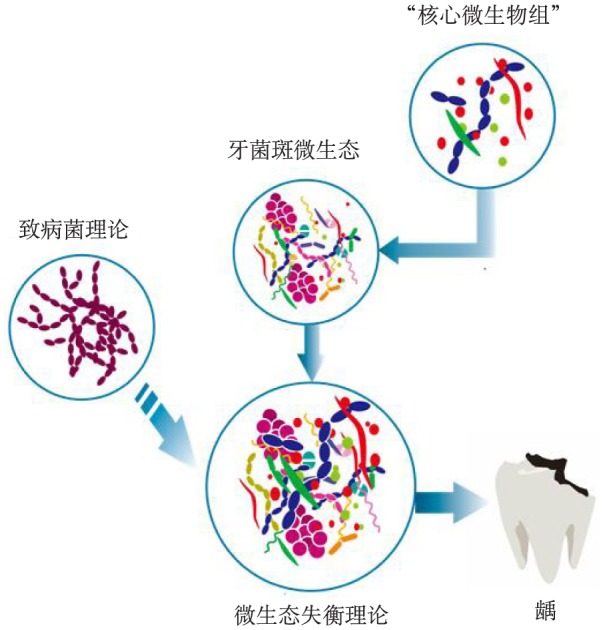
Fig 1 Patten diagram of microbiological etiology studies of dental caries
然而,目前尚缺少对于龋病“核心微生物组”的深入研究。“核心微生物组”是如何调控口腔微生态的;能否寻找有效干预手段进行靶向调节;能否进一步通过靶向抑制龋病“核心微生物组”调节失衡微生态;这一系列问题有待进一步深入研究。
Funding Statement
[基金项目] 国家自然科学基金(81430011)
Supported by: The National Natural Science Foundation of China (81430011).
References
- 1.Vos T, Flaxman AD, Naghavi M, et al. Years lived with disability (YLDs) for 1 160 sequelae of 289 diseases and injuries 1990—2010: a systematic analysis for the Global Burden of Disease Study 2010[J] Lancet. 2012;380(9859):2163–2196. doi: 10.1016/S0140-6736(12)61729-2. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.齐 小秋. 第三次全国口腔健康流行病学调查报告[M] 北京: 人民卫生出版社; 2008. pp. 1–2. [Google Scholar]; Qi XQ. The report of the third national oral health epidemiological survey[M] Beijing: People's Medical Publishing House; 2008. pp. 1–2. [Google Scholar]
- 3.Takahashi N, Nyvad B. Ecological hypothesis of dentin and root caries[J] Caries Res. 2016;50(4):422–431. doi: 10.1159/000447309. [DOI] [PubMed] [Google Scholar]
- 4.Burne RA, Zeng L, Ahn SJ, et al. Progress dissecting the oral microbiome in caries and health[J] Adv Dent Res. 2012;24(2):77–80. doi: 10.1177/0022034512449462. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.Marsh PD. Microbial ecology of dental plaque and its significance in health and disease[J] Adv Dent Res. 1994;8(2):263–271. doi: 10.1177/08959374940080022001. [DOI] [PubMed] [Google Scholar]
- 6.周 学东. 口腔医学史[M] 北京: 人民卫生出版社; 2013. pp. 213–266. [Google Scholar]; Zhou XD. History of stomatology[M] Beijing: People's Medical Publishing House; 2013. pp. 213–266. [Google Scholar]
- 7.Lamont RJ, Jenkinson HF. Oral microbiology at a glance[M] New Jersey: John Wiley & Sons; 2010. pp. 89–117. [Google Scholar]
- 8.Kassebaum NJ, Smith AGC, Bernabé E, et al. Global, regional, and national prevalence, incidence, and disability-adjusted life years for oral conditions for 195 countries, 1990—2015: a systematic analysis for the global burden of diseases, injuries, and risk factors[J] J Dent Res. 2017;96(4):380–387. doi: 10.1177/0022034517693566. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Ewoldsen N, Koka S. There are no clearly superior methods for diagnosing, predicting, and noninvasively treating dental caries[J] J Evid Based Dent Pract. 2010;10(1):16–17. doi: 10.1016/j.jebdp.2009.11.008. [DOI] [PubMed] [Google Scholar]
- 10.Kleinberg I. A mixed-bacteria ecological approach to understanding the role of the oral bacteria in dental caries causation: an alternative to Streptococcus mutans and the specific-plaque hypothesis[J] Crit Rev Oral Biol Med. 2002;13(2):108–125. doi: 10.1177/154411130201300202. [DOI] [PubMed] [Google Scholar]
- 11.Takahashi N, Nyvad B. Caries ecology revisited: microbial dynamics and the caries process[J] Caries Res. 2008;42(6):409–418. doi: 10.1159/000159604. [DOI] [PubMed] [Google Scholar]
- 12.Takahashi N, Nyvad B. The role of bacteria in the caries process: ecological perspectives[J] J Dent Res. 2011;90(3):294–303. doi: 10.1177/0022034510379602. [DOI] [PubMed] [Google Scholar]
- 13.Human Microbiome Project Consortium. Structure, function and diversity of the healthy human microbiome[J] Nature. 2012;486(7402):207–214. doi: 10.1038/nature11234. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Human Microbiome Project Consortium. A framework for human microbiome research[J] Nature. 2012;486(7402):215–221. doi: 10.1038/nature11209. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.石 四箴. 儿童口腔医学[M] 北京: 人民卫生出版社; 2011. pp. 112–113. [Google Scholar]; Shi SZ. Pediatric stomatology[M] Beijing: People's Medical Publishing House; 2011. pp. 112–113. [Google Scholar]
- 16.Jiang W, Jiang Y, Li C, et al. Investigation of supragingival plaque microbiota in different caries status of Chinese preschool children by denaturing gradient gel electrophoresis[J] Microb Ecol. 2011;61(2):342–352. doi: 10.1007/s00248-010-9753-z. [DOI] [PubMed] [Google Scholar]
- 17.Ma C, Chen F, Zhang Y, et al. Comparison of oral microbial profiles between children with severe early childhood caries and caries-free children using the human oral microbe identification microarray[J] PloS One. 2015;10(3):e0122075. doi: 10.1371/journal.pone.0122075. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Luo AH, Yang DQ, Xin BC, et al. Microbial profiles in saliva from children with and without caries in mixed dentition[J] Oral Dis. 2012;18(6):595–601. doi: 10.1111/j.1601-0825.2012.01915.x. [DOI] [PubMed] [Google Scholar]
- 19.Jiang W, Zhang J, Chen H. Pyrosequencing analysis of oral microbiota in children with severe early childhood dental caries[J] Curr Microbiol. 2013;67(5):537–542. doi: 10.1007/s00284-013-0393-7. [DOI] [PubMed] [Google Scholar]
- 20.Tanner AC, Kent RL, Holgerson PL, et al. Microbiota of severe early childhood caries before and after therapy[J] J Dent Res. 2011;90(11):1298–1305. doi: 10.1177/0022034511421201. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.Chalmers NI, Oh K, Hughes CV, et al. Pulp and plaque microbiotas of children with severe early childhood caries[J] J Oral Microbiol. 2015;7:25951. doi: 10.3402/jom.v7.25951. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22.Aas JA, Griffen AL, Dardis SR, et al. Bacteria of dental caries in primary and permanent teeth in children and young adults[J] J Clin Microbiol. 2008;46(4):1407–1417. doi: 10.1128/JCM.01410-07. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.Xiao C, Ran S, Huang Z, et al. Bacterial diversity and community structure of supragingival plaques in adults with dental health or caries revealed by 16s pyrosequencing[J] Front Microbiol. 2016;7:1145. doi: 10.3389/fmicb.2016.01145. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.Johansson I, Witkowska E, Kaveh B, et al. The microbiome in populations with a low and high prevalence of caries[J] J Dent Res. 2016;95(1):80–86. doi: 10.1177/0022034515609554. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.Shay K. Root caries in the older patient: significance, prevention, and treatment[J] Dent Clin North Am. 1997;41(4):763–793. [PubMed] [Google Scholar]
- 26.Ravald N. Root surface caries[J] Curr Opin Periodontol. 1994:78–86. [PubMed] [Google Scholar]
- 27.Preza D, Olsen I, Aas JA, et al. Bacterial profiles of root caries in elderly patients[J] J Clin Microbiol. 2008;46(6):2015–2021. doi: 10.1128/JCM.02411-07. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 28.Preza D, Olsen I, Willumsen T, et al. Microarray analysis of the microflora of root caries in elderly[J] Eur J Clin Microbiol Infect Dis. 2009;28(5):509–517. doi: 10.1007/s10096-008-0662-8. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.Bizhang M, Ellerbrock B, Preza D, et al. Detection of nine microorganisms from the initial carious root lesions using a TaqMan-based real-time PCR[J] Oral Dis. 2011;17(7):642–652. doi: 10.1111/j.1601-0825.2011.01815.x. [DOI] [PubMed] [Google Scholar]
- 30.Chen L, Qin B, Du M, et al. Extensive description and comparison of human supra-gingival microbiome in root caries and health[J] Plos One. 2015;10(2):e0117064. doi: 10.1371/journal.pone.0117064. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 31.Alaluusua S, Kleemola-Kujala E, Nystrom M, et al. Caries in the primary teeth and salivary Streptococcus mutans and lactobacillus levels as indicators of caries in permanent teeth[J] Pediatr Dent. 1987;9(2):126–130. [PubMed] [Google Scholar]
- 32.Teng F, Yang F, Huang S, et al. Prediction of early childhood caries via spatial-temporal variations of oral microbiota[J] Cell Host Microbe. 2015;18(3):296. doi: 10.1016/j.chom.2015.08.005. [DOI] [PubMed] [Google Scholar]
- 33.Yang F, Ning K, Zeng X, et al. Characterization of saliva microbiota's functional feature based on metagenomic sequencing[J] Springerplus. 2016;5(1):2098. doi: 10.1186/s40064-016-3728-6. [DOI] [PMC free article] [PubMed] [Google Scholar]


