Abstract
目的
探讨环状RNA hsa_circ_0002203对口腔鳞状细胞癌(OSCC)细胞系恶性生物学行为的影响。
方法
纳入OSCC患者40例,使用实时荧光聚合酶链反应检测环状RNA hsa_circ_0002203在OSCC组织、癌旁组织、OSCC细胞系和人口腔黏膜角质形成细胞(HOK)中的表达水平。慢病毒感染SCC15和CAL27细胞,实时荧光聚合酶链反应检测环状RNA hsa_circ_0002203的表达,细胞计数(CCK-8)实验检测细胞增殖能力,划痕实验和Transwell迁移及侵袭实验检测细胞迁移侵袭能力,流式细胞凋亡实验检测细胞凋亡水平,蛋白质印迹法检测细胞增殖凋亡侵袭相关蛋白的表达。通过裸鼠成瘤实验观察hsa_circ_0002203对SCC15细胞体外成瘤能力的影响。
结果
环状RNA hsa_circ_0002203在OSCC组织的表达低于癌旁组织(P<0.01),在OSCC细胞系中的表达低于人类角质形成细胞(P<0.001)。慢病毒感染SCC15和CAL27细胞后hsa_circ_0002203的表达增加;SCC15和CAL27细胞的增殖、迁移和侵袭能力下降,凋亡水平增加;裸鼠肿瘤体积、质量减小,生长速度降低。
结论
环状RNA hsa_circ_0002203在口腔鳞状细胞癌中的低表达可以增强肿瘤细胞的增殖、迁移和侵袭能力,抑制肿瘤细胞凋亡。
Keywords: 口腔鳞状细胞癌, 环状RNA hsa_circ_0002203, 增殖, 迁移, 侵袭
Abstract
Objective
To investigate the effect of circular RNA hsa_circ_0002203 on the malignant biological behavior of oral squamous cell carcinoma (OSCC) cell lines.
Methods
Forty patients with oral squamous cell carcinoma were included. Real-time fluorescent polymerase chain reaction (PCR) was used to detect the expression level of circular RNA hsa_circ_0002203 in OSCC and corresponding adjacent tissues, OSCC cell lines, and human oral keratinocytes (HOK). SCC15 and CAL27 cells were transfected with lentivirus. The expression level of circular RNA hsa_circ_0002203 was detected by real-time fluorescent PCR. Cell proliferation was detected by cell counting assay (CCK-8). Cell migration and invasion ability was detected by scratch assay and Transwell migration and invasion assay. Apoptosis level was detected by flow cytometry. The expression of corresponding protein was detected by Western blot. Murine tumor formation experiments were performed to observe the effect of hsa_circ_0002203 on the tumorigenesis of SCC15 cells in vivo.
Results
The expression of circular RNA hsa_circ_0002203 in OSCC tissues was lower than that in adjacent tissues (P<0.01), and the expression in OSCC cell lines was lower than that in HOK (P<0.001). Hsa_circ_0002203 expression increased after the lentiviral infection of SCC15 and CAL27. The proliferation, migration, and invasion of SCC15 and CAL27 reduced, and apoptosis level was promoted. The tumor volume, weight decreased, and growth rate of nude mice decreased.
Conclusion
The low expression of circular RNA hsa_circ_0002203 in oral squamous cell carcinoma can enhance the proliferation, migration, and invasion of cancer cells and inhibit tumor cell apoptosis.
Keywords: oral squamous cell carcinoma, circular RNA hsa_circ_0002203, proliferation, migration, invasion
口腔鳞状细胞癌(oral squamous cell carcinoma,OSCC)是一种常见的头颈部恶性肿瘤,约占全身恶性肿瘤的3%[1]。烟草、酒精以及感染高风险类型的人乳头瘤病毒是OSCC重要的致病诱因[2]。2008年,全世界被诊断为OSCC的患者约为263 900名,因OSCC死亡的患者达到128 000名[3]。2012年,以上数据分别达到300 400名和145 400名[4]。虽然手术治疗、放化疗已在OSCC临床治疗中广泛应用,但由于OSCC较高的颈部淋巴结转移率,其5年生存率依然徘徊在50%左右。
环状RNA(circRNA)是由外显子或内含子剪切组成的环化转录物序列,是自然发生的具有广泛性和多样性的内源性非编码RNA。其通常是稳定的、丰富的、保守的RNA分子,并表现出组织的特异性表达[5]–[7]。环状RNA与多种肿瘤关系密切,包括结肠癌[8]、卵巢癌[9]、胃癌[10]、食道癌[11]和胶质瘤[12] 等。环状RNA是非编码基因组的新成员,具有不同的生物学特性和功能,目前与肿瘤相关的内源性环状RNA的报道逐渐增加。然而,绝大多数环状RNA的功能尚待发现,其在OSCC中的报道更为罕见[13] 。研究[14]报道,circDOCK1在OSCC中上调,生物信息学分析发现circDOCK1可以通过circDOCK1/miR-196a-5p/BIRC3轴调节BIRC3的表达,并参与了OSCC凋亡过程。另有研究[15]表明,circRNA_100290在OSCC组织中上调,circRNA_100290可作为竞争性内源RNA起作用,通过吸附海绵状miR-29b家族成员调节CDK6表达,从而影响OSCC细胞系的增殖能力。
本课题组前期研究通过高通量测序技术筛选出OSCC中差异性环状RNA hsa_circ_0002203,并发现该环状RNA在OSCC细胞系SCC15及CAL27中均低表达。本研究拟通过过表达OSCC细胞系SCC15和CAL27中的hsa_circ_0002203,观察这2种细胞的增殖、迁移、侵袭能力、凋亡水平以及相关蛋白的表达,初步探索hsa_circ_0002203在OSCC恶性生物学行为中的作用,为靶向治疗奠定分子生物学基础。
1. 材料和方法
本研究已通过北京大学伦理审查(项目编号:37923/2-3-2012)。所有患者均知情同意并签署知情同意书。
1.1. 病历资料
本研究纳入2017年3月—2018年10月因OSCC于北京大学深圳医院口腔颌面外科就诊的患者40例,纳入标准:原发性OSCC;术前未作放疗、化疗及生物治疗;无全身系统性疾病,如高血压、糖尿病及免疫系统疾病等。术中切除肿瘤组织及癌旁组织并立即转移至液氮中保存。病理诊断由病理科依据2017年世界卫生组织标准执行。
1.2. 材料和试剂
人类OSCC细胞株CAL27、SCC25、SCC15、SCC9和人口腔黏膜角质形成细胞(human oral keratinocytes,HOK)由武汉大学口腔医学院和北京大学口腔医学院惠赠。达尔伯克改良伊格尔培养基(Dulbecco's modified Eagle medium,DMEM)、胎牛血清(Gibco公司,美国),RNeasy迷你试剂盒(QIAGEN公司,德国),Prime Script RT Master Mix试剂盒(Takara公司,日本),聚合酶链反应(polymerase chain reaction,PCR)Master Mix(2x)试剂盒(Thermo Fisher Scientific公司,美国),B淋巴细胞瘤-2基因抗体(B cell lymphoma/lewkmia-2,Bcl-2)、Bcl-2基因相关X蛋白(Bcl-2 associated X protein,BAX)、基质金属蛋白酶-9(matrix metalloproteinase-9,MMP-9)、细胞周期蛋白D1(CyclinD1)、甘油醛-3-磷酸脱氢酶抗体(glyceraldehyde-3-phosphate dehydrogenase antibody,GAPDH)(CST公司,美国),嘌呤霉素、Transwell小室、V-FITC凋亡测定试剂盒(上海碧云天生物技术有限公司)。慢病毒载体构建于上海汉恒生物有限公司。无胸腺裸鼠购自中国湖南斯莱克景达公司。
1.3. 细胞培养及感染
用含10%胎牛血清的DMEM培养基培养OSCC细胞系(CAL27、SCC9、SCC15、SCC25)和HOK。细胞于37 °C、5%CO2浓度的恒温培养箱中培养。
常规铺板SCC15和CAL27细胞,待细胞贴壁并且融合度为50%时进行慢病毒感染。感染48 h后,用含有适当浓度嘌呤霉素(SCC15:10 µg·mL−1;CAL27:8 µg·mL−1)的DMEM培养基替换以筛选稳定的细胞株。倒置荧光显微镜下观察绿色荧光蛋白(green fluorescent protein,GFP)表达情况。实验组为过表达目的基因hsa_circ_0002203的肿瘤细胞,对照组为仅含空载体的肿瘤细胞。
1.4. 实时荧光PCR检测环状RNA hsa_circ_0002203的表达
用实时荧光PCR检测环状RNA hsa_circ_0002203在OSCC组织、癌旁组织、OSCC细胞系和HOK中的表达水平,以及慢病毒感染SCC15、CAL27后hsa_circ_0002203的表达水平。用RNeasy迷你试剂盒分离总RNA,核糖核酸酶R(ribonuclease R,RNase R)3 U·mg−1在37 °C下处理总RNA 15 min,Prime Script RT Master Mix逆转录提取的RNA。使用PCR Master Mix(2x)进行PCR反应。实时定量PCR的引物序列如下。hsa_circ_0002203:上游5′-ATGTAGGCTGTTACCTCAAGGC-3′,下游5′-AGCACTGAGTCCCAGCTGAA-3′。GAPDH:上游5′-CATCACCATCTTCCAGGAGCG-3′,下游5′-TGACCTTGCCCACAGCCTT-3′。PCR反应条件:预变性95 °C 10 min后,95 °C 30 s,60 °C 1 min,72 °C 30 s,40个循环。使用2-ΔΔCt法量化mRNA表达水平。实验重复3次。
1.5. 细胞计数(cell counting kit-8,CCK-8)实验检测细胞增殖能力
将感染后的SCC15和CAL27细胞以每孔2 000个细胞接种于96孔板。间隔24 h更换100 µL新鲜完全培养基(被测试孔每孔加10 µL CCK-8溶液及90 µL新鲜完全培养物)。将96孔板置于培养箱中继续培养1 h,使用酶标仪检测450 nm双波长的吸光度值(A450)。
1.6. 划痕实验检测细胞迁移侵袭能力
将感染后的SCC15和CAL27细胞铺于六孔板至细胞融合度达90%左右。用200 µL无菌移液器尖端垂直于6孔板划线,磷酸盐缓冲液洗涤,拍照并记为0 h,更换无FBS的DMEM培养基继续培养,24 h后拍照记录。实验重复3次。
1.7. Transwell迁移及侵袭实验检测细胞迁移侵袭能力
用Transwell小室、Transwell预涂覆基质胶小室分别进行迁移和侵袭实验。将均质的感染后SCC15和CAL27细胞悬浮液(无血清培养基重悬)加入到上室中,下室加入含10%FBS的完全培养基分别孵育24 h和48 h。吸去小室内培养基并用PBS洗涤,用棉签擦去上室细胞,4%多聚甲醛固定30 min,PBS洗涤3次,通风橱晾干,0.1%结晶紫染色30 min,PBS洗净干燥后显微镜下拍照。
1.8. 流式细胞凋亡实验检测细胞凋亡水平
将感染后SCC15和CAL27细胞铺于6孔板中,培养48 h后使其融合度接近100%,然后用不含EDTA的胰蛋白酶消化离心。将细胞悬浮于1×膜联蛋白结合缓冲液中,然后将5 µL膜联蛋白V和1 µL 碘化丙啶(propidium iodide,PI)试剂加入100 µL细胞悬浮液中并混合。室温避光孵育15 min。向每个样品中加入400 µL 1×膜联蛋白结合缓冲液终止染色。使用FACSCalibur流式细胞仪测定凋亡率。
1.9. 蛋白质印迹法(Western blot)检测细胞增殖凋亡侵袭相关蛋白的表达
培养感染后SCC15和CAL27细胞至对数生长期。常规提取、定量蛋白质。使用Bio-Rad Bis-Tris凝胶系统(北京索莱宝科技有限公司)建立Western印迹系统。膜在4 °C摇床孵育过夜(用一抗稀释液以1︰2 000的比例稀释一抗原液,一抗分别为GAPDH、BAX、Bcl-2、CyclinD1、MMP-9)。Tris-HCl缓冲盐溶液充分洗涤,室温下孵育二抗1 h(用TBST以1︰1 000的比例稀释二抗原液)。Tris-HCl缓冲盐溶液充分洗涤。使用Millipore化学发光辣根过氧化物底物发光,使用Image J软件(美国国家心理健康研究所)量化条带。
1.10. 裸鼠成瘤实验
随机将4周龄Balb/c无胸腺裸鼠分为实验组和对照组,每组各8只。将感染过表达目的基因hsa_circ_0002203和空载体的SCC15细胞(100 µL PBS中含1×107个细胞)分别注射到实验组和对照组裸鼠腋部皮下。观察测量并计算每周的肿瘤体积:V=πAB2/6,其中A为肿瘤最大直径,B为肿瘤垂直直径,V为肿瘤体积)。6周后,将裸鼠安乐死并称重。
1.11. 统计学方法
使用SPSS 20.0软件进行统计分析。数据经K-S检验证实符合正态分布,以均数±标准差表示。采用GraphPad Prism 5.0软件绘图。组间比较采用t检验。以双侧P<0.05为差异有统计学意义。
2. 结果
2.1. OSCC组织及癌旁组织中hsa_circ_0002203表达水平
OSCC组织及癌旁组织中hsa_circ_0002203的表达水平分别为9.91±0.36、11.80±0.45,OSCC组织的表达低于癌旁组织(t=3.26,P<0.01)。
2.2. OSCC细胞系和HOK中hsa_circ_0002203的表达水平
hsa_circ_0002203在OSCC细胞系中的表达(CAL27:0.29±0.02;SCC9:0.33±0.04;SCC15:0.19±0.03;SCC25:0.32±0.05)显著低于HOK(1.04±0.03)(t值分别为36.28、16.13、28.06、14.92,P值均小于0.001)。
2.3. 慢病毒感染对hsa_circ_0002203表达的影响
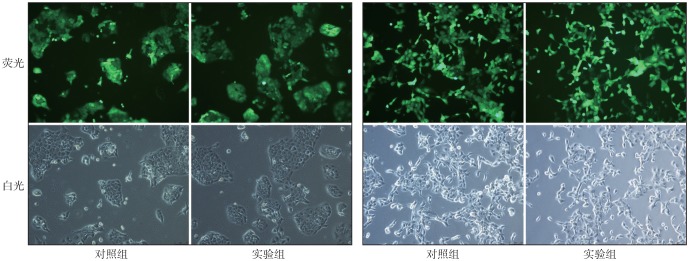
倒置荧光显微镜下观察可见,感染成功的细胞带有GFP绿色荧光(图1)。慢病毒感染SCC15、CAL27后hsa_circ_0002203的表达分别为99.20±3.81和62.55±2.52,高于对照组的表达1.028±0.03和1.06±0.03(t值分别为25.74、24.45,P值均小于0.001)。
图 1. 慢病毒感染后hsa_circ_0002203表达水平 × 200.
Fig 1 Hsa_circ_0002203 expression level after lentivirus infection × 200
左:SCC15;右:CAL27。
2.4. hsa_circ_0002203对SCC15和CAL27细胞增殖能力的影响
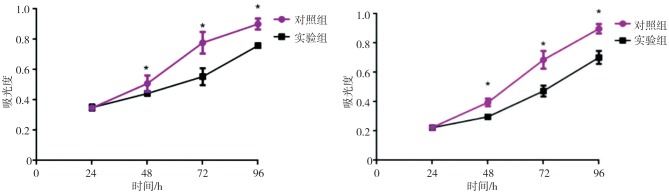
CCK-8实验结果(图2)显示:随时间延长,实验组SCC15和CAL27细胞增殖能力低于对照组(48 h时,tSCC15=8.06,tCAL27=8.00;72 h时tSCC15=16.54,tCAL27=11.21;96 h时tSCC15=14.42,tCAL27=10.86;P值均小于0.05)。
图 2. 慢病毒感染SCC15和CAL27后细胞增殖活性.
Fig 2 Proliferative activity of cells after lentiviral infection of SCC15 and CAL27
左:SCC15;右:CAL27。*同一时间的实验组与对照组相比,P<0.05。
2.5. hsa_circ_0002203对SCC15和CAL27细胞迁移能力的影响
划痕实验结果(图3)显示:实验组SCC15和CAL27细胞的划痕愈合速度均低于对照组(tSCC15=7.27,tCAL27=6.47,P值均小于0.05)。
图 3. 慢病毒感染SCC15和CAL27后划痕实验结果.
Fig 3 Scratch test results after lentiviral infection of SCC15 and CAL27
左:SCC15;右:CAL27。
Transwell迁移实验结果(图4)显示,实验组穿过小室的细胞数(SCC15:188.40±10.39;CAL27:51.40±4.20)均低于对照组(SCC15:335.60±7.99;CAL27:108.00±4.58)(tSCC15=11.24,tCAL27=9.10,P值均小于0.001)。表明实验组SCC15和CAL27细胞的迁移速度低于对照组细胞。
图 4. 慢病毒感染SCC15和CAL27后Transwell迁移结果.
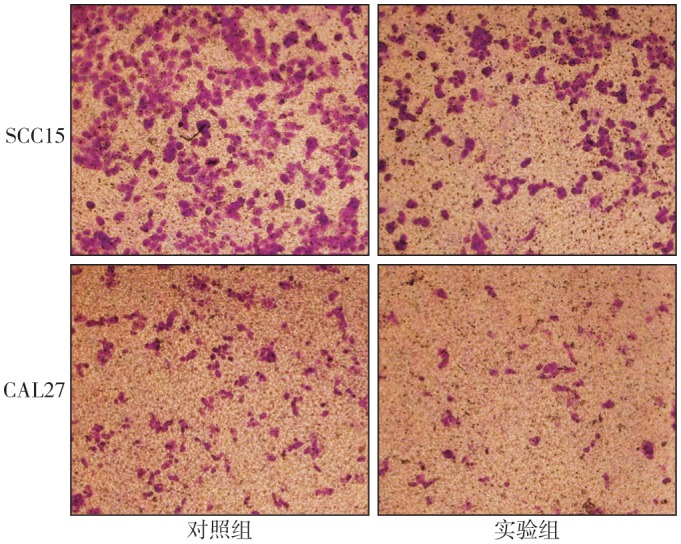
Fig 4 Transwell migration results after lentiviral infection of SCC15 and CAL27
2.6. hsa_circ_0002203对SCC15和CAL27细胞侵袭能力的影响
Transwell侵袭实验结果(图5)显示:实验组穿过小室的细胞数(SCC15:204.20±9.75;CAL27:68.20±3.28)均低于对照组(SCC15:299.40±13.31;CAL27:107.40±5.04)(tSCC15=5.77,tCAL27=6.52,P值均小于0.001)。
图 5. 慢病毒感染SCC15和CAL27后Transwell侵袭结果.
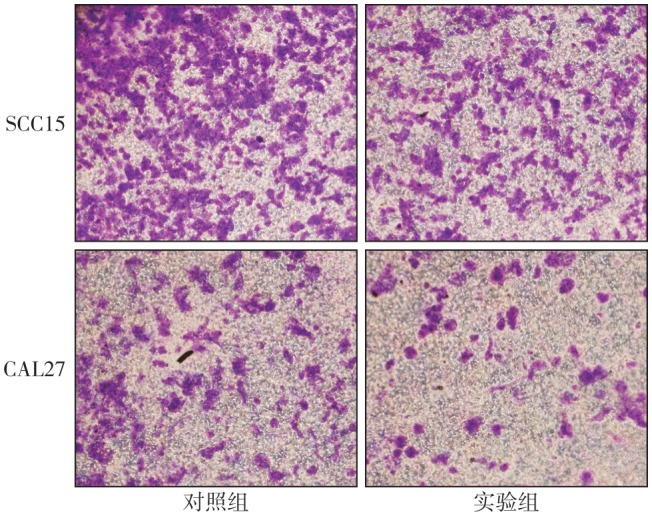
Fig 5 Transwell invasion results after lentiviral infection of SCC15 and CAL27
2.7. hsa_circ_0002203对SCC15和CAL27细胞凋亡能力的影响
流式细胞凋亡实验结果显示:实验组细胞凋亡率(SCC15:0.19%±0.02%;CAL27:0.60%±0.03%)均高于对照组(SCC15:0.05%±0.01%;CAL27:0.04%±0.01%)(tSCC15=6.48,tCAL27=19.02,P值均小于0.001)。
2.8. hsa_circ_0002203对SCC15和CAL27细胞增殖凋亡侵袭相关蛋白表达的影响
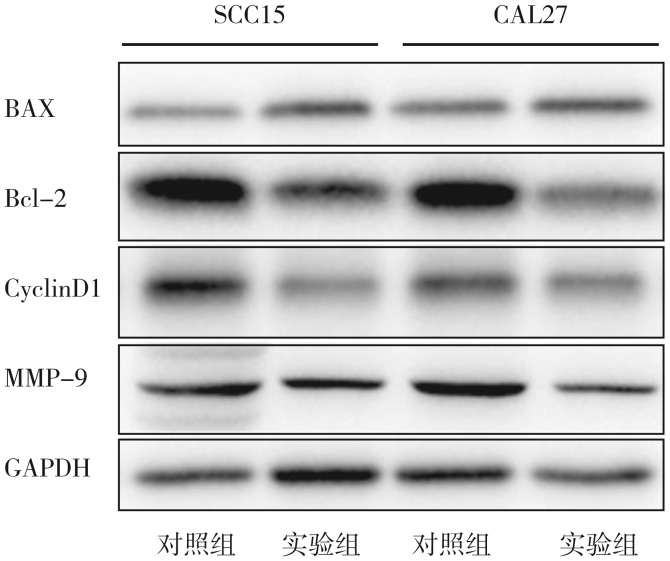
蛋白印迹法检测结果(图6、表1)显示,实验组BAX蛋白表达高于对照组(P<0.01),Bcl-2、CyclinD1、MMP-9蛋白表达低于对照组(P<0.01)。
图 6. 慢病毒感染SCC15和CAL27后细胞增殖凋亡侵袭相关蛋白的表达.
Fig 6 Expression of cell proliferation and apoptosis related proteins after lentiviral infection of SCC15 and CAL27
表 1. 慢病毒感染SCC15和CAL27后细胞增殖凋亡侵袭相关蛋白的表达.
Tab 1 Expression of cell proliferation and apoptosis related proteins after lentiviral infection of SCC15 and CAL27
| 蛋白 | SCC15 |
CAL27 |
||||||
| 实验组 | 对照组 | t值 | P值 | 实验组 | 对照组 | t值 | P值 | |
| BAX | 1.18±0.03 | 1.00±0.01 | 6.55 | <0.01 | 1.40±0.04 | 0.99±0.01 | 9.75 | <0.01 |
| Bcl-2 | 0.51±0.03 | 1.00±0.01 | 16.43 | <0.01 | 0.68±0.03 | 1.00±0.06 | 4.78 | <0.01 |
| CyclinD1 | 0.44±0.02 | 1.03±0.07 | 8.71 | <0.01 | 0.76±0.02 | 1.05±0.05 | 5.44 | <0.01 |
| MMP-9 | 0.46±0.02 | 1.07±0.05 | 11.55 | <0.01 | 0.55±0.03 | 1.02±0.05 | 8.44 | <0.01 |
2.9. hsa_circ_0002203对SCC15细胞体外成瘤能力的影响
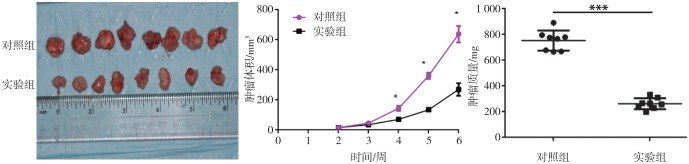
裸鼠成瘤实验结果(图7)显示:实验组裸鼠肿瘤体积小于对照组,肿瘤生长速度慢于对照组,肿瘤质量(262.90 mg±15.09 mg)小于对照组(753.40 mg±27.65 mg)(tSCC15=15.15,P<0.001)。
图 7. 裸鼠成瘤实验.
Fig 7 Results of tumor formation in nude mice
左:6周时肿瘤大小;中:肿瘤体积增长曲线;右:6周时肿瘤质量。*P<0.05,***P<0.001。
3. 讨论
由于口腔颌面部解剖结构的特殊性,OSCC容易侵犯周围组织,导致颈部淋巴结转移以及血行性转移。随着医疗技术的不断发展,尽管在肿瘤诊断和治疗方面已经取得了实质性进展,然而多数晚期OSCC患者仍表现为广泛的远处转移。伴随着发病率的上升和疾病年轻化,OSCC仍然是头颈部致命的疾病[16]。
环状RNA是一类不具有5′末端帽子和3′末端poly(A)尾巴结构,并以共价键形成环形的非编码RNA分子[8]。由于其结构的特殊性,环状RNA的功能也与线性RNA有很大不同。环状RNA可以通过靶向吸附miRNA来调控基因表达[17]。在胶质母细胞瘤中,ciRS-7可以靶向吸附miR-7并显著抑制其活性,上调miR-7靶基因表皮生长因子受体(epidermal growth factor receptor,EGFR)的表达,从而导致肿瘤细胞恶性程度的增高[18]。在乳腺癌中,由FOXO3基因编码的circ-Foxo3表达明显下调,circ-Foxo3的异位表达能够诱导细胞凋亡;与此同时,circ-Foxo3通过促进mdm2诱导的p53泛素化和随后的降解来抑制p53水平,从而影响乳腺癌的发生发展[19]。
目前有关环状RNA在头颈部鳞状细胞癌发生发展机制中的研究报道罕见。本课题组前期研究通过高通量测序实验筛选出差异性表达的环状RNA hsa_circ_0002203[20]。该环状RNA的母基因为钙调蛋白结合转录激活因子1(calmodulin-binding transcriptional activator1,CAMTA1),定位于1号染色体上。在OSCC癌组织中,hsa_circ_0002203的表达水平较癌旁组织显著下降,并且与颈淋巴结转移相关,提示其存在潜在的临床研究意义。同时OSCC细胞株中hsa_circ_0002203表达水平较HOK显著下调,通过慢病毒转染SCC15和CAL27细胞系后,肿瘤细胞的增殖、迁移和侵袭能力明显被抑制,且凋亡水平也相应增高。Bcl-2和BAX是参与细胞增殖和凋亡的关键指标[21]–[22],MMP-9在肿瘤侵袭和转移中起关键作用[23],CyclinD1是参与细胞周期调控的重要蛋白[24]。本研究结果显示,hsa_circ_0002203高表达后,BAX上调,而Bcl-2、MMP-9和CyclinD1下调。这些结果表明,hsa_circ_0002203直接或间接地参与并影响了与肿瘤增殖迁移侵袭相关重要的蛋白质的表达过程。体外裸鼠成瘤实验也证实,高表达hsa_circ_0002203的肿瘤细胞成瘤能力与对照组相比明显被抑制。这说明hsa_circ_0002203在OSCC细胞系中可能起到调控作用。目前关于hsa_circ_0002203在头颈部鳞状细胞癌的研究尚属于探索阶段,其内在机制也有待于后期实验的开展。
Funding Statement
[基金项目] 国家自然科学基金(81572654);深圳市三名工程项目(北京大学口腔医院口腔颌面外科俞光岩教授团队,SZSM 201512036)
Supported by: The National Natural Science Foundation of China (81572654); The Sanming Project for Medicine in Shenzhen (Oral and Maxillofacial Surgery Team, Professor Yu Guangyan, Stomatology Hospital Peking University, SZSM 201512036).
Footnotes
作者声明本文无利益冲突。
References
- 1.Vigneswaran N, Williams MD. Epidemiologic trends in head and neck cancer and aids in diagnosis[J] Oral Maxillofac Surg Clin North Am. 2014;26(2):123–141. doi: 10.1016/j.coms.2014.01.001. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Scully C, Bagan J. Oral squamous cell carcinoma overview[J] Oral Oncol. 2009;45(4/5):301–308. doi: 10.1016/j.oraloncology.2009.01.004. [DOI] [PubMed] [Google Scholar]
- 3.Jemal A, Bray F, Center MM, et al. Global cancer statistics[J] CA Cancer J Clin. 2011;61(2):69–90. doi: 10.3322/caac.20107. [DOI] [PubMed] [Google Scholar]
- 4.Torre LA, Bray F, Siegel RL, et al. Global cancer statistics, 2012[J] CA Cancer J Clin. 2015;65(2):87–108. doi: 10.3322/caac.21262. [DOI] [PubMed] [Google Scholar]
- 5.Salzman J, Gawad C, Wang PL, et al. Circular RNAs are the predominant transcript isoform from hundreds of human genes in diverse cell types[J] PLoS One. 2012;7(2):e30733. doi: 10.1371/journal.pone.0030733. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Lasda E, Parker R. Circular RNAs: diversity of form and function[J] RNA. 2014;20(12):1829–1842. doi: 10.1261/rna.047126.114. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Jeck WR, Sorrentino JA, Wang K, et al. Circular RNAs are abundant, conserved, and associated with ALU repeats[J] RNA. 2013;19(2):141–157. doi: 10.1261/rna.035667.112. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Dou YC, Cha DJ, Franklin JL, et al. Circular RNAs are down-regulated in KRAS mutant colon cancer cells and can be transferred to exosomes[J] Sci Rep. 2016;6:37982. doi: 10.1038/srep37982. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Ahmed I, Karedath T, Andrews SS, et al. Altered expression pattern of circular RNAs in primary and metastatic sites of epithelial ovarian carcinoma[J] Oncotarget. 2016;7(24):36366–36381. doi: 10.18632/oncotarget.8917. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Chen J, Li Y, Zheng Q, et al. Circular RNA profile identifies circPVT1 as a proliferative factor and prognostic marker in gastric cancer[J] Cancer Lett. 2017;388:208–219. doi: 10.1016/j.canlet.2016.12.006. [DOI] [PubMed] [Google Scholar]
- 11.Xia WJ, Qiu MT, Chen R, et al. Circular RNA has_circ_0067934 is upregulated in esophageal squamous cell carcinoma and promoted proliferation[J] Sci Rep. 2016;6:35576. doi: 10.1038/srep35576. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Yang P, Qiu ZJ, Jiang Y, et al. Silencing of cZNF292 circular RNA suppresses human glioma tube formation via the Wnt/β-catenin signaling pathway[J] Oncotarget. 2016;7(39):63449–63455. doi: 10.18632/oncotarget.11523. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Kristensen LS, Hansen TB, Venø MT, et al. Circular RNAs in cancer: opportunities and challenges in the field[J] Oncogene. 2018;37(5):555–565. doi: 10.1038/onc.2017.361. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Wang LP, Wei YX, Yan YY, et al. CircDOCK1 suppresses cell apoptosis via inhibition of miR-196a-5p by targeting BIRC3 in OSCC[J] Oncol Rep. 2018;39(3):951–966. doi: 10.3892/or.2017.6174. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Chen L, Zhang S, Wu J, et al. circRNA_100290 plays a role in oral cancer by functioning as a sponge of the miR-29 family[J] Oncogene. 2017;36(32):4551–4561. doi: 10.1038/onc.2017.89. [DOI] [PMC free article] [PubMed] [Google Scholar] [Retracted]
- 16.Thomson PJ. Oral carcinogenesis[M] In: Thomson PJ, editor. Oral precancer-diagnosis and management of potentially malignant disorders. Chichester: Wiley Blackwell; 2012. pp. 31–47. [Google Scholar]
- 17.Hansen TB, Jensen TI, Clausen BH, et al. Natural RNA circles function as efficient microRNA sponges[J] Nature. 2013;495(7441):384–388. doi: 10.1038/nature11993. [DOI] [PubMed] [Google Scholar]
- 18.Kefas B, Godlewski J, Comeau L, et al. microRNA-7 inhibits the epidermal growth factor receptor and the Akt pathway and is down-regulated in glioblastoma[J] Cancer Res. 2008;68(10):3566–3572. doi: 10.1158/0008-5472.CAN-07-6639. [DOI] [PubMed] [Google Scholar]
- 19.Du WW, Fang L, Yang WN, et al. Induction of tumor apoptosis through a circular RNA enhancing Foxo3 activity[J] Cell Death Differ. 2017;24(2):357–370. doi: 10.1038/cdd.2016.133. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Wang YF, Li BW, Sun S, et al. Circular RNA expression in oral squamous cell carcinoma[J] Front Oncol. 2018;8:398. doi: 10.3389/fonc.2018.00398. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.Campbell KJ, Tait SW. Targeting BCL-2 regulated apoptosis in cancer[J] Open Biol. 2018;8(5):180002. doi: 10.1098/rsob.180002. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22.Liu ZQ, Ding Y, Ye N, et al. Direct activation of bax protein for cancer therapy[J] Med Res Rev. 2016;36(2):313–341. doi: 10.1002/med.21379. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.Jacob A, Jing J, Lee J, et al. Rab40b regulates trafficking of MMP2 and MMP9 during invadopodia formation and invasion of breast cancer cells[J] J Cell Sci. 2013;126(20):4647–4658. doi: 10.1242/jcs.126573. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.Ramos-García P, González-Moles MÁ, González-Ruiz L, et al. Prognostic and clinicopathological significance of cyclin D1 expression in oral squamous cell carcinoma: a systematic review and meta-analysis[J] Oral Oncol. 2018;83:96–106. doi: 10.1016/j.oraloncology.2018.06.007. [DOI] [PubMed] [Google Scholar]