Abstract
Im Dezember 2019 ist erstmalig beim Menschen ein neues Coronavirus in Wuhan, China, aufgetreten, das als SARS-CoV‑2 bezeichnet wird. Die klinischen Verläufe der Erkrankung, die als „coronavirus disease 2019“ (COVID-19) bezeichnet wird, reichen von milden respiratorischen Symptomen bis hin zum schweren Lungenversagen. Das Virus breitet sich aktuell weltweit rasant aus und bringt die Gesundheitssysteme aufgrund des exponentiellen Anstiegs der Fallzahlen an die Grenzen ihrer Kapazitäten. Ursprünglich stammt SARS-CoV aus dem Pool der Fledermaus-Coronaviren und ist im Rahmen eines Speziessprungs in die menschliche Population geraten. Molekulardiagnostische Nachweisverfahren wurden in kürzester Zeit etabliert, und unterschiedliche Studien zum Einsatz verschiedener antiviraler Medikamente werden durchgeführt. Auch werden verschiedene Impfstoffansätze erforscht.
Angesichts der Fallzahlen und einer Sterblichkeit bis zu 9 % ist dringender Handlungsbedarf gegeben. Im vorliegenden Beitrag wird der aktuelle Wissenstand zu Coronaviren zusammengefasst, mit einem Schwerpunkt auf den aktuellen Daten zu SARS-CoV‑2. Aufgrund des sich täglich ändernden Wissensstands reflektiert die Arbeit den Stand bis zum 21.03.2020.
Schlüsselwörter: SARS-Coronavirus-2, COVID-19, Epidemiologie, Antivirale Medikamente, Virale Impfstoffe
Abstract
In December 2019 a new human coronavirus emerged in Wuhan, China, which is known as SARS-CoV‑2. The clinical course of the disease known as coronavirus disease 2019 (COVID-19) ranges from mild respiratory symptoms to severe lung failure. The virus is currently rapidly spreading around the world and pushing health systems to the limits of their capacity due to the exponential increase in the number of cases. The origin of SARS-CoV‑2 lies in the bat coronavirus pool and has now emerged in the human population due to interspecies transmission. Molecular diagnostic methods have been established in a very short time and a number of clinical studies on the effectiveness of different antiviral drugs are ongoing. The development of a vaccine using different approaches is also under investigation.
Considering the high number of cases and mortality rates of up to 9% there is an urgent need for action. This article summarizes the current state of knowledge on human coronaviruses with a strong focus on the current data on SARS-CoV‑2. Due to the daily changing level of knowledge, the article reflects the status up to 21 March 2020.
Keywords: SARS-Coronavirus-2, COVID-19, Epidemiology, Antiviral drugs, Viral vaccines
Im Dezember 2019 ist erstmalig ein neues Coronavirus in Wuhan, China, aufgetreten, das mittlerweile als SARS-CoV‑2 bezeichnet wird. Noch nie wurde ein Erreger so schnell identifiziert und molekularbiologisch analysiert. Die wissenschaftliche Kommunikation erfolgt global, in einem gut funktionierenden Netzwerk, an dem auch Fachzeitschriften via kostenfreie Publikationen und über Onlineportale einen Anteil haben. Im ersten Teil des vorliegenden Beitrags werden die wichtigsten Aspekte zu den klassischen humanen Coronaviren (HCoV) dargestellt und im zweiten Teil mit Stand vom 21.03.2020 die aktuellen Daten zu SARS-CoV‑2 zusammengefasst und bewertet.
Humane Coronaviren
Klinischer Verlauf und Epidemiologie
Humane Coronaviren sind häufige Vertreter der Familie Coronaviridae (Subfamilie Orthocoronavirinae) und verursachen hauptsächlich respiratorische Infektionen („respiratory tract infections“, RTI) unterschiedlicher Schweregrade. Die klassischen Vertreter der humanen Coronaviren sind: HCoV-229E, HCoV-NL63, HCoV-OC43 und HCoV-HKU1. Sie sind für ein Drittel aller Erkältungserkrankungen weltweit verantwortlich. In den meisten Fällen sind Infektionen mit diesen HCoV unkompliziert, es kann aber auch zu schweren Verläufen mit lebensbedrohlicher Pneumonie und Bronchiolitis kommen. Letztere treten v. a. bei immunsupprimierten Patienten und im Senium, aber auch bei Kindern auf. Im Kindesalter sind 9 % aller RTI durch HCoV verursacht. Die Hospitalisierungsraten von HCoV-infizierten Kindern mit Infektionen des unteren Respirationstrakts betragen 1,5 (<5 Jahre) bis 2,8 (<1 Jahr) pro 1000 [1]. Neben der bekannten respiratorischen Symptomatik können neuronale Gewebe und Zellen des Gastrointestinaltrakts infiziert werden, sodass auch mit neurologischen Symptomen und gastrointestinalen Beschwerden zu rechnen ist.
Im Jahr 2002 trat in der Provinz Guangdong (China) erstmalig ein neues Coronavirus in der menschlichen Population auf, das SARS-CoV (SARS: „severe acute respiratory syndrome“). Die Infektion verlief influenzaartig mit Fieber, Kopfschmerzen, Husten und Myalgien. Im Weiteren kam es aber auch zu Pneumonien, die zum lebensbedrohlichen Lungenversagen führten. Beim Ausbruch des SARS-CoV im Zeitraum von 2002–2004 wurden mehr als 8000 Fälle in 29 Ländern registriert. Die Mortalität betrug ca. 10 %. Molekularbiologische Untersuchungen der Genomsequenzen zeigten, dass SARS-CoV aus dem Pool der Fledermaus-Coronaviren stammt und über andere Säuger (z. B. Zibetkatze) als Zwischenwirt auf den Menschen übertragen wurde. Es handelte sich also um ein zoonotisches Virus, das neu in die menschliche Population eingebracht worden war. Die Epidemie konnte gut eingedämmt werden, da die Übertragung von Mensch zu Mensch erst mit Erkrankungsbeginn erfolgte, sodass schon einfaches Temperaturmessen und definierte Kontrollmaßnahmen den Ausbruch stoppen konnten.
Im Jahr 2012 traten klinisch ähnliche Fälle in Saudi-Arabien auf, die ebenfalls mit schwerem Lungenversagen assoziiert waren. Hier zeigte sich, im Gegensatz zu SARS-CoV, das auch über „Superspreader“-Ereignisse übertragen wurde und sich dadurch schnell weltweit verbreitete, ein mehr geografisch limitiertes Auftreten der Infektionen. Ursache dieses Ausbruchs war ein SARS-verwandter Erreger, das MERS-CoV (MERS: „Middle East respiratory syndrome“). Bis heute sind weltweit 2243 Fälle gemeldet worden, 85 % traten in Saudi-Arabien und den Vereinigten Arabischen Emiraten auf. Die restlichen Fälle verteilen sich weltweit auf 25 Länder und sind alle aus dem Nahen Osten importiert worden. Im Gegensatz zu SARS-CoV ist die Mortalität von MERS-CoV mit 35 % jedoch deutlich höher. Auch MERS-CoV stammt aus dem Pool der Fledermaus-Coronaviren und wird über Zwischenwirte, z. B. Dromedare, auf den Menschen übertragen.
Das Ende 2019 in Wuhan, China, erstmals aufgetretene SARS-CoV‑2, das die Erkrankung COVID-19 verursacht, stammt ebenfalls aus dem Pool der Fledermaus-Coronaviren und ist wahrscheinlich erst vor Kurzem entstanden [2].
Molekularbiologie und Pathogenese
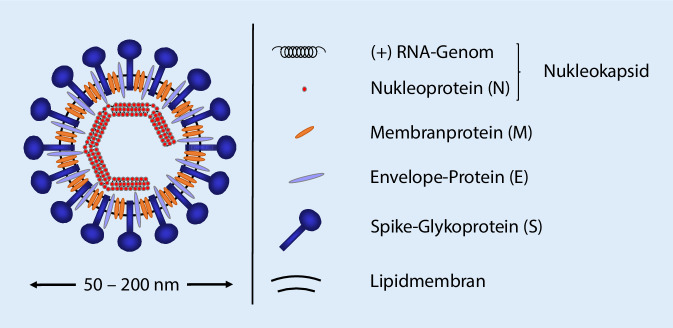
Coronaviren aus der Subfamilie Orthocoronavirinae werden in die Genera α‑, β‑, γ‑ und δ‑Coronaviren eingeteilt und sind im Tierreich weit verbreitet. Es sind Viren mit einem helikalen Nukleokapsid (N), das von einer Lipidmembran umhüllt ist, in der die Strukturproteine E, S und M eingelagert sind (Abb. 1). Das S‑Protein ist für die Bindung an zelluläre Rezeptoren und den nachfolgenden Eintritt in die Zielzellen verantwortlich. Dieser kann nur erfolgen, wenn das S‑Protein durch zelluläre Proteasen funktional gespalten wird. Aufgrund seiner Funktionen ist diese Protein das Ziel für neutralisierende Antikörper.
Die sich schnell entwickelnden Coronavirusvarianten stellen die Basis für einen Speziessprung dar
Coronaviren sind hochvariable RNA-Viren, die mithilfe des Nukleotidaustausches und der Rekombination sehr schnell neue Varianten entwickeln können. Diese stellen die Basis für einen Speziessprung dar. Das (+)-Strang-RNA-Genom der Coronaviren ist zwischen 26 und 32 Kilobasen groß. Zwei Drittel des Genoms bestehen aus 2 überlappenden Leserastern, die den komplexen viralen Replikase-Transkriptase-Enzymkomplex codieren und nach Zelleintritt des Virus direkt vom viralen Genom als Vorläuferpolyprotein translatiert wird. Nach autokatalytischer Spaltung entstehen die Nichtstrukturproteinuntereinheiten, die den funktionsfähigen Transkriptase-Polymerase-Komplex bilden. Seine Aufgaben sind die Transkription viraler subgenomischer „messenger (m)RNA“ und die Replikation der viralen genomischen RNA. Das letzte Drittel des Genoms codiert für die Virusstrukturproteine S, E, M und N, die jeweils mithilfe der entsprechenden subgenomischen mRNA translatiert werden.
Die HCoV infizieren Zellen über unterschiedliche Rezeptoren (Tab. 1). Ziele sind Zellen des oberen und/oder unteren Respirationstrakts, aber auch intestinale Mukosazellen, die Tubuluszellen der Nieren und neuronale Zellen. In der Folge werden HCoV durch respiratorische Sekrete (Tröpfcheninfektion), aber z. T. auch durch Schmierinfektion (Stuhl und Urin) übertragen. Immunzellen, wie dendritische Zellen, Makrophagen und T‑Zellen, gehören ebenfalls zu den Zielzellen von HCoV. Infizierte Immunzellen dienen als Vehikel für die Verbreitung im Organismus, und ihr viral bedingter Verlust wirkt der Aktivität des Immunsystems entgegen. Zur Infektionsstrategie der HCoV gehört die Modulation der angeborenen Immunität, wie die Inhibition des Typ-1-Interferon-Systems [3], der Eingriff in zelluläre Apoptosemechanismen und die Inhibition der Antigenpräsentationsfunktion dendritischer Zellen [4].
| Orthocoronavirinae | Stamm | Entdeckung | Zellrezeptor | Tierischer Wirt |
|---|---|---|---|---|
| α‑Coronaviren | HCoV-229E | 1966 | CD13 | Fledermaus |
| HCoV-NL63 | 2004 | ACE2 | Fledermaus, Zibetkatze | |
| β‑Coronaviren | HCoV-OC43 | 1967 | 9‑O-acetylierte Sialinsäure | Rinder |
| HCoV-HKU1 | 2005 | 9‑O-acetylierte Sialinsäure | Maus | |
| SARS-CoV | 2003 | ACE2 | Fledermaus, Zibetkatze | |
| MERS-CoV | 2012 | DPP4 | Fledermaus, Dromedar | |
| SARS-CoV‑2 | 2019 | ACE2 | Fledermaus, Schuppentiere? |
ACE2 „angiotensin-converting enzyme 2“, CD13 Aminopeptidase N, DPP4 Dipeptidylpeptidase‑4
Diagnostik und Therapie
Die Diagnostik respiratorischer Infektionen erfolgt heute mithilfe der Echtzeit-Polymerase-Kettenreaktion („real-time polymerase chain reaction“, RT-PCR) im Rahmen einer Panel-Diagnostik, die verschiedene Erreger simultan nachweisen kann. Dieses Verfahren erlaubt den zeitnahen, sehr sensitiven und hochspezifischen Nachweis viraler und bakterieller Genome und ist den Antigennachweisverfahren hinsichtlich Sensitivität und Spezifität deutlich überlegen. Infektionen mit HCoV werden weitgehend symptomatisch therapiert. Chloroquin zeigt im Mausmodell bei Infektionen des Zentralnervensystems (ZNS) einige Erfolge [5]. Ein klinischer Nutzen konnte beim Einsatz der eigentlich für die Behandlung von Humane-Immundefizienz(HIV)-Infektionen verwendeten Proteaseinhibitoren Lopinavir und Ritonavir (Kaletra®) zur Therapie des SARS-CoV nachgewiesen werden [6].
Neu aufgetretenes Coronavirus aus Wuhan – SARS-CoV-2
Im Dezember 2019 trat in China erstmalig ein neues Coronavirus auf, das zunächst als 2019-nCoV bezeichnet wurde und nach aktueller Nomenklatur des International Committee on Taxonomy of Viruses (ICTV) nun als SARS-CoV‑2 bezeichnet wird [7]. Die Erkrankung, die das Virus auslöst, erhielt von der World Health Organization (WHO) den Namen „coronavirus disease 2019“ (COVID-19). Betroffen war die Provinz Hubei und hier v. a. die 15 Mio. Einwohner zählende Region der Provinzhauptstadt Wuhan. Bis heute ist das Virus bereits in zahlreiche Länder der Erde importiert worden. Infolgedessen hat die WHO den internationalen Gesundheitsnotstand („public health emergency of international concern“) ausgerufen. Wissenschaftler arbeiten weltweit mit Hochdruck an den zentralen biologischen, medizinischen und epidemiologischen Fragestellungen zu COVID-19 und dem Erreger SARS-CoV‑2; daher ändert sich die Datenlage täglich. Dieser Beitrag erfasst den Datenstand bis zum Zeitpunkt Mitte März 2020.
Ursprung des Virus
Das SARS-CoV‑2 ist das neueste der Coronaviren, die in den letzten 20 Jahren als Zoonoseerreger in die menschliche Population gelangt sind. Im Jahr 2002 trat in China erstmalig das SARS-CoV beim Menschen auf, und im Jahr 2012 erfolgte das erstmalige Auftreten von MERS-CoV in Saudi-Arabien. Aktuelle genetische Vergleichsuntersuchungen geben Hinweise darauf, dass das SARS-CoV‑2 erst vor Kurzem entstanden sein könnte. Wie auch die verwandten Viren SARS-CoV und MERS-CoV stammt SARS-CoV‑2 aus dem Pool der SARS-artigen Coronaviren. Diese zirkulieren in Rhinolophus-Fledermäusen [8]. Die nächsten verwandten Viren von SARS-CoV‑2 in Fledermäusen sind bat-SL-CoVZXC-21 und bat-SLCoVVZC45. Es ist wahrscheinlich, dass die Übertragung von SARS-CoV‑2 von der Fledermaus auf den Menschen durch einen noch unbekannten Zwischenwirt erfolgte. Als Ursprungsort der Epidemie wird der Huanan-Markt in Wuhan vermutet, auf dem, bis zur Schließung durch die lokalen Behörden, zahlreiche lebende Wildtiere gehandelt wurden, sodass eine Interspeziesübertragung durch den engen Kontakt zwischen Mensch und Tier möglich gewesen sein könnte. Inwieweit der Markt in Wuhan jedoch als Ursprungsort der Epidemie angesehen werden kann, ist noch unklar. Es ist genauso möglich, dass die Übertragung auf den Menschen andernorts und evtl. schon früher erfolgte, und der Markt nur als Verstärker und Verteiler diente. Weitere molekularbiologische Untersuchungen werden dies zukünftig aufklären.
Schon im März 2019 wurde die These vertreten, dass es aufgrund der ähnlichen biologischen Eigenschaften von fledermausstämmigen β‑Coronaviren wie SARS-CoV und MERS-CoV sehr wahrscheinlich ist, dass aus diesem Pool weitere Übertragungsereignisse in die menschliche Population erfolgen werden. Gründe sind die hohe Bevölkerungsdichte in China, die hohe Biodiversität im Land, einschließlich der Biodiversität der Fledermäuse und der fledermausstämmigen Coronaviren. Aber auch kulturelle Ernährungs- und Schlachtgewohnheiten tragen dazu bei, dass China ein Hotspot für das Auftreten und Wiederauftreten von zoonotischen Coronaviren in der menschlichen Population ist [9]. Gerade vor diesem Hintergrund ist es sehr bedauerlich, dass die ersten Hinweise chinesischer Ärzte auf das Auftreten eines neuen SARS-artigen Ausbruchs in China von den dort zuständigen lokalen Behörden nicht nur ignoriert, sondern aktiv unterbunden wurden und somit wertvolle Zeit zur Eindämmung des Ausbruchs verloren ging. Dennoch erfolgte die Reaktion der chinesischen Regierung deutlich besser und professioneller als noch beim SARS-Ausbruch 2002.
Epidemiologie, Übertragung und Krankheitsverlauf
Mit Stand vom 21.03.2020 waren weltweit 303.180 bestätigte Fälle der SARS-CoV-2-induzierten Atemwegserkrankung COVID-19 aufgetreten. Bisher sind 12.950 Patienten verstorben. Die Mehrzahl der Fälle sind in China in der Stadt Wuhan und in der Provinz Hubei zu verzeichnen (81.304 Fälle, 3259 Tote, Sterblichkeit 4,0 %). Die Infektion hat sich inzwischen pandemisch auf 188 Länder ausgebreitet. Neben China verzeichnet der Iran die meisten Fälle in Asien (20.610 Fälle, 1556 Tote, Sterblichkeit 7,5 %), gefolgt von Korea (8799 Fälle, 102 Tote, Sterblichkeit 1,2 %). In Europa ist Italien am schwersten betroffen (53.578 Fälle, 4825 Tote, Sterblichkeit 9,0 %), gefolgt von Spanien (25.374 Fälle, 1375 Tote, Sterblichkeit 5,4 %). In Deutschland sind Ende Januar 2020 erstmals Fälle in Bayern aufgetreten. Diese standen im Zusammenhang mit der Schulung bei einem Unternehmen und wurden durch einen chinesischen Gast übertragen. Die Person aus China war während der Schulung wohl noch nicht klinisch respiratorisch erkrankt, hatte aber 4 Mitarbeiter angesteckt. Hier zeigte sich erstmalig, dass das Virus wahrscheinlich schon vor dem Auftreten klinischer respiratorischer Symptome übertragen werden kann [10]. Im Rahmen dieses lokalen Ausbruchs in Bayern wurde das Virus erstmals in Europa von Mensch zu Mensch übertragen. Ende Februar wurden Fälle in Baden-Württemberg und in Nordrhein-Westfalen (NRW) gemeldet, die von einem Ausbruch in Italien importiert wurden. Am 21.03.2020 gab es in Deutschland 22.213 bestätigte Fälle und 84 Todesfälle (Sterblichkeit 0,4 %). Deutschland verzeichnete zu diesem Zeitpunkt somit die dritthöchsten Fallzahlen in Europa.
Bei Erwachsenen sind Vorerkrankungen eindeutige Risikofaktoren bezüglich erhöhter Sterblichkeit
Die Sterblichkeitsrate beträgt weltweit laut WHO durchschnittlich 3,5 % und hängt von unterschiedlichen Faktoren ab. Auf der Basis der Daten des Chinese Center for Disease Control and Prevention (CCDC) haben alte Menschen das höchste Risiko zu versterben (Sterblichkeit >80 Jahre 14,8 %). Auch ist das Sterberisiko bei Männern höher als bei Frauen (2,8 % vs. 1,7 %). Laut einer aktuellen retrospektiven Studie sind in China in der Altersgruppe der 0‑ bis 10-Jährigen 1404 Fälle ohne Todesfall gemeldet worden. In der Altersgruppe 10 bis 19 Jahre sind 748 Fälle und nur ein Todesfall aufgetreten [11]. Somit ist das Sterberisiko für Kinder und Jugendliche insgesamt sehr gering. Auch verlaufen die Erkrankungen bei Kindern und Jugendlichen insgesamt weniger schwer. Bei 94,1 % der Kinder wurde ein asymptomatischer, milder oder moderater Verlauf beobachtet, wobei ca. die Hälfte dieser Gruppe den milden Krankheitsverlauf aufwies. Die Gründe für den insgesamt milderen Verlauf von COVID-19 bei Kindern sind unklar. Daten aus China weisen darauf hin, dass sich Kinder nach Kontakt mit Infizierten weniger leicht anstecken als Erwachsene [12]. Weiterhin kann die höhere Expression des „angiotensin-converting enzyme 2“ (ACE2) bei Kindern im Vergleich zu älteren Personen eine Rolle spielen. Zwar scheint dies zunächst für eine leichtere Infizierbarkeit von Kindern zu sprechen, allerdings spielt ACE2 auch eine wichtige Rolle bei der Immunmodulation, und hohe ACE2-Spiegel schützen vor akuten Lungenschädigungen, infolge von Sepsis und Influenzainfektionen [13]. Dennoch wurden selbst bei Kindern schwere und kritische Verlaufsformen beobachtet. Hierbei fiel auf, dass der Anteil an schweren bzw. kritischen Verlaufsformen bei Kindern im Alter unter einem Jahr mit 10,6 % am höchsten war und mit zunehmendem Alter bis auf 3 % abnahm (>16 Jahre; [11]). Als Risikopatienten sind Säuglinge, die jünger als 3 Monate sind, anzusehen, die entweder eine Vorerkrankung haben (angeborene Herzkrankheit, bronchopulmonale Dysplasie, Fehlbildung der Atemwege, Hämoglobinopathien, schwere Unterernährung usw.) oder immungeschwächt bzw. immunsupprimiert sind [14]. Bei Erwachsenen sind Vorerkrankungen eindeutige Risikofaktoren bezüglich erhöhter Sterblichkeit. Für Patienten mit kardiovaskulären Erkrankungen beträgt die Sterblichkeitsrate 10,5 %, gefolgt von 7,3 % bei Diabetes, 6,3 % bei chronischen Lungenerkrankungen, 6,0 % bei Hypertonie und 5,6 % bei Krebserkrankungen.
Auffällig sind die großen regionalen Unterschiede hinsichtlich der Sterberate. Während z. B. in Deutschland derzeit die Sterberate 0,4 % beträgt (allerdings mit steigender Tendenz), ist die Sterberate in China und Italien mit 4,0 % bzw. 9,0 % bedeutend höher. Die Gründe sind unklar. Zum einen besteht die Möglichkeit, dass in Regionen mit hoher Sterberate nur ein Bruchteil der Infektionen mit SARS-CoV‑2 tatsächlich diagnostiziert wird, insbesondere dann, wenn diese mild oder sogar subklinisch verlaufen. Dass dies eine wesentlicher Grund sein könnte, legen Modellrechnungen nahe, die zu dem Schluss kommen, dass die Dunkelziffer SARS-CoV-2-Infizierter sehr hoch ist [15, 16]. Eine weitere Möglichkeit besteht darin, dass in Regionen mit unterschiedlicher Sterberate SARS-CoV-2-Stämme mit unterschiedlicher Pathogenität zirkulieren. Es gibt erste Hinweise, dass zumindest 2 unterschiedliche SARS-CoV-2-Linien für die bisherigen Humaninfektionen verantwortlich sind [17], wobei derzeit noch nicht eindeutig geklärt ist, ob diese beiden Viruslinien eine unterschiedliche Pathogenität aufweisen.
Modellrechnungen legen eine sehr hohe Dunkelziffer SARS-CoV-2-Infizierter nahe
Des Weiteren konnte gezeigt werden, dass die Bevölkerung in Ostasien, einschließlich China, eine höhere ACE2-Allelfrequenz in den „Expression-quantitative-trait-locus“(eQTL)-Varianten (Variation der mRNA-Expressionslevel) aufweist, die mit einer höheren Expression der Virusrezeptor-ACE2-Varianten im Gewebe assoziiert sind. Dies könnte eine erhöhte SARS-CoV-2-Suszeptibilität bei bestimmten Bevölkerungsgruppen erklären [18]. Für Italien wird vermutet, dass der hohe Altersdurchschnitt der Bevölkerung, verbunden mit der starken Luftverschmutzung, in zahlreichen italienischen Städten zur erhöhten Sterblichkeit beiträgt.
Trotz der epidemiologischen Daten, die vom CCDC erhoben wurden, sind zentrale Fragen offen. Dies betrifft die Rolle möglicher Tierreservoire bei der Verbreitung von COVID-19 und die Frage, wie lange die Patienten vor und nach dem Erkrankungsbeginn infektiös sind. Tong et al. konnten zeigen, dass das Virus mit hoher Wahrscheinlichkeit schon vor dem Ausbruch klinischer Symptome übertragen wird [19]. Untersuchungen am Institut für Mikrobiologie der Bundeswehr haben ergeben, dass das Virus mindestens bis zum Tag 4 nach Krankheitsbeginn in der Zellkultur anzüchtbar (PD Dr. Institut für Mikrobiologie der Bundeswehr Dobler, persönliche Mitteilung) und damit der Patient als infektiös anzusehen ist. Weitere Details zur Übertragungskinetik stehen noch aus. Im Gegensatz zu SARS-CoV, das vorrangig in den tiefen Atemwegen repliziert, vermehrt sich SARS-CoV‑2 effizient im oberen Respirationstrakt und wird von dort über Tröpfchen ausgeschieden. Infektionsepidemiologisch ähnelt der Ausbruch damit mehr einer Influenza als dem SARS-CoV-Ausbruch im Jahr 2002. Es ist wahrscheinlich, dass es eine Reihe von symptomfreien oder gering symptomatischen Patienten gibt, die das Virus effektiv übertragen. Wahrscheinlich müssten deutlich mehr als 60 % der Übertragungen verhindert werden, um den Ausbruch zu stoppen (N. Ferguson, Imperial College, London; pers. Mitteilung). Es ist deshalb bei allen respiratorischen Infektionen an COVID-19 zu denken, und diese wären mithilfe der Molekulardiagnostik (s. Abschn. „Diagnostik“) umgehend sicher auszuschließen oder zu bestätigen. Nur so kann das Ausbreitungsgeschehen wirklich erfasst werden.
Der klinische Verlauf von COVID-19 reicht von leichten respiratorischen Symptomen bis hin zum schweren Lungenversagen und einer Extrakorporale-Membranoxygenierung(ECMO)-pflichtigen Therapie. Für Kinder werden 5 Verlaufsformen definiert [11]: (i) asymptomatisch, (ii) mild, mit Symptomen einer Infektion des oberen Rachentrakts, (iii) moderat mit Fieber, trockenem Husten, Pneumonie und im CT sichtbaren Lungenläsionen, (iv) schwer mit Auftreten von Dyspnoe, einschließlich zentraler Zyanose sowie einer Sauerstoffsättigung weniger als 92 % ca. eine Woche nach Krankheitsbeginn sowie (v) kritisch mit akutem Lungenversagen („acute respiratory distress syndrome“, ARDS).
Gemäß einer von Guan et al. Ende Februar 2020 publizierten Studie beträgt die mittlere Inkubationszeit 4 Tage (Intervall von 2 bis 7 Tagen). Das Durchschnittsalter der Infizierten wird in dieser Studie in China mit 47 Jahren angegeben, wobei 84 % der Patienten der Altersgruppe 15 bis 64 Jahre angehören, 15,1 % der Patienten älter als 65 Jahre und 0,9 % der Patienten jünger als 15 Jahre (15 bis 64 Jahre) sind [20]. Die Symptome dauern in milden Fällen 7 bis 14 Tage, bei schweren Verläufen aber 3 bis 6 Wochen [21]. Angesichts der schnellen Verbreitung von COVID-19 verlaufen glücklicherweise 81 % aller Fälle mit geringer Symptomatik [22]; die durchschnittliche weltweite Sterberate beträgt laut WHO 3,5 %.
Die durchschnittliche weltweite Sterberate beträgt laut WHO 3,5 %
In der Studie von Guan et al. hatten weniger als die Hälfte der Patienten (43,8 %) bei Klinikaufnahme Fieber, jedoch trat Fieber in 88,7 % der Fälle im Verlauf der Erkrankung auf. Das häufigste klinische Symptom war Husten (67,8 %). Selten ist mit einer Diarrhö zu rechnen (3,8 %, [20]). Patienten mit schweren Erkrankungen waren im Mittel 7 Jahre älter als Patienten mit leichten Erkrankungsverläufen. Bei Aufnahme in die Klinik wurde zur Beurteilung der Lungen der Patienten Computertomographien (CT) durchgeführt. Am häufigsten zeigte/zeigten sich eine Milchglastrübung (56,4 %) oder bilaterale fleckige Verschattungen (51,8 %). Nur 2,9 % der Patienten mit schwerer und 17,9 % mit milder Erkrankung wiesen keine CT-Veränderungen auf. In den Laboruntersuchungen fielen Lymphopenie (83,2 %), Thrombopenie (36,2 %) sowie erhöhte Konzentrationen des C‑reaktiven Proteins (CRP) auf. Insgesamt wurden 5 % der Patienten intensivbehandlungspflichtig und 2,3 % mussten beatmet werden. Die Mortalität in dieser Studie betrug 1,4 % [20].
Huang et al. haben folgende Symptome bei stationären Patienten publiziert: Fieber (98 %), Husten (76 %), Myalgien (44 %), Sputumproduktion (28 %), Kopfschmerzen (8 %), Hämoptysen (5 %) und Diarrhö (3 %). Im Verlauf von 8 Tagen entwickelten 55 % der Patienten eine Dyspnoe, und 63 % zeigten im Verlauf eine Lymphopenie. Alle Patienten hatten eine CT-gesicherte Pneumonie [23].
Untersuchungen von Chen et al. konnten zeigen, dass eine Schwangerschaft keinen Einfluss auf den Verlauf der COVID-19-Pneumonie hat. Es ist bisher keine intrauterine Übertragung des Virus auf das Ungeborene in der Spätschwangerschaft beschrieben worden [24]. Aufgrund der noch geringen Fallzahl von 9 Infektionen in der Schwangerschaft können diese Ergebnisse noch nicht als gesichert gelten.
Diagnostik
Die Diagnostik von SARS-CoV, MERS-CoV und SARS-CoV‑2 sowie auch von anderen Coronaviren wird als molekularbiologischer Direktnachweis mithilfe der RT-PCR durchgeführt [25–27]. Als Material sind ein Nasen- und Rachenabstrich sowie ein Sekret der tiefen Atemwege (Sputum, Probe einer bronchoalveolären Lavage [BAL] oder Trachealsekret) zu gewinnen. Hierzu sind spezielle Abstrichtupfer zu verwenden, die entweder trocken oder besser mit einem Virustransportmedium (evtl. auch 500 μl NaCl-Lösung) getränkt sind. Gelhaltige Abstrichtupfer sind für die PCR-Diagnostik unbrauchbar.
Der Ausschluss falsch-negativer Ergebnisse erfordert den PCR-Nachweis von 2 unteschiedlichen viralen Genabschnitten
Verschiedene PCR-Testsysteme sind weltweit im Einsatz. Zurzeit wird in Deutschland v. a. die PCR nach Corman et al. durchgeführt [28]. Die Autoren des vorliegenden Beitrags haben ebenfalls gute Erfahrungen mit der vom amerikanischen CDC publizierten RT-PCR gemacht [29]. Momentan wird an einem molekularen Schnelltest auf der Basis der isothermen Rekombinase-Polymerase-Amplifikation gearbeitet, der einen Vor-Ort-Nachweis in ca. 30 min erlaubt. Die aktuellen Testverfahren weisen unterschiedliche Genabschnitte des Virus nach. Aufgrund der hohen Mutationsraten von RNA-Viren sollte, zum sicheren Ausschluss von falsch-negativen Ergebnissen, die PCR-Diagnostik von zwei Genabschnitten durchgeführt werden.
Zu beachten ist auch, dass mit diesem Testverfahren kein infektiöses Virus nachgewiesen wird, da virale Gene oftmals länger nachgewiesen werden können, als tatsächlich Ansteckungsgefahr besteht; ein Beispiel hierfür ist die Infektion mit Noroviren. In diesem Zusammenhang ist die Tatsache erwähnenswert, dass bei mit SARS-CoV‑2 infizierten Kindern Virusmaterial noch im Stuhl nachgewiesen werden konnte, während zum selben Zeitpunkt erfolgte Rachenabstriche bereits negativ waren. Auch hier allerdings ist unklar, ob es sich um ein infektiöses Virus handelt [12]. Zum Nachweis der Infektiosität muss eine Virusisolierung in der Zellkultur durchgeführt werden. Erste Untersuchungen am Institut der Bundeswehr in München haben gezeigt, dass das Virus mindestens 4 Tage nach Erkrankungsbeginn noch isolierbar (PD Dr. Dobler, Institut für Mikrobiologie der Bundeswehr; persönliche Mitteilung) und damit der Patient infektiös ist. Daten zur Infektionsdosis von SARS-CoV‑2 in Bezug auf die Genomzahlkopien liegen noch nicht vor.
Zum Nachweis der Infektiosität muss eine Virusisolierung in Zellkultur durchgeführt werden
Differenzialdiagnostisch muss die milde Verlaufsform von COVID-19 von anderen viralen Infektionen des oberen Rachentrakts abgegrenzt werden. Die COVID-19-Pneumonie muss von einer Pneumonie durch Influenza‑, Adeno‑, Humanem Respiratorischen Synzytialvirus und anderen viral verursachten Pneumonien sowie von einer durch Mycoplasma pneumoniae verursachten Pneumonie unterschieden werden. Bei Verdachtsfällen sind schnellstmöglich ein „Point-of-care“-Test (POCT) und eine PCR-Diagnostik auf Krankheitserreger der Atemwege durchzuführen [14].
Therapie
Medikamente
Für die antivirale Therapie von COVID-19 stehen bislang weder zugelassene Medikamente noch Impfstoffe zur Verfügung. In begrenztem Umfang bestehen jedoch Erfahrungen bei der Therapie der SARS-CoV-Infektion, die aufgrund der engen Verwandtschaft der Viren herangezogen werden können. So ist der kombinierte Einsatz der Proteaseinhibitoren Lopinavir und Ritonavir bei SARS-CoV-Patienten von klinischem Nutzen [6]. Eine weitere Studie mit MERS-CoV-Infizierten wird in Saudi-Arabien durchgeführt, bei der mit Lopinavir/Ritonavir plus Interferon‑β behandelt wird [30]; Daten zu den Ergebnissen liegen noch nicht vor. Es zeigte sich aber im Mausmodell, dass die Kombination Lopinavir/Ritonavir plus Interferon‑β die Viruslast bei der MERS-CoV-Infektion nur geringgradig senken konnte und wenig Einfluss auf die pathologischen Veränderungen der Lungen hat. Erklären ließe sich dieses Phänomen über die hohe Plasmabindung der Proteaseinhibitoren, sodass die freie Wirkstoffkonzentration für einen antiviralen Effekt gegen Coronaviren evtl. nicht ausreicht. In einer aktuellen Studie zeigte die Behandlung von COVID-19 Patienten mit Lopinavir/Ritonavir keinen therapeutischen Nutzen [31], sodass diese Wirkstoffkombination wohl als Therapiemöglichkeit ausfällt.
Für die COVID-19 Therapie stehen bislang keine zugelassenen Medikamente und Impfstoffe zur Verfügung
Bei der Infektion mit MERS-CoV erzielte der Polymeraseinhibitor Remdesivir in Verbindung mit Interferon‑β in vitro und im Tiermodell eine sehr gute Wirkung. Im Mausmodell konnten eine präventive und eine therapeutische Wirkung nachgewiesen werden. Die Gabe von Remdesivir plus Interferon‑β führte zur deutlichen Reduktion der Viruslast, einer verbesserte Lungenfunktion und verminderter pathologischer Lungenveränderungen [32]. Auch im Affenmodell erzielte Remdesivir sehr gute Erfolge [33]. Hierbei ist zu beachten, dass die frühe Gabe die Wirkung deutlich verbessert. Diese erklärt sich dadurch, dass die Virusreplikation sehr schnell erfolgt und die antivirale Wirkung, wenn der Zell-, und Gewebeschaden bereits gesetzt ist, deutlich geringer ist. Antivirale therapeutische Eingriffe müssen damit so früh wie möglich durchgeführt werden. Remdesivir als RNA-Polymerase-Inhibitor hat auch eine gute Wirkung gegen andere RNA-Viren [34]. Neben Remdesivir besitzt Chloroquin in vitro eine gute antivirale Aktivität gegen Coronaviren, einschließlich SARS-CoV‑2 [5, 34]. Der antivirale Effekt beruht vermutlich auf den folgenden 2 Mechanismen: (1) einer Erhöhung des endosomalen pH-Werts, die zur Störung der Viruszellfusion führt, (2) einer Interferenz mit der Glykosylierung des zellulären Rezeptors ACE2, die mit einer weniger effizienten Bindung von SARS-CoV‑2 einhergeht. Die EC90 von Chloroquin in der Zellkultur, also die Konzentration, bei der die SARS-CoV-2 Replikation zu 90 % gehemmt wird, beträgt 6,9 µM. Dies wäre (wie bei der Therapie der rheumatoiden Arthritis) durch die Chloroquingabe von 3–4 mg/kgKGW und Tag entsprechend der Hydroxychloroquingabe von 6 mg/kgKGW und Tag zu erreichen. Hydroxychloroquin weist zusätzlich eine antiinflammatorische Aktivität auf, die bei der Behandlung von COVID-19 von Vorteil sein könnte [34–36].
Mögliche Wirkstoffe zur medikamentösen Therapie sind Chloroquin bzw. Hydroxychloroquin
Erste klinische Daten aus China legen den therapeutischen Nutzen für Chloroquin nahe [37]. Entsprechend wurde die Behandlung mit Chloroquin in die aktuelle Leitlinie der nationalen chinesischen Gesundheitskommission zum Umgang mit der COVID-19-Epidemie aufgenommen [14]. Allerdings ist keines der angeführten Medikamente aktuell für die COVID-19-Therapie zugelassen. Sollte daher z. B. Chloroquin eingesetzt werden, entspricht dies einer Off-label-Anwendung, und das Einverständnis der Sorgeberechtigten bzw. des Patienten ist einzuholen. Da Remdesivir zurzeit nicht verfügbar ist und sich die Wirksamkeit von Lopinavir/Ritonavir nicht bestätigt hat, bleibt, neben der symptomatischen Therapie, im Wesentlichen die Gabe von Chloroquin bzw. Hydroxychloroquin übrig. Diese scheint von den bisher damit behandelten COVID-19 Patienten gut vertragen zu werden [38]. Schwere Nebenwirkungen wie Makuladegeneration und Kardiotoxizität/Verlängerung des QT-Intervalls, die nach fortgesetzter Einnahme auftreten können, sind aufgrund der kurzen Behandlungsdauer von 7 Tagen weniger wahrscheinlich. Dennoch ist insbesondere bei Patienten mit chronischen Erkrankungen (z. B. mit eingeschränkter Nierenfunktion oder Lebererkrankungen) oder Patienten, die Medikamente erhalten, die zum Auslösen von Arrhythmien beitragen können, Vorsicht geboten [38]. Aus der Behandlung von juveniler rheumatoider Arthritis und systemischem Lupus erythematodes ist bekannt, dass Kinder die zur Behandlung von COVID-19 vorgeschlagenen Chloroquin- und Hydroxychloroquindosen vertragen [39]. Eine Überdosierung ist insbesondere bei Kleinkindern jedoch unbedingt zu vermeiden, da diese tödlich sein kann [40]. Hierbei ist zu beachten, dass bei Übergewicht das Idealgewicht zur Dosierung zugrunde gelegt werden muss. Weitere Behandlungsmöglichkeiten sind laut der aktuellen chinesischen Leitlinie die eher unspezifische alleinige Gabe von Interferon‑α oder in Kombination mit Ribavirin oder die Gabe des Fusionsinhibitors Umifenovir, der in Europa und USA allerdings nicht zugelassen ist.
In einer aktuellen Studie von Hoffmann et al. konnte gezeigt werden, dass die zelluläre Protease TMPRSS2 für die Infektiosität von SARS-CoV‑2 essenziell ist und eine Hemmung dieser Protease mithilfe von Camostat-Mesilat die Vermehrung des Virus u. a. in kultivierten Lungenzellen hemmt [41]. Camostat-Mesilat ist in Japan zur Behandlung von chronischer Pankreatitis und Refluxösophagitis zugelassen und erzielte in Tierexperimenten bereits eine hemmende Wirkung auf die Vermehrung von SARS-CoV [42]. Zudem haben pharmakokinetische Untersuchungen ergeben, dass Camostat-Mesilat nach i.v.-Gabe in größeren Mengen in den Lungen als im Pankreas verfügbar ist [43].
Impfstoff
Mit einem Impfstoff gegen COVID-19 könnte der aktuelle globale Ausbruch eingedämmt werden. Deswegen ist die Impfstoffentwicklung von zentraler Bedeutung. Es ist aber fraglich, ob dieses Ziel unter den gegebenen zeitlichen Umständen und Wahrung aller rechtlichen Zulassungsverfahren in wenigen Monaten erreicht werden kann. Wahrscheinlich kommt der Einsatz eines Impfstoffs für den derzeitigen Ausbruch zu spät. Zur Impfstoffentwicklung werden unterschiedliche Ansätze auf verschiedenen Technologieplattformen von der Coalition for Epidemic Preparedness Innovations (CEPI) gefördert. Hierzu zählen der MERS-DNA-Impfstoffansatz, „Molecular-clamp“-Technologien und die Entwicklung einer mRNA-basierten Vakzine. Alle diese Ansätze benötigen jedoch noch viel Forschungstätigkeiten, und diverse DNA-Impfstoffe waren bisher im Primatenmodell nur wenig erfolgreich.
Ziel eines viralen Impfstoffs sollte es sein, eine nachhaltige Immunantwort zu erzeugen. Die Induktion einer T‑Zell-basierten und einer humoralen virusneutralisierenden Antwort ist von zentraler Bedeutung. Dies wird am besten durch virale Lebendimpfstoffe (z. B. wie Impfstoffe gegen Masern, Mumps und Röteln (MMR) sowie Gelbfieber) erreicht oder als neuere Alternative durch chimäre replikationsfähige Impfviren. Beispiele sind die beiden Entwicklungen eines Impfstoffs gegen das Ebolavirus, die auf dem Vesikulostomatitisvirus (VSV) und dem Pockenimpfstamm modifiziertes Vaccinia-Virus Ankara (MVA) basieren. In beiden Fällen handelt es sich um chimäre Viren, die das Hüllprotein des Ebolavirus tragen, das das Hauptziel der neutralisierenden Immunantwort darstellt. Mithilfe des de VSV-Konstrukts konnten sehr gute Schutzraten erzielt werden [44]. Der Impfstoff Ervebo, der auf diesem chimären VSV basiert, ist deshalb kürzlich von der Food and Drug Administration (FDA) zugelassen worden [45]. Dieses Konzept ist auch mit anderen viralen Hüllproteinen, z. B. bei H5N1-Influenzaviren, erfolgreich getestet worden [46]. Ein S‑Protein-basierter Impfstoff auf VSV-Basis wurde ebenso für MERS-CoV entwickelt und konnte im Primatenmodell neutralisierende Antikörper und eine T‑Zellantwort induzieren [47].
Ein chimäres VSV, das das SARS-CoV-2-Hüllprotein beinhaltet, stellt den besten Impfstoffansatz dar
Eine weitere, sehr erfolgreiche chimäre Vakzine auf der Basis von Adenoviren in Kombination mit dem Pockenimpfvirus MVA wurde ebenfalls zur Bekämpfung von Ebolavirus entwickelt und ist aktuell im Zulassungsverfahren der European Medicines Agency (EMA; [48–50]). Auch zum Schutz vor MERS-CoV-Infektionen wurde eine chimäres MVA-Impfvirus entwickelt, das sich als wirksam in experimentell infizierten Dromedaren erwies [51] und damit als Basis für einen chimären MVA-SARS-CoV-2-Impfstoffansatz dienen könnte. Es ist evident, dass es mit den chimären Vakzineansätzen zeitnah möglich ist, einen Impfstoff gegen SARS-CoV‑2 zu entwickeln. Auf der Basis von Machbarkeit und den Voruntersuchungen bis hin zum Primatenmodell stellt eine Vakzine auf Basis eines chimären VSV, das das SARS-CoV-2-Hüllprotein beinhaltet, wohl den besten Ansatz dar. Dessen Herstellung würde wahrscheinlich die geringste Zeit benötigen und hat vermutlich die besten Erfolgsaussichten.
Risikomanagement – Lagebewertung und Lösungsansätze
Nach anfänglichem Zögern sind in Deutschland und anderen europäischen Ländern weitgehende Reise- und Mobilitätsbeschränkungen angeordnet worden. Diese Maßnahmen sind in einer globalisierten Gesellschaft zwar unpopulär, seuchenhygienisch aber geboten und effizient, wie es die Maßnahmen und Erfahrungen während der Ebola-Epidemien in Westafrika 2015 zeigten [52]. Zudem hat eine Studie anhand von Modellrechnungen nachgewiesen, dass nur die Kombination aus Reise- und Mobilitätsbeschränkungen sowie Quarantänemaßnahmen die Ausbreitung von SARS-CoV‑2 wirkungsvoll eindämmen kann [16]. Dementsprechend wurden in Deutschland öffentliche Veranstaltungen jeglicher Art abgesagt, die Bildungseinrichtungen zeitweise geschlossen, und die persönliche Mobilität der Bewohner ist eingeschränkt.
Bis zur Anordnung dieser Beschränkungen hat die Zahl der Infektionsquellen in Deutschland jedoch bereits sprunghaft zugenommen, wodurch neue Infektionsherde entstanden sind, von denen sich die Coronavirusinfektion epidemisch in der Bevölkerung ausgebreitet hat. Verstärkt wird die Ausbreitung dadurch, dass ein Teil der Infektionen mild und wahrscheinlich auch symptomarm verläuft. Da das Virus vermutlich schon vor oder spätestens beim Beginn der respiratorischen Symptomatik über respiratorische Sekrete ausgeschieden wird, übertragen infektiöse Personen den Erreger längere Zeit unerkannt. Dies spiegelt sich im sehr raschen Anstieg der Fallzahlen in ganz Deutschland seit Mitte März wider. Hinzu kommt, dass sich das Virus in einer hoch empfänglichen immunnaiven Population verbreitet, in der keine ausreichende Herdenimmunität durch früher durchgemachte Infektionen mit einem nahe verwandten Coronavirus besteht. Deswegen gilt es, umgehend alle Personen mit akuter respiratorischer Symptomatik, deren Anamnese auf eine wie auch immer erfolgte mögliche Exposition schließen lässt, auf SARS-CoV‑2 zu testen. So kann ein Bild des Infektionsgeschehens und damit Klarheit darüber erhalten werden, wie lange die ergriffenen Maßnahmen fortgeführt oder sogar verschärft werden müssen. Essenziell ist in diesem Zusammenhang ist die Stärkung des Risikobewusstseins („public awareness“) in der Bevölkerung, da dieses die Reduktion von Kontakten zwischen den Menschen fördert. „Social distancing“ war ein wichtiger Faktor zur Begrenzung der SARS-Epidemie im Jahr 2002 in Hongkong [53]. Hierzu zählt auch das Tragen von Schutzmasken in der Öffentlichkeit, da dies das Risiko einer Übertragung des Erregers minimiert [54].
Neben diesen Notfallmaßnahmen müssen die heutigen wissenschaftlichen und technischen Möglichkeiten dazu genutzt werden, möglichst schnell einen Impfstoff und/oder eine antivirale Therapie zur wirksamen Bekämpfung von COVID-19 zu entwickeln, um in Zukunft die Ausbreitung von SARS-CoV‑2 wirksam bekämpfen zu können.
Das Internet ist angesichts der großen Dynamik des Ausbruchs und des raschen täglichen Datenzuwachses eine unverzichtbare Informationsquelle (Infobox 1 und 2). Mehrfach täglich aktualisierte Daten zum Ausbreitungsgeschehen finden sich beispielsweise auf den Seiten der Johns Hopkins Universität. Aktuelle Informationen zu Klinik‑, Praxis- und Hygienemanagement, zu Labordiagnostik, Meldepflicht sowie zum Personenschutz sind auf den in der Infobox 1 angegebenen Internetseiten des Robert Koch-Instituts (RKI), der Bundeszentrale für gesundheitliche Aufklärung (BZgA) und der Weltgesundheitsorganisation (WHO) zu finden. Weiterhin haben alle wissenschaftlichen und medizinischen Fachverlage den vollständigen Zugriff auf Publikationen im Zusammenhang mit SARS-CoV-2/COVID-19 freigegeben. Exemplarisch soll hier auf die Portale von The Lancet, New England Journal of Medicine, Springer Nature Group und Science verwiesen werden (Infobox 3).
Personen mit akuter respiratorischer Symptomatik und möglicher Exposition sind auf SARS-CoV‑2 zu testen
Tipps für die Praxis zum Umgang mit respiratorisch Erkrankten/COVID-19 finden sich in Infobox 4.
Infobox 1 Allgemeine Informationen, Empfehlungen zu Diagnostik, Klinik‑, Praxis-, und zum Hygienemanagement
Weltgesundheitsorganisation (WHO): https://www.who.int/health-topics/coronavirus
Centers for Disease Control and Prevention (CDC): https://www.cdc.gov/coronavirus/index.html
European Centre for Disease Prevention and Control (ECDC): https://www.ecdc.europa.eu/en/coronavirus
Robert Koch-Institut (RKI): https://www.rki.de/DE/Content/InfAZ/N/Neuartiges_Coronavirus/nCoV.html
ProMED – International Society for Infectious Diseases: https://promedmail.org/coronavirus
Bundeszentrale für gesundheitliche Aufklärung (BZgA): https://www.infektionsschutz.de/coronavirus-sars-cov-2.html
Infobox 2 Echtzeitdaten zum Ausbruchsgeschehen und synoptische Zusammenfassung
Worldometer – real time world statistics: https://www.worldometers.info/coronavirus/
Johns Hopkins University: https://coronavirus.jhu.edu/map.html
Nextstrain: https://nextstrain.org/ncov
Infobox 3 Wissenschaftliche Publikationen
The Lancet: https://www.thelancet.com/coronavirus
New England Journal of Medicine: https://www.nejm.org/coronavirus
Springer Nature Group: https://www.springernature.com/gp/researchers/campaigns/coronavirus
Science: https://www.sciencemag.org/coronavirus-research-commentary-and-news
Infobox 4 Tipps für die Praxis zum Umgang mit respiratorisch Erkrankten/COVID-19
Alle Termine ausschließlich nach telefonischer Voranmeldung vergeben, Kontrolle der Termineinhaltung gewährleisten
Vor Terminvergabe telefonische Evaluation der Anamnese. Bei respiratorisch Erkrankten Falldefinition abfragen und Checkliste durch das Personal erstellen lassen
Sprechzeiten zwischen respiratorisch Kranken und anderen Patienten zeitlich und räumlich strikt trennen
Wenn möglich, trotz zeitlich und räumlicher Trennung separaten Warteraum für respiratorisch Erkrankte/Verdachtsfälle einrichten
Warte- und Praxisräume kontinuierlich lüften, bzw. mehrfach täglich regelmäßig stoßlüften (z. B. feste Zeiten dafür einführen)
Untersuchung von respiratorisch Erkrankten nur mit angelegter persönlicher Schutzausrüstung (PSA) durchführen
Soweit machbar, bei allen respiratorisch Erkrankten Virusnachweis auf SARS-CoV‑2 durchführen
Wenn am Ort vorhanden, Abklärung und Diagnostik immer über ein/einen COVID-19-Zentrum/Stützpunkt durchführen lassen
Hygienemanagement der Praxis optimieren und z. B. verstärkt Wischdesinfektionen durchführen, obligates Tragen von PSA und die Überwachen dieser Maßnahmen sicherstellen
Belehrung der Mitarbeiter und klare Anweisungen zum Verfahren erstellen
Patienten-Hand-out und Schilderinformation zum Praxismanagement erstellen
Auf der Homepage der Praxis die organisatorischen Änderungen publizieren. Bei geschlossener Praxis diese Informationen auch über den Anrufbeantworter mitteilen
Fazit für die Praxis
Angesichts der Fallzahlen von an „coronavirus disease 2019“ (COVID-19) Erkrankten und einer Sterblichkeit bis zu 9 % ist dringender Handlungsbedarf gegeben.
Verstärkt wird die Ausbreitung dadurch, dass ein Teil der Infektionen mild und wahrscheinlich auch symptomarm verläuft.
Da das Virus vermutlich schon vor oder spätestens beim Beginn der respiratorischen Symptomatik über respiratorische Sekrete ausgeschieden wird, übertragen infektiöse Personen den Erreger längere Zeit unerkannt. Deswegen gilt es, umgehend alle Personen mit akuter respiratorischer Symptomatik, deren Anamnese auf eine mögliche Exposition schließen lässt, auf SARS-CoV‑2 zu testen. Abklärung und Diagnostik sollten immer über ein/einen COVID-19-Zentrum/Stützpunkt durchgeführt werden.
Essenziell ist die Stärkung des Risikobewusstseins („public awareness“) in der Bevölkerung und bei den Patienten, da dieses die Reduktion von Kontakten zwischen den Menschen fördert. Das Tragen von Schutzmasken kann hierzu beitragen und verringert die Übertragung von SARS-CoV-2.
Einhaltung ethischer Richtlinien
Interessenkonflikt
F. Hufert und M. Spiegel geben an, dass kein Interessenkonflikt besteht.
Für diesen Beitrag wurden von den Autoren keine Studien an Menschen oder Tieren durchgeführt. Für die aufgeführten Studien gelten die jeweils dort angegebenen ethischen Richtlinien.
Contributor Information
F. Hufert, Email: Frank.Hufert@mhb-fontane.de
M. Spiegel, Email: Martin.Spiegel@mhb-fontane.de
Literatur
- 1.Heimdal I, Moe N, Krokstad S, Christensen A, Skanke LH, Nordbo SA, Dollner H. Human coronavirus in hospitalized children with respiratory tract infections: a 9-year population-based study from Norway. J Infect Dis. 2019;219(8):1198–1206. doi: 10.1093/infdis/jiy646. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Lu R, Zhao X, Li J, Niu P, Yang B, Wu H, Wang W, Song H, Huang B, Zhu N, et al. Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding. Lancet. 2020;395(10224):565–574. doi: 10.1016/S0140-6736(20)30251-8. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.Lim YX, Ng YL, Tam JP, Liu DX. Human Coronaviruses: a review of virus-host interactions. Diseases. 2016;4(3):26. doi: 10.3390/diseases4030026. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Spiegel M, Schneider K, Weber F, Weidmann M, Hufert FT. Interaction of severe acute respiratory syndrome-associated coronavirus with dendritic cells. J Gen Virol. 2006;87(Pt 7):1953–1960. doi: 10.1099/vir.0.81624-0. [DOI] [PubMed] [Google Scholar]
- 5.Niu J, Shen L, Huang B, Ye F, Zhao L, Wang H, Deng Y, Tan W. Non-invasive bioluminescence imaging of HCoV-OC43 infection and therapy in the central nervous system of live mice. Antiviral Res. 2020;173:104646. doi: 10.1016/j.antiviral.2019.104646. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Chu CM, Cheng VC, Hung IF, Wong MM, Chan KH, Chan KS, Kao RY, Poon LL, Wong CL, Guan Y, et al. Role of lopinavir/ritonavir in the treatment of SARS: initial virological and clinical findings. Thorax. 2004;59(3):252–256. doi: 10.1136/thorax.2003.012658. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Gorbalenya AE, Baker SC, Baric RS, et al. The species Severe acute respiratory syndrome-related coronavirus: classifying 2019-nCoV and naming it SARS-CoV-2. Nature. Microbiology. 2020;5(4):536–544. doi: 10.1038/s41564-020-0695-z. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Benvenuto D, Giovanetti M, Salemi M, Prosperi M, De Flora C, Junior Alcantara LC, Angeletti S, Ciccozzi M. The global spread of 2019-nCoV: a molecular evolutionary analysis. Pathog Glob Health. 2020 doi: 10.1080/20477724.2020.1725339. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Fan Y, Zhao K, Shi ZL, Zhou P. Bat Coronaviruses in China. Viruses. 2019;11(3):E210. doi: 10.3390/v11030210. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Rothe C, Schunk M, Sothmann P, Bretzel G, Froeschl G, Wallrauch C, Zimmer T, Thiel V, Janke C, Guggemos W, et al. Transmission of 2019-ncoV infection from an asymptomatic contact in Germany. N Engl J Med. 2020 doi: 10.1056/NEJMc2001468. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Dong Y, Mo X, Hu Y, Qi X, Jiang F, Jiang Z, Tong S. Epidemiological characteristics of 2143 pediatric patients with 2019 Coronavirus disease in China. Pediatrics. 2020 doi: 10.1542/peds.2020-0702. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Xu Y, Li X, Zhu B, Liang H, Fang C, Gong Y, Guo Q, Sun X, Zhao D, Shen J, et al. Characteristics of pediatric SARS-CoV-2 infection and potential evidence for persistent fecal viral shedding. Nat Med. 2020 doi: 10.1038/s41591-020-0817-4. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Zou Z, Yan Y, Shu Y, Gao R, Sun Y, Li X, Ju X, Liang Z, Liu Q, Zhao Y, et al. Angiotensin-converting enzyme 2 protects from lethal avian influenza A H5N1 infections. Nat Commun. 2014;5:3594. doi: 10.1038/ncomms4594. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Chinese Clinical Guidance for COVID-19 Pneumonia Diagnosis and Treatment (7th edition) [http://kjfy.meetingchina.org/msite/news/show/cn/3337.html]
- 15.Li R, Pei S, Chen B, Song Y, Zhang T, Yang W, Shaman J. Substantial undocumented infection facilitates the rapid dissemination of novel coronavirus (SARS-CoV2) Science. 2020 doi: 10.1126/science.abb3221. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Chinazzi M, Davis JT, Ajelli M, Gioannini C, Litvinova M, Merler S, Pastore y Piontti A, Mu K, Rossi L, Sun K, et al. The effect of travel restrictions on the spread of the 2019 novel coronavirus (COVID-19) outbreak. Science. 2020 doi: 10.1126/science.aba9757. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Tang X, Wu C, Li X, Song Y, Yao X, Wu X, Duan Y, Zhang H, Wang Y, Qian Z, et al. On the origin and continuing evolution of SARS-CoV-2. Nat Sci Rev. 2020 doi: 10.1093/nsr/nwaa036. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Cao Y, Li L, Feng Z, Wan S, Huang P, Sun X, Wen F, Huang X, Ning G, Wang W. Comparative genetic analysis of the novel coronavirus (2019-nCoV/SARS-CoV-2) receptor ACE2 in different populations. Cell Discov. 2020;6:11. doi: 10.1038/s41421-020-0147-1. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Tong ZD, Tang A, Li KF, Li P, Wang HL, Yi JP, Zhang YL, Yan JB. Potential presymptomatic transmission of SARS-coV-2, Zhejiang province, China, 2020. Emerg Infect Dis. 2020 doi: 10.3201/eid2605.200198. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Guan WJ, Ni ZY, Hu Y, Liang WH, Ou CQ, He JX, Liu L, Shan H, Lei CL, Hui DSC, et al. Clinical characteristics of Coronavirus disease 2019 in China. N Engl J Med. 2020 doi: 10.1056/NEJMoa2002032. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.Wang D, Hu B, Hu C, Zhu F, Liu X, Zhang J, Wang B, Xiang H, Cheng Z, Xiong Y, et al. Clinical characteristics of 138 hospitalized patients with 2019 novel Coronavirus-infected pneumonia in Wuhan, China. JAMA. 2020 doi: 10.1001/jama.2020.1585. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22.Yanping Z. The epidemiological characteristics of an outbreak of 2019 novel Coronavirus diseases (COVID-19)—China, 2020. CCDC Wkly. 2020;2:113–122. [PMC free article] [PubMed] [Google Scholar]
- 23.Huang C, Wang Y, Li X, Ren L, Zhao J, Hu Y, Zhang L, Fan G, Xu J, Gu X, et al. Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China. Lancet. 2020;395(10223):497–506. doi: 10.1016/S0140-6736(20)30183-5. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.Chen H, Guo J, Wang C, Luo F, Yu X, Zhang W, Li J, Zhao D, Xu D, Gong Q, et al. Clinical characteristics and intrauterine vertical transmission potential of COVID-19 infection in nine pregnant women: a retrospective review of medical records. Lancet. 2020;395(10226):809. doi: 10.1016/S0140-6736(20)30360-3. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.Abd El Wahed A, Patel P, Heidenreich D, Hufert FT, Weidmann M. Reverse transcription recombinase polymerase amplification assay for the detection of middle East respiratory syndrome coronavirus. PLoS Curr. 2013 doi: 10.1371/currents.outbreaks.62df1c7c75ffc96cd59034531e2e8364. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.Weidmann M, Zanotto PM, Weber F, Spiegel M, Brodt HR, Hufert FT. High-efficiency detection of severe acute respiratory syndrome virus genetic material. J Clin Microbiol. 2004;42(6):2771–2773. doi: 10.1128/JCM.42.6.2771-2773.2004. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27.Amer HM, Abd El Wahed A, Shalaby MA, Almajhdi FN, Hufert FT, Weidmann M. A new approach for diagnosis of bovine coronavirus using a reverse transcription recombinase polymerase amplification assay. J Virol Methods. 2013;193(2):337–340. doi: 10.1016/j.jviromet.2013.06.027. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 28.Corman VM, Landt O, Kaiser M, Molenkamp R, Meijer A, Chu DK, Bleicker T, Brunink S, Schneider J, Schmidt ML, et al. Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR. Euro Surveill. 2020 doi: 10.2807/1560-7917.ES.2020.25.3.2000045. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.Coronavirus Disease 2019 (COVID-19) – Information for Laboratories [https://www.cdc.gov/coronavirus/2019-ncov/lab/rt-pcr-detection-instructions.html]
- 30.MERS-CoV Infection tReated With A Combination of Lopinavir /Ritonavir and Interferon Beta-1b (MIRACLE) [https://clinicaltrials.gov/ct2/show/NCT02845843]
- 31.Cao B, Wang Y, Wen D, Liu W, Wang J, Fan G, Ruan L, Song B, Cai Y, Wei M, et al. A trial of lopinavir-ritonavir in adults hospitalized with severe Covid-19. N Engl J Med. 2020 doi: 10.1056/NEJMoa2001282. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 32.Sheahan TP, Sims AC, Leist SR, Schafer A, Won J, Brown AJ, Montgomery SA, Hogg A, Babusis D, Clarke MO, et al. Comparative therapeutic efficacy of remdesivir and combination lopinavir, ritonavir, and interferon beta against MERS-CoV. Nat Commun. 2020;11(1):222. doi: 10.1038/s41467-019-13940-6. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 33.de Wit E, Feldmann F, Cronin J, Jordan R, Okumura A, Thomas T, Scott D, Cihlar T, Feldmann H. Prophylactic and therapeutic remdesivir (GS-5734) treatment in the rhesus macaque model of MERS-CoV infection. Proc Natl Acad Sci U S A. 2020 doi: 10.1073/pnas.1922083117. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 34.Wang M, Cao R, Zhang L, Yang X, Liu J, Xu M, Shi Z, Hu Z, Zhong W, Xiao G. Remdesivir and chloroquine effectively inhibit the recently emerged novel coronavirus (2019-nCoV) in vitro. Cell Res. 2020 doi: 10.1038/s41422-020-0282-0. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 35.Mackenzie AH. Antimalarial drugs for rheumatoid arthritis. Am J Med. 1983;75(6a):48–58. doi: 10.1016/0002-9343(83)90474-6. [DOI] [PubMed] [Google Scholar]
- 36.Liu J, Cao R, Xu M, Wang X, Zhang H, Hu H, Li Y, Hu Z, Zhong W, Wang M. Hydroxychloroquine, a less toxic derivative of chloroquine, is effective in inhibiting SARS-CoV-2 infection in vitro. Cell Discov. 2020;6(1):16. doi: 10.1038/s41421-020-0156-0. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 37.Gao J, Tian Z, Yang X. Breakthrough: chloroquine phosphate has shown apparent efficacy in treatment of COVID-19 associated pneumonia in clinical studies. Biosci Trends. 2020 doi: 10.5582/bst.2020.01047. [DOI] [PubMed] [Google Scholar]
- 38.Coronavirus Disease 2019 (COVID-19)—Information for Clinicians on Therapeutic Options for COVID-19 Patients [https://www.cdc.gov/coronavirus/2019-ncov/hcp/therapeutic-options.html]
- 39.Laaksonen A-L, Koskiahde V, Juva K. Dosage of antimalarial drugs for children with juvenile rheumatoid arthritis and systemic lupus erythematosus: a clinical study with determination of serum concentrations of Chloroquine and Hydroxychloroquine. Scand J Rheumatol. 1974;3(2):103–108. doi: 10.3109/03009747409115809. [DOI] [PubMed] [Google Scholar]
- 40.Smith ER, Klein-Schwartz W. Are 1–2 dangerous? Chloroquine and hydroxychloroquine exposure in toddlers. J Emerg Med. 2005;28(4):437–443. doi: 10.1016/j.jemermed.2004.12.011. [DOI] [PubMed] [Google Scholar]
- 41.Hoffmann M, Kleine-Weber H, Schroeder S, Krüger N, Herrler T, Erichsen S, Schiergens TS, Herrler G, Wu N-H, Nitsche A, et al. SARS-coV-2 cell entry depends on ACE2 and TMPRSS2 and is blocked by a clinically proven protease inhibitor. Cell. 2020 doi: 10.1016/j.cell.2020.02.052. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 42.Zhou Y, Vedantham P, Lu K, Agudelo J, Carrion R, Nunneley JW, Barnard D, Pöhlmann S, McKerrow JH, Renslo AR, et al. Protease inhibitors targeting coronavirus and filovirus entry. Antiviral Res. 2015;116:76–84. doi: 10.1016/j.antiviral.2015.01.011. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 43.Midgley I, Hood AJ, Proctor P, Chasseaud LF, Irons SR, Cheng KN, Brindley CJ, Bonn R. Metabolic fate of 14C-camostat mesylate in man, rat and dog after intravenous administration. Xenobiotica. 1994;24(1):79–92. doi: 10.3109/00498259409043223. [DOI] [PubMed] [Google Scholar]
- 44.Clarke DK, Xu R, Matassov D, Latham TE, Ota-Setlik A, Gerardi CS, Luckay A, Witko SE, Hermida L, Higgins T, et al. Safety and immunogenicity of a highly attenuated rVSVN4CT1-EBOVGP1 Ebola virus vaccine: a randomised, double-blind, placebo-controlled, phase 1 clinical trial. Lancet Infect Dis. 2020 doi: 10.1016/S1473-3099(19)30614-0. [DOI] [PubMed] [Google Scholar]
- 45.First FDA-approved vaccine for the prevention of Ebola virus disease, marking a critical milestone in public health preparedness and response [https://www.fda.gov/news-events/press-announcements/first-fda-approved-vaccine-prevention-ebola-virus-disease-marking-critical-milestone-public-health]
- 46.Furuyama W, Reynolds P, Haddock E, Meade-White K, Le Quynh M, Kawaoka Y, Feldmann H, Marzi A. A single dose of a vesicular stomatitis virus-based influenza vaccine confers rapid protection against H5 viruses from different clades. NPJ Vaccines. 2020;5:4. doi: 10.1038/s41541-019-0155-z. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 47.Liu R, Wang J, Shao Y, Wang X, Zhang H, Shuai L, Ge J, Wen Z, Bu Z. A recombinant VSV-vectored MERS-CoV vaccine induces neutralizing antibody and T cell responses in rhesus monkeys after single dose immunization. Antiviral Res. 2018;150:30–38. doi: 10.1016/j.antiviral.2017.12.007. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 48.Callendret B, Vellinga J, Wunderlich K, Rodriguez A, Steigerwald R, Dirmeier U, Cheminay C, Volkmann A, Brasel T, Carrion R, et al. A prophylactic multivalent vaccine against different filovirus species is immunogenic and provides protection from lethal infections with Ebolavirus and Marburgvirus species in non-human primates. PLoS ONE. 2018;13(2):e0192312. doi: 10.1371/journal.pone.0192312. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 49.Ewer K, Rampling T, Venkatraman N, Bowyer G, Wright D, Lambe T, Imoukhuede EB, Payne R, Fehling SK, Strecker T, et al. A Monovalent chimpanzee adenovirus Ebola vaccine boosted with MVA. N Engl J Med. 2016;374(17):1635–1646. doi: 10.1056/NEJMoa1411627. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 50.Milligan ID, Gibani MM, Sewell R, Clutterbuck EA, Campbell D, Plested E, Nuthall E, Voysey M, Silva-Reyes L, McElrath MJ, et al. Safety and immunogenicity of novel adenovirus type 26- and modified vaccinia ankara-vectored Ebola vaccines: a randomized clinical trial. JAMA. 2016;315(15):1610–1623. doi: 10.1001/jama.2016.4218. [DOI] [PubMed] [Google Scholar]
- 51.Haagmans BL, van den Brand JM, Raj VS, Volz A, Wohlsein P, Smits SL, Schipper D, Bestebroer TM, Okba N, Fux R, et al. An orthopoxvirus-based vaccine reduces virus excretion after MERS-CoV infection in dromedary camels. Science. 2016;351(6268):77–81. doi: 10.1126/science.aad1283. [DOI] [PubMed] [Google Scholar]
- 52.The Ebola Epidemic in West Africa: Proceedings of a Workshop. 2, The Outbreak. [https://www.ncbi.nlm.nih.gov/books/NBK401927/] [PubMed]
- 53.Durham DP, Casman EA. Incorporating individual health-protective decisions into disease transmission models: a mathematical framework. J R Soc Interface. 2012;9(68):562–570. doi: 10.1098/rsif.2011.0325. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 54.Feng S,Shen C, Xia N, Song W, Fan M, Cowling BJ (2020) Rational use of face masks in the COVID-19 pandemic. The Lancet Respiratory Medicine (online ahead of print) [DOI] [PMC free article] [PubMed]