Résumé
Les coronavirus forment un grand groupe de virus infectant les mammifères et les oiseaux ; cinq d’entre eux infectent l’homme : HCoV 229E et OC43, connus depuis les années 1960, SARS-CoV identifié en mars 2003 lors de l’épidémie de syndrome de détresse respiratoire aigu sévère, NL63 et HKU1, identifiés respectivement en 2004 et 2005. Ce sont des virus enveloppés dont le génome est une molécule d’ARN de polarité positive de très grande taille. Leur mode évolutif fait intervenir plusieurs paramètres : la génération de nombreux mutants lors de la réplication, responsable d’une distribution en quasi-espèces de la population virale, la capacité à établir des infections persistantes, la possibilité de délétions importantes, la grande flexibilité du génome due à un fort taux de recombinaisons homologues et hétérologues, la capacité à franchir les barrières d’espèces et à s’adapter au nouvel environnement. Les coronavirus (hors SARS-CoV) sont ubiquitaires et circulent sous forme épidémique. Les différents HCoV co-circulent avec une distribution entre les types qui est variable selon les années et les régions géographiques. Ce sont essentiellement des virus responsables d’infections respiratoires hautes et basses. Le SARS-CoV est, lui, un virus émergent responsable de l’épidémie de pneumopathies atypiques entre novembre 2002 et juillet 2003. Sa circulation a été interrompue grâce à la mise en place de mesures sanitaires drastiques. L’implication des HCoV dans des pathologies digestives et neurologiques reste à préciser. La détection des coronavirus est difficile, et fait appel surtout à des techniques moléculaires. Il n’existe actuellement aucun traitement spécifique des infections à HCoV.
Mots clés: Coronavirus, Infection respiratoire, Syndrome respiratoire aigu sévère (SRAS), Barrière d’espèces, Évolution, Circulation épidémique, Détection moléculaire
Abstract
Coronaviruses are a large group of viruses and infect a lot of species of mammals and birds. Five coronaviruses currently infect humans: HCoVs 229E and OC43, identified in the 1960s, SARS-CoV identified in March 2003 during the SARS epidemic, and the HCoVs NL63 and HKU1, identified in 2004 and 2005 respectively. The genome of the coronaviruses is a linear, non-segmented, positive-sense single-stranded RNA molecule of approximately 30 kb. The evolution of these viruses occurs through some features: the generation of multiple mutants during the replication resulting on a quasispecies structure of the viral population, the demonstrated ability of coronaviruses to establish persistent infections, the flexibility of the genome due to a high frequency of homologue or heterologue recombinations, the ability to jump barrier species and to adapt to the new environment. Two epidemiologic pictures of HCoV infections have to be distinguished: as suggested by recent studies, HCoVs except SARS-CoV, are distributed worldwide and cocirculate during seasonal outbreaks. The distribution of the different HCoV species varies according to the geographic area and season. In contrast, the SARS-CoV is responsible of the first emerging infectious disease of this millennium, infecting more than 8000 people between November 2002 and July 2003. Its circulation has been stopped by drastic public health policy. Human coronaviruses may be also involved in enteric and neurologic diseases. The detection of these viruses is difficult and mainly based on molecular assays (RT-PCR). There is no established specific therapy to date.
Keywords: Coronavirus, Respiratory infection, Severe respiratory acute syndrome (SARS), Barrier species, Evolution, Epidemic circulation, Molecular assays
1. Introduction
La survenue récente, en 2002 à 2003, de l’épidémie de SRAS (ou syndrome respiratoire aigu sévère), et l’identification de l’agent pathogène responsable, un coronavirus émergent dans la population humaine, ont conduit à un vif regain d’intérêt et une intensification importante des recherches sur ces virus. Les premiers coronavirus humains ont été identifiés dans les années 1960 dans le cadre d’infections respiratoires hautes d’allure bénigne. Ils ont été longtemps considérés comme un des agents principaux, avec les rhinovirus, du rhume banal. Même si rapidement, certains travaux ont suggéré leur implication dans des infections respiratoires plus graves, des pathologies entériques et neurologiques, ces virus sont longtemps restés un sujet marginal en médecine humaine. La plupart des données virologiques sur les coronavirus avant le SRAS intéresse le domaine vétérinaire, où ces virus peuvent être à l’origine d’infections graves avec de lourdes conséquences économiques sur les élevages de volailles et de porcs en particulier. Depuis 2003, 24 nouveaux coronavirus ont été identifiés, trois chez l’homme, dix chez les autres mammifères, et 11 chez les oiseaux. Le nombre de séquences de coronavirus référencées dans GenBank en juillet 2007 est de l’ordre de 3000, incluant 264 génomes complets de 25 espèces de coronavirus différents, soit une croissance exponentielle des données génétiques disponibles concernant ces virus [1]. Les recherches menées pour identifier les réservoirs animaux du SARS-CoV ont mis en évidence le fort potentiel évolutif de ces virus, leur très large spectre d’hôtes, et leur importante diversité génétique. L’objectif de cette revue est de présenter, de façon non exhaustive, les principales données virologiques, épidémiologiques et cliniques accumulées depuis plus de 50 ans sur ce groupe de virus.
2. Historique
Le genre « coronavirus » a été créé en 1967 et a regroupé à partir de critères essentiellement morphologiques des virus animaux connus depuis les années 1930 (virus de la bronchite infectieuse ou IBV, virus de l’hépatite murine ou MHV, virus de la gastroentérite porcine ou TGEV) et des virus alors récemment identifiés chez l’homme (souches B814, 229E, OC43, OC48, 692) [2], [3], [4]. Le terme « coronavirus » évoque l’aspect en couronne des virions en microscopie électronique (Fig. 1 ). La taxinomie virale a ensuite été régulièrement revue : l’ordre des Nidovirales, créé en 1996, regroupe actuellement trois familles, les Coronaviridae, les Arteriviridae, et les Roniviridae. Tous ces virus ont en commun l’organisation du génome ARN et la stratégie de réplication, mais ils diffèrent dans leur morphologie, la structure de leur capside, et la taille de leur génome, qui va de 13 000 nucléotides pour les arterivirus à 31 000 nucléotides pour les coronavirus. La famille des Coronaviridae est constituée de deux genres, les coronavirus et les torovirus. Parmi les Nidovirales, seul le genre coronavirus comprend des virus identifiés chez l’homme [5]. Les coronavirus ont été divisés en trois groupes distincts nommés 1, 2 et 3, sur la base de données d’abord sérologiques, puis moléculaires. Le nombre d’espèces de coronavirus décrites a considérablement augmenté depuis 2003 et la classification s’est affinée. Les coronavirus des groupes 1 et 2 infectent les mammifères, dont l’homme ; et les coronavirus du groupe 3 constituent un groupe de virus aviaires. La place du SARS-CoV dans cette classification a beaucoup été débattue, les différentes analyses phylogéniques proposaient soit de le placer dans un quatrième groupe, soit dans un groupe 2 « élargi ». Finalement, cette dernière solution a été adoptée ; actuellement, le groupe 2 est subdivisé en 2a et 2b, et comprend le SARS CoV ainsi que tous les virus « SARS-CoV-like » ou SL-CoV décrits chez les différentes espèces animales [1], [6] (Fig. 3).
Fig. 1.
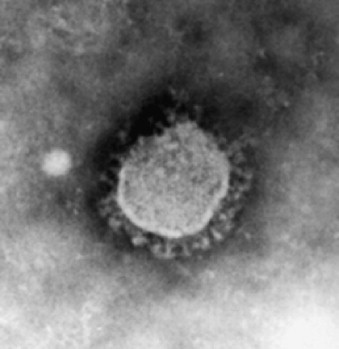
Particule de coronavirus en microscopie électronique (Pierre Lebon, Saint-Vincent de Paul, Paris). L’aspect en couronne est du à la présence de hauts spicules constitués de la protéine de surface S.
Fig. 3.
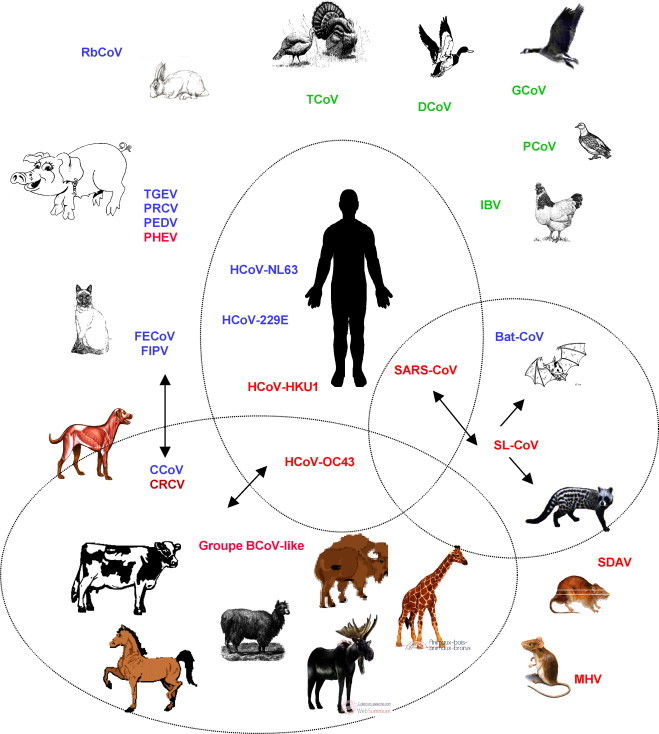
Spectre d’hôtes des différentes espèces de coronavirus. Les coronavirus du groupe 1 sont écrits en bleu, ceux du groupe 2, en rouge, et ceux du groupe 3 (coronavirus aviaires) en vert. Les flèches indiquent les hypothétiques franchissements de barrières d’espèces avec réussite émergentielle.
Les coronavirus humains sont au nombre de cinq : HCoV-229E et HCoV-OC43, décrits dans les années 1960 ; le SARS-CoV, identifié en 2003 lors de l’épidémie de SRAS ; HCoV-NL63, décrit en 2004 aux Pays-Bas, et enfin HCoV-HKU1, découvert en 2005 à Hong-Kong [7], [8], [9], [10]. La chronologie des évènements et l’histoire font que les coronavirus 229E et OC43 sont souvent désignés comme les coronavirus « classiques », alors que les HCoV NL63 et HKU1 sont désignés comme les « nouveaux » coronavirus. Le SARS-CoV, quant à lui, et tous les SL-CoV, forment un groupe à part, et sont à l’origine de la majorité des publications scientifiques. Les HCoV 229E et NL63 appartiennent au groupe 1, les HCoV OC43 et HKU1 au groupe 2a, et les SARS-CoV et SL-CoV au groupe 2b. Tous ces virus ont été identifiés à partir de prélèvements respiratoires, soit après isolement en culture, soit directement à partir des prélèvements cliniques, par biologie moléculaire. HCoV-229E a été isolé en 1966 sur cellules rénales embryonnaires humaines et a été adapté à plusieurs types de cellules, dont les MRC5, cellules largement utilisées dans les laboratoires de virologie. HCoV-OC43 a été isolé en 1967 sur culture de trachée, puis, après passages sur cerveaux de souriceau, à la lignée HRT18 (carcinome rectal humain) ; HCoV-NL63 (souche Amsterdam 1) a été isolé en 2004 sur la lignée LLC-MK2, le SARS-CoV sur cellules Vero E6 ; seul HCoV-HKU1 à ce jour n’a jamais été isolé en culture cellulaire et a été uniquement caractérisé par biologie moléculaire. Il est important de noter qu’en dehors des souches prototypes, les coronavirus restent très difficiles à cultiver et il existe peu d’isolats. Rappelons enfin que ces virus sont des agents de classe 2, sauf le SARS-CoV qui nécessite un laboratoire de confinement de niveau 3 pour sa manipulation.
3. Coronavirus : tropisme et entrée dans les cellules cibles
Les coronavirus sont des particules virales enveloppées pléomorphes de 60 à 200 nm de diamètre. L’aspect en couronne visible en microscopie électronique est dû à la présence sur l’enveloppe virale de spicules en forme de massue de 20 nm de hauteur et constitués de la protéine de surface S (Fig. 1). Les autres glycoprotéines d’enveloppe sont la protéine M, la protéine E et, pour les coronavirus du groupe 2, l’hémaglutinine-estérase HE. La capside virale est de symétrie hélicoïdale, elle est constituée de la protéine N, qui est étroitement liée à l’ARN génomique (Fig. 2 ). La protéine S est une glycoprotéine membranaire de type I, elle est organisée en trimères, et est composée de deux sous-unités nommées S1 (partie globulaire) et S2 (partie en forme de tige). La protéine S joue un rôle primordial dans les premières étapes du cycle viral : elle est responsable de l’attachement du virion à la cellule cible par sa sous-unité S1, et détermine en grande partie le tropisme tissulaire du virus et son spectre d’hôte ; elle est également responsable de la fusion membranaire par sa sous-unité S2. Par ailleurs, elle est la cible principale de la réponse immunitaire cellulaire et humorale et induit la formation d’anticorps neutralisants [11]. De ce fait, comme la plupart des protéines de surface, elle présente des régions hypervariables, lui permettant d’échapper à la pression immunitaire, et, le cas échéant, de pouvoir élargir son tropisme cellulaire. La protéine S des coronavirus possède une faible activité hémagglutinante et se lie aux acides sialiques. Cependant, l’entrée dans les cellules cibles semble requérir l’interaction avec un récepteur protéique spécifique. Ainsi, des récepteurs cellulaires sont identifiés pour certains coronavirus : molécule CEACAM1 pour le virus de l’hépatite murine MHV, l’aminopeptidase N (APN) ou CD13 pour plusieurs coronavirus du groupe 1 (HCoV-229E, coronavirus porcins TGEV et PRCV, coronavirus canins et félins), la molécule ACE2 pour HCoV-NL63 et le SARS-CoV [11], [12], [13], [14]. Les interactions entre protéine S et récepteur semblent complexes, et de nombreuses données restent incomprises : le site de liaison de S à son récepteur (receptor binding domain [RBD]) est localisé dans différentes régions de la protéine selon l’espèce de coronavirus, le clivage de S en ses deux sous-unités S1 et S2 est variable selon le coronavirus et le type cellulaire, le rôle de la liaison aux acides sialiques n’est pas déterminé (corécepteur ?) [11], [15], [16], [17]. Certaines données expérimentales sont inattendues : malgré des séquences en aminoacides conservées au niveau de la protéine S1 des HCoV 229E et NL63, ces deux coronavirus humains utilisent des récepteurs différents (APN et ACE2, respectivement) ; par ailleurs, le SARS-CoV utilise le même récepteur cellulaire que NL63 alors que les séquences S1 sont éloignées, cependant le RBD des deux virus semble proche et il est absent chez les SL-CoV. L’hypothèse est posée d’une acquisition de ce domaine par recombinaison entre le SARS-CoV et un autre coronavirus proche de NL63 lors de son évolution chez l’homme [18]. L’apparente plasticité de la protéine S et du RBD permettrait aux coronavirus de s’adapter à différents récepteurs protéiques ou à des récepteurs hétérologues dans différentes espèces et serait un atout pour émerger chez de nouveaux hôtes.
Fig. 2.
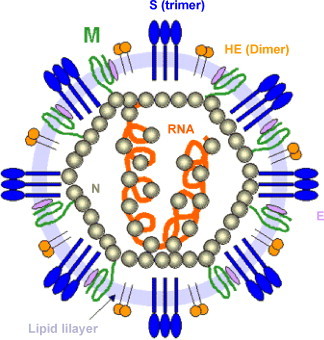
Représentation schématique d’un coronavirus. (pathmicro.med.sc.edu/virol/coronaviruses.htm.) La nucléocapside hélicoïdale est formée de la protéine N, étroitement liée à l’ARN génomique. Les autres protéines structurales comprennent les protéines M et E, et les protéines de surface S et HE. Cette dernière est présente uniquement chez les coronavirus du groupe 2a.
Les coronavirus du groupe 2a sont caractérisés par l’existence d’une protéine HE qui forme une double rangée de petits spicules de 5 nm de hauteur à la surface du virion. Il s’agit d’une protéine dimérique possédant une activité hémagglutinante et acétyl-estérase. Le gène codant cette protéine est caractéristique des CoV du groupe 2a, cependant son expression est très variable. Ainsi, dans la plupart des isolats MHV, des mutations, délétions ou insertions ont conduit à la perte de la phase ouverte de lecture de ce gène [19], [20], [21], [22], [23]. Parmi les coronavirus humains, seules les souches OC43 et HKU1 possèdent le gène codant HE. Il existe une homologie d’environ 28 % entre la protéine de surface HEF du virus influenza C et la protéine HE du HCoV-OC43 et des coronavirus bovins (BCoV). Les virus influenza C et HCoV-OC43 infectant les mêmes tissus chez l’homme, cette homologie suggère l’acquisition de ce gène par recombinaison. Il est à noter que la protéine HEF du virus Influenza C possède une activité de fusion membranaire, absente chez la protéine HE des CoV [19], [21], [24], [25]. Les propriétés biologiques de cette protéine restent obscures. Elle reconnaît les récepteurs cellulaires contenant des acides sialiques 9-O acétylés et induit la formation d’anticorps neutralisants. Elle aurait ainsi une fonction de protéine d’attachement et d’initiation de l’infection, additive à celle de la protéine S. Cependant, sa fonction principale serait l’activité acétyl-estérase. De nombreuses interrogations existent sur le déroulement in vivo des premières étapes du cycle de réplication pour les CoV du groupe 2a. La protéine HE a requis peu d’attention, probablement parce qu’elle serait « accessoire », et aussi parce qu’elle n’est pas présente chez les CoV les plus étudiés jusqu’à présent (IBV, MHV, TGEV, SARS-CoV, HCoV 229E et NL63). Des récepteurs protéiques cellulaires, en plus de la liaison aux acides sialiques ont été décrits pour un certain nombre de coronavirus. Mais certains, comme les BCoV et le HCoV OC43, utiliseraient seulement un acide sialique, le même que celui utilisé par le virus influenza C. Les acides sialiques correspondent au nom générique donné à une grande famille de monosaccharides à neuf carbones, chargés négativement, et représentant les résidus terminaux des glycoconjugués. Ce sont des récepteurs idéaux car ils sont nombreux et facilement accessibles. Cependant, ils permettent aussi la liaison des virus aux cellules non permissives, aux composés sialysés solubles, ou aux mucines sialysés de la barrière muqueuse. Toutes ces liaisons, ajoutées à l’agrégation des virus entre eux, entraînent une possible perte d’infectivité. C’est pour contrecarrer ces effets que quelques virus enveloppés à ARN se sont dotés d’enzymes ayant une activité de destruction des récepteurs associés aux virions (activité receptor destroying enzyme [RDE]) Les RDEs sont de deux types, soit des sialidases ou neuraminidases, trouvés chez les para- et orthomyxovirus ; ou des sialate-O-acetylesterases, trouvés chez le virus influenza C, les coronavirus, et les torovirus. Pour tous les CoV du groupe 2a étudiés jusqu’à présent, la liaison de la protéine S à un acide sialique correspond avec le substrat préférentiel de HE. La protéine HE permettrait donc un usage optimal des acides sialiques comme facteurs d’attachement. Le mode d’entrée des CoV exprimant la protéine HE serait alors proche de celui adopté par les virus influenza A et B, et la balance fonctionnelle décrite entre hémagglutinine et neuraminidase HA/NA pourrait également exister entre S et HE [11], [15], [26], [27].
4. Génome et évolution
Le génome des coronavirus est une molécule d’ARN linéaire, non segmentée, directement infectieuse. Sa caractéristique principale est sa taille qui est de 27 à 31 000 nucléotides. Il s’agit du plus grand ARN viral connu. L’organisation génomique est conservée parmi toutes les espèces de coronavirus. Les deux premiers tiers du génome, soit environ 20 000 nucléotides, sont constitués de deux cadres de lecture ORF1a et 1b chevauchant codant deux précurseurs protéiques d’une taille et d’une complexité sans précédent. Ces précurseurs sont clivés en 15 à 16 fragments qui forment le complexe de réplication. En aval, on trouve les quatre à cinq gènes codant les protéines structurales, dans un ordre précis et conservé (HE-S-E-M-N). Le génome des coronavirus comprend aussi des gènes codant des protéines non structurales dont la fonction n’est pas encore connue. La réplication des coronavirus dans les cellules eucaryotes est entièrement intracytoplasmique, elle fait appel à une stratégie particulière aboutissant à la synthèse discontinue d’ARN subgénomiques de taille décroissante, ayant tous la même extrémité 3’. La taille du génome et la complexité du mode de réplication ont longtemps été un obstacle à l’étude des coronavirus. Les récents progrès réalisés dans les études de génétique inverse et de biochimie structurale permettront probablement de répondre aux nombreuses questions sur la biologie de ces virus [28], [29], [30].
L’importante plasticité du génome des coronavirus fait de ces virus des agents à fort potentiel évolutif. Les deux modes d’évolution majeurs des coronavirus sont les mutations et les recombinaisons (Fig. 3 ). L’ARN génomique est répliqué par une ARN polymérase ARN dépendante (RdRp), dépourvue de système de correction d’erreur, et générant de nombreux mutants. Comme pour tous les virus à ARN, la population virale est hétérogène et possède une distribution en quasi-espèces. Cette distribution peut être vue comme une stratégie d’optimisation, en étant une structure permettant de disposer d’un réservoir de variants possédant les capacités de faire face aux changements environnementaux. Elle a été décrite pour plusieurs coronavirus non seulement dans le cadre d’infections persistantes, mais aussi d’infections aiguës [31], [32], [33], [34], [35].
La très grande taille du génome permet l’émergence de variants présentant de larges délétions, et permet l’utilisation de ces virus comme vecteurs viraux. L’exemple le plus connu est l’émergence du coronavirus porcin respiratoire ou PRCV, dans les années 1980. Le PRCV est un variant spontané du coronavirus porcin entérique ou TGEV. Il présente une délétion en phase de 672 nucléotides (224 acides aminés) dans le gène codant la protéine S1. Une des conséquences biologiques de cette grande délétion est le changement de tropisme du virus qui, d’entérique pour le TGEV, est devenu respiratoire pour le PRCV [36]. Soulignons que, dans ce cas, l’émergence d’un variant a été bénéfique pour l’hôte. L’infection respiratoire à PRCV est peu symptomatique et bénigne, sa transmission respiratoire est facile et efficace, et elle permet l’acquisition d’une immunité croisée avec le TGEV, maladie entérique d’évolution grave chez les porcelets.
L’autre mode évolutif des coronavirus est la recombinaison génétique. Ce phénomène est fréquent chez les virus à ARN positif, et semble favorisé chez les coronavirus, par le mode discontinu de la transcription. La recombinaison correspond à l’échange de matériel génétique, elle peut être homologue si elle a lieu entre deux génomes de coronavirus, ou hétérologue si elle intéresse d’autres gènes viraux ou cellulaires. De nombreuses formes recombinantes ont été décrites in vitro et in vivo chez les coronavirus. Par exemple, Herrewegh et al. ont montré que les coronavirus félins de type II sont le résultat d’une double recombinaison entre coronavirus félins de type I et coronavirus canins, faisant suite à un franchissement de barrière d’espèces dans le sens chien–chat [37]. L’importante capacité évolutive des coronavirus a été mise en lumière par l’émergence du SARS-CoV, dont nous reparlerons plus loin, et a relancé les études chez les autres membres de cette famille.
5. Franchissement de barrières d’espèces et émergence coronavirale
Depuis 2003, de très nombreuses données moléculaires ont été publiées, notamment sur le groupe 2a. Le séquençage complet du génome de HCoV-OC43 en 2005 par Vijgen et al. a montré en outre, que ce coronavirus est très proche des coronavirus bovins, avec plus de 90 % d’identité nucléotidique, et d’autre part, que le gène E aurait été acquis suite à une recombinaison avec le coronavirus porcin du groupe 2, le virus de l’encéphalite hémagglutinante ou HEV. Ces données suggèrent fortement une émergence de HCoV-OC43 dans la population humaine, secondaire à une transmission inter-espèces dans le sens bovins – humains, qui aurait eu lieu à la fin du xix e siècle [38], [39], [40]. Plusieurs coronavirus appartenant au groupe 2a, et au même lignage moléculaire que HCoV-OC43 et BCoV ont été décrits récemment chez l’élan, le buffle, la girafe, le cheval, et le chien [41], [42], [43], [44], [45]. Il semble donc que ces coronavirus du groupe 2a « BCoV-like », aient un spectre d’hôtes très large chez les mammifères sauvages et d’élevage, et possèdent un fort potentiel de passage interspécifique.
De nombreuses études ont été menées à la recherche du réservoir animal du SARS-CoV. Dès l’identification, en mars 2003, du coronavirus responsable de l’épidémie de SARS, l’hypothèse d’un réservoir animal a été retenue. Les premières données sérologiques provenant de la province de Canton, ont montré une séroprévalence significativement plus élevée chez les personnes travaillant au contact des animaux, notamment sur les marchés vivants [46]. Rappelons que ces marchés hébergent pour une période limitée de nombreuses espèces animales ne partageant pas la même écologie dans les conditions naturelles ou les conditions d’élevage, ils constituent donc pour les virus une interface favorable au franchissement interspécifique. La première étude animale a été menée dès mai 2003, et a inclus 25 animaux appartenant à huit espèces différentes. Parmi eux, six civettes masquées (Paguma larvata) et un chien viverrin (Nyctereutes procyonoides) ont été trouvés porteurs de SL-CoV essentiellement par RT-PCR à partir de prélèvements nasaux et fécaux [47]. Dès cette époque, l’alarme a été donnée concernant la consommation de civettes, qui ont été éliminées des menus des restaurants suite à un abattage massif. La consommation de viande de civette est devenue populaire en Chine depuis les années 1980 ; on estime qu’en 2003, environ 40 000 civettes masquées étaient élevées dans 660 fermes sur l’ensemble du territoire chinois [48]. Ces animaux supportent la réplication du SL-CoV sans symptôme apparent et constituent le ou une partie du réservoir animal. Une étude ultérieure a montré que les civettes semblaient être contaminées lors de leur passage dans les marchés, le pourcentage de civettes prélevées dans les fermes d’élevage et SL-CoV positives étant nul [49]. Ces résultats sont cependant controversés [50]. Actuellement, dix génomes complets de SL-CoV provenant de civettes ont été séquencés. Leur comparaison avec les séquences de SARS-CoV humains montrent, sur environ 30 000 nucléotides, un total de 212 positions de variation, dont 209 dans une région codante protéique (73 de ces 209 sont silencieuses) [51]. L’épidémie de SARS a été divisée en trois périodes, précoce, intermédiaire, et tardive, sur la base des données moléculaires des souches prélevées chez les patients : la période précoce s’étend de novembre 2002 à janvier 2003, l’intermédiaire de janvier à février 2003, et la tardive de février à juillet 2003. À cette date, la transmission interhumaine de SARS-CoV a été déclarée interrompue. Les séquences de SARS-CoV de la période précoce sont proches des séquences de SL-CoV des civettes, en particulier l’existence d’une séquence de 29 nucléotides située au niveau de l’ORF8, qui a ensuite disparue lors de l’adaptation du virus à l’homme (délétion de 29 nt) [52]. Au cours de l’évolution du SARS-CoV, la mutation du résidu aminoacide 487 (de serine chez la civette en thréonine chez l’homme) de la protéine S semble avoir contribué de façon importante à l’adaptation du SARS-CoV au récepteur humain ACE2 [53].
Certaines chauves-souris du genre Rhinolophus ont récemment été identifiées comme le réservoir naturel de SL-CoV par deux équipes indépendantes [54], [55]. En particulier, le bat SL-CoV a été détecté par RT-PCR chez l’espèce Rhinolophus sinicus à partir de prélèvements anaux réalisés dans l’habitat naturel. Les séquences complètes de six génomes bat SL-CoV sont disponibles : le pourcentage de similarité des séquences bat SL-CoV entre elles est de 89–90 %, et avec les séquences civets SL-CoV et SARS-CoV, de l’ordre de 87 à 92 %. Les régions les plus variables sont le gène S (similarité 76–78 %) et l’ORF8. La région de 29 nt trouvée chez les civets SL-CoV et les souches humaines de la phase précoce est également trouvée chez les bat SL-CoV. Les différences-clefs entres souches humaines et de civettes d’une part, et les souches de chauve-souris d’autre part, sont concentrées dans la protéine S, qui détermine le spectre d’hôte et le tropisme tissulaire [48].
En dehors des civettes masquées et des chauves-souris, d’autres animaux ont été testés (29 espèces différentes, 13 familles, huit ordres, deux classes), et certains (sept espèces) ont été trouvés porteurs du virus dans certaines circonstances. Les essais d’infection expérimentales, en vue d’obtenir des modèles, notamment pour le développement de vaccins, ont montré que de nombreux mammifères supportaient la réplication du SARS-CoV (singes, chats, furets, souris, porcs, cobayes, hamsters), cependant, la transmission de virus à partir des animaux inoculés semble difficile [48].
En conclusion, l’hypothèse actuellement retenue est l’existence d’un réservoir naturel de SL-CoV chez les chauves-souris. Ces animaux, seuls mammifères volants, constituent à eux seuls 20 % de l’ensemble des mammifères, ils sont répartis sur tout le globe, et sont identifiés comme le réservoir de nombreux virus émergents ou réémergents dans la population humaine : virus Hendra, virus Nipah, lyssavirus, virus Ebola. Il est à noter que les chauves-souris hébergent d’autres coronavirus, notamment un bat-CoV du groupe 1. Ce réservoir naturel serait à l’origine de la contamination de plusieurs espèces animales trouvées sur les marchés, parmi elles, les civettes Paguma Larvata auraient un rôle amplificateur important et serait à l’origine de la contamination des humains (Fig. 4 ).
Fig. 4.
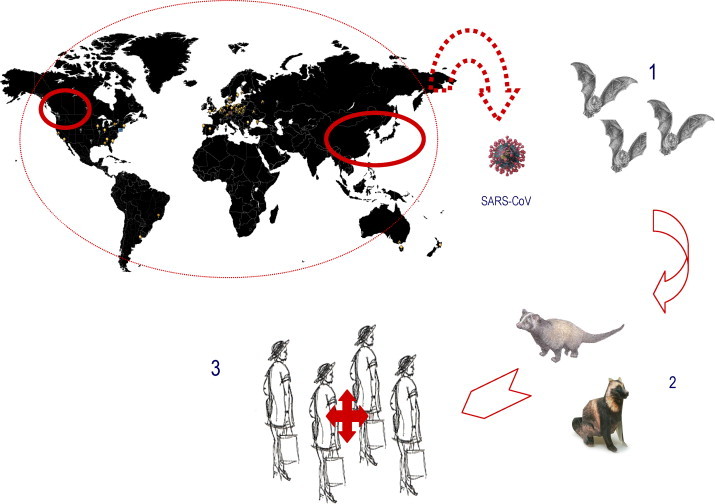
Épidémie de syndrome respiratoire aigu sévère ou SRAS (novembre 2002 à juillet 2003). Le nombre de cas probable de SRAS est de 8000, avec environ 800 décès. L’épidémie a débuté en Chine du Sud (région de Canton). Le Canada a été le pays non asiatique le plus touché par l’épidémie. Le réservoir naturel du virus serait les chauve-souris (genre Rhinolophus)(1). Les civettes masquées paguma larvata, et les chiens viverrins (racoon dog) seraient des hôtes intermédiaires contaminés lors de leur passage sur les marchés vivants, à l’origine de la contamination de l’homme (2,3).
6. Épidémiologie des coronavirus : transmission et circulation
La connaissance des modes de transmission constitue l’un des éléments les plus importants pour la prévention des infections respiratoires virales, et cela d’autant plus qu’il n’existe pas encore de traitement spécifique pour beaucoup d’entre elles. Les durées d’incubation des infections à coronavirus humains sont courtes : de l’ordre de trois jours pour les CoV classiques, et de deux à dix jours pour le SARS-CoV. Les durées d’excrétion virale dans les voies respiratoires sont moins bien connues, l’ARN des HCoV classiques est détectable pendant environ 14 jours dans les voies respiratoires [56]. L’ARN du SARS-CoV peut être détecté par RT-PCR dans les sécrétions respiratoires, les selles, et les urines des patients jusqu’à environ 30 jours après le début des signes cliniques. Cependant, l’isolement en culture de formes infectieuses n’a pas été possible à partir de ces prélèvements après trois semaines de maladie [57]. L’apparente dissociation des résultats moléculaires et cellulaires peuvent être le fait d’un manque de sensibilité des systèmes de culture, ou la neutralisation des formes infectieuses par l’immunité locale.
La transmission des CoV se fait principalement de façon directe par les gouttelettes de sécrétions oropharyngées dispersées par la toux d’une personne infectée et symptomatique. Au cours du SRAS, les personnes infectées étaient essentiellement celles qui avaient eu un contact rapproché avec un cas (vie commune ou prise en charge). Le port de masque et le lavage des mains sont les mesures de prévention les plus efficaces [58]. La dissémination virale aérienne semble peu fréquente ainsi que la transmission indirecte « manu-portée » ; cependant, ces voies de transmission doivent être prises en compte pour le contrôle des épidémies, notamment en milieu de soins. Les études de « survie » ou de maintien de l’infectiosité dans l’air sont rares et difficiles. L’absence de standardisation et de maîtrise de nombreux paramètres rendent difficilement comparables les données de la littérature. Quoi qu’il en soit, les coronavirus perdent leur infectiosité au contact des désinfectants et fixateurs les communément utilisés [59]. Le nombre de cas secondaires à partir d’un cas index n’a été étudié que dans le cadre du SRAS en 2003. Ce virus est modérément contagieux, avec un nombre moyen de cas secondaires estimé de 2,2 à 3,6. Cependant, des évènements de superpropagation avec plusieurs dizaines de cas secondaires ont été décrits, et ont joué un rôle important dans la diffusion de la maladie [60].
Le SRAS constitue une histoire particulière au sein des infections à coronavirus ; cette épidémie a été particulièrement bien documentée. Elle a été divisée rétrospectivement en trois périodes décrites plus haut. La phase précoce consiste en l’émergence de plusieurs cas de pneumopathies atypiques chez des individus sans lien épidémiologique dans la province de Canton. La phase intermédiaire débute par une épidémie nosocomiale hospitalière (106 cas secondaires à partir d’un patient index). La phase tardive correspond à la diffusion mondiale de l’épidémie à partir de l’hôtel Métropole à Hong Kong. Dans cet hôtel, un patient index contaminera 12 personnes logeant au même étage, et en transfert vers des destinations variées [52], [61]. L’alerte mondiale est donnée par l’OMS le 12 mars 2003. Environ 8000 cas probables et 800 décès ont été déclarés, la grande majorité en Chine. Le taux de mortalité estimé est égal à 0 % pour les sujets de moins de 35 ans, de 7 % pour les sujets de 35 à 65 ans, et de 47 % chez les sujets de plus de 65 ans. La fin de la transmission interhumaine est déclarée par l’OMS en juillet 2003, elle a été obtenue grâce aux mesures sanitaires drastiques appliquées dans les différents pays. En France, l’institut de veille sanitaire a été chargé de la surveillance et de l’investigation épidémiologique nationale des cas de SRAS. En mai 2003, 426 cas possibles ont été notifiés, sept ont été considérées comme probables et seulement quatre ont été confirmés par la biologie [62]. Depuis juillet 2003, plusieurs cas de contamination de laboratoire ont été rapportés en Asie. Enfin, les rapports épidémiologiques émanant du Guangdong Center for Disease Control and Prevention ont rapporté qu’en janvier 2004, soit six mois après la fin de l’épidémie, quatre patients ont été hospitalisés pour une infection bénigne par le SARS-CoV. L’étude moléculaire de ces souches a conclu qu’elles dérivaient de la même source que les souches épidémiques de 2002 à 2003 [51]. Ces résultats sont importants car ils montrent que la réémergence du SARS-CoV reste possible, et doit faire l’objet d’une surveillance, notamment dans cette région du monde, où d’autres virus respiratoires potentiellement émergent circulent largement. Le diagnostic virologique des infections respiratoires même bénignes est un élément central de cette surveillance.
Concernant les autres coronavirus humains, des données épidémiologiques anciennes étaient disponibles avant le SRAS pour les coronavirus classiques 229E et OC43. Parmi toutes les souches isolées dans les années 1960, seules celles-ci ont été maintenues en culture et étudiées. Ces données proviennent des grandes études d’épidémiologie descriptive menées notamment aux États-Unis dans les années 1970 [63], [64], [65], [66]. Ces études ont montré que les HCoV représentaient un groupe de pathogènes respiratoires importants pour tous les groupes d’âge. Ils sont, avec les rhinovirus, les principaux agents des infections respiratoires hautes, et sont aussi impliqués dans les infections respiratoires basses (bronchite, bronchiolite, pneumopathies, exacerbations d’asthme). Les primo-infections surviennent dans les premières années, et les réinfections sont fréquentes toute au long de la vie. Ces réinfections sont symptomatiques dans environ 45 % des cas. Le taux d’infection est relativement uniforme dans tous les groupes d’âge ; cette situation est particulière, et diffère de celle observée pour des virus respiratoires comme le virus respiratoire syncytial (VRS), pour lequel les taux d’infection diminuent avec l’âge. Les coronavirus classiques circulent sur un mode épidémique, le plus souvent entre janvier et mai dans les zones à climat tempéré. Le caractère cyclique et alternatif des souches 229E et OC43 avait été souligné, ainsi que l’existence probable d’autres sérotypes, suggérée par l’utilisation combinée de plusieurs techniques sérologiques telles que la fixation du complément, l’inhibition de l’hémagglutination, et la séroneutralisation [67].
Concernant les nouveaux coronavirus NL63 et HKU1, leur identification récente rend compte d’un nombre réduit de données épidémiologiques. Plusieurs points sont à souligner concernant leur découverte. Le HCoV-NL63 est un coronavirus de groupe 1 découvert trois fois par trois équipes différentes [9], [68], [69]. La « paternité » de ce virus revient cependant à Lia van der Hoek et al. qui ont identifié ce virus par une technique moléculaire originale à partir d’un prélèvement respiratoire d’un nourrisson de sept mois hospitalisé pour bronchiolite en janvier 2003. La souche prototype a été appelée Amsterdam 1 [9]. Une autre équipe hollandaise a identifié le même virus à partir d’un prélèvement respiratoire réalisé en 1988 chez un garçon souffrant d’une pneumopathie [69]. Cette observation, et d’autres études rétrospectives ont montré que NL63 n’était pas un virus émergent, mais un virus circulant déjà dans la population humaine, et nouvellement identifié. Certains auteurs pensent qu’il s’agit peut-être de la souche B814, isolé dans les années 1960, et perdue ensuite en laboratoire [9], [69], [70], [71], [72], [73], [74], [75]. Le HCoV-HKU1, lui, est un coronavirus du groupe 2a, découvert à Hong Kong en 2005, chez un patient de 71 ans hospitalisé pour une pneumonie. Ce virus a été caractérisé sur le plan moléculaire, et n’a pas encore été adapté à la culture cellulaire. La même équipe a ensuite déterminé trois génotypes différents A, B, et C de HCoV-HKU1, le génotype C étant un recombinant des génotypes A et B [10], [76]. Là encore, ce virus n’est pas responsable d’une maladie nouvelle, seule sa connaissance est émergente [77], [78], [79]. En 2006 et 2007, un certain nombre d’études a été publié sur la circulation des quatre HCoV hors SARS-CoV. Ces études sont peu comparables dans la mesure où un grand nombre de paramètres diffèrent d’une étude à l’autre : région, saison, population cible (enfants, adultes, hospitalisés ou non, terrain immunitaire), et méthodes de détection moléculaire utilisées. Elles ont néanmoins confirmé le caractère épidémique des infections à coronavirus, à la jonction hiver–printemps (pic en février). Les quatre HCoVs co-circulent, avec cependant des variations dans la distribution des différentes espèces selon la géographie et les années. Le caractère annuel régulier des épidémies reste à étudier. Les coronavirus sont en général placés en quatrième ou cinquième position dans la détection des virus respiratoires, derrière les rhinovirus, le VRS, l’hMPV et les virus influenza ; avec une fréquence équivalente à celle des virus parainfluenza (Fig. 5 ). Le taux de détection moléculaire dans les prélèvements va de 3 à 11 % [80], [81], [82], [83], [84], [85], [86].
Fig. 5.
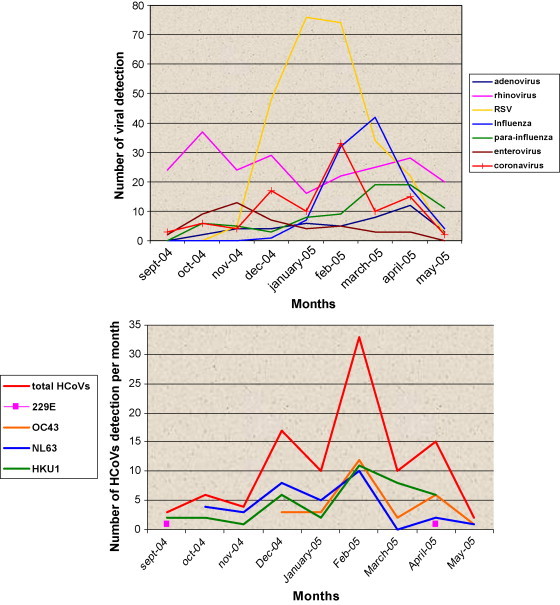
(A) : nombre de virus détectés dans les échantillons respiratoires prélevés de septembre 204 à mai 2005 en Basse-Normandie. (B) : répartition des différents coronavirus humains (229E, OC43, NL63, et HKU1) trouvés dans ces échantillons respiratoires [86].
7. Pathologies liées aux coronavirus humains
Les HCoVs sont essentiellement étudiés dans le cadre d’infections respiratoires. Leur implication précise dans des pathologies digestives et neurologiques chez l’homme est encore controversée. Les HCoV sont détectables dans les selles. Ils ont été mis en cause dans les entérocolites nécrosantes du nourrisson dans les années 1980, et dans les diarrhées aiguës et chroniques, suite à l’observation en microscopie électronique de particules virales dans les selles : ces particules à la morphologie caractéristique ont été alors désignées sous le nom de human enteric coronavirus (HEVC) ou coronavirus-like particles (CVLPs) [87], [88]. L’ARN des HCoV est détectable dans les selles de patients présentant des infections respiratoires à HCoV accompagnées de signes digestifs [78]. S’agit-il seulement d’une excrétion digestive ou de la détection d’un agent responsable des symptômes entériques ? Il faut noter qu’un certain nombre de coronavirus animaux sont responsables d’authentiques pathologies entériques graves, avec une mortalité importante, notamment chez les nouveau-nés. Concernant le système nerveux central, les HCoV font partie de la longue liste d’agents potentiellement responsables de pathologies démyélinisantes, notamment de la sclérose en plaque. Certaines études ont montré le caractère neuroinvasif des HCoV classiques et suggèrent l’existence d’infections persistantes dans le tissu cérébral [89], [90]. Rappelons que le paradigme de la SEP est l’encéphalomyélite induite par l’infection expérimentale de souris par certaines souches neurotropes de coronavirus murins (MHV).
Les HCoV sont essentiellement responsables d’infections respiratoires. Comme de nombreux virus, leur responsabilité n’est pas toujours prouvée par le contrôle des critères du postulat de Koch adapté aux virus par Rivers en 1937 (ensemble de six critères établissant un virus comme cause de la maladie). Cependant, certaines preuves directes ou indirectes ont pu être apportées pour les coronavirus classiques et les infections respiratoires hautes (inoculation à des volontaires sains), pour HCoV-HKU1 et les infections respiratoires basses (séroconversion), et pour le SARS-CoV (inoculation expérimentale à l’animal) [10], [91], [92]. Les études récentes montrent que les patients infectés par un HCoV hors SARS-CoV peuvent présenter une infection respiratoire haute (rhinite, laryngite, otite) ou basse (bronchite, bronchiolite, ou pneumopathies) [71], [72], [73], [74], [77], [80], [81], [82], [84], [85], [86]. Quelques particularités ont été soulignées dans certaines études, comme l’association plus fréquente de l’infection par HCoV-NL63 et la survenue de laryngite [75]. La gravité de ces infections semble surtout déterminée par le terrain du patient. En population générale, les infections à HCoV sont le plus souvent d’évolution bénigne, et ne se distinguent cliniquement pas de l’infection par d’autres virus respiratoires ; en milieu hospitalier, il est à noter qu’un tiers des enfants présentant une infection respiratoire à HCoV est porteur d’un terrain fragilisé (antécédent d’atopie, asthme, immunodépressions diverses, malformations congénitales variées) [80], [82], [86]. Il reste encore des interrogations sur le cadre nosologique précis de l’infection par HCoV-HKU1 : absence d’association avec la survenue de signes de bronchiolite chez l’enfant, signalement de symptômes neurologiques (convulsions), et détection dans les voies respiratoires et les selles chez des sujets asymptomatiques immunodéprimés (infection persistante ?) [83], [85], [86].
Le coronavirus humain le plus pathogène est le SARS-CoV. Au cours de cette infection, après une incubation silencieuse de deux à dix jours, le tableau clinique débute par un syndrome pseudogrippal banal associant de la fièvre et des signes généraux à type de frissons, courbatures, asthénie, et anorexie. Après quatre à six jours, la période d’état s’installe et est marquée par l’apparition de signes respiratoires (toux, dyspnée) accompagnés parfois de signes pharyngés. Les manifestations digestives sont présentes dans environ 30 % des cas (pourcentage variable selon les séries publiées) et associent diarrhées, vomissements, et douleurs abdominales. L’examen et la radiographie pulmonaire révèlent une pneumopathie soit focalisée soit diffuse. Chez un patient sur cinq environ, l’évolution est défavorable et est marquée par l’apparition d’un syndrome de détresse respiratoire grave nécessitant une ventilation assistée. Cette aggravation de la symptomatologie se fait alors qu’une réponse immunitaire s’est mise en place, suggérant un mécanisme immunopathologique. De nombreux patients atteints de SRAS ont reçu divers protocoles incluant ribavirine et corticoïdes. L’issue se fait soit vers la mort par défaillance multiviscérale, soit vers la guérison. Chez certains patients, des signes de fibrose pulmonaire séquellaires visible au scanner ont été décrites après plusieurs mois d’évolution. Il est à noter que des cas d’infections peu symptomatiques par le SARS-CoV ont été décrites [93], [94].
8. Détection des coronavirus humains
La détection des HCoV est réalisée dans un nombre restreint de laboratoires de virologie, et uniquement sur des prélèvements respiratoires. Les techniques moléculaires sont les méthodes de choix pour la détection des HCoV, virus difficilement cultivables, et pour lesquels il n’existe pas d’anticorps spécifiques validés pour une utilisation à visée diagnostique. La détection de l’ARN des HCoV est réalisée par RT-PCR, suivie soit d’une PCR nichée, soit d’une hybridation moléculaire confirmant la spécificité du produit amplifié. Actuellement, comme pour tous les virus, ces techniques ont tendance à être remplacées par des PCR en temps réel, qui présentent l’avantage de réaliser une amplification spécifique en une seule réaction et en tube fermé. Il existe de nombreuses techniques originales publiées pour chaque espèce de HCoV depuis de nombreuses années. Cependant, l’identification de nouveaux coronavirus (HCoV NL63 et HKU1) et la mise en évidence pour certains de différents génotypes rendent de plus en plus complexe la détection de ces virus. La disponibilité de séquences de plus en plus nombreuses et d’origine géographique diverse permet d’apprécier la diversité génétique de ce groupe de virus, et d’affiner le choix des amorces d’amplification. L’objectif principal d’un outil de détection des HCoV est de disposer d’une technique fiable, sensible, et dont le coût et la faisabilité reste raisonnable. Deux principales stratégies sont développées. La première consiste à développer des techniques « consensus », contenant des amorces choisies dans la région du génome la plus conservée au sein des groupes 1 et 2 des coronavirus, soit le premier cadre de lecture (ORF1a/b). Ces amorces contiennent un nombre variable de positions dégénérées, de façon à amplifier en théorie les 4, voire les 5 HCoV. L’autre stratégie est le développement de techniques dites multiplex, utilisant simultanément, dans le même mélange réactionnel plusieurs couples d’amorces spécifiques des différents HCoV. Les régions du génome choisies sont en général les gènes de structure N ou M. Cette stratégie reste encore difficile à appliquer aux techniques temps réel. Elle semble néanmoins plus sensible que les techniques consensus [86], [95]. Concernant le SARS-CoV, il existe deux techniques commerciales de PCR temps réel (RealArt HPA Coronavirus LC kit, Artus, et Light Cycler SARS-CoV quantification kit Roche), ces techniques ont été développées à partir des souches circulant en 2003, elles devront être réévaluées en cas de résurgence de ce virus.
La détection des HCoV hors SARS-CoV sur d’autres prélèvements tels que les selles est possible, mais uniquement dans le cadre d’études visant à clarifier leur rôle dans la survenue de pathologies digestives. Rappelons que les selles ont constitué le meilleur prélèvement pour le diagnostic virologique du SRAS [57]. La détection des HCoV hors SARS-CoV est utile notamment pour un diagnostic virologique précis des infections respiratoires dans le cadre hospitalier, regroupant la majorité des formes sévères, elle reste cependant lourde à mettre en place, et ne constitue pas toujours une priorité, étant donné le nombre grandissant de virus identifiés dont l’étude est importante à développer.
9. Conclusion
Les coronavirus ont bénéficié d’une large couverture médiatique secondaire à l’épidémie de SRAS de 2002 à 2003. De nombreux laboratoires ont alors consacré leur recherche à ce groupe de virus. Les progrès réalisés sur la connaissance de ces virus depuis cinq ans sont très significatifs, et permettent de mieux appréhender leur complexité et leur potentiel évolutif, notamment via le franchissement de barrière d’espèces. Ces connaissances sont importantes pour l’ensemble du monde viral qui comprend la quasi-totalité des malades infectieuses émergentes ; elles permettent aussi d’élargir les collaborations entre différents professionnels, eux-mêmes enfermés dans les différentes « niches écologiques » : chercheurs, virologues médicaux et vétérinaires, cliniciens, zoologistes…
Références
- 1.Huang Y., Lau S.K., Woo P.C., Yuen K.Y. CoVDB: a comprehensive database for comparative analysis of coronavirus genes and genomes. Nucleic Acids Res. 2007:1362–4962. doi: 10.1093/nar/gkm754. Electronic. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Almeida J.D., Tyrrell D.A. The morphology of three previously uncharacterized human respiratory viruses that grow in organ culture. J Gen Virol. 1967;1(2):175–178. doi: 10.1099/0022-1317-1-2-175. [DOI] [PubMed] [Google Scholar]
- 3.Hamre D., Procknow J.J. A new virus isolated from the human respiratory tract. Proc Soc Exp Biol Med. 1966;121(1):190–193. doi: 10.3181/00379727-121-30734. [DOI] [PubMed] [Google Scholar]
- 4.McIntosh K., Dees J.H., Becker W.B., Kapikian A.Z., Chanock R.M. Recovery in tracheal organ cultures of novel viruses from patients with respiratory disease. Proc Natl Acad Sci USA. 1967;57(4):933–940. doi: 10.1073/pnas.57.4.933. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.Cavanagh D. Nidovirales: a new order comprising Coronaviridae and Arteriviridae. Arch Virol. 1997;142(3):629–633. [PubMed] [Google Scholar]
- 6.Gonzalez J.M., Gomez-Puertas P., Cavanagh D., Gorbalenya A.E., Enjuanes L. A comparative sequence analysis to revise the current taxonomy of the family Coronaviridae. Arch Virol. 2003;148(11):2207–2235. doi: 10.1007/s00705-003-0162-1. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Drosten C., Gunther S., Preiser W., van der Werf S., Brodt H.R., Becker S. Identification of a novel coronavirus in patients with severe acute respiratory syndrome. N Engl J Med. 2003;348(20):1967–1976. doi: 10.1056/NEJMoa030747. [DOI] [PubMed] [Google Scholar]
- 8.Rota P.A., Oberste M.S., Monroe S.S., Nix W.A., Campagnoli R., Icenogle J.P. Characterization of a novel coronavirus associated with severe acute respiratory syndrome. Science. 2003;300(5624):1394–1399. doi: 10.1126/science.1085952. [DOI] [PubMed] [Google Scholar]
- 9.van der Hoek L., Pyrc K., Jebbink M.F., Vermeulen-Oost W., Berkhout R.J., Wolthers K.C. Identification of a new human coronavirus. Nat Med. 2004;10(4):368–373. doi: 10.1038/nm1024. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Woo P.C., Lau S.K., Chu C.M., Chan K.H., Tsoi H.W., Huang Y. Characterization and complete genome sequence of a novel coronavirus, coronavirus HKU1, from patients with pneumonia. J Virol. 2005;79(2):884–895. doi: 10.1128/JVI.79.2.884-895.2005. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Wentworth DE HK. Coronavirus binding and entry. In: V. T, ed. Coronaviruses Molecular and cellular biology. Norfolk, UK: Caister Academic Press, 2007:3–31.
- 12.Hofmann H., Pyrc K., van der Hoek L., Geier M., Berkhout B., Pohlmann S. Human coronavirus NL63 employs the severe acute respiratory syndrome coronavirus receptor for cellular entry. Proc Natl Acad Sci USA. 2005;102(22):7988–7993. doi: 10.1073/pnas.0409465102. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Li W., Moore M.J., Vasilieva N., Sui J., Wong S.K., Berne M.A. Angiotensin-converting enzyme 2 is a functional receptor for the SARS coronavirus. Nature. 2003;426(6965):450–454. doi: 10.1038/nature02145. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Yeager C.L., Ashmun R.A., Williams R.K., Cardellichio C.B., Shapiro L.H., Look A.T. Human aminopeptidase N is a receptor for human coronavirus 229E. Nature. 1992;357(6377):420–422. doi: 10.1038/357420a0. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Gallagher T.M., Buchmeier M.J. Coronavirus spike proteins in viral entry and pathogenesis. Virology. 2001;279(2):371–374. doi: 10.1006/viro.2000.0757. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Kubo H., Yamada Y.K., Taguchi F. Localization of neutralizing epitopes and the receptor-binding site within the amino-terminal 330 amino acids of the murine coronavirus spike protein. J Virol. 1994;68(9):5403–5410. doi: 10.1128/jvi.68.9.5403-5410.1994. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Wong S.K., Li W., Moore M.J., Choe H., Farzan M. A 193-amino acid fragment of the SARS coronavirus S protein efficiently binds angiotensin-converting enzyme 2. J Biol Chem. 2004;279(5):3197–3201. doi: 10.1074/jbc.C300520200. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Li W., Sui J., Huang I.C., Kuhn J.H., Radoshitzky S.R., Marasco W.A. The S proteins of human coronavirus NL63 and severe acute respiratory syndrome coronavirus bind overlapping regions of ACE2. Virology. 2007;367(2):367–374. doi: 10.1016/j.virol.2007.04.035. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Brian DA KT. The Coronavirus Hemagglutinin-esterase Glycoprotein. In: S S, ed. The Coronaviridae. New-York: Plenum Press, 1995.
- 20.Kazi L., Lissenberg A., Watson R., de Groot R.J., Weiss S.R. Expression of hemagglutinin esterase protein from recombinant mouse hepatitis virus enhances neurovirulence. J Virol. 2005;79(24):15064–15073. doi: 10.1128/JVI.79.24.15064-15073.2005. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.Kienzle T.E., Abraham S., Hogue B.G., Brian D.A. Structure and expression of the bovine coronavirus hemagglutinin protein. Adv Exp Med Biol. 1990;276:95–102. doi: 10.1007/978-1-4684-5823-7_14. [DOI] [PubMed] [Google Scholar]
- 22.Lissenberg A., Vrolijk M.M., van Vliet A.L., Langereis M.A., de Groot-Mijnes J.D., Rottier P.J. Luxury at a cost? Recombinant mouse hepatitis viruses expressing the accessory hemagglutinin esterase protein display reduced fitness in vitro. J Virol. 2005;79(24):15054–15063. doi: 10.1128/JVI.79.24.15054-15063.2005. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.Yokomori K., Banner L.R., Lai M.M. Heterogeneity of gene expression of the hemagglutinin-esterase (HE) protein of murine coronaviruses. Virology. 1991;183(2):647–657. doi: 10.1016/0042-6822(91)90994-M. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.Vlasak R., Luytjes W., Spaan W., Palese P. Human and bovine coronaviruses recognize sialic acid-containing receptors similar to those of influenza C viruses. Proc Natl Acad Sci USA. 1988;85(12):4526–4529. doi: 10.1073/pnas.85.12.4526. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.Zhang X.M., Kousoulas K.G., Storz J. The hemagglutinin/esterase gene of human coronavirus strain OC43: phylogenetic relationships to bovine and murine coronaviruses and influenza C virus. Virology. 1992;186(1):318–323. doi: 10.1016/0042-6822(92)90089-8. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.Krempl C., Schultze B., Herrler G. Analysis of cellular receptors for human coronavirus OC43. Adv Exp Med Biol. 1995;380:371–374. doi: 10.1007/978-1-4615-1899-0_60. [DOI] [PubMed] [Google Scholar]
- 27.Schwegmann-Wessels C., Herrler G. Sialic acids as receptor determinants for coronaviruses. Glycoconj J. 2006;23(1–2):51–58. doi: 10.1007/s10719-006-5437-9. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 28.Enjuanes LSI, Zuniga S, Moreno JL. Coronavirus RNA synthesis: transcription. In: V T, ed. Coronaviruses Molecular and Cellular Biology. Norkolk, UK: Caister Academic Press 2007;81–107.
- 29.Lai M.M., Cavanagh D. The molecular biology of coronaviruses. Adv Virus Res. 1997;48:1–100. doi: 10.1016/S0065-3527(08)60286-9. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 30.Ziebuhr J SE. The Coronavirus replicase gene: special enzymes for special viruses. In: V T, ed. Coronaviruses Molecular and Cellular Biology. Norfolk, UK: Caister Academic Press, 2007:33–63.
- 31.Adami C., Pooley J., Glomb J., Stecker E., Fazal F., Fleming J.O. Evolution of mouse hepatitis virus (MHV) during chronic infection: quasispecies nature of the persisting MHV RNA. Virology. 1995;209(2):337–346. doi: 10.1006/viro.1995.1265. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 32.Jackwood M.W., Hilt D.A., Callison S.A. Detection of infectious bronchitis virus by real-time reverse transcriptase-polymerase chain reaction and identification of a quasispecies in the Beaudette strain. Avian Dis. 2003;47(3):718–724. doi: 10.1637/6075. [DOI] [PubMed] [Google Scholar]
- 33.Kiss I., Kecskemeti S., Tanyi J., Klingeborn B., Belak S. Preliminary studies on feline coronavirus distribution in naturally and experimentally infected cats. Res Vet Sci. 2000;68(3):237–242. doi: 10.1053/rvsc.1999.0368. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 34.Rowe C.L., Baker S.C., Nathan M.J., Sgro J.Y., Palmenberg A.C., Fleming J.O. Quasispecies development by high frequency RNA recombination during MHV persistence. Adv Exp Med Biol. 1998;440:759–765. doi: 10.1007/978-1-4615-5331-1_98. [DOI] [PubMed] [Google Scholar]
- 35.Vabret A., Dina J., Mourez T., Gouarin S., Petitjean J., van der Werf S. Inter- and intra-variant genetic heterogeneity of human coronavirus OC43 strains in France. J Gen Virol. 2006;87(Pt 11):3349–3353. doi: 10.1099/vir.0.82065-0. [DOI] [PubMed] [Google Scholar]
- 36.Laude H.D.B., Eleouet J.F. Le coronavirus porcin PRCV: un virus émergent pas comme les autres. Virologie. 1998;2:305–316. [Google Scholar]
- 37.Herrewegh A.A., Smeenk I., Horzinek M.C., Rottier P.J., de Groot R.J. Feline coronavirus type II strains 79-1683 and 79-1146 originate from a double recombination between feline coronavirus type I and canine coronavirus. J Virol. 1998;72(5):4508–4514. doi: 10.1128/jvi.72.5.4508-4514.1998. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 38.Vijgen L., Keyaerts E., Lemey P., Maes P., Van Reeth K., Nauwynck H. Evolutionary history of the closely related group 2 coronaviruses: porcine hemagglutinating encephalomyelitis virus, bovine coronavirus, and human coronavirus OC43. J Virol. 2006;80(14):7270–7274. doi: 10.1128/JVI.02675-05. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 39.Vijgen L., Keyaerts E., Lemey P., Moes E., Li S., Vandamme A.M. Circulation of genetically distinct contemporary human coronavirus OC43 strains. Virology. 2005;337(1):85–92. doi: 10.1016/j.virol.2005.04.010. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 40.Vijgen L., Keyaerts E., Moes E., Thoelen I., Wollants E., Lemey P. Complete genomic sequence of human coronavirus OC43: molecular clock analysis suggests a relatively recent zoonotic coronavirus transmission event. J Virol. 2005;79(3):1595–1604. doi: 10.1128/JVI.79.3.1595-1604.2005. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 41.Decaro N., Martella V., Elia G., Campolo M., Maril V., Desario C. Biological and genetic analysis of a bovine-like coronavirus isolated from water buffalo (Bubalus bubalis) calves. Virology. 2008;370(1):213–222. doi: 10.1016/j.virol.2007.08.031. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 42.Erles K., Toomey C., Brooks H.W., Brownlie J. Detection of a group 2 coronavirus in dogs with canine infectious respiratory disease. Virology. 2003;310(2):216–223. doi: 10.1016/S0042-6822(03)00160-0. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 43.Guy J.S., Breslin J.J., Breuhaus B., Vivrette S., Smith L.G. Characterization of a coronavirus isolated from a diarrheic foal. J Clin Microbiol. 2000;38(12):4523–4526. doi: 10.1128/jcm.38.12.4523-4526.2000. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 44.Hasoksuz M., Alekseev K., Vlasova A., Zhang X., Spiro D., Halpin R. Biologic, antigenic, and full-length genomic characterization of a bovine-like coronavirus isolated from a giraffe. J Virol. 2007;81(10):4981–4990. doi: 10.1128/JVI.02361-06. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 45.Jin L., Cebra C.K., Baker R.J., Mattson D.E., Cohen S.A., Alvarado D.E., Rohrmann G.F. Analysis of the genome sequence of an alpaca coronavirus. Virology. 2007;365(1):198–203. doi: 10.1016/j.virol.2007.03.035. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 46.Xu H.F., Wang M., Zhang Z.B., Zou X.Z., Gao Y., Liu X.N. [An epidemiologic investigation on infection with severe acute respiratory syndrome coronavirus in wild animals traders in Guangzhou] Zhonghua Yu Fang Yi Xue Za Zhi. 2004;38(2):81–83. [PubMed] [Google Scholar]
- 47.Guan Y., Zheng B.J., He Y.Q., Liu X.L., Zhuang Z.X., Cheung C.L. Isolation and characterization of viruses related to the SARS coronavirus from animals in southern China. Science. 2003;302(5643):276–278. doi: 10.1126/science.1087139. [DOI] [PubMed] [Google Scholar]
- 48.Shi Z., Hu Z. A review of studies on animal reservoirs of the SARS coronavirus. Virus Res. 2007 doi: 10.1016/j.virusres.2007.03.012. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 49.Kan B., Wang M., Jing H., Xu H., Jiang X., Yan M. Molecular evolution analysis and geographic investigation of severe acute respiratory syndrome coronavirus-like virus in palm civets at an animal market and on farms. J Virol. 2005;79(18):11892–11900. doi: 10.1128/JVI.79.18.11892-11900.2005. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 50.Hu W., Bai B., Hu Z., Chan Z., An X., Tang L. Development and evaluation of a multitarget real-time Taqman reverse transcription-PCR assay for detection of the severe acute respiratory syndrome-associated coronavirus and surveillance for an apparently related coronavirus found in masked palm civets. J Clin Microbiol. 2005;43(5):2041–2046. doi: 10.1128/JCM.43.5.2041-2046.2005. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 51.Song H.D., Tu C.C., Zhang G.W., Wang S.Y., Zheng K., Lei J.C. Cross-host evolution of severe acute respiratory syndrome coronavirus in palm civet and human. Proc Natl Acad Sci USA. 2005;102(7):2430–2435. doi: 10.1073/pnas.0409608102. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 52.Chinese S.A.R.S., Molecular Epidemiology, Consortium Molecular evolution of the SARS coronavirus during the course of the SARS epidemic in China. Science. 2004;333:1666–1669. doi: 10.1126/science.1092002. [DOI] [PubMed] [Google Scholar]
- 53.Li F., Li W., Farzan M., Harrison S.C. Structure of SARS coronavirus spike receptor-binding domain complexed with receptor. Science. 2005;309(5742):1864–1868. doi: 10.1126/science.1116480. [DOI] [PubMed] [Google Scholar]
- 54.Lau S.K., Woo P.C., Li K.S., Huang Y., Tsoi H.V., Wong B.H. Severe acute respiratory syndrome coronavirus-like virus in Chinese horseshoe bats. Proc Natl Acad Sci USA. 2005;102(39):14040–14045. doi: 10.1073/pnas.0506735102. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 55.Li W., Shi Z., Yu M., Ren W., Smith C., Epstein J.H. Bats are natural reservoirs of SARS-like coronaviruses. Science. 2005;310(5748):676–679. doi: 10.1126/science.1118391. [DOI] [PubMed] [Google Scholar]
- 56.van Elden L.J., van Loon A.M., van Alphen F., Hendriksen K.A., Hoepelman A.I., van Kraaij M.G. Frequent detection of human coronaviruses in clinical specimens from patients with respiratory tract infection by use of a novel real-time reverse-transcriptase polymerase chain reaction. J Infect Dis. 2004;189(4):652–657. doi: 10.1086/381207. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 57.Chan K.H., Poon L.L., Cheng V.C., Guan Y., Hung I.F., Kong J. Detection of SARS coronavirus in patients with suspected SARS. Emerg Infect Dis. 2004;10(2):294–299. doi: 10.3201/eid1002.030610. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 58.Seto W.H., Tsang D., Yung R.W., Ching T.Y., Ng T.K., Ho M. Effectiveness of precautions against droplets and contact in prevention of nosocomial transmission of severe acute respiratory syndrome (SARS) Lancet. 2003;361(9368):1519–1520. doi: 10.1016/S0140-6736(03)13168-6. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 59.Rabenau H.F., Kampf G., Cinatl J., Doerr H.W. Efficacy of various disinfectants against SARS coronavirus. J Hosp Infect. 2005;61(2):107–111. doi: 10.1016/j.jhin.2004.12.023. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 60.Shen Z., Ning F., Zhou W., He X., Lin C., Chin D.P. Superspreading SARS events, Beijing, 2003. Emerg Infect Dis. 2004;10(2):256–260. doi: 10.3201/eid1002.030732. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 61.Radun D., Niedrig M., Ammon A., Stark K. SARS: retrospective cohort study among German guests of the Hotel ‘M’, Hong Kong. Euro Surveill. 2003;8(12):228–230. doi: 10.2807/esm.08.12.00436-en. [DOI] [PubMed] [Google Scholar]
- 62.Desenclos J.C., van der Werf S., Bonmarin I., Levy-Buhl D., Yazdanpanah Y., Hoen B. Introduction of SARS in France, March-April, 2003. Emerg Infect Dis. 2004;10(2):195–200. doi: 10.3201/eid1002.030351. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 63.Hambre D., Beem M. Virologic studies of acute respiratory disease in young adults. V. Coronavirus 229E infections during six years of surveillance. Am J Epidemiol. 1972;96(2):94–106. doi: 10.1093/oxfordjournals.aje.a121445. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 64.Kaye H.S., Dowdle W.R. Seroepidemiologic survey of coronavirus (strain 229E) infections in a population of children. Am J Epidemiol. 1975;101(3):238–244. doi: 10.1093/oxfordjournals.aje.a112091. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 65.Kaye H.S., Marsh H.B., Dowdle W.R. Seroepidemiologic survey of coronavirus (strain OC 43) related infections in a children's population. Am J Epidemiol. 1971;94(1):43–49. doi: 10.1093/oxfordjournals.aje.a121293. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 66.Monto A.S., Lim S.K. The Tecumseh study of respiratory illness. VI. Frequency of and relationship between outbreaks of coronavirus infection. J Infect Dis. 1974;129(3):271–276. doi: 10.1093/infdis/129.3.271. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 67.Vabret A., Mourez T., Dina J., Freymuth F. Coronavirus humains. Virologie. 2005;9:273–287. doi: 10.1684/vir.2011.2118. [DOI] [PubMed] [Google Scholar]
- 68.Esper F., Weibel C., Ferguson D., Landry M.L., Kahn J.S. Evidence of a novel human coronavirus that is associated with respiratory tract disease in infants and young children. J Infect Dis. 2005;191(4):492–498. doi: 10.1086/428138. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 69.Fouchier R.A., Hartwig N.G., Bestebroer T.M., Niemeyer B., de Jong J.C., Simon J.H. A previously undescribed coronavirus associated with respiratory disease in humans. Proc Natl Acad Sci USA. 2004;101(16):6212–6216. doi: 10.1073/pnas.0400762101. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 70.Arden K.E., Nissen M.D., Sloots T.P., Mackay I.M. New human coronavirus, HCoV-NL63, associated with severe lower respiratory tract disease in Australia. J Med Virol. 2005;75(3):455–462. doi: 10.1002/jmv.20288. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 71.Bastien N., Robinson J.L., Tse A., Lee B.E., Hart L., Li Y. Human coronavirus NL-63 infections in children: a 1-year study. J Clin Microbiol. 2005;43(9):4567–4573. doi: 10.1128/JCM.43.9.4567-4573.2005. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 72.Chiu S.S., Chan K.H., Chu K.W., Kwan S.W., Guan Y., Poon L.L. Human coronavirus NL63 infection and other coronavirus infections in children hospitalized with acute respiratory disease in Hong Kong, China. Clin Infect Dis. 2005;40(12):1721–1729. doi: 10.1086/430301. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 73.Suzuki A., Okamoto M., Ohmi A., Watanabe O., Miyabayashi S., Nishimura H. Detection of human coronavirus-NL63 in children in Japan. Pediatr Infect Dis J. 2005;24(7):645–646. doi: 10.1097/01.inf.0000168846.71517.ee. [DOI] [PubMed] [Google Scholar]
- 74.Vabret A., Mourez T., Dina J., van der Hoek L., Gouarin S., Petitjean J. Human coronavirus NL63, France. Emerg Infect Dis. 2005;11(8):1225–1229. doi: 10.3201/eid1108.050110. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 75.van der Hoek L., Sure K., Ihorst G., Stang A., Pyrc K., Jebbink M.F. Croup is associated with the novel coronavirus NL63. PLoS Med. 2005;2(8):e240. doi: 10.1371/journal.pmed.0020240. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 76.Woo P.C., Wang M., Lau S.K., Xu H., Poon R.W., Guo R. Comparative analysis of twelve genomes of three novel group 2c and group 2d coronaviruses reveals unique group and subgroup features. J Virol. 2007;81(4):1574–1585. doi: 10.1128/JVI.02182-06. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 77.Sloots T.P., McErlean P., Speicher D.J., Arden K.E., Nissen M.D., Mackay I.M. Evidence of human coronavirus HKU1 and human bocavirus in Australian children. J Clin Virol. 2006;35(1):99–102. doi: 10.1016/j.jcv.2005.09.008. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 78.Vabret A., Dina J., Gouarin S., Petitjean J., Corbet S., Freymuth F. Detection of the new human coronavirus HKU1: a report of 6 cases. Clin Infect Dis. 2006;42(5):634–639. doi: 10.1086/500136. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 79.Woo P.C., Lau S.K., Tsoi H.W., Huang Y., Poon R.W., Chu C.M. Clinical and molecular epidemiological features of coronavirus HKU1-associated community-acquired pneumonia. J Infect Dis. 2005;192(11):1898–1907. doi: 10.1086/497151. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 80.Esposito S., Bosis S., Niesters H.G., Tremolati E., Begliatti E., Rognoni A. Impact of human coronavirus infections in otherwise healthy children who attended an emergency department. J Med Virol. 2006;78(12):1609–1615. doi: 10.1002/jmv.20745. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 81.Garbino J., Crespo S., Aubert J.D., Rochat T., Ninet B., Deffernez C. A prospective hospital-based study of the clinical impact of non-severe acute respiratory syndrome (Non-SARS)-related human coronavirus infection. Clin Infect Dis. 2006;43(8):1009–1015. doi: 10.1086/507898. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 82.Gerna G., Campanini G., Rovida F., Percivalle E., Sarasini A., Marchi A. Genetic variability of human coronavirus OC43-, 229E-, and NL63-like strains and their association with lower respiratory tract infections of hospitalized infants and immunocompromised patients. J Med Virol. 2006;78(7):938–949. doi: 10.1002/jmv.20645. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 83.Gerna G., Percivalle E., Sarasini A., Campanini G., Piralla A., Rovida F. Human respiratory coronavirus HKU1 versus other coronavirus infections in Italian hospitalised patients. J Clin Virol. 2007;38(3):244–250. doi: 10.1016/j.jcv.2006.12.008. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 84.Kuypers J., Martin E.T., Heugel J., Wright N., Morrow R., Englund J.A. Clinical disease in children associated with newly described coronavirus subtypes. Pediatrics. 2007;119(1):e70–e76. doi: 10.1542/peds.2006-1406. [DOI] [PubMed] [Google Scholar]
- 85.Lau S.K., Woo P.C., Yip C.C., Tse H., Tsoi H.W., Cheng V.C. Coronavirus HKU1 and other coronavirus infections in Hong Kong. J Clin Microbiol. 2006;44(6):2063–2071. doi: 10.1128/JCM.02614-05. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 86.Vabret A., Dina J., Gouarin S., Petitjean J., Tripey V., Brouard J. Human (non-severe acute respiratory syndrome) coronavirus infections in hospitalised children in France. J Paediatr Child Health. 2007 doi: 10.1111/j.1440-1754.2007.01246.x. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 87.Chany C., Moscovici O., Lebon P., Rousset S. Association of coronavirus infection with neonatal necrotizing enterocolitis. Pediatrics. 1982;69(2):209–214. [PubMed] [Google Scholar]
- 88.Rousset S., Moscovici O., Lebon P., Barbet J.P., Helardot P., Mace B. Intestinal lesions containing coronavirus-like particles in neonatal necrotizing enterocolitis: an ultrastructural analysis. Pediatrics. 1984;73(2):218–224. [PubMed] [Google Scholar]
- 89.Arbour N., Day R., Newcombe J., Talbot P.J. Neuroinvasion by human respiratory coronaviruses. J Virol. 2000;74(19):8913–8921. doi: 10.1128/jvi.74.19.8913-8921.2000. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 90.Arbour N., Talbot P.J. Persistent infection of neural cell lines by human coronaviruses. Adv Exp Med Biol. 1998;440:575–581. doi: 10.1007/978-1-4615-5331-1_75. [DOI] [PubMed] [Google Scholar]
- 91.Bradburne A.F., Bynoe M.L., Tyrrell D.A. Effects of a “new” human respiratory virus in volunteers. Br Med J. 1967;3(5568):767–769. doi: 10.1136/bmj.3.5568.767. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 92.Fouchier R.A., Kuiken T., Schutten M., van Amerongen G., van Doonum G.J., van den Hoogen B.G. Aetiology: Koch's postulates fulfilled for SARS virus. Nature. 2003;423(6937):240. doi: 10.1038/423240a. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 93.Lee N., Hui D., Wu A., Chan P., Cameron P., Joynt G.M. A major outbreak of severe acute respiratory syndrome in Hong Kong. N Engl J Med. 2003;348(20):1986–1994. doi: 10.1056/NEJMoa030685. [DOI] [PubMed] [Google Scholar]
- 94.Peiris J.S., Chu C.M., Cheng V.C., Chan K.S., Hung I.F., Poon L.L. Clinical progression and viral load in a community outbreak of coronavirus-associated SARS pneumonia: a prospective study. Lancet. 2003;361(9371):1767–1772. doi: 10.1016/S0140-6736(03)13412-5. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 95.Moes E., Vijgen L., Keyaerts E., Zlateva K., Li S., Maes P. A novel pancoronavirus RT-PCR assay: frequent detection of human coronavirus NL63 in children hospitalized with respiratory tract infections in Belgium. BMC Infect Dis. 2005;5(1):6. doi: 10.1186/1471-2334-5-6. [DOI] [PMC free article] [PubMed] [Google Scholar]


