Résumé
La grippe saisonnière est due aux virus influenza A et B. Il s’agit de virus enveloppés dont le génome est constitué de sept à huit fragments d’ARN. Les différents sous-types sont déterminés par la nature des deux glycoprotéines de surface HA et NA. La grippe saisonnière est une maladie épidémique et hivernale dans les zones à climat tempéré. Son épidémiologie est liée à la grande variabilité du virus au cours du temps, nécessitant la mise en place d’un système d’alerte détectant chaque année les variants circulants dominant et déterminant la composition vaccinale. Les symptômes cliniques de la grippe ne sont pas suffisamment spécifiques pour permettre le diagnostic sans examen virologique. Cela est particulièrement vrai en période non épidémique, chez les sujets de plus de 65 ans et chez les enfants de moins de cinq ans. L’enfant représente une cible privilégiée des infections à virus influenza. Le recours à l’hospitalisation est d’autant plus élevé que l’enfant est jeune. Chez le nourrisson, l’infection peut être paucisymptomatique et s’accompagner de manifestations non respiratoires (léthargie, convulsions, malaises). Le diagnostic virologique de la grippe est justifié chez tous les sujets hospitalisés pour un syndrome respiratoire compatible avec une infection à virus influenza. Il existe plusieurs outils permettant une recherche directe du virus dans les sécrétions respiratoires : isolement du virus en culture, détection d’antigènes, détection moléculaire de l’ARN. Le choix de la méthode se fait selon les caractéristiques du test : sensibilité, spécificité, rapidité et simplicité de réalisation, coût.
Mots clés: Virus influenza, Hémagglutinine, Neuraminidase, Variabilité, Épidémie, Infection respiratoire, Immunofluorescence, Tests rapides, RT-PCR
Abstract
Seasonal flu is caused by influenza viruses A and B. These enveloped viruses have a genome made up of seven or eight RNA fragments. The different subtypes are determined by the nature of the two surface glycoproteins HA and NA. Seasonal flu is an epidemic wintertime illness occurring in temperate climate zones. Its epidemiology is linked to the great variability of the virus in time, necessitating an alert system that detects dominating circulating variants each year and that determines the vaccination composition. Clinical flu symptoms are not sufficiently specific to allow for diagnosis with virological tests. This is especially true during non-epidemic periods as well as in subjects older than 65 and younger than five. Children are especially vulnerable to influenza virus infections. Hospitalization occurs more frequently, the younger the child. In children younger than two years, the infection can be pauci-symptomatic and is sometimes detected from non-respiratory symptoms such as lethargy, convulsions, and dizziness. In all cases of respiratory syndrome compatible with influenza virus infection in hospitalized subjects, virological flu diagnosis is of utmost interest. Several tools are available to allow for direct viral detection in respiratory specimens: cell culture isolation, antigenic detection, RNA molecular detection. Choice of method is based on the characteristics of the test: sensibility, specificity, speed and ease of realization, and cost.
Keywords: Influenza virus, Hemagglutinin, Neuraminidase, Variability, Epidemic, Respiratory infection, Immunofluorescence, Rapid test, RT-PCR
1. Introduction
Les virus influenza A et B sont les agents habituels de la grippe saisonnière. Le virus influenza C est beaucoup plus rare [1]. La grippe est une infection respiratoire fréquente, très contagieuse, qui peut être grave et entraîner l’hospitalisation des patients. Cet article présente les éléments du fil conducteur qui peut conduire à un diagnostic de grippe saisonnière : l’épidémiologie et la transmission de l’infection, la symptomatologie chez l’adulte et l’enfant, et l’identification par le diagnostic virologique. Le traitement de la grippe et les vaccins anti-grippaux ne seront pas traités dans ce manuscrit.
2. Épidémiologie
La grippe se manifeste sous trois formes : sporadique, quelques cas dans la population ; épidémique, avec des cas plus nombreux, s’étendant rapidement et se propageant dans une région ; et pandémique, lorsque l’extension de l’infection est rapide et généralisée à l’ensemble de la planète. La grippe dite « saisonnière » est une infection épidémique, fréquente, en général annuelle et hivernale dans les zones à climat tempéré. En général, on observe d’abord des cas sporadiques qui évoluent vers une circulation épidémique. Ainsi, chez les patients hospitalisés au CHU de Caen, les 1770 virus influenza isolés entre 2000 et 2006 représentent 33 % des 5356 virus respiratoires détectés pendant cette période, presque autant que le virus respiratoire syncytial (1819, soit 33,9 %) et plus que les rhinovirus (1118 soit 20,8 %). La grippe est une maladie franchement hivernale : dans cette étude, 49 % des virus influenza A et B sont isolés en janvier et février, contre 25 % en novembre et décembre, et 26 % en mars et avril (Fig. 1 ). L’aspect épidémique est une caractéristique majeure de la grippe. L’épidémie associe le plus souvent les deux types A et B de virus. Les infections à virus influenza A sont les plus fréquentes : 1525 cas (76,7 %) au cours des huit dernières épidémies au CHU de Caen. Les infections à virus influenza B sont moins fréquentes : 463 cas (23,2 %), et très rarement majoritaires (hiver 2005–2006) ; de plus, elles sont souvent décalées de plusieurs semaines en fin d’épidémie (Fig. 2 ). Les épidémies sont d’importance variable. Le nombre moyen de grippes détectées chaque hiver au CHU de Caen est de 248,5, mais il varie de 175 à 406 cas (×2,3) d’une année à l’autre. La variation est plus nette pour les infections à virus influenza A, qui fluctuent entre 67 et 405 cas par hiver (moyenne : 190,6 cas), tandis que les infections à virus influenza B varient entre 0 et 144 cas (moyenne : 57,8 cas).
Fig. 1.
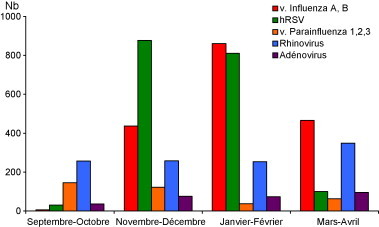
Distribution saisonnière des virus respiratoires. CHU de Caen.
En ordonnées : nombre de virus cumulés sur une période de deux mois chez les patients hospitalisés. En abscisses : septembre et octobre, novembre et décembre, janvier et février, mars et avril, de 2000 à 2006.
Fig. 2.
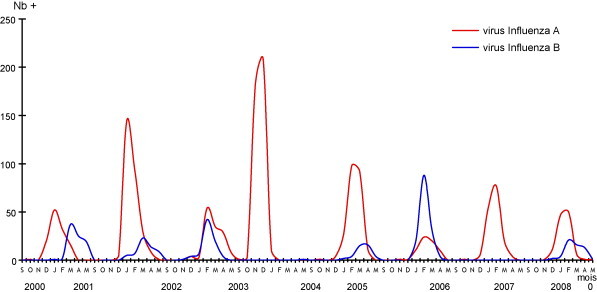
Épidémies à virus influenza. CHU de Caen.
En ordonnées : nombre d’infections à virus influenza A et B chez les patients hospitalisés. En abscisses : épidémies hivernales de 2000 à 2008.
La grippe saisonnière est une affection très contagieuse. La transmission de personne à personne des virus influenza se fait surtout par les aérosols à grosses particules émis lors des éternuements ou de la toux [2]. La contamination nécessite que les individus soient à proximité (moins d’un mètre) les uns des autres, car ces gouttelettes ne restent pas en suspension dans l’air. Elles sédimentent sur les surfaces et les objets, créant ainsi une autre source possible de transmission. La transmission aérienne des virus influenza, par des aérosols de petites particules (< 0,5 mμ) qui resteraient en suspension dans l’air pendant de longues périodes n’est pas vraiment documentée [3], [4]. Les adultes éliminent du virus par voie aérienne, de 24 heures avant le début des symptômes (la durée moyenne d’incubation est de deux jours) jusqu’à cinq à dix jours après le début de la maladie [5]. Cependant, dans des infections humaines expérimentales, on observe que le titre infectieux des sécrétions respiratoires chute notablement dès les trois à cinq premiers jours de la maladie [6], [7]. La transmission du virus grippal de sujet à sujet est donc surtout marquée au tout début de la maladie. Chez les jeunes enfants, la transmission du virus est plus importante car ils éliminent le virus plus tôt et plus longtemps que les adultes [8]. Les patients immunodéprimés peuvent évidemment excréter du virus pendant de longues périodes.
3. Le virus et sa variabilité
Les virus grippaux sont des virus enveloppés dont le génome est constitué d’ARN segmenté en sept ou huit fragments. Ces virus appartiennent à la famille des Orthomyxoviridae, et il en existe trois genres ou types nommés influenzavirus A, B, et C, d’importance décroissante. Pour les virus influenza A, le génome comporte trois segments codant les protéines du complexe de réplication (PB2, PB1, et PA), le quatrième fragment code l’hémagglutinine HA, le cinquième code la nucléoprotéine ou NP, le sixième la neuraminidase ou NA, le septième code les protéines M1 et M2 (protéine de matrice et protéine du canal à ions), et enfin le huitième fragment code les protéines non structurales NS1 et NS2 (Fig. 3 ). Selon la nature des glycoprotéines de surface HA et NA, le type influenza A est divisé en sous-types sérologiques HxNy. Il existe 16 sous-types de HA (H1 à H16) et neuf sous-types de NA (N1 à N9). La nomenclature utilisée pour nommer les souches virales comprend les éléments suivants, toujours dans le même ordre : type / lieu d’isolement / numéro d’enregistrement/ année d’isolement / HxNy. (exemple : A / Brisbane / 59 / 2007 H1N1, la souche vaccinale de la saison 2008/2009). Le réservoir des virus influenza A est aviaire, et c’est chez les oiseaux aquatiques sauvages que l’on observe le plus grand nombre de sous-types de virus de grippe A. Chez l’homme, n’ont circulé que des virus influenza A portant à leur surface les espèces moléculaires H1, H2, H3, et ponctuellement H5, H9, et les neuraminidases N1 et N2 [9]. Même si l’ensemble des protéines structurales et non structurales est indispensable à la réussite de la multiplication du virus dans la cellule, la protéine de surface HA revêt une importance particulière, en jouant un rôle primordial dans les premières phases du cycle de multiplication (entrée du virus dans la cellule), et aussi en portant les principaux sites antigéniques de neutralisation.
Fig. 3.
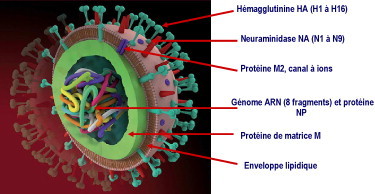
Représentation schématique du virus influenza A. (internet http://farm1.static.flickr.com)
Les huit segments d’ARN génomique sont étroitement liés à la nucléoprotéine NP et codent quatre protéines structurales HA, NA, (M1 et M2), NP et quatre protéines non structurales PB1, PB2, PA, et (NS1et NS2).
Les problèmes les plus complexes de l’épidémiologie de la grippe sont liés à la variabilité du virus influenza. Ces virus varient au cours du temps, de telle sorte que l’organisme n’est pas capable de les reconnaître et donc de se défendre. Ces variations sont de deux ordres. Il peut s’agir de variations mineures dans la séquence de la protéine HA, qualifiées de « glissements », et expliquées par un degré important d’hétérogénéité existant en permanence dans la population virale se multipliant dans l’organisme infecté. Les virus grippaux chez l’homme évoluent rapidement, en subissant une pression de sélection positive (immunosélection) de type darwinien. Certains auteurs qualifient d’espèces « fugitives » les virus de grippe A humains, car ils sont obligés d’évoluer rapidement pour réinfecter la population humaine de façon épidémique la saison hivernale ultérieure. Il peut s’agir d’un changement plus complet de la protéine HA, appelé « cassure », correspondant à un remplacement d’un virus par un autre virus, tout différent dans ses caractères immunologiques. En général, ces nouveaux virus se manifestent sous forme d’une pandémie [9].
Cette diversité et ce grand potentiel évolutif a impliqué le développement de réseaux de surveillance permettant de déceler l’apparition de ces nouveaux virus. Ces réseaux d’alerte sont locaux, régionaux, nationaux, internationaux, et coordonnés au final par l’OMS. Cette organisation permet l’adaptation annuelle de la composition vaccinale. Depuis 2004, il existe également une surveillance de la sensibilité des virus grippaux aux antiviraux actuellement disponibles (oseltamivir et zanamivir). Ces deux molécules constituent les stocks nationaux de traitements anti-grippaux, indispensables en cas de pandémie. Ce réseau a mis en évidence la circulation de virus grippal A résistant à l’oseltamivir lors de l’épidémie saisonnière 2007–2008 en Europe. Lors de cette épidémie, le virus grippal A dominant était le virus influenza A H1N1, ce sous-type représentait par exemple en France 95 % des détections de virus grippal A de la semaine 40 de l’année 2007 à la semaine 20 de l’année 2008 (du 1er octobre 2007 au 11 mai 2008). Il est à noter que, pendant les dix saisons épidémiques antérieures en Europe, le virus influenza A sous-type H3N2 a été dominant lors de neuf saisons, et le virus influenza A H1N1 dominant uniquement lors de la saison 2000–2001. En Europe, cette résistance a été confirmée dans la plupart des pays européens, mais à des taux très variables, allant de 2 % des souches grippales circulantes (Italie, Espagne) à 40 % en Belgique et en France, et plus de 60 % en Norvège. La circulation de ce variant résistant s’est poursuivie dans l’hémisphère sud pendant la saison 2008, représentant jusqu’à 100 % des souches circulantes en Afrique du Sud et en Australie, et à peine 5 % au Japon, alors que ce pays est le plus important prescripteur d’oseltamivir. Cette résistance est liée à une mutation dans la protéine neuraminidase (protéine cible de l’oseltamivir), située à la position 275 (ou 274 dans deux cas), et entraînant le remplacement d’une Histidine par une Tyrosine (mutation H275Y). Cette mutation affecte la liaison de l’oseltamivir, mais non celle du zanamivir. L’apparition de cette résistance a été spontanée et n’est pas le résultat d’une pression de sélection par l’utilisation d’oseltamivir chez les patients infectés ; les souches mutantes ont une capacité de réplication conservée (probablement liée à une meilleure balance fonctionnelle entre les protéines HA et NA H275Y qu’entre les protéines HA et NA non mutée), elles ont co-circulé avec les souches non mutées, sensibles à l’oseltamivir ; enfin, elles peuvent se transmettre facilement. L’analyse des données montre l’absence de corrélation avec des formes cliniques plus graves [10], [11]. Même s’il s’agit d’une curiosité épidémiologique, l’émergence spontanée de ce mutant résistant contrarie le dogme des souches virales dite « sauvages » naturellement sensibles à la molécule antivirale jamais rencontrée. Elle renforce la nécessité d’une surveillance de la sensibilité des souches aux antiviraux disponibles et la nécessité de disposer de substances permettant une éventuelle multithérapie.
4. Symptomatologie de la grippe
4.1. La grippe de l’adulte
La grippe saisonnière est habituellement caractérisée par l’apparition brutale de la fièvre, de céphalées, de myalgies, d’une toux sèche, de maux de gorge et d’une rhinite [12]. La grippe non compliquée se résout spontanément en trois à sept jours, bien qu’une toux et une asthénie puissent persister plus de deux semaines. En période non épidémique, la symptomatologie de la grippe n’est pas suffisamment spécifique pour que la clinique suffise au diagnostic. D’autres virus respiratoires, en particulier les rhinovirus, certains virus parainfluenza, adénovirus ou coronavirus, donnent des signes cliniques comparables à ceux d’une infection à virus influenza. Ainsi, chez des patients de 18 ans et plus, consultant à l’hôpital pour une infection respiratoire aiguë, une étude hollandaise montre qu’un diagnostic clinique de grippe a été posé chez seulement 44 % des sujets infectés par un virus influenza [13]. En revanche, en pleine épidémie, une simple identification clinique de grippe basée sur l’apparition brutale d’une toux et de la fièvre a une valeur prédictive positive de 79 % à 88 % chez les adolescents et les adultes [14], [15]. Le diagnostic clinique de la grippe est plus difficile chez les personnes âgées. Dans une population de patients infectés et non hospitalisés de plus de 60 ans, la valeur prédictive positive de la présence de fièvre, de toux et d’un début brutal ne dépasse pas 30 % [16]. Chez des sujets de plus de 65 ans, porteurs de pathologies chroniques et hospitalisés, cette valeur atteint 53 % sur les critères de fièvre, de toux et d’une maladie de moins de sept jours [17].
Les complications de la grippe sont classiques : infections bactériennes secondaires (pneumonie, sinusite, bronchite, etc.), aggravation d’une pathologie préexistante (cardiopathies, bronchopneumopathie chronique obstructive ou BPCO, etc.), pneumonie à virus influenza. Une large enquête cas-témoins faite en Grande-Bretagne montre que les sujets atteints de grippe ont 3,4 fois plus de risque de faire une complication respiratoire que les témoins ; ce risque est majoré pour les pneumonies : RR = 19,3, et les bronchites : RR = 6,1 [18].
4.2. La grippe de l’enfant
L’enfant représente une cible privilégiée des infections à virus influenza. Il est très exposé car il a les premiers contacts avec les virus de cette famille, et le mode de vie en collectivité des crèches et écoles facilite la contamination. Une étude française montre que le taux d’attaque de grippe saisonnière le plus élevé est observé chez les enfants d’âge scolaire [19]. En Finlande, il a été montré que le taux d’attaque de la grippe saisonnière pouvait atteindre 30 % des enfants entre cinq et 14 ans, et aux États-Unis, les plus forts taux d’hospitalisations pour grippe sont observés chez les enfants de moins de quatre ans et les sujets de plus de 65 ans [20], [21]. Les épidémies de grippe débutent toujours dans les collectivités d’enfants. Déjà en 1958, une étude écossaise montrait que les premiers patients hospitalisés au cours des épidémies étaient des enfants et des adolescents [22]. La grippe est une cause majeure de recours à l’hospitalisation. Une étude récente réalisée chez des enfants de moins de trois ans consultant aux urgences pédiatriques de Lyon en pleine épidémie de grippe montre que 45 % des 630 enfants inclus sont infectés par un virus influenza : 33 % chez les 0–11 mois, 56 % chez les 12–23 mois, et 59 % chez les 24–35 mois [23]. Le nombre d’hospitalisations pour grippe est d’autant plus élevé que l’enfant est plus jeune. Il est de 104 et 50 pour 10 000 chez les nourrissons de moins de six mois et de six à 12 mois, et de 19, neuf et quatre pour 10 000 chez les enfants d’un à trois ans, trois à cinq ans et cinq à 15 ans, respectivement [24].
La grippe de l’enfant de plus de cinq ans est assez proche de celle de l’adulte. Elle débute brutalement avec une forte fièvre, des frissons, un mal de gorge, une asthénie, des courbatures et des céphalées ; les manifestations respiratoires sont rapidement marquées : signes d’irritation laryngotrachéale, bronchique, conjonctivale ; la fièvre et les douleurs durent trois à quatre jours [25]. Deux points sont à souligner. Le premier concerne la grippe du nourrisson. Elle peut être paucisymptomatique, s’accompagner de léthargie, ou être traduite par des manifestations respiratoires ressemblant à celles dues à d’autres virus respiratoires qui circulent à la même période (virus respiratoire syncytial, virus parainfluenza, rhinovirus) : rhinopharyngite, bronchiolite, laryngite, pneumonie, otite moyenne aiguë. Le second concerne les manifestations non respiratoires. Elles sont fréquentes chez le nourrisson. Dans l’étude lyonnaise, 28 % des enfants infectés ont consulté pour des symptômes initiaux non respiratoires : fièvre isolée (22 %), syndrome digestif fébrile (15 %), malaise grave du nourrisson (1 %) [23]. En outre, 5 % des enfants ont présenté des convulsions fébriles, ce qui avait été déjà bien souligné dans une étude caennaise [26].
Les complications de la grippe sont multiples ; on peut observer une pneumonie à virus influenza, l’aggravation d’une pathologique préexistante (cardiopathies, asthme…), une infection bactérienne secondaire (pneumonie, sinusite, otite), la facilitation d’une seconde infection virale (adénovirus, herpès) ou bactérienne [27]. Une étude récente souligne la fréquence des infections invasives à pneumocoque quelques semaines après le pic hivernal de grippes et de bronchiolites [28]. La mortalité due à la grippe est considérée comme faible chez l’enfant. Une estimation portant sur 11 épidémies de grippe en Grande-Bretagne fait état de 78 décès d’enfants d’un mois à 14 ans liés à la grippe [29]. Les formes dites malignes sont extrêmement rares ; elles peuvent se traduire par un décès brutal dans un tableau d’alvéolite hémorragique majeure ou de myocardite [30], [31].
5. Identification d’une infection à virus influenza
L’identification d’une infection à virus influenza A ou B est particulièrement utile en périodes pré- ou post-épidémique. Elle est indispensable chez tous les sujets hospitalisés pour un syndrome respiratoire compatible avec une infection par un virus influenza . Elle est justifiée si l’on recourt à un traitement antiviral spécifique, et elle s’impose en cas d’échec thérapeutique.
Parmi les outils disponibles pour identifier une infection à virus influenza chez un patient, la sérologie a un intérêt très secondaire. Les résultats de l’étude de deux sérums successifs seront, en effet, obtenus trop tardivement pour être utiles au diagnostic. Il existe de nombreux outils permettant une recherche directe du virus ou de ses constituants dans les secrétions respiratoires : isolement du virus en culture, détection d’antigènes par IF ou EIA, recherche d’ARN par RT-PCR.
5.1. L’isolement en culture
L’isolement des virus influenza en culture sur œuf de poule embryonné ou sur des cellules de mammifères cultivées in vitro reste encore aujourd’hui la méthode de référence de l’identification des virus influenza. Le système cellulaire qui est le plus largement employé dans les laboratoires de diagnostic est représenté par les cellules rénales de chien MDCK (Madin-Darby canine kidney). C’est une lignée continue de cellules épithéliales polarisées, possédant des récepteurs spécifiques des virus influenza (de type sialique), qui les rendent sensibles à l’infection par la majorité des virus influenza A, B et C [32]. Les deux éléments qui vont conditionner l’efficacité de ce système pour l’isolement des virus influenza sont l’utilisation de la trypsine, qui active par clivage enzymatique l’hémagglutinine virale et rend les particules infectieuses et, à l’inoculation, la centrifugation des échantillons sur les cultures, qui accroît le rendement de la culture [33].
La culture est un système d’amplification virale puissant, capable, en théorie, de donner un résultat positif à partir d’un seul virus présent dans l’échantillon. Il a l’avantage de procurer des quantités importantes de virus, ce qui peut permettre, par exemple, d’étudier plus finement leur variabilité antigénique et génétique, ou leur profil de sensibilité à un antiviral. Lorsque ces souches sont obtenues en culture sur œuf, elles peuvent éventuellement être retenues dans la composition du vaccin antigrippal. Les inconvénients de la culture sont nombreux, et limitent son efficacité et son utilisation systématique pour le diagnostic. Elle impose des structures de laboratoire, des logistiques et compétences propres à la culture de cellules. La thermolabilité du virus, liée à son enveloppe lipidique et à son génome ARN, génère des conditions particulières de recueil (milieu de transport protecteur) et de transport (rapide) des secrétions respiratoires au laboratoire. Par ailleurs, celles-ci sont naturellement contaminées par les bactéries présentes dans la cavité nasale. Elles peuvent aussi contenir des inhibiteurs de la croissance virale (interférons, anticorps, mucines), qui rendront les cultures inutilisables (lorsque la contamination bactérienne est irréductible), ou faussement négatives. Ainsi, il a été montré que des interférons sont détectables dans les secrétions et le sang des patients atteints de grippes, même banales et non compliquées ; leurs niveaux maxima sont atteints un jour après le pic de l’excrétion virale, et ils diminuent parallèlement à la chute du titre viral [34]. Enfin, le dernier point qui gêne l’utilisation de la culture pour le diagnostic de la grippe est le délai de réponse. Les résultats de la culture ne sont en général pas disponibles avant quatre à six jours, et un délai supplémentaire de quelques jours est souvent nécessaire pour identifier plus finement le sérotype viral. Ainsi, l’isolement en culture des virus influenza n’est pas réalisable dans un laboratoire de biologie polyvalente. Il n’est pas adapté au diagnostic rapide. Il représente un outil spécialisé, réservé aux laboratoires de virologie, mais néanmoins indispensable au suivi de la variabilité des virus influenza et à la stratégie vaccinale.
5.2. Les techniques immunologiques de diagnostic rapide
Les laboratoires non spécialisés peuvent faire aisément le diagnostic virologique de la grippe grâce aux techniques immunologiques directes. La recherche d’antigènes viraux intracellulaires par immunofluorescence (IF) sur l’échantillon respiratoire ou d’antigènes grippaux extraits des prélèvements par méthode immunoenzymatique (EIA) permet d’identifier en une à deux heures une infection à virus influenza A ou B. La méthodologie est simple, rapide et en général globalement peu coûteuse.
5.2.1. Immunofluorescence
Pour l’IF, les cellules d’une aspiration (ou d’un lavage ou d’un écouvillonnage) nasale ou trachéobronchique sont, après lavage, déposées sur une lame microscopique et fixées dans l’acétone. Des anticorps monoclonaux (disponibles commercialement) permettent de marquer les protéines virales. On choisit celles qui sont abondamment exprimées dans les cellules infectées, et qui ne risquent pas de subir de variations antigéniques : protéines NP ou M1 par exemple. La lecture de la lame d’IF se fait soit directement lorsque les anticorps monoclonaux (de souris) antivirus influenza sont eux-mêmes marqués à l’aide d’un fluorochrome, soit indirectement à l’aide d’anticorps fluorescents anti-immunoglobulines de souris. Bien que la sensibilité de l’IF indirecte soit en théorie plus grande, celle-ci dépend surtout du type et de la qualité des anticorps monoclonaux de détection ou de révélation utilisés. Des données déjà anciennes de la littérature ne montrent pas en fait de différences significatives entre les deux approches, et l’IF directe est même parfois plus sensible que la technique indirecte : 65 % versus 45 % [35]. Comparée à l’isolement en cultures de cellules, la sensibilité de l’IF varie selon les études de 65 % à 100 % pour la détection des virus de la grippe hivernale [32], [33], [34]. La majorité de ces études montre qu’une faible fraction des échantillons sont culture-positifs et IF-négatifs. Plusieurs raisons peuvent être évoquées pour expliquer cette discordance. Les épitopes reconnus par les anticorps monoclonaux antivirus influenza peuvent être masqués ou peu exprimés sur certaines souches virales. Plus vraisemblablement, l’absence de cellules infectées visibles en IF tient soit à leur destruction rapide par l’infection elle-même, très cytolytique, ou en raison d’un mauvais conditionnement des échantillons, soit à leur recueil tardif, à un stade où ne persistent que de rares cellules infectées ou virus.
5.2.2. Tests immunoenzymatiques
Des tests EIA « maison » ont été développés par plusieurs laboratoires de virologie pour la recherche des virus influenza. Le plus souvent, la technique est la même, et les différences portent sur la préparation de l’antigène et le type d’anticorps. Les antigènes viraux extraits des prélèvements sont captés par un anticorps monoclonal dans les puits d’une plaque de microtitration, et leur détection se fait par un test EIA classique [33], [36]. Leur sensibilité comparée à l’isolement en culture varie de 50 à 90 % selon les auteurs [37]. Les tests EIA sur membrane représentent une variante technique intéressante [38]. Plusieurs de ces tests sont actuellement commercialisés en France pour la détection des virus influenza A et B de la grippe saisonnière. Leur sensibilité est à peu près équivalente à celle de l’IF. Le caractère unitaire des tests EIA, leurs avantages de rapidité et de facilité d’exécution compensent largement la sensibilité moyenne observée dans certaines études et le coût relativement élevé des réactifs. Ces tests sont peu performants sur les prélèvements de gorge par rapport aux prélèvements nasopharyngés, et l’intensité de la positivité du test n’est pas directement liée au titre infectieux de l’échantillon, mais elle est dépendante de la quantité de cellules infectées de l’échantillon : 20 cellules infectées suffisent pour déclencher la positivité [39]. Cette technologie représente incontestablement l’outil le mieux adapté au diagnostic de la grippe en médecine ambulatoire.
5.3. Les méthodes de virologie moléculaire
Les premières applications des techniques de PCR à la détection des virus influenza A ou B remontent aux années 1991–1992 avec les travaux de Yamada et al., Zhang et Evans, Claas et al. et Donofrio et al. [40], [41], [42], [43]. Le typage des virus influenza A, grâce à l’identification moléculaire de leurs hémagglutines ou neuraminidases a également été proposé à cette époque [41], [44]. La sensibilité de la méthode décrite par Yamada et al. atteint 1,3 à 6 UFP (unités formant plages) de virus influenza A, et l’étude de 23 prélèvements de gorge montre une concordance totale entre les résultats de la culture et de la PCR : 17 échantillons sont positifs et six sont négatifs par les deux méthodes. Zhang et Evans étudient la sensibilité et la spécificité d’une série d’amorces permettant soit la détection des virus influenza A ou B (gène de la protéine de matrice M) ou C (gène de l’hémagglutine), soit l’identification des hémagglutines H1, H2, H3 et des neuraminidases N1 ou N2, mais aucun essai clinique n’est réalisé. L’utilisation de cette PCR nichée dans le diagnostic virologique de la grippe est présentée dans une autre étude [45]. Dans celle-ci, la sensibilité analytique de cette RT-PCR nichée en culture est de 1 à 5 UFP de virus influenza A H1N1, A H3N2 ou B. Sur 619 prélèvements respiratoires analysés pendant l’hiver 1995–1996, 246 (39,7 %) sont positifs par RT-PCR, tandis que 200 (32,3 %) contiennent un virus influenza en culture. La sensibilité supérieure de la technique moléculaire sur la culture est encore plus nette en pleine épidémie grippale où 57,5 % des échantillons sont positifs en RT-PCR contre 38,9 % en culture. La technique rapportée par Donofrio et al. a été utilisée dans plusieurs études. Ces auteurs rapportent les résultats obtenus sur une petite série de prélèvements respiratoires et ne notent pas de différence de sensibilité entre la culture : 14 positifs sur 33, et la RT-PCR : 15 positifs [43]. Atmar et al. retrouvent la même correspondance entre les résultats de la culture et de la RT-PCR : 20 échantillons sont trouvés positifs sur les 72 par les deux approches [46]. Dans une étude sur l’infection expérimentale de volontaires humains par un virus influenza, Cherian et al. observent que cette technique de RT-PCR est particulièrement intéressante pour détecter une infection à virus influenza lorsque les échantillons sont prélevés plus de quatre jours après l’infection : 17 de 45 échantillons prélevés tardivement sont positifs en PCR contre quatre en culture [47]. Enfin, la technique de Wright et al. est une RT-PCR multiplex utilisant plusieurs paires d’amorces dans les gènes NP, NS, H1 et H3 ; elle permet d’identifier à la fois les virus influenza A, B, et les hémagglutinines H1 et H3 [44]. Sur 98 prélèvements respiratoires, 20 sont positifs par culture et 21 par RT-PCR. Nous avons réalisé une étude comparative de la sensibilité de trois techniques de RT-PCR amplifiant des séquences de gènes différents M, NS et NP des virus influenza A, et en comparant les résultats à ceux des techniques conventionnelles d’IF et de culture sur cellules MDCK pour la détection des virus influenza A [48]. L’enquête a porté sur 244 aspirations nasales d’enfants atteints d’infection respiratoire aiguë au cours de l’hiver 1998–1999. Les ARN sont extraits par le thiocyanate de guanidium ; la reverse transcription est faite à l’aide de l’amorce universelle en 3’ ; les amorces sont et les sondes celles des études précédentes [42], [43], [44]. Les résultats que nous avons obtenus montrent la supériorité des méthodes de détection moléculaire par rapport aux outils conventionnels de diagnostic : 74 (30,3 %) échantillons sont trouvés positifs en IF, 100 (40,9 %) sont positifs en culture, 157 (64,3 %) sont positifs avec le système amorces-sondes dans le gène M, 147 (60,2 %) sont positifs avec le système amorces-sondes dans le gène NS, et 110 (45 %) avec la RT-PCR décrite dans le gène NP. Ainsi, le choix du gène à amplifier, guidé par son degré de conservation, doit aussi tenir compte de sa nature et de sa position sur le génome viral.
Ces différentes RT-PCR dites « classiques » (avec détection des produits amplifiés sur gel d’agarose) ont été progressivement remplacées par des techniques de RT-PCR en temps réel, qui présentent l’avantage d’une grande spécificité grâce à l’utilisation d’une sonde et d’une manipulation en tube fermé. À côté des techniques « maison » publiées, peu de trousses commerciales sont disponibles, permettant la détection générique des virus influenza A et B.
Plus récemment, plusieurs industriels ont développé des techniques multiplex permettant la détection simultanée de plusieurs cibles virales (12 à 15 cibles virales différentes) parfois associées à quelques bactéries difficilement cultivables. Ces techniques ont l’avantage d’inclure un contrôle interne permettant de valider le processus d’extraction (séquence ARN encapsidée ou non) et de contrôler l’absence d’inhibiteurs de la polymérase . Ces tests utilisent des technologies variées : PCR en temps réel (société Diagénode), PCR classique avec détection des produits amplifiés par analyse de fragment ou électrophorèse capillaire (société Bionobis, société Pathofinder), par technologie Luminex (société Abbott, société Qiagen), ou par hybridation sur des puces basse densité (société Genomica). Certaines de ces trousses permettent la détection générique des virus influenza A et B, d’autres permettent également le typage moléculaire de l’hémagglutinine. Un des avantages de la PCR en temps réel est la possibilité d’envisager la quantification de la cible virale, en prenant en compte la richesse du prélèvement respiratoire qui, par nature, est hétérogène. Cependant, l’apport de la quantification des virus respiratoires est encore à démontrer et n’a pas, à ce jour, de réalité pratique. En revanche, un des inconvénients de la PCR en temps réel est le nombre limité de cibles détectables en un tube (trois au maximum, dont le contrôle interne), ce qui multiplie le nombre de tubes réactionnels quand la recherche inclut plusieurs virus respiratoires. À l’inverse, avec les trousses utilisant une autre technologie (analyse de fragment, luminex), la réaction « multiplex » se fait en un seul tube, avec cependant l’inconvénient que ce tube n’est pas fermé (ouverture de tube contenant des produits amplifiés avant la détection).
La survenue de la pandémie grippale H1N1v (nouveau variant 2009) en avril dernier a constitué un véritable « booster » pour le diagnostic moléculaire respiratoire, devenu indispensable pour la prise en charge optimale des patients « grippés » hospitalisés. La faible sensibilité clinique des syndromes grippaux et pseudogrippaux explique les limites d’un diagnostic virologique uniquement centré sur les virus influenza. Enfin, même si ces nouvelles techniques constituent un réel progrès dans le diagnostic moléculaire des viroses respiratoires, plusieurs questions attendent des réponses. Certaines sont d’ordre biologique : sensibilité clinique réelle de ces tests pour les différentes cibles virales, du fait de leur sophistication, interprétation clinique des fréquentes co-détections virales mises en évidence par ces techniques multiplex. D’autres questions concernent la prise en charge de ces examens (actes hors nomenclature, formation technique), et leur apport réel dans la prise en charge du patient hospitalisé.
6. Conclusion
Ainsi, il existe plusieurs outils permettant une recherche directe du virus ou de ses constituants dans les secrétions respiratoires : isolement du virus en culture, détection d’antigènes par IF ou EIA, recherche d’ARN par RT-PCR. Le choix de la méthode à utiliser n’est pas forcement simple. Des caractéristiques telles que la rapidité du test, sa simplicité de réalisation, le coût réduit, sont le propre des tests antigéniques ; en revanche, les qualités de sensibilité et de spécificité sont l’apanage des tests moléculaires et de la culture. De plus, d’autres éléments non liés aux techniques interfèrent avec leurs performances : la précocité du recueil de l’échantillon respiratoire au cours de la maladie, le type et la qualité du prélèvement, son transport, etc. Avec l’arrivée des nouvelles possibilités de traitement spécifique de la grippe, son diagnostic au laboratoire est appelé à se développer. La plupart des techniques conventionnelles de détection directe de l’infection par un virus influenza resteront confinées dans les laboratoires de virologie. C’est nécessairement le cas de l’isolement en culture de cellules, dont nous avons montré qu’il demeure la méthode de référence pour détecter la grippe. Les méthodes immunologiques permettant l’identification des antigènes de virus influenza dans les prélèvements respiratoires, IF et EIA, devraient connaître un développement important. Les anticorps existent, la technologie est bien maîtrisée, et la décision de développer des tests unitaires simples et peu coûteux dépend seulement d’une volonté industrielle. Ils envahiront nécessairement le marché de la biologie polyvalente. La place de la virologie moléculaire dans le diagnostic de la grippe et des autres viroses respiratoires chez les patients hospitalisés doit sans doute être évaluée plus précisément avant sa mise en place élargie. Elle constitue un vrai sujet d’actualité en cette année de pandémie grippale.
Conflits d’intérêts
Aucun.
Références
- 1.Gouarin C., Vabret A., Dina J., Petitjean J., Brouard J., Cuvillon-Nimal D. Study of influenza C virus infection in France. J Med Virol. 2008;80:1441–1446. doi: 10.1002/jmv.21218. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Brankston G., Gitterman L., Hirji Z., Lemieux C., Gardam M. Transmission of influenza A in human beings. Lancet Infect Dis. 2007;7:257–265. doi: 10.1016/S1473-3099(07)70029-4. [DOI] [PubMed] [Google Scholar]
- 3.Hall C.B. The spread of influenza and other respiratory viruses: complexities and conjectures. Clin Infect Dis. 2007;45:353–359. doi: 10.1086/519433. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Tellier R. Review of aeosrol transmission of influenza A virus. Emerg Infect Dis. 2006;12:1657–1662. doi: 10.3201/eid1211.060426. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.LeeKha S., Zitterkopf N.L., Espy M.J., Smith T.F., Thompson R.L., Sampathkumar P. Duration of influenza A virus shedding in hospitalized patients and implications for infection control. Infect Control Hosp Epidemiol. 2007;28:1071–1076. doi: 10.1086/520101. [DOI] [PubMed] [Google Scholar]
- 6.Carrat F., Vergu E., Ferguson N.M., Lemaitre M., Cauchemez S., Leach S. Time lines of infection and disease in human influenza: a review of volunteer challenge studies. Am J Epidemiol. 2008;167:775–785. doi: 10.1093/aje/kwm375. [DOI] [PubMed] [Google Scholar]
- 7.Hayden F.G., Fritz R., Lobo M.C., Alvord W., Strober W., Strauss S.E. Local and systemic cytokine responses during experimental human influenza A infection. J Clin Invest. 1998;101:643–649. doi: 10.1172/JCI1355. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Hall C.B., Douglas R.G. Nosocomial influenza infection as a cause of intercurrent fevers in infants. Pediatrics. 1975;55:673–677. [PubMed] [Google Scholar]
- 9.Manuguerra J.C. Écologie, biodiversité et évolution des virus grippaux. Virologie. 2001;5(3):195–205. [Google Scholar]
- 10.Cianco BC, Meerhoff TJ, Kramarz P, Bonmarin I, Borgen K, Boucher CA, et al. Oseltamivir-resistant influenza A (H1N1) viruses detected in Europe during season 2007-8 had epidemiologic and clinical characteristics similar to co-circulating susceptible A (H1N1) viruses. http://www.eurosurveillance.org, 19 novembre 2009. [PubMed]
- 11.Meijer A., Lackenby A., Hungnes O., Lina B., van der Werf S., Schweiger B. Oseltamivir-resistant Influenza Virus A(H1N1) Europe, 2007-8 Season. Emerg Inf Dis. 2009;15(4):552–560. doi: 10.3201/eid1504.081280. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Nicholson K.G. Clinical features of influenza. Semin Respir Infect. 1992;7:26–37. [PubMed] [Google Scholar]
- 13.vd Hoeven A.M., Scholing M., Wever P.C., Fijnheer R., Hermans M., Schneeberger P.M. Lack of discriminating signs and symptoms in clinical diagnosis of influenza of patients admitted to the hospital. Infection. 2007;35:65–68. doi: 10.1007/s15010-007-6112-1. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Boivin G., Hardy I., Tellier G., Maziade J. Predicting influenza infections during epidemics using a clinical case definition. Clin Infect Dis. 2000;31:1166–1169. doi: 10.1086/317425. [DOI] [PubMed] [Google Scholar]
- 15.Monto A.S., Gravenstein S., Elliott M., Colopy M., Schweinle J. Clinical signs and symptoms predicting influenza infection. Arch Intern Med. 2000;160:3243–3247. doi: 10.1001/archinte.160.21.3243. [DOI] [PubMed] [Google Scholar]
- 16.Govaert T.M., Dinant G.J., Aretz K., Knottnerus J.A. The predictive value of influenza symptomatology in elderly poeple. Fam Pract. 1998;15:16–22. doi: 10.1093/fampra/15.1.16. [DOI] [PubMed] [Google Scholar]
- 17.Walsh E.E., Cox C., Falsey A.R. Clinical features of influenza a infection in older hospitalized persons. J Am Geriatr Soc. 2002;50:1498–1503. doi: 10.1046/j.1532-5415.2002.50404.x. [DOI] [PubMed] [Google Scholar]
- 18.Meier C.R., Napalkov P.N., Wegmuller Y., Jefferson T., Jick H. Population-based study on incidence, risk factors, clinical complications and drug utilisation associated with influenza in the united Kingdom. Eur J Clin Microbiol Infect Dis. 2000;19:834–842. doi: 10.1007/s100960000376. [DOI] [PubMed] [Google Scholar]
- 19.Carrat F., Flahault A., Boussard E., Ferran N., Dangoumau L., Valleron A.J. Surveillance of influenza-like illness in France The example of the 1995/1996 epidemic. J Epidemiol Comm Health. 1998;52:S32–S38. [PubMed] [Google Scholar]
- 20.Heikkinen T. Influenza in children. Acta Pediatrica. 2006;95:778–784. doi: 10.1080/08035250600612272. [DOI] [PubMed] [Google Scholar]
- 21.Glezen W.P., Decker M., Perrotta D.M. Survey of underlying conditions of persons hospitalized with acute respiratory disease during epidemics in Houston, 1978-1981. Am Rev Respir Dis. 1987;136:550–555. doi: 10.1164/ajrccm/136.3.550. [DOI] [PubMed] [Google Scholar]
- 22.A combined study group. Some aspects if the recent epidemic of influenza in Dundee. Br Med J 1958;1:908–13. [PMC free article] [PubMed]
- 23.Ploin D., Gillet Y., Morfin F., Fouilhoux A., Billaud G., Liberas S. Influenza burden in febrile infants and young children in a pediatric emergency department. Pediatr Infect Dis J. 2007;26:142–147. doi: 10.1097/01.inf.0000253062.41648.60. [DOI] [PubMed] [Google Scholar]
- 24.Neuzil K.M., Mellen B.G., Wright P.F., Mitchel E.F., Griffin M.R. The effect of influenza on hospitalizations, outpatients visits, and courses of antibiotics in children. N Engl J Med. 2000;342:225–231. doi: 10.1056/NEJM200001273420401. [DOI] [PubMed] [Google Scholar]
- 25.Floret D. La grippe de l’enfant. Rev Prat. 2007;57:1895–1899. [PubMed] [Google Scholar]
- 26.Brouard J., Ribet V., Petitjean J., Freymuth F., Duhamel J.F. Infection à virus influenza A chez l’enfant. Arch Fr Pediatr. 1992;49:693–697. [PubMed] [Google Scholar]
- 27.Schrag S.J., Shay D.K., Gershman K., Thomas A., Craig A.S., Schaffner W. Multistate surveillance for laboratory-confirmed, influenza-associated hospitalisations in children, 2003-2004. Pediatr Infect Dis J. 2006;25:395–400. doi: 10.1097/01.inf.0000214988.81379.71. [DOI] [PubMed] [Google Scholar]
- 28.Ampofo K., Bender J., Sheng X., Korgenski K., Daly J., Pavia A.T. Seasonal invasive pneumococcal disease in children: role of preceding respiratory viral infections. Pediatrics. 2008;122:229–237. doi: 10.1542/peds.2007-3192. [DOI] [PubMed] [Google Scholar]
- 29.Flemming D.M., Pannel R.S., Cross K.M. Mortality in children from influenza and respiratory syncytial virus. J Epidemiol Comm Health. 2005;59:586–590. doi: 10.1136/jech.2004.026450. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 30.Marcorelles P., Freymuth F., Rambaud C., Gardach C., Legrand-Quillien M.C., Lagarde N. Décès brutal et infection à virus influenza A chez un enfant de deux ans : étude d’un cas autopsique. Arch Pediatr. 2002;9:41–44. doi: 10.1016/S0929-693X(01)00693-5. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 31.Letouze N., Jokic M., Maragnes P., Rouleau V., Flais F., Vabret A. Myocardite fulminante de la grippe de type A : cas fatal d’une enfant de 8 ans. Arch Mal Coeur Vaiss. 2006;99:514–516. [PubMed] [Google Scholar]
- 32.Tobita K., Sigiura A., Enomoto C., Furuyama M. Plaque assay and primary isolation of influenza A virus in an established line of canine kidney cells in the presence of trypsin. Med Microbiol Immunol. 1975;162:9–14. doi: 10.1007/BF02123572. [DOI] [PubMed] [Google Scholar]
- 33.Chomel J.J., Remilleux M.F., Marchand P., Aymard M. Rapid diagnosis of influenza A Comparison with immunocapture and culture. J Virol Methods. 1992;37:337–344. doi: 10.1016/0166-0934(92)90033-a. [DOI] [PubMed] [Google Scholar]
- 34.Jao R.L., Wheelock E.F., Jackson G.G. Production of interferon in volunteers infected with asian influenza. J Infect Dis. 1970;121:419. doi: 10.1093/infdis/121.4.419. [DOI] [PubMed] [Google Scholar]
- 35.Dominguez E.A., Taber L.H., Cough R.B. Comparison of rapid diagnostic techniques for respiratory syncytial and influenza A virus respiratory infections in young children. J Clin Microbiol. 1993;31:2286–2290. doi: 10.1128/jcm.31.9.2286-2290.1993. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 36.Grandien M., Pettersson C.E., Gardner P.S., Linde A., Stanton A. Rapid viral diagnosis of acute respiratory infections: comparison of enzyme-linked immunosorbent assay and the immunofluorescence technique for detection of viral antigens in nasopharyngeal secretions. J Clin Microbiol. 1985;22:757–760. doi: 10.1128/jcm.22.5.757-760.1985. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 37.Leonardi G.P., Leib H., Birkhead G.S., Smith C., Costello P., Conron W. Comparison of rapid detection methods for influenza virus and their value in health-care management of institutionalized geriatric patients. J Clin Microbiol. 1994;32:70–74. doi: 10.1128/jcm.32.1.70-74.1994. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 38.Duverlie G., Houbart L., Visse B., Chomel J.J., Manuguerra J.C., Hannoun C. A nylon membrane enzyme immunoassay for rapid diagnosis of influenza A infection. J Virol Methods. 1992;40:77–84. doi: 10.1016/0166-0934(92)90009-3. [DOI] [PubMed] [Google Scholar]
- 39.Ryan-Poirier K.A., Katz J.M., Webster R.G., Kawaoka Y. Application of Directigen Flu-A for the detection of influenza A virus in human and nonhuman primates. J Clin Microbiol. 1992;30:1072–1075. doi: 10.1128/jcm.30.5.1072-1075.1992. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 40.Yamada A., Imanishi J., Nakajima E., Nakajima K., Nakajima S. Detection of influenza viruses in throat swab by using polymerase chain reaction. Microbiol Immunol. 1991;35:259–265. doi: 10.1111/j.1348-0421.1991.tb01555.x. [DOI] [PubMed] [Google Scholar]
- 41.Zhang W., Evans D.H. Detection and identification of human influenza by the polymerase chain reaction. J Virol Methods. 1991;33:165–189. doi: 10.1016/0166-0934(91)90017-t. [DOI] [PubMed] [Google Scholar]
- 42.Claas E.C.J., Sprenger M.J.W., Kleter G.E.M., van Beck R., Quint W.G.V., Masurel N. Type-specific identification of influenza viruses A B and C by the polymerase chain reaction. J Virol Methods. 1992;39:1–13. doi: 10.1016/0166-0934(92)90120-3. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 43.Donofrio J.C., Coonrod J.D., Davidson J.N., Betts R.F. Detection of influenza A and B in respiratory secretions with the polymerase chain reaction. PCR Methods Appl. 1992;1:263–268. doi: 10.1101/gr.1.4.263. [DOI] [PubMed] [Google Scholar]
- 44.Wright K.E., Wilson G.A.R., Novosad D., Dimock C., Tan D., Weber J.M. Typing and subtyping of influenza viruses in clinical samples by PCR. J Clin Microbiol. 1995;33:1180–1184. doi: 10.1128/jcm.33.5.1180-1184.1995. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 45.Ellis J.S., Fleming D.M., Zambon M.C. Multiplex reverse transcription-PCR for surveillance of influenza A and B viruses in England and Wales in 1995 and 1996. J Clin Microbiol. 1997;35:2076–2082. doi: 10.1128/jcm.35.8.2076-2082.1997. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 46.Atmar R.L., Baxter B.D., Dominguez E.A., Taber L.H. Comparison of reverse transcription-PCR with tissue culture and other rapid diagnosis assays for detection of type A influenza virus. J Clin Microbiol. 1996;34:2604–2606. doi: 10.1128/jcm.34.10.2604-2606.1996. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 47.Cherian T., Bobo L., Steinhoff M.C., Karron R.A., Yolken R.H. Use of PCR-enzyme immunoassay for identification of influenza A virus matrix RNA in clinical samples negative for cultivable virus. J Clin Microbiol. 1994;199(32):623–628. doi: 10.1128/jcm.32.3.623-628.1994. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 48.Vabret A., Sapin G., Lezin B., Mosnier A., Cohen J.M., Burnouf L. Comparison of three non-nested RT-PCR for the detection of influenza A viruses. J Clin Virol. 2000;17:167–175. doi: 10.1016/s1386-6532(00)00095-0. [DOI] [PubMed] [Google Scholar]


