Abstract
Alors que la place de la PCR est bien reconnue pour le diagnostic des infections neuroméningées à entérovirus (EV), l’utilisation des techniques de biologie moléculaire dans les infections respiratoires à EV est encore mal définie.
Objectifs
Définir les apports des outils moléculaires (PCR et génotypage) dans le diagnostic et l’épidémiologie des infections respiratoires à EV.
Méthodes
Au cours de l’année 2008, 3612 sécrétions nasales ont eu une recherche virale par IFD et culture MRC5. Dans un second temps, sont réalisés une recherche virale sur culture HuH7 (SN négative en IF) et une PCR duplex entérorhinovirus (PEVRV) sur 816 surnageants de culture HuH7 présentant un ECP. De plus, 327 SN de nouveau-nés ont eu une recherche systématique par la PCR EV temps réel « maison » (PEV) utilisée pour le diagnostic des infections méningées. Un génotypage a été réalisé chez 68 patients présentant une identification d’EV sur surnageants de cultures MRC5 ou HuH7.
Résultats
Trente-cinq EV ont été isolés en culture (0,97 %) ; la combinaison des deux PCR, PEVRV et PEV, a permis d’identifier respectivement, 41 EV, 8 EV-RV et 12 RV supplémentaires, portant le nombre de positif à 96 (2,6 %). Chez les NN, 32 (11,3 %) SN sont positives avec la PEV. Parmi les 98 SN testées par PEV et PEVRV, 27 étaient positives et nous avons détecté dix EV, cinq EV-RV et 12 RV. De janvier à décembre 2008, la distribution saisonnière montre le pic estival habituel d’infections méningées et un pic automnal correspondant d’infections respiratoires. Cinq sérotypes prédominent : 19 EV68 (29,7 %), 12 CB3 (18,7 %), neuf E3 (14,1 %), six CA9 (9,4 %) et six CB1 (9,4 %). Les EV68 sont tous isolés en octobre à novembre et 17/19 (89,5 %) des patients positifs ont été hospitalisés avec des signes d’insuffisance respiratoire aiguë.
Conclusion
Les méthodes de diagnostic moléculaires des infections respiratoires à EV (PCR et génotypage) permettent d’identifier précisément les souches circulantes dans un temps relativement court, apportant un bénéfice épidémiologique, clinique et économique de santé.
Mots clés: Infections respiratoires, Entérovirus, Rhinovirus, Picornavirus, PCR, Génotypage
Abstract
The PCR assays are currently used in diagnosis of enterovirus (EV) meningitis. Nevertheless, the use of molecular diagnosis of EV should be investigated in respiratory tract infections (RTI).
Objectives
To perform enterovirus molecular diagnostic tools, PCR and genotyping, in nasal samples for diagnostic and epidemiologic purposes.
Methods
During 2008, 3612 nasal specimen (NS) were studied by IFD and MRC5 culture. Next, we realised successively viral isolation on HuH7 culture (for NS negative by IFD assay) and a duplex PCR enterovirus-rhinovirus for the 816 HuH7 positive supernatants. Furthermore, 327 NS collected from neonates were systematically tested by a real-time RT-PCR. This assay was used in routine for EV diagnosis setting in cerebrospinal fluid. Enterovirus genotyping was then performed for the 68 positive supernatants.
Results
Thirty-five NS (0.97%) were positive for EV by culture (MRC5). A combination of both PCR assays, PEVRV and PEV, allowed an additional identification of 41 EV, eight EV-RV and 12 RV, increasing the number of positive to 96 NS (2.6%). Among the neonates, 32 NS (11.3%) were positive for EV by PEV. Of the 98 NS tested by the two PCR assays (PEV and PEVRV), 27 were positive and we detected 10 EV, five EV-RV and 12 RV. From January to December 2008, the circulation of EV showed the usual peak in June–July when a small outbreak of aseptic meningitis occurred and an additional autumnal peak corresponding to respiratory tract infections. Five main serotypes were isolated: 19 EV68 (29.7%), 12 CB3 (18.7%), nine E3 (14,1%), six CA9 (9.4%) and six CB1 (9.4%); the 19 EV68 were isolated in October–November and 17/19 (89.5%) of positive patients were hospitalised for severe respiratory diseases.
Conclusion
The use of molecular screening techniques (PCR assays and genotyping) on nasal samples collected from patients with respiratory infections allowed a prospective, effective and precise identification of circulating strains.
Keywords: Respiratory infections, Enterovirus, Rhinovirus, Picornavirus, PCR, Molecular identification
1. Introduction
Première cause de méningite aiguë aseptique bénigne et responsables d’infections plus sévères à type de paralysies flasques aiguës, d’atteintes cardiaques, d’encéphalites et d’infections néonatales graves, les entérovirus (EV) humains sont également responsables d’infections respiratoires [1], [2], [3], [4].
Le diagnostic moléculaire des méningites à EV a largement démontré sa supériorité en termes de sensibilité et de rapidité par rapport à la mise en culture du LCR. Il est devenu une pratique routinière, améliorant le diagnostic de certitude et, par suite, assurant une meilleure prise en charge des patients [2]. Le diagnostic moléculaire des infections respiratoires à EV, plus souvent bénignes, est rarement réalisé en pratique courante. Les EV sont le plus souvent isolés de prélèvements respiratoires à l’occasion d’une recherche non orientée sur culture cellulaire. Les limites des méthodes conventionnelles ont bien été démontrées pour le diagnostic des deux principales viroses respiratoires épidémiques, grippe et infections à virus respiratoire syncytial (VRS) [5]. Le développement récent de techniques multiplex permet, sous un aspect plus rapide et économique, la détection simultanée de plusieurs virus et par conséquent de mieux appréhender l’épidémiologie des infections virales respiratoires [6]. De plus, les techniques PCR associées au séquençage permettent d’affiner les études épidémiologiques avec identification précise des sous-types viraux et étude de la variabilité et diversité génétique des souches [7]. Les EV appartiennent à la famille des Picornaviridae, comme les rhinovirus (RV) dont ils sont très proches génétiquement [8], [9], [10]. Depuis 1970, les sérotypes humains nouvellement identifiés sont classés par numérotation (EV68 à 107). En 2008, la taxonomie internationale au sein de la famille des Picornaviridae change : le genre EV regroupe dix espèces dont six espèces infectant l’homme, quatre espèces EV nommées de A à D et deux espèces RV nommées A et B ; en 2009 apparaît une nouvelle espèce humaine RV désignée C (Fig. 1 ) [11], [12]. Les techniques de diagnostic moléculaire sont basées sur un diagnostic de genre avec amplification dans la région conservée 5′NC du génome viral. Le typage des EV par des méthodes moléculaires permet de détecter et d’identifier précisément les souches circulantes et les souches responsables de manifestations atypiques ou sévères [13], [14]. Il utilise des techniques d’amplification dans les gènes de capside (région VP1, VP2, VP3, VP1-2A, VP1-2C) et l’analyse des séquences (méthode Blast ou analyse phylogénétique) se fait grâce à la mise à disposition d’une large base de données [15], [16].
Fig. 1.

Classification des Picornavirus selon la taxonomie internationale ICTV-v4. a : espèces animales, h : espèces humaines. EV : entérovirus, RV : rhinovirus.
Cette étude a deux objectifs : tout d’abord évaluer l’apport du diagnostic moléculaire des EV en pathologie respiratoire en termes de sensibilité par rapport aux méthodes conventionnelles, puis identifier par génotypage les souches responsables d’infections respiratoires afin de mieux appréhender la circulation régionale des souches d’EV au cours de l’année 2008.
2. Patients et méthodes
2.1. Patients : algorithme de recherche virale sur les prélèvements respiratoires
Au cours de l’année 2008, 3612 sécrétions nasales (SN), dont 76 % d’enfants, ont été reçues au laboratoire de virologie du CHU de Caen et analysées de façon séquentielle par les techniques classiques d’immunofluorescence et de culture, puis par des techniques PCR spécifiques (Fig. 2 ).
Fig. 2.
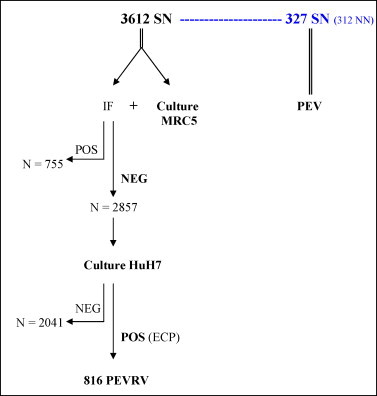
Algorithme de recherche des “picornavirus” sur les 3612 SN. SN : Sécrétions nasales ; NN : nouveau-né ; ECP : Effet cytopathogène. PEV : RT-PCR entérovirus en temps réel « maison ». PEVRV : PCR duplex entérovirus-rhinovirus.
Une recherche virale systématique a été réalisée immédiatement sur ces 3612 SN par une méthode d’immunofluorescence directe (IFD) utilisant des anticorps monoclonaux contre les virus influenza A et B, les virus parainfluenza 1, 2 et 3, les adénovirus (Imagen, Oxoïd France), le VRS, le métapneumovirus (Argène, France) et par isolement sur culture de cellules MRC5. Les SN négatives en IFD ont été secondairement ensemencées sur culture cellulaire de cellules HuH7 en plaques 48 puits. À J + 4, sont réalisés la lecture de l’effet cytopathique (ECP) et un test d’IFD sur les cellules grattées avec les mêmes anticorps monoclonaux que le test d’IFD réalisé directement sur le prélèvement. Sur les surnageants de culture HuH7 des 816 SN présentant un ECP et un test IFD négatif, ont été réalisées une PCR multiplex détectant les quatre coronavirus (OC43, 229E, NL63 et HKU1 et une PCR multiplex détectant le virus influenza C les entérovirus et les rhinovirus (PEVRV) [17].
De plus, sur les prélèvements de 312 nouveaux-nés (NN) de moins de deux mois et chez 15 enfants (deux mois à deux ans) hospitalisés pour une détresse respiratoire ou un syndrome méningé, a été réalisée de façon systématique une RT-PCR temps réel « maison » spécifique entérovirus (PEV) [18].
2.2. Isolement des EV en culture cellulaire
L’isolement des EV en culture est réalisé par inoculation de 250 μl de SN sur des flacons de 25 cm3 de fibroblastes humains embryonnaires (cellule MRC5). Les cultures incubées à 35-36 °C sont conservées pendant 3 semaines et examinées régulièrement. Tout ECP caractéristique d’EV (ECP diffus avec petites cellules arrondies non réfringentes) déclenche un repiquage sur cellules MRC5. La culture cellulaire MRC5 permet également d’isoler les RV avec un ECP typique (cellules arrondies très réfringentes en gouttelettes de mercure). Dans de rares cas, les ECP EV et RV peuvent être difficiles à différencier.
Les SN négatives en IFD sont ensemencées sur les cellules HuH7, lignée continue de cellules épithéliales dérivées d’un hépatocarcinome humain présentant un large spectre de détection des virus respiratoires dont les EV et RV, aucun ECP caractéristique de virus n’est observable sur cette lignée cellulaire [19].
2.3. Techniques de biologie moléculaire
Les amorces et sondes utilisées pour les différentes techniques de PCR et pour le génotypage sont présentées dans le Tableau 1 .
Tableau 1.
Différentes amorces et sonde utilisées dans les PCR (PEVRV et PEV) et le génotypage.
| Amorces et sonde | Séquencea | Localisation | Cible génomique | Références |
|---|---|---|---|---|
| PEVRV | Savolainen et al. [20] | |||
| Anti-sens SRHI1 | GCATCIGGYARYTTCCACCACCANCC | 1083-1058 | VP2 | |
| Sens SRHI2 | GGGACCAACTACTTTGGGTGTCCGTGT | 534-560 | 5’ NC | |
| PEV | Verstrepen et al. [22] | |||
| Anti-sens RNC2 | CACT/CGGATGGCC | 771-760 | 5’ NC | |
| Anti-sens E2 | ATTGTCACCATAAGCAGCCA | 727-708 | 5’ NC | |
| NC1M | CCCTGAATGCGGCTAATCC | 581-599 | 5’ NC | |
| OL27LC | 5’ FAM- AACCGACTACTTTGGGTGTCCGTGTTTC- TAMRA | 664-691 | 5’ NC | |
| Génotypage | Nix et al. [23] | |||
| Anti-sens AN32 | GTYTGCCA | 3009-3002 | VP1 | |
| Anti-sens AN33 | GAYTGCCA | 3009-3002 | VP1 | |
| Anti-sens AN34 | CCRTCRTA | 3111-3104 | VP1 | |
| Anti-sens AN35 | RCTYTGCCA | 3009-3002 | VP1 | |
| Sens 224 | GCIATGYTIGGIACICAYRT | 1977-1996 | VP3 | |
| Anti-sens 222 | CICCIGGIGGIAYRWACAT | 2969-2951 | VP1 | |
| Sens AN 89 | CCAGCACTGACAGCAGYNGARAYNGG | 2602-2627 | VP1 | |
| Anti-sens AN88 | TACTGGACCACCTGGNGGNAYRWACAT | 2977-2951 | VP1 | |
| Sens 232 | CCAGCACTGACAGCA | 2602-2616 | VP1 | |
| Anti-sens 233 | TACTGGACCACCTGG | 2977-2963 | VP1 | |
PEVRV : PCR duplex entéro-rhinovirus.
R = A ou G ; Y = C ou T ; W = A ou T ; I = A, T, G ou C.
2.3.1. Extraction
L’ARN est extrait sur l’automate d’extraction Biorobot M48, Qiagen S.A à partir de 300 μl de surnageant de culture (MRC5 ou HuH7) ou 300 μl de SN (prélèvement direct chez nouveau-nés) au moyen de la trousse Mag Attract viral RNA M48, programme infectious disease-viral RNA selon les instructions du fabricant. Les 50 μl d’éluat obtenus sont utilisés directement dans la PEVRV (PCR 1 étape) ou transcrits en cDNA pour la PEV (PCR 2 étapes) et la PCR semi-nichée du génotypage.
2.3.2. Technique de PCR duplex entérovirus - rhinovirus (PEVRV)
Cette technique d’amplification génique permet la détection simultanée en un seul tube des picornavirus (EV et RV) et du virus influenza C. Il s’agit d’une PCR multiplex « maison » en une étape utilisant la trousse Qiagen one step RT-PCR, deux amorces spécifiques du virus influenza C et deux amorces spécifiques consensus pour EV et RV. Ces deux amorces consensus, SRHI1 et SRHI2, sont choisies respectivement dans la 5′NC et VP2 du génome viral et permettent d’obtenir un produit PCR de taille différente pour chacun des virus : 650 pb pour EV et 530 pb pour RV [20], [21]. La détection des produits amplifiés se fait après migration sur gel d’agarose à 1,25 %.
2.3.3. Technique de RT-PCR entérovirus en temps réel (PEV)
La détection du génome des EV est réalisée depuis 2003 au laboratoire de virologie du CHU de Caen dans les liquides céphalorachidiens (LCR) par une technique de PCR temps réel Taqman « maison » en deux étapes sur le thermocycler LightCyclerR 1,0 -Roche Molecular Biochemicals avec une périodicité bihebdomadaire. Depuis novembre 2004, cette technique s’est étendue aux aspirations nasales des nouveau-nés de moins de deux mois afin d’assurer un diagnostic sensible et rapide des infections néonatales sévères.
Après une première étape de transcription inverse (amorce RNC2), la PCR utilise la trousse LC FastStart DNA master hybridization probe -Roche Molecular Biochemicals, des amorces (E2, NC1M) et une sonde (OL27LC) spécifiques, définies dans la région 5′NC, région hautement conservée dans le génome viral [18], [22]. Cette technique ne comporte pas de contrôle interne. La sensibilité attendue est de 1 TICD50 et l’absence de contamination est contrôlée par l’insertion d’un témoin négatif d’extraction et de PCR dans chaque manipulation.
2.3.4. Technique de PCR en temps réel Enterovirus R-gene, Argene
Cette trousse commercialisée et marquée CE, temps réel 1 étape, a été utilisée pour contrôler les résultats de 22 SN positives en PEV présentant un résultat discordant en PEVRV : dix SN négatives et 12 SN positives RV ou EV-RV.
Elle permet la détection générique des EV par amplification d’un fragment de la région 5′NC et est utilisée selon les recommandations du fabricant. Elle est validée sur les SN et il est stipulé dans la notice de présentation que cette trousse ne détecte pas les RV 9, 39 et 89 mais qu’elle détecte les RV 14, 17 et 22. Nous avons utilisé l’extrait ARN préparé sur l’automate Biorobot M48 après avoir montré sa supériorité sur l’extraction manuelle QIAamp®Viral RNA Mini kit, Qiagen (gain d’un log sur panel de qualité européen QCMD2009).
2.3.5. Génotypage des souches d’EV
Une identification des EV été réalisée sur les surnageants de culture positive, (MRC5 ou HuH7) par génotypage dans la région VP1 conformément à la méthodologie de PCR nichée décrite par Nix et al. [23].
Dans un premier temps, la synthèse de cDNA a été réalisée avec la transcriptase inverse superscript III (Invitrogen, Cergy Pontoise, France) pendant 45 minutes à 50 °C en utilisant 5 μl d’extrait ARN et les quatre amorces anti-sens (AN32, AN33, AN34, AN35). Avec les 10 μl de cDNA résultant, la première PCR (PCR1) a été réalisée dans un volume réactionnel de 50 μl contenant 200 μM de déoxynucléotide triphosphates, 50 pmoles de chacune des deux amorces (222 et 224) et 2,5 U de Fast-start Taq DNA polymérase selon le protocole d’amplification suivant : 95 °C pendant 15 minutes, puis 40 cycles de 30 s à 95 °C, 30 s à 40 °C et 45 s à 60 °C. La seconde PCR (PCR2) a été réalisée avec 1 μl du mélange réactionnel de la première PCR dans un volume de 25 μl contenant 200 μM de déoxynucléotide triphosphates, 40 pmoles de chacune des deux amorces (AN 88 et AN 89) et 2,5U de Fast-start Taq DNA polymérase selon le protocole d’amplification suivant : 95 °C pendant 15 minutes, puis 40 cycles de 30 s à 95 °C, 20 s à 60 °C et 15 s à 72 °C. Les produits amplifiés visualisés sur gel d’agarose à 1,2 % correspondent respectivement à des fragments de 992 pb (PCR1) et 361 pb (PCR2). Le séquençage des produits de PCR purifiés (enzymatique ExoSAP.IP, Amersham) a été réalisé sur le séquenceur SEQ 8000, Beckman à l’aide de la trousse de séquençage GenomeLab™ DTCS-Quick start, Beckman Coulter selon les recommandations du fournisseur et au moyen des amorces 232 et 233 correspondant à la partie consensus des amorces hybrides AN89 et AN88. Les séquences obtenues ont été analysées avec le logiciel Seqman et la détermination du type d’EV a été réalisée par comparaison de la séquence nucléotidique obtenue avec les séquences d’EV disponibles dans Genbank. L’analyse a été réalisée par la méthode BLAST [24].
3. Résultats
3.1. Détection des picornavirus dans les sécrétions nasales reçues au cours de l’année 2008
L’ensemencement des 3612 SN en culture MRC5 a permis d’isoler 35 EV (0,97 %) et 72 RV. Sur les 2852 SN négatives en IFD ensemencées sur cellules HuH7, 816 ont présenté une lyse conduisant à la réalisation de la PEVRV (Fig. 2).
Par cette PCR duplex, ont été détectés : 334 RV (bande unique à 530 pb), 49 EV (bande unique à 650 pb) et dix EV-RV (deux bandes à 650 pb et 530 pb). Ainsi cette PCR a permis d’identifier par rapport à la culture 33 EV et huit EV-RV supplémentaires (Tableau 2 ).
Tableau 2.
Résultats de la PEVRV chez les 816 SN présentant une lyse en culture HuH7.
| Culture MRC5 | PEVRV |
||||
|---|---|---|---|---|---|
| Négative | EV | EV-RV | RV | Total | |
| Négative | 420a | 33b | 8c | 296d | 757 |
| EV | 3 | 16 | 2 | 0 | 21 |
| RV | 0 | 0 | 0 | 38 | 38 |
| Total | 423 | 49 | 10 | 334 | 816 |
PEVRV : PCR duplex entéro-rhinovirus.
Autres virus isolés sur cellules MRC5.
Quatre VRS, 13 adénovirus, cinq CMV.
Un adénovirus, un CMV.
Un adénovirus.
Cinq adénovirus, trois CMV.
La PEV réalisée conjointement de façon systématique sur les 312 SN de nouveau-nés de moins de deux mois et sur 15 SN d’enfants hospitalisés en réanimation pédiatrique a permis d’identifier 37 EV chez 32 NN et cinq nourrissons. Sur ces 37 SN positives en PEV, deux étaient positives en culture MRC5, neuf en PEVRV, six en MRC5 et PEVRV ; la technique PEV permet donc d’identifier huit EV et 12 RV supplémentaires (Tableau 3 ).
Tableau 3.
Résultats de la culture MRC5 et PEVRV chez les 37 SN positives en PEV.
| SN | Âge (j) | Sexe | PEV | Culture MRC5 | PEVRV |
|---|---|---|---|---|---|
| 1. | 6 | M | Positif | Négatif | EV-RHI |
| 2. | 12 | F | Positif | ENTERO | ENTERO |
| 3. | 12 | F | Positif | Négatif | RHINO |
| 4. | 14 | F | Positif | ENTERO | EV-RHI |
| 5. | 14 | F | Positif | Négatif | RHINO |
| 6. | 19 | M | Positif | Négatif | NR |
| 7. | 20 | M | Positif | Négatif | RHINO |
| 8. | 23 | F | Positif | Négatif | RHINO |
| 9. | 25 | F | Positif | Négatif | ENTERO |
| 10. | 25 | F | Positif | Négatif | RHINO |
| 11. | 26 | F | Positif | Négatif | NR |
| 12. | 26 | M | Positif | Négatif | RHINO |
| 13. | 26 | M | Positif | Négatif | RHINO |
| 14. | 29 | F | Positif | ENTERO | ENTERO |
| 15. | 29 | M | Positif | Négatif | NR |
| 16. | 31 | F | Positif | Négatif | NR |
| 17. | 40 | F | Positif | Négatif | NR |
| 18. | 44 | M | Positif | Négatif | NR |
| 19. | 46 | M | Positif | Négatif | ENTERO |
| 20. | 50 | M | Positif | ENTERO | NR |
| 21. | 50 | F | Positif | Négatif | RHINO |
| 22. | 50 | F | Positif | RHINO | RHINO |
| 23. | 51 | M | Positif | ENTERO | NR |
| 24. | 54 | F | Positif | Négatif | NR |
| 25. | 58 | M | Positif | Négatif | ENTERO |
| 26. | 58 | F | Positif | Négatif | NR |
| 27. | 58 | F | Positif | Négatif | RHINO |
| 28. | 60 | M | Positif | Négatif | RHINO |
| 29. | 61 | M | Positif | CMV | ENTERO |
| 30. | 62 | M | Positif | ENTERO | ENTERO |
| 31. | 65 | M | Positif | Négatif | ENTERO |
| 32. | 66 | M | Positif | Négatif | ENTERO |
| 33. | 212 | M | Positif | ENTERO | ENTERO |
| 34. | 384 | M | Positif | ENTERO | EV-RHI |
| 35. | 414 | F | Positif | Négatif | EV-RHI |
| 36. | 493 | M | Positif | Négatif | RHINO |
| 37. | 1819 | M | Positif | Négatif | EV-RHI |
NR : non réalisé.
Au total, les deux techniques PCR, PEVRV et PEV, ont permis d’identifier 61 « picornavirus » supplémentaires (41 EV, huit EV-RV et 12 RV) portant le nombre de positif à 96 (2,6 %).
3.2. Détection des « picornavirus » chez les nouveau-nés
Pour les 327 SN testées par PEV, 37 sont rendues EV positives (11,3 %) dans les trois jours qui suivent le prélèvement alors que la culture MRC5 ne permet d’isoler que huit EV et un RV avec un délai oscillant entre six et 22 jours (Tableau 3). Quatre-vingt-dix-huit SN ont été testées par les deux techniques, PEVRV et PEV : 45 étaient négatives, 26 uniquement positives en PEVRV (25 RV et un EV-RV) et 27 positives avec les deux PCR ; parmi ces 27, ont été identifiés dix EV, cinq EV-RV et 12 RV (Tableau 4 ).
Tableau 4.
Comparaison des résultats PCR (PEVRV et PEV) réalisées sur 98 SN (89 NN et neuf enfants).
| PEV | PEVRV | ||||
|---|---|---|---|---|---|
| Négatif | Positif EV | Positif EV-RV | Positif RV | Total | |
| Négatif | 45 | 0 | 1 | 25 | 71 |
| Positif | 0 | 10 | 5 | 12 | 27 |
| Total | 45 | 10 | 6 | 37 | 98 |
L’identification de 12 RV et cinq EV-RV chez les 37 SN positives en PEV nous a conduits à contrôler 22 SN par la trousse PCR Enterovirus R-gene, Argène France. Vingt et une des 22 SN sont positives avec cette trousse, donnant une concordance de 95,5 % entre les deux PCR EV temps réel amplifiant dans la région 5′NC (Tableau 5 ).
Tableau 5.
Analyse de 22 des 37 SN positives en PEV par la trousse Enterovirus R-gene Argène.
| Enterovirus R-gene | PEVRV | ||||
|---|---|---|---|---|---|
| Non réalisée | Positive | ||||
| EV | EV-RV | RV | Total | ||
| 10 | 10 | 5 | 12 | 37 | |
| Non réalisé | 3 | 10 | 0 | 2 | 15 |
| Positive | 7 | 0 | 5 | 9 | 21 |
| Négative | 0 | 0 | 0 | 1 | 1 |
Les chiffres en gras correspond aux résultats des 22 SN testées par la trousse Enterovirus R-gene Argene.
3.3. Distribution saisonnière des entérovirus
La recherche d’EV réalisée au cours de l’année 2008 sur différents prélèvements biologiques (3612 SN, 1273 selles et 307 LCR) a permis d’identifier respectivement 96 (2,6 %), 27 (2,1 %) et 24 EV (7,8 %). La répartition saisonnière des EV dans les prélèvements de SN et LCR est représentée dans la Fig. 3 . Pour les 23 LCR positifs, nous retrouvons le classique pic estival d’infections méningées (58 % en juin à juillet) ; 52 % des patients sont des enfants (moyenne d’âge = 8,8 ans), 22 % des NN (m = 44 jours) et 26 % des adultes jeunes (m = 35,5 ans). Concernant les 95 SN positives, nous observons un petit pic estival (23 %) et un pic plus important (42 %) en octobre à novembre ; 59 % des patients concernés sont des enfants (m = 2 ans), 34 % des NN (m = 37 jours) et 7 % des adultes (m = 65,7 ans). Sept enfants ont eu une identification conjointe d’EV dans les SN et LCR (deux en juin, deux en juillet, deux en octobre et un en novembre).
Fig. 3.
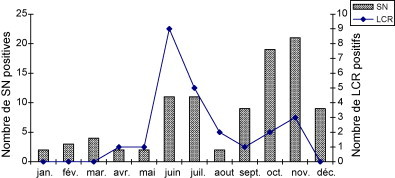
Répartition saisonnière des EV isolés dans les SN et LCR, Caen 2008.
La Fig. 4 montre notre fréquence d’isolement des EV par type de prélèvements en comparaison aux chiffres nationaux publiés par le réseau de surveillance des EV ainsi que la distribution comparée des cas par semaine (institut de veille sanitaire et Centre national de référence pour le EV) [25].
Fig. 4.
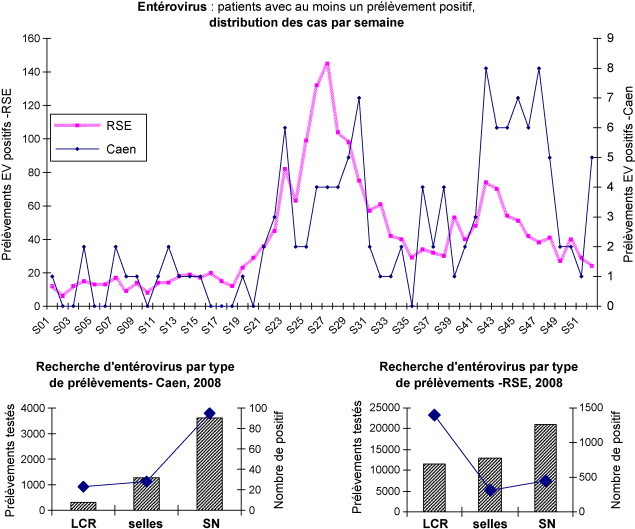
Entérovirus : distribution des cas par semaine et fréquence d’isolements par type de prélèvements – laboratoire virologie Caen et RSE (InVs).
3.4. Génotypage des EV
Le génotypage a été réalisé chez 73 des 96 patients présentant une identification d’EV sur surnageant de culture MRC5 ou surnageant de culture HuH7 (PEVRV positive). Pour cinq surnageants, l’extrait d’ARN n’a pu être amplifié (absence de bande à la PCR nichée) et l’amplification a été possible pour 68 isolats. Le génotypage a été réalisé dans 97 % des cas par séquençage du segment obtenu après la PCR nichée (amorces AN88/AN89, 361 pb), deux souches EV 71 et HRV59 ont été identifiées sur le premier segment amplifié (amorces 222/224, 992 pb). Les 68 génotypes identifiés sont présentés dans le Tableau 6 avec les résultats respectivement obtenus en culture MRC5 et en PEVRV. Les cinq génotypes majoritairement identifiés sont 19 (29,7 %) EV68 (EV68), 12 (18,7 %) coxsackievirus B3 (CB3), neuf (14,1 %) échovirus 3 (E3), six (9,4 %) coxsackievirus A9 (CA9) et six (9,4 %) CB1 ; quatre RV ont été identifiés : HRV59, HRV43, HRV46 et 1 HRV type C. Les 19 EV68 sont isolés entre septembre et décembre, dont 52,6 % au mois de novembre. Dix-sept patients présentent des signes d’insuffisance respiratoire aiguë avec atteintes des voies aériennes inférieures (six bronchiolites, cinq bronchites, cinq pneumopathies, une bronchopneumopathie chronique obstructive « BPCO ») ; sept patients présentent une crise d’asthme sévère.
Tableau 6.
Résultats de la culture MRC5 et PEVRV sur les 73 souches EV génotypées.
| MRC5 | PEVRV-HuH7 | |||||||
|---|---|---|---|---|---|---|---|---|
| n | POS | NEG | EV | EV-RV | RV | NEG | NR | |
| HEV-A 1CA10, 2 CA16, 2 EV71 |
5 | 5 | 0 | 1 | 1 | 0 | 2 | 1 |
| HEV-B | ||||||||
| 6CA9 | 6 | 6 | 0 | 1 | 0 | 0 | 5 | 0 |
| 6CB1, 12CB3, 1CB5 | 19 | 7 | 12 | 17 | 1 | 0 | 0 | 1 |
| 2E25, 9E3, 4E30 | 15 | 15 | 0 | 8 | 0 | 0 | 6 | 1 |
| HEV-D | ||||||||
| EV68 | 19 | 1 | 18 | 14 | 4 | 0 | 1 | 0 |
| HRV | 4 | 0 | 4 | 0 | 1 | 2 | 1 | 0 |
| 3 HRVA et 1 HRVC | ||||||||
| Non typablea | 5 | 0 | 5b | 1 | 0 | 4 | 0 | 0 |
Génotypage : PCR nichée négative.
Quatre négatif et un RV.
Les principaux types d’EV identifiés à Caen chez 64 patients sont comparés aux résultats recensés par le RSE au niveau national chez 589 patients dans la Fig. 5 .
Fig. 5.
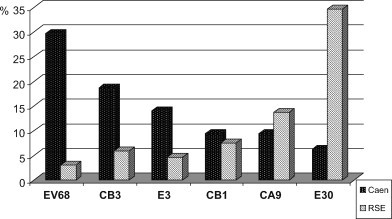
Principaux types d’EV identifiés au cours de l’année 2008. RSE-InVs (589 patients) et Caen (64 patients).
4. Discussion
4.1. Intérêt du diagnostic moléculaire pour la détection des EV
En pratique courante, le diagnostic des infections respiratoires à EV fait appel le plus souvent aux méthodes conventionnelles de culture cellulaire. Il est bien connu que l’utilisation du seul système cellulaire MRC5 pénalise les isolements d’EV et sous-estime très fortement l’implication des EV en pathologie respiratoire. Aucune lignée cellulaire ne permet d’isoler tous les types connus d’EV et les performances de la culture sont améliorées par l’emploi de plusieurs lignées d’origine différente, en privilégiant au moins une lignée simienne (cellules de rein singe : VERO, BGM…). Dans notre laboratoire, a été mis en place un algorithme de recherches virales dans les prélèvements respiratoires (aspirations nasales et sécrétions nasales) avec l’introduction d’un second système cellulaire (cellules HuH7) particulièrement adapté aux isolements des myxovirus et paramyxovirus, et l’introduction en deuxième intention sur les surnageants de HuH7 présentant une lyse, de plusieurs techniques de biologie moléculaire ciblant divers virus. Parmi ces techniques, figure la PCR duplex PEVRV permettant le diagnostic simultané et différentié des EV et RV. Il est actuellement bien reconnu que les techniques PCR améliorent le diagnostic et la prise en charge des méningites virales à EV et nous venons de démontrer que l’addition de deux techniques PCR, séquentielle (PEVRV) ou ciblée (PEV chez les NN), améliore également grandement la sensibilité du diagnostic des infections respiratoires à EV (et de RV, données non montrées) [26], [27]. Au cours de l’année 2008, nous avons pratiquement triplé le nombre d’isolement d’EV. Les techniques de biologie moléculaire permettent de s’affranchir d’un certain nombre de problèmes pénalisant la sensibilité : problème de qualité (richesse cellulaire), de transport et de conservation du prélèvement. De plus, à côté du manque de sensibilité de la culture cellulaire, s’ajoute l’impossibilité pour certains sérotypes d’EV à cultiver (coxsackievirus, EV68 à 71) [28].
Chez les 327 SN de NN, la recherche systématique d’EV par PEV a permis de les détecter rapidement (3e jour) avec une prévalence de 11,3 %, alors que la culture cellulaire ne permettait d’isoler que huit EV et un RV (2,4 %), avec un délai de six à 22 jours. Aucune infection néonatale grave n’a été détectée parmi ces 37 cas, mais le rendu rapide du diagnostic d’infection bénigne à EV peut contribuer à rassurer et faciliter la prise en charge des NN.
Selon l’algorithme de recherche virale sur les SN des NN, la PEVRV a été réalisée conjointement à la PEV chez 98 NN (Tableau 4). Parmi les 27 SN positives en PEV, dix sont positives en EV, cinq sont positives en EV-RV et 12 sont positives en RV ; sur ces 22 SN, 21 sont contrôlées positives (concordance 95,5 %) par la trousse PCR Enterovirus R-gene, Argène. Ces résultats montrent que ces deux PCR (ciblant la région 5′NC) choisies pour le diagnostic des EV détectent également un certain nombre de RV. Lors de l’optimisation de notre technique PEV « maison », n’avaient été testées que deux souches prototypes (HRV9 et HRV31) et, concernant la trousse Enterovirus R-gene, il est signalé dans la notice du fabricant qu’elle ne détecte pas les HRV9, HRV39 et HRV89 mais que sont détectés les HRV14, HRV17 et HRV22 [18]. En général, les techniques PCR EV, qu’elles soient « maison » ou commercialisées sous forme de trousses, sont utilisées dans un cadre d’infections méningées ; toutefois, leur validation sur des prélèvements respiratoires doit remettre en question le rendu des résultats non pas sous le terme de présence d’EV mais de « picornavirus ». Les rhinovirus sont connus depuis longtemps comme présentant une grande diversité génomique. La classification initialement établie sur la base de culture cellulaire suivie de tests de séroneutralisation identifiait 102 sérotypes humains. Par la suite, les techniques moléculaires de séquençage dans diverses parties du génome (VP4-VP2, VP1) ont permis de classer en deux clusters principaux RV-A et RV-B les 102 sérotypes connus de RV à l’exception d’un seul, le RV 87, qui est isolé en un mini cluster intégrant l’EV70 [29]. Très rapidement, la même équipe démontre la très grande similitude entre le RV87 et l’EV68 et les trois espèces RV87, EV68 et EV70 sont rassemblées dans l’espèce HEV-D [9]. Enfin, plus récemment, l’ensemble des analyses phylogénétiques basées sur le séquençage des différentes régions génomiques codant pour les protéines de capside (VP1, VP1-2A, VP3-VP1, VP2, VP4-VP2) ont permis d’établir que la séparation des RV et EV était de moins en moins fondée [7], [8], [10], [11], [12]. Des relations entre les différents virus humains et animaux de la famille des Picornaviridae ont été reconnues, conduisant à une évolution constante de la taxonomie de ces virus. Actuellement, les deux genres EV et RV ne sont plus dissociés ; il existe un seul genre Enterovirus regroupant les six espèces humaines : Human Enterovirus A (HEVA), HEVB, HEVC, HEVD, Human Rhinovirus A (HRVA), HRVB, et HRVC.
4.2. Circulation des EV : intérêt du génotypage moléculaire pour identifier les souches d’EV
Virus nus à ARN, à fort pouvoir de réplication, les EV circulent abondamment dans les collectivités humaines et animales sous plusieurs sérotypes différents conditionnant la survenue d’épidémies et leur grande variabilité génétique [30].
Le profil épidémique des infections à EV et la circulation des sérotypes sont très variables d’une année à l’autre. La surveillance épidémiologique est indispensable pour observer l’évolution au cours du temps des EV non poliomyélitiques connus, identifier les variants et l’émergence de nouveaux types en relation avec les données cliniques et étudier l’émergence de nouvelles pathologies. Cette surveillance est réalisée au niveau national sous forme d’un réseau de surveillance des EV (RSE) grâce à la collaboration entre le Centre national de référence des EV (CNR Lyon-B. Lina) et l’institut de veille sanitaire (InVs-D. Antona) qui regroupent les données fournies par les différents laboratoires de virologie médicale. Chaque année, depuis 2000, les bilans d’activité permettent d’attester l’absence de circulation de poliovirus sauvage, de chiffrer la fréquence de détection des EV dans les échantillons cliniques (LCR, selles, SN), d’authentifier le pic estival de méningite et d’identifier les principaux EV circulants avec un léger bémol du fait d’un nombre restreint de souches typées [31]. Toutefois, ces dernières années, le typage des souches progresse avec principalement l’introduction du génotypage moléculaire. La plupart des méthodes de génotypage sont basées sur l’amplification et le séquençage d’une partie du gène codant pour la protéine de capside VP1, région hypervariable codant pour la majorité des épitopes neutralisants [32], [33], [34], [35]. Peu sensibles, elles sont en général appliquées rétrospectivement sur des surnageants de culture ne reflétant qu’incomplètement les différents sérotypes d’EV circulants. Le génotypage direct des EV dans les prélèvements cliniques (LCR, nasopharynx, sérum, biopsies) commence à se développer ; il présente l’avantage d’identifier des souches d’EV non cultivables (charge virale faible, sérotypes non cultivables) et de répondre rapidement de manière prospective en apportant une valeur pronostique dans les infections graves à EV (infections néonatales, encéphalites, myocardites) [23], [36]. La technique utilisée au laboratoire, basée sur la stratégie consensus degenerate hybride oligonucleotide primers (CODEHOP), permet une diminution des bandes non spécifiques, visualisées fréquemment avec l’utilisation d’amorces dégénérées. La sensibilité de cette PCR nichée permet, théoriquement, l’identification directe dans les prélèvements d’un vaste nombre de génotypes relativement distants au sein des enterovirus [23]. En termes de faisabilité au laboratoire, le génotypage est pour l’instant réalisable dans une démarche rétrospective et de rendu optimal sur les surnageants de culture. Les problèmes de sensibilité observés sur les prélèvements de LCR à faible charge virale sont en cours de résolution avec une optimisation de la technique référencée selon nos conditions opératoires.
Au niveau régional, la distribution par semaine des cas positifs montre pour l’année 2008 un premier pic estival (juin à juillet) et un second pic automnal (octobre à novembre), ce second pic étant nettement plus important que celui observé au niveau national par le RSE (Fig. 4). Alors que le pic estival correspond aux syndromes méningés observés habituellement à cette époque (60 % des LCR EV positif), le pic automnal correspond à une pathologie respiratoire (42 % des SN positives). Bien que l’existence de méningites à EV ait été déjà décrite en hiver sous formes d’épidémies ou de cas sporadiques, nous pouvons l’exclure à notre niveau régional puisque seulement cinq LCR étaient positif en EV [37], [38]. En revanche, nous retrouvons le caractère épidémique des infections respiratoires virales. Alors que les deux principaux virus épidémiques connus et recherchés, VRS et virus influenza A/B, ont une diffusion hivernale majeure, les rhinovirus (picornavirus) circulent plus volontiers en automne [39]. Dans notre pratique quotidienne, la recherche d’EV par PEVRV n’a été faite qu’en procédure dégradée (22,6 % des SN, Fig. 2), sous-estimant probablement l’identification d’EV et de RV. L’arrivée sur le marché de trousses PCR multiplex en temps réel va permettre d’appliquer plus facilement cette recherche systématique de panel respiratoire sur les SN [40]. Il restera à définir, dans un contexte de pathologie respiratoire, s’il faut encore différencier les EV et RV. Dans la nouvelle taxonomie, la séparation phylogénique des RV n’existe plus : ils appartiennent au genre EV et notre étude sur les SN a démontré que les PCR temps réel ciblant la 5′NC ne peuvent identifier spécifiquement EV ou RV. Par conséquent, dans un contexte d’infections respiratoires, il semblerait plus judicieux de rendre « présence de picornavirus ».
L’examen des six EV les plus souvent identifiés au cours de l’année 2008 montre une similitude au niveau régional et national pour cinq d’entre eux : CB3, CB1, E3, CA9 et E30 ; seul l’EV68 se différentie par son identification en tête de liste à Caen avec 29,7 % contre 3,1 % par le RSE (Fig. 5) [25].
En conclusion, le génotypage des souches d’EV d’origine respiratoire a permis de reformuler au niveau régional la circulation des souches d’EV.
4.3. Place de l’EV68 en pathologie respiratoire
La recherche d’EV par des techniques PCR (PEV, PEVRV, génotypage) sur environ le quart des SN reçues au laboratoire a permis de démontrer l’existence d’une petite épidémie d’infections respiratoires à EV68 au cours de l’automne 2008. Parmi les 19 EV68 identifiés, un seul a été isolé en culture cellulaire MRC5, confirmant l’importance des outils PCR pour améliorer le diagnostic de ces infections. Parmi les 19 patients positifs en EV68, 13 (68 %) avaient une atteinte des voies aériennes inférieurs (six bronchiolites, quatre bronchites, deux BPCO, cinq pneumopathies) et sept patients (37 %) présentaient une crise d’asthme sévère (données non montrées). Chez ces 19 patients, trois patients présentent une co-infection virale (deux adénovirus, un coronavirus HKU1) et huit ont eu une recherche de Mycoplasma pneumoniae négative. Principal agent des infections des voies aériennes supérieures, les RV sont bien connus pour leur diffusion dans les voies aériennes inférieures et leur rôle dans le déclenchement de crises d’asthme [41]. De plus, phylogénétiquement nous savons que l’EV68 est très proche des RV (RV87) [9].
L’EV68 n’est que très rarement répertorié dans les bilans d’activité du RSE, probablement du fait que c’est un EV non ou mal cultivable et que la PCR n’est pas de pratique courante sur les prélèvements respiratoires. L’EV68 semble être un sérotype particulièrement relié à un tropisme respiratoire et son implication réelle dans les infections respiratoires basses devra être confirmée par des investigations plus complètes en matière de recherche par PCR et de génotypage direct.
5. Conclusions
Les méthodes de diagnostic moléculaire des infections respiratoires à EV (PCR et génotypage) sont très fiables et permettent une identification précise des souches circulantes dans un temps relativement court. Elles permettent ainsi d’affiner les données taxonomiques, épidémiologiques et cliniques concernant ces infections et de repérer l’émergence de certains sérotypes en pathologie humaine. Grâce aux développements récents, de nombreuses trousses de techniques multiplex sur automates de PCR en temps réel, on peut imaginer pouvoir les utiliser en pratique quotidienne prospective dans un laboratoire hospitalier de virologie avec des bénéfices cliniques, épidémiologiques et économiques de santé en terme de durée d’hospitalisation et de consommation d’actes paracliniques et/ou de médicaments.
Conflit d’intérêt
Aucun.
Remerciements
Nous remercions Denise Antona pour nous avoir fourni l’information détaillée de la distribution hebdomadaire des patients présentant au moins un prélèvement positif au sein du RSE, 2008.
Références
- 1.Rotbart H.A. Enteroviral infections of the central nervous system. Clin Infect Dis. 1995;20:971–981. doi: 10.1093/clinids/20.4.971. [DOI] [PubMed] [Google Scholar]
- 2.Peigue-Lafeuille H., Archimbaud C., De Champs C., Croquez N., Laurichesse H., Clavelou P. Les méningites à entérovirus chez les adultes. Pathologie sous-estimée ? À propos de 30 observations de 1999 à 2000 et évolution des pratiques médicales en 2001. Pathol Biol. 2002;50:516–524. doi: 10.1016/s0369-8114(02)00348-6. [DOI] [PubMed] [Google Scholar]
- 3.Chang L.Y., Huang L.M., Gau S.S.F., Wu Y.Y., Hsia S.H., Fan T.Y. Neurodevelopment and cognition in children after enterovirus 71 infection. N Engl J Med. 2007;356:1226–1234. doi: 10.1056/NEJMoa065954. [DOI] [PubMed] [Google Scholar]
- 4.Huang Y.F., Chiu P.C., Chen C.C., Chen Y.Y., Hsieh K.S., Liu Y.C. Cardiac troponin I: a reliable marker and early myocardial involvement with meningoencephalitis after fatal Enterovirus 71 infection. J Infect. 2003;46:238–243. doi: 10.1053/jinf.2002.1117. [DOI] [PubMed] [Google Scholar]
- 5.Freymuth F., Vabret A., Petitjean J., Gouarin S., Gueudin M., Campet M. Diagnostic des deux principales viroses respiratoires épidémiques : la grippe et les infections à virus respiratoire syncytial. Place de la virologie moléculaire. Med Mal Infect. 2000;30:191–201. [Google Scholar]
- 6.Bellau-Pujol S., Vabret A., Legrand L., Dina J., Gouarin S., Petitjean-Lecherbonnier J. Development of three multiplex RT-PCR assays for the detection of 12 respiratory RNA viruses. J Virol Methods. 2005;126:53–63. doi: 10.1016/j.jviromet.2005.01.020. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Bailly J.L., Mirand A., Henquell C., Archimbaud C., Chambon M., Charbonné F. Génotypage et épidémiologie moléculaire des entérovirus non poliomyélitiques. Virologie. 2008;12(1):53–65. doi: 10.1684/12-1.2011.10007. [DOI] [PubMed] [Google Scholar]
- 8.Laine P., Savolainen C., Blomqvist S., Hovi T. Phylogenetic analysis of human rhinovirus capsid protein VP1 and 2A protease coding sequences confirms shared genus-like relationships with enteroviruses. J Gen Virol. 2005;86:697–706. doi: 10.1099/vir.0.80445-0. [DOI] [PubMed] [Google Scholar]
- 9.Blomqvist S., Savolainen C., Raman L., Roivainen M., Hovi T. Human rhinovirus 87 and Enterovirus 68 represent a unique serotype with rhinovirus and enterovirus features. J Clin Microbiol. 2002;40:4218–4223. doi: 10.1128/JCM.40.11.4218-4223.2002. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Tapparel C., Junier T., Gerlach D., Van Belle S., Turin L., Cordey S.. New respiratory enterovirus and recombinant rhinoviruses among circulating picornaviruses. Emerg Infect Dis. 2009;15(5):719–720. doi: 10.3201/eid1505.081286. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Stanway G., Brown P., Christian P. Family Picornaviridae. In: Fauquet C.M., Mayo M.A., Maniloff J., Desselberger U., Ball L.A., editors. Virus taxonomy, 8th report of the international committee on taxonomy of viruses. Elsevier Academic Press; Amsterdam, The Netherlands: 2005. p. 757–78. [Google Scholar]
- 12.Carstens E.B. Ratification vote on taxonomic proposals to the International Committee on Taxonomy of Viruses (2009) Arch Virol. 2010;155:133–146. doi: 10.1007/s00705-009-0547-x. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Kairis B., Sauter P., Goffard A., Fronval S., Sane F., Hober D. Quand un entérovirus émerge. Pathol Biol. 2009;57:258–267. doi: 10.1016/j.patbio.2008.06.001. [DOI] [PubMed] [Google Scholar]
- 14.Bailly J.L., Chambon M., Henquell C., Icart J., Peigue-Lafeuille H. Genomic variations in echovirus 30 persistent isolates recovered from a chronically infected immunodeficient child and comparison with the reference strain. J Clin Microbiol. 2000;38:552–557. doi: 10.1128/jcm.38.2.552-557.2000. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Brown B., Oberste M.S., Maher K., Pallansch M.A. Complete genomic sequencing shows that poliovirus and members of human enterovirus species C are closely related in the noncapsid coding region. J Virol. 2003;77:8973–8994. doi: 10.1128/JVI.77.16.8973-8984.2003. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Oberste M.S., Maher K., Pallansch M.A. Evidence for frequent recombination within species human enterovirus B based on complete genomic sequences of all thirty-seven serotypes. J Virol. 2004;78:855–867. doi: 10.1128/JVI.78.2.855-867.2004. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Vabret A., Mouthon F., Mourez T., Gouarin S., Petitjean J., Freymuth F. Direct diagnosis of human respiratory coronaviruses 229E and OC43 by the polymerase chain reaction. J Virol Methods. 1997;1–2:59–66. doi: 10.1016/S0166-0934(01)00343-3. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Petitjean J., Vabret A., Dina J., Gouarin S., Freymuth F. Development and evaluation of a real-time RT-PCR assay on the LightCycler for the rapid detection of enterovirus in cerebrospinal fluid specimens. J Clin Virol. 2006;35:278–284. doi: 10.1016/j.jcv.2005.09.006. [DOI] [PubMed] [Google Scholar]
- 19.Freymuth F., Vabret A., Rozenberg F., Dina J., Petitjean J., Gouarin S. Replication of respiratory viruses, particularly influenza virus, rhinovirus, and coronavirus in HuH7 hepatocarcinoma cell line. J Med Virol. 2005;77:295–301. doi: 10.1002/jmv.20449. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Savolainen C., Mulders M.N., Hovi T. Phylogenetic analysis of rhinovirus isolates collected during successive epidemic seasons. Virus Res. 2002;85:41–46. doi: 10.1016/s0168-1702(02)00016-3. [DOI] [PubMed] [Google Scholar]
- 21.Olive D.M., Al-Mufti S., Al-Mulla W., Khan M.A., Pasca A., Stanway G. Detection and differentiation of picornaviruses in clinical samples following genomic amplification. J Gen Virol. 1990;71:2141–2147. doi: 10.1099/0022-1317-71-9-2141. [DOI] [PubMed] [Google Scholar]
- 22.Verstrepen W.A., Kuhn S., Kockx M.M., Van De Vyvere M.E., Mertens A.H. Rapid detection of enterovirus RNA in cerebrospinal fluid specimens with a novel single-tube real-time reverse transcription-PCR assay. J Clin Microbiol. 2001;39:4093–4096. doi: 10.1128/JCM.39.11.4093-4096.2001. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.Nix W.A., Oberste M.S., Pallansch M.A. Sensitive, semi-nested PCR amplification of VP1 sequences for direct identification of all enteroviruses serotypes from original clinical specimens. J Clin Microbiol. 2006;44:2698–2704. doi: 10.1128/JCM.00542-06. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.Altschul D.F., Gish W., Miller W., Meyers E.W., Lipman D.J. Basic local alignment search tool. J Med Biol. 1990:215–403. doi: 10.1016/S0022-2836(05)80360-2. [DOI] [PubMed] [Google Scholar]
- 25.Réseau de surveillance des entérovirus-bilan de l’activité au cours de l’année 2008. D. Antona, Institut de veille sanitaire, département des maladies infectieuses, St.-Maurice, France & B. Lina, Centre national de référence pour les entérovirus, centre de biologie-pôle est, Bron, France.
- 26.Romero J.R. Reverse-transcription polymerase chain reaction detection of the enteroviruses. Overview and clinical utility in pediatric enteroviral infections. Arch Pathol Lab Med. 1999;123:1161–1169. doi: 10.5858/1999-123-1161-RTPCRD. [DOI] [PubMed] [Google Scholar]
- 27.Ramers C., Billman G., Hartin M., Ho S., Sawyer M.H. Impact of a diagnostic cerebrospinal fluid enterovirus polymerase chain reaction test on patient management. JAMA. 2000;283:2680–2685. doi: 10.1001/jama.283.20.2680. [DOI] [PubMed] [Google Scholar]
- 28.Melnick J.L. Enteroviruses: polioviruses, coxsakieviruses, echoviruses, and newer enteroviruses. In: Fields B.N., Knipe D.M., Howley P.M., editors. Fields Virology. 3rd ed. Lippincott-Raven Publishers; Philadelphia, Pa: 1996. pp. 655–712. [Google Scholar]
- 29.Savolainen C., Blomqvist S., Mulders M.N., Hovi T. Genetic clustering of all 102 human rhinovirus prototype strains: serotype 87 is close to human enterovirus 70. J Gen Virol. 2002;83:333–340. doi: 10.1099/0022-1317-83-2-333. [DOI] [PubMed] [Google Scholar]
- 30.Bailly J.L., Chambon M., Peigue-Lafeuille H. Diversité et variabilité des entérovirus. Virologie. 1999;3:281–295. [Google Scholar]
- 31.Antona D., Levêque N., Chomel J.J., Dubrou S., Lévy-Bruhl D., Lina B. Surveillance of enteroviruses in France, 2000–2004. Eur J Clin Microbiol Infect Dis. 2007;26:403–412. doi: 10.1007/s10096-007-0306-4. [DOI] [PubMed] [Google Scholar]
- 32.Oberste M.S., Maher K., Kilpatrick D.R., Flemister M.R., Brown B.A., Pallansch M.A. Typing of human enteroviruses by partial sequencing of VP1. J Clin Microbiol. 1999;37:1288–1293. doi: 10.1128/jcm.37.5.1288-1293.1999. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 33.Caro V., Guillot S., Delpeyroux F., Rainic R. Molecular strategy for ‘serotyping’ of human enteroviruses. J Gen Virol. 2001;82:79–91. doi: 10.1099/0022-1317-82-1-79. [DOI] [PubMed] [Google Scholar]
- 34.Norder H., Bjerregaard L., Magnius L., Lina B., Aymard M., Chomel J.J. Sequencing of ‘untypable’ enteroviruses reveals two new types. EV-77 and EV-78, within human enterovirus type B and substitutions in the BC loop of the VP1 protein for known types. J Gen Virol. 2003;84:827–836. doi: 10.1099/vir.0.18647-0. [DOI] [PubMed] [Google Scholar]
- 35.Mirand A., Archimbaud C., Henquell C., Michel Y., Chambon M., Peigue-Lafeuille H. Prospective identification of HEV-B enteroviruses duting the 2005 outbreak. J Med Virol. 2006;78:1624–1634. doi: 10.1002/jmv.20747. [DOI] [PubMed] [Google Scholar]
- 36.Mirand A., Bailly J.L., Henquell C., Peigue-Lafeuille H. Génotypage direct rapide des entérovirus dans le liquide céphalorachidien : étude prospective de deux ans au sein d’un laboratoire de virologie clinique. Pathol Biol. 2008;56:471–481. doi: 10.1016/j.patbio.2008.08.005. [DOI] [PubMed] [Google Scholar]
- 37.Mori I., Matsumoto K., Hatano M., Sudo M., Kimura Y. An unseasonable winter outbreak of echovirus type 30 meningitis. J Infect. 1995;31:219–223. doi: 10.1016/s0163-4453(95)80030-1. [DOI] [PubMed] [Google Scholar]
- 38.Chambon M., Archimbaud C., Bailly J.L., Henquell C., Regagnon C., Charbonné F. Circulation of enteroviruses and persistence of meningitis cases in winter 1999–2000. J Med Virol. 2001;65:340–347. doi: 10.1002/jmv.2039. [DOI] [PubMed] [Google Scholar]
- 39.Freymuth F., Quibriac M., Petitjean J., Daon F., Amiel M.L. Les virus responsables d’infection respiratoire en pédiatrie. Bilan de 3480 aspirations nasales réalisées chez l’enfant en une période de six ans. Ann Pediatr. 1987;34:493–501. [PubMed] [Google Scholar]
- 40.Reijans M., Dingemans G., Klaassen C.H., Meis J.F., Keijdener J., Mulders B. RespiFinder: a new multiparameter test to differentially identify fifteen respiratory viruses. J Clin Microbiol. 2008;46:1232–1240. doi: 10.1128/JCM.02294-07. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 41.Billaud G., Lina B. Rhinovirus : principal agent des infections aériennes supérieures. Virologie. 2004;8:271–280. [Google Scholar]


