Einleitung
Kinder machen etwa 5 – 6 akute Atemwegserkrankungen im Jahr durch, die mit einer obstruktiven Klinik einhergehen. Es gibt Hinweise, dass genetische Faktoren des Wirts symptomatische Infektionen mit Respirationstraktpathogenen begünstigen, z. B. ein einzelner Nukleotidaustausch im Interleukin-4-Gen 1 .
Merke.
Die große Mehrheit der Atemwegsinfektionen wird in Mitteleuropa von Viren ausgelöst. Trotzdem werden viele Patienten mit Antibiotika behandelt.
Risiken bei Infektion
Virusinfektionen des Respirationstrakts verändern die Zusammensetzung des Bakterioms des oberen Teils und begünstigen die bakterielle Kolonisierung des unteren Respirationstrakts. Beides erleichtert die Entwicklung einer Pneumonie im Rahmen einer bakteriellen Superinfektion 2 . Infektionen mit viralen Respirationstraktpathogenen können auch Erkrankungen beeinflussen, die sich außerhalb des Respirationstrakts manifestieren. So wurde eine Assoziation von Infektionen des oberen Respirationstrakts mit Schüben der Multiplen Sklerose postuliert 3 .
Nachweis neuer respiratorischer Viren
Die Erregerdiagnostik bei Atemwegsinfektionen führt häufig nicht zu einem Erregernachweis. Die beobachtete diagnostische Lücke bei Atemwegsinfektionen wurde in den letzten Jahren durch 2 Entwicklungen verkleinert: Zum einen durch die stärkere Verbreitung sensitiver Verfahren, die ein großes Spektrum viraler Erreger von Atemwegsinfektionen sowie bakterieller Erreger atypischer Pneumonien umfassen. Zum anderen durch die Entdeckung neuer viraler Respirationstraktpathogene. Die Gruppe der seit Langem bekannten relevanten viralen Respirationstraktpathogenen aus dem Formenkreis der Influenzaviren, Rhinoviren, Adenoviren, Enteroviren, Parainfluenzaviren und respiratorischen Synzytialviren (RSV) wurde in den letzten 20 Jahren um mehrere Erreger vergrößert. Neu entdeckt wurden:
Eine regelmäßig nachweisbare Linie des Influenza-B-Virus, für die keine Kreuzprotektion besteht.
Mehrere Vogelgrippeviren, die humane Infektionen verursachen können.
Das variante Influenza-A-Virus H1N1, das die Pandemie von 2009 ausgelöst hat (Schweinegrippe).
humanes Metapneumovirus
4 Coronaviren (HCoV-NL63, HCoV-HKU1, SARS-CoV und MERS-CoV)
humanes Bocavirus
Rhinoviren (Spezies Rhinovirus C)
Für andere, bereits bekannte Viren wurde ein größeres pathogenes Potenzial festgestellt (Rhinoviren, Enteroviren).
Merke.
Zur Orientierung über die jeweils aktuell in Deutschland zirkulierenden respiratorischen Viren und deren typische Saisonalität steht seit einigen Jahren der Internetauftritt des Netzwerks für respiratorische Viren zur Verfügung ( http://rvdev.medical-dpc.com/inhalte/start-viren.html ; Abb. 1 ).
Abb. 1.
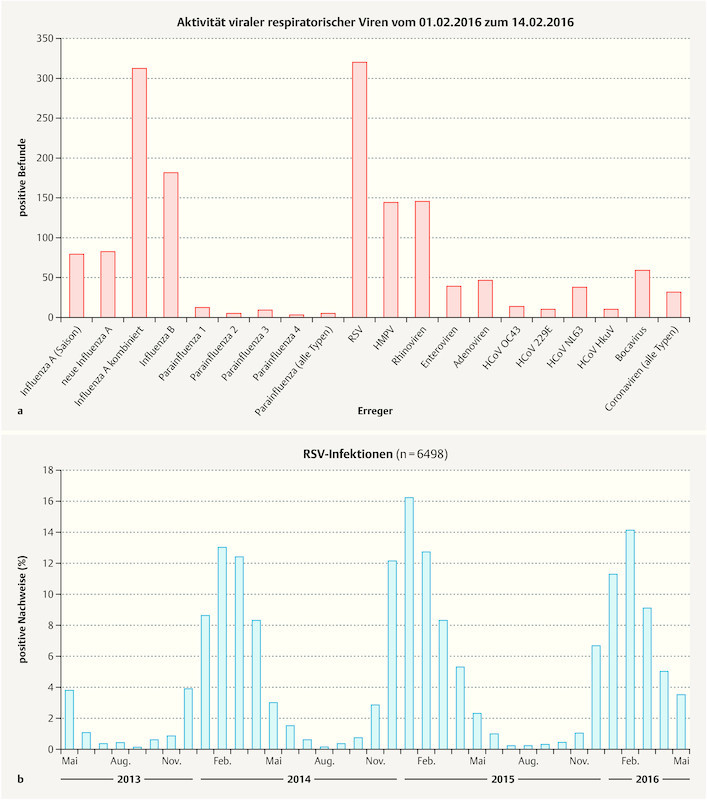
Positive Nachweise respiratorischer Viren in Deutschland für den Zeitraum 1.2. – 14.2.2016 ( a ). Saisonalität von RSV zwischen 2013 und 2016 ( b ). Die Daten wurden durch das Netzwerk für respiratorische Viren erhoben ( http://rvdev.medical-dpc.com/inhalte/start-viren.html ).
Diagnostisches Vorgehen
Früher bewährte Verfahren (z. B. Virusanzucht oder Antikörpernachweis) sind nicht mehr Methode der Wahl, weil sie zeitaufwendig und nicht für alle Erreger im Routinelabor verfügbar sind (Virusanzucht) oder weil Sensitivität und Spezifität für den Nachweis akuter Infektionen nicht ausreichen (Antikörpernachweis). Heutzutage sind Virusdirektnachweise durch Nukleinsäureamplifikationsverfahren, z. B. Polymerasekettenreaktion (PCR) oder isothermale Amplifikationsverfahren, die diagnostischen Methoden der Wahl für den Nachweis respiratorischer Viren.
Merke.
Die Bedeutung der entsprechenden Viren ließ sich nur durch die Anwendung moderner molekularer Methoden erkennen, und sie werden auch am besten durch molekulare Verfahren diagnostisch erfasst.
Neue Nachweisverfahren
In den letzten Jahren hat es eine Weiterentwicklung der verfügbaren Technologien (ausgehend von der für ein einzelnes Virus spezifischen Standard-PCR mit einem Zeitbedarf von 2 – 3 h) in 2 Richtungen gegeben: Zum einen sind nun Multiplexverfahren verfügbar, die organsystemorientiert alle relevanten Erreger für ein definiertes Krankheitsbild in einem Reaktionsansatz nachweisen können, z. B. für bis zu 19 verschiedene respiratorische Viren. Zum anderen sind Point-of-Care-Systeme entwickelt worden, die in wenig störanfälligen geschlossenen Systemen den Nachweis von Viren in sehr kurzer Zeit ermöglichen und geeignet sind für nicht speziell in molekularen Verfahren geschultes Personal.
Merke.
Der Nachweis von Influenzaviren durch Nukleinsäureamplifikation ist mittlerweile in einer Reaktionszeit von 15 min möglich.
Die Bedeutung, die Next-Generation-Sequencing-Verfahren für das Routinelabor gewinnen werden, lässt sich noch nicht abschließend beurteilen 4 , 5 .
Entwicklung von Impfstoffen und Therapeutika
Parallel zur Entdeckung weiterer viraler Respirationstraktpathogene hat es auch Fortschritte im Bereich der Entwicklung antiviraler Wirkstoffe sowie Impfstoffen gegeben. Davon hat die Möglichkeit zum Management von Influenzavirusinfektionen am meisten profitiert. Aber auch für das Management von RSV-Infektionen und Infektionen mit anderen respiratorischen Viren deuten sich substanzielle Fortschritte an. So sind mittlerweile 16 Impfstoffkandidaten für RSV in klinischer Entwicklung. Zusätzlich sind vielversprechende spezifisch antiviral wirkende Substanzen in der Entwicklung, u. a. monoklonale Antikörper 6 , 7 .
Influenzaviren
Biologie
Virustypen
Influenzavirusinfektionen lassen sich weltweit nachweisen. Sie werden ausgelöst durch den Influenza-A-, Influenza-B- und Influenza-C-Virus aus der Familie der Orthomyxoviren. Die genannten Virustypen werden aufgrund der Sequenzinformation des viralen Ribonukleoproteins unterschieden. Influenza-C-Viren sind überwiegend humanspezifisch, wurden aber auch aus Schweinen isoliert 8 . Sie haben im Vergleich zu Influenza-A- und Influenza-B-Viren ein reduziertes pathogenes Potenzial. Auch Influenza-B-Viren lassen sich überwiegend in Menschen nachweisen, aber auch in Seehunden und Pferden 8 . Influenza-A-Viren dominieren in verschiedenen Wasservogelarten, können aber auch Säugetierspezies infizieren. Dazu gehören neben dem Menschen u. a. Pferde, Schweine und Seehunde. Influenza-A-Viren teilt man anhand der Sequenz ihrer Oberflächenantigene Hämagglutinin (H) und Neuraminidase (N) in Subtypen (z. B. H1N1) ein: man unterscheidet 18 H-Varianten und 11 N-Varianten 9 . Die meisten der denkbaren Kombinationsmöglichkeiten von H und N wurden in der Natur bereits beobachtet. Menschliche Infektionen wurden in der Vergangenheit überwiegend durch die Subtypen H1N1, H2N2 und H3N2 ausgelöst. Es ließen sich aber auch humane Infektionen mit H5, H6, H7, H9 und H10 beobachten ( Tab. 1 ).
Tab. 1 Nicht saisonale Influenza-A-Virus-Subtypen, die in den letzten 20 Jahren zu humanen Infektionen geführt haben.
| Subtyp | Jahr und Land | Quelle | Fallzahl (Todesfälle) |
|---|---|---|---|
| H5N1 | 1997 Hongkong | Vögel | 18 (6) |
| H9N2 | 1998 China | Vögel | Einzelfälle (0) |
| H7N7 | 2003 Niederlande | Vögel | 89 (1) |
| H7N2 | 2003 USA | Vögel | Einzelfälle (0) |
| H7N3 | 2004 Kanada | Vögel | Einzelfälle (0) |
| H7N2 | 2007 Großbritannien | Vögel | Einzelfälle (0) |
| H5N1 | seit 2003 Asien, Afrika, Europa | Vögel | 615 (364), Stand 2013 |
| H5N6 | 2010 China | Vögel | Einzelfälle (0) |
| H3N2v | 2012 China | Schweine | Einzelfälle (0) |
| H7N9 | 2013 China | Vögel | 100 (17) |
| H6N1 | 2013 China | Vögel | Einzelfälle (0) |
| H10N8 | 2014 China | Vögel | Einzelfälle (1) |
Antigenic Drift
Influenzaviren sind genetisch sehr variabel. Es kommt häufig zu Punktmutationen, da sie zur Genomreplikation eine fehleranfällige virale RNA-abhängige RNA-Polymerase verwenden. Setzen sich solche Mutationen z. B. in H oder N durch, entstehen neue Virusvarianten. Dieser Mechanismus wird als „Antigenic Drift“ bezeichnet und kommt bei allen Influenzavirustypen vor. Das Genom von Influenzaviren ist segmental aufgebaut, die einzelnen viralen Gene werden durch separate RNA-Moleküle kodiert.
Antigenic Shift
Kommt es zur Infektion einer Wirtszelle durch 2 Virussubtypen, entstehen im Rahmen des zufälligen Zusammenbaus von Tochtervirionen bei der Virusvermehrung Viren mit neuen Kombinationen der Genabschnitte, die sich von den beiden an der Infektion der Wirtszelle beteiligten Ursprungsviren unterscheiden. Bei Influenza-A-Viren kann es wegen der großen Zahl an H- und N-Varianten durch diesen „Antigenic Shift“ genannten Prozess zur Bildung neuer Subtypen kommen.
Merke.
Sowohl Antigenic Drift als auch Antigenic Shift führen zu neuen Influenzavirusvarianten, die den durch den Wirtsorganismus aufgebauten Immunschutz unterlaufen und so zu neuen Ausbrüchen führen können.
Grippewellen
Influenzavirusinfektionen treten saisonal in der kalten Jahreszeit auf, sodass es auf jeder Erdhalbkugel alljährlich eine eigene Grippewelle gibt. Im Rahmen dieser Grippewellen werden schätzungsweise 5 – 20% der Bevölkerung im Ausbruchsgebiet durch Influenzaviren infiziert. Auch außerhalb der klassischen Influenzasaison werden Influenzavirusinfektionen labortechnisch nachgewiesen. Diese können u. U. zu örtlich und zeitlich limitierten Kleinstausbrüchen führen 10 . Grundlage der alljährlichen Grippewelle ist die Antigenic Drift, durch die Virusvarianten immer wieder auf eine durch Vorjahresinfektionen nicht optimal geschützte Bevölkerung treffen.
Influenza-B-Viren sind – je nach Saison – für 1 – 60% aller Influenzavirusinfektionen verantwortlich. Meist schwankt der Anteil der Influenza-B-Viren zwischen 15% und 35%. Der Anteil der Influenza-B-Virus-Infektionen steigt zum Ende der Influenzasaison an. Seit mehr als 20 Jahren zirkulieren 2 durch Antigenic Drift auseinander hervorgegangene Influenza-B-Virus-Linien. Jeweils einer davon dominiert in einer Saison stark. Für die beiden Influenza-B-Virus-Linien besteht keine Kreuzprotektion 11 .
Merke.
Von 2001 – 2011 war nur in 50% der Fälle der dominierende Influenza-B-Virus-Stamm im Impfstoff enthalten.
Pandemie
Tritt ein Antigenic Shift bei Influenza-A-Viren auf, kann es zu einer Grippepandemie kommen, also zu einer Ausbreitung des neuen Virus über die gesamte Welt. Die Ähnlichkeit des neuen Subtyps zu den vorher zirkulierenden Varianten ist ggf. so gering, dass die Bevölkerung im Wesentlichen ungeschützt sein kann. Im 20. Jahrhundert hat es 3 große Influenza-A-Virus-Pandemien gegeben:
Spanische Grippe (1918, H1N1)
Asiatische Grippe (1957, H2N2)
Hongkong-Grippe (1967, H3N2)
Der bis zur neuen Pandemie zirkulierende Influenza-A-Virus-Subtyp wurde jeweils durch den neuen Subtyp verdrängt. Seit 1977 wird eine Kozirkulation eines neu aufgetretenen H1N1-Stamms (Russische Grippe) mit H3N2 beobachtet.
Übertragungsweg
Eine Infektion mit Influenzaviren erfolgt überwiegend von Mensch zu Mensch und kann auf unterschiedlichen Wegen stattfinden. Der wesentliche Übertragungsweg ist die Tröpfcheninfektion: Hierbei werden die Viren durch relativ große Tröpfchen, die beim Husten, Niesen oder Sprechen entstehen, über eine Distanz von wenigen Metern übertragen. Auch eine Übertragung durch Aerosole, Tröpfchen < 5 µm, die länger in der Luft schweben können, wird angenommen. Wichtig ist darüber hinaus die Übertragung durch Schmierinfektion über Kontakt von Händen mit virushaltigem Sekret auf Oberflächen und anschließendem Mund-/Nasenkontakt. Influenzaviren können mehrere Stunden, in Einzelfällen sogar länger, auf Türgriffen, Treppengeländern und ähnlichen Oberflächen infektiös sein.
Cave.
1 ml Rachensekret kann bis zu 1 Mio. Influenzaviren enthalten. Regelmäßige Händehygiene kann daher die Gefahr einer Infektion mit Influenzaviren, aber auch mit anderen respiratorischen Viren, deutlich vermindern.
Infizierte Personen sind bereits 24 h vor Beginn der Symptome infektiös und bleiben dies 3 – 5 Tage, bei Kindern sogar bis zu 10 Tage. Die Inkubationszeit beträgt 1 – 3 Tage, in Einzelfällen sogar bis zu 7 Tagen. Infektionen mit Influenzaviren führen i. d. R. nur in einer Minderheit der Fälle zum Krankheitsbild der Influenza. Bis zu einem Drittel der Infektionen bleiben symptomlos, etwa ein weiteres Drittel der Influenzavirusinfektionen ist von grippalen Infekten nicht zu unterscheiden. Eine Übertragung von Influenzaviren auf andere Menschen mit nachfolgender Influenzasymptomatik ist aber auch in diesen Fällen möglich.
Klinik
Symptome und Verlauf
Typische Symptome einer Influenza sind plötzliches hohes Fieber mit Spitzen bis 40 °C innerhalb des ersten Erkrankungstags, Kopf- und Gliederschmerzen sowie trockener Husten. Auch Bauchschmerzen, Erbrechen und Diarrhö werden beobachtet, vor allem bei Kindern. Nach der Falldefinition des Robert Koch-Instituts müssen für die Diagnose einer Influenza mindestens 2 der folgenden Kriterien vorliegen:
akuter Krankheitsbeginn
Fieber
Husten
Muskel-, Glieder-, Rücken- oder Kopfschmerzen
Die klassischen Influenzasymptome können, insbesondere bei Patienten mit Grunderkrankungen, durch Komplikationen ergänzt werden, z. B. Pneumonie, Myokarditis, Perikarditis, Enzephalitis oder das Guillain-Barré-Syndrom. Bei bis zu 23% der ambulant erworbenen Pneumonien lassen sich Viren nachweisen. Darunter werden Influenzaviren am häufigsten detektiert.
Merke.
Häufig sind Superinfektionen durch Bakterien oder Pilze, die 5 – 10 Tage nach der Influenzavirusinfektion auftreten 10 .
Bronchitis
Eine wichtige Erkrankung aus der Gruppe der grippalen Infekte des unteren Respirationstrakts ist die Bronchitis. Sie ist die zweithäufigste Grundlage für das Ausstellen von Arbeitsunfähigkeitsbescheinigungen. Die Ursache für Bronchitis ist bei Patienten ohne schwere Grunderkrankungen häufig nicht bakteriell. Trotzdem werden oft Antibiotika verschrieben.
Merke.
Eine Auswertung randomisierter Studien ergab nur marginale Vorteile (eine um einen halben Tag verkürzte Hustendauer, aber keine signifikante Verkürzung der Krankheitsdauer) bei mit Antibiotika behandelten Patienten gegenüber Placebogruppen 12 .
Es konnte abgeleitet werden, dass in Deutschland Influenzaviren während der Influenzasaison für bis zu 25% der akuten Bronchitisfälle verantwortlich sind 10 . Auch andere respiratorische Viren spielen hier eine wichtige Rolle.
Neue Influenzaviren
Mutationen des Influenza-B-Virus
Aufgrund des hohen Änderungspotenzials von Influenzaviren durch Antigenic Drift und Antigenic Shift ist das Auftreten neuer humanpathogener Influenzaviren ein regelmäßiges Phänomen. In der Regel handelt es sich nur um geringe Veränderungen durch Punktmutationen von Saison zu Saison. Aber auch diese haben medizinisch relevante Konsequenzen, da eine jährliche Überprüfung und ggf. Anpassung der Impfstoffzusammensetzung nötig ist.
Merke.
Die WHO empfiehlt, den etablierten trivalenten durch einen quadrivalenten Impfstoff zu ersetzen. Grund ist das Neuauftreten der zweiten, nicht kreuzprotektiven Linie des Influenza-B-Virus durch die mangelnde Vorhersehbarkeit des in der nächsten Saison dominierenden Stamms.
Die Saison 2015/2016 hat wieder die Notwendigkeit des quadrivalenten Impfstoffs unterstrichen, da auch in dieser Saison im Impfstoff keine schützende Komponente gegen das Influenza-B-Virus vorhanden war. Möglicherweise ist dies die Grundlage für die beobachtete hohe Zahl an Influenza-B-Virus-Infektionen.
Mutationen des Influenza-A-Virus
Influenza-A-Viren haben aufgrund des hier möglichen Antigenic Shift ein noch viel größeres Änderungspotenzial. Seit 1997 (H5N1, Tab. 1 ) kommt es in kurzer Folge immer wieder zum Auftreten von Influenza-A-Virus-Subtypen, die den Speziessprung z. B. vom Vogel in den Menschen schaffen und dort zu Infektionen mit hoher Letalität führen können. Letzter wichtiger Vertreter dieser Reihe, die von Subtypen mit den Hämagglutininkomponenten H5, H7 und H9 dominiert wird, ist ein Influenza-A-Virus vom Subtyp H7N9, das 2013 in China nachgewiesen wurde und eine größere Zahl humaner Infektionen verursacht hat ( Tab. 1 ). Dieses Virus hat eine Letalität von bis zu 39%. Mehr als 60% der Todesfälle hatten eine Grunderkrankung. Nach 2010 ist es zusätzlich zu vereinzelten humanen Fällen von H5N6, H6N1 und H10N8 gekommen, sowie einer Anzahl von humanen Infektionen mit H3N2v, einem Schweineinfluenzavirus, das sehr verschieden vom humanen H3N2-Subtyp ist 13 .
Merke.
Bisher hat keiner der im vorhergehenden Abschnitt genannten Virusstämme das Potenzial entwickelt, sich effizient von Mensch zu Mensch auszubreiten. Aus diesem Grund ist es bisher noch nicht zu einer Pandemie mit humanen Infektionen mit einem dieser Viren gekommen.
Bei Fledermäusen wurden in den letzten Jahren die Influenza-A-Virus-Subtypen H17N10 und H18N11 neu entdeckt. Zurzeit gibt es keinen Hinweis, dass menschliche Infektionen mit diesen Subtypen möglich sind.
Schweinegrippe
2009 kam es zu einer humanen Influenzapandemie mit dem varianten Influenza-A-Virus-Subtyp H1N1 (Schweinegrippe). In Deutschland wurden mehr als 226 000 Fälle gemeldet. Morbidität und Letalität waren geringer als in den Jahren, die von den bekannten saisonalen Stämmen dominiert waren. Zu letalen Verläufen kam es in Deutschland weit überwiegend bei Patienten mit Grunderkrankungen. Auffällig war allerdings die weltweit hohe Gefährdung von Kleinstkindern und Schwangeren. Nur in Japan gab es keine signifikante Erhöhung der Letalität bei Schwangeren. Hier wurde eine Durchimpfungsrate der Schwangeren von 67% erreicht. 95% der infizierten Schwangeren wurden mit Oseltamivir behandelt, 88% davon innerhalb von 2 Tagen nach Symptombeginn.
Merke.
Als Ursache der besonders guten epidemiologischen Situation bei Schwangeren während der Pandemie 2009 in Japan ist das effiziente Management von Prävention und Therapie der Influenzavirusinfektion anzunehmen 14 .
Diagnostik
Klinische Symptome
Während der Influenzasaison lässt sich eine Influenza i. d. R. anhand von klinischen Symptomen diagnostizieren. Auf eine labordiagnostische Abklärung kann man im ambulanten Bereich dann verzichten. Bei sporadischen Fällen oder zu Beginn der Influenzasaison oder bei schweren Verläufen sollte die Labordiagnostik auf jeden Fall erfolgen. Gute Gründe für die Durchführung der Diagnostik sind ebenso die Möglichkeit zur Durchführung von Kohortierungsmaßnahmen oder die Erfassung der Infektionsepidemiologie (Wirksamkeit der Impfung, Zuordnung von Krankheitslast zu Virustypen und -subtypen).
Anzucht
Goldstandard für die Diagnostik von Influenzavirusinfektionen war lange Zeit die Virusisolation aus Patientenmaterial durch Anzucht auf geeigneten Zellkulturen. Aufgrund der heute zur Verfügung stehenden schnelleren, sensitiveren und automatisierbaren Verfahren ist die Virusanzucht in der täglichen Routine aber in den Hintergrund getreten.
Antikörpernachweise
Serologische Antikörpernachweisverfahren sind ebenfalls seit langer Zeit verfügbar und werden nach wie vor häufig vom klinisch tätigen Arzt angefordert. Aufgrund von eingeschränkter Sensitivität und Spezifität der Antikörpernachweisverfahren ist der positive Vorhersagewert positiver Antikörpernachweise für eine frische Influenzavirusinfektion gering. Somit ist der Antikörpernachweis im Wesentlichen für epidemiologische Fragestellungen oder für Infektionsnachweise in einer späten Erkrankungsphase von Bedeutung, wenn Direktnachweise nicht mehr zum Ziel führen.
Virusdirektnachweis
In der frühen Phase der Influenzavirusinfektion ist der Virusdirektnachweis heute die Methode der Wahl. Zur Verfügung stehen:
Schnelltests (Immunchromatografie)
Antigennachweise durch ELISA
Antigennachweise durch Immunfluoreszenztest (IFT)
Nukleinsäureamplifikationsverfahren (PCR, isothermale Amplifikationsverfahren)
Spezifität und Sensivität
Die Spezifität der genannten Testverfahren liegt in der gleichen Größenordnung ( Tab. 2 ), die Sensitivität ist aber unterschiedlich. Die Sensitivität immunchromatografischer Schnelltests liegt nur bei 63 – 85%, die der PCR bei 92,3%. Die Sensitivität von Antigen-ELISAs ist mit der Sensitivität der Schnelltests vergleichbar, die Sensitivität der Antigen-IFTs liegt zwischen Schnelltest und PCR ( Tab. 2 ).
Tab. 2 Sensitivität und Spezifität von Verfahren zum Direktnachweis von Influenzaviren.
| Testverfahren | Sensitivität | Spezifität |
|---|---|---|
| Schnelltest | 63 – 85% | 97 – 98% |
| Antigen-ELISA | 62 – 74% | 100% |
| Antigen-IFT | 84 – 94% | 97 – 99% |
| PCR | 92% | 99% |
Zeitaufwand und Material
Die am wenigsten zeitintensiven Tests sind die Schnelltests mit ca. 15 min. Eine so schnelle Diagnostik ist mittlerweile aber auch schon mit der sensitiven isothermalen Nukleinsäureamplifikation erreichbar. Diagnostik kann aus Nasen-/Rachenabstrich, Rachenspülwasser oder bronchoalveolärer Lavage (BAL) erfolgen. Eine negative PCR aus Nasen-/Rachenabstrich schließt eine positive PCR aus BAL nicht aus.
Merke.
Für den direkten Nachweis von Influenzaviren besteht eine Meldepflicht nach § 7 IfSG.
Therapie
Bei unkomplizierten Influenzaverläufen oder länger bestehender Erkrankung beschränkt sich die Therapie i. d. R. auf symptomatische Maßnahmen, wie z. B. Bettruhe, Flüssigkeitszufuhr, Schmerzbehandlung, Fiebersenkung, Maßnahmen zum Abschwellen von Schleimhäuten und Verflüssigung von zähem Schleim.
Neben den beschriebenen unspezifischen Maßnahmen stehen aktuell influenzavirusspezifische antivirale Wirkstoffe aus 2 Stoffklassen zur Verfügung:
M2-Inhibitoren
Neuraminidaseinhibitoren
M2-Inhibitoren
Das influenzaviruskodierte M2-Protein ist ein Protonenkanal, der an der Freisetzung der viralen Nukleinsäure aus dem Viruspartikel in das Zytosol in der Frühphase der Infektion beteiligt ist. Die verfügbaren M2-Inhibitoren Amantadin und Rimamantadin blockieren den Protonenkanal und behindern so die Nukleinsäurefreisetzung und die nachfolgende Virusvermehrung. Ihre Wirkung ist auf Influenza-A-Viren beschränkt. Sie führen zur Verkürzung der symptomatischen Phase um einen Tag, ohne die Virusausscheidung signifikant zu behindern. Die Gabe von M2-Inhibitoren ist mit Nebenwirkungen im zentralen Nervensystem verbunden 10 . Resistenzen gegen M2-Inhibitoren sind verbreitet und entstehen schnell, daher ist eine Therapie mit entsprechenden Medikamenten heute nicht mehr Mittel der Wahl. Ein prophylaktischer Einsatz kann aber sinnvoll sein, eine Postexpositionsprophylaxe ist in 70 – 90% der Fälle erfolgreich.
Neuraminidaseinhibitoren
Neuraminsäurereste (= Sialinsäurereste) sind Bindungspartner des viralen Hämagglutinin bei der initialen Bindung zwischen Virus und Wirtszelle. Eine erfolgreiche Freisetzung von Virusnachkommen aus der Wirtszelle ist somit nur nach Spaltung der Neuraminidasereste an der Freisetzungsstelle möglich. Aus diesem Grund haben Influenzaviren eine viral kodierte Neuraminidase in ihrer Hüllmembran. Sie stellt eine erfolgreiche Freisetzung von Tochtervirionen sicher. Neuraminidaseinhibitoren hemmen die Aktivität des Enzyms und hemmen so die Freisetzung von Tochtervirionen und dadurch die Infektion weiterer Wirtszellen. Aufgrund des Wirkmechanismus ist die klinische Wirksamkeit an eine sehr frühe Applikation der Wirkstoffe innerhalb von 48 h nach Symptombeginn geknüpft. Neuraminidaseinhibitoren wirken potenziell gegen alle Influenza-A- und Influenza-B-Viren. In Deutschland zugelassen sind die Neuraminidaseinhibitoren Zanamivir und Oseltamivir. Darüber hinaus bestehen international Zulassungen für Peramivir und Laninamivir.
Zanamivir, das erst ab 12 Jahren zugelassen ist, wird inhalativ appliziert. Es ist für den Einsatz bei Asthmatikern nicht zugelassen. Probleme können bei Patienten auftreten, welche die Inhalationstechnik nicht ausreichend beherrschen 10 . Zwischenzeitlich war Zanamivir in flüssiger Form für den intravenösen Einsatz verfügbar, zurzeit ist für diesen Zweck nur eine flüssige Formulierung von Peramivir auf dem Markt. Oseltamivir wird oral appliziert. Ein Vorteil von intravenöser Applikation gegenüber oraler Applikation ist nicht eindeutig belegt. Die Wirksamkeit der Neuraminidaseinhibitoren ist allerdings nicht unumstritten. Es gibt zahlreiche Studien, die zeigen, dass die symptomatische Phase der Influenza bei frühzeitiger Applikation um bis zu 36 h reduziert wird und sich Komplikationen der Influenza deutlich reduzieren lassen 10 . Bei hospitalisierten Erwachsenen mit schwer verlaufender, komplikationsreicher Influenza kann eine Oseltamivir-Gabe die Mortalität auch noch bei einem Therapiebeginn 4 – 5 Tage nach Symptombeginn reduzieren 15 . Eine Cochrane-Studie zum Thema kommt jedoch zu dem Ergebnis, das die Wirksamkeit der Wirkstoffe nicht bewiesen ist. Dies liegt allerdings an einem Mangel an verfügbaren Studien, welche die Cochrane-Qualitätsanforderungen erfüllen, und nicht daran, dass die publizierten Studien keinen Nachweis der Wirksamkeit von Neuraminidaseinhibitoren zeigen.
Die Effizienz von Oseltamivir zur Prävention der saisonalen Influenza liegt bei 61 – 73%. Trotzdem ist bei Einsatz von Oseltamivir bei einer neuen Influenzaviruspandemie von einer signifikanten Reduktion von Morbidität und Letalität auszugehen 10 . Die Gabe von Oseltamivir innerhalb von 8 Tagen nach Symptombeginn reduziert die Letalität von Infektionen mit dem Influenza-A-Virus-Subtyp H5N1.
Merke.
Je früher die Therapie beginnt, desto höher ist der Effekt. Dies zeigt die Notwendigkeit der frühen Diagnose.
Bei Patienten, die zum Zeitpunkt des Therapiebeginns beatmet waren, ist der positive Effekt der Therapie geringer 13 . Resistenz gegen Oseltamivir kann in 25% der Fälle auftreten. Allerdings sind oseltamivirresistente Stämme i. d. R. in ihrer Vermehrungsfähigkeit eingeschränkt und weiterhin sensitiv gegen Zanamivir.
Andere Wirkstoffe
Weitere spezifisch gegen Influenzaviren wirksame Arzneistoffe sind in Entwicklung. Favipiravir ist ein gegen alle Influenzaviren und andere RNA-Viren wirksamer RNA-Polymeraseinhibitor, der synergistisch mit Oseltamivir zusammenwirkt 16 . Der Wirkstoff ist mittlerweile in Japan zugelassen. Fludase verhindert das Eindringen des Virus in die Wirtszelle, indem dieses Enzym Neuraminsäurereste von Wirtszelloberflächen entfernt und so eine Bindung von Influenzaviren verhindert. Der Wirkstoff befindet sich in einer Phase-II-Zulassungsstudie, wie auch Präparate auf Basis von neutralisierenden monoklonalen Antikörpern, die durch Bindung von Hämagglutinin die Infektion der Wirtszellen beeinträchtigen 13 .
Prophylaxe
Impfstoffe
Neben Maßnahmen der Standardhygiene (z. B. der Hände) stehen zur Prophylaxe einer Influenzavirusinfektion zahlreiche Impfstoffe zur Verfügung. Im Jahr 2015 waren ca. 20 verschiedene Impfstoffe zugelassen. Es handelt sich jeweils um Kombinationsimpfstoffe, die Schutz gegen eine Infektion mit Influenza-A- und Influenza-B-Viren aufbauen.
Merke.
Die Virusstämme, die Grundlage für die Impfstoffentwicklung sind, werden für jede Influenzasaison ca. 6 Monate vor Beginn des empfohlenen Impfzeitraums neu von der WHO festgelegt.
In der Frühphase der Impfstoffentwicklung wurden trivalente (2 Influenza-A-Virus-Stämme, 1 Influenza-B-Virus-Stamm) Ganzvirus-Impfstoffe der 1. Generation zugelassen, die eine optimale Immunantwort auslösten, aber ein großes Nebenwirkungsspektrum zeigten. Die Spaltvirus-Impfstoffe der 2. Generation, die auch heute noch verfügbar sind, zeigen nur noch ein mäßiges Nebenwirkungsspektrum bei suboptimaler Immunantwort. Die Subunit-Impfstoffe der 3. Generation zeigen ein geringes Nebenwirkungsspektrum bei einer zur 2. Generation der Impfstoffe vergleichbaren Immunantwort bzw. Schutzwirkung. Die adjuvantierten Impfstoffe der 4. Generation verbinden starke Immunantwort mit geringem Nebenwirkungsspektrum.
In der Vergangenheit wurden auch Impfstoffe gegen neue Virusvarianten zugelassen, z. B. den Influenza-A-Virus-Subtyp H5N1 (Vogelgrippe), sowie Pandemieimpfstoffe, die eine schnelle Adaptation eines Basisimpfstoffs an einen pandemischen Influenza-A-Virus erlauben sollen.
Impfstoffkonzepte
Basis der großen Zahl zugelassener Influenzavirusimpfstoffe ist die Verwendung unterschiedlicher Impfstoffkonzepte:
Klassischer Impfstoff: Die Impfstoffe basieren auf in Hühnereiern angezüchteten Viren. Die Produktionszeit liegt in der Größenordnung von 6 Monaten.
Impfstoffe auf Zellkulturbasis: Diese Impfstoffe basieren auf in aseptischem Milieu auf Zellkulturbasis angezüchteten Viren. Die Produktionszeit ist mit 3 Monaten wesentlich kürzer als die klassische Virusanzucht in Hühnereiern. Der Impfstoff ist für Hühnereiweißallergiker besonders gut geeignet.
Impfstoff auf Virosomenbasis: Die Virusantigene werden in eine Virushülle ohne RNA-Gehalt inkorporiert. Die Hülle fungiert als Adjuvans. Der Hühnereiweißgehalt ist sehr gering.
Adjuvantierte Impfstoffe: Die Virusantigene für solche Impfstoffe werden mit einem Adjuvans gekoppelt, z. B. MF59. Dadurch wird die Immunogenität des Impfstoffs gesteigert, sodass der Titer protektiver Antikörper höher als bei nicht adjuvantierten Impfstoffen ist. Dies ist insbesondere bei älteren Impflingen von Vorteil.
Lebendimpfstoffe: Im Gegensatz zu den zuvor geschilderten Impfstoffkonzepten für Totimpfstoffe basieren solche Impfstoffe auf lebend attenuierten Viren. Die Schutzwirkung von Lebendimpfstoffen wurde in Studien mit Kindern als der Schutzwirkung von Totimpfstoffen überlegen festgestellt. In kürzlich durchgeführten Studien konnte die Überlegenheit der Lebendimpfstoffe nicht bestätigt werden. Die Ständige Impfkommission hat Mitte 2017 die spezielle Empfehlung von Lebendimpfstoffen zurückgenommen.
Quadrivalente Impfstoffe: Alle zuvor aufgeführten Impfstoffkonzepte wurden zunächst auf Basis trivalenter Impfstoffe zugelassen (2 Influenza-A-Virus-Komponente, 1 Influenza-B-Virus-Komponente). Inzwischen empfiehlt die WHO wegen des seit vielen Jahren kozirkulierenden 2. Influenza-B-Virus-Stamms, für den keine Kreuzprotektion besteht, die Verwendung von quadrivalenten Impfstoffen mit 2 Influenza-A-Virus-Komponenten und 2 Influenza-B-Virus-Komponenten. Entsprechende quadrivalente Impfstoffe sind mittlerweile für mehrere der o. g. Impfstoffkonzepte in Deutschland zugelassen. Für die Saison 2015/2016 war die Verfügbarkeit dieser Impfstoffe aber unbefriedigend. In Einzeldosen konfektionierte quadrivalente Impfstoffe waren wegen eines Produktionsproblems nicht verfügbar, in Gebinden zu 20 Impfdosen konfektionierte quadrivalente Impfstoffe waren sehr früh vergriffen.
Ein auf Basis der Vorteile der verschiedenen Impfstoffkonzepte bzw. der zulassungsbedingten Einschränkungen sinnvoll erscheinende Zuordnung von unterschiedlichen Impfstoffen zu Patientengruppen ist in Tab. 3 dargestellt.
Tab. 3 Sinnvolle Zuordnung unterschiedlicher Influenzaimpfstoffe zu Patientengruppen.
| Zielgruppe | geeignete Impfstoffe (falls verfügbar quadrivalent) |
|---|---|
| gesunde Kinder 6 Monate bis 2 Jahre | nicht adjuvantierter klassischer Impfstoff |
| gesunde Kinder 2 – 6 Jahre | nicht adjuvantierter klassischer Impfstoff, ggfs. Lebendimpfstoff (zugelassen für 2 – 17 Jahre) |
| Kinder ab 6 Monaten mit chronischen Erkrankungen oder Immunsuppression | Virosomenimpfstoff (derzeit nicht im Handel) |
| gesunde Erwachsene < 60 Jahre | nicht adjuvantierter klassischer Impfstoff |
| Erwachsene < 60 Jahre mit Hühnereiweißallergie | zellkulturbasierender Impfstoff |
| Erwachsene < 60 Jahre mit chronischen Erkrankungen oder Immunsuppression | Virosomenimpfstoff, nicht adjuvantierter klassischer Impfstoff |
| Erwachsene > 60 Jahre | adjuvantierter Impfstoff |
Die Impfschutzrate des klassischen Impfstoffs bei gesunden Impflingen unter 65 Jahren liegt bei 73%, bei über 65-Jährigen sinkt sie auf 31% ab.
Durchimpfungsrate
In Deutschland ist die Influenzaimpfung für bestimmte Personengruppen empfohlen. Am höchsten ist die Durchimpfungsrate bei den über 65-Jährigen. Hier lag die Durchimpfung in der Saison 2005/2006 bei ca. 56% 17 . Damit wird das WHO-Durchimpfungsziel für Personen über 65 Jahre für 2006 von 50% noch erreicht, das Durchimpfungsziel für 2010 von 75% aber nicht mehr. Die Durchimpfung bei Risikopersonen liegt in Deutschland mit 40,6% wesentlich niedriger.
Merke.
Am schlechtesten ist die Durchimpfung gegen Influenza in Deutschland bei Beschäftigten des Gesundheitssystems mit 27,5%.
Das RKI berichtet für die Saison 2014/2015 eine Durchimpfungsrate bei älteren Menschen in Deutschland von 36,7%. Hierbei wurde allerdings eine Altersgrenze von 60 Jahren verwendet.
Impfung von Mitarbeitern des Gesundheitssystems
Eine Steigerung der Durchimpfungsrate bei den Mitarbeitern des Gesundheitssystems hätte aufgrund der Minimierung von nosokomialen Infektionen einen bedeutenden Effekt auf die Infektionsrate von Patienten. So konnte gezeigt werden, dass eine Steigerung der Durchimpfung der Mitarbeiter von 4% auf 67% in einem Krankenhaus über einen Zeitraum von 12 Jahren die laborbestätigten Influenzavirusinfektionen der Mitarbeiter von 42% auf 9% absinken ließ. Nosokomiale Infektionen wurden gar nicht mehr nachgewiesen. In Pflegeheimen konnte unabhängig von der Durchimpfung der Bewohner die allgemeine Sterblichkeit in der Influenzasaison um mehr als 40% gesenkt werden.
Impfung von Schwangeren
Als Folge der Schweinegrippe-Pandemie von 2009 hat die STIKO die Empfehlung für Influenzaimpfungen geändert. Seit dieser Änderung ist in Reaktion auf die erhöhte Letalität der Influenzainfektion bei Schwangeren auch die Impfung von Schwangeren empfohlen.
Humanes Metapneumovirus (hMPV)
Biologie
Das humane Metapneumovirus (hMPV) ist ein Paramyxovirus und wurde 2001 in den Niederlanden entdeckt, zirkuliert aber seit mindestens 50 Jahren im Menschen 10 . Man unterscheidet die 4 Genotypen A1, A2, B1 und B2 18 .
Merke.
Das humane Metapneumovirus ist weltweit verbreitet und spielt bei der Mortalität und Morbidität von Säuglingen und Kleinkindern eine große Rolle 10 .
Bis zum 10. Lebensjahr haben fast alle Kinder eine erste Infektion mit dem humanen Metapneumovirus durchgemacht. Der Median von Infektionen mit dem hMPV liegt bei 11,6 Monaten 19 . Erwachsene machen seltener eine schwerwiegende symptomatische Erkrankung durch. Hiervon sind insbesondere Patienten mit chronisch obstruktiven Lungenerkrankungen oder immunkompromittierte Patienten betroffen. Das Infektionsmaximum liegt im Spätwinter bzw. frühen Frühjahr. Verschiedene Virusstämme können an einem Ort kozirkulieren 20 .
Klinik und Diagnostik
Infektionen mit dem humanen Metapneumovirus tragen mit einer Inzidenz von 1,5 – 25% zu Infektionen des oberen und des unteren Respirationstrakts bei. Das Symptomspektrum reicht von einer milden Infektion des oberen Respirationstrakts bis hin zu Bronchiolitis und Pneumonie. Re-Infektionen verlaufen i. d. R. milder 20 .
Cave.
Infektionen mit dem humanen Metapneumovirus sind auf Basis der Symptome i. d. R. nicht von Infektionen mit anderen respiratorischen Viren unterscheidbar.
Virusanzucht ist möglich und Antigennachweisverfahren sind verfügbar ( Tab. 4 ). Wegen des langsamen Viruswachstums bzw. der eingeschränkten Testsensitivität setzt man diese Verfahren aber nicht routinemäßig ein. Die Diagnostik wird von molekularen Nachweisverfahren dominiert, entweder als Einzelnachweis oder im Rahmen einer Multiplexreaktion zusammen mit anderen Respirationstraktpathogenen.
Tab. 4 Verfügbare Nachweisverfahren wichtiger respiratorischer Viren.
| Virus | Schnelltest | Antigen IFT | Antigen ELISA | PCR | Zellkultur |
|---|---|---|---|---|---|
| Influenzaviren | + | + | + | + | + |
| Metapneumovirus | – | + | + | + | + |
| Coronaviren | – | + | – | + | + |
| Bocavirus | – | – | – | + | + |
| Enteroviren | – | – | – | + | + |
| Rhinoviren | – | + | + | + | + |
Therapie und Prophylaxe
Zurzeit erfolgt die Therapie von Infektionen mit dem hMPV symptomatisch. Ribavirin wirkt in Zellkulturen und im Mausmodell gegen das hMPV, ebenso wie die passive Immunisierung mit intravenösen Immunglobulinen (IVIG). Für den Einsatz beim Menschen existieren aber nur Einzelfallberichte 21 , kontrollierte Studien fehlen.
Mehrere monoklonale Antikörper wurden gegen das virale F-Protein entwickelt und im Tiermodell erfolgreich getestet. Fusionsinhibitoren, welche die Übertragung des viralen Genoms in die Wirtszelle inhibieren, wurden entwickelt und zeigen in vitro und im Tiermodell Effektivität gegenüber hMPV. Gleiches gilt für Moleküle, die über den Mechanismus der RNA-Interferenz wirken. Für alle geschilderten Therapiestrategien gibt es noch keine Daten für den Einsatz beim Menschen 18 .
Zurzeit wird an potenziellen Impfstoffen auf Basis von Subunit-Proteinen, Virus-like Particles und lebend attenuierten Viren gearbeitet. Im Tierversuch zeigen einige Kandidaten vielversprechende Resultate. Zur klinischen Erprobung ist es aber noch in keinem Fall gekommen 18 .
Coronaviren
Biologie, Klinik und Diagnostik
Humanpathogene Coronaviren (CoV-OC43 und CoV-229E) können Erkrankungen vom banalem Schnupfen bis hin zu schweren Infektionen des unteren Respirationstrakts auslösen. Obwohl sie seit vielen Jahrzehnten bekannt sind, wurden erst in den letzten Jahren 4 weitere Mitglieder dieser Familie entdeckt, die für respiratorische Erkrankungen verantwortlich sein können.
SARS-CoV
2003 wurde das SARS-CoV in einer international koordinierten Aktion in kurzer Zeit als Ursache des zuvor nicht beschriebenen Severe Acute Respiratory Syndrome (SARS) identifiziert, das eine Letalität von ca. 10% aufwies 10 .
Merke.
Durch strikte Surveillance- und Isolationsmaßnahmen ist es gelungen, eine weltweite Verbreitung des SARS-Coronavirus zu verhindern und es aus der humanen Population zu eliminieren.
CoV-NL63
2004 wurde CoV-NL63 durch eine niederländische Arbeitsgruppe entdeckt 10 . Es lässt sich bei hoher Koinfektionsrate mit weiteren Respirationstraktpathogenen in 1 – 7% der Respirationstraktmaterialien von Patienten mit Respirationstraktinfektion nachweisen. Zudem gibt es Hinweise auf eine Assoziation mit Krupp. Das Virus ist bevorzugt bei chronisch kranken Kindern mit einem schweren Erkrankungsverlauf assoziiert.
CoV-HKU1
2005 wurde CoV-HKU1 erstmals in Hongkong nachgewiesen 10 . Es lässt sich bei hoher Koinfektionsrate mit weiteren Respirationstraktpathogenen in 1 – 19% der Respirationstraktmaterialien von Patienten mit Respirationstraktinfektion nachweisen. Vor allem bei chronisch kranken Kindern ist das Virus mit einem schweren Erkrankungsverlauf assoziiert.
CoV-MERS
2012 wurde eine neue respiratorische Erkrankung beschrieben, die als Middle East Respiratory Syndrome (MERS) bekannt wurde und durch das neu entdeckte CoV-MERS ausgelöst wird 22 . Es gibt Hinweise, dass das Virus über Kamele und Fledermäuse auf den Menschen übertragbar ist und auch von Mensch zu Mensch übertragen werden kann 23 . Die größte Zahl von Erkrankungen wurde bisher von der Arabischen Halbinsel und aus Südkorea gemeldet. CoV-MERS kann respiratorische Erkrankungen bis hin zur Pneumonie auslösen sowie Systemerkrankungen, wie z. B. Nierenversagen, Diabetes und Herzerkrankungen. Die Letalität bei Patienten mit Grunderkrankungen ist hoch. Die Gesamtletalität aller bisher nachgewiesenen MERS-Fälle liegt bei über 30% 24 .
Nachweis
Coronaviren werden durch Nukleinsäureamplifikation nachgewiesen. Andere Verfahren spielen in der Routinediagnostik heute keine Rolle ( Tab. 4 ). CoV-OC43, CoV-229E, CoV-NL63 und CoV-HKU1 sind sowohl durch singuläre PCR erfassbar als auch in gängiger Multiplex-PCR für respiratorische Viren enthalten. CoV-SARS und CoV-MERS sind in der Routinediagnostik nur über eine singuläre PCR nachweisbar.
Therapie und Prophylaxe
Zurzeit ist kein spezifisch gegen Coronaviren wirkendes Medikament zugelassen. Die Therapie erfolgt symptomatisch. Die meisten Coronavirusinfektionen sind selbstlimitierend. Aufgrund der hohen Bedeutung von Coronaviren für Patienten mit Grunderkrankungen und die hohe Morbidität und Letalität von SARS und MERS werden seit Jahren verstärkt Anstrengungen unternommen, spezifisch wirkende antivirale Substanzen zu entwickeln. In Evaluation befinden sich Wirkstoffe, die virale Proteasen, Polymerasen und den Viruseintritt in die Wirtszelle inhibieren 25 .
Zurzeit wird an potenziellen Impfstoffen auf Basis von Subunit-Proteinen, Virus-like Particles, DNA-Vakzinen und lebend attenuierten Viren gearbeitet. Einige Kandidaten zeigen vielversprechende Resultate. Bisher wurde kein Impfstoff zugelassen 26 .
Humanes Bocavirus
Biologie und Klinik
Humane Bocaviren, die zur Familie der Parvoviren gehören, wurden 2005 bei Patienten entdeckt, die an Respirationstraktinfektionen erkrankt waren. Mittlerweile wurden 4 Spezies identifiziert (HBoV1 – 4). Die klinische Bedeutung von Bocaviren ist noch umstritten.
Ätiologische Bedeutung für Respirationstraktinfektionen hat HBoV1. Das HBoV1 ist weltweit und ganzjährig verbreitet und infiziert Kinder und Erwachsene. Eine ätiologische Bedeutung des Virus wurde für Erkrankungen vom banalen Infekt des oberen Respirationstrakts bis hin zur Pneumonie postuliert. Das Virus kann zu Exazerbationen von chronisch obstruktiven Lungenerkrankungen und Asthma führen 27 . Schwerwiegende Erkrankungen können im Wesentlichen bei Kindern und Erwachsenen mit Grunderkrankungen auftreten. HBoV1 wurde bei 3 – 19% aller Kinder mit Respirationstrakterkrankungen nachgewiesen. Die Koinfektionsrate mit anderen Respirationstrakterregern ist sehr hoch. Die Schwere der Symptome steigt mit der nachgewiesenen Viruslast, die Koinfektionsrate sinkt mit steigender Viruslast.
Cave.
Es gibt keine spezifischen Krankheitssymptome, die eine Unterscheidung von Infektionen mit anderen Erregern ermöglichen.
Diagnostik und Therapie
Bocaviren werden durch Nukleinsäureamplifikation nachgewiesen. Andere Verfahren spielen in der Routinediagnostik heute keine Rolle ( Tab. 4 ). Die Viren sind sowohl durch singuläre PCRs erfassbar als auch in gängigen Multiplex-PCRs für respiratorische Viren enthalten.
Merke.
Eine frische Bocavirusinfektion lässt sich nur auf Basis einer mittleren bis hohen Viruslast (> 10 000 Kopien/ml) diagnostizieren.
Die Diagnose wird durch die monatelange Persistenz von Bocavirus-DNA erschwert. Im Fall von Persistenz ist die Viruslast i. d. R. niedrig.
Es gibt keine Arznei- oder Impfstoffe, spezifische Therapeutika oder Vakzine sind entweder nicht zugelassen oder in keinem fortgeschrittenen Entwicklungsstadium 27 .
Enteroviren
Biologie und Klinik
Enteroviren gehören zur Gattung Enterovirus in der Familie der Picornaviren. Die Gattung umfasst 4 Enterovirusspezies (Enterovirus A – D) und 3 Rhinovirusspezies (Rhinovirus A – C).
Die unterschiedlichen Enterovirustypen können Ursache eines großen Erkrankungsspektrums sein, das von exanthematischen Erkrankungen und Fieber über Myokarditis/Perikarditis und Lähmungen bis hin zur Enzephalitis reichen kann.
Merke.
Enteroviren sind die häufigste Ursache viraler Meningitiden.
Einige Enterovirustypen werden nur im Respirationstrakt nachgewiesen. Diese gehören überwiegend zu den Enterovirusspezies C und D. Diese Virustypen sind weltweit verbreitet und können Symptome von der banalen Infektion des oberen Respirationstrakts bis hin zur Pneumonie auslösen 28 . Schwere Verlaufsformen treten überwiegend bei Kindern und Patienten mit Grunderkrankungen aus. Aufgrund der großen Zahl an respiratorischen Enterovirustypen ist die Seroprävalenz hoch.
Enterovirus D68, ein Mitglied der Enterovirusspezies D, wurde erstmals 1962 bei Kindern mit Respirationstrakterkrankungen nachgewiesen. Enterovirus-D68-Nachweise wurden seitdem auf niedrigem Niveau auf der ganzen Welt berichtet. Seit dem Jahr 2000 steigt die Nachweisrate von Enterovirus-D68-Erkrankungsclustern weltweit an. Leichte bis schwere Respirationstrakterkrankungen waren mit dem Virus assoziiert. 2014 wurde ein großer Ausbruch in vielen Staaten der USA nachgewiesen, von dem im Wesentlichen Kinder mit Grunderkrankungen des Respirationstrakts (z. B. Asthma) betroffen waren. Neben respiratorischen Symptomen wurden in diesem Ausbruch häufig auch schwerwiegende ZNS-Symptome berichtet 29 .
Diagnostik
Enteroviren lassen sich durch Nukleinsäureamplifikation nachweisen. Serologische Verfahren spielen eine untergeordnete Rolle ( Tab. 4 ). Die Viren sind sowohl durch singuläre PCRs erfassbar als auch in gängigen Multiplex-PCRs für respiratorische Viren enthalten.
Therapie und Prophylaxe
Es gibt keine spezifischen Arznei- oder Impfstoffe, spezifische Therapeutika oder Vakzine sind für respiratorische Enteroviren nicht zugelassen. Der Wirkstoff Pleconaril ist in vitro gut wirksam, führte in klinischen Studien aber häufig zur Aggravierung der Symptome und hat daher keine Zulassung zur Therapie von Patienten mit respiratorischen Infektionen erhalten. Der Wirkstoff wurde in Einzelfällen erfolgreich bei ZNS-Infektionen durch Enteroviren eingesetzt 10 .
Rhinoviren
Biologie und Klinik
Die neue systematische Einordnung der humanen Rhinoviren fasst sie zu 3 Spezies (Rhinovirus A – C) zusammen und ordnet sie der Gattung Enterovirus in der Familie der Picornaviren zu. Man kann über 100 Rhinovirustypen unterscheiden. Bis 2006 wurden durch molekularbiologische Untersuchungen mehr als 50 nicht anzüchtbare Rhinoviren neu entdeckt. Diese fasste man in der dafür neu geschaffenen Spezies Rhinovirus C zusammen.
Symptome
Rhinoviren sind für etwa die Hälfte aller oberen Atemwegsinfektionen weltweit verantwortlich. Sie können aber auch schwerwiegende Krankheitsbilder auslösen, z. B.
Pneumonie (Inzidenz des Rhinovirusnachweises bei community acquired pneumonia, CAP: 18 – 26%; bis zu 60% bakterielle oder virale Ko-Infektionen) oder
Exazerbationen von Asthma (ungefähr ⅔ aller virusassoziierten Exazerbationen von Asthma sind mit Rhinoviren (nur Spezies Rhinovirus C) assoziiert) oder
Exazerbationen chronisch obstruktiver Lungenerkrankungen (24% der Exazerbationen einer COPD gehen mit der Detektion von Rhinoviren einher) 30 .
Bezüglich Diagnostik, Therapie und Prophylaxe treffen die oben zu ihren nahen Verwandten, den Enteroviren, gemachten Aussagen zu ( Tab. 4 ).
Respiratorisches Synzytialvirus (RSV)
Biologie
Das respiratorische Synzytialvirus (RSV) wurde 1955 entdeckt. Es gehört zur Familie der Paramyxoviren und ist weltweit verbreitet. Man unterscheidet anhand der Antigenstruktur des G-Proteins 2 Serotypen, RSV A und RSV B. Beide Typen kozirkulieren, in den meisten Jahren dominiert RSV A. In den gemäßigten Breiten zeigt RSV eine eindeutige Saisonalität. Das Virus ist zwar ganzjährig nachweisbar, Infektionen dominieren aber im Winter.
RSV wird durch Sekrete des Respirationstrakts übertragen. Eine RSV-Infektion hat typischerweise eine Inkubationszeit von 3 – 5 Tagen, in denen sich das Virus exponentiell vermehrt und die Lunge erreicht. Gleichzeitig ist mit dem Auftreten der Symptome ein starker Rückgang der Viruslast zu beobachten. Re-Infektionen sind häufig; eine persistierende Immunität entsteht nicht.
RSV-Infektionen werden in allen Altersgruppen beobachtet. Schwerwiegende Infektionen dominieren aber bei Kindern. Das Virus ist weltweit für 60% aller Infektionen der unteren Atemwege bei Kindern vor Eintritt in die Schule verantwortlich. Der Schutz durch maternale Antikörper ist nicht vollständig, daher ist RSV für 80% aller Infektionen der unteren Atemwege im 1. Lebensjahr verantwortlich mit einem Inzidenzmaximum im Alter von 2 – 3 Monaten.
RSV-assoziierte Hospitalisierungen nehmen weltweit zu. In den Industrieländern hat die Mortalität durch symptomatische Therapie allerdings abgenommen und liegt zurzeit unter 1%. Risikofaktoren für den Schweregrad einer RSV-Infektion und die Frequenz von RSV-Infektionen sind 31 :
Verzicht auf Stillen
Immunsuppression
chronische Lungenerkrankungen
kongenitale Herzfehler
Frühgeburtlichkeit
Klinik
Eine RSV-Infektion beginnt in der Regel im oberen Respirationstrakt mit Schnupfen, Fieber und/oder Husten. Bei einem Teil der Patienten kommt es zur Beteiligung der unteren Atemwege, die sich als Bronchiolitis, Tracheobronchitis oder Pneumonie manifestieren kann. Typische Symptome der Bronchiolitis sind:
reduzierter Allgemeinzustand
Schwierigkeiten bei der Nahrungsaufnahme
beschleunigte Atmung
Husten
Dyspnoe
Merke.
Bei bis zu 20% der Kinder ist eine Apnoeepisode das erste Symptom einer RSV-Infektion 32 .
Die Infektion der unteren Atemwege durch RSV bei Kleinkindern ist ein signifikanter Risikofaktor für Giemen und Asthma in der ersten Lebensdekade, aber möglicherweise auch in der Adoleszenz und bei Erwachsenen. Die Lungenfunktion kann bei diesen Kindern langfristig gestört sein und zu eingeschränkter Lebensqualität und überdurchschnittlichem Arztkontakt führen 33 .
Diagnostik
Anzucht
Goldstandard für die Diagnostik von RSV war lange Zeit die Virusisolation aus Patientenmaterial durch Anzucht auf geeigneten Zellkulturen. Aufgrund der heute zur Verfügung stehenden schnelleren, sensitiveren und automatisierbaren Verfahren ist die Virusanzucht in der täglichen Routine aber in den Hintergrund getreten.
Antikörpernachweise
Serologische Antikörpernachweisverfahren sind ebenfalls seit langer Zeit verfügbar und werden nach wie vor häufig vom klinisch tätigen Arzt angefordert. Aufgrund von eingeschränkter Sensitivität und Spezifität der Antikörpernachweisverfahren ist der positive Vorhersagewert positiver Antikörpernachweise für eine frische RSV-Infektion gering. Somit ist der Antikörpernachweis im Wesentlichen für epidemiologische Fragestellungen oder für Infektionsnachweise in einer späten Erkrankungsphase von Bedeutung, wenn Direktnachweise nicht mehr zum Ziel führen.
Virusdirektnachweis
In der frühen Phase der RSV-Infektion ist der Virusdirektnachweis heute die Methode der Wahl. Es stehen Schnelltests (Immunchromatografie), Antigennachweise durch ELISA, Antigennachweise durch Immunfluoreszenztest (IFT) und Nukleinsäureamplifikationsverfahren zur Verfügung (PCR, isothermale Amplifikationsverfahren).
Sensitivität und Spezifität
Die Sensitivität der Schnelltests liegt bei 75,3% und die Spezifität bei 98,7% im Vergleich zur PCR-Diagnostik 34 . Die molekulare Diagnostik ist also deutlich überlegen. Mittlerweile sind auch molekulare Verfahren verfügbar, die bezüglich der Geschwindigkeit mit den Schnelltests konkurrieren können (isothermale Amplifikation).
Therapie
Therapie der Wahl ist heute die symptomatische Therapie mit angemessener Flüssigkeits- und Sauerstoffversorgung. Eine Therapie mit Ribavirin kann erwogen werden, ist aber aufgrund der Schwierigkeiten und Gefahren bei der Administration sowie der im Normalfall schon absinkenden Viruslast bei Symptombeginn nur für immunkompromittierte Patienten empfohlen.
In Entwicklung befinden sich die Wirkstoffe GS-5806 (Presatovir) und ALS-008176, die beide Phase-II-Studien erfolgreich abgeschlossen haben. Bei GS-5806 handelt es sich um einen kleinen, oral administrierbaren Fusionsinhibitor, der die Viruslast und die Mukusproduktion reduziert sowie die Gesamtsymptomatik verbessert. ALS-008176 ist ein Cytosin-Nukleosid-Analogon, das als Prodrug oral administrierbar ist. Der Wirkstoff reduziert die Viruslast 32 . Weitere Wirkstoffkandidaten befinden sich in weniger weit fortgeschrittenen Entwicklungsstadien.
Prophylaxe
Passive Immunisierung
Zurzeit steht nur ein einziger Wirkstoff zur Immunprophylaxe zur Verfügung. Es handelt sich dabei um den gegen das F-Protein von RSV gerichteten monoklonalen Antikörper Palivizumab. Der Wirkstoff wird während der RSV-Saison monatlich administriert. Er führt zu einer Reduktion der Hospitalisierungen um 50%. Aufgrund der hohen Kosten wird Palivizumab nur bei definierten Risikopatienten wie ehemaligen Frühgeborenen unter 32 SSW oder Säuglingen mit Herzfehlern eingesetzt. Die Verwendung aktueller epidemiologischer Daten zu RSV, wie sie das RespVir-Netzwerk zu Verfügung stellt, könnte die RSV-Saison präziser definieren und so zu einem ökonomischeren Einsatz von Palivizumab führen.
ALX-0171, ein Nanoantikörper (monomere variable Domäne eines klassischen Antikörpers), reduziert Viruslast und Virusreplikation. Der Wirkstoff hat gerade für Kinder unter 2 Jahren eine Phase-I/II a-Studie erfolgreich durchlaufen, eine Phase-II-Studie beginnt in diesem Jahr. Weiter fortgeschritten ist die klinische Testung von REGN2222, einem humanen monoklonalen IgG-Antikörper, der gegen das F-Protein gerichtet ist. Hier wird im Jahr 2017 eine Phase-II-Studie bei Frühgeborenen abgeschlossen 32 .
Aktive Immunisierung
Zurzeit ist kein aktiver Impfstoff gegen RSV verfügbar. Dies ist auf die Schwierigkeit zurückzuführen, gleichzeitig eine hinreichend lang protektive Immunantwort zu induzieren, aber VED (vaccine induced disease) zu vermeiden. VED wurde in den 1960er-Jahren beim ersten Versuch der Impfstoffentwicklung mit einem formalininaktivierten RSV-Stamm beobachtet.
In Entwicklung befinden sich mehrere lebend attenuierte Impfstoffkandidaten in Phase-I- und Phase-II-Studien, mehrere vektorbasierte Impfstoffkandidaten in Phase-I- und Phase-II-Studien und proteinbasierende Impfstoffkandidaten in Phase-I- bis Phase-III-Studien 32 .
Kernaussagen.
Zahlreiche respiratorische Viren wurden identifiziert, viele davon erst in den letzten 2 Jahrzehnten. Sie sind Grundlage für bis zu 90% der respiratorischen Infektionen weltweit.
Klinisch sind die Infektionen mit den unterschiedlichen respiratorischen Viren zumeist nicht sicher voneinander zu unterscheiden. Hier hilft nur eine spezifische Labordiagnostik aus geeignetem respiratorischen Material.
Höchste Sensitivität und Spezifität haben Nukleinsäureamplifikationsverfahren. Diese stehen sowohl für den Einzelnachweis eines Virus als auch für den syndromorientierten Nachweis aller relevanten Viren durch Multiplexverfahren zur Verfügung. Point-of-Care-Systeme auf Basis von Nukleinsäureamplifikation können die Nachweiszeit auf bis zu 15 min reduzieren.
Nur für Influenzaviren sind spezifische antivirale Wirkstoffe oder effektive Impfstoffe zugelassen. Mit Palivizumab steht ein humanisierter monoklonaler Antikörper zur passiven Immunisierung von Frühgeborenen und kleinen Kindern mit einer Grunderkrankung gegen respiratorische Synzytialviren (RSV) zur Verfügung.
Für einige länger bekannte oder aber auch neue Viren, wie z. B. RSV, humanes Metapneumovirus und bestimmte Coronaviren, ist die Entwicklung geeigneter Therapeutika und Impfstoffe weit fortgeschritten. Bei anderen respiratorischen Viren gibt es in dieser Hinsicht keinerlei positive Entwicklungen.
Erstveröffentlichung.
Dieser Beitrag ist eine aktualisierte Version des Artikels: Kleines M. Virale Atemwegserkrankungen – neue Viren. Krankenhaushygiene up2date 2016; 11: 247 – 262 .
Wissenschaftlich verantwortlich gemäß Zertifizierungsbestimmungen
Wissenschaftlich verantwortlich gemäß Zertifizierungsbestimmungen für diesen Beitrag ist Dr. med. Paul W. Meier, Solothurn.
Über den Autor. Michael Kleines

PD Dr. rer. nat., Jahrgang 1967. 1987 – 1993 Studium der Biologie in Aachen. 1993 – 1997 Promotion am Max-Planck-Institut für Züchtungsforschung in Köln. 1997 – 2010 Institut für Medizinische Mikrobiologie in Aachen. 2007 Habilitation, 2009 Weiterbildung zum Fachvirologen und Oberassistent der Virologie. 2010 – 2013 Leiter der Diagnostik am Institut für Virologie in Innsbruck. Seit 2014 Leiter der Virologie am Labordiagnostischen Zentrum, Aachen.
- Mit welchen Influenza-A-Virus-Subtypen wurden bereits humane Infektionen beobachtet?
- H1 und H4
- H2 und H7
- H8 und H9
- H5 und H8
- H3 und H11
H2 und H7
- Was ist kein Risikofaktor für den Schweregrad einer RSV-Infektion?
- chronische Lungenerkrankungen
- Immunsuppression
- Kaiserschnittgeburt
- Verzicht auf Stillen
- Frühgeburtlichkeit
Kaiserschnittgeburt
- Welchen Influenzaimpfstoff empfiehlt die WHO zur optimalen Protektion?
- trivalenter Impfstoff auf Zellkulturbasis (2-mal A, 1-mal B)
- trivalenter Lebendimpfstoff (2-mal A, 1-mal B)
- adjuvantierter trivalenter Totimpfstoff (2-mal A, 1-mal B)
- nicht adjuvantierter trivalenter Totimpfstoff (2-mal A, 1-mal B)
- quadrivalenter Impfstoff (2-mal A, 2-mal B)
quadrivalenter Impfstoff (2-mal A, 2-mal B)
- Welche Aussage zu viralen Atemwegserkrankungen bei Kindern ist falsch ?
- Kinder machen etwa 5 – 6 akute Atemwegserkrankungen im Jahr durch, die mit einer obstruktiven Klinik einhergehen,und tragen die Hauptlast der Respirationstraktinfektionen.
- Obwohl die meisten Atemwegsinfektionen in Mitteleuropa von Viren ausgelöst werden, werden viele Patienten mit Antibiotika behandelt.
- Virusinfektionen des Respirationstrakts können zu einer bakteriellen Superinfektion führen.
- Die Erregerdiagnostik bei Atemwegsinfektionen führt fast immer zu einem Erregernachweis.
- Nur für Influenzaviren sind spezifische antivirale Wirkstoffe oder effektive aktive Impfstoffe zugelassen.
Die Erregerdiagnostik bei Atemwegsinfektionen führt fast immer zu einem Erregernachweis.
- Welche Aussage zum humanen Metapneumovirus trifft zu?
- Mit Ribavirin steht ein gegen Metapneumoviren hoch wirksamer antiviraler Wirkstoff zur Verfügung.
- Das Infektionsmaximum des humanen Metapneumovirus liegt im Herbst.
- Infektionen mit dem humanen Metapneumovirus können klinisch gut von Infektionen mit respiratorischen Synzytialviren (RSV) abgegrenzt werden.
- Im Tierversuch wurden bereits für Metapneumoviren spezifische monoklonale Antikörper erfolgreich getestet.
- Methode der Wahl zum Nachweis von Metapneumovirus-Infektionen ist der Antigen-Immunfluoreszenztest.
Im Tierversuch wurden bereits für Metapneumoviren spezifische monoklonale Antikörper erfolgreich getestet.
- Welche Aussage zu Impfungen ist falsch ?
- Die Virusstämme werden für jede Influenzasaison neu von der WHO festgelegt.
- Von der Impfung von Schwangeren gegen Influenza wird dringend abgeraten.
- Am schlechtesten ist die Durchimpfung gegen Influenza in Deutschland bei Beschäftigten des Gesundheitssystems.
- In der Vergangenheit wurden auch Impfstoffe gegen die Vogelgrippe zugelassen.
- Klassische Impfstoffe basieren auf in Hühnereiern angezüchteten Viren.
Von der Impfung von Schwangeren gegen Influenza wird dringend abgeraten.
- Welche Aussage zum respiratorischen Synzytialvirus (RSV) ist falsch ?
- RSV gehört zur Familie der Picornaviren und ist weltweit verbreitet.
- Bei Kindern dominieren schwerwiegende RSV-Infektionen.
- Der Schutz durch maternale Antikörper ist nicht vollständig, daher ist RSV für 80% aller Infektionen der unteren Atemwege im 1. Lebensjahr verantwortlich.
- Bei bis zu 20% der Kinder ist eine Apnoeepisode das erste Symptom einer RSV-Infektion.
- Die Infektion der unteren Atemwege durch RSV bei Kleinkindern ist ein signifikanter Risikofaktor für Giemen und Asthma in der ersten Lebensdekade.
RSV gehört zur Familie der Picornaviren und ist weltweit verbreitet.
- Welche Aussage zu Coronaviren ist richtig?
- Die Letalität symptomatischer MERS-Coronavirusinfektionen beträgt mehr als 30%.
- Alle bekannten respiratorischen Coronaviren wurden in den letzten 20 Jahren entdeckt.
- Das Coronavirus HKU1 wurde durch strikte Surveillance- und Isolationsmaßnahmen aus der humanen Population eliminiert.
- Die Letalität von Infektionen mit dem SARS-Coronavirus beträgt mehr als 40%.
- Das Coronavirus NL63 wird nie gemeinsam mit anderen respiratorischen Viren nachgewiesen.
Die Letalität symptomatischer MERS-Coronavirusinfektionen beträgt mehr als 30%.
- Welche Aussage zu Bocaviren ist richtig?
- Auslöser der meisten respiratorischen Bocavirusinfektionen ist die Spezies HBoV3.
- Bei der Diagnostik von Bocavirusinfektionen steht die Serologie im Vordergrund.
- Ein Impfstoff gegen Bocaviren befindet sich in einem fortgeschrittenen Entwicklungsstadium.
- Bocaviren verursachen ausschließlich banale Infektionen des oberen Respirationstrakts.
- Nur eine Viruslast > 10 000 Kopien/ml zeigt eine frische Bocavirusinfektion an.
Nur eine Viruslast > 10 000 Kopien/ml zeigt eine frische Bocavirusinfektion an.
- Welche Aussage zu Entero- und Rhinoviren ist falsch ?
- Es wurden mehr als 100 Rhinovirustypen identifiziert.
- Entero- und Rhinoviren gehören beide zur Familie der Picornaviren.
- Enterovirus D68 ruft leichte bis schwere Respirationstrakterkrankungen hervor.
- Entero- und Rhinoviren lassen sich ausschließlich durch serologische Verfahren nachweisen.
- Es gibt keine antiviralen Impfstoffe gegen Rhino- und Enteroviren.
Entero- und Rhinoviren lassen sich ausschließlich durch serologische Verfahren nachweisen.
Footnotes
Interessenkonflikt Der Autor gibt an, dass kein Interessenkonflikt vorliegt.
Literatur
- 1.Patarcic I, Gelemanovic A, Kirin M. The role of host genetic factors in respiratory tract infectious diseases: systematic review, meta-analyses and field synopsis. Sci Rep. 2015 doi: 10.1038/srep16119. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Lee K H, Gordon A, Foxman B. The role of respiratory viruses in the etiology of bacterial pneumonia. Evol Med Public Health. 2016:95–109. doi: 10.1093/emph/eow007. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.McKay K A, Jahanfar S, Duggan T. Factors associated with onset, relapses or progression in multiple sclerosis: A systematic review. Neurotoxicology. 2016 doi: 10.1016/j.neuro.2016.03.020. [DOI] [PubMed] [Google Scholar]
- 4.Mahony J B, Petrich A, Smieja M. Molecular diagnosis of respiratory virus infections. Crit Rev Clin Lab Sci. 2011;48:217–249. doi: 10.3109/10408363.2011.640976. [DOI] [PubMed] [Google Scholar]
- 5.Sommerville L K, Ratnamohan V M, Dwyer D E. Molecular diagnosis of respiratory viruses. Pathology. 2015;47:243–249. doi: 10.1097/PAT.0000000000000240. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Higgins D, Trujillo C, Keech C. Advances in RSV vaccine research and development – A global agenda. Vaccine. 2016 doi: 10.1016/j.vaccine.2016.03.109. [DOI] [PubMed] [Google Scholar]
- 7.Mejias A, Ramilo O. New options in the treatment of respiratory syncytial virus disease. J Infect. 2015;71:80–87. doi: 10.1016/j.jinf.2015.04.025. [DOI] [PubMed] [Google Scholar]
- 8.Acha P N, Szyfres B. 3. Aufl. Washington DC: Pan American Health Organization; 2003. Zoonoses and communicable Diseases common to Man and Animals. [Google Scholar]
- 9.Tong S, Zhu X, Li Y. New World Bats Harbor Diverse Influenza A Viruses. PLoS Pathogens. 2013;9:e1003657. doi: 10.1371/journal.ppat.1003657. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Ott S, Lepper P, Hauptmeier B. Die Rolle von Viren bei tiefen Atemwegsinfektionen des Erwachsenen – Teil 1: Erreger, Pathogenese und Diagnostik. Pneumologie. 2009;63:709–717. doi: 10.1055/s-0029-1215232. [DOI] [PubMed] [Google Scholar]
- 11.Esposito S, Montinaro V, Groppali E. Live attenuated intranasal influenza vaccine. Hum Vaccin Immunother. 2012;8:115–119. doi: 10.4161/hv.8.1.18809. [DOI] [PubMed] [Google Scholar]
- 12.Smucny J, Fahey T, Becker L. Antibiotics for acute bronchitis. Cochrane Database Syst Rev. 2004;(18):CD000245. doi: 10.1002/14651858.CD000245.pub2. [DOI] [PubMed] [Google Scholar]
- 13.Hurt A C, Hui D S, Hay A. Overview of the 3rd isirv-Antiviral Group Conference – advances in clinical management. Influenza Other Respir Viruses. 2015;9:20–31. doi: 10.1111/irv.12293. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Nakai A, Saito S, Unno N. Review of the pandemic (H1N1) 2009 among pregnant Japanese women. J Obstet Gynaecol Res. 2012;38:757–762. doi: 10.1111/j.1447-0756.2011.01812.x. [DOI] [PubMed] [Google Scholar]
- 15.Hui D S, Zumla A. Emerging respiratory tract viral infections. Curr Opin Pulm Med. 2015;21:284–292. doi: 10.1097/MCP.0000000000000153. [DOI] [PubMed] [Google Scholar]
- 16.Smee D F, Tarbet E B, Furuta Y. Synergistic combinations of favipiravir and oseltamivir against wild-type pandemic and oseltamivir-resistant influenza A virus infections in mice. Future Virol. 2013;8:1085–1094. doi: 10.2217/fvl.13.98. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Blank P R, Schwenkglenks M, Szucs T D. Influenza vaccination coverage rates in five European countries during season 2006/07 and trends over six consecutive seasons. BMC Publ Health. 2008;8:272. doi: 10.1186/1471-2458-8-272. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Wen S C, William J V. New approaches for immunization and therapy against human metapneumovirus. Clin Vaccine Immunol. 2015;22:858–866. doi: 10.1128/CVI.00230-15. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Schuster J E, Williams J V. Human Metapneumovirus. Microbiol Spectr. 2014 doi: 10.1128/microbiolspec.AID-0020-2014. [DOI] [PubMed] [Google Scholar]
- 20.Williams J V, Wang C K, Yang C F. The role of human metapneumovirus in upper respiratory tract infections in children: a 20-year experience. J Infect Dis. 2006;193:387–395. doi: 10.1086/499274. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.Kitanovski L, Kopriva S, Pokorn M. Treatment of severe human metapneumovirus (hMPV) pneumonia in an immunocompromised child with oral ribavirin and IVIG. J Pediatr Hematol Oncol. 2013;35:e311–e313. doi: 10.1097/MPH.0b013e3182915d2d. [DOI] [PubMed] [Google Scholar]
- 22.Báez-Santos Y M, Mielech A M, Deng X. Catalytic function and substrate specificity of the papain-like protease domain of nsp3 from the Middle East respiratory syndrome coronavirus. J Virol. 2014;88:12511–12527. doi: 10.1128/JVI.01294-14. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.Milne-Price S, Miazgowicz K L, Munster V J. The emergence of the Middle East respiratory syndrome coronavirus. Pathog Dis. 2014;71:121–136. doi: 10.1111/2049-632X.12166. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.Prasannavenkatesh D, Batool M, Shah M. Middle East respiratory syndrome coronavirus: transmission, virology and therapeutic targeting to aid in outbreak control. Exp Mol Med. 2015;47:e181. doi: 10.1038/emm.2015.76. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.Fehr A R, Perlman S. Coronaviruses: an overview of their replication and pathogenesis. Methods Mol Biol. 2015;1282:1–23. doi: 10.1007/978-1-4939-2438-7_1. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.Zhang N, Jiang S, Du L. Current advancements and potential strategies in the development of MERS-CoV vaccines. Expert Rev Vaccines. 2014;13:761–774. doi: 10.1586/14760584.2014.912134. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27.Broccolo F, Falcone V, Esposito S. Human bocaviruses: possible etiologic role in respiratory infection. J Clin Virol. 2015;72:75–81. doi: 10.1016/j.jcv.2015.09.008. [DOI] [PubMed] [Google Scholar]
- 28.Daleno C, Greenberg D, Piralla A. A novel human enterovirus C (EV-C118) identified in two children hospitalised because of acute otitis media and community-acquired pneumonia in Israel. J Clin Virol. 2013;56:159–162. doi: 10.1016/j.jcv.2012.10.013. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.Greninger A L, Naccache S N, Messacar K. A novel outbreak enterovirus D68 strain associated with acute flaccid myelitis cases in the USA (2012-14): a retrospective cohort study. Lancet Infect Dis. 2015;15:671–682. doi: 10.1016/S1473-3099(15)70093-9. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 30.Jacobs S E, Lamson D M. George K et al. Human rhinoviruses. Clin Microbiol Rev. 2013;26:135–162. doi: 10.1128/CMR.00077-12. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 31.Royston L, Tapparel C. Rhinoviruses and respiratory enteroviruses: not as simple as ABC. Viruses. 2016;8:E16. doi: 10.3390/v8010016. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 32.Piedimonte G. RSV infection: state of the art. Cleveland Clin J Med. 2015;82:13–18. doi: 10.3949/ccjm.82.s1.03. [DOI] [PubMed] [Google Scholar]
- 33.Rezaee F, Linfield D, Harford T. Ongoing developments in RSV prophylaxis: a clinicianʼs analysis. Curr Op Virol. 2017;24:70–78. doi: 10.1016/j.coviro.2017.03.015. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 34.Fauroux B, Simoes E, Checchia P. The Burden and long-term respiratory morbidity associated with respiratory syncytial virus infection in early childhood. Inf Dis Ther. 2017;6:173–197. doi: 10.1007/s40121-017-0151-4. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 35.Bruning A, Leeflang M, Vos J. Rapid tests for influenza, respiratory syncytial virus, and other respiratory viruses: a systematic review and meta-analysis. Clin Infect Dis. 2017 doi: 10.1093/cid/cix461. [DOI] [PMC free article] [PubMed] [Google Scholar]


