Abstract
La visualización de datos es una herramienta relevante para explorar y comunicar resultados en la investigación médica, en especial cuando se trata de vigilancia epidemiológica. La aplicación web COVID19-Tracker analiza y produce de forma sistemática visualizaciones diarias de los datos de la epidemia de COVID-19 de casos diagnosticados y fallecimientos desde el 24 de febrero de 2020 en adelante. Se han desarrollado tres aplicaciones para: 1) análisis de la tendencia y proyecciones a corto plazo; 2) estimación de la tasa de letalidad; y 3) efecto del estado de alarma sobre la tendencia de datos incidentes. La aplicación online puede ser de utilidad para un mejor conocimiento de la epidemia de SARS-CoV-2 en España.
Palabras clave: SARS-CoV-2, Visualización de datos, Interficie web, Regresión de Poisson
Abstract
Data visualization is an important tool for exploring and communicating findings in medical research, and specially in epidemiological surveillance. The COVID19-Tracker web application systematically produces daily updated data visualization and analysis of SARS-CoV-2 epidemic in Spain. It collects automatically daily data on COVID-19 diagnosed cases and mortality, from February 24th, 2020 onwards. Three applications have already been developed: 1) to analyze data trends and estimating short-term projections; 2) to estimate the case fatality rate; and 3) to assess the effect of the lockdowns on the data trends. The application may help for a better understanding of the SARS-CoV-2 epidemic data in Spain.
Keywords: SARS-CoV-2, Data visualization, Web browser, Poisson regression
Introduccción
Los primeros casos confirmados de enfermedad producida por SARS-CoV-2 en España se identificaron a finales de febrero de 20201. Desde entonces, España se ha convertido en el segundo país del mundo con mayor afectación (148.220 casos diagnosticados) y el tercero en mortalidad (14.792 fallecidos)2. El 16 de marzo se decretó el estado de alarma con medidas orientadas a frenar la curva epidémica, restringiendo el contacto social mediante el confinamiento de la población general, reduciendo los transportes públicos y cerrando comercios y empresas, a excepción de aquellos esenciales para abastecer a la población (Real Decreto 463/2020). Sin embargo, esta medida no fue suficiente para cambiar la tendencia creciente de la epidemia. Por esta razón, se sugirió un confinamiento más restrictivo3, el cual fue decretado el 30 de marzo (Real Decreto-Ley 10/2020).
La visualización de datos resulta relevante para explorar y comunicar resultados en la investigación médica, en especial para la vigilancia epidemiológica. En el contexto actual es importante ofrecer a la comunidad científica, así como a los agentes políticos y de salud pública, herramientas automatizadas para la monitorización de la epidemia, pues el análisis crudo de los datos tabulados puede esconder aspectos esenciales de la propia tendencia. La aplicación web COVID19-Tracker analiza y produce de manera sistemática visualizaciones diarias de los datos de la epidemia de COVID-19 en España desde el 24 de febrero de 2020.
Método
La aplicación se ha desarrollado mediante RStudio (versión 1.2.5033), utilizando la librería Shiny (versión 1.4.0), ya que ofrece la posibilidad de desarrollar interficies gráficas para el usuario que pueden ser cargadas localmente o en línea de forma interactiva. Esto resulta en particular beneficioso para mostrar resultados actualizados a una audiencia amplia.
La aplicación analiza y visualiza datos diarios de COVID-19, correspondientes a casos diagnosticados y fallecidos desde el 24 de febrero de 2020. Estos datos se recogen diariamente de forma automática del repositorio de Datadista en GitHub4. En este repositorio se mantienen actualizados en formato adecuado los datos publicados por el Ministerio de Sanidad, Consumo y Bienestar Social, a través del Instituto de Salud Carlos III2.
Los análisis implementados consideran el modelo de regresión de Poisson5, corregido por sobredispersión6, para estimar la tendencia y obtener proyecciones a 3 días. Todos los análisis estadísticos se han implementado mediante el programa R, versión 3.6.3.
Análisis de tendencia y proyecciones
Para evaluar la tendencia se utiliza el modelo siguiente:
donde t = 1, 2, …, t representa la unidad temporal (desde el primer día hasta el último observado, T días consecutivos en total) y ct es el número de eventos diarios.
Las proyecciones a 3 días, y su intervalo de confianza del 95%, se obtienen a partir de los parámetros estimados por el modelo. También se calculan las tendencias y las proyecciones por grupos de edad (0-39, 40-49, 50-59, 60-69, 70-79 y 80 o más años).
En versiones previas de la aplicación también se consideró un modelo alternativo, incluyendo solo la tendencia lineal, y los modelos se comparaban mediante una prueba de razón de similitudes. Con la evolución de la epidemia observamos que el mejor ajuste lo proporcionaba el modelo cuadrático, descrito anteriormente, por lo que es el modelo que se utiliza en la versión actual. En cualquier caso, la bondad de ajuste de los modelos se evalúa regularmente en caso de ser necesaria una reformulación que pudiese proporcionar un mejor ajuste de los datos durante el transcurso de la epidemia.
Análisis de la letalidad
La tasa de letalidad se define como el cociente entre los fallecidos y los casos diagnosticados7. Para evaluar la tendencia se introduce en el modelo de regresión un término offset con los casos diagnosticados:
donde mt representa el número diario de pacientes fallecidos y ct los casos diagnosticados. También se calculan las tasas de letalidad para los mismos grupos de edad.
En la actualidad no es posible realizar un estimación precisa de la tasa de letalidad debido al grado de subregistro de los casos diagnosticados en las estadísticas oficiales8, si bien es de interés su estimación y seguimiento considerando dicha limitación.
Análisis de intervención
Para evaluar el efecto del estado de alarma sobre la tendencia de datos incidentes utilizamos un diseño de series temporales interrumpidas9, usando un modelo de interacción para estimar los cambios de tendencia:
donde alarma identifica los periodos antes y durante los estados de alarma (0 = antes de 16 marzo, 1 = 16-29 marzo y 2 = después de 30 marzo).
Para facilitar la interpretación en función del incremento diario porcentual de casos incidentes, y su intervalo de confianza del 95%, se asume una tendencia lineal en cada periodo10.
Resultados
La aplicación presenta una estructura amigable e intuitiva que, mediante menús, produce visualizaciones de los datos (fig. 1 ). La aplicación contempla un proceso automatizado por el cual los datos se actualizan cada vez que un usuario se conecta a través de la dirección web: https://ubidi.shinyapps.io/covid19/
Figura 1.
Página web de inicio de la aplicación COVID19-Tracker para la visualización y el análisis automatizado de datos de la epidemia de SARS-CoV-2 en España. Disponible en: https://ubidi.shinyapps.io/covid19/.
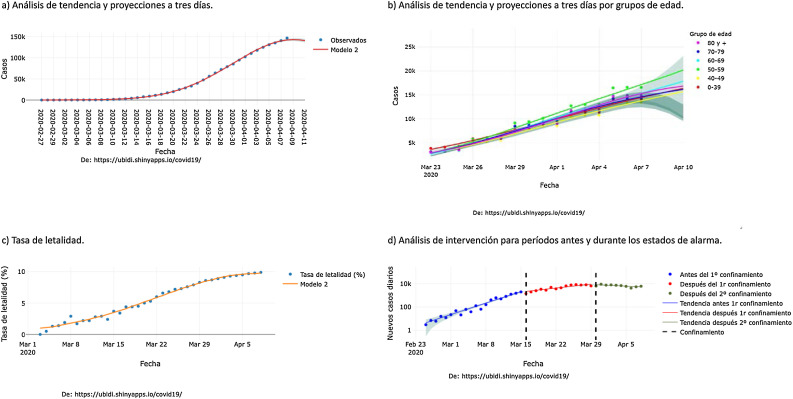
Los menús Proyecciones y Proyecciones por edad producen la visualización de la tendencia de los datos y su proyección a 3 días (fig. 2 a y b, respectivamente) en el ámbito estatal y por comunidades autónomas. Los menús Letalidad y Letalidad por edad permiten visualizar la tendencia en la tasa de letalidad (fig. 2 c) estatal y por comunidades autónomas, aunque por grupos de edad solo en el ámbito estatal. Finalmente, el menú Intervención evalúa el efecto de los estados de alarma en datos incidentes nacionales (fig. 2 d).
Figura 2.
Visualización estándar de la aplicación COVID19-Trarcker (resultados actualizados a 8 de abril de 2020) para el análisis de tendencias y su proyección a 3 días en el ámbito nacional (a) y por grupos de edad (b) de la tasa de letalidad (c), y análisis de intervención para evaluar el efecto de los estados de alarma sobre los datos incidentes (d).
Los gráficos producidos por la aplicación permiten, cuando el usuario señala un punto concreto con el ratón, mostrar el valor observado y el estimado por el modelo. Asimismo, al seleccionar el gráfico permite la opción de descargarlo en formato imagen (portable network graphic, *.png). Todos los menús están disponibles en castellano, catalán e inglés. El menú Otras aplicaciones recoge una colección de aplicaciones Shiny desarrodadas por otros usuarios, también de utilidad para seguir la evolución de la epidemia de COVID-19 en España y globalmente.
Discusión
La aplicación COVID19-Tracker ofrece un conjunto de herramientas para el análisis actualizado y la visualización gráfica que pueden ser de gran utilidad para un mejor conocimiento de la evolución de la epidemia de COVID-19 en España y su vigilancia epidemiológica.
Como limitaciones, hay que indicar que la aplicación no tiene en cuenta los cambios en la definición de caso diagnosticado por COVID-19 ni en los fallecimientos, por lo que se modeliza directamente el número de eventos en lugar de la tasa de incidencia, asumiendo que toda la población está en riesgo, a excepción de la tasa de letalidad. Por otro lado, los análisis no están libres de los sesgos vinculados a la fuente de los datos, proporcionados diariamente por el Ministerio de Sanidad, Consumo y Bienestar Social2 y obtenidos a través del GitHub de Datadista4, donde se pueden consultar detalles sobre los criterios de definición en los datos según las notas publicadas por el Ministerio.
Seguimos planeando mejoras de la aplicación para que incluya nuevos análisis y visualizaciones. Además, la aplicación podría ser extensible para su uso en otros países o áreas geográficas. En resumen, esta aplicación, de uso sencillo, viene a ocupar un espacio en este escenario particular para la visualización y la exploración epidemiológica de los datos de la epidemia de COVID-19 en España.
Editor responsable del artículo
Carlos Álvarez Dardet.
Contribuciones de autoría
Todos los autores han contribuido en la concepción de la herramienta, la obtención de los datos, la propuesta de modelización estadística y su implementación informática. La versión final del manuscrito ha sido aprobada por todos los autores, con una contribución igual en su elaboración.
Agradecimientos
Se agradece a la plataforma Datadista su dedicación por poner a disposición de forma abierta y estructurada los datos diarios acumulados de la epidemia por SARS-CoV-2 en España.
Financiación
Ninguna.
Conflictos de intereses
Ninguno.
Bibliografía
- 1.Saglietto A., D’Ascenzo F., Zoccai G.B. COVID-19 in Europe: the Italian lesson. Lancet. 2020;395:1110–1111. doi: 10.1016/S0140-6736(20)30690-5. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Ministerio de Sanidad, Consumo y Bienestar Social. Instituto de Salud Carlos III. Situación de COVID-19 en España. (Consultado el 8/4/2020.) Disponible en: https://covid19.isciii.es/.
- 3.Mitjà O., Arenas A., Rodó X. Experts’ request to the Spanish Government: move Spain towards complete lockdown. Lancet. 2020;395:1193–1194. doi: 10.1016/S0140-6736(20)30753-4. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Datadista. Extracción, limpieza y normalización de las tablas de la situación diaria acumulada de la enfermedad por el coronavirus SARS-CoV-2 (COVID-19) en España en un formato accesible y reutilizable. (Consultado el 8/4/2020.) Disponible en: https://github.com/datadista/datasets/tree/master/COVID%2019.
- 5.Dyba T., Hakulinen T. Comparison of different approaches to incidence prediction based on simple interpolation techniques. Statistics in Medicine. 2000;19:1741–1752. doi: 10.1002/1097-0258(20000715)19:13<1741::aid-sim496>3.0.co;2-o. [DOI] [PubMed] [Google Scholar]
- 6.Navarro A., Utzet F., Puig P. La distribución binomial negativa frente a la de Poisson en el análisis de fenómenos recurrentes. Gac Sanit. 2001;15:447–452. doi: 10.1016/s0213-9111(01)71599-3. [DOI] [PubMed] [Google Scholar]
- 7.Rothman K., Greenland S. Lippincott-Raven; Philadelphia, PA: 1998. Modern epidemiology. [Google Scholar]
- 8.Battegay M., Kuehl R., Tschudin-Sutter S. 2019-novel Coronavirus (2019-nCoV): estimating the case fatality rate — a word of caution. Swiss Med Wkly. 2020;150:w20203. doi: 10.4414/smw.2020.20203. [DOI] [PubMed] [Google Scholar]
- 9.Bernal J.L., Cummins S., Gasparrini A. Interrupted time series regression for the evaluation of public health interventions: a tutorial. Int J Epidemiol. 2017;46:348–355. doi: 10.1093/ije/dyw098. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Tobías A. Evaluation of the lockdowns for the SARS-CoV-2 epidemic in Italy and Spain after one month follow up. Sci Total Environ. 2020;725:138539. doi: 10.1016/j.scitotenv.2020.138539. [DOI] [PMC free article] [PubMed] [Google Scholar]